Mapeo Génico
Anuncio

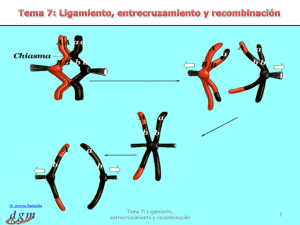
LIGAMIENTO Y MAPEO GENICO Ms. MARIA CRUZ BRICEÑO AREA DE GENETICA Y BIOLOGÍA CELULAR DEPARTAMENTO DE MORFOLOGIA HUMANA LIGAMIENTO GENICO ¿En el ligamiento, se cumple el principio de segregación independiente? GENES SINTENICOS GENES LIGADOS CLASES DE LIGAMIENTO • POR LA LOCALIZACION DE LOS ALELOS EN LOS CROMOSOMAS – ACOPLAMIENTO ó CIS – REPULSION ó TRANS • POR LA DISTANCIA ENTRE LOS LOCI – COMPLETO – INCOMPLETO • POR EL TIPO DE CROMOSOMA – AUTOSOMICO – SEXUAL GRUPOS DE LIGAMIENTO • HAPLOTIPO – Consiste en un conjunto de alelos ligados y próximos que tienden a heredarse juntos en las meiosis, sin separarse por recombinación GRUPOS DE LIGAMIENTO • GAMETOS PARENTALES • GAMETOS REGOMBINANTES FRECUENCIA DE RECOMBINACIÓN Identificar: los individuos NR y R la fase de ligamiento Factores del Entrecruzamiento • Puntos calientes de recombinación • Distancia entre los loci < distancia < entrecruzamiento > ligamiento > distancia > entrecruzamiento < ligamiento Frec. de recombinación ≈Distancia entre loci = cM % Recombinación = 1 cM ≈ 1mll pb Distancia < 50 cM ------- existe ligamiento entre los loci > 50 cM ------ no existe ligamiento Tipo de entrecruzamientos MAPEO GENICO Es la representación esquemática de la ubicación de genes ligados en un cromosoma específico, teniendo en cuenta: cM cM cM A--------------B-------------------------C---------D a--------------b--------------------------c----------d •Secuencia: relación entre los genes •Distancia: se expresa en función de la unidad de Mapa (cM), que equivale a la probabilidad de recombinación entre ambos loci UNIDADES DE MAPA • La unidad de escala de los mapas genéticos es el Centimorgan (cM). • Un cM se define como la fracción de recombinación de 0,01, es decir la aparición de un gameto recombinante entre 100, mientras que los 99 restantes tendrán la configuración parental. • Físicamente 1cM corresponde a una secuencias de DNA de entre 0,7 y 1 Mb. MAPEO GENICO: TIPOS • MAPEO GENÉTICO – USA LA FRECUENCIA DE RECOMBINANTES PARA DETERMINAR LA DISTANCIA ENTRE LOS GENES • MAPEO FÍSICO – USA TÉCNICAS CITOGENETICAS Y MOLECULARES PARA DETERMINAR LA LOCALIZACIÓN DE LOS GENES MAPEO GENETICO • Se basa en la 2ª Ley de Mendel MAPEO GENETICO METODOS DE ESTUDIO • Análisis de descendencia F2 ó Cruce de Prueba – Análisis de Cruzamiento bifactorial – Cruzamiento trifactorial comparar las proporciones fenotípicas esperadas vs observadas • Estudio de familias/ genealogías • Uso de Marcadores de DNA Mapeo Génico:Cruzamiento Bifactorial Color de ojo bw marrones bw+ rojos Venas de las alas hv+ finas hv gruesas • Descendencia de cruce de prueba Ojos rojos, venas finas Ojos marrones, venas gruesas Ojos rojos, venas gruesa 26 Ojos marrones, venas fina 24 124 126 1º Observar la proporción 2º Determinar la frec. de parentales y recombinantes 3º Determinar la distancia: % Recombinación %R= 50/300 x 100 = 16,67 Distancia = 16.67 cM Mapeo Génico: Cruzamiento Trifactorial Color de cuerpo: yellow ( amarillos) Color de ojos: white ( blancos) Forma de ojos:echinus (grandes y rugosos) 1. Determinar la secuencia - Identificar GP y GR - Esquematizar - Realizar cruzamientos Verificar recombiantes simples y dobles 2. Determinar la distancia entre los loci DZ1 = %RSz1 + %RD DZ2 = %RSz2 + %RD Mapeo Génico: Estudio de Familias • Ligamiento al X : método del abuelo d Daltonismo G Déficit de G-6- P DH d D G g Gen d: Daltonismo Gen G: Enz. G-6-P DH normal % R = R/T x 100 Mapeo Génico: Estudio de Familias Ligamiento autosómico GR GT %R %R 2 6 x100 x100 33.33 Fase de ligamiento • La fase determina la manera en que los alelos particulares próximos en la localización de sus loci – en el mismo cromosoma CIS ó – en distinto cromosoma TRANS • Establecer la fase permite distinguir tras la meiosis, si los cromosomas son recombinantes o parentales Mapeo Génico: Estudio de Familias 1. Determinación de la fase de ligamiento N= alelo Neurofibromatosis n= alelo normal RFLP 1= alelo 1 del marcador 1F10 2= alelo 2 del marcador 1F10 %R N 1 -------------------1 x 100 12,25 -------------------8 n 2 2. Determinar los recombinantes y calculo de la fracción de recombinación N 1 --------------------------------------n 2 N 2 --------------------------------n 2 3. Prueba de significancia Puntuación LOD: Z Z = Log 10 Z>3 Ligamiento establecido Indica que la probabilidad a favor del ligamiento es de 1000 veces superior a la probabilidad de distribución independiente Z= 2 Ligamiento muy probable Indica que la probabilidad a favor del ligamiento es de 100 veces superior a la probabilidad de distribución independiente Z=1 Ligamiento posible Z=0 Probabilidad de ligamiento y distribución al azar son aprox. iguales Z= 0 a -1 Posible asociación al azar Z= -2 Asociación al azar establecida Ejemplo N 2 --------------------------------------n 1 n 1 --------------------------------n 1 N 2 --------------------------------------n 1 Fase de ligamiento II 2 Hipótesis • Existe ligamiento (θ=0,0) N 2 -------------------- 1/2 1/2 n 1 -------------------- • Distribución independiente (θ=0,5) 1/4 N 2 -------------------N 1/4 1 -------------------- n 1 1/4 -------------------- 1/4 n 2 -------------------- z (1 / 2) 4 (1 / 4) 4 1 / 16 1 / 256 Log16 1,2 Cálculo de LOD con fase conocida MLS= puntuación máxima de probabilidad LOD score para la mas probable de una serie de alternativas Cálculo de LOD con fase desconocida 1 Calificación de LOD LOD + : pruebas a favor de ligamiento LOD -: prueba en contra el ligamiento Mapeo de múltiples puntos Permite establecer el orden cromosómico de locus en estudio Supera problemas de meiosis no informativas Se elaboran mapas de marco estructural marcador Enf. S. de Waadenburg Problemas en La calificación LOD LOD es un método potente para localizar un gen Dificultades por: •Errores en la genotipificación •Limites computacionales sobre genealogía en análisis •Heterogenidad de locus •Necesidad del modelo genético preciso MAPEO FISICO METODOS: 1. CITOGENETICA A. HETEROMORFISMO: “región no enrollada” G.S. Duffy “ Duffy Fya “ B. DELECIONES La deleción de una región permite definir la región en diferentes pacientes, estructurando así la localización del gen patológico GKD DMD EGC GKD= Deficiencia de glucocinasa DMD= distrofia muscular de Duchmen EGC= Enfermedad granulomatosa crónica OTC= deficiencia de Ornitín transcarbamilasa OTC • Mapeo por Dosificación • : permiten asignar o excluir Análisis – Duplicaciones • Paciente con tres cromosomas 21--- SOD – Deleciones • Pacientes con del en Cromosoma 9 -------- Adenilato ciclasa • Pacientes con del en cromosoma 2 -------- Fosfatasa ácida • TRANSLOCACIONES – Pacientes: t 17;22 – Pacientes: t 1; 17 • Problema : Neurofibromatosis La localización de los puntos de ruptura de esta translocación dio inicio a la clonación de NF1 • Translocación del X – Xp21 Distrofia muscular 2. Técnicas Moleculares • HIBRIDACION IN SITU FISH: Puede mapear genes de 1-2mll pb • HIBRIDACIÓN EN CELULAS SOMATICAS PRUEBA DE SINTENIA Correlación entre la presencia o ausencia de cada cromosoma humano con la presencia o ausencia de cada producto génico Cromosoma Producto A 3 - 5 + 8 - • CLONACION POSICIONAL Enfermedad Secuencia de AA Proteína Secuencia de Nucleótidos Sonda Localización del gen patológico IMPORTANCIA • MAPA GENICO • ANALISIS DE HETEROGENIDAD Y SEGREGACIÓN DE LAS ENFERMEDADES • INFORMACIÓN PARA EL DESARROLLO DE OPTIMAS ESTRATEGIAS EN LA TERAPIA GÉNICA. • HERRAMIENTA PARA EL DIAGNÓSTICO – PRECONCEPCIÓN – PRENATAL – PRESINTOMÁTICO, ETC