Ingeniero Biotecnólogo
Manual de Prácticas
Laboratorio de Biología Molecular
Dr. Alejandro Miguel Figueroa López
M.C. Abel Alberto Verdugo Fuentes
Dr. Ernesto Uriel Cantú Soto
M.I. Andrés Francisco Chávez Almanza
Dra. Libia Zulema Rodríguez Anaya
Dr. José Reyes González Galaviz
Manual de Prácticas de Laboratorio de Biología Molecular
Primera edición, 2018
D.R. © 2018, Instituto Tecnológico de Sonora
Dirección Académica de la División de Recursos Naturales
Departamento de Biotecnología y Ciencias Alimentarias
Av. Antonio Caso s/n Col. Villa ITSON, C.P. 85130
Cd. Obregón, Sonora, México
ISBN:
Impreso y hecho en México
Printed and made in Mexico
Instituto Tecnológico de Sonora
IB plan 2016
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
Prólogo
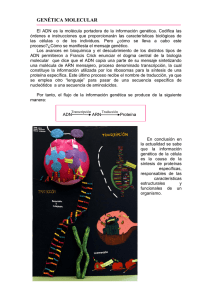
La Biología Molecular es la ciencia que estudia la interacción, estructura, función y
composición de las moléculas de la vida. La finalidad del presente manual es mostrar a
los estudiantes de la carrera de ingeniero biotecnólogo las técnicas de Biología
Molecular más empleadas en un laboratorio, así como lograr la comprensión de las
mismas y la visualización de sus posibles aplicaciones. Dentro de estas prácticas se
adiestrará los estudiantes para realizar una extracción de ácidos nucleicos y la
amplificación de fragmentos de ADN, mediante la técnica de reacción en cadena de la
polimerasa (PCR), además de otras técnias de relevancia que sirven para la
manipulación genética e identificación de microorganismos de interés biotecnológico.
Este manual se elaboró con el propósito de transmitir al alumno el conocimiento acerca
de la manipulación de las moléculas de la vida que codifican la información genética y el
uso de algunas técnicas básicas necesarias requeridas en los distintos campos del
conocimiento en el área de las ciencias de la vida.
Instituto Tecnológico de Sonora
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
Contenido
NORMAS DE SEGURIDAD PARA EL LABORATORIO DE BIOLOGÍA MOLECULAR...................................... 1
ESTRATEGIA DE CONTENCIÓN DE SINIESTROS Y ACCIDENTES EN EL LABORATORIO ............................. 3
PRÁCTICA 1. Uso y manejo correcto de las micropipetas ...................................................................... 5
PRÁCTICA 2. Aislamiento y purificación de ADN genómico de una bacteria Gram positiva. ............... 12
PRÁCTICA 3. Extracción de ARN de bacteria Gram negativa. ............................................................... 19
PRÁCTICA 4. Electroforesis en gel de agarosa para visualización de ácidos nucleicos ......................... 25
PRÁCTICA 5. Cuantificación y estimación de pureza de ADN mediante espectrofotometría .............. 32
PRÁCTICA 6. Reacción en cadena de la polimerasa PCR (replicación artificial del ADN) ..................... 38
PRÁCTICA 7. Enzimas de restricción y su aplicación en la tecnología del ADN recombinante. ............ 45
PRÁCTICA 8. Tranformación bacteriana. .............................................................................................. 51
PRÁCTICA 9. Análisis de secuencias de ADN (secuenciación Sanger)................................................... 58
ANEXO 1. SOLUCIONES Y REACTIVOS. .................................................................................................. 65
Composición de buffer y soluciones para transformación de células competentes JM109 pGEM-TEasy PROMEGA................................................................................................................................. 66
ANEXO 2. FORMATO DE ENTREGA DE REPORTE DE PRÁCTICA PARA EL ALUMNO .............................. 68
REFERENCIAS ........................................................................................................................................ 69
Instituto Tecnológico de Sonora
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
NORMAS DE SEGURIDAD PARA EL LABORATORIO DE BIOLOGÍA
MOLECULAR
1. Revisar y leer las prácticas para comprender los objetivos, conceptos básicos y
los procedimientos ha seguir.
2. Guardar los objetos personales debajo de la mesa o fuera del área de trabajo.
3. Limpiar y mantener el orden del área de trabajo antes, durante y después de
terminar con la práctica.
4. Preparar los reactivos, soluciones, materiales y equipo necesarios para llevar a
cabo la práctica. Revisar las fichas técnicas de las soluciones y reactivos que se
van utilizar en la práctica.
5. Los ácidos y las bases requieren de un manejo cuidadoso, usar el equipo de
protección adecuado como lentes, bata y guantes.
6. Usar los elementos básicos de seguridad como bata, gafas de seguridad, guantes,
máscara de gases (en caso de requerirlo) y zapatos cerrados.
7. No se permite el ingreso y/o consumo de alimentos o bebidas dentro del
laboratorio.
8. Tener buena ventilación en el área.
9. Nunca pipetear con la boca.
10. Tener ubicadas las áreas de regaderas o lavaojos en caso de algún incidente, al
igual que las salidas de emergencia del laboratorio.
11. El material con el que se va a trabajar debe estar completamente limpio.
12. Los tubos deben rotularse perfectamente antes de iniciar con la práctica con
marcador indeleble.
13. Los reactivos se colocarán en el orden indicado en la práctica y no deben de
modificarse a menos que el instructor lo indique.
14. Los reactivos se mantendrán siempre con su tapa para minimizar la
contaminación.
Instituto Tecnológico de Sonora
1
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
15. Solo utilizar la cantidad necesaria de los reactivos y nunca regresar los
excedentes al recipiente original, se puede contaminar.
Instituto Tecnológico de Sonora
2
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
ESTRATEGIA DE CONTENCIÓN DE SINIESTROS Y ACCIDENTES
EN EL LABORATORIO
En este ejercicio se pretende entrenar al alumno para la identificación de riesgos y la
toma de decisiones al momento de ocurrir algún siniestro. Con la finalidad de favorecer
un ambiente seguro y disminuir riesgos en el laboratorio. Se entiende por accidente el
acontecimiento inesperado que altera el curso normal de las cosas, teniendo efectos
negativos o daños a un individuo u objeto. En el laboratorio puede suceder explosiones,
incendios, etc. Por esto es importante demostrar las medidas de contención y mantener
ubicadas las vías de evacuación de los laboratorios.
Para el incremento de la seguridad se plantean las siguientes sugerencias al llevar a
cabo la evacuación:
•
•
•
•
•
•
Indicar las salidas de evacuación.
Conocer las rutas alternas de evacuación.
Evite el uso de elevadores, siempre use las escaleras.
Tener a la mano identificaciones personales.
Dirigirse al punto de reunión establecido por las autoridades.
Poner atención a las indicaciones del personal auxiliar.
Recomendaciones:
Al escuchar la alarma deberá suspender la actividad que se está realizando, mantener la
calma, no correr, no gritar, no empujar, alejarse de las ventanas, y retirarse del
laboratorio hasta el punto de reunión mas cercano, no entrar de nuevo a las
instalaciones, hasta que las autoridades competentes lo indiquen. En caso de quedarse
atrapado dentro del lugar, deberá permanecer bajo una mesa cubriéndose la cabeza.
Después de evacuar:
Verificar que nadie falte una vez reunidas todas las personas en el punto de reunión. El
maestro responsable deberá confirmar con la lista de alumnos.
Instituto Tecnológico de Sonora
3
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
RUTAS DE EVACUACIÓN
Laboratorio LV 700
Aula con 2 salidas:
o Seguir las indicaciones de la ruta de evacuación, hacerlo con calma y evitar tomar
rutas alternas (al menos que se indique).
o Con dos o más rutas se dirigirán hacia dichas salidas en forma paralela y en el
mismo sentido.
o Las rutas de evacuación no deben interceptarse o cruzarse.
Instituto Tecnológico de Sonora
4
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
PRÁCTICA 1. Uso y manejo correcto de las micropipetas
1.1 OBJETIVO
Aprender el uso y manejo adecuado de las micropipetas como instrumentos
indispensables en el laboratorio de Biología Molecular, así como los aspectos de
cuidado, seguridad y control de calidad requeridos.
1.2 INTRODUCCIÓN
El desarrollo de las prácticas en un laboratorio de Biología Molecular requiere que los
parámetros de exactitud, precisión y reproducibilidad se cumplan cabalmente.
Entendemos por exactitud la concordancia que existe entre el valor promedio
aritmético de diversos resultados y el valor real. La precisión es inversamente
proporcional a la dispersión de los resultados obtenidos de ensayos repetitivos sobre
una misma muestra, bajo condiciones constantes y determinadas.
La reproducibilidad se define como la variación que existe entre las medias del conjunto
de mediciones hechas por varios analistas, con los mismos equipos e instrumentos de
medición. Además de estos parámetros, un manejo adecuado de los reactantes
(muestras, reactivos y estándares), la calibración de los equipos, el uso de controles
internos (positivos y negativos) y la evaluación del rango de medición de las técnicas
aplicadas; reflejarán la calidad de los análisis y experimentos que se realicen.
Figura 1. Diferentes tipos de micropipetas utilizadas en el laboratorio de Biología
Molecular.
Seleccionando la micropipeta adecuada
Las micropipetas poseen un rango de volumen máximo y mínimo para la medición de
líquidos. Para cuidar los parámetros de calidad en la medición, se debe evitar rebasar
Instituto Tecnológico de Sonora
5
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
los rangos indicados. Por ejemplo: al usar una micropipeta con un rango de 20 a 200 µL,
se debe evitar tomar un volumen de 15 o 210 µL.
Uso adecuado de micropipetas
1. Sujetar la micropipeta con una mano y con la otra ajustar el micrómetro al
volumen que se desea medir.
2. Colocar la punta en la parte inferior de la micropipeta, antes de tomar el volumen
se deberá cuidar la inclinación, ya que es un factor muy importante para
garantizar la exactitud y precisión.
3. Presionar el émbolo con el pulgar hasta el primer tope, sumergir la punta y
regresar suavemente el émbolo a la posición inicial. Realizar el movimiento
lentamente.
4. Para descargar el líquido, se recarga la punta contra la pared del tubo y se
oprime el émbolo hasta el segundo tope.
5. Para desechar la punta, presionar el botón eyector y tomar una punta nueva de
ser necesario.
Figura 2. Procedimiento de uso y manejo de líquido con micropipetas.
Instituto Tecnológico de Sonora
6
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
Los componentes que integran una micropipeta se describen en la siguiente figura:
Figura 3. Componentes de una micropipeta.
1.3 MATERIALES
•
•
•
•
Micropipetas de 10, 20, 200 y 1000 µl.
Vaso de precipitado con 20 ml de agua destilada
Puntas de micropipetas de 10, 200 y 1000 µl (1 caja de cada volumen)
Guantes de Nitrilo (chicos, medianos y grandes)
1.4 PROCEDIMIENTO
Evaluación de la calidad del pipeteo
Para evaluar la exactitud y repetitividad con precisión del pipeteo, se realizarán 10
pipeteos de un volumen de 150 µl de agua, verificando por el peso y el volumen
pipeteado.
1.
2.
3.
4.
5.
Colocar un vaso de precipitados en la balanza analítica.
Tarar la balanza y verificar que se encuentre balanceada.
Pipetear 150 µl de agua en el vaso y anotar su peso.
Repetir los pasos del 1 al 3, 10 veces.
Realizar una tabla con los valores obtenidos.
Instituto Tecnológico de Sonora
7
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
1.5 RESULTADOS
Anotar en la siguiente tabla los resultados de las mediciones para cada muestra.
Muestra
1
2
3
4
5
Peso en mg
Muestra
6
7
8
9
10
Realizar los siguientes cálculos:
Peso promedio
Volumen promedio
Peso en mg
Coeficiente de variación
Interpretar los resultados y elaborar una conclusión.
1.6 CUESTIONARIO
1. ¿Cuál es la diferencia entre exactitud y precisión?
2. Mencione cuántos tipos de micropipetas existen y cuáles son sus aplicaciones.
Instituto Tecnológico de Sonora
8
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
3. Explique los problemas que pueden ocurrir al modificar el ángulo de inclinación
de la micropipeta al tomar una muestra.
4. ¿Cuáles son las ventajas del uso de micropipetas multicanal?
5. ¿Por qué es importante mantener calibrada una micropipeta?
6. Describe brevemente como realizarías la calibración de una micropipeta.
7. Imagine que tiene que tomar 100 μl de disolución. ¿Qué pipeta utilizaría, una
100, una 200 o una 1000? Justifique su respuesta con base a los resultados
obtenidos desde el punto de vista de la precisión y de la exactitud.
Instituto Tecnológico de Sonora
9
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
Diagrama de flujo Práctica 1
Instituto Tecnológico de Sonora
10
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
Notas Práctica 1:
Instituto Tecnológico de Sonora
11
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
PRÁCTICA 2. Aislamiento y purificación de ADN genómico de una
bacteria Gram positiva.
2.1 OBJETIVO
Realizar y comprender cada una de las etapas de la extracción de ADN genómico
utilizando una bacteria Gram positiva como modelo de estudio.
2.2 INTRODUCCIÓN
Los ácidos nucleicos son moléculas de transmiten la información genética de los
organismos. Esta información contenida en forma de genes dicta como se construye un
organismo. El genoma por sí solo no constituye el organismo, sino que sólo contiene la
información en forma de bases individuales las cuales forman los genes y determinan
estas características. Estas secuencias dan las indicaciones para la síntesis de las
proteínas que componen a todos los organismos vivos. La molécula que almacena la
información hereditaria es el ADN. En todos los organismos vivos es responsable de
transferir esta información de progenitores a hijos. En esta molécula se encuentra las
instrucciones necesarias para formar un organismo. La forma de cómo está dispuesta la
información en el genoma se le conoce como gen o genes. A los monómeros que dan
estructura al ADN se les denomina nucleótidos; los nucleótidos se componen por un
grupo fosfato, una pentosa, la desoxirribosa para el ADN y la ribosa para el ARN; y una
base nitrogenada, las cuales pueden ser adenina, guanina, timina, citosina y uracilo (esta
última en el caso del ARN). Las moléculas de ADN dentro de las células están arregladas
en cromosomas, en la división celular se duplican, este proceso de duplicación del
material genético se le llama replicación.
2.3 MATERIALES Y REACTIVOS
•
•
•
•
•
•
•
•
•
•
•
•
Cultivo de Listeria monocytogenes de 24 h.
Acetato de sodio 3M pH 5.2 (ver anexo 1)
SDS 10% Dodecil sulfato de sodio (SDS) (ver anexo 1)
Proteinasa K (20 mg/mL)
Solución fenol/cloroformo (50/50)
Isopropanol
Etanol al 75% preparado con agua ultra pura.
Puntas para micropipetas de 10, 200 y 1000 µL (1 caja de cada volumen)
Microtubo de 1.5 mLestériles.
Agua ultra pura libre de ADNasas y ARNasas.
Buffer de lisis con lisozima (20mg/mL) (ver anexo 1)
Tubos de 1.5 mL estériles
Instituto Tecnológico de Sonora
12
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
• Gradilla para tubos de 1.5 mL
EQUIPO
•
•
•
•
•
•
Centrifuga para microtubos
Micropipetas de 10, 20, 200 y 1000 µL
Vórtex
Termoblock o baño seco
Baño María
Guantes de Nitrilo (chicos, medianos y grandes)
2.4 PROCEDIMIENTO
•
•
•
•
•
Preparar todas las soluciones necesarias antes de iniciar la práctica.
Para llevar a cabo el procedimiento se tendrá que contar con todo el equipo
necesario de seguridad, el cual incluye, gafas, guantes y bata.
Un día antes, realizar un cultivo de Listeria monocytogenes en un matraz de 250
ml con 20 ml de caldo nutritivo, dejar crecer en agitación durante toda la noche a
35 °C y 200 rpm.
Procedimiento modificado y adaptado para bacterias Gram positivas (Cox, 1968).
Etiquetar adecuadamente los tubos de 1.5 mL que se usarán en la extracción.
Lisis y separación de fases
1. Hacer un pellet bacteriano tomando 1.5 mL del cultivo celular de 24 h y colocarlo
en un microtubo de 1.5 mL, después centrifugar por 5 min a 8000 rpm y
descartar el sobrenadante.
2. Agregar 200 µLde buffer de lisis (ver anexo 1) en el tubo donde se realizó el
pellet celular. Lisar las células por pipeteo suave de arriba-abajo e incubar a 37
°C por 30 min.
3. Se agregan 30 µL de SDS (10 %) y 3 µL de proteinasa K, se mezcla y se incuba a
65 °C por 30 min.
4. Agregar 230 µL de fenol/cloroformo y mezclar bien por inversión hasta
homogenizar bien las fases. CUIDADO el fenol causa quemaduras en la piel, usar
gafas protectoras, guantes, bata y tapar bien los tubos.
5. Centrifugar por 10 min a 10000 x g a 4 °C o a temperatura ambiente. Después
colocar el sobrenadante en un tubo nuevo y etiquetado de 1.5 mL.
Precipitación del ADN
6. Agregar 50 µL de acetato de sodio 3 M y 0.5 mL de isopropanol agitar y colocarlo
durante 15 min en hielo. Se observa el ADN como un precipitado. NOTA: EN
ESTE PUNTO SE PUEDE PARAR EL PROCEDIMIENTO INCUBANDO LAS
MUESTRAS EN EL REFRIGERADOR A 4 °C Y CONTINUAR LA SIGUIENTE SESIÓN.
Instituto Tecnológico de Sonora
13
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
7. Centrifugar a 13,000 rpm por 10 min a 4 °C para terminar de precipitar todo el
ADN.
8. Retirar el sobrenadante con una pipeta de 20 µL cuidadosamente del lado
contrario del tubo de donde se encuentra el precipitado de ADN.
Lavado del ADN
9.
Agregar 1 mL de etanol al 70 % para lavar el ADN por 30 segundos, centrifugar
y remover por decantación el etanol. Si quedan residuos de etanol retirarlos
cuidadosamente con la pipeta de 20 µL, repetir este paso una vez más.
10. Secar el ADN dejando el tubo abierto por 15 segundos después de remover
etanol.
Solubilización de ADN
11. Solubilizar el ADN agregando 50 µL de agua ultra pura libre ADNasas y
ARNasas.
12. Analizar la integridad de la molécula con una electroforesis en gel de agarosa al
1 %. Guardar el ADN a -20°C (congelador). El profesor determinará la sesión
pertinente para cuantificar por Nanodrop el ADN extraído para conocer la
concentración.
Protocolo de extracción de ADN de hongos modificado (Raeder & Broda, 1985).
1. Colocar 100 mg de micelio fresco en un tubo eppendorff de 1.5 mL
2. Agregar 500 µL de buffer de extracción (200 mM Tris HCl pH 8.0, 250 mM NaCl,
25 mM EDTA, 0.5 % SDS) resuspender el pellet celular, mezclar bien en vórtex
durante 5 min.
3. Añadir 350 µL de fenol frío (4 °C) y mezclar bien en vórtex a velocidad máxima
durante 30 segundos.
4. Añadir 150 µL de cloroformo frío y mezclar bien en vórtex a velocidad máxima
durante 30 segundos.
5. La suspensión obtenida se centrifuga durante 30 min a 13,000 rpm a 4 °C.
6. Tomar la fase acuosa y transferirla a un tubo eppendorff de 1.5 mL
7. Agregar 1 volumen de cloroformo, mezclar en vórtex durante 30 segundos.
8. Posteriormente, la mezcla se centrifuga por 10 min a 13, 000 rpm
9. La fase acuosa (400 µL aprox.) se transfiere a un tubo eppendorff de 1.5 mL y se
agregan 0.6 volúmenes de isopropanol (500 µL aprox.).
10. Homogenizar la mezcla por inversión (10 veces) y centrifugar por 5 min. a
13,000 rpm.
11. Descartar el sobrenadante.
Instituto Tecnológico de Sonora
14
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
12. Lavar el pellet obtenido con 500 µLde etanol al 70%, resuspender el pellet con
pequeños golpecitos con el dedo. Centrifugar a 13,000 rpm por 2 minutos a 4°C.
Descartar el sobrenadante. Repetir este paso 2 veces.
13. Secar el pellet con la tapa abierta del tubo por 1 min a temperatura ambiente y
resuspender en 40-70 µL de agua ultra pura.
14. Analizar la integridad de la molécula con una electroforesis en gel de agarosa al
1 %. Guardar a -20 °C.
2.5 RESULTADOS
Documentar todos los comportamientos de las soluciones durante la realización del
proceso de extracción. Definir la función de cada uno de los componentes en el proceso
de extracción de ADN y reportar las cifras de la cuantificación y pureza del producto.
2.6 CUESTIONARIO
1. ¿Para qué es útil una extracción de ADN?
2. ¿Cuál es el principio básico de cada uno de los componentes utilizados para la
extracción de ADN?
3. ¿Cuáles otros protocolos existen para la extracción de ácidos nucleicos?
4. ¿Cuál es el fundamento de cada una de las etapas de la extracción de ADN?
Instituto Tecnológico de Sonora
15
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
5. ¿Cuál es la importancia del uso de la lisozima en la extracción de ADN en
bacterias Gram positivas?
Instituto Tecnológico de Sonora
16
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
Diagrama de flujo Práctica 2
Instituto Tecnológico de Sonora
17
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
Notas Práctica 2:
Instituto Tecnológico de Sonora
18
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
PRÁCTICA 3. Extracción de ARN de bacteria Gram negativa.
3.1 OBJETIVO
Realizar extracción de ácido ribonucleico (ARN) utilizando el método convencional a
través de la utilización de fenol, cloroformo y alcohol isoamílico. Posteriormente se
evaluará calidad y cantidad utilizando un equipo Nanodrop y su integridad a través de
un gel de agarosa.
3.2 INTRODUCCIÓN
Las células existentes en los organismos vivos, guardan su información hereditaria en
forma de ADN de doble cadena, son dos polímeros paralelos no ramificados integrados
por cuatro tipos de monómeros. Estos monómeros son adenina (A), timina (T), citosina
(C) y guanina (G); formando una larga secuencia unidos entre sí y codifican la
información genética contenida en la célula. A diferencia del ADN, en el ARN se
encuentran los mismos monómeros, a excepción de la timina (T) que es sustituida por el
uracilo (U). La molécula de ARN es un intermediario en la función de transportar la
información genética, esta información pasa de estar contenida en el ADN a una
secuencia de nucleótidos de ARN. Esta información, aunque se encuentra copiada en
otra molécula, sigue estando escrita en el mismo lenguaje que en el ADN, sigue estando
en nucleótidos, de ahí viene el nombre de transcripción.
Recomendaciones para el manejo de ARN
•
Realice todos los pasos a temperatura ambiente (20-25 °C) a menos que se
indique lo contrario.
•
Use Fenol:Cloroformo:Alcohol Isoamílico frío (FCA).
•
Use material de plástico estéril desechable, envuelto individualmente y
puntas de pipeta estériles desechables libres de ARNasas y tubos.
•
Procure utilizar material estéril. Se deberán utilizar puntas y tubos libres de
ARNasas, así como agua destilada estéril o grado biología molecular.
•
Use guantes desechables mientras maneja reactivos y muestras de RNA
para prevenir la contaminación por ARNasas de la superficie de la piel;
cambie los guantes con frecuencia, particularmente como el protocolo
progresa de extractos crudos a los materiales más purificados.
•
Trabajar asépticamente cuando se manipula ARN.
Instituto Tecnológico de Sonora
19
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
3.3 MATERIALES
• Cultivo bacteriano (E. coli)
• Cloroformo
• Etanol (100% y 70%)
• Buffer de lisis con Lisozima (20 mg/mL) (ver anexo 1)
• Fenol:Cloroformo:Alcohol isoamílico (FCA 25:24:1) (ver anexo1)
• SDS (10%)
• Acetato de Sodio 3 M (pH 5.2) (ver anexo 1)
• Agua libre de ARNasas
• Puntas para micropipetas de 10, 200 y 1000 µL (1 caja de cada volumen)
• Tubos de 1.5 mL estériles
• Gradilla para tubos de 1.5 mL
EQUIPO
• Incubadora
• Centrífuga para microtubos
• Micropipetas de 10, 200 y 1000 µL.
• Nanodrop
• Baño maría a 65 °C
• Vórtex
• Termoblock o baño seco
• Guantes de Nitrilo (chicos, medianos y grandes)
3.4 PROCEDIMIENTO
• Preparar y tener lista las soluciones un día antes de iniciar la práctica.
• Un día antes realizar un cultivo de E. coli en un matraz de 250 mL con 20 mL de
caldo nutritivo y dejar crecer en agitación durante toda la noche.
• Para llevar a cabo el procedimiento se tendrá que contar con todo el equipo
necesario de seguridad, el cual incluye, gafas, guates y bata.
Lisis y separación de fases
1. Tomar 1 mLdel cultivo bacteriano y colocarlo en un tubo de 1.5 mL estéril,
centrifugar las células a 8000 rpm por 2 min a 4 °C. Retirar por decantación el
medio de cultivo y repetir este paso en el mismo tubo una vez más para
incrementar la biomasa celular.
2. Agregar 400 µL de buffer de lisis con lisozima 20 mg/mL(ver anexo 1) e
incubar por 2 min a temperatura ambiente (omitir este paso si se trabaja con
bacterias Gram negativas).
3. Adicionar 15 µLde NaOAc (pH 5.2) 3 M y 45 µL de 10% de SDS a las células y
dar vórtex por 10 seg.
4. Agregar 400 µL de FCA 25:24:1. Rápidamente dar vórtex.
Instituto Tecnológico de Sonora
20
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
5. Incubar 10 min a 65 °C, dar vórtex a cada tubo por 5-10 segundos. Regresar el
tubo a 65 °C cada vez. Al terminar incubar los tubos por 5 min en hielo.
6. Centrifugar a 14,000 rpm por 5 min a temperatura ambiente.
7. Después de la centrifugación, agregar 400 µL de cloroformo mezclar
suavemente.
8. Centrifugar a 14,000 rpm por 5 min a temperatura ambiente.
9. Transferir la fase acuosa que contiene el ARN a un tubo nuevo colocando la
muestra en un ángulo de 45° y pipetear la solución con mucho cuidado sin
transferir ni tocar la interfase o la fase orgánica.
Precipitación del ARN
10. Agregar 0.5 mL de isopropanol a la fase acuosa por cada 1 mL usado de FCA en
la lisis. Incubar 10 minutos a temperatura ambiente. NOTA: EN ESTE PUNTO SE
PUEDE PARAR EL PROCEDIMIENTO INCUBANDO LAS MUESTRAS EN EL
REFRIGERADOR A 4°C Y CONTINUAR LA SIGUIENTE SESIÓN.
11. Centrifugar por 10 min a 12000 rpm a 4 °C. El ARN se observa como un
pequeño pellet transparente en el fondo del tubo. Descartar el sobrenadante
con una micropipeta.
Lavado de ARN
12. Resuspender el ARN en 1 mL de etanol 75% por cada 1 mL de FCA usado en la
lisis.
13. Dar vórtex brevemente a la muestra y centrifugar por 5 min a 10000rpm a 4 °C.
Descartar el sobrenadante con una micropipeta.
14. Secar la muestra a temperatura ambiente por 5-10 min.
Solubilizar el ARN
15. Resuspender el pellet en 20-50 µL de agua libre de ARNasas. Incubar en baño
maría o termoblock a 55-60 °C por 10-15 min.
16. Cuantificar usando el espectrofotómetro Nanodrop y analizar la integridad de
la molécula con una electroforesis en gel de agarosa al 1%.
3.5 RESULTADOS
Documentar todos los comportamientos de las soluciones durante la realización del
proceso de extracción. Definir la función de cada uno de los componentes en el proceso
de extracción de ARN.
Instituto Tecnológico de Sonora
21
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
3.6 CUESTIONARIO
1. ¿Para qué es útil una extracción de ARN? ¿Qué tipos de análisis son posibles de
realizar posterior a la extracción de ARN?
2. ¿Cuáles otros protocolos existen para la extracción de ARN?
3. ¿Cuál es el fundamento de cada una de las etapas de la extracción de ARN?
4. ¿Cuántos tipos de ARN existen y cuáles son sus funciones?
5. ¿Cuáles con las principales diferencias entre el ARN y el ADN?
Instituto Tecnológico de Sonora
22
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
Diagrama de flujo Práctica 3
Instituto Tecnológico de Sonora
23
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
Notas Práctica 3:
Instituto Tecnológico de Sonora
24
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
PRÁCTICA 4. Electroforesis en gel de agarosa para visualización
de ácidos nucleicos
4.1 OBJETIVO
A través de esta práctica el alumno conocerá los principios y las aplicaciones de la
técnica de electroforesis.
4.2 INTRODUCCIÓN
El alumno conocerá y será capaz de describir, explicar los principios básicos y
principales aplicaciones de la electroforesis de ADN en geles de agarosa, así como de
realizar esta técnica de forma rutinaria.
La electroforesis en gel de agarosa es una técnica que se utiliza para la separación de
fragmentos de ADN que varían en tamaños desde 10 pb a 25 kb (Sambrook & Russell,
2001). La agarosa es un compuesto que se obtiene de las algas marinas pertenecientes a
los géneros Gelidium y Gracilaria, se compone de subunidades repetidas de agarobiosa
(L- y D-galactosa) (Lee et al., 2012).
Al gelificar, los polímeros de la agarosa se asocian de forma no covalente y forman una
red con tamaños de poro variable. El uso de la electroforesis en gel de agarosa ha
revolucionado la forma como se separa el ADN. La separación del ADN en agarosa, se
hace cargando el ADN dentro de los pozos preformados y posteriormente se aplica un
corriente eléctrica (Lee et al., 2012). Los grupos fosfatos en la molécula de ADN y ARN le
confieren una carga negativa y al estar dentro de un campo eléctrico, estas moléculas
tienden a migrar al ánodo cargado positivamente, por lo tanto, se van a separar por
tamaños dependiendo su peso molecular (Helling et al., 1974). La tasa de migración de
la moléculas de ADN o ARN a través del gel está determinada por las siguientes
características: El tamaño de la molécula, la concentración de la agarosa, la
conformación del ADN o ARN, el voltaje aplicado, la presencia de bromuro de etidio, el
tipo de agarosa y el buffer para la electroforesis (Figura 1) (Lee et al., 2012).
El tamaño de la molécula
Las moléculas de mayor tamaño se mueven más lentamente debido a la fricción en la
matriz porosa, teniendo más dificultad de migración que las moléculas pequeñas. El
ADN súper-enrollado migra más rápido que el ADN lineal en función de la naturaleza de
la molécula.
Instituto Tecnológico de Sonora
25
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
Concentración de agarosa
Una concentración menor hace que los poros sean de un mayor tamaño, permitiendo
que las moléculas chicas se muevan más rápido (Ver Tabla 1).
Tabla 1. Concentraciones de agarosa según la longitud de los fragmentos a separar extraída de Sambrook
y Russell (2001).
Concentración de agarosa (% [p/v])
0.3
0.6
0.7
0.9
1.2
1.5
2.0
Rangos de separación de moléculas
lineales de ADN (kb)
5-60
1-20
0.8-10
0.5-7
0.4-6
0-2-3
0.1 - 2
Voltaje aplicado
Con voltajes bajos, la velocidad de migración de los fragmentos de ADN es proporcional
al voltaje aplicado. Sin embargo, el intervalo efectivo de separación disminuye al
incrementarse el voltaje, por lo que no debe de aplicarse más de 5 u 8 V/cm.
Buffer de electroforesis
La movilidad electroforética del ADN es influenciada por la composición y la fuerza
iónica del buffer de electroforesis. En ausencia de iones (agua des ionizada) la
conductividad eléctrica es mínima y el ADN migra lentamente. En un buffer con una
fuerza iónica demasiado alta, la conductividad eléctrica es muy buena, aún con voltajes
moderados y se generan grandes cantidades de calor, lo que podría fundir el gel y
desnaturalizar el ADN.
Figura 1. Representación de una cámara de electroforesis y el gel de agarosa.
Esta imagen fue tomada de Khan Academy (bajo la licencia).
4.3 MATERIALES
• Matraz Erlenmeyer de 250 mL
• Agarosa grado biología molecular
Instituto Tecnológico de Sonora
26
Manual de Prácticas de Laboratorio de Biología Molecular
•
•
•
•
•
•
•
•
•
•
IB plan 2016
Buffer TAE o TBE de corrida de elerectroforesis 0.5 X
Buffer de carga 6 X
Muestras de ADN de diferentes tamaños, a concentraciones conocidas
Marcadores de peso molecular (1Kb Plus Ladder de Invitrogen)
SYBR® SAFE Gel ADN Stain 10,000 X
Puntas para micropipetas de 10, 200 y 1000 µL(1 caja de cada volumen)
Papel encerado (Parafilm)
Contenedor CrioSafe para tubos de 1.5 mL
Gradilla para tubos de 1.5 mL
Guantes de Nitrilo (chicos, medianos y grandes)
EQUIPO
• Cámara de electroforesis horizontal
• Fuente de poder
• Micropipetas de 10 y 20 µL
• Horno de microondas
• Transiluminador UV o fotodocumentador
4.4 PROCEDIMIENTO
Antes de iniciar la práctica, consulte las recomendaciones de seguridad especificadas
para cada sustancia utilizada (ver “Seguridad en el laboratorio de Biología Molecular” al
final de este Manual). Utilice bata y guantes en todo momento, en caso de requerirse
utilice lentes de seguridad con filtro UV. Verificar que haya suficiente cantidad de buffer
TAE antes de iniciar.
1. Lavar la base y las peinetas con agua corriente y jabón neutro, enjuagar con
abundante agua destilada, mínimo tres veces. Secar con mucho cuidado con papel
secante. Armar la base donde se va preparar el gel y colocarlo en una posición
horizontal.
2. Preparar la solución de agarosa con buffer TAE 0.5 X y para la electroforesis a una
concentración de 1 X. Agregar la cantidad de requerida de agarosa en gramos a un
matraz Erlenmeyer exclusivo para la preparación de geles que contiene la cantidad
en ml apropiada de buffer TAE 0.5 X.
3. Colocar el matraz con la suspensión en el horno de microondas por 30 segundos a la
máxima potencia, después sacarlo con cuidado y agitar para ayudar a disolver.
Posteriormente se coloca de nuevo en el horno por 30 segundos. Verificar que se
haya disuelto completamente toda la agarosa, enfriar a una temperatura que se
puedan soportar en la palma de la mano.
4. Agregar el bromuro de etidio a una concentración de 0.5 µg/ml, agitar para
homogenizar y vaciar en el molde. Evitar vaciar el gel caliente, ya que se daña el
Instituto Tecnológico de Sonora
27
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
molde para realizar los geles. EL BROMURO DE ETIDIO ES CANCERÍGENO Y
MUTAGÉNICO DEBE MANEJARSE CON MUCHO CUIDADO, evitar derramarlo.
5. Evitar la formación de burbujas en el gel al momento de vaciar. Dejar gelificar a
temperatura ambiente por 15 min.
6. Colocar la base del gel dentro de la cámara en dirección de corrida (de negativo a
positivo) y agregar buffer hasta cubrir el gel completamente y remover la peineta
con cuidado para no romper el gel.
7. Colocar 1 µL de buffer de carga 6X en un papel encerado (parafilm) y mezclarlo con
5 µL de ADN.
8. Con mucho cuidado cargar la mezcla en los pozos del gel sumergido usando una
micropipeta de 20 µL. Al final, colocar marcador de peso molecular en el primer
pozo.
9. Colocar la tapa de la cámara de electroforesis y verificar que los electrodos estén
debidamente conectados y en la posición correspondiente. Aplicar el voltaje de 1-5
V/cm (medido como la distancia entre el ánodo y cátodo). Correr el gel hasta que los
colorantes bromofenol azul y el xilen cianol migren a una distancia apropiada en el
gel (cuidar que durante la migración no salgan del gel).
10. Una vez terminada la electroforesis apagar la fuente de poder, retirar la tapa de la
cámara, examinar el gel en el transiluminador o fotodocumentador y capturar una
fotografía.
4.5 RESULTADOS
Evalúe las imágenes de los geles al comparar la migración de las bandas de cada carril y
comente el efecto de la concentración de agarosa. Determine el peso molecular aparente
de cada banda.
4.6 CUESTIONARIO
1. ¿Cuál es el fundamento de la electroforesis?
2. ¿Qué característica tienen los ácidos nucleicos que los hacen migrar?
Instituto Tecnológico de Sonora
28
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
3. ¿Cómo se calcula el voltaje aplicado a la cámara de electroforesis?
4. ¿Qué otros polímeros se usan para separar moléculas por electroforesis?
5. Menciona otro tipo de moléculas que se pueden separar por esta técnica
6. ¿A qué se debe que los colorantes se separen en la electroforesis?
Instituto Tecnológico de Sonora
29
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
Diagrama de flujo Práctica 4
Instituto Tecnológico de Sonora
30
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
Notas Práctica 4:
Instituto Tecnológico de Sonora
31
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
PRÁCTICA 5. Cuantificación y estimación de pureza de ADN
mediante espectrofotometría
5.1 OBJETIVO
Determinar mediante espectrofotometría la concentración y la pureza de los ácidos
nucleicos.
5.2 INTRODUCCIÓN
La espectrofotometría a través de la densidad óptica (DO) medida por la absorbancia
(ABS) se usa para evaluar la cantidad de ADN y su pureza. La molécula de ADN tiene una
absorción de luz a una longitud de onda de 260 nm (A260). Por medio de estas
mediciones de ABS se puede entonces calcular la concentración de ADN. Los valores de
las lecturas de DO tienen que mostrar un rango de 0.1 a 1. Hay otras moléculas como el
ADN que presentan absorbancia a 260 nm. Otras moléculas como el ARN de igual forma
absorbe luz a 260 nm, moléculas como los aminoácidos aromáticos absorben a 280 nm.
Estas otras moléculas pueden interferir en la medición de los ácidos nucleicos debido a
su ABS. Por esto no sólo se puede tomar la medida de 260 nm. La determinación de la
pureza del ADN se tiene que realizar en un rango de 230 a 320 nm para poder descartar
otras moléculas contaminantes durante la extracción.
Relación 260/280.
La relación de la absorbancia a 260 nm y 280 nm se utiliza para medir la pureza de ADN
y ARN. Una relación de 1.8 generalmente es aceptada para ADN, en cambio para el ARN
la relación aceptada es 2.0. Si estas relaciones son bajas, podría indicar la presencia de
proteínas, fenol o algún otro contaminante que absorba cerca de los 280 nm.
Por otro lado, sabemos que los nucleótidos que componen al ADN y ARN muestran una
amplia variación en las razones 260/280. Los siguientes datos representan las
proporciones estimadas para cada nucleótido si se midieran independientemente:
Guanina: 1.15
Adenina: 4.50
Citosina: 1.51
Uracilo: 4.00
Timina: 1.47
Instituto Tecnológico de Sonora
32
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
La relación 260:280 resultante para el ácido nucleico que se estudia será
aproximadamente igual al promedio de las relaciones de 260/280 para los cuatro
nucleótidos presentes. Es importante tener en cuenta que las relaciones generalmente
aceptadas de 1.8 y 2.0 para el ADN y el ARN, respectivamente, dependerá de la
composición del ácido nucleico. Nota: El ARN normalmente tendrá una relación
260/280 más alta debido a la mayor proporción de uracilo en comparación con la de
timina.
Relación 260/230.
Esta relación es utilizada como una medida secundaria de pureza. Los valores de
260/230 para ácidos nucleicos “puros” normalmente son más altos que los valores para
260/280. El rango esperado comúnmente es 2.0–2.2, si esta relación es baja podría
indicar la presencia de contaminantes que absorben a 230 nm como el EDTA,
carbohidratos y cloruro de guanidinio utilizados para el aislamiento de ADN, mientras
que el isotiocinato de guanidina se utiliza para la extracción de ARN y absorbe a 260 nm.
Lecturas a 320 nm.
Podrían indicar si hay turbidez en la solución, otra indicación de una posible
contaminación. Por lo tanto, analizar las lecturas del espectrofotómetro entre 230 nm y
320 nm da mayor información de la calidad de la muestra.
La cantidad de ADN puede se puede calcular midiendo la absorbancia a 260 y
multiplicándola por el factor de dilución y la relación A260 de 1.0 = 50 μg/mLde ADN:
𝐴𝐷𝑁 (𝜇𝑔⁄𝑚𝐿) = {(𝐴260 × 𝐴320) × (𝑑𝑖𝑙𝑢𝑐𝑖ó𝑛)} × 50
La cantidad total obtenida se calcula multiplicando la concentración de ADN obtenida en
la ecuación anterior y se multiplica por el volumen máximo de la muestra:
Cantidad total ADN (μg) = ADN (µg/mL) x (volumen final de la muestra)
La pureza de la muestra se obtiene mediante la relación A260/A280, después de ajustar
con el valor de turbidez:
Pureza ADN (A260/A280) = (A260-A320) / (A280-A320)
Instituto Tecnológico de Sonora
33
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
5.3 MATERIALES
• Agua grado molecular
• Puntas para micropipetas de 10, 200 y 1000 µL (1 caja de cada volumen)
• Tubos de 1.5 mL estériles
• Celdas para espectrofotómetro
• Gradilla para tubos de 1.5 mL
EQUIPO
•
•
•
•
•
Incubadora
Centrífuga para microtubos
Micropipetas
Nanodrop/Espectrofotómetro
Guantes de Nitrilo (chicos, medianos y grandes)
5.4 PROCEDIMIENTO
Utilice bata y guantes en todo momento.
1. Descongelar en hielo las muestras de ADN.
2. Agitar suave dando golpes con la yema del dedo índice (sin vórtex).
3. Realizar una dilución tomando 5 μL de la muestra y mezclar con 995 μL de agua
ultra pura (previamente rotulados).
4. Dar vórtex para homogenizar las diluciones.
5. Colocar las diluciones en celdas para medir en espectrofotómetro.
6. Ajustar a cero la ABS usando como control agua ultra pura.
7. Hacer un barrido entre 230 nm y 320 nm. En caso de no poder realizar barridos,
registrar los valores de absorbancia a 230 nm, 260 nm, 270 nm, 280 nm y 320
nm.
8. Después de obtener la concentración de las muestras de ADN, hacer los cálculos
necesarios para cargar 1 μg de ADN por pozo en un gel de agarosa y realizar la
electroforesis como en la práctica previa.
5.5 RESULTADOS
Documentar los valores de absorbancia en el espectrofotómetro, realizar un gráfico con
los valores obtenidos en el barrido y hacer una comparación con los valores obtenidos
en el gel de electroforesis de la próxima práctica.
Instituto Tecnológico de Sonora
34
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
5.6 CUESTIONARIO
1. ¿Por qué se recomienda medir la absorbancia a 280 nm?
2. Espectrofotométricamente no es posible distinguir una muestra pura de ADN a
una contaminada con ARN. Proponga un método que permita observar esta
contaminación.
3. ¿Cuál es la importancia de conocer los valores de concentración y pureza del
ADN aislado?
Instituto Tecnológico de Sonora
35
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
Diagrama de flujo Práctica 5
Instituto Tecnológico de Sonora
36
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
Notas Práctica 5:
Instituto Tecnológico de Sonora
37
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
PRÁCTICA 6. Reacción en cadena de la polimerasa PCR
(replicación artificial del ADN)
6.1 OBJETIVO
Conocer los principios básicos de la reacción en cadena de la polimerasa y llevar a
cabo la amplificación de un fragmento de ADN con oligonucleótidos universales para
bacterias.
6.2 INTRODUCCIÓN
El ADN es una macromolécula que almacena la información genética en todos los
organismos vivos, responsable de transferir esta información de progenitores a hijos.
En esta molécula se encuentra las instrucciones necesarias para formar un organismo.
La forma de cómo está dispuesta la información se le llaman genes y es la forma como
se dispone la información genética. A los monómeros que dan estructura al ADN se les
denomina nucleótidos; cada nucleótido está formado por un grupo fosfato, un azúcar
(Desoxirribosa en el ADN y Ribosa en el ARN) y una base nitrogenada (Adenina,
Guanina, Timina, Citosina y Uracilo (solo ARN). Las moléculas de ADN dentro de las
células están arregladas en cromosomas, en la división celular se duplican, este
proceso de duplicación del material genético se le llama replicación.
La técnica de reacción en cadena de la polimerasa es una emulación del proceso de
replicación en el laboratorio (in vitro), esta se lleva a cabo en equipo llamado
termociclador, ya que permite realizar ciclos de temperatura para la amplificación o
copiado de una secuencia de ADN (llamada templado o molde). Esta técnica permite
copiar una secuencia de ADN de forma selectiva y específica dentro de la muestra. Un
punto importante a considerar es la selección adecuada de los oligonucleótidos
(primers o cebadores) que flanquean la secuencia deseada o el gen diana. Estos deben
presentar ciertas características como: el tamaño óptimo entre 18 a 25 nucleótidos,
tener un contenido de guaninas y citosinas (GC) entre 40 y 60 %, no tener variaciones
superiores a los 5 °C entre las temperaturas de anillamiento de ambos oligos y los
extremos 3’ de los oligonucleótidos no debe ser complementarias entre sí. Esta es una
herramienta ampliamente utilizada en el área de la Biología Molecular principalmente
en el mapeo de genes, clonación, detección y reacciones de secuenciación.
La reacción de PCR consiste en cambios secuenciales de 3 temperaturas en forma
cíclica:
Instituto Tecnológico de Sonora
38
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
Paso 1. Desnaturalización. Se ajusta la temperatura entre 94-98 °C para romper los
enlaces puente de hidrógeno del ADN templado para que las cadenas se abran y
permitan la entrada de los oligonucleótidos.
Paso 2. Alineamiento. Se ajusta la temperatura entre 55-65 °C, tomando en cuenta el
Tm de los oligonucleótidos. En este paso los oligonucleótidos se hibridan con sus
respectivas secuencias complementarias.
Paso 3. Elongación, síntesis o polimerización. En este paso se ajusta la temperatura
a entre 70- 75 °C, esta es la temperatura óptima en la cual trabaja la enzima, en este
paso se lleva a cabo la síntesis de la nueva cadena de ADN que se quiere amplificar o
copiar.
La secuencia de estos pasos en conjunto son 1 ciclo de temperaturas, en una reacción
de PCR estándar se usan hasta 30 ciclos; permitiendo aproximadamente amplificar
1073 millones de copias.
6.3 MATERIALES
• Tubos de PCR 0.2 mL
• Buffer PCR 10 X
• MgCl 50 mM
• dNTPs 10 mM
• Oligonucleótidos (Forward y Reverse)
• Taq ADN polimerasa 5 U/µL
• Agarosa
• Buffer TAE para electroforesis
• Buffer de carga 6 X
• Marcador de peso molecular
• Parafilm
• ADN molde (Muestra de ADN extraído en la práctica 3)
• Agua grado molecular
• Micropipetas
• Puntas para micropipeta (1 caja de cada volumen)
Instituto Tecnológico de Sonora
39
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
EQUIPO
• Termociclador
• Transiluminador
• Fuente de poder
• Cámara de electroforesis
• Horno de Microondas
• Guantes de Nitrilo (chicos, medianos y grandes)
6.4 PROCEDIMIENTO
1. Etiquetar cuatro tubos de PCR de 0.2 mL, se utilizarán dos para la muestra
problema y dos para el control negativo (colocar agua ultra pura en lugar de ADN).
2. Descongelar los reactivos de PCR en hielo buffer de PCR, dNTPs, los primers y el
ADN. Mantener el agua en hielo, agitar todos los reactivos menos la enzima. NOTA:
la enzima se retira del congelador hasta su uso y mantenerla en hielo en todo
momento. Regresar la enzima al congelador inmediatamente después de usar.
3. Etiquetar un tubo de 1.6 mL nuevo estéril y colocar en hielo, agregar los
ingredientes para hacer la mezcla de reacción necesaria para las cuatro reacciones
(muestra y control negativo por duplicados), tomando en cuenta realizar los
cálculos con un excedente de media reacción más para que no falte. Agregar los
componentes en el orden como se muestran en la Tabla 1, iniciando con el agua,
terminando con la enzima y omitiendo el ADN, este se agrega al final a cada tubo
de 0.2 mL por separado. También se muestra los volúmenes necesarios para una
reacción y calcular los volúmenes requeridos para 5 reacciones.
4. Distribuir en cada tubo la mezcla de reacción y después el ADN, agua ultra pura e
lugar de ADN para el control negativo.
5. Dar unos golpes suaves a cada tubo para mezclar los componentes y centrifugar
por 10 segundos para que todo el líquido se vaya al fondo del tubo.
6. Colocar el programa en el termociclador con las condiciones para amplificar
correspondientes a los oligos.
7. Para ver la amplificación realizar un gel de agarosa al 1% y correrlo a 85V/45 min.
Instituto Tecnológico de Sonora
40
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
Tabla 1. Componentes de la mezcla para PCR.
Componente
Volumen para 25 µL
Agua Ultra Pura
Buffer PCR 10 X
MgCl2 50 mM
10 mM dNTPs
10 µM Forward
10 µM Reverse
Taq DNA Pol.
ADN (10 ng/µL)
17.65
2.5
0.75
0.5
1.25
1.25
0.1
1
Volúmenes para 5 rxn’s
Tabla 2. Condiciones para la amplificación
Pasos
Desnaturalización
Desnaturalización
Alineamiento
Extensión
Extensión final
Final
Ciclos
1
30
1
1
Temperatura
95 °C
95 °C
60 °C
72 °C
72 °C
18 °C
Tiempo
5 min
20s
20s
1:30
5 min
∞
6.5 RESULTADOS
Describir detalladamente la imagen observada del gel de agarosa e interpretar y
determinar los tamaños moleculares (pb) de los fragmentos amplificados.
6.6 CUESTIONARIO
1. ¿Cuál es la función de los componentes usados en una reacción de PCR?
Describa cada uno de ellos.
Instituto Tecnológico de Sonora
41
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
2. ¿Por qué se llama Taq ADN polimerasa? ¿Cuál es su característica principal?
3. Explique el fundamento del uso de un primer para cada extremo de la región
de ADN que se desea amplificar
4. ¿A qué se debe que los primers tengan diferentes temperaturas de
anillamiento? ¿Cómo se resuelve esto?
5. ¿Qué importancia tiene el contenido de G-C en el diseño de primers?
Instituto Tecnológico de Sonora
42
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
Diagrama de flujo Práctica 6
Instituto Tecnológico de Sonora
43
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
Notas Práctica 6:
Instituto Tecnológico de Sonora
44
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
PRÁCTICA 7. Enzimas de restricción y su aplicación en la
tecnología del ADN recombinante.
7.1 OBJETIVO
Comprender el fundamento, utilidad y potencial de las enzimas de restricción en la
tecnología del ADN recombinante.
7.2 INTRODUCCIÓN
La tecnología del ADN recombinante permitió a los científicos estudiar y experimentar
con ADN. Esta tecnología permite copiar, leer sus secuencias e introducirlo en nuevas
células. Estas técnicas son ampliamente usadas para transformar plantas, tratar y
diagnosticar enfermedades. Cuando una molécula de ADN de dos diferentes
organismos se combina, la molécula unida se llama ADN recombinante. Las bacterias
producen enzimas llamadas endonucleasas de restricción que cortan el ADN en piezas
más pequeñas (Figura 1). Las enzimas de restricción fueron descritas por Werner
Arber junto con Stuart Linn, las descubrieron mientras estudiaban un fenómeno que
ocurre en bacterias llamado restricción de bacteriófagos que es controlada por el
huésped. Los bacteriófagos son partículas virales que invaden las bacterias y replican
su ADN de forma independiente al ADN cromosomal bacteriano (Pray, 2008). Arber
propuso que las células bacterianas fueron capaces de protegerse contra ADN extraño
por medio de un tipo de mecanismo de defensa enzimático (Arber, 1969).
Arber tenía la teoría que solo aquellos bacteriófagos que habían estado en contacto
con la misma cepa bacteriana anteriormente, podrían infectar con éxito nuevas células
huésped, generando con esta interacción alguna protección contra la restricción. Los
fagos con su ADN sin modificar, por otra parte, fueron inmediatamente lisados por las
enzimas de restricción bacterianas. Las bacterias reconocen el ADN de los fagos como
extraño, lo rompen en sitios específicos interrumpiendo su desarrollo. Con estas
teorías llegaron a la conclusión Arber y Linn que las responsables eran las
endonucleasas de restricción y la llamaron “endonucleasa R” (Pray, 2008).
Instituto Tecnológico de Sonora
45
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
Figura 1. Ejemplificación de corte de un plásmido con el uso de enzimas de
restricción y da como resultado un corte con extremos pegajosos.
Estas enzimas reconocen sitios específicos para cortar, llamadas sitios de restricción.
Al cortar el ADN en sitios de restricción igual, pero en moléculas de ADN diferentes,
estas se pueden combinar.
7.3 MATERIALES
• Productos de PCR de la práctica anterior.
• Buffer TAE 0.5 X
• Buffer de carga 6 X
• Enzima EcoRI
• Buffer de restricción 10 X para EcoRI
• Marcador de peso molecular
• Tubos de 1.5 mL
• Micropipetas y puntas de 200, 20 y 10 µL
• Agarosa al 1%
• Matraz Erlenmeyer de 250 mL
• Solución de bromuro de etidio (10 mg/mL)
EQUIPO
• Incubadora o termociclador a 37 °C
• Cámara de electroforesis
• Fuente de poder
• Horno de microondas
• Guantes de nitrilo (chicos, medianos y grandes)
7.4 PROCEDIMIENTO
1. Etiquetar 3 tubos, dos para la muestra problema con EcoRI y uno para control
negativo (sin enzima).
2. Calcular los volúmenes de reacción a utilizar de acuerdo con la Tabla 1 y
colocar 500 ng de ADN.
Instituto Tecnológico de Sonora
46
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
3. Cambiar siempre las puntas entre cada reactivo.
Tabla 1. Reacción estándar de restricción.
Enzima de restricción
ADN
10X Buffer de enzima
Agua ultra pura
Volumen total
0.5 μL
1 μL
1 μL
7.5 μL
10 μL
4. Mezclar todos los componentes en cada tubo previamente etiquetado,
mantener todos los componentes en hielo. Las enzimas sacarse del congelador
e inmediatamente después regresarse al momento de tomar el volumen
necesario.
5. Una vez preparada la reacción, dar unos golpes suaves con el dedo para
mezclar los componentes.
6. Incubar a 37 °C por 90 min.
7. Preparar un gel de agarosa al 1 % con TAE 0.5 X y correr una electroforesis
para visualizar las digestiones.
7.5 RESULTADOS
Interpretar la imagen y los patrones de bandeo obtenidos. Identificar los pesos
moleculares de cada una de las bandas observadas.
7.6 CUESTIONARIO
1. ¿Para qué son importantes las enzimas de restricción?
2. ¿Cuál es su implicación en los sistemas de transformación bacteriana?
Instituto Tecnológico de Sonora
47
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
3. De un ejemplo diferente al de esta práctica de una secuencia palíndrome
4. Investigue 3 ejemplos de otras enzimas restricción que existen.
Instituto Tecnológico de Sonora
48
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
Diagrama de flujo Práctica 7
Instituto Tecnológico de Sonora
49
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
Notas Práctica 7:
Instituto Tecnológico de Sonora
50
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
PRÁCTICA 8. Tranformación bacteriana.
8.1 OBJETIVO
Realizar una ligación de un fragmento de ADN en un vector para su posterior
inserción en células competentes de Escherichia coli.
8.2 INTRODUCCIÓN
Los organismos procariotes, donde se incluyen bacterias y microorganismos unicelulares
llamados Archaea, usualmente pueden pasar ADN cromosomal a su descendencia de forma
asexual. La célula bacteriana se reproduce por simple replicación de su cromosoma y se
divide en sus dos células hijas. Las células hijas que resultan de esta división son
genéticamente idénticas una a la otra. La reproducción bacteriana entonces puede generar
miles de millones de células, las cuales son genéticamente idénticas a la célula parental
original.
Dada su reproducción asexual, esto puede causar que pensemos que las bacterias acrecen de
variación genética, pero aquí no es el caso. Las células procarioticas han desarrollado
diversos métodos para recombinar su material genético, lo que a su vez, contribuye a su
variación genética. Los métodos más comunes son transformación, conjugación y
transducción. No todas las bacterias son capaces de llevar a cabo los tres procesos.
La Transformación es un proceso por el cual una bacteria susceptible o competente
adquiere material genético nuevo de su ambiente. Hay dos tipo de transformación: natural y
artificial.
La transformación natural, como su nombre lo implica, es un mecanismo natural usado por
las células bacterianas para tomar ADN del ambiente. Este ADN alguna vez formó parte de
otra bacteria. Por ejemplo, cuando una bacteria muere y se desintegra, su cromosoma se
libera al medio ambiente. Los fragmentos de este ADN permanecen en el medio ambiente y
están disponibles para otras células vivas, incluidas las bacterias. Este material genético
puede entrar a una bacteria viva a través de la membrana. Si es ADN de doble cadena, una
de las cadenas pasará a través de la membrana, y la otra cadena se disolverá o será
hidrolizada. Partes de la molécula monocatenaria recién introducida se recambiará en
regiones similares en el cromosoma bacteriano e incorporarse al genoma de la bacteria.
En contraste, durante la transformación artificial, la adquisición del ADN por la célula
hospedera ocurre en condiciones de laboratorio. En el laboratorio, los científicos introducen
ADN exógeno en células bacterianas vía transformación para estudiar genes específicos y
sus funciones. Típicamente, estos investigadores usa células de E. coli que han sido
químicamente tratadas para que sus membranas celulares externas sean permeables al ADN
exógeno. Adicionalmente, la transformación puede ser inducida por electroporación, un
proceso que facilita el paso de las moléculas de ADN a través de la membrana mediante la
aplicación de un campo eléctrico (Figura 1). El choque térmico es otra forma de realizar
Instituto Tecnológico de Sonora
51
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
una transformación bacteriana, en donde las células hospederas son expuestas a
temperaturas extremas que también permiten que la membrana celular permita
temporalmente la entrada de moléculas de ADN dentro de la célula.
Figura 1. Mapa del vector pGEM®-T Easy.
8.3 MATERIALES
• Productos de PCR purificados de la práctica 6.
• Hielera o contenedor con hielo en fragmentos pequeños.
• Buffer de ligación 2 X
• Vector de clonación pGEMT-Easy
• T4 ADN ligasa de promega.
• Células competentes de Escherichia coli.
• IPTG
• X-Gal
• Ampicilina solución
• Caldo LB (Luria Bertani) (100 mL)
• Placas de LB
• Medio SOC
• Micropipetas y puntas de 200, 20 y 10 µL
Instituto Tecnológico de Sonora
52
Manual de Prácticas de Laboratorio de Biología Molecular
•
•
IB plan 2016
Tubos ependorf de 0.2 mL
Tubos ependorf de 1.5 mL
EQUIPO
• Incubadora a 37 °C
• Baño maría a 42 °C
• Fuente de poder
• Horno de microondas
• Guantes de nitrilo (chicos, medianos y grandes)
8.4 PROCEDIMIENTO
Nota: Las soluciones, reactivos y medios de cultivo necesarios para llevar a cabo esta
práctica se preparan como se indica en el Anexo 1.
Ligación. Se realizará un la ligación de productos de PCR con el vector pGEM-T EASY
(Procedimiento Modificado).
1.-Seguir estas recomendaciones, centrifugar el vector y ADN problema brevemente,
antes de realizar las reacciones dar vórtex al buffer, mezclar las reacciones usando un
pipeteo lento.
2.-Montar la siguiente reacción de ligación para cada una de las muestras:
Componente
Buffer de ligación 2X
Vector diluir a 50ng/uL
Producto de PCR
T4 DNA Ligasa 3U/uL
Volumen total
Volumen
5.0 uL
0.5 uL
3.5 uL
1.0 uL
10.0 uL
3.-Incubar toda la noche a 4ºC para obtener el mayor número de transformantes.
4.-Proceder a transformar.
Transformación. Transformación de células competentes JM109 o DH5-alpha con el vector
pGEM-T- Easy PROMEGA (MODIFICADO).
1.-Antes de iniciar preparar dos placas de LB adionado con Amp/IPTG/X-Gal (Anexo 1)
para cada reacción de ligación.
2.-Centrifugar los tubos con las reacciones de ligación para colectar el contenido en el
fondo y colocarlos en hielo.
Instituto Tecnológico de Sonora
53
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
4.-Descongelar los tubos de células competentes en hielo, no agitar las células competentes,
evitar cualquier movimiento brusco.
5.- Colocar 1 uL de la ligación en un tubo de células manteniéndolas en hielo.
6.-Mezclar mediante golpes ligeros con los dedos y colocar en hielo por 20min.
7.-Dar choque térmico por 45-50 seg a EXACTAMENTE A UNA TEMPERATURA DE
42°C (NO AGITAR).
8.-Colocar inmediatamente en hielo por 2 min.
9.-Agregar 989 uL de medio SOC (ver Anexo 1).
10.-Incubar por 1.5 horas a 37°C con agitación (150-200 rpm).
11.-Centrifugar a 4000 RPM por 2 min, eliminar el sobrenadante dejando 50 uL para
resuspender el paquete celular y plaquear el total de células en las placas de LB
amp/IPTG/X-Gal previamente preparadas.
12.-Incubar toda la noche (16-18 horas) a 37°C. Y posteriormente se tiene que analizar las
transformantes.
8.5 RESULTADOS
Realizar una extracción de ADN plasmídico a 3 clonas positivas tranformadas
siguiendo el protocolo del anexo 1, verificar los plásmidos con una sola reacción de
restricción.
En el reporte se entregará como resultado en análisis de las tranformantes en los geles
de agarosa con su debida descripción.
8.6 CUESTIONARIO
1. En la transforción bacteriana, ¿Qué es un vector?
2. Para poder persistir y mantenerse de manera estable en la célula, ¿una
molécula de ADN plasmídica debe contener qué?
Instituto Tecnológico de Sonora
54
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
3. ¿Cuál es el propósito del cloruro de calcio en la transformación bacteriana?
4. Indica los métodos de tranformación bacteriana.
5. Define los métodos de la pregunta 4.
6. ¿Todas las bacterias son competentes para tranformación? Fundamenta tu
respuesta.
Instituto Tecnológico de Sonora
55
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
Diagrama de flujo Práctica 8
Instituto Tecnológico de Sonora
56
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
Notas Práctica 8:
Instituto Tecnológico de Sonora
57
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
PRÁCTICA 9. Análisis de secuencias de ADN (secuenciación
Sanger)
9.1 OBJETIVO
Aplicar herramientas bioinformáticas básicas por medio del análisis de secuencias de
ácidos nucleicos para la identificación molecular de microorganismos.
9.2 INTRODUCCIÓN
La bioinformática es conceptualizar la biología en términos de las macromoléculas y
aplicar técnicas informáticas (derivadas de disciplinas como las matemáticas
aplicadas, ciencia computacional y estadística) para comprender y organizar toda
información asociada a estas moléculas a gran escala. Entre los años 70 y 80 empezó
una acumulación de información biológica creando la necesidad de desarrollar bases
de datos de secuencias de ADN. Debido a esto, se fundaron GenBank (1979), EMBL
(1980) en el Laboratorio Europeo de Biología Molecular y el DDBJ (1984) (Banco de
datos de ADN de Japón). Posteriormente, herramientas de búsqueda como Entrez
(Sistema de búsqueda global en base de datos cruzada) se desarrollaron para permitir
búsquedas rápidas en bases de datos vía web y palabras clave. Por lo tanto, con la
primera base de datos de secuencias proteicas y los trabajos pioneros de homología
de proteínas surgió un gran interés en detectar homología y similitud
estadísticamente significativa entre proteínas lejanamente relacionadas, creando
herramientas y algoritmos para los alineamientos de secuencias (Gonzalez et al.,
2017).
Dos secuencias que comparten un ancestro común son secuencias homologas. Dos
proteínas homologas provienen de dos genes homólogos y comparten la misma
estructura tridimensional y la misma función biológica. Normalmente se confunde
homología con similitud, pero la homología es cualitativa y solo admite dos estados: o
ser homologo o no serlo, en cambio la similitud es cuantificable. Sin embargo, el grado
de similitud entre dos secuencias nos puede ayudar para inferir la existencia o no de
homología entre ambas. Actualmente la comparación de secuencias es una
herramienta muy poderosa para comprender la función biológica de una secuencia
desconocida mediante la búsqueda en bases de datos de secuencias homologas
previamente caracterizadas. Con los avances en las tecnologías de secuenciación de
ADN, la disponibilidad de bases de datos de secuencias, junto con el desarrollo de
eficientes algoritmos de comparación de secuencias han creado un cambio sustancial
en la biología molecular y celular, ya que actualmente cuando se descubre un nuevo
gen, no se va directamente al laboratorio a realizar experimentos para describir su
Instituto Tecnológico de Sonora
58
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
función celular, simplemente se introduce su secuencia en un ordenador esperando
encontrar genes homólogos de otros organismos ya caracterizados (Abascal et al.,
2014).
Para esta práctica, utilizaremos alineamientos con secuencias provenientes de
secuenciación Sanger, las cuales se encuentran en un archivo con formato “.ab1”
llamado electroferograma (gráfico que arroja el secuenciador después de analizar la
electroforesis) junto con la información de calidad de cada base. Una inspección
detallada de los electroferogramas permite descubrir de una manera sencilla algunos
de los problemas comunes que pueden tener las secuencias para determinar si se
necesita realizar alguna edición. Un electroferograma con una buena calidad muestra
picos únicos, bien definidos y con poco ruido de fondo (Figura 1).
Figura 1. Ejemplo de electroferograma con buena calidad.
En la figura 2 y 3 se muestran electroferogramas con una alta señal de fluorescencia
inicial que después disminuye rápidamente. Esto ocurre en aquellos casos con
muestras con poca cantidad de ADN o con concentraciones muy bajas de primers de la
reacción de secuenciación. También, se observan este tipo de resultados cuando
existen contaminantes o cuando los primers son poco específicos.
Figura 2. Ejemplo de electroferograma con mala calidad.
Instituto Tecnológico de Sonora
59
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
Figura 3. Ejemplo de electroferograma de mala calidad por contaminación en la
muestra.
9.3 MATERIALES
Electroferorogramas (formato .ab1)
EQUIPO
Computadora con internet y el programa Chromas instalado, el cual se puede
encontrar de manera gratuita en la web (https://technelysium.com.au/wp/).
9.4 PROCEDIMIENTO
Análisis de electroferograma y edición de secuencia
1. Para visualizar el electroferograma debes tener instalado el programa Chromas en
tu computadora. Una vez instalado, ábrelo y carga el archivo del electroferograma que
te proporcione el instructor.
2. El siguiente paso es analizar la señal de fluorescencia mostrada en tu gráfica y las
calidades para cada nucleótido asignado.
3. Hay que tomar decisiones sobre la edición de la secuencia. Por lo general, la calidad
es baja a los extremos por lo que estos nucleótidos se seleccionan para eliminarlos.
También, hay que verificar que las bases indicadas en la secuencia correspondan a los
picos con mayor señal en la gráfica. Si estos no corresponden, se selecciona la base
incorrecta y se sustituye por la correcta.
4. Al terminar con la edición de la secuencia, se guarda en formato FASTA para
proceder al alineamiento en la base de datos.
Alineación de secuencias en BLAST.
1. Para alinear tu secuencia de ADN ingresa a la página de BLAST
https://blast.ncbi.nlm.nih.gov/Blast.cgi
Instituto Tecnológico de Sonora
60
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
2. Elegir la opción de alineamiento de “nucleótido – nucleótido” (blastn).
3. Ingresar la secuencia que se desea alinear en el apartado “Enter query sequence”, ya
sea copiando y pegando o subiendo tu archivo fasta desde “seleccionar archivo”.
4. Una vez cargada la secuencia, elegir la opción BLAST para que el programa realice
los alineamientos.
Instituto Tecnológico de Sonora
61
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
5. Interpretar los resultados revisando los parámetros entre cada alineamiento:
cobertura de secuencia, E-value (nos interesa un valor cercano a 0), % de identidad,
etc., ya que estos nos indican que tan similares son las secuencias alineadas y la
probabilidad de error que se puede obtener al haberse realizado estos alineamientos
al azar.
9.5 RESULTADOS
Describe las observaciones que te llevaron a decidir si identificaste o no al
microorganismo con el que trabajaste durante este análisis.
9.6 CUESTIONARIO
1. Describe el fundamento de la Secuenciación Sanger.
2. Investiga el significado de los valores de calidad phred.
3. Investiga los genes más utilizados para la identificación molecular de bacterias,
hongos y otros eucariotas.
Instituto Tecnológico de Sonora
62
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
Diagrama de flujo Práctica 9
Instituto Tecnológico de Sonora
63
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
Notas Práctica 9:
Instituto Tecnológico de Sonora
64
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
ANEXO 1. SOLUCIONES Y REACTIVOS.
A1. Fenol:Cloroformo:Alcohol isoamílico (FCA) (25:24:1)
Antes de hacer esta solución, el fenol tiene que ser ajustado a pH > 7.8 ya que el ADN
se dirige a la fase orgánica en pH ácidos. El fenol debe ser guardado a -20 °C. Mezclar
volúmenes iguales de fenol equilibrado y cloroformo. Guardar la solución en una
botella con la tapa no muy apretada a 4 °C. Tener en cuenta las medidas necesarias de
seguridad antes de manipular el fenol.
A2. Buffer de lisis
Lisozima
100 mM Tris-NaCl (pH 7.6)
100 mM EDTA (pH 8.0)
Esterilizar las soluciones por autoclave por 15 min a 15 psi. Almacenar el buffer a
temperatura ambiente. Preparar 10 mL de buffer de lisis, agregar a un tubo de 15 ml,
1.2 ml de Tris HCl, 0.4 mL de EDTA, 80 mg de lisozima y 2.4 mL de agua ultra pura;
estos volúmenes se obtuvieron si los alumnos hacieron 12 extracciones en total (3 por
equipo en caso de ser 4 equipos). La lisozima se agrega justo antes de usar a una
concentración de 20 mg/mL.
A3. Acetato de Sodio
Para preparar una solución 3 M: Disolver 408.3 g de acetato de sodio•3H2O en 800 mL
de H2O. Ajustar el pH a 5.2 con ácido acético glacial y ajustar el pH a 7.0 con ácido
acético diluido. Ajustar el volumen a 1 litro con H2O. Hacer alícuotas y esterilizar en
autoclave.
A4. SDS 10% (p/v) (10 ml)
Colocar 1 g de SDS en 8 ml de agua ultra pura y agitar con agitador magnético.
Calentar un poco para solubilizar más rápido, si requiere ajustar el pH usar una
solución de HCl y ajustar a 7.2, y ajustar el volumen a 10 mlL. Almacenar a
temperatura ambiente. Utilice bata y guantes en todo momento para preparar las
soluciones.
A5. Fenol:cloroformo (1:1) (v/v) (10 mL)
Antes de hacer esta solución, el fenol se ajusta el pH > 7.8, ya que el ADN se dirige a la
fase orgánica en pH ácidos. El fenol debe ser guardado a -20 °C. Mezclar volúmenes
iguales de fenol equilibrado y cloroformo. Guardar la solución en una botella con la
tapa no muy apretada a 4 °C. Tener en cuenta las medidas necesarias de seguridad
Instituto Tecnológico de Sonora
65
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
antes de manipular el fenol.
A6. EDTA 0.5 M (pH 8)
Agregar 186.1 g de EDTA sal disódica•2H2O en 800 mL de agua destilada. Agitar
vigorosamente con un agitador magnético. Ajustar el pH con NaOH (agregar aprox. 20
g de perlas de NaOH). Separar en alícuotas y esterilizar por autoclave. La sal disódica
de EDTA no se disolverá por completo hasta alcanzar un pH cercano a 8 agregando
NaOH.
A7. Buffer TAE 50X (1L)
Disolver 242 g de Tris base, 57.1 mL de ácido acético glacial, 100 mL de 0.5 M de EDTA
(ph 8) en 600 ml de agua destilada.
A8. Buffer de carga 6 x (5 mL)
0.25% (p/v) de azul de bromofenol, 0.25% de xilen-cianol (p/v), 15% (p/v) de Ficoll o
glicerol, en agua destilada. Dividir en alícuotas de 1 mL en tubos de 1.6 mL. Conservar
en congelación a -20 °C.
A9. Bromuro de etidio (10 mg/mL)
Será preparado por el profesor.
Composición de buffer y soluciones para transformación de células
competentes JM109 pGEM-T- Easy PROMEGA
A10. IPTG, sol. Stock (0.1 M)
1.2 g de IPTG, llevar a 50 mL con agua destilada, esterilizar por filtración y almacenar a 4
°C.
A11. X-Gal (2 ml)
100 mg de 5-bromo-4-chloro-3-indolyl-β-D-galactoside. Disolver en 2 mL de N,N’dimethyl formamide. Cubrir con papel aluminio y almacenar a -20 °C.
A12. Medio LB (por litro)
10 g de bacto triptona
5 g de extracto de levadura
5 g de NaCl
Ajustar el pH a 7.0 con NaOH
A13. Placas de LB con Ampicilina
Agregar 20 g de agar por litro de medio LB. Esterilizar a 121 °C / 15 min. Dejar enfriar el
medio a 50 °C aprox. y agregar ampicilina a una concentración final de 100 ug/mL.
Almacenar a 4 °C por un mes o a temperatura ambiente por una semana.
Instituto Tecnológico de Sonora
66
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
A14. Placas LB/ampicilina/IPTG/X-Gal
Preparar las placas con ampicilina y distribuir sobre ellas 100 µL de IPTG 100 mM y 20 µL
de X-Gal 50 mg/mL y dejar absorber por 30 minutos antes de su uso a 37 °C.
A15. Medio SOC (100mL)
2.0 g de bactotriptona
0.5 g de extracto de levadura
1.0 mL de NaCl 1M
0.25 mL de KCl 1M
1.0 mL de Mg2+ 2M esterilizado por filtración
1.0 mL de glucosa 2M esterilizado por filtración
Añadir la bactotriptona, el extracto de levadura, NaCl y KCl a 97 mL de agua destilada.
Disolver. Autoclavear y enfriar a temperatura ambiente. Agregar el Mg2+ 2M y la glucosa
2 M a una concentración final de 20 mM. Llevar a 100 mL con agua destilada estéril. El pH
deberá ser de 7.0
A16. Mg2+ 2M (stock)
20.33 g de MgCl2 * 6H2O
24.65 g de MgSO4 * 7H2O
Agregar agua a 100 mL. Esterilizar por filtración
Instituto Tecnológico de Sonora
67
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
ANEXO 2. FORMATO DE ENTREGA DE REPORTE DE PRÁCTICA
PARA EL ALUMNO
El reporte de cada práctica se entregará sin falta la clase siguiente después de
terminar la práctica. Se realizará un reporte por cada equipo. El reporte será
redactado en ARIAL 12 (títulos y subtítulos en negritas), sin faltas de ortografía,
interlineado 1.15 y con número de página (excepto en portada). El reporte será
estructurado de la siguiente manera:
a. Portada.
b. Palabras clave (5).
c. Introducción (1 cuartilla mínimo, describir las generalidades del tema
de la práctica).
d. Objetivos (¿qué, cómo, para qué?).
e. Materiales y métodos utilizados
f. Resultados (describir con gran detalle los resultados obtenidos e
interpretarlos comparándolos con lo encontrado en la literatura)
g. Conclusión
h. Bibliografía (Formato APA, mínimo 5 bibliografías de fuentes
CONFIABLES, mínimo 2 en inglés).
En el apartado de “Resultados” se deberá hacer el análisis y la descripción
fundamentada de los resultados obtenidos, basado en la información que se encuentre
en bibliografía (artículo científico, revista, libro, etc.). Formato de citas APA
(ejemplos):
Libro:
•
•
González, R. (2010). Introducción a la psicología contemporánea. San José, Costa
Rica: Editorial ULACIT.
González, R., y Ramírez, J.L. (2007). La teoría de los valores. México:
PrenticeHall.
Artículos en revistas:
•
•
Mora, H., y Domínguez, L.A. (2010). La psicología cognoscitiva y su relación con
las ciencias del cerebro. Actualidades en Ciencias Cognoscitivas, 14, 330- 337.
González, R. (2012). Teorías contemporáneas del aprendizaje. Revista
Costarricense de Psicología, 7(2), 24-31.
Instituto Tecnológico de Sonora
68
Manual de Prácticas de Laboratorio de Biología Molecular
IB plan 2016
REFERENCIAS
Abascal F., Aguirre J, Andres-Leon, E., Bajic D., Bau D., Bueren-Calabuig J. A., CortesCabrera A., Dotu I., Fernandez J. M., Santos H. G. D., Garcia-Jimenez B., Guantes
R., Irisarri I., Jimenez-Lozano N., Klett J., Mendez R., Morreale A., Pascual-Garcia
A, Perona A., Sebastian A., Stich M., Tarazona S., Yruela I., Zardoya R. (2014).
Bioinformática con ñ. Libro autoeditado e impreso por CreateSpace.
Arber, W., & Linn, S. (1969). DNA modification and restriction. Annual Review of
Biochemistry, 38, 467-500.
Cox, R. A. (1968). Methods in Enzymology. In L. Grossmann & E. Moldave (Eds.), (Vol.
12, pp. 120-129). Orlando, FL.: Academic Press.
González M., A., Alvarez Y., A. C., López A., D., Quintero, A., Orozco A., S., Guyot, R.,
Cristancho A., M. A., Zuluaga, M., Botero, K., Mosquera R., J., Garavito, A.
(2017). Una aproximación conceptual a las ciencias ómicas. Manizales, Colombia:
Editorial Blanecolor S.A.S.
Helling, R. B., Goodman, H. M., & Boyer, H. W. (1974). Analysis of endonuclease
R·EcoRI fragments of DNA from lambdoid bacteriophages and other viruses by
agarose-gel electrophoresis. Journal of Virology, 14(5), 1235-1244.
Lee, P. Y., Costumbrado, J., Hsu, C.-Y., & Kim, Y. H. (2012). Agarose Gel Electrophoresis
for the Separation of DNA Fragments. (62), e3923. doi:doi:10.3791/3923
Pray, L. (2008). Restriction enzymes. Nature Education, 1(1), 38.
Raeder, U., & Broda, P. (1985). Rapid preparation of DNA from filamentous fungi. Letters
in Applied Microbiology, 1, 17-20.
Sambrook, J., & Russell, D. W. (2001). Molecular Cloning, (3 ed.).
Instituto Tecnológico de Sonora
69