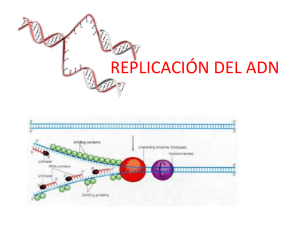
ColágenoQueratina Elastina.Trombina Ovoalbúmina Ferritina Mucoproteínas Insulina Hemocianina Clase 5 del segundo parcial Replicación del ADN en células eucariotas Diferencias replicación células eucariotas y procariotas -La principal diferencia del genoma de las células procariotas es que es circular y de las células eucariotas es lineal -para explicar la replicación en células eucariotas se divide por fases del ciclo celular -en las células procariotas siempre las moléculas se encuentran activas y en las células eucariotas es necesario activar y desactivar moléculas -En células procariotas existe un único origen de replicación y en células eucariotas existen muchos orígenes de replicación -G1 (antes de las síntesis de la replicación, por ende, las polimerasas), preparación de la célula para la replicación -en la cadena existen dos sitios: un sitio que es rico en timina y adenina (en donde se van a unir las helicasas) y otro sitio en donde se unen las proteínas (del complejo ORC 1-6) (está conformado por 6 proteínas, ORC 1 a la 6) * en los gráficos lo que está pintado esta de forma activa y lo que está sin pintar de forma inactiva * organismos eucariotas inferiores como levaduras, el complejo ORC 1-6 detecta una secuencia específica de bases; pero en organismos superiores como en plantas, animales y humanos detecta solo estructuras tridimensionales en base a la condensación de la cromatina -por consiguiente, una vez ya establecido el origen de la replicación intervienen 2 proteínas que son la CDC6 y la CDT1 (análogas a las proteínas DNA tipo c, ayudan a que la helicasa se una) -después, de la unión del complejo ORC 1-6 este da una señal para que intervenga una proteína análoga a la helicasa (que también está conformada por 6 proteínas) el complejo mcm 2-7 -después, en el complejo mcm 2-7 se observa 2 proteínas análogas a la helicasa una a la derecha y otra a la izquierda -después, se forma el complejo el complejo de pre-replicación: en donde la proteína CDC-6 (estado activo) se une al complejo ORC 1-6 (estado activo) y la proteína CDT1 (estado activo) se une al complejo mcm 2-7 (en estado inactivo) *es importante que la mcm 2-7 este en estado inactivo porque si no directamente empezaría la síntesis de ADN, que en la fase G1 no se requiere (ya que en esta fase solo se prepara el material para poder posteriormente empezar la síntesis del ADN en la fase S) Fase S (síntesis) -aquí las enzimas más importantes son las cdk (ciclinas dependientes de quinasas) que van a fosforilar y desfosforilar elementos de algunas proteínas (lo que hace que se activen o desactiven las proteínas) -una vez que aumentan los niveles de cdk, esto da una señal a la célula para que entre en fase S -para que inicie la síntesis de ADN, las dos proteínas izquierda y derecha que forman el complejo mcm 27 tienen que activarse, por lo cual, se une una cdk en cada una y fosforila estas proteínas, en consecuencia, se activan -adicionalmente existen 2 moléculas que se unen al complejo mcm 2-7, la CDC45 y GINS (activas) y una vez se unen estas, se denomina complejo helicasa CMG (que va a romper los puentes de hidrogeno y separar las dos hebras) -Finalmente, la cdk fosforila el ORC 1-6 y se desactiva, adicionalmente otras cdk fosforilan las proteínas CDC6 y CDT1 y se desactivan *si el ORC 1-6 no se desactiva va a continuar reconociendo sitios de origen replicación y va a empezar una nueva síntesis de proteínas y también va a volver a llamar a formar el complejo helicasa CMG (y se va a sintetizar otra cadena de ADN adicional) que no es necesaria ya que solo se necesita 2 copias de ADN *-En células procariotas existe un único origen de replicación y en células eucariotas existen muchos orígenes de replicación, por ende, también muchas burbujas de replicación porque el genomas es más largo (de esta manera la replicación se hace de mas rápida) *la fase de síntesis se demora de 3-4 horas *los complejos ya vienen pegados en el ADN (solo esperan a ser fosforilados para iniciar la síntesis) Diferencia entre células procariotas y eucariotas en la elongación En elongación de las células procariotas (existen polimerasas I, II, Y III) y en células eucariotas existen 5 tipos de polimerasas: alfa, beta (reparación), delta (síntesis), ípsilon (síntesis), gama (es aparte porque se encarga de la replicación del material genético en las mitocondrias) Elongación -en esta fase es importante la actividad de las polimerasas y existen varias: alfa, beta (reparación), delta (síntesis), ípsilon (síntesis), gama (es aparte porque se encarga de la replicación del material genético en las mitocondrias). Cadena continua -Lo primero, que sucede es que la polimerasa alfa coloca el primer (o cebador) de bases complementarias y a partir de esto la misma polimerasa alfa (añade nucleótidos) y comienza a sintetizar la nueva cadena líder (continua) en sentido 5”—3” * la actividad que (en células procariotas esto realiza la primasa) en células eucariotas esto realiza la polimerasa alfa (que tiene actividad primasa y de síntesis) -después, de añadir 20 nucleótidos la polimerasa alfa se separa (y ya no puede seguir sintetizando) -después, viene otra polimerasa (delta o ípsilon) a continuar con la síntesis (generalmente en la hebra líder termina la síntesis la polimerasa ípsilon) y (en la hebra retardada termina la síntesis la polimerasa delta) -para que las polimerasas (ípsilon) se aseguren a la hebra líder hasta que se termine la síntesis, actúan dos proteínas: una externa RFC y otra interna PCNA (que forman una pinza) y de esta manera se logra añadir todos los nucleótidos hasta terminar la síntesis -además, el complejo helicasa CMG (va continuar rompiendo los puentes de hidrógeno y separando las cadenas) -además, interviene la topoisomerasa que corta 1 o 2 cadenas para desenredar (molécula de ADN) Cadena discontinua - -Lo primero, que sucede es que la polimerasa alfa coloca el primer (o cebador) de bases complementarias y a partir de esto la misma polimerasa alfa (añade nucleótidos) y comienza a sintetizar la nueva cadena (discontinua) en sentido 5”—3” --después, de añadir 20 nucleótidos la polimerasa alfa se separa (y ya no puede seguir sintetizando) --después, viene otra polimerasa (delta o ípsilon) a continuar con la síntesis (en la hebra retardada termina la síntesis la polimerasa delta) -para que las polimerasas (delta e ípsilon) se aseguren a la hebra retardada hasta que se termine la síntesis, actúan dos proteínas: una externa RFC y otra interna PCNA (que forman una pinza) y de esta manera se logra añadir los nucleótidos de los fragmentos de Okazaki - Nuevamente, a medida que se va abriendo la burbuja de replicación la polimerasa alfa va de nuevo colocando más primers (o cebadores) y sintetiza pocos nucleótidos y se separa, y después actúa la polimerasa delta a continuar con la síntesis - para que las polimerasas (delta e ípsilon) se aseguren a la hebra retardada hasta que se termine la síntesis, actúan dos proteínas: una externa RFC y otra interna PCNA (que forman una pinza) y de esta manera se logra añadir los nucleótidos de los fragmentos de Okazaki Semejanza entre células procariotas y eucariotas en la terminación Terminación en las células procariotas y eucariotas es igual Terminación -Una RNAasa elimina los primers (o cebadores) de ARN -Después, actúa la polimerasa delta (hebra retardada) o ípsilon (hebra líder) para sintetizar los fragmentos de ADN que faltan -Después, la ligasa va a unir los extremos 5” y 3” que quedaron separados