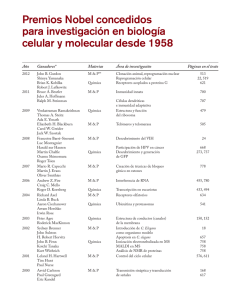
BIOQUIMICA Seminario Estructura y función de los Ácidos Nucleicos OBJETIVOS: Comprender la estructura química monomérica y polimérica del material genético, el ácido desoxirribonucleico o DNA (Deoxyribonucleic Acid), que se encuentra dentro del núcleo de las células eucariotas. Explicar por qué el DNA genómico es de doble cadena y con carga altamente negativa. Comprender el esquema de cómo se puede duplicar fielmente la información genética del DNA. Describir cómo la información genética del DNA se transcribe o se copia, en una infinidad de formas distintas de ácido ribonucleico (RNA, ribonucleic acid). Apreciar que una forma de RNA rico en información, el llamado RNA mensajero (mRNA, messenger RNA), puede traducirse posteriormente en proteínas, las moléculas que forman las estructuras, las formas y, en última instancia, las funciones de las células individuales, los tejidos y los órganos. DESARROLLO Realizar la lectura del material adjunto y complementarlo que consultas propias en diferentes fuentes para posteriormente discutir en una plenaria los siguientes tópicos: 123456789- ¿Cual es la importancia biomédica de los ácidos nucleicos? ¿De que manera está contenida la información genética en el ADN? ¿En que consiste la desnaturalización del ADN? ¿En que consiste la replicación del ADN? ¿En que consiste la Transcripción de la información genética? Desde el punto de vista químico, que diferencias existen entre ADN y ARN? ¿Cuáles son los tipos de ARN? ¿cuáles son las funciones de los diferentes tipos de ARN? ¿Qué son las nucleasas y cuales su función? C A P Í T U L O Estructura y función del ácido nucleico P. Anthony Weil, PhD OBJETIVOS: Después de estudiar este capítulo, usted deberá ser capaz de: ■ ■ ■ ■ ■ 34 Comprender la estructura química monomérica y polimérica del material genético, el ácido desoxirribonucleico o DNA (Deoxyribonucleic Acid), que se encuentra dentro del núcleo de las células eucariotas. Explicar por qué el DNA genómico es de doble cadena y con carga altamente negativa. Comprender el esquema de cómo se puede duplicar fielmente la información genética del DNA. Describir cómo la información genética del DNA se transcribe o se copia, en una infinidad de formas distintas de ácido ribonucleico (RNA, ribonucleic acid). Apreciar que una forma de RNA rico en información, el llamado RNA mensajero (mRNA, messenger RNA), puede traducirse posteriormente en proteínas, las moléculas que forman las estructuras, las formas y, en última instancia, las funciones de las células individuales, los tejidos y los órganos. IMPORTANCIA BIOMÉDICA El descubrimiento de que la información genética está codificada a lo largo de una molécula polimérica compuesta por sólo cuatro tipos de unidades monoméricas, fue uno de los principales logros científicos del siglo XX. Esta molécula polimérica, el ácido desoxirribonucleico (DNA), es la base química de la herencia y está organizada en genes, las unidades fundamentales de la información genética. Ha sido dilucidada la vía de información básica, es decir, el DNA, que dirige la síntesis del RNA, la cual a su vez dirige y regula la síntesis de proteínas. Los genes no funcionan de manera autónoma; más bien, su replicación y función están controladas por diversos productos génicos, a menudo en colaboración con componentes de varias vías de transducción de señales. El conocimiento de la estructura y función de los ácidos nucleicos es esencial para comprender la genética y muchos aspectos de la fisiopatología, así como la base genética de la enfermedad. EL DNA CONTIENE LA INFORMACIÓN GENÉTICA La demostración de que el DNA contenía la información genética, fue hecha por primera vez en 1944 en una serie de experimentos por Avery, MacLeod y McCarty. Mostraron que la determinación genética del carácter (tipo) de la cápsula de una bacteria neumocócica específica podría transmitirse a otro de diferente tipo capsular, introduciendo el DNA purificado del anterior neumococo en el último. Estos autores se refirieron al agente (que luego se demostró que era el DNA) que logró el cambio, como “factor transformador”. Posteriormente, este tipo de manipulación genética se ha converti- do en algo común. Experimentos conceptualmente similares se realizan de forma regular en la actualidad utilizando una variedad de células eucariotas, incluyendo las células humanas y los embriones de mamíferos como receptores y DNA clonado molecularmente como el donante de la información genética. El DNA contiene cuatro desoxinucleótidos La naturaleza química de las unidades desoxinucleótidas monoméricas de DNA —desoxiadenilato, desoxiguanilato, desoxicitidilato, y timidilato— se describe en el capítulo 32. Estas unidades monoméricas de DNA se mantienen en forma polimérica mediante enlaces 3',5'-fosfodiéster que constituyen una sola cadena, como se representa en la figura 34-1. El contenido informacional del DNA (el código genético) reside en la secuencia en que estos monómeros —desoxirribonucleótidos de purina y pirimidina— se ordenan. El polímero como se representa posee una polaridad; un extremo tiene un 5'-hidroxilo o fosfato terminal, mientras que el otro tiene un 3'-fosfato o hidroxilo terminal. La importancia de esta polaridad se hará evidente. Dado que la información genética reside en el orden de las unidades monoméricas dentro de los polímeros, debe existir un mecanismo de reproducción o replicación de esta información específica con un alto grado de fidelidad. Ese requisito, junto con los datos de difracción de rayos X de la molécula del DNA, generada por Franklin, y la observación de Chargaff de que en las moléculas del DNA la concentración de los nucleótidos de desoxiadenosina (A) es igual a la de los nucleótidos de timidina (T) (A = T), mientras que la concentración de los nucleótidos de desoxiguanosina(G) es igual a la de los nucleótidos de desoxicitidina (C) (G = C), condujeron a que Watson, Crick y Wilkins propusieran a principios de la década de 1950 un modelo de una molécula de DNA de CAPÍTULO 34 Estructura y función del ácido nucleico 339 O N G 5′ CH2 O– – O O N NH NH2 NH2 N O C P H H H O H CH2 H O– O N N O H3C O T P O O H H O H H O– N CH2 H O NH O N O A P O O NH2 H H H – H CH2 H O O N O P O O N N H H H H H O– O– P O 3′ O FIGURA 34-1 Un segmento de una cadena de una molécula de DNA, en la que las bases de purina y pirimidina, guanina (G), citosina (C), timina (T) y adenina (A) se mantienen juntas mediante un esqueleto de fosfodiéster entre las partes 2' desoxirribosilo unidas a las nucleobases mediante un enlace N-glucosídico. Tenga en cuenta que el esqueleto de fosfodiéster está cargado negativamente y tiene una polaridad (es decir, una dirección). La convención dicta que una secuencia de DNA de cadena simple se escribe en la dirección 5' a 3' (es decir, pGpCpTpAp, donde G, C, T y A representan las cuatro bases y p representa los fosfatos interconectados). doble cadena. El modelo que propusieron se muestra en la figura 34-2. Las dos cadenas de esta hélice de doble cadena se mantienen unidas mediante enlaces de hidrógeno entre las bases de purina y pirimidina de las respectivas moléculas lineales y por interacciones de Van der Waals hidrofóbicas entre los pares de bases adyacentes apiladas. Las coincidencias entre los nucleótidos de purina y pirimidina en las cadenas opuestas son muy específicos y dependen del enlace de hidrógeno de A con T y G con C (figura 34-2). Los pares de bases A-T y G-C a menudo se denominan pares de bases de Watson-Crick. Se dice que esta forma común de DNA es diestra porque cuando uno mira desde arriba hacia abajo de la doble hélice, los residuos de la base forman una espiral en el sentido de las agujas del reloj. En la molécula de doble cadena, las restricciones impuestas por la rotación sobre el enlace fosfodiéster, la favorecida anti-configuración del enlace glucosídico (véase figura 32-5) y los tautómeros predominantes (véase figura 32-2) de las cuatro bases (A, G, T y C) permiten que A se empareje sólo con T, y G sólo con C, como se representa en la figura 34-3. Estas restricciones de emparejamiento de bases explican la observación anterior de que en una molécula de DNA de cadena doble el contenido de A es igual al de T, y el contenido de G es igual al de C. Las dos cadenas de la molécula de doble hélice, cada una de las cuales posee una polaridad, son antiparalelas; es decir, una cadena corre en la dirección de 5'a 3' y la otra en la dirección de 3' a 5'. Dentro de un gen particular en las moléculas de DNA de doble cadena, la información genética reside en la secuencia de los nucleótidos en una cadena, la cadena plantilla. Esta es la cadena de DNA que se copia, o transcribe, durante la síntesis del ácido ribonucleico (RNA). A veces se denomina cadena no codificadora. La cadena opuesta se considera la cadena codificadora porque coincide con la secuencia del RNA transcrito (pero que contiene uracilo en lugar de timina, figura 34-8) que codifica la proteína. Las dos cadenas, en las que las bases opuestas se mantienen juntas mediante enlaces de hidrógeno entre las cadenas, giran alrededor de un eje central en forma de una doble hélice. En el tubo de ensayo, el DNA de doble cadena puede existir en al menos seis formas (A-E y Z). Estas diferentes formas del DNA difieren con respecto a las interacciones intra e inter cadenas e implican reordenamientos estructurales dentro de las unidades monoméricas del DNA. La forma B generalmente se encuentra bajo condiciones fisiológicas. Un solo giro de la forma B-DNA alrededor del eje largo de la molécula, contiene 10 pares de bases (bp). La distancia que abarca una vuelta de B-DNA es de 3.4 nm (34 Å). El ancho (diámetro helicoidal) de la doble hélice en el B-DNA es de 2 nm (20 Å). Como se muestra en la figura 34-3, tres enlaces de hidrógeno (véase figura 2-2), formados por hidrógeno unido a átomos electronegativos N u O, unen el nucleótido desoxiguanosina con el nucleótido desoxicitidina, mientras que el otro par, el par A-T, se mantiene unido por dos enlaces de hidrógeno. Obsérvese que las cuatro bases de nucleótidos de DNA, purinas ([dG, dA] y pirimidinas [dT, dC]; véanse figura 32-1 y cuadro 32-1] son planas, moléculas planas. Estas propiedades fundamentales de las bases de 340 SECCIÓN VII Estructura, función y replicación de macromoléculas informacionales Surco menor A T P S S P S T S A C P P o S G G S C 34 A P S P S Surco mayor La desnaturalización del DNA se usa para analizar su estructura o 20 A FIGURA 34-2 Una representación diagramática del modelo de Watson y Crick de la estructura de doble hélice de la forma B del DNA. La flecha horizontal indica el ancho de la doble hélice (20 Å), y la flecha vertical indica la distancia que abarca un giro completo de la doble hélice (34 Å). Un giro de B-DNA incluye 10 pares de bases (bp), por lo que el aumento es de 3.4 Å por bp. El eje central de la doble hélice está indicado por la barra vertical. Las flechas cortas designan la polaridad de las cadenas antiparalelas. Se representan las surcos mayor y menor. (A: adenina; C: citosina; G: guanina; P: fosfato; S: azúcar [desoxirribosa]; T: timina). Los enlaces de hidrógeno entre las bases A/T y G/C se indican mediante líneas horizontales cortas, rojas. CH3 O H N N N H H N N O Timina N N Adenina H N N H O N H O Citosina H N H FIGURA 34-3 nucleótidos les permiten apilarse estrechamente dentro del DNA doble (figura 34-2). Los átomos dentro de las bases heterocíclicas aromáticas son altamente polarizables, y, junto con el hecho de que muchos de los átomos dentro de las bases contienen cargas parciales, permite que las bases apiladas formen las interacciones electroestáticas y Van der Waals. Estas fuerzas se conocen colectivamente como fuerzas o interacciones de apilamiento de bases. Las interacciones de apilamiento de bases entre pares de bases adyacentes G-C (o C-G) son más fuertes que los pares de bases A-T (o T-A). Por tanto, las secuencias de DNA rico en G-C son más resistentes a la desnaturalización, o separación de cadenas, denominadas “fusión”, que las regiones de DNA ricos en A-T. N N N N Guanina El emparejamiento clásico de bases del DNA de Watson-Crick entre desoxinucleótidos complementarios implica la formación de enlaces de hidrógeno. Dos de tales enlaces H se forman entre la adenina y la timina, y se forman tres enlaces H entre la citidina y la guanina. Las líneas discontinuas representan los enlaces H. La estructura de doble cadena del DNA se puede separar en dos cadenas componentes en solución, incrementando la temperatura o disminuyendo la concentración de sal. Las dos pilas de bases no sólo se separan, sino que las bases mismas se desapilan mientras están aún conectadas con el polímero por el esqueleto de fosfodiéster. Concomitante con la desnaturalización de la molécula de DNA, está el aumento en la absorbancia óptica de las bases de purina y pirimidina, un fenómeno denominado hipercromicidad de desnaturalización. Debido al apilamiento de las bases y el enlace de hidrógeno entre las pilas, la doble molécula de DNA exhibe propiedades de una varilla rígida, y en solución es un material viscoso que pierde su viscosidad tras la desnaturalización. Las cadenas de una molécula de DNA dada se separan en un rango de temperatura. El punto medio se llama temperatura de fusión, o Tm. La Tm está influenciada por la composición de base del DNA y por la concentración de sal (u otros solutos, véase a continuación) de la solución. El DNA rico en pares G-C, que tienen tres enlaces de hidrógeno, se funde a una temperatura más alta que el DNA rico en pares A-T, que tienen dos enlaces de hidrógeno. Un aumento de 10 veces la concentración de catión monovalente aumenta la Tm en 16.6 °C al neutralizar la repulsión intercadena intrínseca entre los fosfatos con carga altamente negativa del esqueleto de fosfodiéster. Por el contrario, el solvente orgánico formamida, que se usa comúnmente en experimentos de DNA recombinante, desestabiliza el enlace de hidrógeno entre las bases, disminuyendo así la Tm. Además, la formamida permite que las cadenas de DNA o los híbridos DNA-RNA se separen a temperaturas mucho más bajas y minimiza la ruptura del enlace fosfodiéster y el daño químico a los nucleótidos, que pueden ocurrir tras la incubación prolongada a temperaturas más altas. La renaturalización del DNA requiere el emparejamiento de pares de bases Es importante destacar que las cadenas separadas de DNA se renaturalizarán o reasociarán, cuando se alcancen las condiciones fisiológicas apropiadas de temperatura y sal; este proceso de reasociación a menudo se denomina hibridación. La tasa de reasociación depende de la concentración de las cadenas complementarias. La reasociación de las dos cadenas complementarias del DNA de un cromosoma después de la transcripción, es un ejemplo fisiológico de renaturalización (véase a continuación). A una temperatura y concentración de sal dadas, una cadena particular de ácido nucleico se asociará estrechamente sólo con una cadena complementa- CAPÍTULO 34 ria. Las moléculas híbridas también se formarán en condiciones apropiadas. Por ejemplo, el DNA formará un híbrido con un DNA complementario (cDNA, complemetary DNA) o con un RNA complementario afin (p. ej., mRNA, véase a continuación). Cuando la hibridación se combina con técnicas de electroforesis en gel que separan los ácidos nucleicos por tamaño, junto con el marcaje de sonda complementaria radiactiva o fluorescente para proporcionar una señal detectable, las técnicas analíticas resultantes se denominan electrotransferencia Southern (DNA/DNA) y Northern (RNA-DNA), respectivamente. Estos procedimientos permiten la identificación muy clara y de alta sensibilidad de especies de ácidos nucleicos específicas a partir de mezclas complejas de DNA o RNA (véase capítulo 39). Existen surcos en la molécula del DNA El análisis del modelo representado en la figura 34-2 revela un surco mayor y un surco menor enrollado a lo largo de la molécula paralela a los esqueletos de fosfodiéster. En estos surcos, las proteínas a menudo interactúan específicamente con los átomos expuestos de los nucleótidos (a través de interacciones hidrófobas e iónicas específicas), reconociendo y uniéndose así a secuencias de nucleótidos específicas, así como a las formas únicas formadas a partir de ellas. La unión generalmente ocurre sin interrumpir el emparejamiento de bases de la molécula de DNA de doble hélice. Como se discutió en los capítulos 35, 36 y 38, las proteínas reguladoras que controlan la replicación, reparación y recombinación del DNA, así como la transcripción de genes específicos ocurren a través de tales interacciones proteína-DNA. El DNA existe en formas relajadas y superenrolladas. En algunos organismos, como bacterias, bacteriófagos, muchos virus de animales que contienen DNA, así como en organelos como las mitocondrias (véase figura 35-8), los extremos de las moléculas de DNA se unen para crear un círculo cerrado con extremos no covalentemente libres. Esto, por supuesto, no destruye la polaridad de las moléculas, pero elimina todos los grupos fosforilo e hidroxilo 3' y 5' libres. Los círculos cerrados existen en formas relajadas o superenrolladas. Las superenrolladas se presentan cuando un círculo cerrado se retuerce alrededor de su propio eje, o cuando una pieza lineal de DNA doble, cuyos extremos son fijos, se tuerce. Este proceso que requiere energía pone a la molécula bajo tensión de torsión, y cuanto mayor es el número de superenrollados, mayor es el estrés o la torsión (prueba esto al retorcer una goma elástica). Los superenrollados negativos se forman cuando la molécula se tuerce en la dirección opuesta al sentido de las agujas del reloj de la doble hélice derecha que se encuentra en el B-DNA. Se dice que dicho DNA está infraenrollado. La energía requerida para lograr este estado está, en cierto sentido, almacenada en los superenrollados. La transición a otra forma que requiere energía se ve facilitada por el infraenrollamiento (véase figura 35-19). Una de esas transiciones es la separación de cadenas, que es un requisito previo para la replicación y la transcripción del DNA. El DNA superenrollado es por tanto una forma preferida en los sistemas biológicos. Las enzimas que catalizan los cambios topológicos del DNA se denominan topoisomerasas. Las topoisomerasas pueden relajar o insertar superenrollados, usando ATP como una fuente de energía. Los homólogos de esta enzima existen en todos los organismos y son Estructura y función del ácido nucleico 341 blancos importantes para la quimioterapia contra el cáncer. Los superenrollados también pueden formarse dentro de los DNA lineales, si segmentos particulares de DNA son forzados a interactuar estrechamente con proteínas nucleares que establecen dos sitios límites que definen un dominio topológico. EL DNA PROPORCIONA UNA PLANTILLA PARA LA REPLICACIÓN Y LA TRANSCRIPCIÓN La información genética almacenada en la secuencia de nucleótidos del DNA tiene dos propósitos. Es la fuente de información para la síntesis de todas las moléculas de las proteínas de la célula y el organismo, y proporciona la información heredada por las células hijas o la descendencia. Ambas de estas funciones requieren que la molécula de DNA sirva como plantilla, en el primer caso para la transcripción de la información en RNA y en el segundo caso para la replicación de la información en moléculas de DNA hijas. Cuando cada cadena de la molécula de DNA de doble cadena original se separa de su complemento durante la replicación, cada una sirve independientemente como una plantilla sobre el que se sintetiza una nueva cadena complementaria (figura 34-4). Las dos moléculas hijas de DNA de doble cadena recién formadas, cada una con una cadena (pero complementaria más que idéntica) a partir de la molécula de DNA de doble cadena original, son divididas entre las dos células hijas durante la mitosis (figura 34-5). Cada célula hija contiene moléculas de DNA con información idéntica a la que poseía la original; sin embargo, en cada célula hija, la molécula de DNA de la célula original ha sido sólo semiconservada. LA NATURALEZA QUÍMICA DEL RNA DIFIERE DE LA DEL DNA El RNA es un polímero de ribonucleótidos de purina y pirimidina unidos por enlaces 3',5'-fosfodiéster análogos a los del DNA (figura 34-6). Aunque comparte muchas características con el DNA, el RNA posee varias diferencias específicas: 1. En el RNA, la parte del azúcar al que están unidos los fosfatos y las bases de purina y pirimidina es la ribosa en lugar de la 2'-desoxirribosa del DNA (véanse figuras 19-2 y 32-3). 2. Los componentes de pirimidina del RNA pueden diferir de los del DNA. Aunque el RNA contiene los ribonucleótidos de adenina, guanina y citosina, no posee timina, excepto en el raro caso mencionado a continuación. En lugar de timina, el RNA contiene el ribonucleótido de uracilo. 3. El RNA típicamente existe como una sola cadena, mientras que el DNA existe como una molécula helicoidal de doble cadena. Sin embargo, dada la adecuada secuencia de bases complementarias con polaridad opuesta, la única cadena de RNA —como se muestra en las figuras 34-7 y 34-11— es capaz de replegarse sobre sí misma como una horquilla y así adquirir características de doble cadena: emparejándose G con C y A con U. 4. Dado que la molécula de RNA es una cadena única complementaria de sólo una de las dos cadenas de un gen, su contenido de guanina no es necesariamente igual a su contenido de 342 SECCIÓN VII Estructura, función y replicación de macromoléculas informacionales VIEJA 5′ G C G VIEJA 3′ C C Molécula madre original G A T A A T G C G C A A T T G G T C 3′ T A C A G C A C 5′ G C Primera generación de moléculas hijas A C A T A T T T T A T A G C C G G A Segunda generación de moléculas hijas A G T A T FIGURA 34-5 FIGURA 34-4 La síntesis del DNA mantiene la secuencia y la estructura del modelo original del DNA. La estructura del DNA de cadena doble y la función de una plantilla de cada cadena parental vieja (naranja) sobre la que se sintetiza una nueva cadena hija complementaria (azul). La replicación del DNA es semiconservativa. Durante una ronda de replicación, cada una de las dos cadenas del DNA se usa como una plantilla para la síntesis de una nueva cadena complementaria. La naturaleza semiconservativa de la replicación del DNA tiene implicaciones para el control bioquímico (véase figura 35-16), citogenético (véase figura 35-2) y epigenético de la expresión génica (véanse figuras 38-8 y 38-9). citosina, ni su contenido de adenina es necesariamente igual a su contenido de uracilo. que reemplaza a T) es la misma que la de la cadena codificadora del gen (figura 34-8). 3′ VIEJA T T A A 5′ NUEVA T 3′ NUEVA T A A 5′ VIEJA 5. El RNA se puede hidrolizar mediante álcali a diésteres cíclicos 2', 3' de los mononucleótidos, compuestos que no pueden formarse a partir del DNA tratado con el álcali debido a la ausencia de un grupo 2'-hidroxilo. La labilidad alcalina del RNA es útil tanto diagnóstica como analíticamente. La información dentro de la única cadena de RNA está contenida en su secuencia (“estructura primaria”) de nucleótidos de purina y pirimidina dentro del polímero. La secuencia es complementaria a la cadena plantilla del gen del cual fue transcrito. Debido a esta complementariedad, una molécula de RNA puede unirse específicamente mediante las reglas de emparejamiento de bases a su cadena de DNA plantilla (A-T, G-C, C-G, U-A, la base de RNA en negrita); no se unirá (“hibridará”) con la otra cadena (codificadora) de su gen. La secuencia de la molécula de RNA (excepto para U CASI TODAS LAS VARIADAS ESPECIES DE RNA ABUNDANTES ESTABLES ESTÁN INVOLUCRADAS EN ALGUNOS ASPECTOS DE LA SÍNTESIS DE PROTEÍNA Aquellas moléculas de RNA citoplasmáticas que sirven como unas plantillas para la síntesis de proteínas (es decir, que transfieren información genética desde el DNA a la maquinaria que sintetiza proteínas) se denominan RNAs mensajeros (mRNAs). Muchas otras moléculas de RNA citoplasmático muy abundantes (RNA ribosómico [rRNAs, ribosomal RNA]) tienen roles estructurales que CAPÍTULO 34 Estructura y función del ácido nucleico 343 O N NH G CH2 5′ P O N NH2 NH2 N N O H H C H CH2 H O HO N O O O P H O H U H O HO O NH2 N O P O N CH2 H NH H H A H CH2 H O HO N O P O N N H H H H HO P 3′ O FIGURA 34-6 Un segmento de una molécula de ácido ribonucleico (RNA), en la cual las bases de la purina y la pirimidina —guanina (G), citosina (C), uracilo (U), y adenina (A)— se apoyan juntas por los enlaces de fosfodiéster entre las partes deribosil unidos a las nucleobases por enlaces N-glucosídico. Nótese que la(s) carga(s) negativa(s) en el esqueleto fosfodiéster no están ilustradas (es decir, figura 34-1) y que el polímero tiene una polaridad como se indica por los fosfatos unidos a 3′ y 5′ etiquetados. Asa C C G A A A U U C G U U U U U C G 5 FIGURA 34-7 G G C U U U G G C C A A C A G G C Tallo 3 Representación diagramática de la estructura secundaria de una molécula de RNA de cadena simple, en la cual un tallo con asa, u “horquilla,” ha sido formada. La formación de esta estructura es dependiente de la pareja base intramolecular indicada (las bases coloreadas entre líneas horizontales). Nótese que el par G con C es como en el DNA, pero en el RNA el par A se empareja con U a través de los enlaces de hidrógeno. contribuyen a la formación y función de los ribosomas (el organelo maquinaria para la síntesis de proteínas) o sirven como moléculas adaptadoras (RNA de transferencia [tRNAs, transfer RNA]) para la traducción de información de RNA en secuencias específicas de aminoácidos polimerizados. Curiosamente, algunas moléculas de RNA tienen actividad catalítica intrínseca. La actividad de estas enzimas de RNA, o ribozimas, a menudo implica la escisión de un ácido nucleico. Dos ribozimas son, la peptidil transferasa que cataliza la formación del enlace peptídico en el ribosoma, y las ribozimas implicadas en el empalme del RNA. En todas las células eucariotas, hay especies nucleares pequeñas de RNA (snRNA, small nuclear RNA) que no están directamente involucradas en la síntesis de proteínas, pero desempeñan una función fundamental en el procesamiento del RNA, particularmente en el procesamiento del mRNA. Estas moléculas relativamente pequeñas varían en tamaño desde 90 hasta aproximadamente 300 nucleótidos (cuadro 34-1). Las propiedades de las diversas clases de RNA celulares se detallan a continuación. El material genético para algunos virus de animales y vegetales es el RNA en lugar del DNA. Aunque algunos virus de RNA nunca transcriben su información en una molécula de DNA, muchos virus de RNA en los animales –específicamente, los retrovirus (p. ej., el de la inmunodeficiencia humana, o virus (HIV, human immunodeficiency virus), se transcriben por la DNA polimerasa dependiente de RNA viral, la llamada transcriptasa inversa, para producir una copia de DNA de doble cadena de su genoma de RNA. En muchos casos, la transcripción de DNA de doble cadena resultante se integra en el genoma del huésped y, posteriormente, sirve como una plantilla para la expresión génica y a partir del cual se pueden transcribir nuevos genomas de RNA viral y mRNA virales. La in- 344 SECCIÓN VII Estructura, función y replicación de macromoléculas informacionales Cadenas de DNA: Codificación Plantilla 5′ 3′ T GGA A T T G T GAGCGGA T A A C A A T T T C A C A C AGGA A A C AGC T A T GA CC A T G A CC T T A A C A C T CGCC T A T T G T T A A AG T G T G T CC T T T G T CGA T A C T GG T A C 3′ 5′ RNA transcrito: 5′ ppp A U U G U G A G C G G A U A A C A A U U U C A C A C A G G A A A C A G C U A U G A C C A U G 3′ FIGURA 34-8 La relación entre las secuencias de un RNA transcrito y su gen, en el que las cadenas de codificación y modelo se muestran con sus polaridades. El RNA transcrito con una polaridad de 5' a 3' es complementario a la cadena plantilla con su polaridad de 3' a 5'. Tenga en cuenta que la secuencia en la transcripción de RNA y su polaridad es la misma que en la cadena de codificación, excepto que la U de la transcripción reemplaza la T del gen; el nucleótido iniciador de los RNA contiene un terminal 5-trifosfato (es decir, pppA-arriba). serción genómica de tal integración de moléculas de DNA “províricas”, dependiendo del sitio involucrado pueden ser mutagénicas, inactivar un gen o desregular su expresión (véase figura 35-11). EXISTEN VARIAS CLASES DISTINTAS DE RNA Como se señaló anteriormente, en todos los organismos procariotas y eucariotas, existen cuatro clases principales de moléculas de RNA: mRNA, tRNA, rRNA, y pequeños RNA. Cada uno difiere de los demás por la abundancia, tamaño, función y estabilidad general. RNA mensajero Esta clase es la más heterogénea en abundancia, tamaño y estabilidad; por ejemplo, en la levadura de la cerveza, los mRNA específicos están presentes en 100 s/célula, en promedio, ≤0.1/mRNA/ célula en una población genéticamente homogénea. Como se detalla en los capítulos 36 y 38, los mecanismos transcripcionales y postranscripcionales específicos contribuyen a este amplio rango dinámico en el contenido del mRNA. En las células de los mamíferos, la abundancia específica de mRNA probablemente varía en un rango de 104 veces. Todos los miembros de esta clase de RNA fun- cionan como mensajeros que transmiten la información en un gen a la maquinaria sintetizadora de proteínas, donde cada mRNA sirve como una plantilla sobre el que se polimeriza una secuencia específica de aminoácidos para formar una molécula de proteína específica, el producto génico final (figura 34-9). Los mRNA eucarióticos tienen características químicas únicas. El terminal 5' del mRNA es “cubierto” por un 7-metilguanosina trifosfato que está unido a un 2'-O-metil ribonucleósido adyacente a su 5'-hidroxilo a través de tres fosfatos (figura 34-10). Las moléculas de mRNA con frecuencia contienen 6-metiladenina interna y otros nucleótidos metilados 2'-O-ribosa. La cubierta está involucrada en el reconocimiento del mRNA por la maquinaria de traducción, y también ayuda a estabilizar el mRNA al prevenir el ataque nucleolítico por la 5'-exorribonucleasas. La maquinaria sintetizadora de proteínas empieza a traducir al mRNA en proteínas comenzando corriente abajo del terminal 5' o casquetado. En el otro extremo de casi todas las moléculas de mRNA en eucariotas, el terminal 3'-hidroxilo tiene un adjunto, un polímero de residuos de adenilato de 20 a 250 nucleótidos de longitud no genéticamente codificado. La “cola” poli (A) en el terminal 3' de los mRNA mantiene la estabilidad intracelular del mRNA específico evitando el ataque de las 3'-exorribonucleasas y también facilita la traducción (véase figura 37-7). Tanto la cubierta como la “cola poli (A)” se añaden después DNA 5′ 3′ CUADRO 34-1 Algunas de las especies de RNA pequeños y estables que se encuentran en las células de los mamíferos Longitud (nucleótidos) Moléculas por células U1 165 1 × 106 Nucleoplasma U2 188 5 × 105 Nucleoplasma U3 216 3 × 105 Nucléolo 139 5 Nucleoplasma 5 Nucleoplasma 5 Gránulos de pericromatina Nombre U4 U5 118 1 × 10 2 × 10 106 3 × 10 4.5S 95 3 × 105 Núcleo y citoplasma 5 Núcleo y citoplasma 7SK 280 5 × 10 mRNA 5′ Localización U6 3′ 5′ 3′ Síntesis de proteína sobre una plantilla de mRNA 3′ 5′ Ribosoma N N N N C Molécula de N proteína completa FIGURA 34-9 Se muestra la expresión de información genética dentro del DNA en la forma de un transcrito de mRNA con una polaridad de 5' a 3' y luego en una proteína con polaridad de N a C. El DNA se transcribe en mRNA que posteriormente se traduce por los ribosomas en una molécula de proteína específica que exhibe polaridad, N-terminal (N) a C-terminal (C). CAPÍTULO 34 HC OH OH C C H H Estructura y función del ácido nucleico 345 CH O N H2N N O– H2C 5′ NH2 O P HN N O O N O P O O– CH3 O P O 5′ CH2 O CAP N O– N N O HC H H 3′ C C CH 2′ O OCH3 O Terminal 5′ mRNA O O NH 5′ CH2 P O– N O O HC H H 3′ C C CH OH O O P O– FIGURA 34-10 La estructura de la cubierta fija unida al terminal 5' de la mayoría de las moléculas de RNA mensajero eucariotas. Una 7-metilguanosina trifosfato (negro) se une en el extremo 5' del mRNA (rojo), que generalmente también contiene un nucleótido 2'-O-metilpurina. Estas modificaciones (la cubierta y el grupo metilo) se agregan después de que el mRNA se transcribe a partir del DNA. Nótese que los γ y β fosfatos del GTP (guanosine triphosphate): trifosfato de guanosina, añadido para formar la cubierta (negra en la figura) se pierden al añadir la cubierta, mientras que el γ fosfato del nucleótido iniciador (aquí un residuo A, rojo en la figura) se pierde durante la cubierta adicional. de la transcripción mediante enzimas no dirigidas por plantillas a moléculas precursoras de mRNA (pre-mRNA). El mRNA representa de 2 a 5% del RNA celular eucariótico total. En las células de mamíferos, incluidas las células de los humanos, las moléculas de mRNA presentes en el citoplasma no son los productos de RNA inmediatamente sintetizados a partir de una plantilla de DNA, sino que deben formarse mediante el procesamiento del precursor, o pre-mRNA antes de entrar en el citoplasma. Por tanto, en los núcleos de las células de los mamíferos, los productos inmediatos de la transcripción génica (transcritos primarios) son muy heterogéneos y pueden ser de 10 a 50 veces más largos que las moléculas de mRNA maduras. Como se discutió en el capítulo 36, las moléculas de pre-mRNA se procesan para generar moléculas de mRNA, que luego ingresan al citoplasma para servir como unas plantillas para la síntesis de proteínas. RNA de transferencia Las moléculas de tRNA varían en longitud desde 74 a 95 nucleótidos, como muchos otros RNA, también se generan mediante el 346 SECCIÓN VII Estructura, función y replicación de macromoléculas informacionales procesamiento nuclear de una molécula precursora (véase capítulo 36). Las moléculas de tRNA sirven como adaptadores para la traducción de la información en la secuencia de nucleótidos del mRNA de aminoácidos específicos. Existen al menos 20 especies de moléculas de tRNA en cada célula, al menos una (y a menudo varias) correspondientes a cada uno de los 20 aminoácidos necesarios para la síntesis de proteínas. Aunque cada tRNA específico difiere de los demás en su secuencia de nucleótidos, las moléculas de tRNA como clase tienen muchas características en común. La estructura primaria —es decir, la secuencia de nucleótidos— de todas las moléculas de tRNA permite un extenso plegamiento y una complementariedad intracadena para generar una estructura secundaria que aparece en dos dimensiones, como una hoja de trébol (figura 34-11). Todas las moléculas de tRNA contienen cuatro brazos o tallos principales de doble cadena, conectados por asas de una cadena, llamados así por su respectiva composición o función de nucleótidos. El brazo aceptor termina en los nucleótidos CpCpAOH. Estos tres nucleótidos se agregan después de la transcripción por una enzima específica nucleotidil transferasa. El aminoácido tRNA apropiado se fija, o “carga”, en el grupo 3'-OH del resto A del brazo aceptor mediante la acción de aminoacil tRANA sintetasas específicas (véase figura 37-1). Los D, TΨC, y brazos extra ayudan a definir un tRNA específico. Los tRNA componen aproximadamente 20% del RNA celular total. RNA ribosómico Un ribosoma es una estructura de nucleoproteína citoplásmica que actúa como la maquinaria para la síntesis de proteínas a partir de unas plantillas de mRNA. En los ribosomas, las moléculas de mRNA y tRNA interactúan para traducir la información transcrita del gen durante la síntesis del mRNA en una proteína específica. Durante los periodos de la síntesis de proteína activa, muchos ribosomas pueden estar asociados con cualquier molécula de mRNA para formar un ensamblaje llamado polisoma (véase figura 37-7). Los componentes del ribosoma en los mamíferos, que tiene un peso molecular de aproximadamente 4.2 × 106 y un coeficiente de velocidad de sedimentación de 80S (S = unidades Svedberg, un parámetro sensible del tamaño y forma molecular) se muestran en el cuadro 34-2. El ribosoma de los mamíferos contiene dos subunidades principales de nucleoproteína, una más grande con un peso molecular de 2.8 × 106 (60S) y una subunidad más pequeña con un peso molecular de 1.4 × 106 (40S). La subunidad 60S contiene un rRNA de 5S, un rRNA de 5.8S y un rRNA de 28S; también hay más de 50 polipéptidos específicos. La subunidad 40S es más pequeña y contiene un único rRNA 18S y aproximadamente 30 cadenas polipeptídicas distintas. Todas las moléculas de rRNA, excepto el rRNA 5S, que se transcribe independientemente, se procesan a partir de una única molécula precursora de RNA de 45S en el nucléolo (véase capítulo 36). Las moléculas de rRNA altamente metiladas se empaquetan en el nucléolo con las proteínas ribosómicas específicas. En el citoplasma, los ribosomas permanecen bastante estables y capaces de muchos ciclos de traducción. Las funciones exactas de las moléculas de rRNA en la partícula ribosómica no se conocen por completo, pero son necesarias para el ensamblaje ribosómico y también desempeñan una función clave en la unión del mRNA a los ribosomas y su traducción. Estudios recientes indican que el componente grande del rRNA realiza la actividad peptidil transferasa y, por tanto, es una ribozima. Los rRNA (28S + 18S) representan aproximadamente 70% del RNA celular total. RNA pequeño Una gran cantidad de pequeñas especies de RNA discretas, y muy conservadas se encuentran en las células eucariotas; algunos son bastante estables. La mayoría de estas moléculas están compuestas con proteínas para formar ribonucleoproteínas y se distribuyen en el núcleo, el citoplasma, o ambos. Varían en tamaño de 20 a 1 000 nucleótidos y están presentes en 100 000 a 1 000 000 de copias por célula, colectivamente representan ≤5% de RNA celular. RNA pequeños nucleares Los snRNA, un subconjunto de los RNA pequeños nucleares (cuadro 34-1), están significativamente implicados en el procesamiento del rRNA y mRNA y la regulación génica. De los varios snRNA, el U1, U2, U4, U5 y U6 están involucrados en la eliminación del intrón y el procesamiento de precursores de mRNA en mRNA dentro del núcleo (véase capítulo 36). El snRNA U7 está involucrado en la producción de los extremos 3' correctos de la histona del mRNA, que carece de una cola de poli (A). El RNA 7SK se asocia con varias proteínas para formar una ribonucleoproteína compleja, denominada P-TEFb, que modula la elongación de la transcripción del gen de mRNA por la RNA polimerasa II (véase capítulo 36). Los RNAs reguladores no codificantes grandes y pequeños: Los micro-RNA (miRNAs, micro-RNAs), los RNA silenciadores (siRNAs, silencing RNAs), los RNA largos no codificadores (lncRNAs, long noncoding RNAs) y los RNA circulares (circRNAs, circular RNAs) Uno de los descubrimientos más interesantes y no anticipados en la última década de la biología reguladora eucariota ha sido la identificación y caracterización de los RNA reguladores no codificantes de proteínas (ncRNAs, nonprotein coding RNAs). Los ncRNA existen en dos clases generales de tamaño, grande (50-1 000nt) y pequeña (20-22nt). Los ncRNA reguladores se han descritos en la mayoría de los eucariotas (véase capítulo 38). Los ncRNA pequeños denominados miRNA y siRNA típicamente inhiben la expresión génica a nivel de la producción de una proteína específica por un mRNA blanco a través de uno de los variados mecanismos diferentes. Los miRNA se generan por procesamiento nucleolítico específico de los productos de distintas unidades genes/transcripción (véase figura 36-17). Los precursores de miRNA, que están 5'-casquetados y 3'-poliadenilados, generalmente varían en tamaño aproximadamente de 500 hasta 1 000 nucleótidos. Por el contrario, los siRNA se generan por el procesamiento nucleolítico específico de grandes dsRNA que se producen de otros RNA endógenos, o dsRNA introducidos en la célula por virus RNA, por ejemplo. Tanto los siRNA como los miRNA se hibridan mediante la formación de hibridación RNA-RNA con sus mRNA blancos (véase figura 38-19). Hasta la fecha, se han descrito cientos de miRNA y siRNA distintos en los humanos; las estimaciones sugieren que hay ~ 1 000 genes que codifican miRNA humanos. Dada su exquisita especificidad genética, tanto los miRNA como los siRNA representan nuevos y excitantes agentes potenciales para el desarrollo de fármacos terapéuticos. Los siRNA se usan con frecuencia para disminuir o “noquear” concentraciones de proteínas específicas (por medio de degradación de mRNA dirigida hacia homología de siRNA) en contextos experimentales en el laboratorio, una herramienta extremadamente útil y poderosa a la tecnología Asa anticodón U Gm A C 5 mC U G 7 50 U GUG m G G G U 5 mC40 ψ A Y A A C C A G30 A Cm A D G A C U C m2G10 D G G A G C2 G20 m2G G A 30 G C Asa variable Tψ 40 50 60 70 Estructura terciaria (3°) o o Estructura de un tRNA maduro y funcional, fenilalanil-tRNA de levadura (tRNAPhe, phenylalanyl-tRNA). Se muestran las estructuras primaria 1o), secundaria 2 ) y terciaria 3 ) (superior, inferior izquierda e inferior derecha, respectivamente) de tRNAPhe. Los números debajo de la estructura primaria prolongada de tRNAPhe de 76 nucleótidos indican la numeración de nucleótidos desde el extremo 5' (+1) al terminal 3' (+76) de la molécula. Obsérvese que el nucleótido +1 contiene un resto 5' fosfato (P), mientras que el nucleótido 3' tiene un grupo libre 3' hidroxilo (OH). Las bases subrayadas en negrita dentro de la secuencia de tRNAPhe modifican fuertemente a los nucleótidos que se muestran en la representación estructural 2o de tRNAPhe. Esta estructura a menudo se denomina como “hoja de trébol”. Algunos de estos nucleótidos tienen nombres de ribonucleótidos no canónicos, como se representa en el modelo estructural 2o. Dentro de tRNAPhe, los nucleótidos U16 y U17 se modifican a D16, D17; G37 a Y37; U39 y U55 a Ψ; y U54 a T54 (véase texto a continuación para más detalles). Las líneas rectas entre las bases dentro de la estructura secundaria del tRNA representan los enlaces de hidrógeno formados entre las bases (A-U; G-C). Nótese que estas regiones de estructura secundaria se forman con la misma polaridad de cadena (es decir, 5' a 3' y 3' a 5') como regiones del DNA emparejadas por pares. Las tres bases del asa del anticodón se muestran en rojo. En el caso de los tRNA cargados con aminoácidos, una parte aminoacil se esterifica al terminal 3'-CCAOH (carmelita, en este caso el aminoácido sería la fenilalanina, no se muestra). El tipo azul resalta los nucleótidos no tradicionales introducidos por modificación postraduccional, abreviados de la siguiente manera: m2G = 2-metilguanosina; D = 5,6-dihidrouridina; m22 G = N2- dimetilguanosina; Cm = O2'-metilcitidina; Gm = O2'-metilguanosina; T = 5-metiluridina; Y = wybutosina; Ψ = pseudouridina; m5C = 5-metilcitidina; m7G = 7-metilguanosina; m1A = 1-metiladenosina. Esencialmente, todos los tRNA se pliegan en estructuras terciarias 3o) con características similares como se muestra, debajo a la derecha. Las porciones distintas de la molécula en 2o (inserción) y configuraciones de 3o están codificadas por colores en esta imagen para mayor claridad. El tRNAPhe fue el primer ácido nucleico cuya estructura se determinó mediante cristalografía de rayos X. Dichas estructuras de tRNA tridimensionales distintas se unen específicamente a sitios funcionales importantes tanto en la aminoacil tRNA sintetasas como en los ribosomas durante la síntesis de proteínas (véase capítulo 37). Las imágenes de estructura secundaria y terciaria de tRNAPhe son figuras de código abierto de Wikipedia (Wikipedia.org/Wikipedia/commons/b/ba/ TRNA_Phe_yeast_1ehz.png). FIGURA 34-11 D-asa 5’ pG1 C G G A U U U 20 3’ A -OH C C A Aminoácido Brazo aceptor/tallo C G C70 U U Tψ C asa A A C60U 1 G AC A C mA Estructura secundaria (2°) 10 5′ PGCGGAUUUAGCUCAGUUGGGAGAGCGCCAGACUGAAGAUCUGGAGGUCCUGUGUUCGAUCCACAGAAUUCGCACCAOH 3’ Estructura primaria (1°) CAPÍTULO 34 Estructura y función del ácido nucleico 347 348 SECCIÓN VII Estructura, función y replicación de macromoléculas informacionales CUADRO 34-2 Componentes de los ribosomas de los mamíferos Proteína Componente Subunidad 40S Subunidad 60S Masa (MW) 6 1.4 × 10 2.8 × 10 6 Número 33 50 RNA Masa Tamaño 5 7 × 10 1 × 10 18S 6 Masa Bases 5 7 × 10 1 900 4 3.5 × 10 120 5.8S 4.5 × 104 160 28S 1.6 × 106 4 700 5S Nota: Las subunidades ribosomales se definen según su velocidad de sedimentación en unidades Svedberg (S) (40S o 60S). Se detallan el número de proteínas únicas y su masa total (MW) y los componentes del RNA de cada subunidad en tamaño (unidades de Svedberg), masa y número de bases. de bloqueo de genes (véase capítulo 39). De hecho, varios ensayos clínicos terapéuticos basados en siRNA están en progreso para probar la eficacia de estas nuevas moléculas como fármacos para tratar enfermedades humanas. Otras recientes observaciones interesantes en el ámbito del RNA son la identificación y caracterización de dos clases de RNA no codificadoras más grandes, los RNA circulares (circRNAs) y los RNA largos que no codifican, o lncRNA. Muchos circRNA han sido recientemente descubiertos y caracterizados. Los circRNA parecen producirse por reacciones tipo empalme de RNA a partir de una amplia gama de precursores de RNA, tanto precursores de mRNA como precursores de lncRNA no proteico (véase a continuación para más información sobre los lncRNA). Aunque no es una clase abundante de moléculas de RNA en la mayoría de las células, se han detectado circRNA en todos los eucariotas analizados, particularmente en los metazoos. Mientras las funciones de los circRNA todavía se están elucidando, parecen ser particularmente abundantes en las células del sistema nervioso. De forma similar a los lncRNA, estas moléculas probablemente desempeñan una función importante en la biología celular mediante la regulación de la expresión génica en múltiples niveles. Los lncRNA, que como su nombre lo indica, no codifican proteínas, y varían en tamaño desde ≈300 a 1 000 s de nucleótidos en longitud. Estos RNA se transcriben típicamente a partir de las grandes regiones de genomas eucarióticos que no codifican para proteína (es decir, los genes que codifican mRNA). De hecho, los análisis de los transcriptomas indican que >90% de todo el DNA genómico eucariota se transcribe. Los ncRNA constituyen una parte importante de esta transcripción. Los ncRNA desempeñan muchas funciones que van desde la contribución a los aspectos estructurales de la cromatina hasta la regulación de la transcripción del gen de mRNA por la RNA polimerasa II. El trabajo futuro caracterizará aún más a esta clase de moléculas de RNA importantes, recién descubiertas. Curiosamente, las bacterias también contienen RNA reguladores pequeños y heterogéneos denominados sRNA. Los sRNA bacterianos varían en tamaño de 50 a 500 nucleótidos, y de forma similara los mi/si/lncRNA eucariotas, también controlan una gran variedad de genes. Los sRNA a menudo reprimen, pero a veces activan la síntesis de proteínas uniéndose a un mRNA específico. LAS NUCLEASAS ESPECÍFICAS DIGIEREN ÁCIDOS NUCLEICOS Se han reconocido durante muchos años las enzimas capaces de degradar los ácidos nucleicos. Estas nucleasas se pueden clasificar en varias formas. Aquellas que exhiben especificidad por el DNA se denominan desoxirribonucleasas. Aquellas nucleasas que hidrolizan específicamente el RNA son ribonucleasas. Algunas nucleasas degradan tanto el DNA como el RNA. Dentro de estas dos clases hay enzimas capaces de escindir enlaces fosfodiéster internos para producir terminales 3'-hidroxilo y 5'-fosforilo o terminales 5'-hidroxilo y 3'-fosforilo. Estas se conocen como endonucleasas. Algunas son capaces de hidrolizar ambas cadenas de una molécula de doble cadena, mientras que otros sólo pueden cortar cadenas simples de ácidos nucleicos. Algunas nucleasas pueden hidrolizar sólo cadenas únicas no apareadas, mientras que otras son capaces de hidrolizar cadenas simples que participan en la formación de una molécula de doble cadena. Existen clases de endonucleasas que reconocen secuencias específicas en el DNA. Una clase de estas enzimas de escisión de DNA, las endonucleasas de restricción, también denominadas enzimas de restricción, lo hacen directamente uniendo pares de bases de DNA contiguos (típicamente 4, 5, 6 u 8 bp) y escindiendo ambas cadenas de DNA, generalmente el DNA dentro del elemento secuencia de reconocimiento/unión. La segunda clase de enzimas, que son ribonucleoproteínas complejas, utiliza un “RNA guía” de una secuencia de nucleótidos específica que tiene a una nucleasa como blanco para escindir secuencias distintas de DNA o RNA. Estas son las familias de enzimas CRISPR-Cas. Ambas clases de enzimas que escinden el DNA se describen con mayor detalle en el capítulo 39. Estas enzimas representan herramientas críticamente importantes en genética molecular y ciencias médicas. Algunas nucleasas son capaces de hidrolizar un nucleótido sólo cuando está presente en un terminal de una molécula; estas se conocen como exonucleasas. Las exonucleasas actúan en una dirección (3 '→ 5' o 5 '→ 3') solamente. En las bacterias, una exonucleasa 3 '→ 5' es una parte integral de la maquinaria de replicación del DNA y sirve para editar —o corregir— el desoxinucleótido agregado más recientemente por errores de emparejamiento de bases. RESUMEN ■ El DNA consiste en cuatro bases —A, G, C y T— que se mantienen en un orden lineal mediante enlaces fosfodiéster a través de las posiciones 3' y 5' de las partes de desoxirribosa adyacentes. El DNA se organiza en dos cadenas mediante el emparejamiento de las bases A con T y G con C en cadenas complementarias. Estas cadenas forman una doble hélice alrededor de un eje central. ■ Las ~3 × 109bp de DNA en los humanos están organizadas en el complemento haploide de 23 cromosomas. La secuencia exacta de estos 3 mil millones de nucleótidos define la singularidad de cada individuo. ■ CAPÍTULO 34 El DNA proporciona una plantilla para su propia replicación y, por tanto, el mantenimiento del genotipo y para la transcripción de aproximadamente 25 000 genes humanos que codifican proteínas, así como una gran variedad de ncRNA reguladores que no codifican proteínas. ■ El RNA existe en muchas estructuras diferentes de una sola cadena, la mayoría de las cuales están directa o indirectamente implicadas en la síntesis de proteínas o su regulación. El conjunto lineal de nucleótidos en el RNA consiste en A, G, C y U, y el resto de azúcar es ribosa. ■ Las principales formas de RNA incluyen al mRNA, rRNA, tRNA y snRNA y los ncRNA reguladores. Ciertas moléculas de RNA actúan como catalizadores (ribozimas). ■ REFERENCIAS Cech TR, Steitz JA. The noncoding RNA revolution-trashing old rules to forge new ones. Cell 2014;157:77. Estructura y función del ácido nucleico 349 Mayerle M, Guthrie C. Genetics and biochemistry remain essential in the structural era of the spliceosome. Methods 2017;125:3 Muruhan K, Babu K, Sundaresan R, et al. The revolution continues: newly discovered systems expand the CRISPR-Cas toolkit. Molecular Cell 2017;68:15. Noller HF. The parable of the caveman and the Ferrari: protein synthesis and the RNA world. Philos Trans R Soc Lond B Biol Sci 2017;372(1716):20160187. Rich A, Zhang S. Timeline: Z-DNA: the long road to biological function. Nat Rev Genet 2003;4:566. Salzman J. Circular RNA expression: its potential regulation and function. Trends Genet 2016;32:309. Watson JD, Crick FH. Molecular structure of nucleic acids: a structure for deoxyribose nucleic acid. Nature 1953;171:737.