Universidad Nacional de Ingeniería - Facultad de Ingeniería Industria y de Sistemas
Sección de Post Grado. Doctorado en Ingeniería Industrial. Curso: Programación Matemática.
Pedro C. Espinoza H.
3. RNA NO SUPERVISADAS (KOHONEN O SOM)
Introducción
La sigla SOM viene del ingles: Self-Organizing Feature Maps o brevemente SelfOrganizing-Maps. El aprendizaje de este tipo de RNA es el no supervisado.
3.1 La topología de capas de neuronas
Las neuronas en la capa de un SOM pueden ser representadas por puntos o posiciones de un
plano bidimensional o plano coordenado XY, formando los vértices de un grafo, donde los
vértices o nodos, son las posiciones de las neuronas y las aristas o arcos son los enlaces entre
estos. Los grafos adoptan diversas formas o geometrías, denominadas Topologías. En
MATLAB existen funciones que ayudan a crear estas Topologías: gridtop, hextop o randtop,
estas tres funciones presentan a las neuronas en forma de una malla rectangular, hexagonal,
o en forma de un grafo aleatoria, respectivamente.
a) La topología gridtop
Crea una sucesión de puntos del plano XY, que son las posiciones de las neuronas y que son
los vértices de una malla o rejilla rectangular.
Ejemplo 1
p=gridtop(4,3)
p =
0
0
1
0
2
0
3
0
0
1
1
1
2
1
3
1
0
2
1
2
2
2
3
2
Aquí la entrada(4,3)significa que se trata de una malla con 3 filas y 4 columnas de
posiciones, como puede apreciarse en el gráfico adjunto generado con:
plotsom(p) % grafo en forma de malla rectangular
Fig.1
Neuron Positions
2
1.5
position(2,i)
El orden en que aparecen las posiciones
de p en la malla, es de abajo hacia arriba
y de izquierda a derecha. En la primera
fila debajo de la malla, están (0,0),
B(1,0), C(2,0) y (3,0). En la segunda
fila están E(0,1) etc.
Nota: para un gridtop(3,4) , con los
argumentos invertidos, se tiene un
arreglo diferente y por ende un grafo
de 4 fila con 3 columnas de vértices.
1
0.5
b) La topología hextop
Con esta función se crean las posiciones
de las neuronas, que son los vértices de un
grafo, pero que tienen un patrón hexagonal.
Ejemplo 2
0
0
0.5
1
1.5
position(1,i)
2
2.5
q=hextop(4,3)
q =
0 1.0000 2.0000 3.0000 0.5000 1.5000 2.5000 3.5000
0 0
0
0
0.8660 0.8660 0.8660 0.8660
107
0
1.0000 2.0000 3.0000
1.7321 1.7321 1.7321 1.7321
3
Universidad Nacional de Ingeniería - Facultad de Ingeniería Industria y de Sistemas
Sección de Post Grado. Doctorado en Ingeniería Industrial. Curso: Programación Matemática.
Pedro C. Espinoza H.
plotsom(q)
Fig.2
Neuron Positions
2
1.5
position(2,i)
Las posiciones de las neuronas están
en q,el orden en que aparecen en el
grafo, es de abajo hacia arriba y de
izquierda a derecha.
Nota hextop es el patrón por defecto
para las redes SOM generado con
newsom.
1
0.5
c) La topología randtop
Crea grafos para una capa de
0
neuronas con un patrón aleatorio, es
decir los vértices del grafo ocupan
-0.5
posiciones aleatorias del plano XY,
0
las aristas en forma aleatoria. El
código siguiente genera un grafo con
nodos aleatorios (posiciones de las neuronas).
0.5
1
1.5
2
position(1,i)
2.5
3
3.5
Ejemplo 3
Para crear y graficar una capa de 8x10 neuronas, en una topología de randtop se utiliza el
siguiente código:
pos3 = randtop(8,10); % matriz de 2 filas y 80 columnas= 80 puntos en XY
plotsom(pos3)
% grafo aleatorio de 80 vértices o puntos
Fig. 3
Neuron Positions
6
5
position(2,i)
4
3
2
1
0
0
1
2
3
position(1,i)
4
5
6
3.2 Las métricas o nociones de distancia entre neuronas
La noción de distancia más común entre dos puntos de un espacio es la denominada distancia
“euclideana” que es la longitud del segmento de recta que une estos puntos. Desde el punto
de vista matemático, existen infinitas nociones de distancia, que se definen a continuación.
Pero estas nociones de distancia se basan en la noción de norma o longitud de un vector,
108
Universidad Nacional de Ingeniería - Facultad de Ingeniería Industria y de Sistemas
Sección de Post Grado. Doctorado en Ingeniería Industrial. Curso: Programación Matemática.
Pedro C. Espinoza H.
puesto que dos puntos definen un vector. Entonces se pasará a definir una clase de normas,
donde algunas de ellas se utilizarán en las nociones de distancia para redes neuronales SOM.
Definición
Si x = ( x1 , x2 ,......, xN ) es un vector de n , la longitud o norma de este vector, en el
sentido de los L p (Minkowski), se denota y define como:
1/ p
x
p
N
= | xi | p
i =1
donde 1 p es cualquier número real.
Propiedades de la norma o longitud L p de un vector
a) x
b) x
p
p
c) x
0
=0 x=0
p
d) x + y
= x
p
x
p
p
+ y
p
La distancia o métrica L p entre dos vectores x = ( x1 , x2 ,......, xn ) y = ( y1 , y2 ,......, yn ) se
denota y define como d ( x, y ) = x − y
p
Propiedades
a) d ( x, y ) 0
b) d ( x, y ) = 0 x = y
c) d ( x, z ) d ( x, y ) + d ( y, z ) desigualdad triangular
Casos especiales de las métricas L p
◊ Cuando p = 1 la distancia se llama de Mahattan y coincide con la distancia que existe
entre dos puntos de una ciudad cuyas calles forman una malla rectangular.
◊ Cuando p = 2 , se tiene la distancia Euclideana, que es la longitud del segmento que une
estos puntos. Este es único caso en que la norma provienen de un producto interno. Como se
recordará (sección 2.5 del capítulo 1) el producto interno o escalar de dos vectores
x = ( x1 , x2 ,......, xn ) y = ( y1 , y2 ,......, yn ) se denota y define como el número
x • y = x1 y1 + x2 y2 + ...... + xn yn
◊ La distancia L (Chebychev). Este límite es igual a la distancia del máximo y se define
como sigue:
Si x = ( x1 , x2 ,......, xN ) e y = ( y1 , y2 ,......, y N ) son dos vectores de dimensión N , entonces
la distancia L entre estos dos vectores viene a ser
d ( x, y) = x − y = max | xi − yi |
i =1, 2,.. N
109
Universidad Nacional de Ingeniería - Facultad de Ingeniería Industria y de Sistemas
Sección de Post Grado. Doctorado en Ingeniería Industrial. Curso: Programación Matemática.
Pedro C. Espinoza H.
Ejemplo
Fig.1
Sean los puntos 𝑝 = (2,2) y 𝑞 = (6,5).
Entonces la distancia entre 𝑝 y 𝑞:
a) La Euclideana es 5
b) La de Manhatan es 4+3=7
c) Del máximo es 4
La noción de distancia entre las neuronas viene mayormente de la noción de distancia
entre vectores que se ha resumido al inicio de esta sección, sin embargo hay otras que
vienen de otro tipo de conceptos, como la del enlace. Hay cuatro funciones de distancia
que emplea MATLAB: dist, boxdist, linkdist y mandist, que son las distancias:
euclideana, del máximo, del enlace y de Mahattan respectivamente.
Como hay tres topologías: gridtop, hextop o randtop podemos hacer combinaciones de
estas con la cuatro distancias, por ejemplo a la topología gridtop asociarle la distancia
eucideana con dist, es decir hacer un plotsom(gridtop,dist), otro podría ser un
plotsom(hexatop,mandist) etc.
Fig. 2
a) gridtop y dist
Esta función de distancia es la euclidiana.
Ejemplo 1
Retomemos el ejemplo 1 de la sección anterior, esto
es:
>> p = gridtop(2,3)
p =
0
0
1
0
0
1
1
1
0
2
1
2
plotsom(p)
Esta topología proporciona 6 neuronas, con sus
posiciones en el plano coordenado XY que son los
vértices o nodos de una malla rectangular. (Fig. 4)
La lectura de los nodos es de abajo hacia arriba y de izquierda a derecha. El primer nodo es
A(0,0), el siguiente es B(1,0) y así sucesivamente. Esto se resume en la siguiente:
Tabla 1
A
(0,0)
B
(1,0)
C
(0,1)
D
(1,1)
E
(0,2)
F
(1,2)
Para esta topología, la matriz de distancias entre las 6 neuronas, con la distancia euclideana
dist, es una matriz de 6x6 que tiene la siguiente forma:
A
B
A
B
d(A, A) d(A, B)
d(B, A) d(B, B)
C
d(A, C)
d(B, C)
D
d(A, D)
d(B, D)
110
E
d(A, E)
d(B, E)
F
d(A, F)
d(B, F)
Universidad Nacional de Ingeniería - Facultad de Ingeniería Industria y de Sistemas
Sección de Post Grado. Doctorado en Ingeniería Industrial. Curso: Programación Matemática.
Pedro C. Espinoza H.
C
D
E
F
d(C, A)
d(D, A)
d(E, A)
d(F, A)
d(C, B)
d(D, B)
d(E, B)
d(F, B)
d(C, C)
d(D, C)
d(E, C)
d(F, C)
d(C, D)
d(D, D)
d(E, D)
d(F, D)
d(C, E)
d(D, E)
d(E, E)
d(F, E)
d(C, F)
d(D, F)
d(E, F)
d(F, F)
En efecto, en la topología de la malla rectangular, la distancia euclideana entre A(0,0) y
D(1,1) es d(A, D)=||(1,1)-(0,0)||= 2^(0.5)=1.414, la distancia de B(1,0) a F(1,2) es d(B,
F)=||(1,2)-(1,0)||=4^(0.5)=2 etc.
Esta matriz de distancias se obtiene con la función:
dp = dist(p)
dp =
0
1.0000
1.0000
1.4142
2.0000
2.2361
1.0000
0
1.4142
1.0000
2.2361
2.0000
1.0000
1.4142
0
1.0000
1.0000
1.4142
1.4142
1.0000
1.0000
0
1.4142
1.0000
2.0000
2.2361
1.0000
1.4142
0
1.0000
2.2361
2.0000
1.4142
1.0000
1.0000
0
b) hextop y linkdist
Ejemplo 2
r = hextop(3,3);
r=
0 1.0000 2.0000 0.5000 1.5000 2.5000
0 0
0
0.8660 0.8660 0.8660
A
B
0
0
C
1
0
D
E
F
0
1.0000 2.0000
1.7321 1.7321 1.7321
G
H
I
2
0.5
1.5
2.5
0
1
2
0 0.866 0.866 0.866 1.732 1.732 1.732
plotsom(r)
Neuron Positions
linkdist(r)
1.8
1.6
1
0
1
1
1
2
2
2
2
2
1
0
2
1
1
3
2
2
1
1
2
0
1
2
1
1
2
2
1
1
1
0
1
2
1
1
3
2
1
2
1
0
3
2
1
2
2
3
1
2
3
0
1
2
2
2
2
1
1
2
1
0
1
3
2
2
2
1
1
2
1
0
1.4
1.2
position(2,i)
dr =
dr =
0
1
2
1
2
3
2
2
3
Fig. 3
1
0.8
F
0.6
0.4
0.2
A
0
0
0.5
1
1.5
position(1,i)
2
2.5
Por ejemplo la distancia con linkdist entre A(0,0) y F(2.5,0.866), es d(A,F)=1+1+1=3
c) randtop y mandist
Fig. 4
111
Universidad Nacional de Ingeniería - Facultad de Ingeniería Industria y de Sistemas
Sección de Post Grado. Doctorado en Ingeniería Industrial. Curso: Programación Matemática.
Pedro C. Espinoza H.
Ejemplo 4
Neuron Positions
1.5
La matriz rm de las posiciones de 6 neuronas en la
topología aleatoria randtop se obtiene haciendo:
rm =
0.2221 1.0272 0.3006 1.2519
0
0
1.5809 1.4460
0.0511 0.6531 0.7598
1
position(2,i)
rm=randtop(2,3)
0.8621
0.5
plotsom(rm)
la distancia de Mahttan, entre todas las columnas de la
0
0
0.5
position(1,i)
1
matriz r es
drm = mandist(rm)
drm=
0
0.8563
0.7317
1.7897
1.8030
2.0861
0.8563
0
1.3287
0.9334
2.5571
1.5600
0.7317
1.3287
0
1.0580
1.2284
1.3544
1.7897
0.9334
1.0580
0
2.0730
1.0760
1.8030
2.5571
1.2284
2.0730
0
0.9971
2.0861
1.5600
1.3544
1.0760
0.9971
0
Observando las columnas de drm y la topología randtop se verifica que la distancia de
Manhattan mandist de la neurona A(0.2221,0) a la neurona D(1.2519, 0.7598) es d(A,
D)=||(1.2519, 0.7598)-(0.2221,0)||=1.0298+0.7598 =1.7896
3.3 Vecindad o esfera de una neurona
Dada una topología T y una distancia d, la vecindad o esfera de radio r, de una neurona i*,
se denota N(i*, r) y se define como el conjunto de todas las neuronas i que están en T a una
distancia menor o igual que r , brevemente:
Fig. 1
N(i*, r) = { i en T, tal que d(i, i*)<= r }
Weight Vectors
4
Para ilustrar el concepto de vecindad, considere la siguiente
figura, que es la distribución de las neuronas con gridtop,
W(i,2)
es decir con la topología de la malla rectangular y con la
3
2
distancia de Manhattan: mandist.
1
0
112
0
1
2
W(i,1)
3
4
Universidad Nacional de Ingeniería - Facultad de Ingeniería Industria y de Sistemas
Sección de Post Grado. Doctorado en Ingeniería Industrial. Curso: Programación Matemática.
Pedro C. Espinoza H.
El diagrama adjunto muestra una vecindad bidimensional de radio r = 1 y con centro la
neurona (2,2). Esto se ha logrado con los códigos:
Q = gridtop(5,5);%
Fig.2
D = mandist(Q);
Weight Vectors
plotsom(Q',D);
hold on
V1=[ 2 2 1 3 2;1 2 2 2 3];
D1= mandist(V1);
plot(V1(1,:),V1(2,:),'x','markersize',20);
plot(V1(1,:),V1(2,:),'o','markersize',20);
4
Weight Vectors
W(i,2) W(i,2)
4
3
El diagrama de la Fig. 2 muestra la vecindad de la neurona
(2,2) con la misma noción de distancia, pero de radio r = 2.
3
2
2
1
1
0
Esta se consigue agregando a lo anterior los códigos:
0
V2=[ 2 2 1 3 2 2 3 4 1 0 1 2 3 4;
1 2 2 2 3 0 1 2 1 2 3 4 3 2];
plot(V2(1,:),V2(2,:),'x','markersize',20);
plot(V2(1,:),V2(2,:),'o','markersize',20);
0
1
0
1
2
W(i,1)
2
W(i,1)
3
4
3
4
Fig. 3
Q = gridtop(5,5);
D = mandist(Q);
V3=[2 2 1 3 2 3 1 1 3;1 2 2 2 3 1 1 3 3
];
plotsom(Q',D);;
hold on
Weight Vectors
4.5
4
3.5
3
2.5
W(i,2)
d) El gráfico muestra una neurona central en una capa
de neuronas bidimensionales (gridtop). La neurona
central de coordenadas (2,2) , tiene una vecindad de
radio 1, con la distancia boxdist. Por ejemplo la neurona
de coordenadas (1,3), está a la distancia 1 por que el
máximo entre |2-1| y |2-3| es 1. La gráfica se ha
logrado con los códigos:
2
1.5
1
0.5
0
-0.5
0
plot(V3(1,:),V3(2,:),'x','markersize',20);
plot(V3(1,:),V3(2,:),'o','markersize',20);
1
2
W(i,1)
3
4
Esta vecindad de radio 1 incluye la neurona central y sus vecinos inmediatos.
3.4 La arquitectura
Esta arquitectura no emplea sesgos, lo demás es como la de una red cualquiera, con la
salvedad de que sus funciones de transferencia son como las de las RNA competitivas.
113
Universidad Nacional de Ingeniería - Facultad de Ingeniería Industria y de Sistemas
Sección de Post Grado. Doctorado en Ingeniería Industrial. Curso: Programación Matemática.
Pedro C. Espinoza H.
Se puede elegir entre varias topologías para desarrollar los grafos de las capas de neuronas
y también las diversas distancias para calcular las neuronas que están cerca de la neurona
ganadora.
3.5 Creación de una red selforgmap (“newsom” en la versión Matlab 2010)
Sintaxis: se crea esta red neuronal con la función
selforgmap(dimensions, coverSteps, initNeighbor, topologyFcn, distanceFcn)
donde los parámetros tienen el siguiente significado:
dimensions=número de neuronas de la capa (puede ser [d1, d2], d1xd2 neuronas)
coverSteps= Número de pasos de entrenamiento. (Por defecto=200)
initNeighbor= Tamaño inicial de la vecindad. (Por defecto =3)
topologyFcn= describe la topología. (Por defecto toma la topología hextop)
distanceFcn= describe la distancia. (Por defecto toma la distancia linkdist)
Ejemplo 1
Se tiene los siguientes datos de entrada:
P=[5 5 4 6 12 11 18 17 1 3 3 4 12 11 18 17
5 4 6 5 3 1 3 2 18 18 16 18 19 19 17 18];
Es una matriz P de 2 filas y 16 columnas.
Supongamos que se desea trabajar con 6 neuronas, con la topología hextop, la distancia
linkdist y los demás parámetros por defecto. En este caso simple el código es:
net = selforgmap([2,3]);
Todos los parámetros que ingresan a la función selforgmap, con excepción de la
dimensión, se completan por defecto: coverSteps=200; initNeighbor=3;
topologyFcn=hextop; distanceFcn=linkdist.
3.6 El entrenamiento de la red neuronal
La neurona ganadora y el algoritmo de Kohonen.
Cuando ingresa un vector p a la red neuronal net, lo que ocurre es que se identifica una
neurona ganadora i* y se actualizan los pesos de todas las neuronas que se encuentran
dentro de una determinada vecindad N(i*, r) de la neurona ganadora i*.
Si w = [ w1 , w2 ,......., wN ] es la matriz de pesos de una de la neuronas “ i ” en la iteración k ,
entonces en la iteración (k + 1) la actualización de estos valores para cada neurona de la
vecindad N (i*, r ) = { i T : d (i, i*) r} se realiza mediante el siguiente algoritmo:
114
Universidad Nacional de Ingeniería - Facultad de Ingeniería Industria y de Sistemas
Sección de Post Grado. Doctorado en Ingeniería Industrial. Curso: Programación Matemática.
Pedro C. Espinoza H.
w = w + ( p'− w) donde 0 1
[ p − w]
En consecuencia, después de muchas iteraciones las
neuronas de una vecindad N(i*, r) tendrán pesos similares
p
w( k + 1)
entre sí.
w(k )
Continuando con el ejemplo 1 del parágrafo 3.5. El entrenamiento de la red se da con 200
iteraciones:
net=train(net,P);
luego de correr, sale una interface con 6 botones:
SOM Topology
2
1
0
-1
-1
0
1
2
El botón SOM Topology, genera una gráfica que muestra la topología de la red neuronal.
115
Universidad Nacional de Ingeniería - Facultad de Ingeniería Industria y de Sistemas
Sección de Post Grado. Doctorado en Ingeniería Industrial. Curso: Programación Matemática.
Pedro C. Espinoza H.
SOM Neighbor Connections
2.5
2
1.5
1
0.5
0
-0.5
-1
-1
0
1
2
En el gráfico Hits se observa 6 clústeres. Haciendo la lectura de abajo hacia arriba y de
izquierda a derecha, ellos son: 1, 2, 3, 4, 5 y 6. Los números que aparecen en cada hexágono
indican el número de elementos que tiene cada clúster. Así en el clúster 1 hay 2; en el 2 hay
2, el clúster 3 es vacío. Así sucesivamente.
b) Los pesos o coordenadas de las neuronas (luego del entrenamiento)
La gráfica de los agrupamientos (SOM Weight Positions) salen con mayor nitidez,
mediante el siguiente código:
plot(P(1,:),P(2,:),'.b','markersize',20); hold on
plotsom(net.iw{1,1},net.layers{1}.distances);
hold on
grid
116
Universidad Nacional de Ingeniería - Facultad de Ingeniería Industria y de Sistemas
Sección de Post Grado. Doctorado en Ingeniería Industrial. Curso: Programación Matemática.
Pedro C. Espinoza H.
Esta gráfica muestra aproximadamente las coordenadas de las neuronas (puntos rojos) en el
plano XY. Pero con el siguiente programa se muestra exactamente las posiciones (pesos) de
cada neurona.
Cluster=1:1:6;
[net.IW{1}, Cluster']
ans =
x
17.5000
12.0000
10.9167
2.7500
14.5000
5.0000
y
clúster
17.5000 1.0000
19.0000 2.0000
10.5000 3.0000
17.5000 4.0000
2.2500 5.0000
5.0000 6.0000
3.7 Los Clúster y sus miembros
En esta parte se verá exactamente qué miembros de la data de ingreso P pertenecen a uno u
otro clúster. Se consiguen con el siguiente código:
distances = dist(P',net.IW{1}');
[d,cndx] = min(distances,[],2);
Con este se genera una tabla, donde las dos primeras columnas son las coordenadas de los
puntos y la tercera el número del clúster al que pertenece el punto:
TABLA=[P',cndx]
5
5
4
6
12
11
18
17
5
4
6
5
3
1
3
2
6
6
6
6
5
5
5
5
117
Universidad Nacional de Ingeniería - Facultad de Ingeniería Industria y de Sistemas
Sección de Post Grado. Doctorado en Ingeniería Industrial. Curso: Programación Matemática.
Pedro C. Espinoza H.
1
3
3
4
12
11
18
17
18
18
16
18
19
19
17
18
4
4
4
4
2
2
1
1
4. Agrupamiento de una base de datos con 4 variables
4.1 La base de datos
La base de datos que se utilizará en esta oportunidad, se obtiene activando en Matlab:
load fisheriris;
Luego de activado aparecen dos matrices, una denominada “species”, que es una matriz
columna de 150 componentes, formada por caracteres, en las 50 primeras componentes se
repite la palabra Setosa, en las 50 siguientes Versicolor y las 50 últimas Verginica. La
segunda matriz es “meas” que es de 150x4, donde las 50 primeras filas son los registros de
las medidas del ancho y largo del sépalo y del pétalo de cada flor de la especie Setosa, las
50 filas siguientes de Versicolor y las 50 últimas de Verginica.
Con la finalidad de tener un control de cada una de las 3 especies, le asignaremos un código
a cada una de ellas. A Setosa el número 1, a Versicolor 2 y a Vergínica 3. Entonces la matriz
Esp=[ones(50,1); 2*ones(50,1); 3*ones(50,1)];
sustituye perfectamente a la matriz de las especies. Pero la matriz que ingresará a la red
neuronal será solo la transpuesta de “meas”, esto es: P=meas';
4.2 Creando la red neuronal SOM y su entrenamiento
Se creará una red neuronal SOM eligiendo la dimensión, la topología y la distancia de la
función “selforgmap” que tiene por sintaxis:
selforgmap(dimensions, coverSteps, initNeighbor,topologyFcn,distanceFcn)
La red neuronal SOM es
net=selforgmap([2 2], 200, 3,'topologyFcn','gridtop','distanceFcn','mandist');
El entrenamiento
P=meas';
net=train(net,P);
118
Universidad Nacional de Ingeniería - Facultad de Ingeniería Industria y de Sistemas
Sección de Post Grado. Doctorado en Ingeniería Industrial. Curso: Programación Matemática.
Pedro C. Espinoza H.
SOM Neighbor Connections
2
1.5
1
0.5
0
-0.5
-1
-1
-0.5
0
0.5
1
1.5
2
2.5
Los pesos de las neuronas (luego del entrenamiento)
net.IW{1}
5.0093
6.9760
5.5400
6.2857
3.4140
3.1120
2.6400
2.8976
1.4674
5.8840
3.9840
4.8929
0.2535
2.1680
1.2360
1.6619
119
Universidad Nacional de Ingeniería - Facultad de Ingeniería Industria y de Sistemas
Sección de Post Grado. Doctorado en Ingeniería Industrial. Curso: Programación Matemática.
Pedro C. Espinoza H.
4.3 Los clústeres y sus miembros
En esta parte se verá exactamente qué miembros de los datos de ingreso P=meas' son los que
conforman los clústeres. Según el gráfico Hits, se observa que hay 4 clústeres. Haciendo la
lectura de abajo hacia arriba y de izquierda a derecha, ellos son los clústeres: 1, 2, 3 y 4. El
clúster 1 tiene 27 miembros, el 2 tiene 50, el 3 tiene 45 y 4 tienen 28 elementos.
Pero los miembros concretos de estos clústeres se consiguen con el siguiente código:
distances = dist(P',net.IW{1}');
[d,cndx] = min(distances,[],2);
cndx es una matriz columna, con igual número de filas que P' que indica el clúster al que
pertenece cada punto (fila) de P. Para esto se tiene la siguiente tabla:
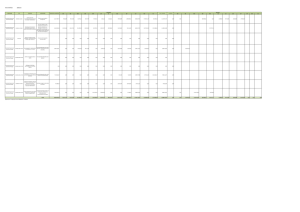
TABLA=[Esp, P', cndx]
La tabla se pasa al Excel, se ordena según los clústeres, luego se ordena en cada clúster
según las especies, de este modo se sabe cuántas especies hay en cada clúster. El resultado
se muestra en el cuadro que sigue:
Especie SepLarg
SepAnch
PetLarg PetAnch Clúster
1
5.1
3.5
1.4
0.2
1
1
4.9
3.0
1.4
0.2
1
1
4.7
3.2
1.3
0.2
1
1
4.6
3.1
1.5
0.2
1
1
5.0
3.6
1.4
0.2
1
1
5.4
3.9
1.7
0.4
1
1
4.6
3.4
1.4
0.3
1
1
5.0
3.4
1.5
0.2
1
1
4.4
2.9
1.4
0.2
1
1
4.9
3.1
1.5
0.1
1
1
5.4
3.7
1.5
0.2
1
1
4.8
3.4
1.6
0.2
1
1
4.8
3.0
1.4
0.1
1
1
4.3
3.0
1.1
0.1
1
1
5.8
4.0
1.2
0.2
1
1
5.7
4.4
1.5
0.4
1
1
5.4
3.9
1.3
0.4
1
1
5.1
3.5
1.4
0.3
1
1
5.7
3.8
1.7
0.3
1
1
5.1
3.8
1.5
0.3
1
1
5.4
3.4
1.7
0.2
1
1
5.1
3.7
1.5
0.4
1
1
4.6
3.6
1.0
0.2
1
1
5.1
3.3
1.7
0.5
1
1
4.8
3.4
1.9
0.2
1
1
5.0
3.0
1.6
0.2
1
1
5.0
3.4
1.6
0.4
1
120
Universidad Nacional de Ingeniería - Facultad de Ingeniería Industria y de Sistemas
Sección de Post Grado. Doctorado en Ingeniería Industrial. Curso: Programación Matemática.
Pedro C. Espinoza H.
1
5.2
3.5
1.5
0.2
1
1
5.2
3.4
1.4
0.2
1
1
4.7
3.2
1.6
0.2
1
1
4.8
3.1
1.6
0.2
1
1
5.4
3.4
1.5
0.4
1
1
5.2
4.1
1.5
0.1
1
1
5.5
4.2
1.4
0.2
1
1
4.9
3.1
1.5
0.2
1
1
5.0
3.2
1.2
0.2
1
1
5.5
3.5
1.3
0.2
1
1
4.9
3.6
1.4
0.1
1
1
4.4
3.0
1.3
0.2
1
1
5.1
3.4
1.5
0.2
1
1
5.0
3.5
1.3
0.3
1
1
4.5
2.3
1.3
0.3
1
1
4.4
3.2
1.3
0.2
1
1
5.0
3.5
1.6
0.6
1
1
5.1
3.8
1.9
0.4
1
1
4.8
3.0
1.4
0.3
1
1
5.1
3.8
1.6
0.2
1
1
4.6
3.2
1.4
0.2
1
1
5.3
3.7
1.5
0.2
1
1
5.0
3.3
1.4
0.2
1
3
6.3
3.3
6.0
2.5
2
3
7.1
3.0
5.9
2.1
2
3
6.5
3.0
5.8
2.2
2
3
7.6
3.0
6.6
2.1
2
3
7.3
2.9
6.3
1.8
2
3
6.7
2.5
5.8
1.8
2
3
7.2
3.6
6.1
2.5
2
3
6.8
3.0
5.5
2.1
2
3
7.7
3.8
6.7
2.2
2
3
7.7
2.6
6.9
2.3
2
3
6.9
3.2
5.7
2.3
2
3
7.7
2.8
6.7
2.0
2
3
6.7
3.3
5.7
2.1
2
3
7.2
3.2
6.0
1.8
2
3
6.4
2.8
5.6
2.1
2
3
7.2
3.0
5.8
1.6
2
3
7.4
2.8
6.1
1.9
2
3
7.9
3.8
6.4
2.0
2
3
6.4
2.8
5.6
2.2
2
3
7.7
3.0
6.1
2.3
2
3
6.3
3.4
5.6
2.4
2
3
6.9
3.1
5.4
2.1
2
121
Universidad Nacional de Ingeniería - Facultad de Ingeniería Industria y de Sistemas
Sección de Post Grado. Doctorado en Ingeniería Industrial. Curso: Programación Matemática.
Pedro C. Espinoza H.
3
6.7
3.1
5.6
2.4
2
3
6.9
3.1
5.1
2.3
2
3
6.8
3.2
5.9
2.3
2
3
6.7
3.3
5.7
2.5
2
3
6.7
3.0
5.2
2.3
2
2
5.5
2.3
4.0
1.3
3
2
5.7
2.8
4.5
1.3
3
2
4.9
2.4
3.3
1.0
3
2
5.2
2.7
3.9
1.4
3
2
5.0
2.0
3.5
1.0
3
2
5.9
3.0
4.2
1.5
3
2
6.0
2.2
4.0
1.0
3
2
5.6
2.9
3.6
1.3
3
2
5.6
3.0
4.5
1.5
3
2
5.8
2.7
4.1
1.0
3
2
5.6
2.5
3.9
1.1
3
2
6.1
2.8
4.0
1.3
3
2
5.7
2.6
3.5
1.0
3
2
5.5
2.4
3.8
1.1
3
2
5.5
2.4
3.7
1.0
3
2
5.8
2.7
3.9
1.2
3
2
5.4
3.0
4.5
1.5
3
2
5.6
3.0
4.1
1.3
3
2
5.5
2.5
4.0
1.3
3
2
5.5
2.6
4.4
1.2
3
2
5.8
2.6
4.0
1.2
3
2
5.0
2.3
3.3
1.0
3
2
5.6
2.7
4.2
1.3
3
2
5.7
3.0
4.2
1.2
3
2
5.7
2.9
4.2
1.3
3
2
5.1
2.5
3.0
1.1
3
2
5.7
2.8
4.1
1.3
3
3
4.9
2.5
4.5
1.7
3
2
7.0
3.2
4.7
1.4
4
2
6.4
3.2
4.5
1.5
4
2
6.9
3.1
4.9
1.5
4
2
6.5
2.8
4.6
1.5
4
2
6.3
3.3
4.7
1.6
4
2
6.6
2.9
4.6
1.3
4
2
6.1
2.9
4.7
1.4
4
2
6.7
3.1
4.4
1.4
4
2
6.2
2.2
4.5
1.5
4
2
5.9
3.2
4.8
1.8
4
2
6.3
2.5
4.9
1.5
4
2
6.1
2.8
4.7
1.2
4
122
Universidad Nacional de Ingeniería - Facultad de Ingeniería Industria y de Sistemas
Sección de Post Grado. Doctorado en Ingeniería Industrial. Curso: Programación Matemática.
Pedro C. Espinoza H.
2
6.4
2.9
4.3
1.3
4
2
6.6
3.0
4.4
1.4
4
2
6.8
2.8
4.8
1.4
4
2
6.7
3.0
5.0
1.7
4
2
6.0
2.9
4.5
1.5
4
2
6.0
2.7
5.1
1.6
4
2
6.0
3.4
4.5
1.6
4
2
6.7
3.1
4.7
1.5
4
2
6.3
2.3
4.4
1.3
4
2
6.1
3.0
4.6
1.4
4
2
6.2
2.9
4.3
1.3
4
3
5.8
2.7
5.1
1.9
4
3
6.3
2.9
5.6
1.8
4
3
6.5
3.2
5.1
2.0
4
3
6.4
2.7
5.3
1.9
4
3
5.7
2.5
5.0
2.0
4
3
5.8
2.8
5.1
2.4
4
3
6.4
3.2
5.3
2.3
4
3
6.5
3.0
5.5
1.8
4
3
6.0
2.2
5.0
1.5
4
3
5.6
2.8
4.9
2.0
4
3
6.3
2.7
4.9
1.8
4
3
6.2
2.8
4.8
1.8
4
3
6.1
3.0
4.9
1.8
4
3
6.3
2.8
5.1
1.5
4
3
6.1
2.6
5.6
1.4
4
3
6.4
3.1
5.5
1.8
4
3
6.0
3.0
4.8
1.8
4
3
5.8
2.7
5.1
1.9
4
3
6.3
2.5
5.0
1.9
4
3
6.5
3.0
5.2
2.0
4
3
6.2
3.4
5.4
2.3
4
3
5.9
3.0
5.1
1.8
4
Conclusiones
1) La red neuronal SOM que se ha credo con 4 neuronas, luego de 400 iteraciones de
entrenamiento, ha permitido descubrir 4 clases o grupos de registros.
2) El clúster 1 tiene 50 miembros y está conformado por la especie Setosa. Este clúster
identificó perfectamente a la especie Setosa, pues ninguno de los clústeres restantes contiene
esta especie.
3) El clúster 2 tiene 27 registros y está formado exclusivamente por la especie Vergínica(3).
4) El clúster 3 está constituido por 28 miembros, 1 de la especie Verginica (3) y 27 registros
de la especie Versicolor(2).
123
Universidad Nacional de Ingeniería - Facultad de Ingeniería Industria y de Sistemas
Sección de Post Grado. Doctorado en Ingeniería Industrial. Curso: Programación Matemática.
Pedro C. Espinoza H.
5) El clúster 4 está constituido por 45 miembros, 27 de la especie Verginica (3) y 23 registros
de la especie Versicolor(2).
Referencia en Internet
[1] “Redes Neuronales Aplicadas a los Negocios: análisis de un caso” Autor: M. S. Gómez
de Lima, Instituto Tecnológico de Buenos Aires.
https://ri.itba.edu.ar/bitstream/handle/123456789/1062/Tesis%20Manuel%20G%C3%B3m
ez%20de%20Lima.pdf?sequence=1&isAllowed=y
[2] “Tutorial de redes Neuronales”. Autores: M. Acosta, C.A. Zuluaga, H. Bazalar,
Universidad Tecnológica de Pereira. http://ohm.utp.edu.co/neuronales
[3] “Programación Matemática”
Separata del curso del mismo nombre. Universidad
Nacional de Ingeniería-FIIS. 2019.
124