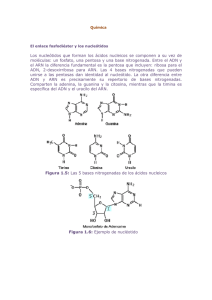
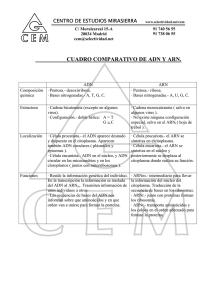
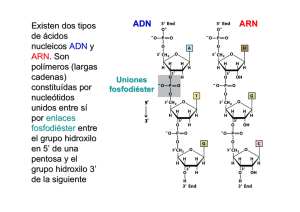
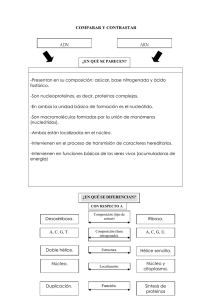
LOS ÁCIDOS NUCLEICOS Son biomoléculas orgánicas formadas, principalmente, por C, H, O, N y P. Los Ác. nucleicos son el ADN y el ARN, están constituidas por la unión de unos monómeros llamados nucleótidos. Cada nucleótido consta de: 1. Un Ac. fosfórico. Interviene en la unión de nucleótidos, responsable del carácter ácido. 2. Una pentosa (glúcido). + Ribosa: forma nucleótidos que constituyen el ARN. + Desoxirribosa: forma nucleótidos que constituyen el ADN. 3. Una base nitrogenada. Responsable del carácter básico: + Púricas: Adenina (ADN y ARN) y Guanina (ADN y ARN). + Pirimidínicas: Citosina (ADN y ARN), Timina (ADN) y Uracilo (ARN) Nucleósidos Moléculas formadas por la unión, mediante enlace N-glucosídico, del C1 de una ribosa (o desoxirribosa) con el N1 de una base nitrogenada pirimidínica (o el N9 de una base nitrogenada púrica). Nucleótidos Moléculas formadas por la unión, mediante enlace fosfoéster, de un nucleósido con una molécula de Ác. Fosfórico: la unión tiene lugar entre el grupo hidroxilo [-OH] del carbono 5’ de la pentosa del nucleósido y el propio Ác. fosfórico. Unión de nucleótidos: las cadenas de nucleótidos presentan dos extremos. 1. Extremo 5’; con un grupo fosfato unido al carbono 5’ del primer nucleótido 2. Extremo 3’, con el radical hidroxilo [-OH] del carbono 3’ del ultimo nucleótido libre. Los Ác. nucleicos se sintetizan desde el extremo 5’ hacia su extremo 3’; el enlace entre el radical [-OH] del carbono 3’ del último nucleótido y el radical fosfato del carbono 5’ del nucleótido que se añade recibe el nombre de enlace (3’, 5’) fosfodiéster. EL ADN. Clasificación de las moléculas de ADN. A. Según su forma: lineal o circular. B. Según el número de cadenas: monocatenario o bicatenario. C. Según la naturaleza de los organismos: Células eucariotas + ADN NUCLEAR: es bicatenario y lineal. Se asocia a histonas. Bacterias Arqueobacterias Virus + ADN en MITOCONDRIAS y CLOROPLASTOS: similar a la de bacterias. ADN bicatenario y circular. No está asociado a histonas. Es igual que el ADN nuclear, pero con histonas diferentes. Puede ser circular o lineal, monocatenario o bicatenario y puede estar o no asociado a histonas. El ADN presenta 3 niveles estructurales 1. Estructura primaria (Ep): Secuencia de nucleótidos de una sola cadena en la cual se distingue un eje de nucleótidos del que sobresalen bases nitrogenadas. A través de la secuencia de nucleótidos es posible almacenar la información genética. 2. Estructura secundaria (Es): Corresponde a la disposición que dos cadenas de polinucleótidos relacionadas adoptan en el espacio. Según el modelo de la doble hélice (de Watson y Crick), las características del ADN son: 1. 2. 3. 4. Consta de dos cadenas antiparalelas. Las cadenas son complementarias: las bases nitrogenadas se enfrentan A-T y G-C. Están enrolladas con giro dextrógiro: la estructura gira a la derecha a medida que avanza. El enrollamiento es plectonémico. Para separar las cadenas hay que girar una respecto a la otra. Su estabilidad se debe a los puentes de hidrógeno [H] que se establecen entre las bases nitrogenadas complementarias y a las fuerzas de Van der Waals que se dan entre los grupos hidrófobos de las bases. Las bases nitrogenadas se disponen hacia el interior de la doble hélice, mientras que las pentosas y los grupos fosfato quedan en el exterior. El ADN se puede desnaturalizar al calentar una dispersión de fibras de ADN. Si posteriormente se enfría el medio, el ADN sufre una renaturalización (técnica empleada en pruebas de paternidad). 3. Estructura terciaria o ADN superenrollado. Dado en ADN bacteriano o mitocondrial Empaquetamiento. Para caber en el núcleo de una célula, el ADN se empaqueta sobre un grupo de moléculas. Según el tipo de moléculas que sirven de soporte en el empaquetamiento del ADN, existen: - ADN asociado a histonas formando nucleosomas: se encuentra en el núcleo de las células eucariotas y en las arqueobacterias. ADN asociado a protaminas (proteínas básicas): se encuentra en el núcleo de los espermatozoides. ADN asociado a otro tipo de proteínas: El ADN se asocia a proteínas parecidas a las histonas (sin formar nucleosomas), es decir, proteínas no histónicas. 1. Fibra de cromatina de 100 A (de diámetro) o NUCLEOFILAMENTO Corresponde a la sucesión de nucleosomas (100 A de diámetro) que se asemeja a un collar de perlas. Esta estructura se encuentra en el núcleo EN REPOSO de toda célula eucariota (menos en espermatozoides). Cada nucleosoma consta de un octámero de histonas (8 moléculas de 4 tipos de histonas -H2A, H2B, H3, H4- sobre el que se enrolla un fragmento de la doble hélice de 200 pb de longitud. Esta longitud incluye los dos extremos de unión con los nucleosomas anterior y posterior. El ADN entre un octámero y otro se llama ADN espaciador (forma laxa). 2. Fibra de cromatina de 300 A o SOLENOIDE Se forma cuando las histonas H1, que componen la forma compacta de la fibra de cromatina de 100 A, se agrupan entre sí constituyendo el eje central de la misma. Se agrupan 6-8 nucleosomas por vuelta, la longitud del collar de perlas se acorta hasta 5 veces. En el nucléolo tienen lugar dos situaciones: (1) durante la interfase la cromatina se encuentra en forma de fibras de 100 A que recibe el nombre de eucromatina; en los cromosomas: el nivel más bajo de empaquetamiento es la fibra de 300 A. 3. Dominios en bucle. Las fibras de cromatina de 300 A, en los cromosomas, dan lugar a dominios estructurales en forma de bucle (20.000-70.000 pb). Estos quedan anclados al andamio proteico del cromosoma, constituido por histonas H1 o proteínas no histónicas (topoisomerasas). El ARN Tanto en células eucariotas como en procariotas predomina sobre el ADN. Pueden ser bicatenarios (algunos virus) pero la mayoría son monocatenarios. Según su función se distinguen varios tipos de ARN: ARNm (monocatenario y lineal) Su función es copiar la información contenida en un fragmento determinado de ADN (gen). - En células eucariotas: Se forma a partir de un pre-ARNm el cual presenta exones o trozos de ADN con información separados entre sí por trozos sin información o intrones. En el proceso de maduración del pre-ARNm las ribonucleoproteínas eliminan los intrones y los exones quedan unidos. En eucariotas el ARNm es monocistrónico, es decir, lleva información para sintetizar una proteína. Capucha o cap: molécula que constituye la señal de inicio en la síntesis de la proteína y bloquea la acción de las enzimas exonucleasas, que pueden destruir el ARN. A continuación, hay un intrón seguido de un exón que suele empezar con la secuencia AUG. Cola de poli A: en el extremo final (3’) se localizan numerosos nucleótidos de adenina, que sirve para retardar la llegada de las enzimas exonucleasas al último exón. - En células procariotas: No presenta intrones, carece de capucha, de cola de poli-A y puede ser policistrónicos. ARNt (mono/bicatenario y lineal): https://www.youtube.com/watch?v=FumTJz-_aeQ Tiene una conformación en forma de L y se encuentra en el citoplasma. Su función es captar y transportar aminoácidos hasta los ribosomas. Existen unos 50 tipos de ARNt El ARNt presenta zonas con estructura secundaria en doble hélice y zonas monocatenarias que forman asas o bucles. En el extremo 5’ se localiza siempre una guanina y en el extremo 3’, que es donde se enlaza el aminoácido, se encuentra siempre el triplete: 5’… C-C-A 3’ ARNr (presenta tanto segmentos monocatenarios como en doble hélice) Constituye los ribosomas. Se une a proteínas específicas, constituyendo el 60% del peso de estos orgánulos. Los ribosomas son proteínas globulares e intervienen en la síntesis de proteínas en función de la secuencia de nucleótidos del ARNm a partir de los aminoácidos del ARNt. La masa de los ARNr se expresa en unidades Svedberg: las células procariotas presentan ribosomas 70 S mientras que las células eucariotas presentan ribosomas 80 S. ARN nucleolar (monocatenario) El ARNn forma parte del nucléolo. Se origina a partir de la región del ADN denominada región organizadora nucleolar. Este ARN de 45 S se asocia a proteínas procedentes el citoplasma para formar las subunidades de los ribosomas. Otros tipos de ARN. ARN pequeño nuclear: se encuentra en el núcleo de células eucariotas. Se une a proteínas formando las ribonucleoproteínas nucleares, que eliminan los intrones en la maduración del pre-ARNm. ARN de interferencia: es utilizado por enzimas para reconocer determinados ARNm y posteriormente degradarlos impidiendo que originen proteínas. Principales funciones del ARN. 1. Transmisión de la información genética desde el ADN a los ribosomas (transcripción) Las enzimas ARN polimerasas sintetizan, por complementariedad de bases con una secuencia de ADN, un ARNm monocistrónico. El mensaje del ARNm llegará después hasta los ribosomas para sintetizar una proteína determinada (traducción) 2. Maduración del ARNm. El ARNpn asociado a proteínas (ribonucleoptoreínas) actúa eliminando los intrones del pre-ARNm. 3. Conversión de la secuencia de del ARNm en una secuencia de aminoácidos o traducción. Lo realizan los ribosomas. En el proceso intervienen todos los tipos de ARN. 4. Almacenamiento de la información genética. Algunos virus carecen de ADN y guardan su información biológica en moléculas de ARN. (23’) https://www.youtube.com/watch?v=7sDU21ffbo8&t=14s https://www.youtube.com/watch?v=YqjbmrQcyfM Duplicación o replicación del ADN Replicación semiconservativa: tras la duplicación cada hebra de la doble hélice sirve de molde para que se sintetice una hebra nueva mediante la complementariedad de bases. | Hip. Conservativa | |Hip. Semiconservavita| | Hip. Dispersiva | Síntesis de ADN in vitro (experimental) (Arthur Kornberg) ADN POLIMERASA. Formada por unos mil aminoácidos, se encuentra en el núcleo, mitocondrias oyen cloroplastos. Para la síntesis de una nueva hebra, la enzima necesita un desoxiribonucleótido (desoxirrubonucleótidos-5-fosfato), iones Mg2+ y un fragmento de ADN en el cual se ha quitado un sector de una de las cadenas. A. Kornberg aisló la ADP polimerasa, como la enzima reguladora del proceso de síntesis de la hebra complementaria de ADN. Tiene varios lugares donde se fijan los sustratos. Quedan ocupados por el ADN patrón, el ADN cebador y el nucleótido añadido. La ADN polimerasa actúa añadiendo nucleótidos al extremo de una cadena preexistente con un nucleótido en su C3 libre, por eso la cadena de ADN solo puede crecer en sentido 5’ 3’. La nueva hebra sintetizada es antiparalela y complementaria a la hebra patrón. https://www.um.es/molecula/dupli00.htm Duplicación del ADN in vivo (en interfase) 1. En células procariotas (bacterias). Fase de iniciación. La región denominada “origen de replicación” actúa como señal de inicio del proceso. A partir de la señal de inicio se separan las hebras complementarias del ADN gracias a la acción de la enzima helicasa cuya acción bidireccional inicia a su vez la formación de las horquillas de replicación. Las enzimas topoisomerasas, (como las girasas) eliminan las tensiones y los superenrollamientos de la doble hélice mientras las proteínas estabilizadoras – o proteínas SSBmantienen las hebras complementarias separadas. Fase de elongación. En primer lugar, una ARN polimerasa, la primasa, sintetiza un fragmento corto de ARN o primer (unos 5-15 nucleótidos), que actúa como cebador (nucleótidos de ARN que permiten que la ADN polimerasa III comience la síntesis de la nueva cadena de ADN). Partiendo del cebador la ADN polimerasa III sintetiza la nueva hebra de ADN en sentido 5’3’ (dirección 3’ 5’ de la hebra molde). Esta nueva habrá sintetizada, denominada hebra conductora, tiene un crecimiento continuo. Sobre la otra hebra de ADN antiparalela la ARN polimerasa (primasa) sintetiza unos 40 nucleótidos de ARN –primer- a unos 1000 nucleótidos del origen de replicación. A partir de estos, la ADN polimerasa III sintetiza los 1000 nucleótidos de ADN en dirección 5’3’ (dirección 3’5’ de la hebra molde) formando el segmento denominado fragmento de Okazaki. Este proceso se va repitiendo a medida que se van separando las dos hebras de patrón. Seguidamente, la ADN polimerasa I retira los segmentos de ARN (del primer) y añade nucleótidos de ADN en su lugar (uracilos por timinas). Por último, la ADN ligasa une los fragmentos de ADN sintetizados (los fragmentos de Okazaki y los nucleótidos sintetizados por la ADN polimerasa I). Esta nueva hebra, denominada hebra retardada, tiene un crecimiento discontinuo (lento). 2. En células eucariotas. -. Al formarse las horquillas de replicación, la hebra que sirve de patrón a la hebra conductora se queda con las histonas y ambas cadenas se enrollan sobre los nucleosomas preexistentes antiguos. La hebra retardada y la hebra que le sirve de patrón se enrollan sobre nuevos nucleosomas. -. Los fragmentos de Okazaki son más pequeños y el ADN de un cromosoma presenta, distribuidos de manera irregular, varios orígenes de replicación – conjunto denominado replicón- que se activan coordinadas. En los virus con ARN como material genético (retrovirus); el ARN funciona como molde para la síntesis de ADN mediante la enzima transcriptasa inversa. Transcripción del ADN Proceso por el cual, a partir de la secuencia de nucleótidos de un fragmento determinado de ADN (gen), se sintetiza un ARN concreto. Este proceso se lleva a cabo en el núcleo o en el citoplasma. En el proceso intervienen: un gen (fragmento de ADN molde), ribonucleicos trifosfatos de A, G, C y Uracilo y enzimas ARN polimerasas. 1. TRANSCRIPCION EN PROCARIOTAS (en el citoplasma). 1. Iniciación. En el segmento de ADN (gen) que se va a transcribir, la existencia de una región inicial que no se transcribe, denominada promotor, determina cuál de las dos cadenas del fragmento se va a transcribir. Este tiene una secuencia de consenso (nucleótidos) a la que se asocia la ARN polimerasa, que posteriormente desenrolla la hélice e inicia la polimeración de ARN. 2. Elongación. A medida que la ARN polimerasa recorre la hebra de ADN patrón, sintetiza una hebra de ARN antiparalela en dirección 5’3’ (dirección 3’5’ a la hebra patrón). 3. Finalización. Se produce cuando la ARN polimerasa llega a la secuencia “terminador”, formado por G y C seguido de varias T. Finalmente, el ADN recupera su forma de doble hélice. 4. Maduración. Si se sintetiza un ARNm, no hay maduración. No obstante, si es un ARNt o ARNr, sí experimenta un proceso de maduración eliminando los intrones. 2. TRANSCRIPCIÓN EN EUCARIOTAS (en el núcleo) En eucariotas existen tres tipos de ARN polimerasa: ARN polimerasa I; que transcribe genes de ARNr, ARN polimerasa II, que transcribe genes de proteínas (sintetiza ARNm); y ARN polimerasa III, que trascribe ARNt y ARNr (tipo S). Siempre es necesario la maduración del ARN transcrito (cualquiera que este sea). En genes que se transcriben continuamente (como el ARNr), el ADN siempre está extendido. En otros, siempre está formando nucleosomas, y en otros, hay transición durante la transcripción. 1. Iniciación. La ARN polimerasa se fija al promotor, que en muchos genes consta de dos secuencias de consenso: CAAT o TATA. Para que la ARN polimerasa se pueda fijar, antes se deben fijar en dichas secuencias los factores de transcripción (proteínas que ubican la ARN-polimerasa) 2. Elongación. La síntesis continúa en dirección 5’3’ (dirección 3’5’ a la hebra molde). Un mismo gen puede ser transcrito por varias ARN-pol a la vez. Al término de la síntesis se añade una caperuza (CAP) en el extremo inicial 5’ de la nueva cadena ARN sintetizada. 3. Finalización. A continuación, unas enzimas separan el pre-ARN de la ARN polimerasa y queda libre en el núcleo. Finalmente, interviene la enzima poliA polimerasa, que añade ribonucleótidos de adenina al extremo final 3’ de la nueva hebra, la cola de poliA. Esto retarda la acción destructiva de las enzimas exonucleasas. 4. Maduración. Varias enzimas ribonucleoproteína pequeña nuclear (RNPpn) se asocian a proteínas y forman la espliceosoma: complejo formado por proteínas y ARN que cataliza la eliminación de intrones (segmentos del gen sin información). A continuación, la ARN ligasa empalma los exones. PROCESO DE TRADUCCIÓN DEL ARNm https://www.youtube.com/watch?v=YoyFpumWtHo Proceso por el cual, en los ribosomas, se sintetiza una proteína mediante la unión de los aminoácidos indicados en la secuencia de nucleótidos de un ARNm. Durante la fase intervienen: Aminoácidos Diversos tipos de ARN -. ARNmensajero: lleva la información genética transcrita de los genes a los ribosomas. -. ARNtransporte: transporta los aminoácidos a los ribosomas. -. ARNrribosómico: forma parte esencial del propio ribosoma Factores proteicos de traducción. Nucleótidos trifosfato, donadoras de energía. En el proceso de traducción se distinguen las siguientes etapas: A. Activación previa dea los aminoácidos B. Traducción propiamente dicha: INICIACIÓN. Una vez activados los aminoácidos, el proceso continúa en el complejo ribosomal: 1. Centro A (acceptor): sirve de entrada a nuevos ARNtransferentes cargados con aminoácidos o aminoacil ARNt 2. Centro P (peptidil): región ocupada por el peptidil ARNt. Es el ARNt que lleva la cadena polipeptídica creciente. Iniciada la traducción, aquí se sitúa el primer aminoacil ARNt (la metionina) 3. Centro E (exit): indica la salida de los ARNt después de haber dejado el aminoácido. Ubica el ARNt que acaba de dar su aminoácido y que saldrá del ribosoma. El ARNm se une a la subunidad menor de un ribosoma por la secuencia inicial 5’UTR o región líder, que no se traduce. Después, la subunidad menor se mueve respecto al ARNm hasta que se encuentra el codón de iniciación AUG. Al codón se asocia al aminoacil ARN que presenta el anticodón (en eucariotas transporta la metionina). A continuación, se une la subunidad ribosómica mayor para formar el complejo activo o ribosomal. En este proceso intervienen los factores de iniciación, que consumen la energía del GTP. ELONGACIÓN. El radical carboxilo de la metionina se une con el radical amino del segundo aminoácido (mediante enlace peptídico) y una enzima, la peptidil transferasa, cataliza la unión. Así, el centro P es ocupado por un ARNt sin aminoácido y después se produce la translocación ribosomal; este ARNt pasa al centro E. El aminoacil ARNt pasa al centro P y el centro A queda libre, a la espera de un nuevo aminoacil ARNt. En el proceso intervienen los factores de elongación, que consumen ATP. TERMINACIÓN El final del proceso está determinando por un UGA, UAG o UAA para las que no hay ningún anticodón complementario, pero si son reconocidos por los factores de liberación, que consumen energía del GTP. Esto provoca que, en el centro A, la peptidil transferasa haga interaccionar el grupo carboxilo del ultimo aminoácido con el agua, liberándose la cadena polipeptídica (por hidrólisis) del complejo ribosomal. El ARNm y las subunidades ribosómicas también se separan. C. Asociación de grupos proteicos y grupos prostéticos para formar proteínas complejas. TEORÍA UN GEN-UNA ENZIMA Cuando se altera la secuencia de nucleótidos de un gen se impide la síntesis de una enzima. Como las enzimas controlan las sustancias y las características de los organismos, se establece un paralelismo entre genes y enzimas. Un mutante, organismo carente de una enzima, solo sobrevive si a su medio se le añade la sustancia que no podían sintetizar. EL CÓDIGO GENÉTICO El código genético es el conjunto de reglas que define cómo se traduce una secuencia de nucleótidos (ARN) a una secuencia de aminoácidos (proteína). Existen un total de 64 tripletes o codones posibles, de los cuales: 1) Varios codifican para un mismo aminoácido, fenómeno denominado degeneración del código genético. Esto resulta ventajoso, pues, si se producen errores en la copia de un nucleótido, se puede mantener la colinealidad entre el triplete y el aminoácido. 2) Los codones UGA, UAG y UAA solo indican el final del proceso de traducción y el codón AUG actúa como señal de inicio del proceso. EN LAS CÉLULAS EXISTEN SISTEMAS DE REGULACIÓN DE LA EXPRESIÓN GÉNICA. Como la cantidad de proteínas que se debe sintetizar depende de la cantidad de ARNm presente en el citoplasma, y su estabilidad es muy corta, la cantidad de ARNm sintetizado regula los niveles enzimáticos del medio (el metabolismo celular) CONTROL DE LA EXPRESIÓN GÉNICA EN EUCARIOTAS Aunque todas las células de un organismo tienen el mismo ADN, no se manifiesta la misma información (diferenciación celular). Esto se sabe a que algunas enzimas: Añaden grupos acetilos a las histonas (acetilación de histonas): de esta forma los nucleosomas no se unen y la cromatina se descondensa favoreciendo la transcripción. Metilan las bases nitrogenadas (metilación del ADN): provoca que no se expresen algunos genes. Cada tipo de célula tiene receptores de membrana diferentes, así, solo algunas pueden ser diana para determinadas hormonas: - Hormonas lipídicas: por su composición, pasan fácilmente por la membrana plasmática. - Hormonas proteicas: por el tamaño y la naturaleza de sus moléculas se deben unir a receptores específicos de membrana para poder atravesar la bicapa.