Tema 12.2 Naturaleza del ácido nucleico terapéutico
Anuncio
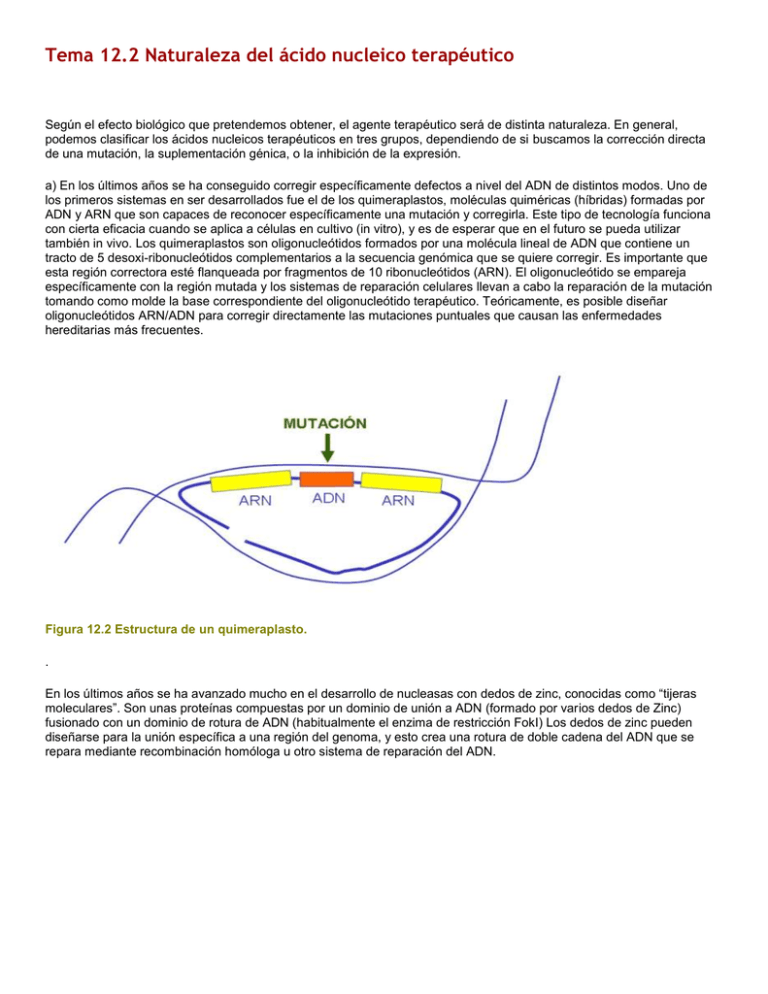
Tema 12.2 Naturaleza del ácido nucleico terapéutico Según el efecto biológico que pretendemos obtener, el agente terapéutico será de distinta naturaleza. En general, podemos clasificar los ácidos nucleicos terapéuticos en tres grupos, dependiendo de si buscamos la corrección directa de una mutación, la suplementación génica, o la inhibición de la expresión. a) En los últimos años se ha conseguido corregir específicamente defectos a nivel del ADN de distintos modos. Uno de los primeros sistemas en ser desarrollados fue el de los quimeraplastos, moléculas quiméricas (híbridas) formadas por ADN y ARN que son capaces de reconocer específicamente una mutación y corregirla. Este tipo de tecnología funciona con cierta eficacia cuando se aplica a células en cultivo (in vitro), y es de esperar que en el futuro se pueda utilizar también in vivo. Los quimeraplastos son oligonucleótidos formados por una molécula lineal de ADN que contiene un tracto de 5 desoxi-ribonucleótidos complementarios a la secuencia genómica que se quiere corregir. Es importante que esta región correctora esté flanqueada por fragmentos de 10 ribonucleótidos (ARN). El oligonucleótido se empareja específicamente con la región mutada y los sistemas de reparación celulares llevan a cabo la reparación de la mutación tomando como molde la base correspondiente del oligonucleótido terapéutico. Teóricamente, es posible diseñar oligonucleótidos ARN/ADN para corregir directamente las mutaciones puntuales que causan las enfermedades hereditarias más frecuentes. Figura 12.2 Estructura de un quimeraplasto. . En los últimos años se ha avanzado mucho en el desarrollo de nucleasas con dedos de zinc, conocidas como “tijeras moleculares”. Son unas proteínas compuestas por un dominio de unión a ADN (formado por varios dedos de Zinc) fusionado con un dominio de rotura de ADN (habitualmente el enzima de restricción FokI) Los dedos de zinc pueden diseñarse para la unión específica a una región del genoma, y esto crea una rotura de doble cadena del ADN que se repara mediante recombinación homóloga u otro sistema de reparación del ADN. La Figura 12.3 ilustra el funcionamiento de nucleasas de dedos de zinc. b) En el caso de mutaciones que simplemente producen pérdida de función, puede ser suficiente la suplementación génica, es decir, suplementar las células con copias normales del gen sin preocuparnos de corregir las mutaciones presentes en el ADN genómico. Lo más habitual en estos casos es que el agente terapéutico sea una unidad de expresión en la que la transcripción del gen en cuestión viene regulada por promotores virales potentes, por promotores regulables o por promotores específicos de un tipo celular concreto. La Figura 12.4 muestra un plásmido de expresión para suplementación génica. c) Cuando el defecto genético que tratamos de corregir origina una ganancia de función, como suele suceder en mutaciones con efectos oncogénicos, lo lógico es intentar inhibir la expresión del gen que está causando el fenotipo aberrante. Los agentes terapéuticos más utilizados en estas circunstancias son las moléculas antisentido o los ribozimas. Los oligonucleótidos antisentido pueden inhibir directamente la expresión de un gen al unirse a la doble hélice de ADN mediante enlaces tipoHoogsteen, formado una triple hélice que impide la transcripción mediada por la ARN polimerasa II. Estos oligonucleótidos suelen estar modificados químicamente para aumentar su estabilidad y eficacia, siendo las principales modificaciones los grupos fósforo-tioato en el esqueleto desoxi-ribosa-fosfato de los oligonucleótidos, o bien los ácidos nucleicos en los que el enlace fosfo-diéster viene substituido por un enlace peptídico (denominados Acidos Nucleicos Peptídicos, PNA). Los ARNm antisentido son capaces de emparejarse con los ARN sentido y formar moléculas bicatenarias de ARN que no pueden ser traducidas y son digeridas por la RNAsa H celular. Los ribozimas, pequeñas moléculas de ARN con actividad catalítica, pueden ser diseñados para reconocer un ARNm específico y degradarlo merced a su actividad endonucleolítica específica. En general, la utilización de moléculas antisentido y ribozimas está limitada por la baja eficacia que muestran todavía en modelos in vivo. La Figura 12.5 muestra los distintos tipos de moléculas utilizadas para inhibir la expresión génica. Hoy en día, las mejores perspectivas de inhibición eficaz y específica de la expresión génica se basan en la interferencia de ARN, un mecanismo de silenciamiento génico post-transcripcional descrito en plantas y C. elegans que consiste en la degradación de ARN mensajeros endógenos por la presencia de moléculas pequeñas de ARN de doble cadena homólogas al mensajero que se destruye. Este proceso depende de una RNAasa tipo III llamada DICER, responsable de la formación de los ARN pequeños, y de un complejo llamado RISC (RNA Induced Silencing Complex) que contiene unas proteínas del complejo ARGONAUTA y los ARN pequeños que dirigen el complejo a la diana. Se ha visto que este mecanismo también juega un papel importante en la regulación génica en humanos, ya que el análisis del genoma ha revelado la presencia de genes que codifican ARN pequeños (aproximadamente 75 nucleótidos) que forman horquillas y que son procesados por DICER y proteínas del complejo Argonauta para generar ARN intereferentes pequeños, de unos 21-22 nucleótidos. Estos genes endógenos se denominan miRNA (microRNA), para diferenciarlos de los siRNA que provienen de moléculas bicatenarias de ARN de procedencia externa. Actualmente se piensa que ambos tipos de moléculas (miRNA y siRNA) tienen la misma vía efectora, que actúa impidiendo la traducción (si la homología de la molécula interferente con el ARN mensajero no es total), o bien eliminando los mensajeros si la homología es total. Sorprendentemente, también se ha visto que las repeticiones centroméricas y los transposones, cuando se transcriben, dan lugar a ARN interferentes pequeños que utilizan este mecanismo para modificar la cromatina, induciendo metilación en las histonas ó en el ADN y provocando la formación de heterocromatina o el silenciamiento transcripcional. En los últimos años se ha demostrado que se pueden utilizar siRNAs ó microRNAs para inhibir la expresión de genes endógenos, generando moléculas interferentes específicas para un gen concreto. Por ejemplo, se ha conseguido silenciar genes endógenos en células humanas mediante siRNAs dirigidos específicamente frente a promotores concretos. Dicho silenciamiento se lleva a cabo por metilación de los dinucleótidos CpG de esos promotores y por metilación de la lisina 9 de la histona H3 de esa región. El avance más espectacular usando esta tecnología tuvo lugar en 2004, cuando un grupo consiguió reducir hasta un 40% los niveles de colesterol en ratón, usando unos siRNA sintéticos conjugados con colesterol que fueron injectados directamente en la vena de la cola. Los siRNA fueron captados eficazmente por receptores del hígado, yeyuno y otros órganos y provocaron la degradación de los ARNm endógenos de apolipoproteína B, con el consiguiente descenso en los niveles de LDL y colesterol total. Figura 12.6 El video ilustra el funcionamiento de los microARNs .
