QST*R - Elucigene
Anuncio

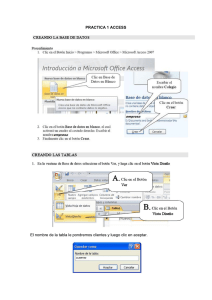
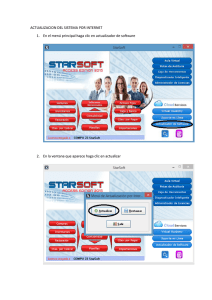
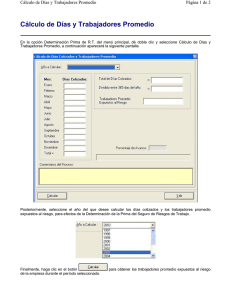
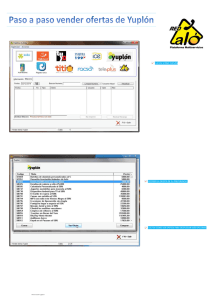
Productos Elucigene® QST*R® Guía del software de análisis Fabricado por: Gen-Probe Life Sciences Ltd. Oaks Business Park Crewe Road Wythenshawe Manchester M23 9HZ, Reino Unido Para ventas, servicio al cliente y asistencia técnica: T: +49 (0) 6122 7076451 F: +49 (0) 6122 7076155 E: [email protected] E: [email protected] AN000GSES Rev.10/2013 Página 1 de 29 Análisis GeneMapper Introducción La sección siguiente describe los procedimientos de análisis de muestras de resultados de Elucigene QST*R usando los paquetes de software GeneMapper de Applied Biosystems (versión 3.7 o superior) y la forma de transferir datos de tablas en la plantilla de informe de QST*R de Excel. Proceso de análisis El proceso para el análisis de muestras se resume en el diagrama de flujo siguiente: AN000GSES Rev.10/2013 Página 2 de 29 GeneMapper Abra el archivo del programa GeneMapper e importe los archivos .fsa requeridos haciendo clic en el botón «add samples to project» (añadir muestras al proyecto) . Navegue a la carpeta del ciclo deseado usando el árbol de carpetas del lado izquierdo de la ventana. Seleccione la carpeta y haga clic en el botón «Add to List >>» (Añadir a la lista >>). La carpeta del ciclo aparecerá ahora en la ventana «Samples To Add» (Muestras para añadir). Haga doble clic en el icono de la carpeta del ciclo en esta ventana para ver los archivos .fsa individuales que se van a importar (figura 1). Las muestras se añaden entonces haciendo clic en el botón «Add» (Añadir) . Figura 1: Muestras listas para añadirse al proyecto AN000GSES Rev.10/2013 Página 3 de 29 Importación de la configuración del análisis QST*R para GeneMapper al GeneMapper Manager Es necesario importar la configuración de QST*R para GeneMapper. Este proceso se controla a través de la interfaz de «GeneMapper Manager». La configuración de QST*R para GeneMapper está disponible en el sitio web de GenProbe: www.gen-probe.com/global/products-services/ 1. Abra el programa GeneMapper Manager haciendo clic en el icono (figuras 2 y 3). Figura 2: Abrir GeneMapper Manager Figura 3: Interfaz de GeneMapper Manager 2. Seleccione la ficha «Analysis Methods» (Métodos de análisis) y luego haga clic en el botón Import (Importar). AN000GSES Rev.10/2013 Página 4 de 29 3. Navegue a los archivos de configuración del análisis QST*R: 3130 o 3500, e importe los archivos requeridos (figura 4). Figura 4: Importación de métodos de análisis 4. Repita el proceso seleccionando la ficha adecuada e importando los archivos correspondientes para: • Table Settings (Ajustes de la tabla) • Plot Settings (Ajustes del gráfico) • Size Standards (Estándares de tamaño) Nota: No es necesario importar archivos para: Cluster Plot Settings (Ajustes de gráficos de grupos), Matrices (Matrices), SNP Sets (Conjuntos SNP) y Report Settings (Ajustes de informes). AN000GSES Rev.10/2013 Página 5 de 29 Importación de los ajustes del panel QST*R en Panel Manager Es necesario importar los ajustes de los paneles y de los «bines» QST*R para GeneMapper. Este proceso se controla a través de la interfaz del «Panel Manager» (Administrador de paneles). Los ajustes de los paneles y de los «bines» QST*R para GeneMapper están disponibles en el sitio web de Gen-Probe: www.gen-probe.com/global/products-services/ 1. Abra el programa Panel Manager (Administrador de paneles) haciendo clic en el icono . 2. Haga clic en «Panel Manager» (Administrador de paneles) en la ventana de navegación de la izquierda. El administrador de paneles aparecerá ahora resaltado en azul. 3. Seleccione «File/Import Panels» (Archivo/Importar paneles). Navegue al archivo del panel GeneMapper «anan000 gmbf*.txt», e impórtelo (figura 5). 4. El archivo del panel se mostrará ahora en la ventana de navegación de la izquierda. Haga clic en el archivo del panel y asegúrese de que quede resaltado en azul. 5. Seleccione «File/Import Bin Set’ (Archivo/Importar juego de «bines»). Navegue al archivo del «bin» GeneMapper «anan000 gmbf*.txt’, e impórtelo (figura 6). 6. Haga clic en «Apply» (Aplicar) y luego en «OK» (Aceptar). Figura 5: Importación del archivo del panel QST*R AN000GSES Rev.10/2013 Página 6 de 29 Figura 6: Importación del archivo de juego de «bines» QST*R Modificación del archivo de parámetros del análisis Puede que sea necesario modificar los «Analysis Ranges» (Rangos del análisis) predeterminados en la configuración del análisis QST*R para tener en cuenta la varianza local en las condiciones del ciclo. El rango mínimo del análisis dependerá del capilar y del polímero usados durante la recogida de datos. Para ver la configuración del análisis actual: 1. Abra «GeneMapper Manager». 2. Seleccione la ficha «Analysis Methods» (Métodos de análisis). El archivo QST*R importado figurará en la lista como «QSTR Analysis» (Análisis QSTR). 3. Haga clic en «QSTR Analysis» (Análisis QSTR). La fila aparecerá ahora resaltada. 4. Haga clic en el botón «Open» (Abrir) y seleccione la ficha «Peak Detector» (Detector de picos) (figura 7). De forma predeterminada, los rangos del análisis se establecen para empezar en 2000 y terminar en 18.000. AN000GSES Rev.10/2013 Página 7 de 29 Figura 7: Rangos del análisis Para encontrar el rango de análisis correcto para su laboratorio: 1. Desde la ventana principal de GeneMapper, haga doble clic en la carpeta del ciclo importado para ver la lista de archivos .fsa que contiene. 2. Seleccione un archivo .fsa. 3. Al hacerse clic en la ficha «Raw data» (Datos sin procesar), aparece el electroferograma de los datos sin procesar. 4. Empleando el primer pico del estándar de tamaño (p. ej., 75 pb de GS500LIZ) como guía, seleccione un punto de datos que sea aproximadamente 100 puntos de datos más grande (figura 8). Esto determina el punto más bajo del rango analizable. 5. Asegúrese de que el rango de análisis máximo incluya el pico más grande del estándar de tamaño (p. ej., 500 pb de GS500LIZ o 600 pb de GS600LIZv2). 6. Introduzca los nuevos valores en el archivo de análisis QST*R (acceda a él según se ha descrito anteriormente). AN000GSES Rev.10/2013 Página 8 de 29 Figura 8: Búsqueda del rango mínimo usando datos sin procesar de la muestra Análisis de archivos QST*R importados 1. En la ventana principal de GeneMapper, seleccione «QST*R Table Settings» (Ajustes de la tabla QST*R) (figura 9). Figura 9: Menú desplegable de los ajustes de la tabla QST*R 2. En la ventana principal de GeneMapper, seleccione la primera celda bajo «Analysis Method» (Método de análisis) y seleccione «QSTR Analysis 3130» (Análisis QSTR 3130) o «QSTR Analysis 3500» (Análisis QSTR 3500) del menú desplegable. Repita el proceso para el estándar de tamaño seleccionando «QSTRLIZ500» o «QSTRLIZ600» del menú desplegable. 3. Seleccione el panel «QSTR gm*» y haga clic en el subpanel adecuado (figura 10). 4. Rellene cada columna seleccionando el encabezado de la columna y pulsando teclas «Ctrl-D». 5. Haga clic en para iniciar el análisis de la muestra. Asigne un nombre al proyecto cuando se le pida. AN000GSES Rev.10/2013 Página 9 de 29 Figura 10: Selección de los ajustes del panel QST*R Revisión de los datos de QST*R 1. Seleccione la muestra que desee analizar (resalte la fila de la muestra). 2. Haga clic en para «Display Plots» (Mostrar gráficos). 3. Seleccione los «QST*R Plot settings» (Ajustes del gráfico QST*R) (figure 11). Figura 11: Menú desplegable de los ajustes del gráfico QST*R. 4. La ventana del gráfico mostrará el perfil de la muestra con los datos tabulados (figura 12). GeneMapper etiquetará automáticamente un máximo de dos picos por cada marcador. Si hay tres alelos presentes para un marcador, el tercer pico no etiquetado deberá etiquetarse manualmente (consulte: Modificación manual de perfiles). Nota: Los rangos de tamaño de los alelos de cada marcador se basan en datos observados previamente. Puede haber alelos infrecuentes que caigan fuera del rango de tamaño de un marcador determinado, en cuyo caso podrá ser necesario ajustar consiguientemente el juego de «bines». 5. Se recomienda que la ventana del gráfico tenga la opción «Single click editing» (Modificación con un clic) activada. Para hacer esto, seleccione «Alleles/set click editing» (Alelos/definir modificación con clic) y compruebe que esta opción está marcada. AN000GSES Rev.10/2013 Página 10 de 29 Figura 12: Ventana del gráfico de la muestra mostrando los datos de las trazas etiquetadas y la tabla de genotipo correlacionada AN000GSES Rev.10/2013 Página 11 de 29 Modificación manual de perfiles ¡ADVERTENCIA! GeneMapper solamente asignará etiquetas a un máximo de 2 picos por marcador. Por lo tanto, es posible que sea necesario la modificación manual de los perfiles, p. ej., cuando haya que asignar etiquetas a terceros picos (si los hay) o retirar etiquetas de picos de bandas sombra («stutter peaks»). Para añadir una etiqueta de pico, haga clic izquierdo en el pico sin etiquetar. Se le ofrecerá la opción de añadir un comentario de alelo. Haga clic en «OK» (Aceptar). El pico estará ahora etiquetado con su tamaño en pares de bases y su área de pico. La tabla incorporará automáticamente el pico recién etiquetado. Para eliminar una etiqueta de pico, haga clic izquierdo en ella. Se le ofrecerá la opción de eliminar un comentario de alelo. Haga clic en «OK» (Aceptar). La etiqueta de pico se eliminará ahora. La tabla eliminará automáticamente el dato de pico borrado. Para obtener más información sobre la puntuación de muestras QST*R, consulte las Instructions for Use (Instrucciones de uso) y la Guide to Interpretation (Guía de la interpretación) de Elucigene QST*R disponibles en el sitio web de Gen-Probe: www.gen-probe.com/global/products-services/ Copia de datos de tablas 1. Resalte todas las filas de la tabla situada en la parte inferior de la ventana Plot (Gráfico). 2. Copie las filas seleccionadas pulsando teclas «Ctrl+C». Plantilla de informe QST*R Las plantillas de informes QST*R pueden utilizarse para determinar las razones de picos de un marcador. La plantilla de informe de cada uno de los productos de la línea QST*R está disponible en el sitio web de Gen-Probe: www.gen-probe.com/global/products-services/ 1. Abra el archivo de plantilla de informe adecuado. 2. Si la plantilla de informe muestra una advertencia de seguridad que indica que las macros se han deshabilitado, haga clic en el botón de opciones como se indica en la figura 13. 3. Aparecerá una ventana de opciones de seguridad (figura 14). Seleccione la opción «Enable this content» (Habilitar este contenido) y haga clic en «OK» (Aceptar). AN000GSES Rev.10/2013 Página 12 de 29 Figura 13: Advertencia de seguridad (macros deshabilitadas) Figura 14: Ventana de opciones de seguridad (para habilitar macros) 4. Seleccione la hoja «Import GM» (Importar GM) y asegúrese de que esté seleccionada la celda A1. 5. Pegue los datos de la tabla copiados de GeneMapper pulsando teclas «Ctrl+V» y haga clic inmediatamente en el botón «Sort» (Ordenar) (figura 15). Nota: Para que la función «Sort» (Ordenar) funcione, DEBEN estar seleccionados todos los datos pegados. 6. Seleccione la ficha «QSTR-GM» (- indicará qué hoja de cálculo está en uso en ese momento). El informe de resultados contendrá ahora toda la información y las razones de picos de la muestra (figura 16). AN000GSES Rev.10/2013 Página 13 de 29 Figura 15: Ejemplo de tabla de GeneMapper importada para QST*Rplusv2 Figura 16: Ejemplo de informe de QST*Rplusv2 AN000GSES Rev.10/2013 Página 14 de 29 Puntuación del informe 1. La trisomía se determina por una de las dos condiciones siguientes: a) Dos picos de altura desigual debido a que uno de los picos representa dos alelos comunes a ambos padres. En este caso, la razón entre los dos picos se clasificará como 2:1 o 1:2. Donde A1/A2 dará un resultado en la región de 1,8 a 2,4 cuando el pico que represente el alelo de menor longitud tenga un área mayor que el pico que represente el alelo de mayor longitud, o donde A1/A2 dará un resultado en la región de 0,45 a 0,65 cuando el pico que represente el alelo de menor longitud tenga un área inferior a la del pico que represente el alelo de mayor longitud. En ambos casos aparecerá «Ratio» (Razón) en la columna «Warning» (Advertencia). b) Presencia de tres picos de altura similar. La razón de los picos se clasificará como 1:1:1 y sus valores caerán dentro del rango normal de 0,8 a 1,4 (aunque para los alelos separados por más de 24 pb es aceptable una razón alélica de hasta 1,5). En este caso aparecerá «3 Alleles» (3 alelos) en la columna «Warning» (Advertencia). 2. Para interpretar un resultado como anormal, (esto es, con presencia de trisomía) es necesario que al menos dos marcadores informativos sean indicativos de un genotipo trialélico y que todos los demás marcadores sean no informativos. No se recomienda interpretar un resultado como anormal sobre la base de información de un solo marcador. 3. Para interpretar un resultado como normal, es necesario que al menos dos marcadores informativos sean indicativos de un genotipo dialélico y que todos los demás marcadores sean no informativos. Un resultado normal indica el complemento normal de dos de los cromosomas analizados. 4. Las razones de áreas de picos que caigan entre los rangos normal y anormal se clasifican como no concluyentes. Los resultados no concluyentes pueden resolverse utilizando los kits de cromosoma único. 5. Cuando no haya datos de picos para un marcador, la columna de advertencia mostrará ‘Absent’ (Ausente). Esta advertencia se observará siempre que no haya marcadores de cromosoma Y. AN000GSES Rev.10/2013 Página 15 de 29 Análisis GeneMarker Introducción La sección siguiente describe los procedimientos de análisis de muestras de resultados de Elucigene QST*R usando los paquetes de software GeneMarker de SoftGenetics (versión 1.65 o superior). Las imágenes utilizadas en esta sección se han tomado de la versión 1.85 del software GeneMarker. Proceso de análisis El proceso para el análisis de muestras se resume en el diagrama de flujo siguiente: AN000GSES Rev.10/2013 Página 16 de 29 Adición de archivos de muestras a GeneMarker Abra el archivo del programa GeneMarker y, cuando se le pida, seleccione «Open Data» (Abrir datos). Aparecerá el cuadro Open Data Files (Abrir archivos de datos). Haga clic en el botón «Add» (Añadir). Aparecerá el cuadro de diálogo Open (Abrir). Navegue al directorio que contiene los archivos de datos sin procesar; • • Seleccione todos los archivos mediante CTRL+A o utilice las teclas CTRL y/o MAYÚS para seleccionar muestras individuales Haga clic en el botón «Open» (Abrir) en el cuadro de diálogo Open (Abrir) Los archivos seleccionados aparecerán en el campo Data File List (Archivos de datos) (figura 1). Figura 1: Muestras añadidas a la lista Data File List (Archivos de datos) Haga clic en el botón «OK» (Aceptar) en el cuadro Open Data Files (Abrir archivos de datos) y las muestras se transferirán al GeneMarker. El software abrirá entonces automáticamente la ventana Raw Data Analysis (Análisis de datos sin procesar) (figura 2). AN000GSES Rev.10/2013 Página 17 de 29 Figura 2: Ventana de Raw Data Analysis (análisis de datos sin procesar) AN000GSES Rev.10/2013 Página 18 de 29 Importación de los ajustes del panel QST*R para GeneMarker Es necesario importar los ajustes del panel QST*R para GeneMarker. Este proceso se controla a través de la interfaz del «Panel Editor» (Editor de paneles). Los ajustes del panel QST*R para GeneMarker están disponibles en el sitio web de Gen-Probe: www.gen-probe.com/global/products-services • Abra «Panel Editor» (Editor de paneles) en el menú desplegable «Tools» (Herramientas). Figura 3: Selección de Panel Editor (Editor de paneles) • Seleccione «Import Panels» (Importar paneles) en el menú desplegable «File» (Archivo). Figura 4: Importación de paneles • Navegue al panel, p. ej., anan0pl_gupf***.xml, e impórtelo. (Nota: * Indica un número de versión de tres dígitos (p. ej., anan0pl_gupf002.xml) • Repita el proceso como sea necesario para otros archivos de paneles relevantes. AN000GSES Rev.10/2013 Página 19 de 29 Procesamiento de datos Una vez cargados en GeneMarker, los archivos de datos sin procesar están listos para procesarse. El paso de procesamiento consiste en la aplicación de un estándar de tamaño, la filtración de los picos de ruido y la comparación con un panel alélico conocido si se desea. GeneMarker combina todos estos pasos en una simple herramienta denominada «Run Wizard» (Asistente de ciclos) (figura 5). Para acceder al Run Wizard (Asistente de ciclos), simplemente haga clic en el icono para «Run Project» (ejecutar el proyecto) en la barra de herramientas principal. Run Wizard – Creating a Run Template (Asistente de ciclos – Creación de una plantilla de ciclo) La primera vez que se utilice este software para analizar datos de QST*R será necesario crear una plantilla de ciclo. Esto se hace con «Run Wizard» (Asistente de ciclos). Para acceder al Run Wizard (Asistente de ciclos), simplemente haga clic en el icono «Run Project» (ejecutar el proyecto) en la barra de herramientas principal. • Asigne un nombre a la plantilla, p. ej., QSTR • Seleccione los valores de Panel, Size Standard (Estándar de tamaño), Standard Color (Color estándar) y Analysis Type (Tipo de análisis) como se ilustra en la figura 5 siguiente • Haga clic en «Save» (Guardar) para almacenar la plantilla para futuros análisis • Haga clic en «Next» (Siguiente) para continuar Figura 5: Ventana Run Wizard – Template Selection (Asistente de ciclos – Selección de plantillas) AN000GSES Rev.10/2013 Página 20 de 29 Run Wizard – Data Process (Asistente de ciclos – Proceso de datos) La ventana Data Process (Proceso de datos) de Run Wizard (Asistente de ciclos) permite al usuario seleccionar los parámetros de filtración de picos. Seleccione la configuración del análisis adecuada en la ventana Data Process (Proceso de datos) como se ilustra en la figura siguiente. Haga clic en «Next» (Siguiente) para continuar. Nota: El ajuste del rango del análisis del cuadro de análisis de datos sin procesar variará dependiendo del polímero utilizado durante la recogida de datos. El usuario deberá seleccionar un valor de punto de datos inicial que incluya el pico estándar de 75 pb de tamaño. Figura 6: Ventana Run Wizard – Data Process (Asistente de ciclos – Proceso de datos) Nota: Para datos del 3500, aumente el valor de Minimum Intensity (Intensidad mínima) a 150. Run Wizard – Additional Settings (Asistente de ciclos – Ajustes adicionales) La realización del análisis QST*R no requiere ajustes adicionales. Haga clic en «OK» (Aceptar) para continuar. AN000GSES Rev.10/2013 Página 21 de 29 Figura 7: Run Wizard – Additional Settings (Asistente de ciclos – Ajustes adicionales) Cuadro Data Processing (Procesamiento de datos) Después de hacer clic en «OK» (Aceptar) en el cuadro Run Wizard - Additional Settings (Asistente de ciclos - Ajustes adicionales), aparece el cuadro Data Processing (Procesamiento de datos) (figura 8). Los datos sin procesar se procesan y se mide su tamaño; a continuación, se aplican los parámetros de filtración y el panel QST*R seleccionado. Haga clic en «OK» (Aceptar) en el cuadro Data Processing (Procesamiento de datos) cuando el análisis haya terminado. Figura 8: Cuadro Data Processing (Procesamiento de datos) AN000GSES Rev.10/2013 Página 22 de 29 Ventana principal de análisis Ventana principal de análisisLa ventana principal de análisis (figura 9) de GeneMarker presenta un diseño intuitivo. Este diseño incluye: • La lista de archivos de muestra: en el lado izquierdo de la ventana. • La imagen de gel sintética: en la parte superior de la ventana. • Los electroferogramas de datos: debajo de la imagen de gel. • La tabla del informe: en el lado derecho de la ventana. En esta ventana es importante comprobar que se han identificado correctamente todos los picos pertinentes en cada perfil. Haga doble clic en cada una de las muestras del árbol de archivos de muestras en el lado izquierdo de la pantalla. Haga clic con el botón derecho en cada pico en cuestión y elija una de las opciones del cuadro de diálogo (p. ej., editar o eliminar alelo, confirmar o anular confirmación) según corresponda. Desde la ventana principal de análisis, seleccione la opción de menú desplegable «Applications» (Aplicaciones) en la parte superior de la pantalla. Seleccione «Trisomy Analysis» (Análisis de trisomías). Se abrirá el cuadro Trisomy Analysis Settings (Configuración del análisis de trisomías) (figura 10). Figura 9: Ventana principal de análisis AN000GSES Rev.10/2013 Página 23 de 29 Figura 10: Cuadro Trisomy Analysis Settings (Configuración del análisis de trisomías) Nota: Para datos del 3500, aumente el valor de Minimum Intensity (Intensidad mínima) a 150. Trisomy Analysis Settings (Configuración del análisis de trisomías) El cuadro Trisomy Analysis Settings (Configuración del análisis de trisomías) tiene dos fichas: • Ficha Analysis (Análisis) • Ficha Statistics Plot (Gráfico estadístico) Ficha Analysis (Análisis) La ficha Analysis (Análisis) ofrece opciones de ajuste de umbrales para el análisis de trisomías. Asegúrese de que esté seleccionado «BPG» en el cuadro de análisis y de que se muestren los siguientes ajustes. • Peak Height (Altura de pico) 50: Altura mínima de 50 para los picos que se vayan a identificar. (150 si se están utilizando datos del 3500) • Height Ratio (Razón de alturas) 30 %: Porcentaje máximo del pico principal al que debe llegar el segundo pico para que se puedan identificar dos alelos. • Quantification by Peak Area (Cuantificación por Área de pico). • Shorter Length/Longer Length (Longitud menor/longitud mayor) seleccionada. • Los umbrales de Trisomy Ratio (Razón de trisomías) son de 0,80 a 1,40. • Opción Apply Linear Correction (Aplicar corrección lineal) deseleccionada. Haga clic en «OK» (Aceptar). AN000GSES Rev.10/2013 Página 24 de 29 Ventana Trisomy Analysis (Análisis de trisomías) La ventana Trisomy Analysis (Análisis de trisomías) (figura 11) permite al usuario revisar datos de muestras QST*R, mostrar la razón de picos de cada marcador y acceder al informe GeneMarker. Hay una serie de pantallas que facilitan el análisis de datos al usuario: Dichas pantallas son: • Lista de muestras • Electroferograma • Gráfico de razones • Tabla de informe Figura 11: Ventana Trisomy Analysis (Análisis de trisomías) Para obtener más información sobre las características del análisis de trisomías y su uso, consulte el GeneMarker Manual (Manual del GeneMarker). AN000GSES Rev.10/2013 Página 25 de 29 Informe GeneMarker GeneMarker incluye una plantilla de informe compatible con los kits Elucigene QST*R. Para acceder al informe, haga clic en el icono «Print» (Imprimir) situado en la barra de herramientas de la parte superior izquierda de la ventana Trisomy Analysis (Análisis de trisomías). Esto abrirá la ventana Trisomy Print Settings (Ajustes de impresión de trisomías) (figura 12). Ventana Trisomy Print Settings (Ajustes de impresión de trisomías) La ventana Trisomy Print Settings (Ajustes de impresión de trisomías) contiene las opciones para incluir y visualizar datos de muestras en el informe GeneMarker. Seleccione las opciones como se ilustra en la figura 12. Asegúrese de que «Custom Size Range» (Rango de tamaño personalizado) esté ajustado a 98 pb (Start [Inicio]) y 510 pb (End [Fin]). Figura 12: Ventana Trisomy Print Settings (Ajustes de impresión de trisomías) AN000GSES Rev.10/2013 Página 26 de 29 Vista previa del informe GeneMarker Haga clic en «Preview» (Vista previa) para ver el informe GeneMarker (figura 13). Desde esta ventana, el usuario puede revisar e imprimir los datos de los picos de cada muestra correspondientes a todos los marcadores, para así obtener un sencillo informe de muestras de una o dos páginas. Figura 13: Informe GeneMarker El informe GeneMarker incluye lo siguiente: • Encabezado del informe: Contiene información sobre el análisis, el proyecto, la muestra y los parámetros. • Cuadro para la firma: Espacio para que los revisores del informe introduzcan la fecha y sus iniciales. • Electroferograma: De forma similar a la ventana Trisomy analysis (Análisis de trisomías), muestra los colores de todos los colorantes de la traza de la muestra. • Tabla del informe: Muestras los valores de los picos y marcadores seleccionados de la muestra actual. Las identificaciones de trisomías están resaltadas en gris. Se proporciona una columna de control adicional para las iniciales de los revisores. • Gráfico de razones corregidas: Contiene los puntos del gráfico de toda la serie de datos correspondientes a todos los marcadores del color del colorante. Las formas de los símbolos representan diferentes marcadores y pueden descifrarse empleando la fila «Symbol» (Símbolo) de la tabla «Report» (Informe). Los símbolos con relleno amarillo representan los puntos de datos de la muestra actual. Los símbolos con contorno rojo representan las identificaciones de trisomías. Nota: El gráfico de razones corregidas aparece en una segunda página para cada muestra solamente cuando se ha seleccionado Ratio Plot (Gráfico de razones) en el cuadro Trisomy Print Settings (Ajustes de impresión de trisomías). AN000GSES Rev.10/2013 Página 27 de 29 Etiquetas de los picos Las etiquetas de los picos se muestran con códigos de color de acuerdo con la calidad del emparejamiento entre el pico detectado y los «bines» del panel observados. En el electroferograma, los marcadores son barras grises horizontales y el centro de los picos está marcado con una barra gris vertical. Los picos que caen fuera de los marcadores o «bines» del panel se marcan en rojo para indicar que están «fuera de escala» («OL», Off Ladder). Nota: Las varianzas en el tipo de polímero y el estándar de tamaño del aparato podrían requerir la optimización de los «bines» de los marcadores para adaptar el software a las condiciones de ciclos que se estén utilizando. Puede encontrarse más información sobre cómo modificar los «bines» del panel en el GeneMarker Manual (Manual del GeneMarker) suministrado con el software. Puntuación del informe Para obtener unas pautas generales de análisis, consulte la sección «Analysis and Interpretation of Results» (Análisis e interpretación de los resultados) de las Instructions for Use (Instrucciones de uso) disponibles en el sitio web de Gen-Probe: www.gen-probe.com/global/products-services 1. La trisomía se determina por una de las dos condiciones siguientes: a) Dos picos de altura desigual debido a que uno de los picos representa dos alelos comunes a uno o a ambos padres. En este caso, la razón entre los dos picos se clasificará como 2:1 o 1:2. Donde A1/A2 dará un resultado en la región de 1,8 a 2,4 cuando el pico que represente el alelo de menor longitud tenga un área mayor que el pico que represente el alelo de mayor longitud, o donde A1/A2 dará un resultado en la región de 0,45 a 0,65 cuando el pico que represente el alelo de menor longitud tenga un área inferior a la del pico que represente el alelo de mayor longitud. b) Presencia de tres picos de altura similar. La razón de los picos se clasificará como 1:1:1 y sus valores caerán dentro del rango normal de 0,8 a 1,4 (aunque para los alelos separados por más de 24 pb es aceptable una razón alélica de hasta 1,5). Nota: Los marcadores estarán sombreados en gris en la tabla del informe si: • • La razón de picos de un marcador cae fuera de los límites predefinidos (0,8 y 1,4). Se detectan tres alelos. 2. Para interpretar un resultado como anormal, (esto es, con presencia de trisomía) es necesario que al menos dos marcadores informativos sean indicativos de un genotipo trialélico y que todos los demás marcadores sean no informativos. No se recomienda interpretar un resultado como anormal sobre la base de información de un solo marcador. 3. Para interpretar un resultado como normal, es necesario que al menos dos marcadores informativos sean indicativos de un genotipo dialélico y que todos los demás marcadores sean no informativos. Un resultado normal indica el complemento normal de dos de los cromosomas analizados. 4. Las razones de áreas de picos que caigan entre los rangos normal y anormal se clasifican como no concluyentes. Los resultados no concluyentes pueden resolverse utilizando los kits de cromosoma único. AN000GSES Rev.10/2013 Página 28 de 29 ELUCIGENE y QST*R son marcas comerciales de Gen-Probe Life Sciences Ltd. GENEMAPPER es una marca comercial de Life Technologies Corporation. GENEMARKER es una marca comercial de SoftGenetics Corporation. Nota al comprador: Licencia limitada Los polinucleótidos marcados con los colorantes VIC, NED y PET, así como su uso, podrían estar cubiertos por una o más de las patentes propiedad de Applied Biosystems, LLC. El precio de compra de este producto incluye derechos limitados, no transferibles, bajo determinadas reclamaciones de ciertas patentes propiedad de Applied Biosystems, LLC para usar solo esta cantidad del producto únicamente para actividades del comprador en detección de dianas dentro del campo del diagnóstico humano. No se transfieren otros derechos. Si desea información adicional sobre la compra de licencias en referencia a los colorantes anteriormente mencionados, póngase en contacto con el director de licencias en la dirección siguiente: Director of Licensing, Applied Biosystems, 850 Lincoln Centre Drive, Foster City, California 94404, EE.UU. Copyright 2013 Gen-Probe Life Sciences Ltd. AN000GSES Rev.10/2013 Página 29 de 29