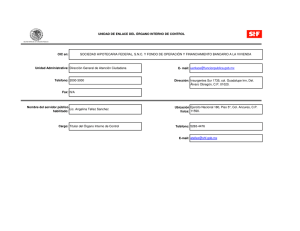
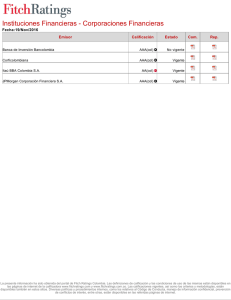
Tesis Doctoral Noelia Foresi
Anuncio

La vitalidad se revela no solamente en la capacidad de persistir sino en la de volver a empezar. Francis Scott Fitzgerald II AGRADECIMIENTOS - al Dr. Lorenzo Lamattina por enseñarme, acompañarme y darme la oportunidad de realizar esta tesis. - a la Dra. Claudia Casalongué por sus sugerencias y correcciones. - a la Dra Natalia Correa por todo, por su companía, por compartir sus conocimientos, por la gran ayuda en estos años. - a la gente del Laboratorio 8, por la comprensión, por el buen ambiente de trabajo. - al IIB y sus integrantes, por su generosidad - al CONICET y a la Universidad Nacional de Mar del Plata. - a las personas con la que colaboramos para que este trabajo pueda llevarse a cabo. Dra. Graciela Salerno, Lic. Gonzalo Caló, Dr. Gustavo Parisi, Dr.Jerome Santolini, Dr. Néstor Carrillo, Dra. Anabella Lodeyro y Lic.Martín Mayta. - a Nico por estar siempre a mi lado y por la familia que estamos formando. - a mi familia: mis hermanos, mamita, abuelas y a mi suegros - a mis amigos de lejos y de cerca. - a mi papá III ___________________________________________________________Índice ÍNDICE TEMÁTICO RESUMEN 1 INTRODUCCIÓN GENERAL 1. El óxido nítrico (NO). Propiedades físicas y químicas. 3 2. Biosíntesis de NO en animales y plantas 4 2.1. Biosíntesis de NO en animales. 4 2.2. Biosíntesis de NO en plantas. 4 2.2.1. Producción de NO a partir de L-arg. 4 2.2.2. Producción de NO a partir de NO2-. 5 2.2.3. Producción de NO a partir de poliaminas. 5 2.2.4. Producción de NO no enzimática. 5 3. Efectos biológicos del NO 6 3.1. Efectos biológicos del NO en animales. 6 3.2. Efectos biológicos del NO en plantas. 7 3.3. NO y estrés en plantas. 8 3.3.1. Estrés abiótico 9 Estrés hídrico. 9 Estrés salino. 9 Deficiencia de hierro. 9 Otros tipos de estreses abióticos. 10 3.3.2. Estrés biótico. 10 OBJETIVOS 12 RESULTADOS CAPÍTULO I: Estudio de la estructura y secuencia de la proteína Oxido Nítrico Sintasa de Ostreococcus tauri (OtNOS). Análisis filogenético de OtNOS. 13 I.1. Estudio de la estructura y secuencia de la proteína OtNOS. 14 i ___________________________________________________________Índice I.2. Análisis filogenético de la proteína OtNOS. 20 CAPÍTULO II: Estudio de las propiedades catalíticas y mecanismo de acción de la proteína Oxido Nítrico Sintasa de Ostreococcus tauri (OtNOS) expresada en forma recombinante. 28 II.1. Estudio de las propiedades catalíticas de OtNOS 29 II.2. Mecanismo de acción molecular de OtNOS. 34 II.3. Actividad de OtNOS expresada en bacterias E. coli. 37 CAPÍTULO III: Producción y función biológica del NO en cultivos celulares de Ostreococcus tauri. 41 III.1. Producción de NO en cultivos celulares de O. tauri. 42 III.2. Función biológica del NO en cultivos celulares de O. tauri 46 CAPÍTULO IV: Transformación de Nicotiana tabacum y Arabidopsis thaliana con el ADNc de OtNOS. 53 IV.1. Transformación de plantas de N. tabacum PH con el vector pCHF3:OtNOS. 53 IV.2. Transformación de plantas de A. thaliana con el vector pCHF3:OtNOS. 59 IV.3. Transformación de plantas de A. thaliana con el vector pBI-PEC:OtNOS. 62 DISCUSIÓN GENERAL 73 MATERIALES Y MÉTODOS. 78 1. Material biológico. 78 1.1 Bacterias. 78 1.1.1. Escherichia coli. 78 1.1.2. Agrobacterium tumefaciens. 78 1.2. Plantas. 78 ii _________________________________________________________Índice 1.3. Cultivo de Ostreococcus tauri. 78 2. Cebadores utilizados. 78 3. Construcciones realizadas. 79 3.1. pUC57OtNOS. 79 3.2. pET24bOtNOS. 80 3.3. pET15bOtNOSoxi. 80 3.4. pCHF3. 81 3.4.1 pCHF3:OtNOS. 81 3.5. pBI-PEC:GUS. 82 3.5.1. pBI-PEC:OtNOS. 82 4. Purificación de ADN plasmídico. 83 4.1. Electroforesis de ADN en gel de agarosa. 83 4.2. Estimación de la concentración de ADN. 83 5. Transformación de bacterias. 83 5.1. Obtención de células electrocompetentes y electroporación de A. tumefaciens. 83 5.1.1. Obtención de células electrocompetentes de A. tumefaciens. 83 5.1.2. Electrotransformación de A. tumefaciens. 84 5.2. Obtención de células competentes y transformación de E. coli. 84 5.2.1. Obtención de células CaCl2 competentes de E. coli. 84 5.2.2. Transformación de E. coli. 84 6. Expresión y purificación de proteínas en E. coli. 85 6.1. Expresión de OtNOS completa: 85 6.2. Expresión dominio oxi de OtNOS (OtNOSoxi). 85 7. Determinación de la actividad NOS. 86 7.1. El método de oxihemoglobina. 86 7.2. Formación de L-citrulina. 86 iii ___________________________________________________________Índice 7.3. Oxidación de NADPH. 87 8. Modelado por Homología. 87 9. Análisis filogenético. 88 10. Análisis estadísticos. 88 11. Números de Accesos. 88 12. Cuantificación de NO en cultivos de O. tauri y E. coli. 88 13. Análisis de la muerte celular en E. coli. 89 14. Extracción de ADN, ARN y proteína de O. tauri. 89 14.1. Extracción de ADN de O. tauri. 89 14.1.1 Verificación de la presencia del gen que codifican para OtNOS en O. tauri. 89 14.2. Extracción de ARN de O. tauri. 90 14.3. Síntesis del ADN complementario (Retrotranscripción). 90 14.4. Amplificación de ADN complementario específico mediante PCR 90 14.5. Extracción de proteínas de O. tauri. 90 14.5.1. Separación de proteínas mediante electroforesis en gel de poliacrilamida. 91 14.5.2. Transferencia e inmunodetección de proteínas en membranas de nitrocelulosa (Western Blot). 91 15. Generación de plantas transgénicas. 92 15.1. Transformación transiente de Nicotiana tabacum. 92 15.2. Crecimiento y transformación estable de las plantas de Nicotiana tabacum. 92 15.2.2. Esterilización de las semillas de N. tabacum. 93 15.2.3. Cultivo de N. tabacum en macetas. 93 15.3. Crecimiento y transformación estable de las plantas. de A. thaliana. 93 15.3.1. Crecimiento de A. thaliana. 93 iv ___________________________________________________________Índice 15.3.2. Transformación estable de A. thaliana. 94 15.3.3. Esterilización de las semillas de A. thaliana. 94 15.3.4. Selección de plantas transformantes de A. thaliana. 94 16. Análisis de las plantas transformadas. 95 16.1. Evaluación estadística 95 16.2. Extracción de ADN genómico a partir de tejido foliar. 95 16.3. Verificación de la presencia del transgén que codifican para OtNOS en las plantas transformadas. 95 16.4. Verificación de la presencia de la proteína OtNOS en las plantas transformadas. 95 16.4.1. Extracción de proteínas 96 16.5. Verificación mediante RT-PCR de la presencia del transcripto del transgén que codifica para OtNOS. 96 16.5.1. Extracción de ARN. 96 16.5.2. Estimación de la concentración y el grado de pureza del ARN. 96 16.5.3. Verificación de la integridad del ARN. 96 16.5.4. Tratamiento del ARN con nucleasas de ADN (ADNsa). 97 16.6. Cuantificación de nitritos. 97 17. PCR 97 18. TRABAJOS PRESENTADOS 98 19. REFERENCIAS BIBILIOGRÁFICAS 102 v ______________________________________________________Abreviaturas ABREVIATURAS ACE: elemento de control auto inhibitorio AmpR: marcador de selección que confiere resistencia al antibiótico ampicilina ATS: solución para Arabidopsis thaliana BCIP: 5-bromo-4-cloro-3-indolil-fosfato CaM: calmodulina CaMV: virus del mosaico del coliflor cGMP: GMP cíclico Col-0: Columbia 0 ADNsa: nucleasa de ADN eNOS: Óxido Nítrico Sintasa endotelial HGT: transferencia horizontal de genes GSNO: nitrosoglutatión GSNOR: nitrosoglutatión Reductasa GUS: β-glucuronidasa H4B: tetrahidrobiopterina iNOS: oxido nítrico sintasa inducible Intrón Cox 5c: intrón de la sub-unidad 5c de la citocromo c oxidasa de A. thaliana L-cit: L-citrulina MS-0: medio Murashige-Skoog MV: metil viológeno NBT: azul de nitrotetrazolio Ni-NOR: nitrito-NO reductasa nNOS: Óxido Nítrico Sintasa neuronal NO: óxido nítrico NOHA: N-ω-hidroxi-L-arginina NOS: óxido nítrico sintasa NOS-like: actividad enzimática similar a NOS de mamíferos NOSoxy: dominio oxigenasa de la Óxido Nítrico Sintasa NOSred: dominio reductasa de la Óxido Nítrico Sintasa NiNOR: nitrito NO reductasa NR: nitrato reductasa OtNOS: Óxido Nítrico Sintasa de Ostreococcus tauri PAGE: electroforesis en gel de poliacrilamida PEC: alelo corto del promotor del gen Hahb4 de girasol PH: Petit Havana vi ______________________________________________________Abreviaturas pLac: promotor del gen lacZ PM NR: nitrato reductasa unida a la membrana plasmática PTGS: silenciamiento génico post-transcripcional pUC ori: origen de replicación de la serie de vectores pUC pVS1 ori: origen de replicación del tipo Col E1 ROS: especies reactivas del oxigeno SA: ácido salicílico SAR: respuesta sistémica adquirida SNP: nitroprusiato de sodio RNS: especies reactivas del nitrógeno Rubisco: ribulosa-1,5-bisfosfato carboxilasa-oxigenasa SMC: sitio de múltiple clonado SN: sobrenadante T-ADN: ADN de transferencia tEF-1α: Factor de Elongación de N. tabacum Ter 35S: terminador transcripcional del gen 35S del virus del mosaico del coliflor Terminador NOS: señal de poliadenilación de la nopalina sintasa de A. tumefaciens THF: tetrahidrofolato tRBCs: secuencia terminadora del gen de la sub-unidad menor de la Rubisco TGS: silenciamiento génico transcripcional WB: Western Blot χ2: chi-cuadrado X-Gal: 5-bromo-4-cloro-3-indolil-β-D-galactósido vii _________________________________________________________Resumen RESUMEN El óxido nítrico (NO) se consideró, durante mucho tiempo, sólo como un contaminante ambiental. El descubrimiento en animales de la enzima que sintetiza NO, una Óxido Nítrico Sintasa (NOS) y el papel del NO como molécula señal regulando diversos procesos pato-fisiológicos, condujeron a estudiar sus efectos en plantas, incluyendo un posible rol en la respuesta a diferentes tipos de estreses. Los análisis de los genomas de plantas superiores no han revelado la presencia de genes homólogos a NOS descriptos previamente en animales. Por lo tanto, aún se desconoce la identidad génica de la actividad tipo NOS detectada en extractos vegetales. Sin embargo, luego de la secuenciación del genoma de Ostreococcus tauri, un alga verde unicelular marina perteneciente al reino Plantae, se encontró la primera secuencia con un alto porcentaje de similitud a las NOS de animales, en un organismo fotosintético. Mediante un análisis ‘in silico’ de la secuencia proteica de la NOS de O. tauri (OtNOS) se predijo su estructura terciaria y cuaternaria, y su posible origen evolutivo. Se demostró que la enzima OtNOS es una NOS genuina que comparte todas las características estructurales presentes en las NOS animales. En conjunto, las evidencias indican que O. tauri podría haber adquirido el gen OtNOS por un proceso de transferencia horizontal. El estudio y caracterización de OtNOS recombinante indican que es una enzima activa que presenta parámetros bioquímicos similares a las enzimas NOS de animales. Los resultados indican que la enzima OtNOS y la producción de NO participarían de la respuesta adaptativa de O. tauri a diferentes intensidades de luz. Por otro lado, la expresión de OtNOS recombinante en E. coli aumenta la tolerancia de la bacteria al estrés oxidativo. La transformación de plantas de Arabidopsis thaliana y Nicotiana tabacum con OtNOS bajo el promotor fuerte de CaMV:35S mostró un cambio en el fenotipo de las plantas transgénicas, acelerando la floración. Cuando la expresión de OtNOS estuvo bajo el control de un promotor inducible por ABA, NaCl y sequía, las plantas transgénicas presentaron un crecimiento normal sin estrés, y un mayor desarrollo radical en condiciones de estrés abiótico generado por los inductores antes señalados. Este trabajo de tesis presenta evidencias acerca del posible origen evolutivo y funciones de la enzima NOS en organismos fotosintéticos, y confirma la participación del NO en procesos esenciales de la fisiología de las plantas como el crecimiento y desarrollo de las raíces. 1 _________________________________________________________Resumen ABSTRACT Nitric oxide (NO) has been considered as an environmental pollutant for a long time. The discovery of the Nitric Oxide Synthase (NOS) enzyme, the biosynthetic NO pathway in animals and its central role in a broad spectrum of patho-physiological processes have led to develop studies on the NO effects in plants, including the putative NO role/s in different types of stresses. Analyses of higher plant genomes did not revealed the presence of genes with homology to animal NOS. Therefore, it’s still unknown the nature of the NOS-like activity described in plants. However, after sequencing the whole genome of the marine unicellular algae Ostreococcus tauri belonging to the Plant Kingdom, it was found the first NOS sequence with high similarity to animal NOS in a photosynthetic organism. In silico analysis of the structure of NOS from O. tauri (OtNOS) showed that it is a genuine NOS and it shares almost identical features with animals NOS. Phylogenetic studies suggest that the presence of OtNOS in O. tauri genome is due to horizontal gene transfer. OtNOS recombinant was expressed in E. coli as a soluble active enzyme and it showed biochemical parameters similar to animal NOS. The expression of recombinant OtNOS in E. coli confers the bacteria higher tolerance to oxidative stress. Results also indicate that the expression of OtNOS in O. tauri is associated to the adaptive response of the algae to different light irradiance. Transgenic Arabidopsis thaliana and Nicotiana tabacum plants expressing OtNOS under the control of the constitutive and strong promoter CaMV:35S exhibit a general accelerated development and early flowering. When Arabidopsis plants were transformed with OtNOS under the control of a stress inducible promoter, the transgenic plants showed normal development without stress and increased root growth under ABA, salinity and drought stress treatments. This PhD thesis contributes to the understanding of the possible evolutionary origin and functions of the NOS enzyme in photosynthetic organisms, and it confirms that NO is involved in essential plant physiological processes like root growth and development. 2 _______________________________________________________Introducción INTRODUCCIÓN GENERAL 1. El óxido nítrico (NO). Propiedades físicas y químicas El NO, óxido de nitrógeno (II) o monóxido de nitrógeno es una molécula simple que consiste en un átomo de oxigeno (O) unido a un átomo de nitrógeno (N) (Figura 1). Es una de las 10 moléculas estables más pequeñas que se conocen. Es un radical paramagnético, gaseoso, incoloro, de carga neutra, que posee gran afinidad hacia metales de transición (Fe, Cu, Zn) siendo capaz de formar aductos con ellos. Debido a que posee un electrón desapareado, tiene una gran reactividad química y reacciona - rápidamente con el radical superóxido (O2˙¯ ) para formar peroxinitrito (ONOO ) y posteriormente ácido peroxinitroso (ONOOH), que luego se descompone y forma el radical hidroxilo (OH˙¯ ) y NO2. Tanto el ONOO como el ONOOH, pueden dañar a las células al ser capaces de oxidar grupos tioles presentes en lípidos, lipoproteínas y ADN, entre otros (Wendehenne y col., 2001). El NO también puede reaccionar con O2 para formar varios óxidos de N y puede ser oxidado o reducido para formar las especies nitrosonio (NO+) o nitroxilo (NO-) respectivamente, importantes intermediarios en la química del NO (Gow e Ischiropoulos, 2001). El NO es soluble tanto en ambientes hidrofóbicos como hidrofÍlicos, aunque prefiere los primeros. Esta propiedad, junto a su alta difusividad en H2O (4.8 x105 cm2 s-1), le confiere la capacidad de atravesar fácilmente las membranas biológicas y en consecuencia difundir extra e inter-celularmente con relativa facilidad. Su vida media es corta (menos de un segundo) pero su gran reactividad química le confiere las características de un potente efector biológico (Bruckdorfer, 2005). Figura 1. Fórmulas y modelos moleculares del óxido nítrico. http://quimicausc.blogspot.com.ar 3 _______________________________________________________Introducción 2. Biosíntesis de NO en animales y plantas. 2.1. Biosíntesis de NO en animales En animales, la biosíntesis de NO es principalmente catalizada por la enzima NO sintasa (NOS; EC 1.14.13.39). Existen tres isoformas de NOS: dos constitutivas, NOS neuronal (nNOS) y NOS endotelial (eNOS) y una inducible (iNOS) (Alderton y col. 2001). Las enzimas NOS actúan como homodímeros catalizando la formación de Lcitrulina (L-cit) y NO a partir del sustrato L-arginina (L-arg) utilizando NADPH y O2 como cosustratos (Alderton y col., 2001). Además, requiere de los cofactores flavina mononucleótidos (FMN), flavina adenina dinucleótido (FADH) y tetrahidrobiopterina (H4B). Las NOS de animales poseen dos dominios: oxigenasa (conteniendo un centro hemo) y reductasa (conteniendo sitios de unión para NADPH, FAD y FMN). Ambos dominios están conectados por un sitio de unión a calmodulina (CaM) (Alderton y col., 2001). 2.2. Biosíntesis de NO en plantas En plantas, sin embargo, existen varias fuentes de producción de NO, enzimáticas y no enzimáticas. La producción enzimática de NO en plantas podría dividirse de acuerdo al sustrato: i) producción de NO a partir de L-arg ii) producción de NO a partir de nitrito NO2 (iii) Producción de NO mediada por poliaminas (Figura. 2). 2.2.1. Producción de NO a partir de L-arginina. La existencia de la actividad enzimática de tipo NOS en plantas (NOS-like) ha sido inferida mediante el uso de anticuerpos anti-NOS de animales (Kuo y col., 1995, Sen y Cheema 1995), detección de la actividad en extractos vegetales (Cueto y col., 1996, Ribeiro y col., 1999, Caro y Puntarulo 1999., Simontacchi y col., 2004., Corpas y col., 2009) y mediante el uso de inhibidores específicos (Cueto y col., 1996, Bright y col., 2006, Valderrama y col., 2007). A nivel subcelular, NOS ha sido asociada a peroxisomas (Barroso y col., 1999, Corpas y col., 2001), cloroplastos (Jasid y col., 2006), mitocondrias (Kaiser y col. 2007) y citoplasma (Ribeiro y col. 1999). Sin embargo, a pesar de los indicios sobre la existencia de NOS en plantas, aún no se ha identificado el gen o la proteína responsable de dicha actividad. Dos trabajos acerca de la identificación de la proteína NOS en plantas han sido retractados o fuertemente 4 _______________________________________________________Introducción cuestionados (Klessig y col., 2004, Zemojtel y col., 2006) entendiéndose claramente que es aún un tema sin resolver. 2.2.2. Producción de NO a partir de nitrito (NO2 ). En plantas la enzima nitrato reductasa (NR, EC 1.6.6.2) es una fuente importante de producción de NO a partir de nitrito NO2. Klepper (1990) descrbió que durante los tratamientos con herbicidas, las plantas de soja mutantes en NR emitían menores niveles de NO que las plantas salvajes. La capacidad de la NR de producir NO a partir de nitrito y NADH se ensayó in vitro (Yamasaki y col., 1999, Rockel y col., 2002) e in vivo (Rockel y col., 2002). Los ensayos indicaron que la producción de NO por la enzima NR resulta importante cuando las plantas se someten a condiciones de anoxia (Rockel y col., 2002). Otras enzimas también han mostrado capacidad de producir NO. Stöhr y col. (2001) detectaron producción de NO dependiente de nitrito en extractos de raíces de tabaco. La enzima responsable se encontraría unida a membrana plasmática y se la designó como nitrito-NO reductasa (Ni-NOR) (Stöhr y col., 2001). 2.2.3. Producción de NO a partir de poliaminas La participación de las poliaminas en la síntesis del NO es otro aspecto importante a ser considerado. Incrementos en los niveles de ciertas poliaminas (espermina y espermidina) inducen una liberación de NO en plántulas de A. thaliana. La producción de NO se detectó en la zona de elongación de la raíz y en las hojas primarias, especialmente en las venas y tricomas. Sin embargo la localización y el mecanismo de esta vía aún no sido dilucidado (Tun y col., 2006) 2.2.4. Producción no-enzimática de NO La producción no-enzimática de NO a partir de formas oxidadas del nitrógeno también ocurre en plantas. Los pigmentos carotenoides son capaces de producir NO a partir de NO2 -, en una reacción dependiente de la luz (Cooney y col. 1994). Además, se observó la generación de NO a partir de nitrito en el apoplasto de células de la aleurona bajo condiciones reductoras y a un pH de 5,5 (Bethke y col., 2004). La contribución de las distintas fuentes de NO en plantas dependería de la especie, célula o tejido involucrado, condiciones de crecimiento y desarrollo de las plantas (Neill y col., 2003; Gupta y col., 2011). 5 _______________________________________________________Introducción Por otra parte, el tipo de fuente de producción de NO, especie molecular generada: NO· (radical libre sin carga), NO+ (ión nitrosonio), NO- (ión nitroxilo) y localización subcelular podrían inducir una señal específica y desencadenar una respuesta fisiológica definida en plantas. ? Like Figura 2. Síntesis enzimática de NO y algunas reacciones químicas que contribuyen con el balance del NO en plantas. El NO se sintetiza a partir de L-arg por la actividad de la enzima Óxido Nítrico Sintasa (NOS). El signo de pregunta (?) indica que la enzima no ha sido aún identificada en plantas. El NO además puede ser producido a partir de nitrito catalizado por la enzima nitrato reductasa (NR) o a partir de poliaminas. Una vez producido, el NO puede - interactuar con superóxido (O2˙¯ ), formar peroxinitrito (ONOO ) y nitrar residuos tirosina (TyrNO2). El NO es además capaz de S-nitrosilar residuos cisteína (Cys-NO) presentes en proteínas. En presencia de oxígeno, el NO es oxidado a NO2, el cual reacciona con NO para dar N2O3 (Corpas y col., 2011). 3. Efectos biológicos del NO 3.1. Efectos biológicos del NO en animales El NO fue descubierto hace ya más de 200 años por John Priestley, pero recién hacia fines del siglo XX, se demostró que es una molécula clave en los sistemas biológicos. Furschgott y Zawadzki en 1980 descubrieron en animales que una sustancia formada en el endotelio podía relajar el músculo liso y la llamó EDRF (factor relajante derivado del endotelio). 6 _______________________________________________________Introducción Intensas investigaciones se realizaron en todo el mundo hasta que en el año 1992, se presentó al NO como la molécula del año en la revista Science (Koshland, 1992). En 1998 Furschgott, Murad e Ignarro, quienes descubrieron y describieron la actividad biológica del NO, fueron galardonados con el premio Nobel de Medicina. El NO se ha descripto con funciones en varios procesos biológicos tales como, por ejemplo mantener la presión sanguínea (Moncada y col., 1991), estimular las defensas en el sistema inmune (Snyder y Bredt, 1992), regular la neurotransmisión (Jeffrey y Snyder, 1995), entre otras numerosas funciones. 3.2. Efectos biológicos del NO en plantas Durante mucho tiempo el NO se consideró como un producto de las descargas eléctricas producidas en la atmósfera (Levine, 1984), y más recientemente un componente de los gases de combustión y contaminante atmosférico (Bruckdorfer, 2005). Por lo tanto, la investigación sobre los efectos del NO en plantas se concentró durante décadas en sus efectos tóxicos (Rowland y col., 1985; Leshem, 1988; Mc Kersie y Leshem, 1994). El descubrimiento de efectos benéficos en modelos animales condujo a numerosas investigaciones en plantas. Desde que se descubrió la relación entre el NO y la germinación hasta ahora, se han descripto la acción del NO en prácticamente todos los aspectos de la fisiología vegetal, convirtiendo al NO en una fitohormona no convencional. Entre sus principales efectos se pueden detallar: • Favorece la germinación. Semillas de lechuga que requieren luz para germinar cuando son tratadas con dadores de NO, tales como el nitroprusiato de sodio (SNP) y S-nitroso-N-acetilpenicilamina pueden germinar en oscuridad (Beligni y Lamattina, 2000). • En granos de cebada regula la muerte celular programada dependiente de ácido giberélico en la capa de aleurona, promoviendo la movilización de reservas para alimentar al embrión (Beligni y col., 2002b). • Favorece la des-etiolación. Es decir incrementa los niveles de clorofila en plantas que crecen en oscuridad (Beligni y Lamattina., 2000). • Promueve la cicatrización de heridas (Huang y col., 2004, París y col., 2007) a través de una comunicación cruzada con las hormonas ácido jasmónico y ácido salicílico. 7 _______________________________________________________Introducción • Permite el reconocimiento del polen por el estigma (Hiscock y col., 2007) y la reorientación del tubo polínico (Prado y col., 2004). • En tomate a bajas concentraciones es un componente esencial en la cascada de señales dirigida por auxinas que afecta la arquitectura radical y el desarrollo de raíces adventicias (Pagnussat y col., 2002, 2004). También puede inducir el desarrollo de raíces laterales (Correa-Aragunde y col., 2004). • En arveja, a bajas concentraciones promueve la expansión foliar, mientras que la inhibe a elevadas concentraciones (Quiao y Fan., 2008). • En soja es requerido para el desarrollo de nódulos funcionales que permitan la correcta fijación simbiótica de N2 (Leach y col., 2010) • Retrasa la senescencia de hojas (Leshem y col., 1998; Mishina y col., 2007) y frutos (Duan y col., 2007), la maduración de frutos (Manjunatha y col., 2010) y cuando es aplicado exógenamente reprime la floración (He y col., 2004). Este último efecto es opuesto al del etileno y sugiere que el balance de ambos gases sería clave en el establecimiento de la senescencia. • Participa en la vía de señalización que controla el movimiento de los estomas resultando fundamental en la regulación de la transpiración y el intercambio gaseoso necesario para la fotosíntesis (García-Mata y Lamattina, 2001, 2003). 3.3. NO y estrés en plantas La presencia de un electrón desapareado en la molécula del NO le confiere alta reactividad y también es el origen de su dualidad en su efecto biológico. A altas concentraciones, el NO es generalmente tóxico. Esto se debería a que - la combinación del mismo con pequeñas cantidades de anión O2˙¯ forma ONOO , un potente oxidante que puede causar daños en lípidos, proteínas y ADN (Goldestein y col., 2008). Sin embargo, durante la sobreproducción de ROS debido a un estrés, el NO tendría una función protectora eliminando el exceso de ROS y frenando la oxidación de lípidos mediada por radicales. Por ello, es posible que el NO participe en el sistema antioxidante de las células (Beligni y Lamattina., 1999a). Según lo expuesto, el NO posee diferentes reactividades bioquímicas para explicar su habilidad de actuar como mediador de vías de señalización o según su química ‘’per se” en funciones fisiológicas y en mecanismos de respuesta a enfermedades. 8 _______________________________________________________Introducción 3.3.1. Estrés abiótico Estrés hídrico La sequía es uno de los factores más importantes que limitan la productividad de los cultivos. Este estrés promueve la producción de NO (Gould y col., 2003). A su vez el NO aplicado externamente a través de un dador químico como el nitro prusiato de sodio (SNP) no sólo evita la pérdida de agua de las hojas, sino que también disminuye las fugas de iones y la tasa de transpiración, e induce el cierre de los estomas, aumentando con ello la tolerancia de las plantas al estrés hídrico (García-Mata y Lamattina 2001). El cierre de los estomas inducido por NO sería a través de la modulación del Ca2+ intracelular en las células de la guarda. Se ha informado que el NO activa selectivamente canales intracelulares de Ca2+ en células de la guarda a través de una vía de señalización dependiente de cGMP / cADPR (Garcia-Mata y col., 2003). Además, el NO también participaría del mantenimiento del agua en hojas de plántulas de trigo mediante la estimulación de la biosíntesis de ABA (Xing y col. 2004). El NO tiene la capacidad de aliviar el daño oxidativo causado por el estrés osmótico. Los niveles de anión O2˙¯ se reducen significativamente luego del tratamiento con el dador SNP, en microsomas aislados de ejes embrionarios de soja (Caro y Puntarulo., 1998). Estrés salino La participación del NO en la resistencia a estrés salino ha llamado mucho la atención en los últimos años. La función del NO en la tolerancia a la salinidad, se demostró en muchas especies de plantas. El pretratamiento SNP es capaz de proteger plántulas de arroz, resultando en un mejor crecimiento y viabilidad de las plantas bajo condiciones de estrés salino (Uchida y col., 2002). Las plantas mutantes de A. thaliana Atnoa con niveles reducidos de NO endógeno son más sensible al estrés por salinidad que las plantas salvaje (Guo y col., 2003; Zhao y col., 2007). El tratamiento con SNP de la mutante Atnoa logró aliviar el daño oxidativo causado por el NaCl (Zhao y col., 2007). El NO puede mejorar la tolerancia a la salinidad en las plantas mediante un incremento en la expresión del antiportador Na+/H+ y la H+-ATPasa unidos a la membrana citoplasmática, requeridos para la homeostasis de Na+ y la adquisición de K+ (Quiao y Fan., 2008). Deficiencia de hierro 9 _______________________________________________________Introducción El hierro es un cofactor esencial involucrado en procesos vitales como la respiración, fotosíntesis y fijación del nitrogeno (N). El NO posee una gran afinidad por el hierro y es capaz de evitar los síntomas de clorosis, alteración de cloroplastos y déficit de crecimientos observados durante la deficiencia del metal. Se ha hipotetizado que el NO aumenta la bio-disponibilidad del metal dentro de la planta, convirtiéndose así en una molécula clave en el sensado, homeostasis y disponibilidad del hierro por parte de las plantas (Graziano y col., 2002). Otros tipos de estreses abióticos. El NO incrementa la supervivencia de plantas de tabaco expuestas a altas temperaturas, probablemente disminuyendo los niveles altos de ROS causados por el calor (Quiao y Fan, 2008). También participa en la aclimatación y tolerancia al frío a través de la activación de genes de respuesta al frío y formación de fosfoesfingolípidos (Cantrel y col., 2011). El NO protege contra los efectos tóxicos de herbicidas como diquat, paraquat, glifosatos y atrazina, disminuyendo la toxicidad a través de efectos protectores sobre la membrana del cloroplasto, la pérdida de clorofila y aumentando las actividades de las enzimas antioxidantes tales como superoxido dismutasa, peroxidasa y glutatión reductasa (Beligni y Lamattina 1999b, Huang y col., 2002). Mackerness y col. (2001) informaron que plantas de A. thaliana producen NO y ROS cuando se exponen a radiación UV-B y también se observó un incremento en la actividad NOS de hipocótilos de maíz luego de la radiación UV-B (Zhang y col., 2003). Los flavonoides son compuestos que absorben UV-B y su concentración aumenta cuando las células vegetales se irradian con dosis elevadas de UV-B. Se demostró que el NO y los flavonoides son sistémicamente inducidos en las plántulas de maíz irradiadas con UVB (Tossi y col., 2012). El NO también confiere tolerancia a metales pesados como Cu, As, Cd y Al. Su efecto protector se debería a la reducción del estrés oxidativo causado por estos metales y a una disminución de la acumulación de los mismos en el interior de las plantas (Xiong y col., 2010). 3.3.2. Estrés biótico El NO es producido en respuesta a patógenos biotróficos, necrotróficos y virus. Se propone que el NO coordina un amplio rango de respuestas, desde la regulación de genes de defensa como la producción de hormonas durante el desarrollo de la 10 _______________________________________________________Introducción hipersensible (HR) (Asai y Yoshioka, 2009; Delledonne y col., 1998; Durner y col., 1998, Shi y col., 2008). Durante la HR el NO tiene un rol en la señalización junto a los ROS y el SA y participa en la muerte celular programada dando lugar a la condensación de la cromatina y la fragmentación del ADN (Clarke y col., 2000). El NO también participa en la señalización para la acumulación de fitoalexinas (Able, 2003) y en la activación de la respuesta sistémica adquirida (SAR) induciendo la producción de SA, necesaria para la activación de la proteína de defensa NPR-1 (Arasimowicz y Floryszak-Wieczorek, 2007). Para inducir SAR, el NO sería transportado intra e intercelularmente en el floema como nitroso glutatión (GSNO) (Durner y Klessig, 1999). Los cambios en los estado redox que ocurren durante la SAR regulan la actividad biológica de NPR1. La S-nitrosilación de NPR1 por GSNO induce la oligomerización de NPR1 y su translocación al núcleo (Tada y col., 2008; Lindermayr y col., 2010). A pesar de la gran cantidad de fenómenos biológicos regulados por NO, aún quedan por responder varios aspectos sobre la naturaleza de sus fuentes y producción en plantas. 11 _________________________________________________________Objetivos Objetivo general: Contribuir al conocimiento del origen evolutivo y funciones de la enzima Óxido Nítrico Sintasa (NOS) en organismos fotosintéticos. Objetivos particulares 1) A partir de la secuencia del gen y del ADNc correspondiente a la NOS del alga unicelular O. tauri se propone clonar la secuencia del ADNc, secuenciarla y caracterizar los diferentes dominios. Por otra parte realizar un estudio filogenético que permita identificar el origen evolutivo de OtNOS. Hipótesis 1: La secuencia génica correspondiente a OtNOS publicada en la base de datos NCBI es correcta. El gen codifica para una proteína NOS y se expresa en O. tauri. 2) Expresar la proteína OtNOS recombinante en E. coli y caracterizar su actividad. Dado que esta secuencia aún no ha sido analizada y no se sabe si corresponde a una NOS genuina, es de interés expresarla en forma recombinante y analizar sus parámetros bioquímicos. Hipótesis 2: El gen OtNOS codifica para una proteína NOS funcional en O. tauri. 3) Estudiar las funciones biológicas de OtNOS en O. tauri. Hipótesis 3: El NO generado a partir de OtNOS cumple funciones en el metabolismo celular del alga. 4) Realizar construcciones con el ADNc de OtNOS bajo el control de un promotor constitutivo y un promotor inducible por estrés para transformar plantas de Nicotiana tabacum y Arabidopsis thaliana. Analizar la regulación de la producción de NO bajo distintos tipos de estreses (salino e hídrico) y caracterizar los diferentes fenotipos que se obtengan. Hipótesis 4: La expresión regulada de OtNOS conferirá una cierta protección y tolerancia a diferentes tipos de estrés en plantas genéticamente transformadas. 12 ________________________________________________________Capítulo I Introducción En animales, se han identificado cuatro isoformas de proteínas NOS llamadas: NOS neuronal (nNOS), NOS endotelial (eNOS), NOS inducible (iNOS) y NOS mitocondrial (similar a la iNOS). Todas ellas tienen una identidad de secuencia entre 50% y 60% y poseen un dominio oxigenasa (NOSoxy) N-terminal y un dominio reductasa (NOSred) C-terminal. El dominio NOSoxy contiene un grupo hemo, tipo citocromo P-450 y un sitio de unión para el cofactor tetrahidrobiopterina (H4B). El dominio NOSred contiene sitios de unión para NADPH, FAD y FMN y posee alta homología de secuencia con la NADPH citocromo P-450 reductasa. Ambos dominios están conectados por un sitio de unión a calmodulina (CaM) que se encuentra entre NOSoxi y NOSred. La extensión Nterminal es característica de cada NOS y determina su localización intracelular. En su forma activa las NOS oxidan L-arg a L-cit y NO (Alderton y col., 2001). En bacterias gram positivas se ha identificado la presencia de una NOS cuya estructura está sólo constituida por el dominio oxigenasa. Dicha NOS recibiría los electrones necesarios para la catálisis desde otras reductasas celulares (Crane y col., 2010). Una excepción es la NOS encontrada en Sorangium celullosum la cual presenta además del dominio NOSoxi, un dominio NOSred en el N terminal (Agapie y col., 2009) (Figura 3). Figura 3. Dominios y organización de las proteínas Oxido Nítrico Sintasas. Las proteínas NOS se definen por la presencia de un dominio básico oxigenasa (azul) que une un grupo hemo y el cofactor H4B, es el centro catalítico de la enzima. En la mayoría de las bacterias este dominio oxigenasa es la proteína completa, y recibe los electrones de otras reductasas celulares. La única excepción conocida es la NOS de Sorangium cellulosum (scNOS, centro), que tiene un dominio amino terminal reductasa (rojo). En los metazoos (Metazoan NOS) la estructura es más compleja, desde el extremo amino al extremo carboxilo, comprende un dominio de isotipo de orientación específica (amarillo), un sitio de unión a Zn (celeste), que estabiliza el dominio oxigenasa (azul), un dominio de unión a calmodulina (CaM, negro) y un dominio reductasa (rojo). El dominio reductasa de scNOS es homólogo a un dominio ferredoxina reductasa/ ferredoxina de fusión, mientras que el dominio reductasa de la NOS de metazoos es homólogo al dominio reductasa del citocromo P450. Tanto scNOS y NOS de metazoos pueden recibir electrones desde NADPH . Adaptada de Sudhamsu y Crane. (2009). 13 ________________________________________________________Capítulo I Resultados I.1. Estudio de la estructura y secuencia de la proteína OtNOS. El objetivo principal de este Capítulo es demostrar mediante el análisis de secuencia primaria y de estructuras moleculares que el gen NOS encontrado en O. tauri (OtNOS) presenta un alto porcentaje de homología con las NOS hasta el momento caracterizadas. La secuencia del ADN y ADNc correspondientes a OtNOS se obtuvo a partir de la información del genoma completo de O. tauri publicado en 2006. La secuencia de aminoácidos se predijo y anotó como una enzima NOS (Derelle y col., 2006). Para identificar los dominios conservados en la secuencia primaria se utilizaron los programas informáticos Interpro y PFAM. Se encontró que OtNOS contiene los dominios característicos NOSoxy y NOSred (Sheta y col., 1994; Lowe y col., 1996). Estos resultados también se apoyaron por la predicción de estructura terciaria resultante de la asignación y uso de Hhpred y FFAS03. Se identificaron así, un dominio NOSoxy (AP código 1d0c con una puntuación de -289,00, una puntuación de menos de -9,5 lo cual es significativo) y un dominio NOSred (1tll código PDB con una puntuación de -132,00). La Figura 4a muestra el alineamiento entre las secuencias de aminoácidos de la enzima OtNOS (Derrelle y col., 2006) y las enzimas eNOS, iNOS y nNOS humanas. La similitud de secuencia entre los segmentos alineados de OtNOS y eNOS es del 44%, mientras que entre OtNOS e iNOS o nNOS es del 45%. En las secuencias completas el porcentaje de similitud de OtNOS es de 41,6; 42,7 y 34,3% para eNOS, iNOS y nNOS, respectivamente. Los sitios de unión del grupo hemo y cofactores H4B, FMN, FAD, y NADPH están altamente conservados en todas la secuencias alineadas (Figuras 4a y 4b). Los primeros 90 aminoácidos son diferentes con respecto a otras NOS a excepción del sitio de unión a Zn el cual está presente en OtNOS y contiene un motivo C-(x) 3-C (en el cual C es Cys y x es otro aminoácido). Este motivo es diferente del motivo de unión a Zn presente en las NOS animales (Figuras 4a y 4b). OtNOS presenta un sitio putativo de unión a CaM que no está totalmente conservado (Figuras 4a y 4b). Mediante un análisis de estructura con HHPred se observó que esta región es similar a la región de unión a CaM de nNOS de Gallus gallus (AP código 2o60, Evalor. 0,0036) (Söding y col., 2005). En la Figura 4a se muestra que las regiones involucradas en la 14 ________________________________________________________Capítulo I dimerización de proteínas NOS (I, II, III y IV) también se conservan en OtNOS (Bird y col., 2002). 15 ________________________________________________________Capítulo I Figura 4. Alineamiento de las secuencias aminoacídicas de OtNOS y NOS de humanos. a) Las secuencias OtNOS y NOS humana, eNOS, nNOS, iNOS se alinearon mediante los programas ClustalX y GENEDOC. Las cajas negras indican los residuos conservados en las cuatro secuencias; las cajas de color gris oscuro representan los residuos conservados en tres secuencias, y de color gris claro representan los residuos conservados en dos secuencias. Los aminoácidos que no comparten ninguna similitud no se sombrearon. Los sitios putativos de unión a cofactores como el Zn, el hemo, H4B, CaM, FMN, FAD pirofosfato, FAD isoalloxazina, NADPH ribosa, NADPH adenina, y el dominio C-terminal de NADPH se indican con su nombre. Las regiones que participan en la dimerización (I, II, III y IV) están encuadradas. b) Comparación en la arquitectura de los dominios de las diferentes NOS. Las enzimas NOS eucariotas tienen una región de unión a Zn, un dominio NOSoxy que une un grupo hemo, Larg, y H4B, una región de unión a CaM, y un dominio NOSred con subdominios que unen FMN, FAD y NAD. Las enzimas NOS bacterianas tienen sólo un dominio NOSoxy. El signo de interrogación indica que el motivo de unión a Zn no se conserva por completo. H4B/THF significa que aún no está claro a cual de los dos cofactores corresponde. Se realizó una predicción de la estructura terciaria utilizando el programa Modeller el cual predice un modelo estructural basándose en los mejores templados de proteínas cristalizadas. 16 ________________________________________________________Capítulo I Figura 5. Predicción de la estructura terciaria de los dominios de OtNOS. a) Diagrama de cintas del dominio OtNOSoxy según las coordenadas de la eNOS cristalizada de Bos taurus. Se muestra magnificado el sitio catalítico que contiene el grupo hemo, H4B y L-arg. b) Diagrama de cintas de la eNOS cristalizada de Bos taurus. c) Superposición de los residuos en el sitio activo de OtNOS (verde) y eNOS (rojo). Las uniones por puentes de hidrógeno se indican como líneas de puntos. d) Alineamiento estructural entre el modelo de OtNOSred (verde) y el nNOSred de Rattus norvegicus (rojo). OtNOS tiene un bucle CD2A (contribuye a la 2+ dependencia de Ca en la unión de CaM) más corto y carece de ACE (Elemento de Control Auto Inhibitorio). Las Figuras 5a y 5b muestran la estructura tridimensional del dominio OtNOSoxy basado en las coordenadas de eNOSoxy de Bos taurus. Se evaluó la calidad del modelo con PROSA II y dio lugar a un z-score de 29.06, lo cual indica que es similar al dominio oxi de la NOS de Bos Taurus (Wiederstein y Sippl, 2007). Los sitios de unión al sustrato en el dominio OtNOSoxy y eNOSoxy de B.taurus son idénticos (Figuras 5a y 5b, inserciones). Además, los residuos que interactúan con el sustrato L-arg, el cofactor H4B y grupo hemo están completamente conservados en la secuencia OtNOS (Figura5c). 17 ________________________________________________________Capítulo I Las NOS pertenecen a la familia de proteínas hemo Cys-coordinados en el que el ligando proximal al Fe del grupo hemo es un átomo de S de una Cys intrínseca (Rousseau y col., 2005). En OtNOS, el grupo hemo es estabilizado por unión al átomo de S de la Cys-125 (Figura 5c). El sustrato L-arg por encima del átomo de Fe se estabiliza por la unión que implica los aminoácidos Trp-301, Tyr-302 y Glu-306 que interactúan con el átomo de nitrógeno terminal de la L-arg y un átomo de oxígeno terminal de guanidinio y nitrógeno, respectivamente (Figura 5c). L-arg y H4B están unidos entre sí por uniones puente de hidrogeno entre dos grupos de propianato del hemo. A su vez el H4B se estabiliza por una interacción con el oxígeno del Trp-392 (Figura 5c). La predicción de estructura para el dominio reductasa de OtNOS (OtNOSred) también se conserva respecto a los dominios NOSred de animales. La Figura 5d muestra la superposición de OtNOSred y nNOSred en Rattus norvegicus. La Figura 5d indica dos diferencias principales entre los dominios; OtNOSred no presenta el elemento de control auto inhibitorio (ACE) que bloquea el flujo de electrones en ausencia de CAM, característico de la NOS constitutivas (eNOS y nNOS) cuya actividad se regula por Ca2+. Esta diferencia se pone de manifiesto también en la secuencia primaria (Figura 4a). El bucle CD2A contribuye a la regulación de la actividad de NOS por Ca2+, y es más corto en aquellas NOS cuya actividad es independiente de Ca2+. Dicho bucle también es más corto en OtNOS (Knudsen y col., 2003). La secuencia OtNOS contiene el dominio oxigenasa y reductasa unidos por un dominio de unión a CaM. La predicción de la estructura dimérica de OtNOS indica que OtNOS es capaz de dimerizar, tal como se describió para la eNOS de Bos taurus (Figura 6a y 6b). 18 ________________________________________________________Capítulo I Figura 6. Predicción de la estructura correspondiente al dímero de OtNOS. a) Diagrama de la estructura dimérica de OtNOS según las coordenadas de eNOS de Bos Taurus cristalizada. b) Diagrama de la estructura dimérica de eNOS de Bos taurus cristalizada. O. lucimarinus, otra especie del género Ostreococcus, también contiene un gen con alta homología a las NOS de animales; cuya expresión se validó por análisis de ESTs (Lanier y col., 2008). Considerando que la secuencia NOS de O. lucimarinus (OlNOS) sólo tiene un intrón (Lanier y col., 2008) y la predicción de la secuencia de OtNOS (Derelle y col., 2006) contiene un segundo micro-intrón (41 nucleótidos), se verificó si la secuencia OtNOS publicada corresponde a la OtNOS nativa o hay un error en la secuencia OtNOS cargada en la base de datos (GenBank/EMBL, número de acceso de OtNOS: CAL57731). En consecuencia se preparó ADNc de O. tauri y mediante cebadores específicos OtNOS2203Fw y OtNOS2537Rv (Materiales y Métodos, inciso 2, Tabla IV). Se amplificó la región de interés. Los resultados de la secuenciación del ADNc indicaron que existió un error de un nucleótido en la secuenciación del genoma de O. tauri que cambió el marco de lectura del gen OtNOS. Durante la predicción del ADNc de OtNOS, probablemente se consideró que correspondía a un intrón de 41 nucleótidos. Indicando que la predicción del segundo micro-intrón en la secuencia OtNOS situado entre las regiones de unión de pirofosfato FAD y FAD isoalloxazina, correspondientes a la posición de los aminoácidos 783 - 796, es incorrecta, resultando dichos nucleótidos parte de la secuencia exónica (Figura 7a y 7b). 19 ________________________________________________________Capítulo I Figura 7. Identificación de un error en la secuencia publicada del gen OtNOS. a) Alineamiento de la secuencia del ADNc de O. tauri amplificado con el cebador OtNOS2203Fw (OtNOSsec) y la secuencia del gen OtNOS correspondientemente publicada en GenBank/EMBL, número de acceso de: CAL57731 indicada en la Figura como OtNOSpub. El recuadro negro indica que el nucleótido G en la posición 2351 presente en OtNOSpub y no en OtNOSsec. b) Alineamiento de las secuencia de aminoácidos provenientes de la traducción a aminoácidos del producto de la secuenciación del ADNc de O. tauri (OtNOSsec), la secuencia de aminoácidos publicada de OtNOS (OtNOSpub) y la secuencia de aminoácidos de la NOS de O. lucimarinus (OlNOS). El recuadro negro indica la secuencia de aminoácidos deducida en OtNOSsec y en OlNOS y ausente en la secuencia de OtNOS publicada GenBank/EMBL (OtNOSpub). Los siguientes estudios se realizaron con la enzima OtNOS cuya secuencia corresponde a la publicada (Derelle y col., 2006; Foresi y col., 2010). El error en la secuencia no compromete ningún dominio crítico de unión a cofactores. I.2 Análisis filogenético de la proteína OtNOS. Las proteínas homólogas a la NOS de mamíferos se encuentran en todos los reinos de la vida (Figura 8). Las NOSs son muy abundantes en los organismos vivos. Sin embargo, poco se sabe acerca de la evolución de esta gran familia de genes. El origen 20 ________________________________________________________Capítulo I evolutivo de la NOS en metazoos y los eventos responsables de la especialización funcional y estructural de las diferentes isoformas siguen siendo una incógnita. Los análisis genómicos y funcionales indican que el gen NOS están presente en bacterias (Crane y col., 2010). La enzimas bacterianas son generalmente más pequeñas que las formas encontradas en metazoos, ya que sólo tienen el dominio oxigenasa, el cual es altamente homólogo al de mamíferos y utilizan reductasas celulares inespecíficas para la producción de NO (Gusarov y col., 2008). Las funciones biológicas de NOS en bacterias no están totalmente caracterizadas. Las NOSs bacterianas se han encontrado en más de 20 géneros de bacterias, incluyendo Bacillus, Staphylococcus y Deinococcus, Rhodococcus, Streptomyces y otras ( Wang y col., 2007; Adak y col., 2002a; Cohen y Yamasaki, 2003; Cohen y col., 2005; Gusarov y Nudler., 2005). Dichas formas procarióticas de NOS se han considerado como los precursores de la NOS de metazoos que se originaron durante la evolución por la fusión de un dominio oxigenasa a un dominio reductasa. Recientemente, se describió una NOS de la bacteria Sorangium cellulosum la cual contiene un dominio reductasa unido covalentemente, pero con una disposición diferente respecto a las encontrados en metazoos, sugiriendo la ocurrencia de eventos evolutivos independientes en los linajes procariotas y eucariotas (Agapie y col., 2009). Dentro de los metazoos, las mayorías de las especies de invertebrados tienen un único gen (Regulski y Tully, 1995; Yuda y col., 1996.; Nighorn y col., 1998; Luckhart y Rosenberg, 1999; Davies, 2000; Imamura y col., 2002; Scheinker y col., 2005; Godoy y col., 2006; Moroz y Kohn, 2007; Matsuo y col., 2008). 21 ________________________________________________________Capítulo I Figura 8. Árbol filogenético correspondiente a las NOS presentes en todos los Reinos de la vida. Adaptada de Gusarov y col. (2007). Este único gen presente en los invertebrados parece codificar una NOS constitutiva similar a la NOS neuronal de mamíferos, lo que sugiere que una función sináptica podría ser ancestral en la familia de la NOS. Por otra parte, el gen NOS del insecto Bombyx mori se expresa en bajo nivel en los tejidos larvales y está fuertemente inducido en los cuerpos grasos después de la infección bacteriana (Imamura y col., 2002). La NOS del cnidario Striata discosoma carece del elemento ACE responsable de la dependencia de Ca2+, típico de la NOS constitutiva, en apoyo a los antiguos orígenes del comportamiento inducible de la enzima (Moroz y Kohn, 2007). 22 ________________________________________________________Capítulo I En el Reino Protista, dos secuencia NOS han sido encontradas y caracterizadas en Physarum polycephalum, las cuales presentan un 82% de identidad entre si y corresponden a NOSs genuinas que se inducen durante la esporulación (Golderer y col., 2001). En este punto nos interesó conocer el posible origen evolutivo de la única NOS encontrada hasta el momento en organismo fotosintéticos. Se realizó una búsqueda mediante BLAST en la base de datos de proteínas no redundantes disponible en GenBank para recolectar putativos homólogos del gen OtNOS. Las secuencias recuperadas principalmente pertenecieron al super Reino Eucariota y al Reino Animalia. Se encontraron pocas excepciones, tal como una NOS en el protista (Amoebozoa) P. polycephalum, en la cianobacteria Synechococcus sp (S. sp) y una secuencia altamente homóloga en el alga unicelular O. lucimarinus. La Figura 9 muestra el árbol filogenético obtenido a partir de 1000 repeticiones de máxima probabilidad. Los números cerca de los nodos internos en la Figura 9 indican el apoyo relativo resultante de un test no paramétrico (bootstrap). Los nodos con valores iniciales de menos del 40% se descartaron. El árbol se puede dividir en dos partes principales. Un grupo principal contiene las tres isoformas NOS de organismos vertebrados (rojo, Figura 9). El patrón de agrupamiento representa la diversificación funcional de los tipos de NOS (eNOS, nNOS y la iNOS) (Figura 9). El segundo grupo contiene las secuencias NOS de invertebrados (filo Placozoa, Cnidaria, Artrópodos y Moluscos). En este grupo, también se encuentran representantes de las algas verdes (O. tauri y O. lucimarinus), bacterias (S. sp), y Amoebozoa (P. polycephalum). El análisis filogenético revela que la proteína OtNOS pertenece a un grupo que incluye las NOS de cianobacterias y Amoebozoa como un grupo fuera de los metazoos. Mientras que el patrón de agrupamiento entre estos organismos está bien soportado, su inserción en el grupo que contiene otros representantes de metazoos (color azul, Figura 9) no presenta el apoyo adecuado. Por lo tanto es difícil establecer profundas relaciones evolutivas entre las enzimas NOS dentro de este cluster. El estudió de la relación filogenética entre OtNOS y las secuencias conocidas de NOS sugiere que OtNOS ha evolucionado de un pariente cercano de la NOS de los animales. OtNOS aparece en un grupo bien soportado con Synechococcus sp y P. polycephalum, un representante de las cianobacterias y Amoebozoa, respectivamente. 23 ________________________________________________________Capítulo I Un hallazgo interesante fue que el gen NOS sólo está presente en uno de los 13 genomas de Synechococcus que han sido completamente secuenciados hasta el momento. De manera similar, una secuencia homóloga a la NOS de P. polycephalum no se ha encontrado en el genoma de Dictyostelium discoideum una especie estrechamente relacionada y completamente secuenciada (Messner y col., 2009). Figura 9. Árbol filogenético de las secuencias aminoacídicas de NOS. Representación radial del árbol filogenético. Los colores indican las principales divisiones taxonómicas: vertebrados (rojo), invertebrados (azul), plantas (verde), bacterias (amarillo) y Amoebozoa (azul claro). El número de acceso de cada secuencia se indica al lado del nombre de la especie. Los valores en las ramas indican el porcentaje de “bootstrap” por encima del 40%. Se estudió la posible transferencia horizontal (HGT) del gen OtNOS en Ostreococcus para lo cual se analizó el porcentaje de G+C en el genoma de O. tauri y se comparó con el porcentaje de G+C presentes en el gen OtNOS que se encuentra en el cromosoma 17. También se analizó el contenido de G+C de los genes adyacentes a OtNOS. 24 ________________________________________________________Capítulo I Se calcularon los porcentajes para dos genes NOS de P. policephalum (Figura 10). Los parámetros analizados no permitieron afirmar que se trató de un evento HGT. En apoyo a la posible transferencia horizontal del gen OtNOS existe el antecedente de que el gen NOS sólo esta en dos especies del genero Ostreococcus. Figura 10. Contenido de G+C en OtNOS, en los genes adyacentes al gen OtNOS y en el genoma de O. tauri. Como ejemplo de genes NOS que se postulan haber sido transferidos horizontalmente se indican los porcentajes de G+C de dos genes NOS identificados en el genoma de Physarum policephalum. 25 ________________________________________________________Capítulo I Discusión En la primer parte del Capítulo I se demostró que la secuencia predicha correspondiente a la proteína OtNOS (Derrelle y col., 2006) presenta alto porcentaje de similitud con las proteínas NOS de animales. Contiene los dominios característicos oxigenasa y reductasa unidos por un sitio de unión a CaM y conserva todos los sitios de unión a cofactores encontrados en las NOS anteriormente estudiadas. Con algunas excepciones que le confieren más interés para su estudio como por ejemplo, el sitio de unión a CaM el cual está parcialmente conservado. La presencia de las cuatro regiones implicadas en la interface de dimerización soporta la hipótesis de que la forma activa de OtNOS es un dímero (Bird y col., 2002). El dímero del dominio oxigenasa de OtNOS fue modelado y se asemeja al de la NOS de animales. El motivo C-(x) 4-C de unión a Zn está conservado en las secuencias NOS y es necesario para la estabilidad del dímero (Hemmens y col., 2000). El átomo de Zn está tetrahedricamente coordinado a dos C de cada subunidad NOS. En OtNOS encontramos en cambio, un motivo C-(x) 3-C. Un motivo C-(x) 3-C hasta ahora sólo ha sido descrito en las proteínas OtNOS y OlNOS. Sin embargo, el dedo de Zn presente en el dominio de la familia GATA de factores de transcripción es C-(x) 2-C (Aita y col., 2000), y el dominio de dedo de Zn de la citocromo oxidasa es un motivo C-(x) 3-C (Jaksch y col., 2001), indicando que el motivo C-(x) 3-C presente en OtNOS podría ser funcional, unir Zn y estabilizar el dímero de OtNOS. Además, los aminoácidos P y R que están presentes dentro del motivo (x) 3, entre los dos residuos de C en los supuestos dominios de unión de Zn [C-(x) 3-C] de las NOSs de Ostreococcus, se conservan entre la mayoría de las proteínas eNOS descritas hasta la fecha. No obstante, tanto la dimerización de las subunidades OtNOS como la unión de Zn, aún no se han verificado experimentalmente. En la segunda parte de este Capítulo se estudió el posible origen evolutivo de OtNOS, los resultados del análisis filogenético podrían sugerir que el gen NOS puede ser móvil dentro de los diferentes organismos unicelulares como resultado de HGT. HGT es considerado como una importante fuerza evolutiva que modula los genomas eucariotas (Keeling y Palmer, 2008). En O. tauri, dos cromosomas (2 y 19) son diferentes de los 18 restantes en términos de organización y contenido G + C. Se ha sugerido que todo el cromosoma 19 puede haber sido derivado de una fuente exógena por HGT (Derelle y col., 2006). 26 ________________________________________________________Capítulo I Un hallazgo interesante es que el gen NOS sólo está presente en Sy PCC 7335 uno de los 13 genomas de Synechococcus que han sido completamente secuenciados hasta el momento. Los altos niveles de diversidad de genes y la eficacia de eventos HGT se han postulado que pueden jugar papeles importantes en la diversidad de la población costera en el género Synechococcus (Páleník y col., 2009). Sy PCC 7335 parece ser un organismo unicelular inusual, ya que es la única cianobacteria capaz de fijar N2 sin la formación de heterocistos (Bergman y col., 1997). Será interesante estudiar el papel del NO dependiente de la actividad NOS en la cepa Sy PCC 7335. Un homólogo al gen NOS de Sy PCC 7335 se encontró en la bacteria de agua dulce Spirosoma linguale pero no así en otros organismos relacionados. No se ha podido validar la HGT del gen OtNOS en O. tauri por ser un organismo muy apto a la incorporación de nuevos genes y poseer un porcentaje de G+C altamente variable en su genoma. Se ha encontrado en O. tauri una gran cantidad de virus que modifican su genoma por la incorporación de nuevos genes. Un último estudio indicó que estos prasinovirus son especie específico (Clerissi y col., 2012). De todos modos, siendo que O. tauri comparte el ancestro con las plantas superiores no puede ser descartado un acontecimiento de pérdida de genes como una posible explicación de la ausencia de una NOS homóloga a OtNOS en las plantas (Archibald, 2009). Tampoco puede descartarse que un evento HGT muy temprano haya sido responsable de la presencia de OtNOS en el genoma de O. tauri y que durante la evolución del genoma de O. tauri haya ocurrido una homogenización del contenido de G+C en el cromosoma 17 que contiene el gen OtNOS. Las diferentes estructuras de los genes NOS están presentes en organismos terrestres y acuáticos. Sin embargo, actualmente no hay ninguna información para establecer una correlación entre los hábitats, la presencia y la estructura de genes NOS en diferentes organismos. Por lo tanto, sería muy interesante investigar las fuerzas evolutivas que relacionan los nichos ecológicos con la evolución molecular de la NOS y tratar de establecer, en la medida de lo posible alguna, correlación genómica funcional. 27 ________________________________________________________Capítulo II Introducción En su forma activa las NOS oxidan L-arg a L-cit y NO. En la Figura 11 se muestra un modelo de la enzima NOS y su regulación. Figura 11. Esquema de la estructura de la NOS y sus cofactores. Los electrones (e-) son donados por el NADPH al dominio reductasa y vía FAD y FMN al dominio oxigenasa donde interactúan con el Fe del grupo hemo y la H4B para generar L-cit y NO a partir de L-arg y O2. La CaM funciona como un puente molecular entre ambos dominios de la NOS permitiendo el paso de los electrones de las flavinas al Fe del grupo hemo. Adaptada de Alderton y col. (2001). En condiciones de baja disponibilidad de L-arg y/o H4B, las NOS también producen O2˙¯ y NO, dando lugar a la producción de ONOO-, que es citotóxico y el responsable de muchos efectos negativos del NO (Wendehenne y col., 2001). El sustrato L-arg es convertido en NO y L-cit por medio de dos reacciones de monooxigenación consecutivas (Figura 12) dentro del dominio NOSoxy de la NOS (Alderton y col., 2001). El intermediario N-ω-hidroxi-L-arginina (NOHA) generalmente se mantiene unido en el sitio activo durante la catálisis (Daff, 2010). La reacción comienzan con la unión de un O2 al hemo ferroso (Fe+2) formado el compuesto oxi-ferroso (Fe+2=O2). En el primer paso un sólo átomo de O2 se inserta en el sustrato. Dos electrones originados de la des-hidrogenación del NADPH son transportados a través de los co-factores FAD y FMN del dominio NOSred y pasan al hemo en el dominio NOSoxy para activar al di-oxigeno del Fe+2=O2 y formar Fe+2=O2-2 este último compuesto se forma solo en presencia del sustrato L-arg. Alli los electrones interactúan con el Fe del grupo hemo y el H4B en el sitio activo para catalizar la reacción del O2 con L-arg, generándose L-cit y NO como productos finales. 28 ________________________________________________________Capítulo II El segundo paso de síntesis del NO requiere solo un electrón, pero también involucra la activación del di-oxígeno (Griffith y Stuehr, 1995). Aunque existe mucha incertidumbre sobre el mecanismo exacto de acción molecular de NOS, hay bastante evidencia que apoya la formación de un complejo oxiferroso como agente mono-oxigenante, la reducción de este por un electrón de H4B y liberación del NO de un complejo Fe ferroso-NO por reducción de un radical H4B (Daff, 2010). Figura 12. Reacción catalizada por la enzima NOS. a) Esquema de transferencia de electrones a través de los cofactores presentes en la enzima NOS. b) L-arg es convertida en Lcit en una reacción de 2 pasos: oxigenación de L-arg a NOHA, seguido de la oxigenación de NOHA a L-cit y NO. La reacción requiere O2 y NADPH. Adaptada de Santolini (2011). Resultados II.1 Estudio de las propiedades catalíticas de la proteína OtNOS expresada en forma recombinante. La proteína recombinante OtNOS se expresó en E. coli y se detectó como una banda de 119 kDa en un gel de SDS-PAGE 12% (Figura 13, calle 2), de acuerdo con la masa molecular esperada según la secuencia de aminoácidos. No se detectó proteínas en 29 ________________________________________________________Capítulo II esta región del gel para las células de E. coli transformadas con el vector vacío (Figura 13, calle 1). OtNOS se purificó mediante cromatografía utilizando una columna de ADP agarosa, la cual retiene aquellas proteínas que tienen la capacidad de unir ADP. Esta columna cromatográfica ha sido utilizada en la purificación de otras NOS debido a la capacidad de estas proteinas de unir NADPH (Martasek y col. 1996). La proteína purificada migró como una única banda en un gel de SDS-PAGE (Figura 13a, calle 3) y exhibió inmunoreactividad con un anticuerpo anti-OtNOS (Figura 13b). Bajo las condiciones descriptas en Materiales y Métodos inciso 6, aproximadamente 1,5 mg de proteína recombinante OtNOS se lograron purificar a partir de 1 litro de cultivo de células E. coli BL21. Figura 13. Expresión de OtNOS recombinante en E. coli. a) Se transformó E. coli BL21 con el vector pET24b y pET24bOtNOS, se indujo la expresión con IPTG 0,5 mM durante 24 hs. Calle 1) Extracto total proteico bacteriano de un cultivo transformado con el vector vacío pET24b e inducido con IPTG; Calle 2) Extracto total proteico bacteriano de un cultivo transformado con el vector pET24bOtNOS e inducido con IPTG; Calle 3) proteína purificada mediante columna de ADP-agarosa. b) Western Blot correspondiente a la proteína purificada mediante columna de ADP-agarosa e identificada con un anticuerpo especifico anti-OtNOS. La actividad de la enzima OtNOS recombinante purificada se evaluó mediante dos métodos: la producción de NO por el ensayo de la oxihemoglobina y la formación de L-citrulina (Materiales y Métodos, incisos 7.1 y 7.2) A partir de el ensayo de la oxihemoglobina, se observó una fase de producción de NO (150 s de 0,49 +/- 0,03 µM NO.min-1), seguido por un largo período de lenta liberación de NO (33% de la fase inicial). Un resultado similar se obtuvo por el método de detección de L-cit (0,53 +/- 0,05 µM L-cit. min-1) en la reacción que contenía L-arg, el cofactor H4B, CaM y OtNOS (Tabla 1). 30 ________________________________________________________Capítulo II Tabla 1. Actividad de OtNOS recombinante purificada. La producción de NO se cuantificó mediante el ensayo de oxihemoglobina. La actividad se cuantificó durante 3 min a 25º C con oxihemoglobina 20 µM, DTT 5mM, L-arg 100 µM, NADPH 1 mM, CaCl2 10 mM, CaM 10 µM, H4B 100 µM, 100 U/ml de catalasa y OtNOS 0,5 µM o iNOS 3 comercial 0,5 µM (Sigma Aldrich). La formación de [ H] L-cit se cuantificó durante 30 min en 3 6 una reacción con [ H] L-arg 1 µCi (1.10 cpm), L-arg 50 µM, NADPH 100 µM, CaCl2 2 mM, CaM 10 µM, H4B 100 µM y OtNOS 0,5 µM o iNOS 0,5 µM. Formación de -1 Formación de NO.min % L-cit.min-1 % FAD, FMN 0,49 ± 0,03 100 0,53 ± 0,05 100 FAD, FMN, CaM 0,35 ± 0,01 71 0,37 ± 0,01 69 FAD, FMN, H4B < 0,001 < 1,0 < 0,001 < 1,0 0,10 ± 0,10 20 < 0,01 < 1,0 0,59 ± 0,13 120 0,41 ± 0,14 77 OtNOS (cofactores omitidos) OtNOS (en presencia del inhibidor) L-NAME iNOS (cofactores omitidos) FAD, FMN Los ensayos de actividad indican que la OtNOS recombinante contiene los cofactores FAD y FMN dado que la enzima retiene su actividad sin la adición de los mismos en la reacción, tal como fue descripto para eNOS bovina de origen recombinante (Martasek y col., 1996). En ausencia de CaM, la OtNOS retiene más del 70% de la actividad. La formación de NO y L-cit no se detectó cuando se omitió H4B. Así mismo se inhibió en un 80% por el análogo inactivo de L-arg, L-nitro arginina metilester (L-NAME) (Tabla 1). Los resultados obtenidos a partir de ensayos realizados con la iNOS comercial fueron comparables a los obtenidos para OtNOS (Tabla 1). Para validar la formación de L-cit se realizó una cromatografía de capa delgada (Figura 14) e inequívocamente se demostró que estamos en presencia de una actividad NOS genuina. 31 ________________________________________________________Capítulo II F Figura 14: Detección de L-cit como producto de la actividad OtNOS. a) Cromatografía de capa delgada (TLC) correspondiente a los productos de reacción en Tris-HCl 50mM pH 7.4 con L-arg 50 µM, [ H] L-arg 1 µCi (1.10 cpm), NADPH 100 µM, FAD 10 µM, CaCl2 2 mM, CaM 10 3 6 µM y H4B 10 µM en un volumen final de 40 µl. La reacción enzimática se inició por la adición de OtNOS 0,5 µM o H2O (control). Luego de 30 min a 25ºC, la reacción se detuvo con 40 µl de acetato de sodio 20 mM, pH 5,5, conteniendo EDTA 2 mM y EGTA 0,2 mM. Luego se sembró en la resina Dowex AG 50W-X8 (la cual retiene L-arg). La L-cit se eluyó por centrifugación y luego se separó por TLC. Los aminoácidos se tiñeron con ninhidrina (0,1 % etanol/ácido acético, en una relación 5:1 v/v). Los aminoácidos sembrados como estándar correspondieron a L-arg 0,2 mol, L-cit 0,2 mol y la mezcla de 0,1 mol de L-arg mas 0,1 mol de L-cit. Se estimó el factor de retención (RF) correspondiendo para L-arg: 0,21 y L-cit: 0,59. b) Los aminoácidos se removieron de las placas de TLC y la radioactividad se cuantificó por un contador de 3 3 centelleo líquido. Los valores se expresaron como la relación de [ H] L-cit / [ H] L-arg en la reacción que contiene o no la enzima OtNOS. La Km de OtNOS se estimó por dobles reciprocas mediante el ensayo de oxihemoglobina y fue de 12 +/- 5 µM (Figura 15). 32 ________________________________________________________Capítulo II Figura 15: Gráfica de dobles reciprocas. La producción de NO se cuantificó mediante el ensayo de oxihemoglobina. La reacción se realizó durante 3 min a 25ºC en presencia de oxihemoglobina 20 µM, DTT 5mM, NADPH 1 mM, CaCl2 10 mM, CaM 10 µM, H4B 100 µM, 100 U/ml de catalasa y OtNOS 0,5 µM. El ensayo se repitió con diferentes concentraciones L-arg (5, 50, 100 µM). Para cada concentración de L-arg se determinó la velocidad inicial de la reacción y se graficaron las dobles reciprocas. El NADPH es el dador inicial de electrones de NOS. Por lo tanto, el tercer método que se utilizó para medir la actividad de OtNOS se basó en cuantificar la tasa de oxidación de NADPH. Una oxidación basal de NADPH se detectó en ausencia de Larg (0,42 +/- 0,2 min-1) la cual se sustrajo en cada una de las mediciones. Una oxidación basal similar fue descripta para las mediciones de nNOS y eNOS (Heinzel y col., 1992; Martasek y col., 1996). En una reacción conteniendo L-arg y todos los cofactores, OtNOS 0,5 µM oxida 1,36 +/- 0,18 mM NADPH min-1. Se espera una relación de 1,5 NADPH oxidados por cada NO producido. Esta relación estequiométrica es característica de las NOS (Santolini, 2011). En el caso de OtNOS nosotros observamos una relación aproximada de 2,5 NADPH consumidos por cada NO producido, esto podría corresponder a un parcial desacople de ambas reacciones. La oxidación de NADPH por OtNOS fue completamente bloqueada en ausencia de H4B y por la adición de L-NAME. Se demostró que las enzimas NOSs pueden ser inhibida por una interacción del NO con el grupo hemo, actuando como mecanismo autoregulatorio (Alderton y col., 2001). 33 ________________________________________________________Capítulo II Es por ello que en la reacción también se agregó mioglobina la cual actúa como un secuestrante de NO y esto evita la inhibición por producto de la actividad NOS. En nuestro caso la omisión de mioglobina en la mezcla de reacción inhibió completamente la oxidación de NADPH. II.2. Mecanismo de acción de OtNOS Se analizó el mecanismo catalítico de OtNOS para lo cual se clonó el dominio oxi de OtNOS (OtNOSoxi). Se generó una construcción de OtNOSoxi fusionado a una señal de histidina y luego se realizó la expresión y detección mediante Western Blot. El dominio OtNOSoxi se identificó con el anticuerpo específico anti-OtNOS (Figura 16). A partir de 4 litros de cultivo se lograron purificar 40 mg de proteína con una pureza superior al 80% mediante una columna de afinidad de Níquel (Materiales y Métodos, inciso 6.2). Figura 16: Expresión de OtNOSoxi recombinante en E. coli. Se transformó E. coli BL21 con el vector pET15b y pET15bOtNOSoxi y se indujo la expresión con IPTG 1mM por 24 hs. Western Blot correspondiente a la proteína OtNOSoxi identificada con el anticuerpo específico anti-OtNOS. La masa molecular esperada del dominio OtNOSoxi, según la secuencia de aminoácidos es de 53 kDa. Calle1) Extracto total proteico bacteriano de un cultivo transformado con el vector vacío pET15b e inducida con IPTG. Calle 2) Extracto total proteico bacteriano de un cultivo transformado con el vector pET15bOtNOSoxi e inducidas con IPTG. 34 ________________________________________________________Capítulo II Para analizar el mecanismo catalítico de OtNOS se analizaron los espectros de absorción en el UV-visible. Los cambios en los picos de absorción corresponden a la transición electrónica de los diferentes estados de energía del grupo hemo y son indicativos del estado de oxidación y de la coordinación del Fe del grupo hemo. El dominio OtNOSoxy mostró un pico de absorción centrado en 405 nm, exhibiendo una mezcla de alto spin correspondiente al Fe del grupo hemo coordinado por 5 ligandos y spin bajo correspondiente al Fe coordinado por 6 ligandos. La adición de L-arg cambió el pico de absorción en 398 nm cambiando a alto el spin y tambien la coordinación del Fe del grupo hemo. No se observó cambio significativo en el spin de resonancia con el agregado de H4B. La banda α/β características de grupos hemo oxidados se observa a los 540 nm (Figura 17a). Este mismo comportamiento fue observado en NOS de expresión constitutiva de mamíferos (Chen y col., 1996). El espectro de absorción de OtNOS reducida con ditionito de sodio es diferente al de la mayoría de las NOS de mamíferos. OtNOS reducida presenta un pico alrededor de 422 nm y una banda α/β con dos hombros (530/558) con una fuerte banda en 558 nm, lo que podría sugerir una especie de spin bajo hexacoordinado. La banda a 650 nm desaparece con la reducción con ditionito de sodio (Figura 17 b). La adición de CO da lugar al bien descripto equilibrio p420/p450 (Gorren y Mayer., 2007). El pico principal se encuentra en 421 nm, y corresponde a un complejo penta coordinado FeIICO que ha perdido su ligando tiolato proximal. Un pico menor en 445 nm correspondió a las especies tiolato hexacoordinado de FeIICO. Este último complejo se convierte en el complejo pentacordinado FeIICO rápidamente. La reducción de OtNOS requiere una alta concentración de ditionito de sodio y gran tiempo de equilibrio similar a lo que ha sido observado en eNOS (Figura 17 b) (Sorlie y col., 2003). La unión del NO es poco eficiente y de baja afinidad. Se induce la degradación de OtNOSoxi probablemente por la S-nitrosilación del ligando proximal. En efecto, la reducción de la fracción de OtNOSoxi-NO conduce a una especie pentacordinado FeII NO (el NO se une fácilmente al complejo ferroso induciendo el desplazamiento del equilibrio termodinámico y la captura de FeIINO). La huella espectral fue similar a las de NOSs de mamíferos: FeIIINO exhibe un pico alrededor de 438 nm y una doble banda α/β a 545/581 nm. La especie FeIINO exhibió un pico en 396 nm y una sola banda de α/β alrededor de 570 nm. Sin embargo, en presencia de L-arg, el FeIIINO no pudo ser visto y el pico se observó a 395 nm con un pico adicional a 374 nm (dato no mostrado). La reducción por ditionito de sodio indujo la degradación de la proteína y 35 ________________________________________________________Capítulo II convirtió en una especie pentacordinada FeIINO (396 nm y una gran banda α/β en 570 nm) (Figura 17 c). OtNOSoxi fue sensible a altas concentraciones de NO. Probablemente una Snitrosilación pueda alterar la integridad del grupo hemo. Figura 17: Efecto de L-arg, H4B, CO y NO en el espectro de absorción de OtNOSoxi: a) Se estudió la unión del sustrato L-arg y el cofactor H4B a OtNOSoxi mediante el cambio espectal en UV-vis. Los espectros se realizaron después de dos ciclos de dilución / concentración de la proteína solubilizada en tampon kPI 0,1 M pH 7,4, L-arg 10 mM y H4B 80 µM. b) Reducción de OtNOSoxi por la unión de ditionito de sodio y CO. Se analizó el cambio espectral luego del agregado de ditionito de sodio 5 mM, en dos ciclos de reducción (10mM final) y CO mediante burbujeo en condiciones de anaerobiosis. c) Interacción de NO con OtNOSoxi. Se analizó el cambio espectral de OtNOSoxi luego del agregado de NO y la reducción con ditionito de sodio 5 mM en condiciones de anaerobiosis. 36 ________________________________________________________Capítulo II II.3. Actividad de OtNOS expresada en bacterias E. coli. El tercer objetivo de este Capítulo fue estudiar in vivo la actividad de OtNOS en E. coli que expresan OtNOS recombinante. La capacidad de producción de NO por parte de las células de E. coli transformadas con OtNOS se midió mediante el agregado de sustrato L-arg, el cual se añadió al momento de la inducción por isopropil tiogalactósido (IPTG). Luego, la producción de NO se cuantificó durante un período de 120 min. Las bacterias se preincubaron con la sonda 4-amino-5-metilamino-2',7'-difluorofluoresceina diacetato (DAF-FM DA) durante 20 min y se cuantificó la fluorescencia en un fluorometro. Las bacterias de E. coli transformadas con pET24b (vector vacío) o con el vector pET24bOtNOS no produjeron una cantidad significativa de NO sin el agregado de Larg (Figura 18a y 18b). La adición de L-arg al medio de cultivo de células transformadas con el vector vacío provocó una pequeña producción de NO comparándolo sin el agregado de L-arg. Sin embargo, las bacterias que expresan OtNOS recombinante produjeron hasta 2,5 veces más NO que las células control (Figuras 18a y 18b). El enantiómero inactivo D-arg no indujo la formación de NO en ningún cultivo analizado (Figura 18 b). Gusarov y Nudler (2005) demostraron que el NO confiere citoprotección a estrés oxidativo en bacterias. Para investigar la tolerancia al estrés oxidativo de las bacterias que expresan OtNOS, se analizó la inducción de muerte celular por la exposición a peróxido (H2O2.). La Figura 18c muestra que las bacterias transformadas con el vector vacío poseen un índice mayor de muerte celular (30%) luego del tratamiento con H2O2. Sin embargo, las bacterias que expresan OtNOS presentan una mayor viabilidad que las bacterias transformadas con el vector vacío, aún sin el tratamiento con H2O2. La proporción de muerte celular en las bacterias que expresan OtNOS es similar entre las tratadas y no tratadas con H2O2. Indicando que la protección por NO es independiente de H2O2 en células de E. coli. Como se muestra en la Figura 18d, las células transformadas con OtNOS presentaron mayores niveles de NO comparadas con las células controles. De todos modos, se midieron altos niveles de NO luego del tratamiento con H2O2, en ambas cepas transformadas con el vector vacío y con OtNOS, sugiriendo que el H2O2 promueve la producción de NO independientemente de la presencia de OtNOS recombinante. 37 ________________________________________________________Capítulo II Figura 18. Producción de NO y tolerancia a H2O2 en bacterias E. coli que expresan OtNOS recombinante. a) Los cultivos de E. coli se indujeron con IPTG en presencia o ausencia de 1 mM de L-Arg. La fluorescencia se determinó en un fluorómetro utilizando la sonda DAF-FM DA. Los datos se expresan como unidades arbitrarias por minutos con respecto a E. coli transformada con el vector vacío pET24b. Se muestra un gráfico representativo de tres experimentos independientes. b) Cultivos de E. coli inducidos con IPTG (que expresan OtNOS o transformados con un vector pET24b vacío) se trataron con 1 mM de L-arg o D-arg. La fluorescencia se determinó en presencia de DAF-FM DA durante un período de 2 hs. Los datos son expresados como número de veces, respecto al control. Letras diferentes indican diferencias estadísticamente significativas (ANOVA, P <0,05). Las barras de error indican el error estándar (n = 3). c) Los cultivos de E. coli se trataron con H2O2 30 mM durante 30 min en presencia de L-Arg 1mM. La muerte celular se determinó usando la sonda Sytox Green. Los valores se expresan como aumento de número veces con respecto al control. El asterisco indica diferencia estadísticamente significativa entre el tratamiento con H2O2 y el control (test de t, p <0,05). Las barras de error indican el error estándar (n = 5). d) El contenido de nitritos, como medida indirecta de NO se cuantificó con el reactivo de Griess, el NO espontáneamente se oxida a nitrito y este se acumula intra e intercelularmente. Letras diferentes indican diferencia estadísticamente significativa (ANOVA, P <0,05). Las barras de error indican SE (n = 3). 38 ________________________________________________________Capítulo II Discusión La proteína OtNOS recombinante producida en E. coli correspondió a una proteína soluble y funcional, con una actividad enzimática similar a la iNOS de macrófagos de ratón (Tabla 1; Stuehr y col., 1991). El valor de Km para L-arg de Ot NOS es de 12 +/5 µM, el cual está dentro del rango de Km de las enzimas NOS previamente caracterizadas (1 a 22 µM) (Bredt y Snyder, 1990; Stuehr y col., 1991; Roman y col., 1995; Gerber y col., 1997). No podemos afirmar si la enzima OtNOS es activa catalíticamente como un monómero o dímero. Algunos estudios indican que las eNOS en animales pueden ser activas en forma de monómero (Bredt y Snyder, 1990; Mayer y col., 1991), mientras que iNOS de macrófagos sólo es activa como un dímero (Stuehr y col., 1991). La presencia de una proteína del tamaño esperado del dímero OtNOSoxi en la Figura 16 indicaría una gran estabilidad de esta estructura cuaternaria de OtNOS. Sumado a esto la presencia de todos los sitios involucrados en la dimerización discutidos en el Capítulo 1, confirmarían la hipótesis de una estructura dimérica para OtNOS. La capacidad de unir CaM es muy importante en las enzimas NOS, dado que forma parte de su regulación catalítica. Por ejemplo, la iNOS de Macrófagos une CaM, incluso a muy bajas concentraciones de Ca2+. Por lo tanto, la actividad de la iNOS es Ca2+/CaM independiente (Mayer y Hemmens., 1997; Aoyagi y col., 2003). Por el contrario, la eNOS y nNOS cuyas actividades son reguladas por Ca2+ unen el complejo Ca2+/CaM cuando los niveles de Ca2+ son significativamente altos. Es por ello que estas enzimas son Ca2+/CaM dependientes. Los resultados obtenidos a partir de la medición de actividad y en el Capítulo 1 sobre la acción del segmento ACE que bloquea el flujo de electrones en ausencia de CAM y no está presente en OtNOS, sugieren que la actividad de la OtNOS es principalmente Ca2+/CaM independiente. En apoyo de este supuesto el segmento ACE tampoco esta presente en la iNOS y si está presente en la nNOS y eNOS (Salerno y col., 1997). El cofactor H4B se describió como esencial en la actividad de todas las NOS estudiadas en animales hasta el momento (Alderton y col., 1996). OtNOS mostró una dependencia total de H4B, ya que en ausencia de este cofactor no mostró actividad detectable para ninguno de los métodos utilizados. Cuando se analizó el mecanismo molecular no se observó un cambio significativo en el espectro correspondiente al estado oxidativo del Fe del grupo hemo de OtNOS luego del agregado de H4B probablemente, debido a una baja afinidad por el cofactor H4B. 39 ________________________________________________________Capítulo II La proteína eNOS bovina expresada en E. coli no tiene modificaciones posttraduccionales que se encuentran en la enzima nativa y es libre de H4B (RodríguezCrespo y col., 1996). En presencia de H4B, eNOS tiene un máximo de absorción en 400 nm que se desplaza a 395 nm cuando el sustrato L-arg es agregado. eNOS libre de H4B es completamente inactiva, pero la actividad catalítica se recupera cuando H4B es agregado a la reacción. Es de particular interés el hallazgo que muestra que eNOS libre de H4B existe en un equilibrio monómero-dímero muy similar a lo observado cuando la proteína es reconstituida con la adición de H4B. Los resultados establecen que el H4B influye en el entorno del hemo y estabiliza la proteína con respecto a la pérdida del hemo, pero no se requiere para la formación de dímero (Rodriguez-Crespo y col., 1996). Los resultados observados para el mecanismo catalítico de OtNOS se corresponden con los observados en eNOS, sin embargo lo que es mucho mas interesante es que el dominio reductasa es similar a una iNOS y su actividad es mayormente Ca2+/CaM independiente. En conclusión, nuestros datos indicarían que OtNOS es una quimera entre los dominios reductasa y oxidasa de una NOS inducible y una NOS constitutiva, respectivamente. En la última parte de este Capítulo analizamos in vivo la actividad de OtNOS. La expresión de OtNOS en E. coli generó un incremento en los niveles de NO en las bacterias. Se ha demostrado que el NO producido por las NOS bacterianas se requiere para mantener una tasa normal de crecimiento al principio de la fase estacionaria (Gusarov y col., 2008). La adición de H2O2 indujo un aumento en la producción de NO en E. coli transformadas con el vector vacío, sin embargo, este aumento de NO basal no logró proteger las células contra los efectos nocivos del H2O2. Dado que E. coli carece de NOS, la producción de NO después del tratamiento con H2O2 podría ser debido a la capacidad de ciertas NR para reducir nitritos a NO. La producción de NO por cultivos de E. coli ya ha sido informada previamente (Hutchings y col, 2000.; Corker y Poole, 2003). Por otra parte, el H2O2 es capaz de generar NO por vía no enzimatica a partir de L-arg o D-arg (Nagase y col., 1997; Gotte y col., 2002). En E. coli se describieron un conjunto complejo de 80 proteínas inducibles reguladas por un factor transcripcional llamado regulador redox SoxR frente a las respuestas a H2O2 y O2˙¯ (Greenberg y col., 1990; Liochev y col., 1994). Curiosamente en E. coli, el regulador SoxR se encontró que puede ser inducido por NO y conferir resistencia bacteriana a macrófagos múrinos (Nunoshiba y col., 1993). Los resultados permitieron concluir que OtNOS es activa en E. coli y sugieren que E. coli presenta todos los cofactores necesarios para la actividad de OtNOS. 40 _______________________________________________________Capítulo III Introducción O. tauri es un alga verde unicelular marina del clado Prasinophyceae que pertenece al grupo Chlorophyta del Reino Plantae y es un componente esencial de fitoplancton. Prasinophyceaes divergió tempranamente a partir de Chlorophyta y, en consecuencia del linaje verde (Bhattacharya y Medlin, 1998). O. tauri tiene una posición clave en el árbol filogenético de la vida eucariótica siendo un modelo potencialmente importante para diferenciar entre los procesos que son comunes a todos los eucariotas. O. tauri es la célula eucariota más pequeña de vida libre descripta hasta el momento. Posee un diámetro de no más de 1 µm. Por otra parte, O. tauri tiene un mínimo de organización celular, con un núcleo, un cloroplasto, y una mitocondria (Figura19) (Piganeau y col., 2006). Posee un pequeño genoma de 12,5 Mb completamente secuenciado y conocido (Derelle y col., 2006; Keeling, 2007). Tres diferentes ecotipos o especies potenciales de Ostreococcus se han definido en base a su adaptación a la intensidad de la luz. O. lucimarinus está adaptado a altas intensidades de luz y corresponde a una cepa aislada de la superficie del océano. El segundo ecotipo (RCC809) se definió como sensible a la luz e incluye la cepa aislada a los 100 metros de profundidad en la columna de agua. El tercer ecotipo (O. tauri) corresponde a la cepa aislada a partir de una laguna costera y se puede considerar como luzpolivalente (Six y col., 2007, 2008). Análisis comparativos de diferentes ecotipos de Ostreococcus sp ayudarían a comprender la colonización de diferentes nichos por los eucariotas unicelulares. Figura 19: O. tauri. a) Imagen de microscopía electrónica correspondiente a O. tauri. b) Imagen en dos dimensiones de una crioelectro- tomografía de O. tauri. N (núcleo), M (mitocondria), G (aparato de golgi) C (cloroplasto). c) Predicción de la estructura interna en tres dimensiones de O. tauri. Adaptada de Koning y col. (2009). 41 _______________________________________________________Capítulo III Resultados III.1. Producción de NO en cultivos celulares de Ostreococcus tauri. Para analizar si O. tauri produce NO en vivo, se incubaron suspensiones celulares con el fluoróforo específico de NO, DAF-FM DA. La producción de NO se midió durante 60 min con o sin el agregado de L-arg. La Figura 20a muestra que, dentro de los primeros 30 min, L-arg induce la producción de NO de una manera dosis dependiente (L-arg 0,5 a 5 mM), alcanzando la máxima producción de NO a una concentración de L-arg 5 mM (8 veces de inducción). Los inhibidores de la NOS de mamíferos L-NAME, G-monometil L-arginina (L-NMMA), y NG-nitro-L-arginina (L-NNA) se utilizaron para confirmar la fuente de producción de NO en las células de O. tauri tratadas con L-arg. La Figura 20b muestra que los inhibidores de NOS y el secuestrante de NO, cPTIO redujeron los niveles de NO luego del tratamiento con L-arg. Por otra parte, el antagonista de CaM trifluoperazina diclorhidrato (TFP) causó una reducción del 30% en la producción de NO y este resultado se correlacionó con la actividad de la proteína purificada OtNOS medida en ausencia de CaM (Tabla 1, Capítulo 2). 42 _______________________________________________________Capítulo III Figura 20. Producción de NO en O. tauri. a) La fluorescencia emitida por las células O. tauri (crecidas a 100 µmol.m .s de irradiancia de luz) se determinó usando la sonda DAF-FM DA -2 -1 en presencia de diferentes concentraciones de L-arg. Los datos se expresan en veces con respecto al control (unidades arbitrarias por minuto). Recuadro: gráfico representativo de tres experimentos independientes. La flecha indica el punto utilizado para el cálculo de la producción de NO. Los asteriscos indican una diferencia estadísticamente significativa (test de t, p <0,05). Las barras corresponden al error estándar (n = 3). b) La fluorescencia se determinó en un cultivo de células de O. tauri tratado con L-arg 5mM, en presencia y ausencia de los inhibidores de la NOS, L-NAME 10 mM, L-NMMA 10 mM, y L-NNA 10 mM, el secuestrante específico de NO, CPTIO 20 µM, o el antagonista de CaM, TFP 100 µM. Los datos se expresan como veces respecto al control (unidades arbitrarias por minuto). Letras diferentes indican diferencias estadísticamente significativas (ANOVA, P <0,05). Las barras corresponden al error estándar (n = 3). c) Células de O. tauri tratados o no con L-arg 5mM durante 20 min en presencia de DAF-FM DA. La producción de NO se visualizó mediante un microscopio de flourescencia (panel superior). El panel inferior corresponde a el campo claro de cada una de las imágenes (Materiales y Metodós, inciso 12). Barra = 50 µm. 43 _______________________________________________________Capítulo III El análisis de microscopía de fluorescencia mostró que la sonda DAF-FM DA (excitación 495 nm, emisión 515 nm) incremento la emisión de fluorescencia cuando las células se trataron con L-arg 5 mM comparando con el control (Figura 20c). También se observó fluorescencia roja, como autofluorescencia de la clorofila (Figura 20c). Por otra parte, el análogo inactivo de L-arg (D-arg) no fue capaz de inducir una respuesta como la observada para L-arg. El cPTIO redujo parcialmente los niveles de NO inducidos por L-arg corroborando por microscopía la cuantificación estimada con el fluorómetro (Figura 21). Figura 21. Producción de NO en O. tauri. Células de O. tauri tratadas con L-arg 5mM, D-Arg 5mM, CPTIO 20 µM y L-arg + cPTIO durante 20 min en presencia de DAF-FM DA. La producción de NO se visualizó mediante un microscopio de flourescencia (panel superior). El panel inferior corresponde a el campo claro de cada una del las imágenes. Barra = 50 µm. Con el objetivo de corroborar la fuente de producción de NO, se analizó el efecto del tungstato sódico, un inhibidor inespecífico de la enzima NR, sobre la producción de NO inducida con L-arg. 44 _______________________________________________________Capítulo III Figura 22: Producción de NO en un cultivo de O. tauri pre-tratado con tungstato sódico. La fluorescencia se determinó con la sonda DAF-FM DA en un cultivo de células pre-tratadas durante 15 min con diferentes concentraciones de tungstato sódico y luego tratadas o no con L-arg 5 mM. Los datos se expresan como número de veces respecto al control y corresponden a unidades arbitrarias por minuto. Letras diferentes indican diferencias estadísticamente significativas (ANOVA, P <0,05). Las barras corresponden al error estándar (n = 3). La Figura 22 muestra que el tungstato sódico en ningúna de las concentraciones probadas inhibió la producción de NO inducida por L-arg. Este resultado es otra evidencia que la fluorescencia detectada luego del agregado de L-arg correspondería a una actividad de tipo NOS y no a una actividad de NR. Se analizó la presencia y expresión del gen OtNOS mediante ensayos de PCR y RT-PCR. La Figuras 23a y 23b muestran la presencia del gen OtNOS en O. tauri y su expresión en condiciones basales ya que se detecta el ARN mensajero correspondiente (Figura 23b). Este resultado se corresponde con la detección de actividad NOS en estas condiciones. 45 _______________________________________________________Capítulo III Figura 23: Producto de amplificación del gen OtNOS a partir de ADN genómico y del ADNc por RT-PCR de O. tauri. a) Amplificación de ADN genómico de O .tauri. Calle 1) ADN genómico de O. tauri amplificado con los cebadores OtNOS632Fw y OtNOS632Rv (tamaño de amplificación esperado 632 pb. Calle 2) Control (-), control de reactivos. Calle 3) ADN genómico de Anabaena 7120 amplificado con los cebadores OtNOS632Fw y OtNOS632Rv. Calle 4) Control (+), ADN genómico de O. tauri amplificado con los cebadores Ot18SrFw y Ot18SrRv para el gen del ARNr 18S (tamaño de amplificación esperado 149 pb). b) Producto de amplificación del ARN de O. tauri por RT-PCR. Calle 1) RT-PCR amplificado con los cebadores OtNOS2203Fw y OtNOS2537Rv (tamaño de amplificación esperado 334 pb). Las secuencias de los cebadores utilizados se muestran en la Tabla 2, inciso 2 en Materiales y Métodos. III.2. Función biológica del NO en cultivos celulares de Ostreococcus tauri. Se analizó la producción de NO dependiente de L-arg en las distintas fases del cultivo y diferentes condiciones de irradiación de luz en cultivos de O. tauri. La Figura 24a muestra que la producción de NO fue mayor en la fase de crecimiento exponencial y rápidamente cayó al comienzo de la fase estacionaria. El cambio de radiación de luz de 40 µmol.m-2.s-1 a 100 µmol.m-2.s-1 provocó un aumento en la producción de NO, tanto en presencia como en ausencia de L-arg (Figura 24b). Mediante Western Blot utilizando un anticuerpo comercial anti-iNOS se observó que la adición de L-arg no modificó el nivel de la proteína OtNOS, mientras que el cambio de baja a alta irradiación indujo un aumento del 30% de OtNOS (Figura 24c). La especificidad del anticuerpo anti-iNOS contra OtNOS se confirmó por inmunoprecipitación (Figura 25). 46 _______________________________________________________Capítulo III Figura 24. En O. tauri la producción de NO depende de la irradiación de luz y la fase de crecimiento. a) O. tauri se cultivó con una irradiación de 40 µmol.m .s . El crecimiento del -2 -1 cultivo se determinó midiendo la densidad óptica a 660 nm. La producción de NO se determinó utilizando DAF-FM DA con o sin la adición de L-arg 5 mM. Los asteriscos indican una diferencia estadísticamente significativa (test de t, p <0,05). Las barras corresponden al error estándar (n = 3). b) Células de O. tauri crecidas a 40 µmol.m .s de irradiación de luz durante 10 días -2 -1 fueron sometidas a una irradiación de 100 µmol.m .s durante 24 hs. La producción de NO se -2 -1 determinó en presencia o ausencia de L-arg 5 mM usando DAF-FM DA. Los datos se expresan como número de veces (unidades arbitrarias por minuto) con respecto al control. Los asteriscos indican una diferencia estadísticamente significativa (test de t, p <0,05). Las barras corresponden al error estándar (n = 3). c) Análisis de la expresión de OtNOS en un cultivo de 2, 5 y 10 días de crecimiento a una irradiación de luz de 40 µmol.m .s y luego de un cambio a -2 -1 100 µmol.m .s por 3 días. En otro experimento, un cultivo de 10 días irradiado con 100 -2 µmol.m .s -2 -1 -1 se trató con o sin L-arg durante 1 h. La proteína OtNOS se detectó con un anticuerpo comercial anti-iNOS. Se incluyó como control de la carga de proteínas la banda correspondiente a RuBisCo (RbcL). La cuantificación relativa de la OtNOS y rbcL se realizó con el programa Image J y se muestra en el gráfico de barras. d) Células de O. tauri crecidas a 40 µmol.m .s se transfirieron a 400 µmol.m .s durante 1 h y luego se las regresó a 40 µmol.m -2 2 -1 -2 -1 - -1 .s durante 18 h. La producción de NO se analizó utilizando la sonda fluorescente DAF-FM DA. Los datos se expresan como número de veces (unidades arbitrarias por minuto) con respecto a las células cultivadas a 40 µmol.m .s . Letras diferentes indican diferencias -2 -1 estadísticamente significativas (ANOVA, P <0,05). Las barras corresponden al error estándar (n = 3). 47 _______________________________________________________Capítulo III La exposición de organismos fotosintéticos a una intensidad de irradiación de luz fotoinhibitoria resulta en un estrés oxidativo debido al desequilibrio entre la energía de la luz absorbida y el máximo de energía que puede ser utilizado en la fotosíntesis. Se analizó la producción de NO en un cultivo de O. tauri sometido a una irradiación fotoinhibitoria (cambio de 40 a 400 µmol.m-2.s-1). Un aumento significativo y repentino en la radiación de luz provoca en O. tauri una fotoinhibición la cual es reversible (Six y col., 2009). La Figura 24d muestra que la producción de NO en cultivos celulares de O. tauri expuestas a 400 µmol.m-2.s-1 es 1,75 veces en comparación con el cultivo que se mantuvo a 40 µmol.m-2.s-1. La producción de NO alcanzó niveles casi basales cuando el cultivo se retornó a las condiciones controles 40 µmol.m-2.s-1 durante 18 hs (Figura 24d). Figura 25. Inmmunoprecipitación de la proteína OtNOS utilizando el anticuerpo anti OtNOS. Los extractos de proteína totales de O. tauri fueron incubadas en ausencia (-) o presencia (+) del anticuerpo anti-OtNOS durante 18 hs. Las proteínas OtNOS acopladas al anticuerpo anti-OtNOS se inmunoprecipitaron utilizando una matriz de proteína A-Sepharosa. La fracción sobrenadante (SN) se recuperó. La presencia de OtNOS en el SN se analizó luego por Western Blot (WB) utilizando el anticuerpo anti-iNOS. La subunidad mayor de la RuBisCo (RbcL) se utilizó como control de carga, teñida con Ponceau. Se analizó el efecto del agente oxidante Metilviologeno (MV) o Paraquat, en la producción de NO en O. tauri. Estos compuestos son utilizados normalmente en agronomía como herbicidas. Ellos causan un exceso en la producción de ROS, dentro de los cloroplastos, sometiendo a la planta a un estrés oxidativo severo. En algunos estudios en algas se ha usado este herbicida para simular un estrés severo por luz. A partir de estas evidencias decidimos estudiar el efecto del MV sobre la producción de NO y ROS en O. tauri. 48 _______________________________________________________Capítulo III Figura 26. Producción de NO y H2O2 en células de O. tauri tratados con Metilviologeno (MV). a) La fluorescencia correspondiente al NO se determinó con la sonda fluorescente DAFFM DA en un cultivo de O. tauri tratado por 1 h en luz con diferentes concentraciones de MV. La fluorescencia correspondiente al H2O2 se determinó con la sonda fluorescente 2',7'dichlorodihydrofluoresceina diacetato (H2DCF DA) en un cultivo de O. tauri tratado por 2 hs en luz bajo las mismas concentraciones de MV. Los datos se expresan como número de veces (unidades arbitrarias por minuto) con respecto a las células sin tratar. Letras diferentes indican diferencias estadísticamente significativas (ANOVA, P <0,05). Las barras corresponden al error estándar (n = 3). b) Se analizó la fuente de producción de NO inducida por MV en cultivos de O. tauri con los inhibidores L-NAME 10 mM y tungstato sódico 100 µM. Los datos se expresan como número de veces (unidades arbitrarias por minuto) con respecto a las células sin tratar. Letras diferentes indican diferencias estadísticamente significativas (ANOVA, P <0,05). Las barras corresponden al error estándar (n = 3). En la Figura 26a, se observa que el MV indujo la producción de NO y H2O2, de manera dosis dependiente. Se evaluó la fuente de producción de NO, inducida por MV para la cual se midió la producción de NO en presencia del inhibidor especifico de NOS, LNAME, el inhibidor no logró inhibir el efecto del MV indicando que una fuente de producción de NO diferente a la NOS estaría involucrada (Figura 26b). El genoma de O. tauri presenta un gen con alta porcentaje homología al gen de la NR encontrada en algas y plantas superiores (GenBank/EMBL, número de acceso de NR: CAL56049.1). 49 _______________________________________________________Capítulo III NR es capaz de producir NO a partir de nitrito, y podría ser la fuente enzimática involucrada en la producción de NO frente a un fuerte estrés foto-oxidativo. La Figura 26b muestra que el tungstato sódico inhibió la producción de NO inducida por MV. Discusión O. tauri mostró un alto nivel de producción de NO durante la fase de crecimiento exponencial del cultivo. Dicha producción de NO puede aumentar mediante la adición exógena de L-arg. Varios estudios demuestran que la disponibilidad de L-arg es un factor importante en la producción de NO en animales (El-Gayar y col., 2003; Lee y col., 2003). La disponibilidad endógena de L-arg puede ser regulada por (1) aumento en la síntesis de “novo” de L-arg, (2) aumento en el transporte a través de la membrana celular, y (3) la reducción de la actividad de arginasas (Hallemeesch y col., 2002; Flores y col., 2008). Varios informes confirman que las microalgas excretan aminoácidos libres al medio (Martin-Jézequel y col., 1988; Penteado y col., 2009). Nuestros resultados demuestran que el aumento de la concentración de L-arg en el medio de cultivo incrementa la producción de NO en O. tauri. Esto podría estar relacionado con el desarrollo y la ecología de las floraciones de algas en el picofitoplancton (Mayali y Azam., 2004; Tillmann y col., 2004). El NO podría participar en diferentes niveles durante las sucesiones de las floraciones algales. La expresión de proteínas relacionadas con la muerte celular en la diatomea marina Skeletonema costatum es inducida por el NO, alta irradiación, y compuestos fotoinhibitorios (Chung y col., 2008). Vardi y col. (2006) demostraron que, en diatomeas marinas, los aldehídos promueven la producción de grandes cantidades de NO (a través de una actividad NOS) y la correspondiente muerte celular. Vardi y col. (2008) también demostraron que un alto nivel de la producción de NO fue el responsable de la reducción del crecimiento y el deterioro de la eficiencia fotosintética de la diatomea Phaeodactylum tricornutum. Varias líneas de evidencia indican que incrementos en los niveles de NO están estrechamente relacionados con la regulación de las floraciones de algas. Por lo tanto, una producción de NO dependiente de L-arg durante el ciclo de vida de O. tauri y la liberación de aminoácidos durante dichas floraciones podrían tener un impacto potencial en el ciclo del N en los océanos (Morel, 2008). Los organismos marinos fotosintéticos enfrentan cambios en el medio ambiente tales como los cambios de luz y temperatura. La irradiación de un organismo fotosintético con intensidad de luz fotoinhibitoria provoca la fotoinactivación del 50 _______________________________________________________Capítulo III fotosistema II (PSII) y genera una respuesta de estrés oxidativo que puede dañar biomoléculas. Como resultado de la fotoinactivación en O. tauri, la proteína D1 del PSII se degrada y se reemplaza a través de la síntesis de “novo” de D1 y el montaje del PSII (Six y col., 2009). En plantas superiores el NO es un potente antioxidante que ha demostrado proteger del daño fotooxidativo generado por herbicidas a los lípidos, proteínas incluyendo la proteína D1, y ácidos nucleicos (Beligni y Lamattina., 2002a). La inducción de la producción de NO en O. tauri tras la exposición a una intensidad fotoinhibitoria de luz permitiría reducir el daño oxidativo y promover la protección y/o reparación del aparato fotosintético. Por lo tanto, tenemos dos efectos posibles y opuestos del NO. Uno estaría relacionado con la inhibición del crecimiento, el deterioro de la actividad fotosintética y a la posible muerte celular durante las floraciones algales, tal como se ha descripto en Diatomeas. El otro se refiere a la capacidad antioxidante del NO y su efecto protector sobre las proteínas cloroplásticas asociadas a la fotosíntesis y al sostenimiento de los niveles de clorofila, cuando el organismo esta sometido a un fuerte estrés oxidativo. Este comportamiento dual hace referencia a la naturaleza química del NO y a su versatilidad en procesos biológicos. Una pregunta que aún queda por responder es cual sería el cofactor utilizado por la NOS de O. tauri en reemplazo de H4B, dado que este organismo carece de las enzimas involucradas en la biosíntesis de este H4B. En animales, el H4B es sintetizado a partir de GTP mediante tres reacciones enzimáticas (GTP ciclohidrolasa I, 6-piruvoiltetrahidropterina sintasa, y sepiapterina reductasa) (Thony y col., 2000). O. tauri carece de estas tres enzimas. Se ha postulado que las NOS bacterianas son capaces de utilizar otro cofactor llamado tetrahidrofolato (THF) (Adak y col., 2002b). En la Figura 27 se muestran las estructuras químicas de H4B y THF. Figura 27: Estructura química de los cofactores tetrahidrobiopterina (H4B) y tetrahidrofolato (THF) 51 _______________________________________________________Capítulo III O. tauri sintetiza THF y por lo tanto, puede ser que el THF sustituya al H4B en la síntesis de NO (Figura 28). No existen aún evidencias que confirmen o retracten esta hipótesis. , Figura 28. Biosíntesis de Folatos Diagrama de las enzimas y compuestos involucrados en la biosíntesis de folatos. En verde se indican las enzimas presentes en O. tauri. Los recuadros rojos indican la ruta de biosíntesis de H4B y de THF. Según la base de datos KEGG (http://www.genome.jp/kegg/pathway.html). 52 _______________________________________________________Capítulo IV Introducción Tras descubrir los importantes roles del NO en plantas, muchos investigadores comenzaron a buscar la presencia de una enzima NOS en plantas. Esta búsqueda ha sido larga, complicada e infructuosa, ya que la identidad de esta proteína permanece aún oculta. Aunque los proyectos genómicos no han arrojaron una secuencia con suficiente homología a la NOS de animales, varios candidatos se han analizados. Inicialmente, se describió en tabaco una proteína tipo NOS, inducible por patógenos, identificada como una variante de la proteína P del complejo glicina decarboxilasa de mitocondria. Sin embargo, no se pudo reproducir su actividad de manera confiable y el trabajo tuvo que ser retractado (Gas y col., 2009). Más tarde, a través de una búsqueda de mutantes de A. thaliana deficientes en la acumulación de NO en la raíz, se identificó el gen AtNOS1 que codifica para una proteína con homología de secuencia a una NOS hipotética del caracol Helix pomatia y que reacciona con anticuerpos anti-NOS de mamífero. Aunque ninguna de las dos proteínas presentó similitud a las NOS animales típicas, cuando se sobre-expresaron en E. coli produjeron un aumento de la síntesis de NO dependiente de L-arg. Sumado a esto, la evidencia de que plantas sin AtNOS1 tuvieron menor actividad NOS y menos NO, reforzó la idea de que era una verdadera NOS (Guo y col., 2003). Sin embargo, varios grupos demostraron recientemente que esta proteína no es una NOS per se, sino una GTPasa y estaría asociada de manera indirecta a la acumulación de NO (Zemojtel y col., 2006; Moreau y col., 2010). Por esta razón AtNOS1 fue renombrada como AtNOA1 o simplemente NOA (proteína asociada a NO). Dado que la producción de NO dependiente de una actividad NOS en plantas aún no está caracterizada, resultó de interés estudiar el comportamiento de plantas superiores que expresen el gen OtNOS que proviene de otro organismo fotosintético. Resultados IV.1. Transformación de Nicotiana tabacum con el vector pCHF3:OtNOS El primer objetivo del Capítulo fue realizar construcciones con OtNOS bajo el control de un promotor constitutivo para transformar plantas de N. tabacum (Petit Havana) y analizar en ellas la producción de NO. 53 _______________________________________________________Capítulo IV La transformación de las plantas con OtNOS y el análisis se realizó en colaboración con el Lic. Martin Mayta, la Dra. Anabella Lodeyro y el Dr. Néstor Carrillo del IBR, Universidad Nacional de Rosario, Argentina. Las plantas de N. tabacum PH (NtPH) se transformaron con la cepa de A. tumefaciens GV3101:pMP90 transformada con el vector pCHF3:OtNOS. La nomenclatura utilizada para las líneas transgénicas que expresan OtNOS citosólica es NtC (por N. tabacum citosólica), seguido de un número que indica los diferentes eventos de transformación. Como resultado de la transformación, se generaron 8 brotes transgénicos T0 independientes. Las plantas T0 mostraron un fenotipo de floración muy temprana con respecto a las plantas control NtPH (Figura 29a). Esto se observó desde la etapa de enraizamiento en caja Magenta, donde produjeron meristema de inflorescencia (Figura 29b). Como consecuencia, varias plantas no sobrevivieron la rusticación y murieron al poco tiempo de ser transplantadas en tierra. Por ello es que a partir de los 8 brotes que luego produjeron raíz verdadera, sólo 4 sobrevivieron y se extrajo tejido foliar para realizar los ensayos. Luego de 3,5 meses se cosecharon las semillas T1. Figura 29. Plantas de N. tabacum PH transformadas con OtNOS. a) Fotografía de las plantas T0 transformados con el vector pCHF3:OtNOS rusticados y creciendo en tierra. Las plantas transgénicas florecieron con un crecimiento vegetativo mucho menor que las plantas controles N. tabacum PH (NtPH). b) Brote transgénico en etapa de enraizamiento en caja Magenta. El asterisco indica la aparición temprana de un meristema de inflorescencia (ver recuadro amarillo). Se realizó la extracción de ADN genómico, para detectar la presencia del transgén OtNOS mediante PCR en las plantas T0 y líneas NtC5, NtC6, NtC7 y NtC8 (según se indica en Materiales y Métodos inciso 16.2). Las muestras de ADN fueron separadas por electroforesis en gel de agarosa al 0,7 % (p/v) para verificar la calidad de la extracción. Las condiciones de la reacción de PCR se describen en Materiales y 54 _______________________________________________________Capítulo IV Métodos. Los cebadores utilizados fueron OtNOS632Fw y OtNOS632Rv para amplificar una secuencia interna de 632 pb. Se incluyó un control negativo de PCR sin el agregado de templado y un control de amplificaciones inespecíficas empleando el ADN genómico de una planta N. tabacum PH sin transformar (control). En el control positivo el templado utilizado fue el plásmido pCHF3:OtNOS utilizado para transformar y que posee la secuencia codificante de OtNOS bajo control del promotor 35S del CaMV. Figura 30. Presencia del transgén en plantas de N. tabacum PH transformadas con pCHF3:OtNOS. Electroforesis en gel de agarosa al 2 % (p/v) para verificar mediante PCR la presencia del transgén OtNOS en las plantas T0. Se incluyó un control positivo (+) para el cual se utilizó el vector pCHF3:OtNOS y uno negativo (-) en el cual no se puso ADN. Los cebadores utilizados corresponden a OtNOS632Fw y OtNOS632Rv cuyas secuencias se indican en la Tabla IV inciso 2 en Materiales y Métodos. En la Figura 30 se observa que el fragmento del transgén de 632 pb se detectó en cada una de las líneas analizadas y en los controles negativos no se detectó ningún producto de amplificación. Determinación de la expresión de OtNOS en las plantas transgénicas. A partir de tejido foliar de las líneas NtC3, NtC5 y NtC6 se prepararon extractos de proteínas totales. Las proteínas se analizaron por SDS-PAGE, electrotransferencia e inmunodetección con anticuerpos comerciales anti-iNOS con el fin de analizar los niveles de proteína OtNOS. No se detectaron niveles de OtNOS en ninguna de las plantas analizadas (dato no mostrado). Se utilizó entonces un anticuerpo anti-OtNOS diseñado a partir de un péptido (RPQTPGTNDGPRIWC) presente en la secuencia OtNOS. Nuevamente no se evidenció la presencia de la proteína en ninguna de las líneas transgénicas analizadas (dato no mostrado). 55 _______________________________________________________Capítulo IV Debido a los resultados obtenidos se decidió evaluar mediante RT-PCR la presencia del transcripto OtNOS en las mismas plantas. Se extrajo ARN a partir de plantas NtPH sin transformar y de las plantas transgénicas T0. Mediante electroforesis en gel de agarosa al 1,5 % (p/v) se determinó la integridad del ARN total (Figura 31a). Los cebadores utilizados en la reacción de PCR fueron los mismos que los utilizados para analizar la presencia del gen OtNOS en las plantas transgénicas. Se incluyó el control negativo de PCR utilizando los mismos reactivos pero sin el agregado del templado y como control positivo se amplificó el gen OtNOS clonado en el plásmido pCHF3:OtNOS. Como control de contaminación con ADN genómico se realizaron reacciones de PCR a partir de muestras de ARN total de las líneas NtC5 y NtC6. Como control interno de la amplificación se usaron cebadores para el transcripto del gen tEF-1α (Factor de Elongación de N. tabacum). Figura 31. Análisis de la expresión del transgén OtNOS por RT-PCR en líneas transgénicas de NtPH. a) Electroforesis en gel de agarosa al 1,5 % (p/v) para determinar la integridad del ARN extraído de las plantas T0 de tabaco transformadas con pCHF3OtNOS. Se sembró 1 µg de cada muestra. b) Electroforesis en gel de agarosa al 2 % (p/v) de los productos de RT-PCR obtenidos a partir de ARN total proveniente de las líneas NtC5, NtC6, NtC7, NtC8. Los cebadores utilizados corresponden a OtNOS632Fw y OtNOS632Rv para amplificar el gen OtNOS y como control interno se utilizaron los cebadores Fw-tEF-α y Rv-tEF-α cuyas secuencias se muestran en Tabla IV, inciso 2 en Materiales y Métodos. En la reacción de PCR con los cebadores específicos para OtNOS se obtuvo un producto de 632 pb en todas las líneas transgénicas y en el control positivo, pero en el control NtPH, en los controles de contaminación con ADN genómico y en el control negativo no se obtuvo amplificación. La PCR con los cebadores para el control interno tEF-1α generó un producto del tamaño esperado (100 pb) en todas las líneas incluyendo el control NtPH. Por lo tanto el transgén OtNOS se expresó a nivel transcripcional en todas las líneas analizadas. 56 _______________________________________________________Capítulo IV Se realizó la estimación de los niveles endógenos de NO a partir de la cuantificación de nitritos provenientes del tejido foliar de las líneas NtC5, NtC5, NtC6, NtC7, NtC8 y NtPH. Se prepararon extractos según se indica en Materiales y Métodos inciso 16.6. Se cuantificó mediante el reactivo de Griess la concentración de nitritos como medida indirecta de los niveles de NO en las plantas. Sin embargo, no se detectaron diferencia estadísticamente significativa entre las líneas analizadas y el control NtPH (dato no mostrado). Análisis de segregación en la primera generación de las plantas NtPH transgénicas. Para estimar el número de loci en que se integró el T-ADN, se sembraron semillas T1 esterilizadas en placas de Petri con MS-0 con Kanamicina y se cultivaron en fitotrón. Se asumió que si la proporción de segregación KanR: KanS es 3:1, entonces el T-ADN se insertó en un solo sitio del genoma vegetal (Murgia y col., 2004). El recuento de plántulas resistentes y sensibles al antibiótico se realizó alrededor de los 30 días después de la siembra. La segregación 3:1 del gen marcador se evaluó estadísticamente por análisis χ2 con un nivel de significación de 0,05. NtC5 NtC6 NtC7 NtC8 χ2 observado Nº Pántulas Línea T0 (segregación 3:1) Total 191 243 217 252 R Kan 139 217 167 187 Kan 52 26 50 65 ¿χ observado <χ 2 (χ 0,05 = 3,84) 2 2 S 0,43 26,81 0,39 0,08 SI NO SI SI 0,05? Nº sitios de inserción del T-ADN 1 >1 1 1 Tabla II. Análisis de la segregación del gen de resistencia a Kanamicna en las plántulas T1. Para cada línea se indica el número total de plántulas que germinaron y cuántas fueron o no resistentes a Kanamicina. Las líneas NtC5, NtC7 y NtC8 segregaron con una relación 3:1 (Tabla II), sin embargo, la línea NtC6 no cumplió con la hipótesis de segregación 3:1 del gen de resistencia a Kanamicina, lo que implica que posee más de una inserción del T-ADN en su genoma. Transformación transiente de plantas de N tabacum PH con el gen OtNOS. 57 _______________________________________________________Capítulo IV Dado que no se logró detectar la proteína OtNOS en las plantas transgénicas y a que existen evidencias de un incremento en la estabilidad de las enzimas NOS en presencia del cofactor H4B (Klatt y col., 1995; Tzeng y col., 1995) sumado al hecho de que las plantas superiores al igual que O. tauri carecen de las enzimas involucradas en la biosíntesis de H4B. Se decidió expresar transientemente el gen OtNOS en NtPH en presencia del cofactor H4B. Para ello se realizó la coinfiltración con A. tumefaciens transformado con el vector pCHF3:OtNOS y el cofactor H4B de acuerdo a la metodología estándar ( Materiales y Métodos, inciso 15.1). Con el objeto de identificar si las plantas agroinfiltradas expresaron el transgén OtNOS, se analizó la presencia del transcripto en el tejido foliar mediante RT-PCR. Se extrajo ARN a partir de plantas transformadas con el vector vacío pCHF3 y con el vector pCHF3:OtNOS en presencia o ausencia del agregado exógeno del cofactor H4B. Figura 32. Análisis de la expresión del transgén OtNOS por RT-PCR en plantas NtPH transformadas de manera transientes. a) Electroforesis en gel de agarosa 1 % (p/v) para determinar la integridad del ARN extraído de las plantas NtPH agroinfiltradas. La nomenclatura utilizada corresponde a pCHF3:OtNOS para plantas NtPH agoinfiltradas con A. tumefaciens transformado con el vector anteriormente mencionado; pCHF3:OtNOS /H4B, plantas NtPH coinfiltradas con A. tumefaciens transformado con el vector pCHF3:OtNOS y con el cofactor H4B y pCHF3, plantas NtPH agroinfiltradas con A. tumefaciens transformado con el vector vacío pCHF3. Las extracciones de ARN se realizaron luego de 1 y 3 días de realizada la agroinfiltración. b) Electroforesis en gel de agarosa al 1% (p/v) de los productos de RT-PCR obtenidos a partir de ARN de plantas NtPH agroinfiltradas como se detalla anteriormente. Los cebadores utilizados corresponden a OtNOS632Fw y OtNOS632Rv cuyas secuencias se muestran en la Tabla IV inciso 2 en Materiales y Métodos. La figura 32b muestra el resultado de RT-PCR. Se observa el producto de amplificación tanto en presencia como en ausencia de H4B. Los resultados indican que 58 _______________________________________________________Capítulo IV el transcripto OtNOS está presente en las plantas agroinfiltradas, indicando que el gen se estaría expresando correctamente. Nuevamente se extrajo proteínas de cada una de las plantas y se realizó un Western Blot, utilizando el anticuerpo específico anti-OtNOS y el anticuerpo comercial anti-iNOS. En ninguno de los dos casos analizados se logró detectar la proteína (dato no mostrado), sugiriendo que el H4B no logró estabilizar la proteína o mejorar su detección en nuestras condiciones experimentales. IV.2. Transformación de plantas de A. thaliana con el vector pCHF3:OtNOS. Con la intención de estudiar la expresión de OtNOS en el sistema experimental modelo de A. thaliana, un total de 6 plantas T0 ecotipo Col-0 y 12 plantas Col-0 rdr6 se transformaron mediante la técnica de “floral dip” (Clough y Bent, 1998), con la cepa de A. tumefaciens GV3101:pMP90 que posee el vector pCHF3:OtNOS. Se utilizaron plantas Col-0 rdr6, pues permiten obtener niveles más altos de expresión de los transgenes (Butaye y col., 2004) ya que son deficientes en el silenciamiento posttranscripcional (PTGS) (Luo y col., 2007). Luego de 4 semanas se cosecharon las semillas T1, según las condiciones de crecimiento descriptas en Materiales y Métodos inciso 15.3.2. La nomenclatura utilizada para las líneas transgénicas de A. thaliana que expresan OtNOS en citosol de manera constitutiva fue AtC (por A. thaliana y citosólica), seguido de un número que indica diferentes eventos de transformación o líneas. Selección de plantas transgénicas A. thaliana por resistencia a Kanamicina Se plaquearon entre 150 y 300 semillas T1 estériles por placa de Petri en MS-0 suplementado con Kanamicina 50 µg/ml. Las plántulas transformadas que expresaron el gen NPTII resistieron la presencia de Kanamicina y desarrollaron las primeras hojas verdaderas. Sin embargo, las semillas sin transformar germinaron y quedaron en el estadio de dos cotiledones completamente cloróticos sin llegar a desarrollar hojas verdaderas. Luego de 3 semanas, cada una de las plantas T1 resistentes a Kanamicina (KanR) se transfirieron a tierra. 59 _______________________________________________________Capítulo IV Figura 33: Floración en líneas de A. thaliana transformadas con pCHF3OtNOS. Fotografía de algunas líneas de A. thaliana Col-0 rdr6 transformadas con el vector pCHF3:OtNOS. La floración fue más rápida en las plantas transgénicas (OtNOS) que en las control (rdr6 wt). AtC1, AtC5, AtC8, AtC13, AtC17, AtC20, AtC23: distintas líneas de A. thaliana transformadas con OtNOS. rdr6 WT corresponde a las plantas de A. thaliana deficientes en silenciamiento (sin transformar). Las plantas transformadas con el pCHF3:OtNOS mostraron un fenotipo de floración temprana (Figura 33) aunque mucho menos pronunciado que las plantas NtPH transformadas con OtNOS (Figura 29). También se observó que algunas plantas produjeron silicuas pequeñas, sin semillas (dato no mostrado). Análisis de la presencia del transgén de OtNOS en la generación T1 de A. thaliana Col-0 rdr6 transformadas con el pCHF3:OtNOS. Se realizó la extracción de ADN genómico, según se indica en Materiales y Métodos inciso 16.2, para detectar la presencia del transgén de OtNOS mediante PCR en las plantas AtC1, AtC5, AtC8, AtC13, AtC17, AtC20 y AtC23 de la generación T1. Se incluyó un control negativo sin el agregado de ADN templado y un control de bandas inespecíficas empleando ADN genómico de una planta Col-0 rdr6. En el control positivo el templado utilizado fue el plásmido pCHF3:OtNOS. 60 _______________________________________________________Capítulo IV Figura 34. Presencia del transgén OtNOS en plantas de A. thaliana transformadas con pCHF3:OtNOS. Electroforesis en gel de agarosa al 2 % (p/v) para verificar mediante PCR la presencia del transgén OtNOS en las en las líneas AtC de la generación T1. Se incluyó un control positivo (+) para el cual se utilizó el vector pCHF3:OtNOS y uno negativo (-) en el cual no se puso ADN. Los cebadores utilizados corresponden a OtNOS632Fw y OtNOS632Rv cuyas secuencias se muestran en la Tabla IV, inciso 2 en Materiales y Métodos. Los productos de amplificación se analizaron en gel de agarosa al 2 % (p/v). Se obtuvo una amplificación del tamaño esperado de 632 pb para cada una de las líneas analizadas lo que demuestra la presencia del transgén OtNOS. Las plantas Col-0 rdr6 no evidencian la presencia de dicho amplicón (Figura 34). Análisis de segregación del gen OtNOS en la generación T2 de las plantas transgénicas. Las líneas T1 evidenciaron la presencia del transgén OtNOS se autopolinizaron y se cosecharon las semillas T2. Éstas se germinaron en MS-0 en presencia de Kanamicina para determinar la segregación de la resistencia al antibiótico. La segregación 3:1 del gen marcador se evaluó estadísticamente por análisis χ2 con un nivel de significación de 0,05. Línea T1 χ2 observado Nº Pántulas R S AtC1 AtC5 AtC8 AtC13 AtC20 Total 116 155 78 34 110 Kan 90 118 69 26 79 Kan 26 37 9 8 31 AtC23 127 0 127 (segregación 3:1) 0,41 0,14 8,14 0,15 0,43 _ ¿χ observado <χ 2 2 (χ 2 0,05 = 3,84) SI SI NO SI SI _ 0,05? Nº sitios de inserción del T-ADN 1 1 >1 1 1 _ Tabla III. Análisis de segregación de la característica KanR en las plántulas de las líneas de A. thaliana Col-0 rdr6 que expresan OtNOS. Para cada línea se indica el número total de plántulas que germinaron y cuántas fueron o no resistentes a Kanamicina. 61 _______________________________________________________Capítulo IV La línea AtC17 no dío semillas y por lo tanto no se pudo utilizar para este análisis. Se aceptó la hipótesis de segregación 3:1 para las líneas AtC1, AtC5, AtC13 y AtC20 pero no para la línea AtC8. La línea AtC8 evidenció una segregación 15:1, que corresponde a la presencia del T-ADN en dos loci (el valor de χ2 observado para segregación 15:1 fue de 3,42). La progenie de la planta AtC23 resultó ser totalmente sensible a Kananamicina. Dado que la misma dio positiva para el transgén OtNOS, se interpreta que haya ocurrido un silenciamiento génico transcripcional (Elmayan y col., 1996). La Tabla III presenta un resumen del análisis de segregación de las líneas transformadas de A. thaliana en la generación T2. Determinación de la presencia de la proteína OtNOS en la generación T2 de las plantas transgénicas. Para identificar las plantas que expresan la proteína de interés, a partir de tejido foliar de las líneas AtC1, AtC5, AtC8 y AtC13 se prepararon extractos proteicos y se analizaron por SDS-PAGE y Western Blot con anticuerpos anti-OtNOS . Figura 35. Análisis de expresión de la proteína OtNOS en A. thaliana mediante Western Blot. Se obtuvieron extractos de proteínas provenientes de las diferentes líneas de A. thaliana transformada pCHF3:OtNOS. Como control positivo se utilizó la proteína OtNOSoxi expresada en forma recombinante en E. coli. La proteína fue reconocida mediante Western Blot con el anticuerpo específico anti-OtNOS. El tamaño esperado del dominio OtNOSoxi según la secuencia de aminoácidos es de 53 kDa. En las plantas transgénicas se espera un producto de 119 kDa correspondiente a la proteína OtNOS completa. La Figura 35 muestra que el anticuerpo anti-OtNOS identificó a la proteína recombinante OtNOSoxi expresada en E. coli. Sin embargo no se logró detectar la proteína OtNOS en ninguna de las líneas de plantas transgénicas. IV.3. Transformación de A .thaliana con el vector pBI-PEC:OtNOS. 62 _______________________________________________________Capítulo IV Dado que las plantas transgénicas de NtPH y A. thaliana que expresaron OtNOS bajo el control del promotor CaMV:35S mostraron un fenotipo de floración temprana, se decidió realizar la sobreexpresión a partir de un vector de expresión en plantas que tenga un promotor inducible. Ello se logró utilizando el vector pBI-PEC:GUS diseñado por Cabello y col. (2007). Este vector permite clonar el gen de interés bajo un promotor inducible por NaCl, ABA y sequía. Este es el promotor PEC del gen Hahb4 de girasol. Este vector fue gentilmente cedido por la Dra. Raquel Chan de la Universidad Nacional del Litoral (UNL), Santa Fe, Argentina. Se realizó una construcción en la cual se clonó el gen OtNOS (pBIPEC:OtNOS) bajo el control del promotor PEC (Materiales y Métodos, inciso 3.5.1). Se transformaron seis plantas Col-0 rdr6 y seis plantas Col-0 con las cepas de A. tumefaciens GV3101:pMP90 que posee el vector pBI-PEC:OtNOS, mediante la técnica de “floral dip” (Clough y Bent, 1998), cuando aún tenían las inflorescencias secundarias inmaduras y una altura entre 1 y 10 cm. Luego de 4 semanas se cosecharon las semillas T1. Nuevamente se utilizaron plantas Col-0 rdr6, pues permiten obtener niveles más altos de expresión de los transgenes (Butaye y col., 2004) La nomenclatura utilizada para las líneas transgénicas de A. thaliana que expresan OtNOS en citosol de manera inducible es rdr6 o Col-0 seguido de un número que indica los diferentes eventos de transformación o líneas. Selección de plantas transformadas por resistencia al antibiótico Kanamicina y análisis del transcripto OtNOS en las plantas transformadas con el vector pBIPEC:OtNOS luego de la inducción con NaCl y ABA. Con el objeto de identificar las líneas que expresan el transgén OtNOS, se decidió comprobar la presencia del transcripto en A. thaliana luego de la inducción. Las plantas se crecieron en medio ATS-agar en presencia de Kanamicina por 5 días, luego se transfirieron durante 5 días mas a los tratamientos NaCl 100 mM y ABA 10 µM. Se extrajo ARN de plantas Col-0 rdr6 transformadas con el vector vacío pCHF3 y de las plantas Col-0 rdr6 transformadas con el vector pBI-PEC:OtNOS. Se amplificó el gen OtNOS mediante RT-PCR, los cebadores utilizados en la PCR fueron los mismos que se utilizaron para determinar la presencia del gen OtNOS en el resto de las plantas transgénicas. 63 _______________________________________________________Capítulo IV Figura 36. Análisis de la expresión del transgén OtNOS por RT-PCR en líneas de A. thaliana transformadas con pBI-PEC:OtNOS. a) Electroforesis en gel de agarosa al 1% (p/v) de los productos de RT-PCR obtenidos a partir de ARN de A. thaliana Col-0 rdr6 transformada con pBI-PEC:OtNOS. Las líneas rdr6(1), rdr6(2), rdr6(3), rdr6(4), rdr6(6), rdr6(7) corresponden a los diferentes eventos de transformación con el vector pBI-PEC:OtNOS, el control rdr6pCHF3 corresponde a A. thaliana Col-0 rdr6 transformada con el vector pCHF3. Las plantas se trataron con NaCl 100mM y ABA 10µM durante 5 días. b) Como control interno se amplificó el gen de Actina. Los cebadores utilizados corresponden a OtNOS632Fw / OtNOS632Rv y ActinaFw(At) / ActinaRv(At) cuyas secuencias se indican en la Tabla IV en Materiales y Métodos, inciso 2. Las Figuras 36a y 36b indican que la valoración semicuantitativa por PCR evidenció un aumento en los niveles de los transcriptos de OtNOS inducidos por el tratamiento con NaCl en la mayoría de las líneas analizadas. En el caso de el tratamiento con ABA se observó un incremento en los niveles de los transcriptos OtNOS en las líneas rdr6(3), rdr6(4), rdr6(6). Fenotipo de plantas transgénicas de A. thaliana transformadas con pBIPEC:OtNOS luego de la inducción con NaCl. Con el objeto de analizar si existió un cambio en el fenotipo de las plantas transgénicas luego de la inducción del gen OtNOS se analizó el área foliar de las plantas luego del tratamiento con NaCl 100 mM. 64 _______________________________________________________Capítulo IV Figura 37. Efecto del NaCl sobre el área foliar de plantas de A. thaliana transformadas con pBI-PEC: OtNOS. Fotografía de plantas A. thaliana Col-0 rdr6 transformadas con el vector pBI-PEC:OtNOS denominadas rdr6OtNOS(6) y rdr6OtNOS(7). Como control se usaron plantas transformadas con el vector pCHF3 denominadas rdr6pCHF3. Crecidas en medio MS-Agar por 5 días y luego transferidas a NaCl 100 mM por 5 días más. Barra de escala = 2 cm. En las fotos se muestra la cuantificación del área foliar promedio de cada planta para cada tratamiento. La cuantificación se realizó con el Programa Image J. Las barras corresponden al error estándar. El asterisco indica una diferencia estadísticamente (test de t, p <0,05). En el panel superior se muestra la línea rdr6OtNOS(6) y en el panel inferior la línea rdr6OtNOS(7). La Figura 37 muestra que las líneas rdr6OtNOS(6) y rdr6OtNOS(7) evidenciaron un mayor desarrollo foliar luego del tratamiento con NaCl comparado con las plantas transformadas con el vector pCHF3. Dado que un mayor desarrollo foliar podría corresponder a un mayor crecimiento radical, se analizó el fenotipo de las raíces de las plantas transgénicas luego de la inducción del gen OtNOS con NaCl. 65 _______________________________________________________Capítulo IV La longitud de la raíz primaria y la densidad de raíces laterales determinan la arquitectura radical (Malamy y Benfey, 1997). Los procesos que afectan a la arquitectura general del sistema de la raíz son: (1) la división celular en el meristema de raíz primaria que permite el crecimiento continuo mediante la adición de nuevas células, la formación y elongación de raíces laterales lo que aumenta la capacidad exploratoria del sistema radical (Lopez-Bucio y col., 2003). Los parámetros analizados correspondieron al largo de la raíz primaria, número y largo de raíces laterales. Figura 38: Fenotipo de raíces de plántulas de A. thaliana transformadas con el vector pBI-PEC: OtNOS y tratadas con NaCl. a) Fotografía de plantas de A. thaliana rdr6OtNOS(6) y plantas control rdr6pCHF3. Las plantas se crecieron en medio MS-Agar por 5 días y luego se transfirieron a un medio MS-Agar con NaCl 100 mM por 5 días más. En el panel superior se muestran las plantas crecidas en medio MS y en el panel inferior las plantas transferidas a NaCl. Barra de escala = 1 cm. b) En el panel superior se muestra la cuantificación del número de raíces laterales para los tratamientos anteriormente indicados. Las líneas utilizadas fueron A. thaliana Col-0 rdr6 transformadas con el vector pBI-PEC:OtNOS: líneas rdr6OtNOS(6) y rdr6OtNOS(7). A. thaliana Col-0 transformadas con el vector pBI-PEC:OtNOS denominadas: líneas Col0OtNOS(1) y Col0OtNOS(2). A. thaliana de ambos genotipos transformadas con el vector pCHF3 como control. En el panel inferior se muestra la cuantificación de la longitud de raíces primarias de A. thaliana sometidas a los tratamientos anteriormente indicados. Letras diferentes indican diferencias estadísticamente significativas (ANOVA, P <0,05). Las barras corresponden al desvío estándar. 66 _______________________________________________________Capítulo IV Las Figuras 38a y 38b muestran que el tratamiento con NaCl 100mM indujo una inhibición de la raíces laterales y de la raíz primaria correspondiendo con lo descripto en plantas de A. thaliana sometidas a esta concentración de NaCl (Mitler y col., 2006). Las plantas transgénicas mostraron una menor inhibición de el sistema radical comparado con las plantas controles. Esta tendencia fue más evidente en las líneas transgénicas de genotipo Col-0 rdr6, las cuales son deficientes en silenciamiento postranscripcional, esto podría indicar una mayor expresión del transgén OtNOS en estas líneas. Se cuantificó también el largo de las raíces laterales pero no se observaron diferencias estadísticas significativas en ninguna de las líneas analizadas (dato no mostrado). Fenotipo de plantas transgénicas de A. thaliana transformadas con pBIPEC:OtNOS luego de la inducción con ABA. Dado que se observó la inducción del transgén OtNOS con ABA (Figura 36), se analizó el fenotipo de las raíces del las plantas transgénicas luego de la inducción con ABA 10µM. Se utilizaron la líneas transgénica de A. thaliana genotipo Col-0 rdr6 ya que mostraron diferencias significativas en los parámetros anteriormente analizados. 67 _______________________________________________________Capítulo IV Figura 39: Fenotipo de raíces de plántulas A. thaliana transformadas con el vector pBIPEC:OtNOS y tratadas con ABA. a) Fotografía de plántulas de A. thaliana Col0rdr6 (6) y plantas control rdr6 pCHF3. Las plantas se crecieron en medio ATS-Agar por 5 días y luego se transfirieron a medio ATS-Agar con ABA 10 µM durante otros 5 días más. En el panel superior se muestran las plántulas crecidas en medio ATS y en el panel inferior las plántulas transferidas a medio ATS con ABA 10 µM. Barra de escala= 1cm. b) En el panel superior se muestra la cuantificación del largo promedio de las raíces laterales crecidas en medio ATSAgar por 5 días y luego transferidas al tratamiento ABA 10 µM. Líneas rdr6OtNOS (3), rdr6OtNOS (4), rdr6OtNOS (6), rdr6OtNOS (7) y rdr6pCHF3 como control. En el panel inferior se muestra la cuantificación del número de raíces laterales por planta de A. thaliana sometidas al tratamiento anteriormente indicado. Letras diferentes indican diferencias estadísticamente significativas (ANOVA, P <0,05). Las barras corresponden al desvío estándar. Se observó una diferencia significativa en el número y largo de las raíces laterales luego del tratamiento con ABA 10 µM en la mayoría de las líneas analizadas. La Figura 39b muestra que el ABA indujo un drástico descenso del número de las raíces laterales en las plantas que no expresan OtNOS como ya ha sido informado anteriormente (DeSmet y col., 2007; Dongliang y col., 2009). Sin embargo en la mayoría de las líneas transgénicas se observa que la reducción del número de las raices laterales es menor al de las plantas controles luego del tratamiento con ABA (Figuras 39b). Por otro lado, se observa un aumento significativo en la longitud de las raíces laterales luego del la inducción con ABA en las líneas transgénicas (Figura 39a). 68 _______________________________________________________Capítulo IV Cuantificación de los niveles de nitritos en las plantas transformadas con el vector pBI-PEC:OtNOS . A partir de tejido foliar de las líneas Col0OtNOS(1), Col0OtNOS(2), rdr6OtNOS(6) y rdr6OtNOS(7) se prepararon extractos según se indica en Materiales y Métodos inciso 16.6 y se cuantificó mediante el reactivo de Griess la concentración de nitritos como medida indirecta de los niveles de NO en las plantas. Figura 40. Cuantificación de nitritos en plantas transgénicas. La cuantificación de nitritos se realizó con el reactivo de Griess en plántulas crecidas en medio ATS-Agar por 5 días y luego transferidas a medio ATS-Agar con ABA 10 µM por 5 días más. Las líneas analizadas correspondieron a Col0OtNOS(1), Col0OtNOS(2), rdr6OtNOS(6), rdr6OtNOS(7) y Col0pCHF3. La Figura 40 muestra que el ABA indujo un incremento en los niveles de nitritos de 4 veces aproximadamente en las plantas transformadas con el vector vacío pCHF3. En las plantas transgénicas los niveles cuantificados fueron mayores para todas las líneas analizadas. Resta por analizar los niveles de nitritos en plantas genotipo Col-0 rdr6 transformadas con el vector pCHF3. Efecto de la sequía en las plantas transgénicas. Dado que el NO ejerce un efecto protector frente a la sequía (Tian y col., 2006) y además el promotor PEC se induce por estrés hídrico (Dezar y col., 2005) se evaluó el comportamiento de las plantas transgénicas sometidas a estrés por sequía. 69 _______________________________________________________Capítulo IV Figura 41. Efecto de la sequía en plantas de A. thaliana genotipo Col-0 rdr6 transformadas con pBI-PEC:OtNOS. Las plántulas se crecieron durante 3 semanas en un sustrato de perlita/vermiculita/tierra con riego normal. En el panel superior se muestran las plántulas luego de 8 días de suspendido el riego. En el panel inferior se muestran las plántulas luego de 10 de días de riego suspendido más 10 días de recuperación con riego normal. La Figura 41 muestra que las líneas rdr6OtNOS(6) y rdr6OtNOS(7) presentaron mayor tolerancia a la sequía. La línea rdr6OtNOS(6) mostró un porcentaje de supervivencia del 33 %, la línea rdr6OtNOS(7) el porcentaje fue del 10%, sin embargo la línea rdr6OtNOS(2) mostró una supervivencia del 0% al igual que las plantas control rdr6pCHF3 luego del estrés. Este resultado se corresponde con los mostrados en la Figura 36, la línea rdr6OtNOS(2) no mostró expresión del transgén luego de los tratamientos con ABA y NaCl, si bien los niveles de transcriptos mostrados provienen de una RT-PCR no cuantitativa. 70 _______________________________________________________Capítulo IV Discusión En el presente Capítulo se demostró que las plantas transformadas con el gen OtNOS bajo un promotor CaMV:35S presentaron un fenotipo de floración temprana, si bien no se pudo conocer las causas de dicho fenotipo, el mismo se contradice con el fenotipo descripto por He y col. (2004). En ese trabajo, se informó que el NO reprime la transición floral en A. thaliana. Las plantas tratadas con NO, así como una sobreproducción de NO en la mutante (nox1), mostraron una floración tardía, mientras que un mutante que produce menos NO (Atnoa1) florece mas temprano que las plantas salvajes (He y col., 2004). La plantas transgénicas CaMV:35SOtNOS podrían presentar un silenciamiento postrancripcional de algún gen endógeno y el fenotipo observado podría estar relacionado con este tipo de silenciamiento. Estudios previos indicaron que en general plantas transgénicas que sobreexpresan un transgén bajo un promotor CaMV:35S, podían evidenciar diferentes tipos de silenciamientos (Stam y col., 1997). Estos se clasifican en silenciamiento transcripcional y silenciamiento postranscripcional de genes endógenos. En el primer caso corresponde a la inserción del transgén en un promotor o en un gen endógeno impidiendo la expresión del gen modificado. Por otra parte, el silenciamiento postrancripcional se refiere al silenciamiento de genes por homología de secuencia, cuando existe una muy alta expresión del transgén. Se ha postulado el rol de los ARNs complementarios (ARNc) sintetizados en plantas por ARN polimerasas dependientes de ARN presentes en el citoplasma (RdARN). Estas RdARN sintetizan pequeños ARN a partir de ARN como templado y estos pequeños ARN podrían hibridar y las dobles cadenas de ARN serían degradadas por ARNasas específicas. Estas plantas transgénicas presentan el mismo fenotipo de plantas mutantes en el gen silenciado. Un ejemplo particular se da en plantas de tabaco transgénicas que sobreexpresan el gen NR (Nitrato Reductasa) bajo un promotor CaMV:35S cuyo fenotipo es el opuesto al esperado con plantas más pequeñas y cloróticas. Los autores sugieren que este fenotipo se debe a un silenciamiento del gen endógeno de la NR (Dorlhac de Borne y col., 1994). Por otra parte, las plantas de A. thaliana Col-0 rdr6 que se transformaron con el vector pBI-PEC:OtNOS, luego de la inducción con ABA o NaCl desarrollaron un mayor sistema radical, lo cual podría aportar una mayor tolerancia a situaciones de estrés. El comportamiento de dichas plantas a nivel radical también se correspondió con un mayor desarrollo de la parte aérea. 71 _______________________________________________________Capítulo IV En este sentido, Correa Aragunde y col. (2007) y Gouvea y col. (1997), describieron que el NO proporcionado a través SNP promovió el desarrollo de raíces laterales en plantas de tomate y maíz. Otras evidencias indicaron que el SNP disminuyó el efecto inhibitorio del ABA sobre la densidad de raíces laterales sugiriendo que el NO actúa río abajo del ABA. (Sivananthan, 2006). El NO producido por la sobreexpresión de OtNOS podría secuestar los ROS generados durante el estrés como ha sido previamente demostrado por Caro y Puntarulo (1998). Durante la sobreproducción de ROS debido a un estrés, el NO tendría una función protectora eliminando el exceso de ROS y frenando la oxidación de lípidos mediada por radicales (Beligni y Lamattina, 1999a). El pretratamiento de plántulas de Lupinus luteus con SNP produce una reducción efectiva de los daños producidos por estrés abiótico sobre el crecimiento y morfología de la raíz y el efecto protector del NO sobre las raíces estresadas puede ser debido a la estimulación de la actividad de SOD y/o al secuestro directo del anión O2˙¯ por parte del NO (Kopyra y col., 2003). En un trabajo recientemente publicado se demostró que plantas transgénicas de A. thaliana que sobrexpresaron la enzima nNOS de rata en forma constitutiva bajo el promotor CaMV:35S, presentaron mayor resistencia a sequía y salinidad. La mayor tolerancia se adjudicó a cambios en parámetros fisiológicos incluyendo una reducción en la tasa de pérdida de agua, una menor apertura estomática, y una alteración en los niveles prolina y malondialdehído (Shi y col., 2012). En su conjunto, estos resultados mostrarían que, además del efecto del NO de promover el cierre de los estomas, previniendo la perdida de agua en condiciones de estrés hídrico (Garcia Mata y Lamattina., 2001), el NO podría actuar a nivel de la regulación de la homeostasis redox. Las plantas transgénicas generadas durante esta tesis permitirán abordar el estudio de los mecanismos del NO involucrados en la respuesta a estrés por sequía y salinidad, debido a que las plantas transgénicas presentan una regulación de expresión controlada por estos tipos de estreses ambientales. 72 ___________________________________________________Discusión General DISCUSIÓN GENERAL Las funciones del NO se han diversificado y especializado durante la evolución con la aparición de organismos pluricelulares. Es probable que el NO se haya ido involucrando progresivamente en la señalización inter e intracelular. En bacterias además de ser el NO un producto del metabolismo del N (Zumft y col., 1993), el NO es también generado por procesos oxidativos a partir del aminoácido L-arg y mediante la actividad de la enzima NOS. Las bacterias presentan una NOS truncada, con un dominio oxidasa que es capaz de producir NO (Crane y col., 2010). En eucariontes, las NOS adquirieron un dominio reductasa y un sitio de unión a CaM alcanzando una mayor eficiencia y sofisticación de la regulación de la actividad enzimática, ya que pueden ser activadas por aumento de los niveles de Ca2+ (Alderton y col., 2001). OtNOS posee un sitio de unión a CaM, el cual no es muy conservado respecto a las NOS estudiadas en animales y posee una dependencia parcial de CaM. Mediante un análisis filogenético se determinó que OtNOS se encuentra en el mismo grupo que la NOS de Synechococcus sp y P. polycephalum. Por otra parte, un análisis ‘in sílico’ de la secuencia NOS de Synechococcus sp (SyNOS) se observó que conserva los dos dominios oxigenasa y reductasa característicos de las NOS de mamíferos, pero carece del sitio de unión a CaM (Correa-Aragunde, comunicación personal). A su vez, la proteína NOS de P. polycephalum muestra una actividad independiente de Ca2+/CaM (Golderer y col., 2001). Todos estos datos podrían estar indicando cuando las NOS adquirieron el sitio de unión a CaM a lo largo de la evolución biológica. Las formas de NOS que presentan una regulación por Ca2+/CaM se encuentran en organismos pluricelulares complejos demostrando la aparición de estructuras más sofisticadas de NOS en organismos más evolucionados (relación estructura/función). Otra evidencia en el mismo sentido es la proteína NOS en Sorangium celullosum, la cual es la primera NOS bacteriana conocida hasta el momento que presenta un dominio reductasa fusionado a un dominio oxidasa. Al igual que todas las NOS bacterianas anteriormente analizadas carece del sitio de unión a CaM (Agapie y col., 2009). La función del NO en mamíferos está relacionada con la señalización intercelular. La primera NOS que se identificó fue encontrada en el tejido neuronal y corresponde a nNOS, la eNOS fue la tercera identificada. Se clasificaron originalmente como de expresión constitutiva y Ca2+ sensibles, pero ahora se sabe que están presentes en muchos tipos de células diferentes y que la expresión está regulada por determinadas condiciones fisiológicas (Alderton y col., 2001). 73 ___________________________________________________Discusión General Concentraciones fisiológicas de Ca2+ regulan la unión de CaM a nNOS y eNOS, iniciando así la transferencia de electrones desde las flavinas al grupo hemo. No ocurre lo mismo con la iNOS en la que la CaM se mantiene fuertemente unida resultando en una isoforma insensible a Ca2+ y actuando como una subunidad de esta isoforma (Alderton y col., 2001). iNOS es expresada en macrófagos los cuales producen altos niveles de NO que resultan citotóxicos para bacterias y para las células que lo producen. La expresión de iNOS depende de la exposición de las células a citoquinas y productos bacterianos (Stuehr y col., 1991). Puede concluirse entonces que O. tauri es un organismo eucariota unicelular muy sencillo, con un solo cloroplasto y una sola mitocondria y posee una NOS con un sitio de unión a CaM. Esto confirma que en las NOS estudiadas hasta el momento, la adquisición de un sitio de unión a CaM corrrespondería a organismos eucariotas. La producción de NO dependiente de NOS puede regularse por sí mismo, modulando el NO la expresión y la actividad de la NOS. Específicamente, se ha demostrado que el NO desempeña un papel importante en la retroalimentación negativa en eNOS. Este proceso ocurre mediante la S-nitrosilación de eNOS que inhibe reversiblemente su actividad en las células endoteliales vasculares. Este mecanismo de regulación puede ser importante ya que está modulado por las condiciones redox celulares y por lo tanto puede proporcionar una explicación para asociar el "estrés oxidativo" y la disfunción endotelial (Alderton y col., 2001). OtNOS muestra una fuerte inhibición por NO. Estos resultados podrían estar indicando una auto-regulación de la actividad por un proceso de S-nitrosilación tal como ha sido caracterizado para eNOS. Será interesante investigar este punto en el futuro. En bacterias, las NOS están relacionadas con la regulación del estrés oxidativo. Por ejemplo, se sabe que la NOS de Bacillus subtilis está relacionada con la resistencia de las bacterias al estrés oxidativo generado por H2O2, o algunos antibióticos como la ampicilina (Gusarov y col., 2009). Nuestras evidencias indican que OtNOS se induce en O. tauri en respuesta a un incremento en la intensidad de luz y esta inducción de NO vía NOS podría estar involucrada en la protección del alga frente a un estrés fotoxidativo o en procesos de desarrollo regulados por luz. En un estudio previo se ha demostrado que la luz controla el desarrollo del hongo Phycomyces blakesleeanus (Maier y col., 2001). La inhibición de la biosíntesis de H4B inhibe la fotomorfogénesis en el hongo. 74 ___________________________________________________Discusión General Se ha demostrado que la luz induce la producción y emisión de NO en el mismo orden de magnitud que la formación de L-cit a partir de L-arg. El dador de NO SNP puede sustituir el efecto de la luz sobre el desarrollo del hongo y los inhibidores de NOS reprimirlos, sugiriendo que una NOS está implicada en el desarrollo y en la señalización por luz en este hongo (Maier y col., 2001). Por otro lado se ha encontrado que la radiación UV-B induce la producción de NO en el alga Chlorella pyrenoidosa. El agregado de SNP fue capaz de prevenir la pérdida de clorofila mediada por UV-B. Además, las suspensiones de algas irradiadas por UV-B aumentaron la actividad de las enzimas antioxidantes catalasa y superóxido dismutasa. El UV-B también mostró ser un potente inductor de la producción de NO el cual, conjuntamente con las actividades antioxidantes, logró proteger el alga contra el daño oxidativo (Chen y col., 2003). Por lo tanto, el conjunto de estos resultados muestra que el NO puede estar involucrado tanto en la regulación de los procesos del desarrollo como en la defensa antioxidante de O. tauri. El NO ha surgido como una molécula clave implicada en muchos procesos fisiológicos en las planta. Si bien no se ha encontrado en plantas superiores ningún gen, ADNc o proteína con homología de secuencia a las NOS animales, numerosos estudios provenientes de diferentes laboratorios demuestran una producción de NO dependiente de una actividad tipo NOS en plantas. En esta tesis se transformaron plantas de A. thaliana y N. tabacum con el gen OtNOS para estudiar su comportamiento en situaciones de estrés. Las plantas transgénicas que sobreexpresan OtNOS bajo un promotor inducible muestran mayor desarrollo radical y tolerancia a estrés salino. En la mutante Atnoa1 con niveles reducidos de NO la germinación y tasa de supervivencia se reduce en presencia de sal. Plantas mutantes Atnoa1 mostraron mayores niveles de H2O2 que las plantas salvajes en estrés salino y el tratamiento con SNP revirtió la sensibilidad a la sal (Zhao y col., 2007). Nuestros resultados se encuentran en el mismo sentido que las respuestas de las mutantes Atnoa1 al estrés salino. Por lo tanto, los resultados de esta tesis proporcionan una nueva línea de evidencia indicando que la producción de NO se asocia con tolerancia a la salinidad en A. thaliana. La acumulación de ABA y el estrés oxidativo son dos respuestas comunes de las plantas al estrés ambiental y objeto de intensos estudios. Zhao y col. (2001) analizaron los efectos de los ROS y el NO en la síntesis de ABA en puntas de raíces de plántulas de trigo (Triticum aestivum) bajo estrés por sequía. Las raíces fueron sometidas a estrés hídrico por evaporación y las actividades de producción de O2˙¯ y 75 ___________________________________________________Discusión General la actividad tipo NOS aumentaron después del tratamiento. La acumulación de ABA por sequía se bloqueó con el inhibidor de la NOS (L-NAME) sugiriendo que los ROS y el NO pueden ser las señales primarias que la planta percibe en la condición de sequía y tal vez otros estreses abióticos. El conocimiento sobre el NO y su papel en el crecimiento de raíces y los procesos de desarrollo de las plantas se ha incrementado en los últimos años (Pagnussat y Lamattina, 2002; Correa y col., 2007). El aumento transitorio de la concentración de NO demostró estar involucrado en el desarrollo de raíces inducida por auxinas (Pagnussat y col., 2002). Los autores sugieren que el NO podría mediar la respuesta a auxina en este proceso. Mas recientemente, se ha demostrado que el NO S-nitrosila el receptor de auxina TIR1 y regula la señalización disparada por auxinas, especialmente la activación de genes que responden a auxinas (Terrile y col., 2012). Finalmente, los estudios sobre la expresión del transgén OtNOS en plantas indicaron cambios en las respuestas del desarrollo tal como el crecimiento de raíces laterales, además de una mayor tolerancia al estrés salino. Aún restan por estudiar los mecanismos moleculares que operan río abajo del NO en la regulación de los procesos fisiológicos. En conclusión, el conjunto de las evidencias que se describen en esta tesis permitieron evaluar las hipótesis planteadas en los objetivos. Hipótesis 1: La secuencia génica correspondiente a OtNOS publicada en la base de datos NCBI codifica para una proteína NOS y se expresa en O. tauri. Mediante la secuenciación del ADNc correspondiente a OtNOS encontramos un error de un nucleótido en la secuencia publicada en NCBI. Sin embargo, el error no implicó ningún dominio crítico de unión a cofactores y la proteína heteróloga se expresó correctamente. Hipótesis 2: El gen OtNOS codifica para una proteína NOS funcional en O. tauri. Los análisis de secuencia y estructura mostraron que OtNOS presenta una similitud del 45% con las NOS humanas, todos los sitios de unión a cofactores se encuentran conservados y el dominio catalítico mostró ser idéntico a eNOS de Bos turus. La proteína OtNOS recombinante mostró parámetros bioquímicos similares a las enzimas NOS animales en cuanto a actividad, Km y dependencia de cofactores. 76 ___________________________________________________Discusión General Por otra parte los análisis del mecanismo catalítico indicaron que OtNOS conserva las características de formación de un compuesto oxido ferroso en el grupo hemo al igual que todas las NOS estudiadas. Hipótesis 3: El NO generado a partir de OtNOS cumple funciones en el metabolismo celular del alga. Se observó una producción de NO dependiente de L-arg en el cultivo de O. tauri. Esta producción de NO dependiente de L-arg fue bloqueada por los inhibidores de NOS animales, indicando la presencia de una actividad tipo NOS en O. tauri. Los resultados mostraron que la producción de NO a través de OtNOS estaría involucrada en procesos de adaptación de O. tauri a diferentes intensidades de luz. Hipótesis 4: La expresión regulada de OtNOS conferirá una cierta protección y tolerancia a diferentes tipos de estreses en plantas genéticamente transformadas. Las plantas transformadas con el vector pBI-PEC:OtNOS mostraron mayor desarrollo del sistema radical y mayor tolerancia al estrés salino. Por otra parte cuando estas plantas fueron inducidas con ABA lograron evadir el efecto inhibitorio de la hormona sobre el desarrollo de las raíces laterales. Se observó que las plantas transgénicas mostraron una mayor supervivencia luego de ser sometidas a estrés hídrico. Perspectivas. Continuar con los estudios del mecanismo catalítico de OtNOS, centrados principalmente en el análisis de la función del cofactor THF reemplazando el cofactor H4B ausente en Ostreococcus y plantas superiores. Profundizar los estudios sobre la función fisiológica del NO y de OtNOS en O. tauri mediante el silenciamiento génico de OtNOS. Estudiar en profundidad los procesos que involucran el NO en respuestas al estrés hídrico y salino en A. thaliana y N. tabacum utilizando las herramientas generadas como las plantas transgénicas que expresan OtNOS bajo un promotor inducible por los estreses mencionados. 77 _________________________________________________Materiales y Métodos MATERIALES Y MÉTODOS 1. Material biológico. 1.1 Bacterias. 1.1.1. Escherichia coli. Para propósitos de clonado y obtención de plásmidos, se utilizó la cepa DH5α DE3 (Life Technologies), Para expresión de proteínas recombinantes, se utilizó la cepa Bl21-codon plus (DE3)-RIL (genotipo F- ompT hsds (rB- mB-) dcm + Tet R gal R (DE3) endA Hte (argU ileY lleuW Cam )) adquirido en Novagen. 1.1.2. Agrobacterium tumefaciens. Para transformar plantas de A. thaliana, y N. tabacum, se empleó la cepa GV 3101 pMP90 (Koncz y Schell, 1986), con resistencia a los antibióticos rifampicina (codificación genómica) y gentamicina (codificada por el plasmido pMP90). 1.2. Plantas. En el presente estudio se utilizaron las siguientes especies: A. thaliana ecotipo Columbia (Col 0). A. thaliana, ecotipo Columbia, deficientes en silenciamiento transcripcional rdr6 (Luo y col., 2007). Nicotiana Tabacum ecotipo Petit Havana. Todas ellas fueron cedidas por el Dr. Nestor Carrillo. IBR, Universidad Nacional de Rosario, Argentina 1.3. Cultivo de Ostreococcus tauri. El cultivo de O. tauri (cepa OTTH 0595) fue adquirido de la Colección de Cultivos de Roscoff (Station Biologique, Roscoff, Francia), y se cultivaron en Erlenmeyers conteniendo medio K (Keller y col, 1987.) A 18 ºC bajo fotoperiodo 12 h: 12 h (luz: oscuridad). Los últimos fueron optimizados en colaboración con la Dra Graciela Salerno y el Lic. Gonzalo Caló. FIBA, Mar del Plata. Los experimentos con luz se llevaron a cabo con irradiaciones que van desde 40 a 400 µmoles. m-2. s-1. 2. Cebadores utilizados. En la Tabla IV se muestra las secuencias de los oligonucleótidos utilizados como cebadores en el presente trabajo, la finalidad que tuvieron y características particulares de alguno de ellos. 78 _________________________________________________Materiales y Métodos Tabla IV. Lista oligonucleótidos. Secuencia Finalidad Observación OtNOS662 Fw Nombre GCTGGGCGCCGGAAAAGAC OtNOS2674Rv GCGCCGGCCGAAACTCAAC OtNOSNdeIFw GGAATTCCCATATGGCGAGCGTTGGCTCGGGCGCGACC NdeI OtNOSXho RV CCGCCTCGAGAGCATCCTTTGCGCGAGGCAGAAGGGAGCC OtNOS632 Fw CTACACCATGCGGTTGTTTG PCR/ control de transformación PCR/ control de transformación Amplificación dominio oxi Amplificación dominio oxi RT-PCR OtNOS632 Rv CCCTGTGAAGAGTCGAAAGC RT-PCR OtNOS2203Fw CTTGTTCCGAAGAGTGATGCG OtNOS2537Rv GAGCTTGAGACGGAATAATAGC OtNOSXbaIPCFw GTCTAGAGGATCCCCGATGGCGAGCGTTG control de secuenciación control de secuenciación PCR/RT-PCR XbaI OtNOSSacIPCRv CGAGCTCTCACGGCGACGACAAGCACAAC PCR/Rt-PCR SacI Fw tEF-1α TTCAGGAGCATGCGTCAAACTG Control interno gen de Nicotiana tabacum Rv tEF-1α TCTTCTTCTGAGCAGCCTTGGT Actina Fw(At) AATCTCCGGCGACTTGACAG Actina Rv (At) AAACCCTCGTAGATTGGCACA XhoI Control interno gen de A. thaliana. 3. Construcciones realizadas. 3.1. pUC57OtNOS. Este vector fue diseñado por la empresa Gen Script Corporation a pedido del Dr. Lamattina. Contiene la secuencia codificante de la OtNOS clonada en el sitio EcoRV del sitio de múltiple clonado (SMC) del pUC57 (entre BamHI y XbaI), a la cual se le mutó un sitio interno BamHI para facilitar luego su clonado y se agregaron 5 bases protectoras antes de su ATG inicial. Esquema del vector pUC57+OtNOS. Representación de las diferentes secuencias del vector, identificadas por colores. AmpR: marcador de selección que confiere resistencia al antibiótico ampicilina. pUC ori: origen de replicación de la serie de vectores pUC. LacZ: subunidad α de la β-galactosidasa. OtNOS: secuencia codificante de la NOS de O. tauri. El número central indica el tamaño del plásmido en pares de bases. 79 _________________________________________________Materiales y Métodos 3.2. pET24bOtNOS. El vector pET-24b (Novagen) posee una secuencia N-terminal que codifica para tag T7. El marcador de selección es la Kanamicina. En este vector se clonó la secuencia completa del ADNc de OtNOS utilizando las enzimas de restricción BamHI y SacI (Promega). El fragmento se obtuvo a partir del vector pUC57+OtNOS, digerido con las mismas enzimas. Esquema del vector pET24b. Representación de las diferentes secuencias del vector, identificadas por colores. KanR: marcador de selección que confiere resistencia al antibiótico Kanamicina. Promotor T7 y terminador T7. El número central indica el tamaño del plásmido en pares de bases. 3.3. pET15bOtNOSoxi. El vector pET-15b (Novagen) posee una secuencia C-terminal que codifica para un tag de histidina. El marcador de selección es la ampicilina. En este vector se clonó la secuencia del dominio oxi de OtNOS incluyendo el sitio de unión a CaM del ADNc de OtNOS utilizando las enzimas de restricción NdeI y XhoI en fase con la señal de histidina. Se diseñaron cebadores específicos que permitieron amplificar la secuencia del dominio OtNOSoxi, a estos oligonucleótidos se les adicionó los sitios de restricción correspondientes OtNOSNdeIFW y OtNOSXhoIRv. 80 _________________________________________________Materiales y Métodos Esquema del vector pET15b. Representación de las diferentes secuencias del vector. Ap: marcador de selección que confiere resistencia al antibiótico ampicilina. En negrita se indican los sitios restricción para el clonado en fase con la señal de histidina. El número central indica el tamaño del plásmido en pares de bases 3.4. pCHF3. Es un vector binario apto para transformación de plantas por medio de A. tumefaciens. Posee un marcador de resistencia a espectinomicina para selección en bacterias y el gen NPTII que otorga resistencia a Kanamicina dentro del T-ADN en plantas. Contiene el promotor CaMV:35S y la secuencia terminadora del gen de la subunidad menor de la Rubisco (tRBCs) para la expresión constitutiva de genes en plantas. Cedido gentilmente por el Dr. Eduardo Zabaleta. Esquema del plásmido pCHF3. Representación de las diferentes secuencias del vector, identificadas por colores. Se muestran en gris algunos sitios reconocidos por enzimas de restricción relevantes. Los números entre paréntesis indican la posición de los mismos. En celeste se muestra el sitio tRBCs y en verde el promotor CaMV. 3.4.1 pCHF3:OtNOS. 81 _________________________________________________Materiales y Métodos Vector construido al clonar la secuencia de OtNOS entre los sitios BamHI y XbaI del pCHF3. El fragmento se obtuvo a partir del vector pUC57+OtNOS, digerido con las enzimas anteriormente mencionadas. Este plásmido se utilizó para la transformación de plantas y permite la expresión de OtNOS en el citosol, de manera constitutiva. 3.5. pBI-PEC:GUS. Es un derivado del vector binario pBI101.3 y fue diseñado por Cabello y colaboradores (2007). Contiene el gen de la enzima β-glucuronidasa (GUS) de E. coli, como gen reportero, bajo control transcripcional del alelo corto del promotor del gen Hahb4 de girasol (PEC). Este promotor es inducible por estrés hídrico, estrés salino, ABA (Dezar y col., 2005), etileno (Manavella y col., 2006), metil-jasmonato, daño mecánico (Manavella y col., 2008a) y oscuridad (Manavella y col., 2008b). Este vector posee además el intrón de la sub-unidad 5c de la citocromo c oxidasa de A. thaliana. Este vector fue cedido por la Dra. Raquel Chan. Universidad Nacional del Litoral, Argentina Esquema del plásmido pBI-PEC:GUS. Se detallan los principales sitios reconocidos por enzimas de restricción. Posee dos genes que confieren KanR, uno para su uso en plantas, que se detalla en el mapa (NPTII) y otro para selección del vector en bacterias (no mostrado) fuera del T-ADN. 3.5.1. pBI-PEC: OtNOS. Vector construido al clonar la secuencia de OtNOS entre los sitios XbaI y SacI del pBI-PEC en reemplazo del gen GUS. Se diseñaron cebadores específicos que permitieron amplificar la secuencia de OtNOS. A estos oligos se les adicionó los sitios 82 _________________________________________________Materiales y Métodos de restricción correspondientes (OtNOSXbaIPCFw y OtNOSSACIPCFw). Este plásmido se utilizó para la transformación de plantas y permite la expresión de OtNOS en el citosol y es inducible por estrés hídrico, estrés salino, ABA, etileno, metil-jasmonato, daño mecánico y oscuridad. 4. Purificación de ADN plasmídico. Se utilizaron dos métodos distintos para obtener ADN plasmídico. El método de lísis alcalina se utilizó para obtener ADN para reacciones de PCR y digestiones con enzimas de restricción durante la búsqueda de colonias transformadas con los plásmidos recombinantes de interés. Para la preparación de vectores para clonados se utilizó un sistema comercial. Para la lisis alcalina se utilizó el protocolo descrito por Maniatis y col. (2001). En la purificación de ADN plasmídico de A. tumefaciens se utilizó el protocolo descripto para el método de lisis alcalina “Midiprep” según Sambrook y col. (2001). 4.1. Electroforesis de ADN en gel de agarosa. Se empleó una cuba de inmersión horizontal con tampón de corrida 1X TAE (40 mM Tris acetato, pH 8, 1 mM EDTA) a 80 mA. A las muestras se les agregó solución colorante para ácidos nucleicos 10X [0,4 % (p/v) azul de bromofenol, 0,1 % (p/v) xilencianol y 50 % (v/v) glicerol] y fueron sembradas en geles de agarosa preparado en tampón 1X TAE y suplementado con una dilución 1:25000 de Saber safe (Invitrogen). La concentración de agarosa varió entre 0,8 y 2% (p/v) según el tamaño de los fragmentos que se deseaba separar. El ADN se visualizó por fluorescencia con un transiluminador luz azul. 4.2. Estimación de la concentración de ADN. Las muestras se separaron por electroforesis y se estimó la concentración por comparación de la intensidad de las bandas con las del ADN del fago λ digerido con la enzima Hind III que genera cantidades conocidas de fragmentos de distintos tamaños. 5. Transformación de bacterias. 5.1. Obtención de células electrocompetentes y electroporación de A. tumefaciens 5.1.1. Obtención de células electrocompetentes de A. tumefaciens. 83 _________________________________________________Materiales y Métodos Se cultivó un preinoculo de A. tumefaciens GV3101:pMP90 en 2 ml de medio de cultivo 2YT (16 g/l extracto de peptona, 10 g/l extracto de levadura y 5 g/l NaCl) suplementado con gentamicina 50 µg/ml y rifampicina100 µg/ml durante 24 h a 29°C bajo agitación constante. El preinóculo fue diluído en 100 ml del mismo medio en un Erlenmeyer de 500 ml. Se lo cultivó hasta alcanzar una DO600 entre 0,4 y 0,6. El cultivo fue transferido a tubos de centrífuga, incubado 10 min en hielo y centrifugado por 15 min a 4000 g y 4°C. Se realizaron 4 lavados del sedimento celular adicionando en cada caso 20 ml, 10 ml, 10 ml y 1 ml de glicerol frío 10 % (v/v), resuspendiéndolo con vortex y centrifugándolo 15 min a 4000 g y 4°C. Finalmente las células se resuspendieron en 1 ml de glicerol frío 10 % (v/v), se fraccionó en alícuotas de 40 µl y se congelaron a 80°C. 5.1.2. Electrotransformación de A. tumefaciens. Se mezclaron como máximo 40ng de ADN con 50 µl de bacterias competentes. La mezcla se transfirió a una cubeta de electroporación de 0,2 cm (BIO-RAD) fría, y a continuación se aplicaron 2,5 kV empleando el electroporador E. coli pulser (BIORAD). Inmediatamente se agregaron 600 ul de medio LB a la cubeta. Se mezcló por inversión y se transfirió a un tubo eppendorf de 1,5 ml. Por último, las bacterias se plaquearon en medio LB con el antibiótico adecuado. La conservación de las cepas transformadas de E. coli y A. tumefaciens a largo plazo fueron guardadas a -80°C con una concentración final de glicerol del 20 % (v/v). 5.2. Obtención de células competentes y transformación de E. coli. 5.2.1. Obtención de células por CaCl2 competentes de E. coli. Se cultivó un preinoculo de E. coli en 2 ml de medio de cultivo LB durante 24 h a 37°C bajo agitación constante. El preinóculo se diluyo en 100 ml del mismo medio en un Erlenmeyer de 500 ml. Se lo cultivó hasta alcanzar una DO600 entre 0,4 y 0,6. El cultivo se transfirió a tubos de centrífuga, y se centrifugó por 10 min a 5000 g y 4°C. Se resuspendió en 2,5 ml de CaCl2 0,1 M con 15% (v/v) glicerol frío, se fraccionó en alícuotas de 200 µl y se congelaron a -80°C. Cada una sirve para realizar una transformación. 5.2.2 .Transformación de E. coli. Se mezcló el plásmido deseado en 100 µl de CaCl2 0.1M en tubos eppendorf de 1,5 ml y se agregó 200 µl de células competentes. Luego se incubó durante 30 min en hielo. Se realizó un shock por 2 min a 42ºC, rápidamente se agregó 700 µl de 84 _________________________________________________Materiales y Métodos medio LB y se incubó por 30 min a 37ºC. Finalmente, se plaqueó en placas de LB agar con el antibiótico adecuado (Hanahan y col., 2003). 6. Expresión y purificación de proteínas en E. coli. 6.1. Expresión de OtNOS completa: En Erlenmeyers conteniendo 1 litro de LB modificado (20 g de extracto de levadura, 10 g de bactotriptona, 2,65 g de KH2PO4, 4,33 g de Na2HPO4, y 4 ml de glicerol) y Kanamicina (50 mg/ml) se creció 1 ml de cultivo y se agitó a 190 rpm a 30ºC. La expresión de la proteína recombinante se indujo a un DO600 = 0,6 mediante la adición de 0.5 mM IPTG. Se añadieron los precursores del grupo hemo y de flavinas, ácido aminolevulínico 450 µM y riboflavina 3 µM en el momento de la inducción (List y col., 1996). Las células se recogieron después de 40 h de inducción y se resuspendieron en 30 ml de tampón (Tris-HCl 100 mM, pH 7,4, EDTA 1 mM, de DTT 1 mM, 10% [v/v] glicerol, PMSF 1 mM, 5 mg / ml de leupeptina, y pepstatina 5 mg/ml). El cultivo inicial se lisó por sonicación mediante pulsos (seis ciclos de 20 s). Los restos celulares se eliminaron por centrifugación, y el sobrenadante se aplicó a una columna de 1 ml de 2,5-ADP-4B agarosa equilibrada en tampón B (Tris-HCl 50 mM, pH 7,4, EDTA 0,1 mM, DTT 0,1 mM, 10% (v/v) de glicerol, y NaCl 100 mM). La columna se lavó extensamente con 10 volúmenes de tampón B y finalmente con tampón B y NaCl 500 mM. La proteína se eluyó con tampón B, NaCl 500 mM y 2'-AMP 25 mM. Las proteínas se cuantificaron mediante la técnica de Bradford (Kruger y col., 2002). 6.2. Expresión dominio oxi de OtNOS. Erlenmeyers conteniendo 4 litros de LB modificado (20 g de extracto de levadura, 10 g de bactotriptona, 2,65 g de KH2PO4, 4,33 g de Na2HPO4, y 4 ml de glicerol) y ampicilina 100 mg/ml) se inocularon con 1 ml de cultivo y se agitó a 190 rpm a 37ºC. La expresión de la proteína recombinante se indujo a un DO600 = 1 mediante la adición de IPTG 1 mM a 25ºC durante toda la noche. El precursor del grupo hemo, ácido aminolevulínico 500 mM se añadió al momento de la inducción. Las células se centrifugaron durante 20 min a 4ºC a 6000 rpm después de 18 hs de inducción y se resuspendieron en 200 ml de tampón de lisis (Tris-HCl 100 mM, pH 7,4, EDTA 1 mM, NaCl 150 mM, PMSF 1 mM, 1 mg / ml de lisozima, D-Nasa 50 U/ml). El producto de la lisis se sometió a una prensa francesa, en el cual 8,5 ml de extracto se sometieron a 1 bar de presión. Luego se centrífugó a 4ºC durante 45 min a 16000 rpm y se recuperó 85 _________________________________________________Materiales y Métodos el sobrenadante. Se realizó una precipitación con sulfato de amonio durante 30 min y se aplicó la siguiente relación: M (sulfato de amonio) (g)=0,331 x V (en ml de lisado). Se centrifugó a 16.000 rpm durante 30 min a 4 °C, el sedimento se resuspendió en tampón de unión Tris-HCl 0,1 M, pH 8-9, 10% de glicerol, NaCl 0,25M, PMSF 1mM. El sobrenadante se cargó en una columna de Niquel ProBond (Invitrogen) previamente lavada con tampón de unión más imidazol 10 mM (tampón A). La columna se lavó con 10 volúmenes de tampón A. La proteína unida se eluyó con tampón de unión que contenía 200 mM de imidazol. Las distintas fracciones de la columna se agruparon y se concentraron utilizando Centriprep (Millipore, Bedford, MA) con centrifugaciones a 5000 rpm. Los concentrados de proteínas se dializaron a 4º C frente a 2 litros de tampón KPi 0,1M pH 8-9, que contiene 10% (v/v) de glicerol, NaCl 0,25M, PMSF 1mM y DTT 3mM. Después de un cambio de tampón durante la noche, se verificó el contenido de hemo y la pureza de la proteína por SDS-PAGE y la proteína se almacenó a -80 ° C. Todos los espectros de UV-visible se registraron a temperatura ambiente en un Uvikon XL (Secomam Ales, Francia). 7. Determinación de la actividad NOS. 7.1. El método de oxihemoglobina. Se realizó según lo descrito por Ghafourifar y col. (2005). La hemoglobina fue completamente reducida a oxihemoglobina con ditionito de sodio. La concentración de oxihemoglobina en la solución se determinó usando el coeficiente de extinción molar 131 mM-1cm-1 a 415 nm. Una reacción de 500 µl que contiene oxihemoglobina 20 mM, HEPES-NaOH 7,5 mM pH 7,5, DTT 5 mM, L-arg 100 mM, NADPH 1mM, CaCl2 10 mM, CaM 10mM, H4B 100 µM, y catalasa 100 U/ml. La reacción fue iniciada por el agregado de la proteína purificada OtNOS 0,5 mM. La conversión de oxihemoglobina a metahemoglobina se analizó en un espectrofotómetro (Ultrospec 1100 Pro, Amersham Biosciences) por barrido entre 380 y 450 nm. Un coeficiente de extinción de 100 mM-1cm-1 entre el pico a 401 nm y el valle a 420 nm se utilizó para cuantificar la producción de NO. 7.2. Formación de L-citrulina. 86 _________________________________________________Materiales y Métodos La formación de L-cit se determinó según Bredt y Snyder (1990). Las reacciones enzimáticas se llevaron a cabo a 25ºC en Tris-HCl 50 mM, pH 7,4, conteniendo L-arg 100 mM, 1 mCi [3H] Arg (40 a 70 Ci / mmol; Perkin-Elmer), NADPH 100 mM, FAD 10 mM, CaCl2 2mM, CaM 1mg, y 100 µM de H4B en un volumen final de 40 µl. Las reacciones enzimáticas se iniciaron mediante la adición de OtNOS 0,5 mM y finalizaron después de 30 min mediante la adición de 400 ml de acetato sódico 20 mM, pH 5,5, conteniendo L-citrulina 1 mM, EDTA 2 mM y EGTA 0,2 mM (tampón de corte). Las muestras se aplicaron a columnas con 1 ml Dowex AG50W-X8, forma Na+ (Bio-Rad), previamente equilibrada con tampón de corte. La L-cit se eluyó con 2 ml de agua destilada. Alícuotas de 0,5 ml del eluído se disolvieron en 10 ml de líquido centelleo, y se midió la radiactividad en un contador Beckman LS 3801 en un sistema de centelleo líquido. La formación de L-citrulina se verificó mediante cromatografía en capa delgada. 7.3. Oxidación del NADPH. La oxidación de NADPH se cuantificó en un volumen de 500 µl conteniendo OtNOS 0,5 mM, Tris-HCl 50 mM, pH 7,6, DTT 5 mM, L-arg 100 mM, NADPH 0,5 mM, CaCl2 10mM, CaM 10 mM, H4B 10 µM, y 100 unidades/ml de catalasa. La tasa de disminución de la absorbancia en 340 nm se evaluó durante 10 min a 25º C utilizando un espectrofotómetro (Ultrospec 1100 Pro, Amersham Biosciences). Un coeficiente de extinción de 6,22 µM-1cm-1 en 340 nm se utilizó para calcular la tasa de oxidación de NADPH. 8. Modelado de estructuras por homología. Los modelos estructurales se construyeron con el programa de MODELERv 9.5 (Sali y Blundell., 1993) utilizando ClustalX y FFAS03 derivados. La plantilla se seleccionó con FFAS03 y correspondió a la estructura cristalina de la región de NOSoxy Bos taurus eNOS y de la región NOSred de nNOS de Rattus norvegicus (Jaroszewski y col., 2005). El modelo se validó utilizando un programa de PROSA II (Wiederstein y Sippl, 2007), y las cifras se extrajeron utilizando el programa Web Lab ViewerLite 3,20 (Molecular Simulations). La superposición estructural de OtNOS y B. taurus eNOS se realizó con el programa Superpose (Maiti y col., 2004). OtNOS y las secuencias de NOS de humanos fueron alineados utilizando el programa ClustalX (versión 1.81; Thompson y col., 1997) y se editó con el programa GENEDOC (versión 2.5.010). Las secuencias de proteína con similitud a OtNOS (E-valor <1.102110) se recuperaron 87 _________________________________________________Materiales y Métodos utilizando BLAST y la base de datos no redundante de GenBank. El programa CDHIT se utilizó para eliminar todo el intercambio de secuencias > 90% de identidad (Huang y col., 2010). Las secuencias fueron alineadas con ClustalX y editadas con el programa GENEDOC. La organización del dominio oxi se analizó con InterPro (Hunter y col., 2009) y PFAM (Finn y col., 2008). 9. Análisis filogenético de OtNOS. El análisis filogenético se realizó con PHYML (Guindon y Gascuel, 2003). El mejor modelo evolutivo para la inferencia filogenética se estimó utilizando ModelTest (Posada y Crandall, 1998). Un “bootstrapping” no paramétrico (1000 repeticiones) se utilizó para evaluar el apoyo de las ramificaciones del árbol. El programa de iTool (Letunic y Bork, 2007) se utilizó para mostrar los árboles filogenéticos (http://itol.embl.de/). 10. Análisis estadísticos. Los resultados se expresaron como error y desvío estándar. Los análisis estadísticos se realizados con Sigma Stat (Jandel Scientific), se realizó también un análisis de varianza para comparaciones múltiples (ANOVA) y un test de t para la comparación entre pares. 11. Números de Accesos. Las secuencias de aminoácidos utilizadas en este trabajo se encuentran GenBank/EMBL con los siguientes números de accesos: O. tauri NOS (CAL57731), eNOS (NP_000594), nNOS (NP_000611), iNOS (AAI30284) y Spirosoma linguale NOS (YP_003391026). Las estructuras cristalizadas utilizadas en este trabajo se encuentran en Protein Data Bank (www.rcsb.org) según los siguientes códigos: R. norvegicus nNOSred (1tll) y B. taurus eNOSoxy (1fop). 12. Cuantificación de NO en cultivos de O. tauri y E. coli. Los cultivos O. tauri se crecieron en Erlenmeyers con medio K (Keller y col, 1987) a 18º C bajo fotoperíodo 12 h: 12 h (luz: oscuridad).Los experimentos con luz se llevaron 88 a cabo con irradiaciones que van desde 40 a 400 µmol.m-2.s-1. El contenido de NO en E. coli o en O. tauri se cuantificó utilizando la sonda fluorescencia DAF-FM DA _________________________________________________Materiales y Métodos (Invitrogen). DAF-FM DA 10µM se añadió al medio de cultivo 20 min antes de la medición. La producción de NO fue iniciada por la adición del sustrato L-arg. La intensidad de fluorescencia (excitación de 480 nm, emisión de 525 nm) se midió utilizando un lector de placas de fluorescencia (Fluoroskan Ascent, Thermo Electron) o visualizada en un microscopio invertido de fluorescencia (Nikon Eclipse TI). La formación de NO en E. coli también se midió utilizando reactivo Griess para la medición de nitrito como según Xu y col. (2000). 13. Análisis de la muerte celular en E. coli. Los cultivos de E. coli se incubaron con H2O2 30 mM durante 30 min. Las células se tiñeron con el colorante nuclear Sytox Green (Molecular Proves) el cual es impermeable a la membrana de células vivas. Las células se incubaron durante 10 min en oscuridad a temperatura ambiente y luego se lavaron con 1,5 volúmenes de medio LB fresco. La intensidad de fluorescencia (excitación 480 nm; emisión 525 nm) se midió utilizando un lector de placas de fluorescencia (Fluoroskan Ascent, Thermo Electron). 14. Extracción de ADN, ARN y proteína de Ostreococcus tauri. 14.1. Extracción de ADN de O. tauri. Se precipitaron 50 ml de cultivo de O. tauri y se descartó el sobrenadante, se trtituró el precipitado utilizando un pilón para tubos de microcentrífuga. Se adicionó a cada tubo 400 µl de solución de extracción Tris-HCl 200 mM, pH 7,5, NaCl 250 mM, EDTA 25 mM y SDS 0,5% (p/v) y el homogenato fue resuspendido con vortex por 5 s. El extracto se centrifugó a 13000 g y 4ºC durante 2 min. Se transfirieron 300 µl del sobrenadante a un tubo nuevo y se le adicionaron 300 µl de isopropanol frío. Se mezclaron los contenidos por inversión suave seguida de 2 min de incubación a temperatura ambiente. Después se centrifugó a 13.000 g a 4º C por 5 min. Se descartó el sobrenadante y al sedimento se le agregaron 400 µl de etanol 70 % (v/v). Se resuspendió el sedimento en vortex por 5 min. Posteriormente se centrifugó a 13.000 g por 1 min. Este lavado remueve impurezas que acompañan al ADN. El lavado se repitió una vez más y a continuación el precipitado fue secado a temperatura ambiente y se resuspendió en 100 µl de agua mili-Q estéril. 89 14.1.1. Verificación de la presencia del gen que codifica para OtNOS en O. tauri. _________________________________________________Materiales y Métodos El ADN genómico previamente extraído se calentó a 95° C durante 5 min en termobloque y luego se colocó rápidamente en hielo. Para cada reacción de PCR se utilizó 1 µl de la suspensión de ADN genómico y se utilizaron los cebadores OtNOS632Fw y OtNOS632Rv. 14.2. Extracción de ARN de O. tauri. Para la extracción de ARN a partir de O. tauri se utilizó el Kit ARNeasy (Qiagen), aplicando el protocolo diseñado para purificación de ARN total de cultivos celulares de plantas y hongos filamentosos. Es esencial usar la cantidad correcta de material de partida a fin de obtener un óptimo rendimiento y pureza. Se precipitaron 100 ml de cultivo de O. tauri. Se resuspendió en 450 µM de tampón RLT (Qiagen), y se continuó con el protocolo descripto en el manual de ARNeasy 14.3. Síntesis del ADN complementario (Retrotranscripción). En un tubo de microcentrífuga se agregaron 2 pmoles de cebador oligo (dT) comercial, 12 µl del ARN tratado con ADNasa y 1 µl de mezcla de dNTPs a una concentración de 2,5 mM cada uno, resultando un volumen final de 13 µl. Se incubó durante 5 min a 65°C e inmediatamente después se colocó en hielo, durante al menos 1 min. Luego se agregaron 4 µl de First Strand Tampón 5X (Invitrogen), 1 µl 0,1 M DTT, 1 µl de inhibidor de ARNsa (Invitrogen) y 0,5 µl de transcriptasa reversa SuperScript III (200 unidades/µl) (Invitrogen). Posteriormente se incubó por 60 min a 55°C. La reacción se detuvo por incubación a 70° C durante 15 min. 14.4. Amplificación de ADN complementario específico mediante PCR. Se utilizaron los cebadores OtNOS2203Fw y OtNOS2537Rv para amplificar el cADN de OtNOS (Tabla IV). Para cada reacción se utilizó 1 µl de la muestra de cADN, 1,25 U de Taq ADN polimerasa Go Taq™ (Promega), 0,2 µM de cada cebador, 0,2 mM de cada dNTP, MgCl2 2 mM, y tampón 1X provisto por el fabricante de la enzima, en un volumen final de 25 µl. El ARN genómico previamente extraído se calentó a 95° C durante 5 min en termobloque y se colocó rápidamente en hielo. Para cada reacción de PCR se utilizó 1 µl de la suspensión de ADN. 14.5. Extracción de proteína de O. tauri. 90 Se precipitaron 2 ml de un cultivo de O. tauri por centrifugación. Se descartó el sobrenadante. A los precipitados se les adicionó solución de siembra Tris-HCl 60 mM pH 6,8, ditiotreitol (DTT) 500 mM, SDS 2 % (p/v), glicerol 10 % (v/v), azul de _________________________________________________Materiales y Métodos bromofenol 25 µg/ml. Se los desnaturalizó durante 5 min a 100°C en un termobloque y luego se los congeló en nitrógeno liquido. Se realizaron 3 ciclos de cambios de temperatura. El homogenato se centrifugó a 4°C y 10000 rpm durante 5 min. Se recuperó el sobrenadante de los tubos. 14.5.1. Separación de proteínas mediante electroforesis en gel de poliacrilamida, SDS-PAGE. Las muestras se corrieron en geles de concentración de 5 % (p/v) acrilamida 5%/ bisacrilamida 0,13 % en una solución de Tris-HCl 0,12 M, pH 6,8, SDS 0,1 % (p/v) y en geles de separación de 12 % (p/v) acrilamida 15% /bisacrilamida 0,4% en TrisHCl 0,75 M pH 8, SDS 0,1 % (p/v), persulfato amónico 0,1 % y Temed 0,05%. Las electroforesis fueron desarrolladas a una intensidad constante de 20-25 mA con tampón de corrida Tris-HCl 25 mM, glicina 0,2 M pH 8,3, EDTA 1 mM y SDS 0,05 % (p/v). Los géles fueron teñidos con azul de Coomassie R-250 0,2 % (p/v) en etanol: ácido acético: agua (50:10:40) durante 1 h. 14.5.2. Transferencia e inmunodetección de proteínas en membranas de nitrocelulosa por Western Blot. Las proteínas separadas electroforéticamente en geles de poliacrilamida fueron electrotransferidas a membranas de nitrocelulosa en una cuba de transferencia (Transblot, Bio-Rad). La transferencia se llevó a cabo en una solución conteniendo Tris-HCl 20 mM, glicina 150 mM, pH 8,0, SDS 0,02 % (p/v) y metanol 20 % (v/v), durante 1 h con agitación, a una intensidad constante de 100 volt y a 4°C. Una vez completada la transferencia, la membrana fue bloqueada con solución PBS/leche Na2HPO4 80 mM , NaH2PO4 20 mM pH 7,5, NaCl 100 mM y leche descremada 5 % (p/v). Se realizaron 4 lavados de 15 min cada uno, con agitación. A continuación se incubó la membrana durante 2 h con el anticuerpo primario (IgG de conejo anti-iNOS de macrófago murino, Sigma Aldrich nº N7782 o IgG de conejo anti-OtNOS, Gen Script) diluidos 1:1000 en 12 ml de solución PBS/leche. Después se realizaron 4 lavados de 15 min con solución de bloqueo y posteriormente la membrana se incubó durante 1 h con el anticuerpo secundario Immun-StartTM (BioRad) anti IgG de conejo conjugado a la enzima fosfatasa alcalina (dilución 1:5000). Luego se realizaron 4 lavados de 15 min con solución PBS/leche y un lavado de 10 min con una solución conteniendo Tris-HCl 100 91 mM pH 9,5, NaCl 100 mM, MgCl2 5 mM. Finalmente, la membrana fue sumergida en una solución de igual composición con el agregado de 5-bromo-4-cloro-3- indolilfosfato (BCIP) 0,01 % (p/v) y azul de nitrotetrazolio (NBT) 0,01 % (p/v) e incubada en oscuridad a 37° C hasta el desarrollo de color. _________________________________________________Materiales y Métodos 15. Generación de plantas transgénicas. 15.1. Transformación transiente de Nicotiana tabacum. Se cultivó un preinóculo de la cepa GV3101:pMP90 de A. tumefaciens transformada con el plásmido de interés en 2 ml de medio 2YT suplementado con 100 µg/ml de rifampicina, 50 µg/ml de gentamicina y 50 µg/ml de cloranfenicol durante 12 h a 28°C bajo agitación contante. Se realizó una dilución 1/100 del cultivo saturado en 10 ml de 10 mM 2YT–MES (pH 5,6) suplementado con acetosiringona 20 µM y con las mismas concentraciones de antibióticos del paso anterior con agitación a 28°C hasta alcanzar un DO600 de 1-2. El cultivo fue centrifugado a 1000 g durante 20 min a 25°C. Luego se resuspendió en una solución MES 10 mM pH 5,6, MgCl2 10 mM y acetosiringona 100 µM llevando a una DO600 final de 0,2–0,5. Se dejó 2 a 3 h a 25°C sin agitación y luego se infiltraron hojas de N. benthamiana con esa solución. Luego de 1 y 3 días en la cámara de crecimiento se cosecharon las hojas para su análisis. Para las plantas co infiltradas con H4B 100µM, se agregó el cofactor a la solución de agroinfiltración anteriormente mencionada. 15.2. Crecimiento y transformación estable de las plantas de Nicotiana tabacum. La transformación de plantas de N. tabacum se realizó siguiendo el método descrito por Gallois y Marinho (1995). Se cultivó un preinóculo de la cepa GV3101:pMP90 de A. tumefaciens transformada con el plásmido de interés en 2 ml de medio 2YT suplementado con 100 µg/ml de rifampicina, 50 µg/ml de gentamicina y 80 µg/ml de espectinomicina durante 12 h a 28–30°C bajo agitación constante. Se realizó una dilución 1/100 del cultivo saturado en 10 ml de medio 2YT suplementado con las mismas concentraciones de antibióticos del paso anterior, bajo agitación a 28– 30°C hasta DO600 de 1. Se colocaron 2,5 ml del cultivo en una caja de Petri con 50 ml de medio líquido (MS-0) Murashige-Skoog (1962). Se usaron plántulas de N. tabacum cultivar Petit Havana (PH) SR1 de 6 a 8 semanas crecidas en cajas Magenta con MS-0, agar 0,8 % (p/v). Se cortaron sus hojas, se descartó la nervadura central y se obtuvieron fragmentos de aproximadamente 1,2 cm de lado. Los cortes se realizaron sobre papel de filtro estéril mojado sobre medio MS-0 92 líquido. Los fragmentos de hojas fueron transferidos a la caja de Petri con el A. tumefaciens y se incubaron por 20 min. _________________________________________________Materiales y Métodos Los fragmentos se removieron y escurridos sobre papel de filtro y transferidos a caja de Petri con MS-10 (MS-0 suplementado con 0,1 mg/ml de kinetina y 0,1 mg/ml de auxina), agar 0,8 % (p/v), apoyando la parte superior de la hoja sobre el medio. Se colocaron entre 8 y 10 trozos de hoja por placa. El control de regeneración consistió en fragmentos de hoja sin inocular incubados en MS-10, agar 0,8 % (p/v) y el control de selección en trozos de hoja sin inocular incubados en MS-10, agar 0,8 % (p/v) con Kanamicina 100 µg/ml. Los trozos de hojas se cultivaron a 25°C durante 3 días en oscuridad. Después se transfirieron los fragmentos a placas con medio MS-10, agar 0,8 % (p/v) suplementado con Kanamicina 100 µg/ml y trifamox 300 µg/ml. Se cultivó durante una semana y luego los trozos de hojas fueron transferidos a placas con medio MS-10, 0,8 % (p/v) agar suplementado con Kanamicina 100 µg/ml y cefotaxime 300 µg/ml. Se cultivaron durante 4 a 6 semanas a 25° C y 16 h de luz, realizándose transferencias a nuevas placas con idéntico medio cada una semana. Cuando se formaron vástagos de 1–2 cm de longitud, fueron transferidos a cajas Magenta con MS-0, agar 0,8 % (p/v) y Kanamicina 100 µg/ml. Los vástagos que formaron raíces fueron posteriormente transferidos a tierra. 15.2.2. Esterilización de las semillas de N. tabacum. Se trabajó en condiciones de esterilidad en flujo laminar horizontal. Las semillas se incubaron durante 15 min en Tween 20 0,05 % (v/v), hipoclorito de sodio comercial 1,5 % (v/v). Luego se descartó el sobrenadante y se realizaron 4 lavados sucesivos con agua destilada estéril. 15.2.3. Cultivo de N. tabacum en macetas. Las plantas de tabaco se cultivaron individualmente en macetas de plástico de 13 cm de diámetro con tierra en cámara de cultivo con fotoperíodo de día largo (16 h de luz y 8 h de oscuridad), una temperatura de 29°C/23°C durante el ciclo luz/oscuridad, 150 µE/m2s de intensidad de luz y una humedad de 90 % durante el período de oscuridad y 40 % durante el período de luz. Las plantas se regaron con agua cada 2-3 días. 93 15.3. Crecimiento y transformación estable de las plantas de Arabidopsis thaliana. 15.3.1. Crecimiento de A. thaliana. _________________________________________________Materiales y Métodos Las semillas se sembraron por dispersión sobre potes con tierra orgánica, previa estratificación de las mismas a 4ºC por 2-4 días en oscuridad. El sustrato se humedeció con agua por capilaridad desde una bandeja. Luego los potes fueron cubiertos con “film” plástico durante 15 días. Se realizaron riegos con agua cada 4 días. Las plantas se mantuvieron en cámara de cultivo con un flujo de luz de 130-150 µE/m2s, en un fotoperíodo de 16 h de luz y 8 h de oscuridad (día largo). La temperatura de crecimiento fue de 22°C/18°C durante el ciclo luz/oscuridad y la humedad se mantuvo en 80 %. 15.3.2. Transformación estable de A. thaliana. Se empleó la técnica de “floral dip” (Clough y Bent, 1998) para transformar los ecotipos Columbia 0 (Col-0) y Col-0 rdr6 (Luo y col., 2007) de A. thaliana. La cepa GV3101:pMP90 de A. tumefaciens transformada con el plásmido de interés fue crecida en 250 ml de medio 2YT suplementado con rifampicina100 µg/ml, gentamicina 50 µg/ml y espectinomicina100 µg/ml hasta DO600 mayor a 2. El cultivo se centrifugó a 5000 g durante 10 min a 25°C y resuspendido en 500 ml de medio de infiltración sacarosa 5 % (p/v) y Silwet L-77 0,02 % (v/v). Las inflorescencias de las plantas de A. thaliana (regadas el día anterior) se sumergieron en la suspensión de bacterias durante 30 s. Las plantas se mantuvieron en cámara de cultivo en posición horizontal sobre bandejas cubiertas con papel absorbente para retener la humedad luego de la inoculación y mantenerlas en oscuridad. Luego de 24 h se volvieron a la posición vertical y a las condiciones normales de crecimiento hasta el momento de la cosecha de las semillas (entre 3 y 4 semanas). 15.3.3. Esterilización de las semillas de A. thaliana. Se trabajó en condiciones de esterilidad en flujo laminar horizontal. Las semillas se incubaron durante 15 min en solución de Tween 20 0,05 % (v/v) y hipoclorito de sodio 30 % (v/v). Luego se hizo se lavó las semillas 6 veces con agua. 15.3.4. Selección de plantas transformantes de A. thaliana. Para seleccionar las plantas transformadas y resistentes a Kanamicina, las semillas estériles fueron dispersadas en placas de Petri con medio MS-0 al medio sin 94 sacarosa con agar 0,8 % (p/v) (0,5X MS-0) y Kanamicina 100 µg/ml A los 10 días las plantas fueron transferidas a potes con tierra orgánica en cámara de cultivo. Las plántulas para los tratamientos con NaCl 100mM y ABA 10 µM se crecieron en medio ATS con KNO3 5 mM; Ca (NO3)2 2 mM; MgSO4.7H2O 2mM; KPO4 _________________________________________________Materiales y Métodos 2,5 mM; H3BO3 70 µM; y los micronutrientes: MnCl2 14 µM; ZnSO4 1 µM; CuSO4 0,5 µM; Na2MoO4 0,2 µM; NaCl 10 µM; CaCl2 0,01 µM; y Fe-EDTA 50 µM. 16. Análisis de las plantas transformadas. 16.1. Evaluación estadística. La segregación 3:1 del gen marcador seleccionable NPTII, que confiere resistencia a Kanamicina, fue evaluada estadísticamente por análisis de chi-cuadrado (χ2). Los tests se realizaron con un nivel de significación de 0,05 (Murgia y col., 2004). 16.2. Extracción de ADN genómico a partir de tejido foliar. Se pulverizaron 2-3 discos de hoja de 1cm de diámetro de cada planta de N. tabacum o 1-2 hojas (según el tamaño) de cada planta de A. thaliana utilizando un pilón para tubos de microcentrífuga con nitrógeno líquido en cantidad necesaria. Se adicionó a cada tubo 400 µl de solución de extracción Tris-HCl 200 mM (pH=7,5), NaCl 250 mM, EDTA 25 mM, y SDS 0,5% (p/v) y el homogenato fue resuspendido con vortex por 5 s. Luego fue centrifugado a 13000 g y 4ºC por 2 min. Se transfirieron 300 µl del sobrenadante a un tubo nuevo y se le adicionaron 300 µl de isopropanol frío. Se mezclaron los contenidos por inversión suave seguida de 2 min de incubación a temperatura ambiente. Después se centrifugó a 13.000 g y 4ºC por 5 min. Se descartó el sobrenadante y al sedimento se le agregó 400 µl de etanol 70 % (v/v). Se resuspendió el sedimento en vortex por 5 min. Posteriormente se centrifugó a 13.000 g por 1 min. El lavado se repitió una vez más y a continuación el sedimento se secó a temperatura ambiente para ser resuspendido en 100 µl de agua mili-Q estéril a 4ºC. Finalmente se hizo una centrifugación a temperatura ambiente durante 2 min y se transfirieron 90 µl a un tubo nuevo. Se usaron 1-2 µl de esta preparación en un volumen final de reacción de PCR de 20 µl. 16.3. Verificación de la presencia del transgén que codifica para OtNOS en las plantas transformadas. El ADN genómico previamente extraído se calentó a 95° C durante 5 min en termobloque y luego colocado rápidamente en hielo. Para cada reacción se utilizó 1 µl 95 de la suspensión de ADN genómico. Para analizar los productos se efectuó una separación electroforética en un gel de agarosa al 1% (p/v) y las muestras fueron teñidas con Saber Safe (Invitrogen). 16.4. Verificación de la presencia de la proteína OtNOS en las plantas transformadas. _________________________________________________Materiales y Métodos 16.4.1. Extracción de proteínas. Las muestras de tejido foliar fresco o congelado fueron pulverizadas utilizando un pilón para tubos de microcentrífuga con nitrógeno líquido en cantidad necesaria. Luego se adicionó solución de homogenización con la siguiente composición: Tris-HCl 50 mM pH 8, MgCl2 1mM, EDTA 3mM, fenil metil sulfonil fluoruro (PMSF) 1mM, dtiotreitol (DTT) 1mM, agregando 2 ml de la solución por g de tejido fresco. El homogenato fue centrifugado a 4°C y 5000 g durante 2 min. Se recuperó el sobrenadante de los tubos para ser analizado por electroforesis en gel de poliacrilamida en condiciones desnaturalizantes (SDS-PAGE). Según las condiciones descriptas en los incisos 14.5.1 y 14.5.2. 16.5. Verificación mediante RT-PCR de la presencia del transcripto del transgén que codifica para OtNOS. 16.5.1. Extracción de ARN. Se maceraron 100 mg de tejido foliar de cada planta con nitrógeno liquido en mortero. Luego se agregó 0,8 ml de TRIzol (Invitrogen) y se centrifugó a 12000 g y 4°C por 10 min. El homogenato fue transferido a un nuevo tubo, en el cual se agregaron 400 µl de cloroformo. Se invirtió vigorosamente durante 15 s. Se centrifugó a 12000 g y 4°C durante 15 min. La fase superior fue transferida a nuevo tubo, se le agregaron dos volúmenes de isopropanol y se incubó por 30 min a 25°C. Posteriormente se centrifugó a 12000 g y 4°C durante 10 min y se descartó el sobrenadante. Después se agregó 1 ml de etanol frío 70 % (v/v) y se agitó. Se centrifugó a 7500 g y 4°C durante 5 min y se descartó el sobrenadante. El lavado con etanol 70 % (v/v) fue repetido. El sedimento fue secado dejando el tubo abierto a 25° C durante 5–10 min. Finalmente se resuspendió en 40 µl de H2O mili-Q previamente tratada con dietil pirocarbonato (DEPC). 16.5.2. Estimación de la concentración y el grado de pureza del ARN. Se midió absorvancia a 260 nm en un espectrofotómetro Ultrospec en cubeta de cuarzo para estimar la concentración de ARN total. La relación entre las absorvancia en 260 y 280 nm se determinó para descartar la contaminación con 96 proteínas. Una muestra relativamente pura presentó una relación cercana a 2. Se midió también la relación entre las absorvancias en 260 y 230 nm para comprobar la ausencia de contaminación con compuestos orgánicos como fenol. Un valor cercano a 2 indica una muestra relativamente pura. 16.5.3. Verificación de la integridad del ARN. _________________________________________________Materiales y Métodos El grado de integridad del ARN se determinó mediante electroforesis en gel de agarosa 1,5 % (p/v). Previo a la siembra, las muestras se calentaron a 65° C durante 5 min y llevadas inmediatamente a hielo. Se sembró 1 µg de cada una, agregándosele solución colorante para ácidos nucleicos (Saber safe). Se utilizó agarosa al 1,5 % (p/v), preparada en tampón 1X TAE. 16.5.4. Tratamiento del ARN con nucleasas de ADN (ADNsa). El tratamiento del ARN total con ADNasa permite degradar el ADN genómico contaminante que no se ha podido eliminar completamente durante la extracción del ARN. Se añadieron en un tubo de microcentrífuga 4 µg de ARN total, 2 µl de tampón ADNasa (Promega), 1 µl de ADNasa (Promega) y una cantidad suficiente de H2O miliQ previamente tratada con DEPC para un volumen final de 20 µl. Se incubó 30 min a 37°C. Luego se agregó 1 µl de ADNasa Stop Solution (Promega) y se incubó por 10 min a 65° C. Se realizó la síntesis del ADN complementario y la amplificación del ADN complementario especifico mediante PCR según lo descripto en los puntos 14.3 y 14.4. 16.6. Cuantificación de nitritos. La cuantificación de nitritos se realizó con el reactivo de Griess (Sun y col., 2003). Para lo cual se procesaron 5 plántulas de A. thaliana o 100 mg de tejido de hojas de N. tabacum. Las muestras de tejido foliar fresco o congelado se pulverizaron utilizando un pilón para tubos de microcentrífuga con nitrógeno líquido en cantidad necesaria. Luego se adicionó solución de homogenización de la siguiente composición: Tris-HCl 50 mM pH 7,5 y NaCl 10 mM agregando 2 ml de la solución por g de tejido fresco. El homogenato se centrifugó a 4°C y 5000 g durante 2 min. Se recuperó el sobrenadante de los tubos. Se realizó la cuantificación de proteínas mediante el reactivo de Bradford según Kruger (2002) y luego se relativizó la cantidad de Nitritos cuantificados por miligramos de proteínas. 17. PCR 97 En líneas generales la reacción en cadena de la Taq ADN polimerasa se realizó con las condiciones estandarizadas. Es decir la solución de reacción provista por el vendedor MgCL2 1,5mM, dNTPs 0,5 mM de cada uno, cebadores 0,25 µM de cada uno y unidades de ADN taq polimerasa de acuerdo a lo sugerido por el proovedor. Las reacciones se realizaron en un termociclador Mastercycler (Eppendorf). Para las reacciones de PCR realizadas para analizar colonias de bacterias se utilizó Taq polimerasa (Invitrogen), para los productos de PCR que se clonaron se _________________________________________________Materiales y Métodos utilizó Pfu (Promega), la cual posee capacidad de control de calidad y reduce la probabilidad de introducir un error durante la amplificación. 98 _________________________________________________Trabajos presentados TRABAJOS PRESENTADOS PUBLICACIONES EN REVISTAS CON REFERATO Foresi N, Claudia V, Tonon CV, Casalongué CA, Lamattina L (2007) Extracellular ATP induces nitric oxide production in tomato cell suspensions. Plant Physiology 145:589– 592. Foresi N, Sueldo DJ, Casalongué CA, Lamattina L (2010) Phosphatidic acid formation is required for extracellular ATP-mediated nitric oxide production in suspension cultured tomato cells. New Phytology 185:909–916. Foresi N, Correa-Aragunde N, Parisi G, Caló G, Salerno G y Lamattina L (2010) Characterization of a Nitric Oxide Synthase (NOS) from the plant kingdom: The NO generation from the green algae Ostreococcus tauri is light irradiance- and growth phase dependent. Plant cell 22:3816–3830. CAPÍTULOS DE LIBRO Foresi N, Tonón C, Gonorazky G, Lamattina L, Laxalt AM, Casalongué CA. Extracellular ATP and Nitric Oxide Impact on Cell Viability in Tomato Suspension Cells. Nova Publishers (2010) 13. CONGRESOS NACIONALES Laxalt A, Tonón C, Foresi N, Casalongué C,A Lamattina L (2006) Extracellular ATP induces NO production in tobacco and tomato cultured cells. XLII Reunión Anual de la Sociedad Argentina de Investigaciones en Bioquímica y Biología Molecular. Rosario, Santa Fe, Argentina. 99 Foresi N, Casalongué C, Lamattina L, Laxalt A (2007) Extracellular ATP (eATP) activates phospholipid signalling in tomato cell suspensions. XLIII Reunión Anual de la Sociedad Argentina de Investigaciones en Bioquímica y Biología Molecular. Mar del Plata, Buenos Aires, Argentina. _________________________________________________Trabajos presentados Foresi N, Sueldo D, Casalongué C, Lamattina L, Laxalt A (2008) Extracellular ATP (eATP) induces Phosphatidic Acid and Nitric Oxide in tomato cell suspensions. XXV Reunión Argentina de Fisiología Vegetal. Rosario, Buenos Aires, Argentina. Sueldo D, Foresi N, Casalongué C, Lamattina L, Laxalt A (2008) Extracellular ATP (eATP)-signalling in plants: role of Phopholipase, Ca2+ and NO. XLIV Reunión Anual de la Sociedad Argentina de Investigaciones en Bioquímica y Biología Molecular. Villa Carlos Paz, Córdoba, Argentina. Foresi N, Correa-Aragunde N, Casalongué C, Lamattina L (2009) First characterization of a Nitric Oxide Synthase from the plant kingdom. XLV Reunión Anual de la Sociedad Argentina de Investigaciones en Bioquímica y Biología Molecular, San Miguel de Tucumán, Argentina. Foresi N, Correa-Aragunde N, Calo G, Salerno G, Lamattina L (2010) Nitric oxide generation by the unicellular marine green alga Ostreococcus tauri. XLVI Reunión Anual de la Sociedad Argentina de Investigaciones en Bioquímica y Biología Molecular, Puerto Madryn, Argentina. Correa-Aragunde N, Foresi N, Lamattina L (2010) Nitric oxide and auxin regulates ascorbate peroxidase 1 activity through nitrosylation. Reunión Anual de la Sociedad Argentina de Investigación en Bioquímica y Biología Molecular. Potrero de los Funes, San Luis, Argentina. Foresi N, Correa-Aragunde N, Lamattina L (2011) ¿Es posible expresar una Óxido Nítrico Sintasa activa en plantas? Biólogos en red. Mar del Plata, Buenos Aires, Argentina. Foresi N, Mayta M, Lodeyro A, Casalongué C, Carrillo N, Lamattina L (2012) Sobreexpresión de Óxido Nítrico Sintasa de Ostreococcus tauri (OtNOS) en 100 Arabidopsis y tabaco. Comportamiento de las plantas transgénicas frente a estrés. XXIX Reunión Argentina de Fisiología Vegetal. Mar del Plata, Buenos Aires, Argentina CONGRESOS INTERNACIONALES _________________________________________________Trabajos presentados Foresi N, Correa-Aragunde N, Lamattina L (2010) Characterization of a Nitric Oxide Synthase from a member of plant kingdom, the algae Ostreococcus tauri. XXXIX Reunión Anual de la Sociedad Brasileira de Bioquímica e Biología Molecular, Foz de Iguazú, Brasil. Correa-Aragunde N, Foresi N, Lamattina L (2010) Auxin partially inhibits APX1 activity through denitrosylation leading to hydrogen peroxidase accumulation and lateral root formation in Arabidopsis. The 7th International Conference on the Biology, Chemistry and Therapeutic Applications of Nitric Oxide. Edinburgh, Scotland. 101 ______________________________________________Referencias Bibliográficas REFERENCIAS BIBLIOGRÁFICAS Able AJ (2003) Role of reactive oxygen species in the response of barley to necrotrophic pathogens. Protoplasma 221: 137-143. Adak S, Bilwes AM, Panda K, Hosfield D, Aulak KS, McDonald JF, Tainer JA, Getzoff ED, Crane BR, Stuehr DJ (2002a) Cloning, expression, and characterization of a nitric oxide synthase protein from Deinococcus radiodurans. Proc Natl Acad Sci USA 99: 107-112. Adak S, Aulak KS, Stuehr, DJ (2002b) Direct evidence for nitric oxide production by a nitric-oxide synthase-like protein from Bacillus subtilis. J Biol Chem 277: 16167-16171. Adoutte A, Balavoine G, Lartillot N, Lespinet O, Prud’homme B, y de Rosa R (2000) The new animal phylogeny: Reliability and implications. Proc Natl Acad Sci USA 97: 4453-4456. Agapie T, Suseno S, Woodward JJ, Stoll S, Britt RD, Marletta MA (2009) NO formation by a catalytically self-sufficient bacterial nitric oxide synthase from Sorangium cellulosum. Proc Natl Acad Sci USA 106: 16221-26. Aita VM, Ahmad W, Panteleyev AA, Kozlowska U, Kozlowska A, GilliamTC, Jablonska, S y Christiano, AM (2000) A novel missense mutation (C622G) in the zinc-finger domain of the human hairless gene associated with congenital atrichia with papular lesions. Exp. Dermatol 9: 157-162. Albakri Q.A. y Stuehr DJ (1995) Intracellular assembly of inducible NO synthase is limited by nitric oxide-mediated changes in heme insertion and availability. J Biol Chem 271: 5414-5421. Alderton WK, Cooper CE y Knowles RG (2001) Nitric oxide synthases: Structure, function and inhibition. Biochem J 357: 593-615. 102 Aoyagi M, Arvai AS, Tainer JA y Getzoff ED (2003). Structural basis for endothelial nitric oxide synthase binding to calmodulin.EMBO J 22: 766-775. ______________________________________________Referencias Bibliográficas Arasimowicz M y Floryszak-Wieczorek J (2007) Nitric oxide as a bioactive signaling molecule in plant stress responses. Plant Sci 172: 876-887. Archibald JM (2009) Green evolution, green revolution. Science 324: 191-192. Arnold WP, Mittal CK, Katsuki S, Murad F (1977) Nitric oxide activates guanylate cyclase and increase guanosine 3´, 5´-cyclic monophosphate levels in various tissue preparations. Proc Natl Acad Sci USA 74: 3203-3207. Asai S y Yoshioka H (2009) Nitric oxide as a partner of reactive oxygen species participates in disease resistance to necrotrophic pathogen Botryis cinerea in Nicotiana benthamiana. Mol Plant-Microbe Interact 22: 619-629. Barroso JB, Corpas FJ, Carreras A, Sandalio LM, Valderrama R, Palma JM, Lupianezi JA, del Rio LA (1999) Localization of nitric oxide synthase in plant peroxisomes. J Biol Chem 274: 36729-36733. Beligni MV y Lamattina L (1999a) Is nitric oxide toxic or protective? Trends Plant Sci 4: 299-300. Beligni MV y Lamattina L (1999b) Nitric oxide counteracts cytotoxic processes mediated by reactive oxygen species in plant tissues. Planta 208: 337-344. Beligni MV y Lamattina L (2000) Nitric Oxide stimulates seed germination and deetiolation and inhibits hypocotyl elongation, three light-inducible responses in plants. Planta 210: 215-221. Beligni MV, Fath A, Bethke PC, Lamattina L y Jones RL (2002) Nitric oxide acts as an antioxidant and delays programmed cell death in barley aleurone layers. Plant Physiol 129: 1642-1650. Beligni MV y Lamattina L (2002) Nitric oxide interferes with plant photo- oxidative stress by detoxifying reactive oxygen species. Plant Cell Environ 25: 737-748. 103 Benamar A, Rolletschek H, Borisjuk L, Avelange-Macherel M, Curien G, Mostefai HA, Andriantsitohaina R. y Macherel D (2008). Nitrite-nitric oxide control of mitochondrial respiration at the frontier of anoxia. Biochim Biophys Acta 1777: 1268-1275. ______________________________________________Referencias Bibliográficas Bergman B, Gallon JR, Rai AN y Stal LJ (1997) N2 Fixation by non-heterocystous cyanobacteria. FEMS Microbiol Rev19 139-185. Bethke PC, Badger MR y Jones RL (2004) Apoplastic synthesis of nitric oxide by plant tissues. Plant Cell 16: 332-341. Bhattacharya D y Medlin L (1998) Algal phylogeny and the origin of land plants. Plant Physiol 116: 9-15. Bird LE, Ren J, Zhang J, Foxwell N, Hawkins AR, Charles IG y Stammers DK (2002) Crystal structure of SANOS, a bacterial nitric oxide synthase oxygenase protein from Staphylococcus aureus. Structure 10: 1687-1696. Bredt DS y Snyder SH (1990) Isolation of nitric oxide synthetase, a calmodulin requiring enzyme. Proc Natl Acad Sci USA 87: 682-685. Bright J, Desikan R, Hancock JT, Weir IS y Neill SJ (2006) ABA-induced NO generation and stomatal closure in Arabidopsis are dependent on H2O2 synthesis. Plant J 45:113-122. Bruckdorfer R (2005) The basics about nitric oxide. Mol Aspects Med 26: 3-31. Butaye KMJ, Goderis IJWM, Wouters PFJ, Pues JMT, Delauré S L, Broekaert WF, Depicker A, Cammue BPA y de Bolle MFC (2004) Stable high level transgene expression in Arabidopsis thaliana using gene silencing mutants and matrix attachment regions. Plant J 39:440-449. Butt YKC, Lum JHK y Lo SCL (2003) Proteomic identification of plant proteins probed by mammalian nitric oxide synthase antibodies. Planta 216: 762-771. Cabello JV, Dezar CA, Manavella PA y Chan RL (2007) The intron of the Arabidopsis thaliana COX5c gene is able to improve the drought tolerance conferred by the sunflower Hahb-4 transcription factor. Planta 226:1143-1154. 104 Cantrel C, Vazquez T, Puyaubert J, Rezé N, Lesch M, Kaiser WM, DutilleulC, Guillas I, Zachowski A y Baudouin E (2011) Nitric oxide participates in cold responsive ______________________________________________Referencias Bibliográficas phosphosphingolipid formation and gene expression in Arabidopsis thaliana. New Phytol 189: 415-427. Caro A y Puntarulo S (1998) Nitric oxide decreases superoxide anion generation by microsomes from soybean embryonic axes. Physiol Plant 104: 357-364. Caro A y Puntarulo S (1999) Nitric oxide generation by soybean embryonic axes. Possible effect on mitochondrial function. Free Radic Res. 31 (suppl.) S205-S212. Chen K, Feng H, Zhang M, Wang X (2003) Nitric oxide alleviates oxidative damage in the green alga Chlorella pyrenoidosa caused by UV-B radiation. Folia microbiologica 48, 3:389-393. Chen P, Tsai A, Berka V y Wu KW (1996) Endothelial Nitric-xide Synthase: Evidence for bidomain structure and successful reconstitution of catalytic activity from two separate domains generated by a baculovirus expression system. J Biol Chem 271, 24:14631-5. Chung CC, Hwang SPL y Chang J (2008) Nitric oxide as a signaling factor to upregulate the death-specific protein in a marine diatom, Skeletonema costatum, during blockage of electron flow in photosynthesis. Appl. Environ Microbiol 74: 65216527. Clarke A, Desikan R, Hurst RD, Hancock JT y Neill ST (2000) NO way back: nitric oxide and programmed cell death in Arabidopsis thaliana suspension cultures. Plant J 4: 667-677. Clerissi C y Desdevises Y (2012) Prasinoviruses of the marine green alga Ostreococcus tauri are mainly species-specific. Journall of Virology 86, 8: 4611-4619. Clough SJ y Bent AF (1998) Floral dip: a simplified method for Agrobacterium mediated transformation of Arabidopsis thaliana. Plant J 16: 735-743. 105 Cohen MF y Yamasaki H (2003) Involvement of nitric oxide synthase in sucroseenhanced hydrogen peroxide tolerance of Rhodococcus sp strain APG1, a plantcolonizing bacterium. Nitric Oxide 9: 1-9. ______________________________________________Referencias Bibliográficas Cohen MF, Yamasaki H y Mazzola M (2005) Brassica napus seed meal soil amendment modifies microbial community structure, nitric oxide production and incidence of Rhizoctonia root rot. Soil Biol Biochem 37: 1215-1227. Cooney RV, Harwood PJ, Custer LJ y Franke AA (1994) Light-mediated conversion of nitrogen dioxide to nitric oxide by carotenoids. Environ Health Perspect 102: 460-462. Corker H y Poole RK (2003) Nitric oxide formation by Escherichia coli. Dependence on nitrite reductase, the NO-sensing regulator Fnr, and flavohemoglobin Hmp. J Biol. Chem 278: 31584-31592. Correa-Aragunde N, Graziano M y Lamattina L (2004) Nitric oxide plays a central role in determining lateral root development in tomato. Planta 218: 900-905. Corpas F.J, Barroso JB y del Rio LA (2001) Peroxisomes as a source of reactive oxygen species and nitric oxide signal molecules in plant cells. Trends Plant Sci 6 145150. Corpas FJ, Palma JM, del Río LA y Barroso JB (2009) Evidence supporting the existence of L-arginine-dependent nitric oxide synthase activity in plants. New Phytol 184: 9-14. Corpas FJ, Leterrier M, Valderrama R, Airaki M ,Chaki M, Palma JM y Barroso JB (2011) Nitric oxide imbalance provokes a nitrosative response in plant sunder abiotic stress. Plant Science 181, 5: 604-611. Crane BR, Sudhamsu J y Patel BA (2010) Bacterial nitric oxide synthases. Annu Rev Biochem 79: 445-470. Cueto M, Hernández-Perera O, Martín R, BenturaML, Rodrigo J, Lamas S y Golvano MP (1996) Presence of nitric oxide synthase activity in roots and nodules of Lupinus albus. FEBS Lett 398: 159-164. 106 Curi GC, Chan RL y González DH (2005). The leader intron of Arabidopsis thaliana genes encoding cytochrome c oxidase subunit 5c promotes high-level expression by increasing transcript abundance and translation efficiency. J Exp Bot 56: 2563-2571. ______________________________________________Referencias Bibliográficas Daff S (2010) NO synthase: structures and mechanisms. Nitric Oxide 23: 1-11. Davies SA (2000) Nitric oxide signalling in insects. Insect Biochem Mol Biol 30: 11231138. Delledonne M, Xia Y, Dixon R A. y Lamb C (1998) Nitric oxide functions as a signal in plant disease resistance. Nature 394: 585-588. Derelle E, Ferraz C, Rombauts S, Rouzé P, Wordenf AZ, Robbensd S, Partenskyg F, Degroeved S, Echeyniéc S, Cookei R, Saeysd Y, Wuytsd J, Jabbarij K, Bowler C, Panaudi O, Piégui B, Ball SG, Ral JP, Bouget FY, Piganeau G, De Baetsh B., Picard, A, Delsenyi M, Demaillec J, van de Peer Y y Moreau H (2006) Genome analysis of the smallest free-living eukaryote Ostreococcus tauri unveils many unique features. Proc Natl Acad Sci USA 103:11647-11652. DeSmet I, Signora L, Beeckman T, Inzé D, Foyer CH y Zhang HM (2003) An abscisic acid-sensitive checkpoint in lateral root development ofArabidopsis. Plant J 33: 543555. Dezar CA, Fedrigo GV y Chan RL (2005) The promoter of the sunflower HD-Zip protein gene Hahb4 directs tissue-specific expression and is inducible by water stress, high salt concentrations and ABA. Plant Sci 169:447-456. Díez B, Pedrós-Alio C y Massana R (2001) Study of genetic diversity of eukaryotic picoplankton in different oceanic regions by small-subunit rRNA gene cloning and sequencing. Appl Environ Microbiol 67: 2932-2941. Guo D, Liang J y Li L (2009) Abscisic acid (ABA) inhibition of lateral root formation involves endogenous ABA biosynthesis in Arachis hypogaea. Plant growt regulation 58, 2:173-179. Dorlhac de Borne FD, Vincentz M, Chupeau Y y Vaucheret H (1994) Cosuppression 107 of nitrate reductase host genes and transgenes in transgenic tobacco plants. Molecular & General Genetics 243:613-621. ______________________________________________Referencias Bibliográficas Duan X, Su X, You Y, Qu H, Li Y. y Jiang Y (2007) Effect of nitric oxide on pericarp browning of harvested longan fruit in relation to phenolic metabolism. Food Chem 104: 571-576. Durner J, Wendehenne D y Klessig DF (1998) Defense gene induction in tobacco by nitric oxide, cyclic GMP, and cyclic ADP-ribose. Proc Natl Acad Sci USA 95:1032810333. Durner J y Klessig DF (1999) Nitric oxide as a signal in plants. Curr Opin Plant Biol 2: 369-374. El-Gayar S, Thüring-Nahler H, Pfeilschifter J, Röllinghoff M y Bogdan C (2003). Translational control of inducible nitric oxide synthase by IL-13 and arginine availability in inflammatory macrophages. J Immunol 171: 4561-4568. Elmayan T y Vaucheret H (1996) Expression of single copies of a strongly expressed 35S transgene can be silenced post-transcriptionally. Plant J 9: 787-797. Feldman PL, Griffith OW y Stuehr DJ (1993) The surprising life of nitric oxide. Chem Eng News 71: 26-38. Finn RD, Tate J, Mistry J, Coggill PC, Sammut SJ, Hotz HR, CericG, Forslund K, Eddy, SR, Sonnhammer EL, y Bateman A (2008) The Pfam protein families database. Nucleic Acids Res 36: D281-D288. Flores T, Todd CD, Tovar-Mendez A, Dhanoa PK, Correa- Aragunde N, Hoyos ME, Brownfield DM, Mullen RT, Lamattina L y Polacco JC (2008) Arginase-negative mutants of Arabidopsis exhibit increased nitric oxide signaling in root development. Plant Physiol 147: 1936-1946. Foresi N, Correa-Aragunde N, Parisi G, Caló G, Salerno G y Lamattina L (2010) Characterization of a nitric oxide synthase from the plant kingdom: NO generation from 108 the green alga Ostreococcus tauri is light irradiance and growth phase dependent. Plant Cell 22: 3816-3830. ______________________________________________Referencias Bibliográficas Furchgott RF y Zawadzki JV (1980) The obligatory role of endothelial cells in the relaxation of arterial smooth muscle by acetylcholine. Nature 288: 373-376. Gallois P y Marinho P (1995) Leaf disk transformation using Agrobacterium tumefaciens–expression of heterologous genes in tobacco. Plant Gene Transfer and Expression Protocols (Jones, H. ed.) 39-48. García-Mata C y Lamattina L. (2001) Nitric oxide induces stomatal closure and enhances the adaptive plant responses against drought stress. Plant Physiol 126: 1196-1204. García-Mata C y Lamattina L (2002) Nitric Oxide and ABA cross-talk in guard cells. Plant Physiol 128: 790-792. García-Mata C, Gay R, Sokolowski S, Hills A, Lamattina L y Blatt MR (2003) Nitric oxide regulates K+ channels in guard cells through a subset of ABA-evoked signaling pathways. Proc Natl Acad Sci USA 100: 11116-11121. Gas E, Flores-Pérez U, Sauret-Güeto S y Rodríguez-Concepción M (2009) Hunting for plant nitric oxide synthase provides new evidence of a central role for plastids in nitric oxide metabolism. Plant Cell 21: 18-23. Gerber NC, Nishida CR y Ortiz de Montellano PR (1997) Characterization of human liver inducible nitric oxide synthase expressed in Escherichia coli. Arch Biochem Biophys 343: 249–253. Ghafourifar P, Asbury M.L, Joshi SS y Kincaid ED (2005) Determination of mitochondrial nitric oxide synthase activity. Methods Enzymol 396: 424-444. Godoy L, González-Duarte R, Albalat R (2006) S-Nitrosogluthathione reductase activity of amphioxus ADH3: insights into the nitric oxide metabolism. Int J Biol Sci 2:117-124. 109 Golderer G, Werner ER, Leitner S, Gröbner P, Werner-Felmayer G (2001) Nitric oxide synthase is induced in sporulation of Physarum polycephalum. Genes 15,10: 12991309. ______________________________________________Referencias Bibliográficas Goldestein S, Gabor Merényi (2008) The Chemistry of Peroxynitrite: Implications for Biological Activity. Methods in Enzymology 436: 49-61. Gorren ACF y Mayer B (2007) Nitric-oxide synthase: A cytochrome P450 family foster child. Biochim Biophys Acta 1770: 432–445. Gotte G, Amelio E, Russo S, Marlinghaus E, Musci G y Suzuki H (2002) Short-time non-enzymatic nitric oxide synthesis from L-arginine and hydrogen peroxide induced by shock waves treatment. FEBS Lett 520: 153-155. Gould KS, Klinguer A, Pugin A y Wendehenne D (2003) Nitric oxide production in tobacco leaf cells: a generalized stress response? Plant Cell Environ 26: 1851-1862. Gouvea CMCP, Souza JF, Magalhaes CAN, Martins IS (1997) NO-releasing substances that induce growth elongation in maize root segments. Plant Growth Regulation 21:183-187. Gow AJ e Ischiropoulos H (2001) Nitric oxide chemistry and cellular signaling. J Cell Physiol 187: 277-282. Graziano M, Beligni V y Lamattina L (2002) Nitric oxide improves the iron deficiency phenotype in maize Plant Physiol 130:1852-1859. Greenberg JT, Monach P, Chou JH, Josephy PD y Demple B (1990) Positive control of a global antioxidant defense regulon activated by superoxide-generating agents in Escherichia coli. Proc Natl Acad Sci USA 87: 6181-6185. Griffith OW y Stuehr DJ (1995) Nitric oxides synthases properties and catalytic mechanism. Annu Rev Physiol 57: 707-736. Guindon S, and Gascuel O (2003) A simple, fast, and accurate algorithm to estimate large phylogenies by maximum likelihood. Syst Biol 52: 696-704. 110 Guo FQ, Okamoto My Crawford NG (2003) Identification of a Plant Nitric Oxide Synthase Gene Involved in Hormonal Signaling. Science 302, 5642: 100-103. Guo FQ y Crawford NM (2005) Arabidopsis nitric oxide synthase1 is targeted to ______________________________________________Referencias Bibliográficas mitochondria and protects against oxidative damage and dark-induced senescence. Plant Cell 17: 3436-3450. Gupta KJ, Stoimenova M y Kaiser WM (2005) In higher plants, only root mitochondria, but not leaf mitochondria reduce nitrite to NO, in vitro and in situ. J Exp Bot 56: 26012609. Gupta KJ, Fernie AR, Kaiser WM y van Dongen JT (2011) On the origins of nitric oxide. Trends Plant Sci 16: 160-168. Gusarov I y Nudler E (2005) NO-mediated cytoprotection: Instant adaptation to oxidative stress in bacteria. Proc Natl Acad Sci USA 102: 13855-13860. Gusarov I, Starodubtseva M, Wang ZQ, McQuade L, Lippard SJ, Stuehr DJ y Nudler E (2008) Bacterial nitric-oxide synthases operate without a dedicated redox partner. J Biol Chem 283: 13140-13147. Gusarov I, Shatalin Y, Starodubtseva M y Nudler E (2009) Endogenous Nitric Oxide Protects Bacteria Against a Wide Spectrum of Antibiotics. Science 325, 5946: 13801384. Hajdukiewicz P, Svab Z y Maliga P (1994) The small, versatile pPZP family of Agrobacterium binary vectors for plant transformation. Plant Mol Biol 25: 989-994. Hallemeesch MM, Lamers WH y Deutz NE (2002) Reduced arginine availability and nitric oxide production. Clin Nutr 21: 273-279. Halliwell B y Gutteridge JMC (2000) Free Radicals In Biology and Medicine, 3rd edn. Oxford University Press, Oxford, UK. Hanahan D (1983) Studies on transformation of Escherichia coli with plasmids. J Mol Biol 166: 557-580. 111 He Y, Tang RH, Hao Y, Stevens RD, Cook CW, Ahn SM, Jing L, Yang Z, Chen L, Guo F., Fiorani F, Jackson RB, Crawford NM y Pei ZM (2004) Nitric oxide represses the Arabidopsis floral transition. Science 305:1968-1971. ______________________________________________Referencias Bibliográficas Heinzel B, John M, Klatt P, Böhme E y Mayer B (1992) Ca2+/calmodulin-dependent formation of hydrogen peroxide by brain nitric oxide synthase. Biochem J 281: 627630. Hemmens B, Goessler W, Schmidt K y Mayer B (2000) Role of bound zinc in dimer stabilization but not enzyme activity of neuronal nitric-oxide synthase. J Biol Chem 275: 35786-35791. Hiscock S, Bright J, Mc Innis SM, Desikan R y Hancock JT (2007) Signaling on the stigma: potential new roles for ROS and NO in plant cell signaling. Plant Signal Behav 2: 23-24. Huang X, Rad U y Durner J (2002) Nitric oxide induces transcriptional activation of the nitric oxide-tolerant alternative oxidase in Arabidopsis suspension cells. Planta 215: 914-923. Huang X, Stettmaier K, Michel C, Hutzler P, Mueller MJ y Durner J (2004) Nitric oxide is induced by wounding and influences jasmonic acid signaling in Arabidopsis thaliana. Planta 218: 938-946. Huang Y, Niu B, Gao Y, Fu L y Li W (2010) CD-HIT Suite: A web server for clustering and comparing biological sequences. Bioinformatics 26: 680-682. Hunter S, Apweiler R, Attwood TK, Bairoch A, Bateman A, Binns D, Bork P, Das U, Daugherty L, Duquenne L (2009) InterPro: the integrative protein signature database. Nucleic Acids Res 37: D211-D215. Hutchings MI, Shearer N, Wastell S, van Spanning RJM y Spiro S (2000) Heterologous NNR-mediated nitric oxide signaling in Escherichia coli. J Bacteriol 182: 6434-6439. 112 Ignarro LJ, Buga GM, Wood KS, Byrns RE y Chaudhuri G (1987a) Endothelium derived relaxing factor produced and released from artery and vein is nitric oxide. Proc Natl Acad Sci USA 8: 9265-9269. Ignarro LJ, Byrns RE, Buga GM y Wood KS (1987b) Endothelium-derived relaxing factor from pulmonary artery and vein possesses pharmacologic and Chemicals properties identical to those of nitric oxide radical. Circ Res 61: 866-879. ______________________________________________Referencias Bibliográficas Imamura M, Yang J, Yamakawa M (2002) cDNA cloning, characterization and gene expression of nitric oxide synthase from the silkworm, Bombyx mori. Insect Mol Biol 11: 257-265. Jaksch M, Paret C, Stucka R, Horn N, Muller-Hocker J, Horvath R, Trepesch N, Stecker G, Freisinger P, Thirion C, Muller J, Lunkwitz R, Rodel G, Shoubridge EA, Lochmuller H (2001) Cytochrome c oxidase deficiency due to mutations in SCO2, encoding a mitochondrial copper-binding protein, is rescued by copper in human myoblasts. Hum Mol Genet 10: 3025-3035: Jaroszewski L, Rychlewski L, Li Z, Li W y Godzik A (2005) FFAS03: a server for profileprofile sequence alignments. Nucleic Acids Res 33 (Web Server issue): W284-W288. Jasid S, Simontacchi M, Bartoli CG y Puntarulo S (2006) Chloroplasts as a nitric oxide cellular source. Effect of reactive nitrogen species on chloroplastic lipids and proteins. Plant Physiol 142: 1246-1255. Jeffrey SR y Snyder SH (1995) Nitric oxide: a neural messenger. Annu Rev Cell Dev Biol 11:417-440. Kaiser W, Gupta K y Planchet E (2007) Higher plant mitochondria as a source for NO. En: Nitric Oxide in Plant Growth, Development and Stress Physiology Springer-Verlag 1-14. Kalendar R, Lee D y Schulman AH (2009) Fast PCR software for PCR primer and probe design and repeat search. Genes Genomes Genomics 3: 1-14. Keeling PJ (2007) Ostreococcus tauri: seeing through the genes to the genome Trends Genet 23:151-154. 113 Keeling PJ y Palmer JD (2008) Horizontal gene transfer in eukaryotic evolution. Nat Rev Genet 9: 605-618. Keller MD, Selvin RC, Claus W y Guillard RRL (1987) Media for the culture of oceanic ultraphytoplankton. J Phycol 23: 633–638. ______________________________________________Referencias Bibliográficas Klatt P, Schmidt K, Lehner D, Glatter O, Bachinger HP y Mayer, B (1995) Structural analysis of porcine brain nitric oxide synthase reveals a role for tetrahydrobiopterin and L-arginine in the formation of an SDS-resistant dimer. EMBO J 14: 3687-3695. Klepper L (1990) Comparison between NO(x) evolution mechanisms of wild-type and Nr (1) mutant soybean leaves. Plant Physiol 93: 26-32. Klessig DF, Ytterberg AJ y van Wijk KJ (2004) The pathogen-inducible nitric oxide synthase (iNOS) in plants is a variant of the P protein of the glycine decarboxylase complex. Cell 119: 445. Knudsen GM, Nishida CR, Mooney SD y Ortiz de Montellano PR (2003) Nitric-oxide synthase (NOS) reductase domain models suggest a new control element in endothelial NOS that attenuates calmodulin-dependent activity. J Biol Chem 278: 31814-31824. Koncz C y Schell J (1986) The promoter of the TL-ADN gene 5 controls the tissuespecific expression of chimeric genes carried by a novel type of Agrobacterium binary vector. Mol Gen Genet 204: 383-396. Koning RI y Koster AJ (2009) Cryo-electron tomography in biology and medicine. Ann Anat 427-445. Kopyra M, Gwozdz E (2003) Nitric oxide stimulates seed germination and counteracts the inhibitory effect of heavy metals and salinity on root growth of Lupinus luteus. Plant Physiology and Biochemistry 41 (11-12): 1011-1017. Koshland Jr DE (1992) The Molecule of the Year. Science 258:1861. 114 Kruger NJ (2002) The Bradford method for protein quantitation. The proteína protocols handbook. Springer Kuo WN, Ku TW, Jones DL y Baptiste J (1995) Nitric oxide synthase immunoreactivity in baker's yeast, lobster, and wheat germ. Biochem Arch 11: 73-78. Lamattina L, García-Mata C, Graciano M y Pagnussat G (2003) NITRIC OXIDE: The versatility of an extensive signal molecule. Annu Rev Plant Biol 54:109-136. ______________________________________________Referencias Bibliográficas Lanier W, Moustafa A, Bhattacharya D y Comeron JM (2008) EST analysis of Ostreococcus lucimarinus, the most compact eukaryotic genome, shows an excess of introns in highly expressed genes. PLoS ONE 3: e2171. Leach J, Keyster M, Du Plessis M y Ludidi N (2010) Nitric oxide synthase activity is required for development of functional nodules in soybean. J Plant Physiol 167: 15841591. Lee J, Ryu H, Ferrante RJ, Morris SMJr y Ratan RR (2003) Translational control of inducible nitric oxide synthase expression by arginine can explain the arginine paradox. Proc Natl Acad Sci USA 100: 4843-4848. Leshem YY (1988) Plant senescence processes and free radicals. Free Rad Biol Med 5: 39-44. Leshem YY, Wills RBH y Ku VVV (1998) Evidence for the function of the free radical gas nitric oxide (NO) as an endogenous maturation and senescence regulating factor in higher plants. Plant Physiol Biochem 36: 825-833. Letunic I y Bork P (2007) Interactive Tree Of Life (iTOL): An online tool for phylogenetic tree display and annotation. Bioinformatics 23:127-128. Levine J S (1984) Water and the photochemistry of the troposphere In A. HendersonSellers (ed.). Satellite sensing of a cloudy atmosphere: observing the third planet. Taylor & Francis, Ltd., London 123-166. Lindermayr C, Sell S, Müller B, Leister D, Durner J (2010) Redox regulation of the NPR1-TGA1 system of Arabidopsis thaliana by nitric oxide. Plant Cell 22: 2894–2907. 115 Liochev SI, Hausladen A, Beyer WF Jr y Fridovich I (1994) NADPH: Ferredoxin oxidoreductase acts as a paraquat diaphorase and is a member of the soxRS regulon. Proc. Natl. Acad. Sci. USA 91: 1328-1331. List BM, Klatt P, Werner ER, Schmidt K y Mayer B (1996) Overexpression of neuronal nitric oxide synthase in insect cells reveals requirement of haem for tetrahydrobiopterin binding. Biochem J 315: 57-63. ______________________________________________Referencias Bibliográficas Lopez-Bucio J, Cruz-Ramirez A, Herrera-Estrella L (2003) The role of nutrient availability in regulating root architecture. Current Opinion in Plant Biology 6:280-287. Lowe PN, Smith D, Stammers DK, Riveros-Moreno V, Moncada S, Charles I y Boyhan A (1996) Identification of the domains of neuronal nitric oxide synthase by limited proteolysis. Biochem J 314: 55-62. Luckhart S, Rosenberg R (1999) Gene structure and polymorphism of an invertebrate nitric oxide synthase gene. Gene 232: 25-34. Luo Z y Chen Z (2007) Improperly terminated, unpolyadenylated mRNA of sense transgenes is targeted by RDR6-mediated RNA silencing in Arabidopsis. Plant Cell 19: 943-958. Mackerness SAH, John CF, Jordan B y Thomas B (2001) Early components in ultraviolet-B responses: distinct roles for different reactive oxygen species and nitric oxide. FEBS Let 489: 237-242. Maiti R, Van Domselaar GH, Zhang H y Wishart DS (2004) SuperPose: A simple server for sophisticated structural superposition. Nucleic Acids Res. 32 (Web Server issue): W590-W594. Maier J, Hecker R, Rockel P y Ninnemann H (2001). Role of nitric oxide synthase in the light-induced development of sporangiophores in Phycomyces blakesleeanus. Plant Physiol 126:1323-1330. Malamy JE, Ryan KS (2001) Environmental regulation of lateral root initiation in Arabidopsis. Plant Physiology 127: 899-909. 116 Manavella PA, Arce AL, Dezar CA, Bitton F, Renou JP, Crespiand M. y Chan RL (2006) Cross-talk between ethylene and drought signaling pathways is mediated by the sunflower Hahb-4 transcription factor. Plant J 48: 125-137. Manavella PA., Dezar CA, Bonaventure G, Baldwin IT y Chan RL (2008a) HAHB4, a sunflower HD-Zip protein, integrates signals from the jasmonic acid and ethylene pathways during wounding and biotic stress responses. Plant J 56: 376-388. ______________________________________________Referencias Bibliográficas Manavella PA, Dezar CA, Ariel FD, Drincovich MF. y Chan RL (2008b) The sunflower HD-Zip transcription factor HAHB4 is up-regulated in darkness, reducing the transcription of photosynthesis-related genes. J Exp Bot 59:3143-3155. Manjunatha G, Lokesh V y Neelwarne B (2010) Nitric oxide in fruit ripening: trenes and opportunities. Biotechnol Adv 28: 489-499. Martasek P, Liu Q, Liu J, Roman LJ, Gross SS, Sessa WC y Masters BS (1996) Characterization of bovine endothelial nitric oxide synthase expressed in E. coli. Biochem Biophys Res Commun 219: 359-365. Martin-Jezequel V, Poulet SA, Harris RP, Moal J y Samain JF (1988) Interspecific and intraspecific composition and variation of free amino acids in marine phytoplankton. Mar Ecol 44: 303-313. Matsuo R, Misawa K, Ito E (2008) Genomic structure of nitric oxide synthase in the terrestrial slug is highly conserved. Gene 415:74-81. Mayali X y Azam F (2004) Algicidal bacteria in the sea and their impact on algal blooms. J Eukaryot Microbiol 51: 139-144. Mayer B, John M, Heinzel B, Werner ER, Wachter H, Schultz G y Bohme E (1991) Brain nitric oxide synthase is a biopterin- and flavin-containing multi-functional oxidoreductase. FEBS Lett 288: 187-191. Mayer B y Hemmens B (1997) Biosynthesis and action of nitric oxide in mammalian cells. Trends Biochem Sci 22: 477-481. 117 Messner S, Leitner S, Bommassar C, Golderer G, Grobner P, Werner ER y WernerFelmayer G (2009) Physarum nitric oxide synthases: Genomic structures and enzymology of recombinant proteins. Biochem J 418: 691-700. Mc Kersie B y Leshem YY (1994) Stress and Stress Coping in Cultivated Plants. Dordrecht: Kluwer Academic Publishers. Mishina TE, Lamb C y Zeier J (2007) Expression of a nitric oxide degrading enzyme induces a senescence programme in Arabidopsis. Plant Cell Environ 30: 39-52. ______________________________________________Referencias Bibliográficas Moncada S, Palmer RMJ y Higgs FA (1991) Nitric oxide: physiology, patophysiology and pharmacology. Pharmacol Rev 43: 109-142. Moreau M, Lindermayr C, Durner J y Klessig DF (2010) NO synthesis and signalling in plants - where do we stand? Physiol Plant 138: 372-383. Morel FM (2008) The co-evolution of phytoplankton and trace element cycles in the oceans. Geobiology 6: 318-324. Moroz LL, Kohn AB (2007) On the comparative biology of nitric oxide (NO) synthetic pathways: parallel evolution of NO mediated signalling. In: Trimmer B, Tota B, editors. Advances in experimental biology. London: Elsevier 1-44. Murashige T y Skoog F (1962) A revised medium for rapid growth and bioassays with tobacco tissue cultures. Physiol Plant 15: 473-497. Murgia I, Tarantino D, Vannini C, Bracale M, Carravieri S y Soave C (2004) Arabidopsis thaliana plants overexpressing thylakoidal ascorbate peroxidase show increased resistance to paraquat-induced photooxidative stress and to nitric oxideinduced cell death. Plant J 38: 940-953. Nagase S, Takemura K, Ueda A, Hirayama A, Aoyagi K, Kondoh M y Koyama A. (1997) A novel nonenzymatic pathway for the generation of nitric oxide by the reaction of hydrogen peroxide and D- or L-arginine. Biochem Biophys Res Commun 233:150153. Neill SJ, Desikan R y Hancock JT (2003) Nitric oxide signalling in plants. New Phytol 159: 11-35. 118 Neill SJ, Desikan R y Clarke A (2002) Hydrogen peroxide and nitric oxide assignaling molecules in plants J Exp Bot 53: 1237-1242. Neill S, Barros R, Bright J, Desikan R, Hancock J, Harrison J, Morris P, Ribeiro D y Wilson I (2008) Nitric oxide, stomatal closure, and abiotic stress. J Exp Bot 59: 165176. ______________________________________________Referencias Bibliográficas Nighorn A, Gibson NJ, Rivers DM, Hildebrand JG, Morton DB (1998) The nitric oxidecGMP pathway may mediate communication between sensory afferents and projection neurons in the antennal lobe of Manduca sexta. J Neurosci 18:7244–7255. Nunoshiba T, de Rojas-Walker T, Wishnok JS, Tannenbaum SR y Demple B (1993) Activation by nitric oxide of an oxidative estress response that defends Escherichia coli against activated macrophages. Proc Natl Acad Sci USA 90: 9993-9997. Pagnussat G, Simontachi M, Puntarulo S y Lamattina L (2002) Nitric oxide is required for root organogenesis. Plant Physiol 129: 954-956. Pagnussat G, Lanteri L, Lombardo MC y Lamattina L (2004) Nitric oxide mediates the indole acetic-acid induction of a mitogen-activated protein kinase cascade in adventitious root development. Plant Physiol 135: 279-286. Palenik B, Ren Q, Tai V y Paulsen IT (2009) Coastal Synechococcus metagenome reveals major roles for horizontal gene transfer and plasmids in population diversity. Environ Microbiol 11: 349–359. París R, Lamattina L y Casalongue CA (2007) Nitric oxide promotes the woundhealing response of potato leaflets. Plant Physiol Biochem 45: 80-86. Penteado JC, Rigobello-Masini M, Liria CW, Miranda MT y Masini JC (2009). Fluorimetric determination of intra- and extracellular free amino acids in the microalgae Tetraselmis gracilis (Prasinophyceae) using monolithic column in reversed phase mode. J Sep Sci 32: 2827-2834. 119 Petracek ME, Lefebvre PA, Silflow CD, Berman J (1990) Chlamydomonas telomere sequences are A1T-rich but contain three consecutive G-C base pairs. Proc Natl Acad Sci USA 87: 8222-8226. Piganeau G y Moreau H (2007) Screening the Sargasso Sea metagenome for data to investigate genome evolution in Ostreococcus (Prasinophyceae, Chlorophyta). Gene 406: 184-190. Prado AM, Porterfield DM. y Feijó JA (2004) Nitric oxide is involved in growth regulation and re-orientation of pollen tubes. Development 131: 2707-2714. ______________________________________________Referencias Bibliográficas Posada D y Crandall KA (1998) MODELTEST. Testing the model of ADN substitution. Bioinformatics 14: 817-818. Qian H, Chen W, Li J, Wang J, Zhou Z Liu W y Fu Z (2009) The effect of exogenous nitric oxide on alleviating herbicide damage in Chlorella vulgaris. Aquatic Toxicology, 92: 250-257. Quiao W y Fan LM (2008) Nitric oxide signaling in plant responses to abiotic stresses. JIntegr Plant Biol 50: 1238-1246. Radi R, Beckman JS, Bash KM y Freeman RA (1991) Peroxynitrite-induced membrane lipid peroxidation: cytotoxic potential of superoxide and nitric oxide. Arch Biochem Biophys 228: 481-487. Regulski M y Tully T (1995) Molecular and biochemical characterization of dNOS: a Drosophila Ca2+/calmodulin-dependent nitric oxide synthase. Proc Natl Acad Sci USA 92:9072–9076. Rhoads AR y Friedberg F (1997) Sequence motifs for calmodulin recognition. FASEB J 11: 331-340. Ribeiro EA, Jr Cunha FQ, Tamashiro WM y Martins IS (1999) Growth phasedependent subcellular localization of nitric oxide synthase in maize cells. FEBS Lett. 445: 283-286. Rockel P, Strube F, Rockel A, Wildt J y Kaiser WM. (2002) Regulation of nitric oxide (NO) production by plant nitrate reductase in vivo and in vitro. J Exp Bot 53: 103-110. 120 Rodríguez-Crespo I, Gerber NC, Ortiz de Montellano PR (1996) Endothelial nitric-oxide synthase. Expression in Escherichia coli, spectroscopic characterization, and role of tetrahydrobiopterin in dimer formation. J Biol Chem 271, 19: 11462-11467. Rodrigo J, Springall DR, Uttenthal O, Bentura ML, Abadia-Molina F, Riveros-Moreno V, Martinez-Murillo R, Polak JM y Moncada S (1994) Location of nitric oxide synthase in the adult rat brain. Phil Trans R Soc Lond B 345: 175-221. ______________________________________________Referencias Bibliográficas Roman LJ, Sheta EA, Martasek P, Gross SS, Liu Q y Masters BS (1995) High-level expression of functional rat neuronal nitric oxide synthase in Escherichia coli. Proc. Natl Acad Sci USA 92: 8428–8432. Rousseau DL, Li D, Couture M y Yeh SR (2005) Ligandprotein interactions in nitric oxide synthase. J. Inorg. Biochem 99: 306–323. Rowland A, Murray AJ y Welburn AR (1985) Oxides of nitrogen and their impact upon vegetation. Rev Environ Health 5: 295-342. Rumer S, Gupta KJ y Kaiser WM (2009) Plant cells oxidize hydroxylamines to NO. J Exp Bot 60: 2065-2072. Salerno JC, Harris DE, Irizarry K, Patel B, Morales AJ, Smith SM, Martasek P ,Roman, LJ, Masters BS, Jones CL, Weissman BA, Lane P, Liu Q y Gross SS (1997) An autoinhibitory control element defines calcium regulated isoforms of nitric oxide synthase. J Biol Chem 272: 29769-29777. Sali A y Blundell TL (1993) Comparative protein modelling by satisfaction of spatial restraints. J Mol Biol 234: 779-815. Sambrook J, Fritsch EF y Maniatis T (2001) Molecular Cloning: a Laboratory Manual, 2nd edn. Cold Spring Harbor Laboratory Press, Cold Spring Harbor, New York. Sang J, Jiang M, Lin F, Xu S, Zhang A y Tan M (2008) Nitric oxide reduces hydrogen peroxide accumulation involved in water stress-induced subcellular anti-oxidant defense in maize plants. J Integr Plant Biol 50: 231-243. 121 Santolini J (2011) The molecular mechanism of mammalian NO-synthases: a story of electrons and protons. J Inorg Biochem 105:127–141. Scanlan DJ, Ostrowski M, Mazard S, Dufresne A, Garczarek L, Hess WR, Post AF, Hagemann M, Paulsen I y Partensky F (2009) Ecological genomics of marine picocyanobacteria. Microbiol Mol Biol Rev 73: 249–299. Sheta EA, McMillan K y Masters BS (1994) Evidence for a bidomain structure of constitutive cerebellar nitric oxide synthase. J Biol Chem 269: 15147–15153. ______________________________________________Referencias Bibliográficas Sen S y Cheema IR (1995) Nitric oxide synthase and calmodulin immunoreactivity in plant embryonic tissue. Biochem Arch 11: 221-227. Shi FM y Li YZ (2008) Verticillium dahliae toxins-induced nitric oxide production in Arabidopsis is major dependent on nitrate reductase. BMB Rep 41: 79- 85. Shi HT, Li RJ, Cai W, Liu W, Wang CL, Lu YT (2012) Increasing nitric oxide content in Arabidopsis thaliana by expressing rat neuronal nitric oxide synthase resulted in enhanced stress tolerance. Plant Cell Physiol 2012 53: 344-57 Simontacchi M, Jasid S y Puntarulo S (2004) Nitric oxide generation during early germination of sorghum seeds. Plant Sci 167: 839-847. Sivananthan M (2006) An examination of the relationship between NO, ABA and auxin in lateral root initiation and root elongation in tomato. Master of Science in Plant Biotechnology. University of Canterbury Six C, Finkel ZV, Irwin AJ, Campbell DA (2007) Light variability illuminates nichepartitioning among marine picocyanobacteria. PLoS One 2: e1341. Six C, Finkel Z, Rodriguez F, Marie D, Partensky F, Campbell DA (2008) Contrasting photoacclimatation costs in ecotypes of the marine picoplankter Ostreococcus. Limnol Oceanogr 53: 255-265. Six C, Sherrard R, Lionard M, Roy S y Campbell DA (2009) Photosystem II and pigment dynamics among ecotypes of the green alga Ostreococcus. Plant Physiol. 151: 379-390. 122 Snyder SH y Bredt A (1992) The biological role of nitric oxide. Sci Am 216: 27-35. Söding J, Biegert A y Lupas AN (2005) The HHpred interactive server for protein homology detection and structure prediction. Nucleic Acids Res 33 (Web Server issue): W244-W248. Sørensen HP y Mortensen KK (2005) Advanced genetic strategies for recombinant protein expression in Escherichia coli. J Biotechnol 115: 113-128. ______________________________________________Referencias Bibliográficas Sorlie M, Gorren AC, Marchal S, Shimizu T, Lange R, Andersson KK, Mayer B (2003) Single-turnover of nitric-oxide synthase in the presence of 4-amino-tetrahydrobiopterin: proposed role for tetrahydrobiopterin as a proton donor. J Biol Chem 278: 4860248610. Stam M, Mol N, Kooter J (1997) The Silence of genes in Transgenic Plants. Annals of Botany 79: 3-12. Scheinker V, Fiore G, Di Cristo C, Di Cosmo A, d’Ischia M, Enikolopov G, Palumbo A. 2005. Nitric oxide synthase in the nervous system and ink gland of the cuttlefish Sepia officinalis: molecular cloning and expression. Biochem Biophys Re Commun 338: 1204-1215. Stöhr C, Strube F, Marx G, Ullrich WR y Rockel P (2001) A plasma membrane bound enzyme of tobacco roots catalyses the formation of nitric oxide from nitrite. Planta 212: 835-841. Stuehr DJ, Cho HJ, Kwon NS, Weise MF y Nathan CF (1991) Purification and characterization of the cytokine-induced macrophage nitric oxide synthase: An FADand FMN-containing flavoprotein. Proc Natl Acad Sci USA 88: 7773–7777. Sudhamsu J, Crane BR (2009) Bacterial nitric oxide synthases: what are they good for? Trends Microbiol 17: 212-218. Tada Y, Spoel SH, Pajerowska-Mukhtar K, Mou Z, Song J, Wang C, Zuo J, Dong X (2008) Plant immunity requires conformational changes of NPR1 via S-nitrosylation and thioredoxins. Science 321: 952-956. 123 Thompson JD, Gibson TJ, Plewniak F, Jeanmougin F y Higgins DG (1997) The CLUSTAL-X windows interface: Flexible strategies for multiple sequence alignment aided by quality analysis tools. Nucleic Acids Res 25: 4876-4882. Tillmann U (2004) Interactions between planktonic microalgae and protozoan grazers. J Eukaryot Microbiol 51: 156-168. Thony B, Auerbach G, Blau N (2000). "Tetrahydrobiopterin biosynthesis, regeneration and functions". Biochem J 341, 1: 1-16. ______________________________________________Referencias Bibliográficas Tian X, Lei Y (2006) Nitric oxide treatment alleviates drought stress in wheat seedlings. Biol. Plant 50: 775-778. Tossi V, Lombardo C, Cassia R, Lamattina L (2012) Nitric oxide and flavonoids are systemically induced by UV-B in maize leaves. Plant Sci 193-194: 103-109. Tun NN, Santa-Catarina C, Begum T, Silveira V, Handro W, Floh EIS y Scherer GFE (2006) Polyamines induce rapid biosynthesis of nitric oxide (NO) in Arabidopsis thaliana seedlings. Plant Cell Physiol 47: 346-354. Tzeng E, Billiar TR, Robbins PD, Loftus M. y Stuehr DJ (1995) Expression of human inducible nitric oxide synthase in a tetrahydrobiopterin (H4B)-deficient cell line: H4B promotes assembly of enzyme subunits into an active dimmer. Proc Natl Acad Sci USA 92: 11771-11775. Valderrama R, Corpas FJ, Carreras A, Fernández-Ocaña A, Chaki M, Luque F, Gómez-Rodríguez MV, Colmenero-Varea P, Del Río LA y Barroso JB (2007) Nitrosative stress in plants. FEBS Lett 581: 453-461. Vardi A, Bidle KD, Kwityn C, Hirsh DJ, Thompson SM, Callow JA, Falkowski P y Bowler C (2008) A diatom gene regulating nitric-oxide signaling and susceptibility to diatom-derived aldehydes. Curr Biol 18: 895-899. Vardi A, Formiggini F, Casotti R, De Martino A, Ribalet F, Miralto A y Bowler C (2006) A stress surveillance system based on calcium and nitric oxide in marine diatoms. PLoS Biol 4: e60. 124 Wang ZQ, Lawson RJ, Buddha MR, Wei CC, Crane BR, Munro AW y Stuehr DJ (2007) Bacterial flavodoxins support nitric oxide production by Bacillus subtilis nitric-oxide synthase. J Biol Chem 282: 2196-2202. Wendehenne D, Pugin A, Klessig DF y Durner J (2001) Nitric oxide: comparative synthesis and signaling in animal and plant cells Trends Plant Sci 6: 177-183. Wiederstein M y Sippl MJ (2007) ProSA-web: Interactive web service for the recognition of errors in three-dimensional structures of proteins. Nucleic Acids Res 35 (Web Server issue): W407-W410. ______________________________________________Referencias Bibliográficas Xing H, Tan L, An L, Zhao Z, Wang S y Zhang C (2004) Evidence for the involvement of nitric oxide and reactive oxygen species in osmotic stress tolerance of wheat seedlings: inverse correlation between leaf abscisic acid accumulation and leaf water loss. Plant Growth Regul 42 61-68. Xiong J, Fu G, Tao L y Zhu C (2010) Roles of nitric oxide in alleviating heavy metal toxicity in plants. Xu J, Xu X y Verstraete W (2000) Adaptation of E. coli cell method for micro-scale nitrate measurement with the Griess reaction in culture media. J Microbiol Methods 41: 23-33. Yamasaki H, Sakihama Y y Takahashi S (1999) An alternative pathway for nitric oxide production in plants: New features of an old enzyme. Trends Plant Sci 4: 128-129. Yuda M, Hirai M, Miura K, Matsumara H, Ando K, Chinzei Y (1996) ADNc cloning, expression and characterization of nitric-oxide synthase from the salivary glands of the blood-sucking insecto Rhodnius prolixus. Eur J Biochem 242: 807-812. Zemojtel T, Frohlich A, Palmieri MC, Kolanczyk M, Mikula I, Wyrwicz LS, Wanker EE, Mundlos S, Vingron M, Martasek P y Durner J (2006) Plant nitric oxide synthase: a never-ending story? Trends Plant Sci 11: 524-525 Zhang M, An L, Feng H, Chen T, Chen K, Liu Y, Tang H, Chang J, Wang X (2003) The cascade mechanisms of nitric oxide as a second messenger of ultraviolet B in inhibiting mesocotyl elongations. Photochemistry and Photobiology 77: 219-225 125 Zhao MG, Tian QY, Zhang WH (2007) Nitric oxide synthase dependent nitric oxide production is associated with salt tolerance in Arabidopsis. Plant Physiology 144: 206217. Zumft WG (1993) The biological role of nitric oxide in bacteria. Arch Microbiol 160: 253264. 126