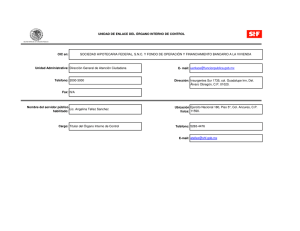
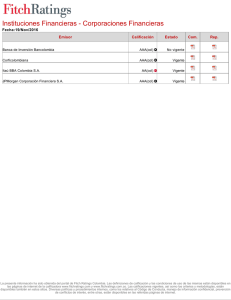
articulo en formato PDF - Coordinación General de Estudios de
Anuncio

2011 Volumen 3, No. 6 Revista Científica de la Universidad Autónoma de Coahuila EXPRESIÓN GENÉTICA EN LA DIGESTIÓN ANAEROBIA: UN PASO ADELANTE EN LA COMPRENSIÓN DE LAS INTERACCIONES TRÓFICAS DE ESTA BIOTECNOLOGÍA Almeida, A1., Nafarrate-Rivera, E1., Alvarado, A1., Cervantes-Ovalle A1., Luevanos, M.P.E1., Oropeza R2., Balagurusamy, N1* 1 Laboratorio de Biorremediación de la Escuela de Ciencias Biológicas de la Universidad Autónoma de Coahuila. Carretera Torreón-Matamoros km 7.5, Torreón, Coahuila. 2 Instituto de Biotecnología/ UNAM, Cuernavaca, Morelos. * [email protected] RESUMEN La digestión anaerobia es una estrategia promisoria para la generación de compuestos de valor agregado a partir de materiales de desecho, principalmente metano. En este proceso se conoce la participación e interacción de al menos 11 grupos microbianos. Sin embargo, el potencial de estos microorganismos involucrados en la degradación anaerobia aún no se ha entendido completamente. Aunque existen varios reportes en el potencial de la biometanización de la biomasa y la relación entre los factores ambientales que afectan a los microorganismos, detalles de su diversidad, sus capacidades metabólicas e interacciones no han sido revelados satisfactoriamente. Los recientes avances en ecología molecular aún buscan esclarecer estos cruciales detalles. Estudios sobre expresión genética de las especies presentes en los reactores anaerobios revelan importantes datos sobre los mecanismos que detrás de las respuestas de los organismos a las diferentes condiciones de estrés. Este trabajo trata sobre los grupos de microorganismos anaerobios involucrados en la generación de metano a partir de biomasa, su metabolismo, dinámica, interacciones y la relación con la expresión genética de los mismos. Un conocimiento profundo de los principios y funciones que controlan en última instancia las respuestas a la inhibición ayudarán a entender el proceso y modificarlo a fin de incrementar los rendimientos. Además, se demuestra que los estudios de expresión genética pueden empelarse como indicadores confiables del estado general de los reactores y procesos anaerobios. INTRODUCCIÓN Las conversiones anaerobias están entre los más viejos procesos biológicos empleados por la humanidad, inicialmente, para obtener alimentos y bebidas; sin embargo, la necesidad de la sociedad actual de desarrollar procesos sustentables y ecológicamente responsables, está incrementando la aplicación de los procesos anaerobios basándose en dos características primordiales de éstos: el tratamiento de los residuos orgánicos y la recuperación de energía. El tratamiento de los desechos provee un beneficio económico y ambiental. Además, el metano, presente en el biogas producido por los sistemas anaerobios, puede ser recuperado y aprovechado para la generación de energía, reduciendo el consumo de combustibles fósiles e impactando de manera positiva sobre la reducción de las emisiones de gases de efecto invernadero (Kansal y col., 2004). Nagamani y Ramasamy (1999) señalan tres puntos básicos en el proceso de digestión anaerobia: a) que las bacterias más importantes involucradas en el proceso de producción de biogas son anaerobias y tienen un crecimiento lento; b) que se observa un alto grado de especialización metabólica en estos microorganismos anaerobios; y c) que la mayoría de la energía libre presente en el sustrato se encuentra en el producto final metano. Debido a que la digestión anaerobia no es una tecnología mecánica sino biológica que involucra una compleja comunidad microbiana íntimamente relacionada que metaboliza productos generados entre los diversos grupos, es necesario un constante monitoreo para dar un buen rendimiento del proceso. Las interacciones que se dan entre los grupos tróficos dependen de los genes que los organismos en la comunidad expresan durante el proceso de digestión. Tecnología para estudiar la expresión diferencial de genes puede ser utilizada para elucidar redes metabólicas en comunidades microbianas. Una profunda comprensión de la regulación metabólica bajo diferentes factores puede ayudar a mejorar el rendimiento de metano en digestores anaerobios. En el siguiente artículo de revisión, se cubre de manera profunda la comunidad microbiana de la digestión anaerobia, compuestos que causan http://www.postgradoeinvestigacion.uadec.mx/divulgacionAQM.html 14 2011 Volumen 3, No. 6 Revista Científica de la Universidad Autónoma de Coahuila inhibición de grupos tróficos y los estudios de expresión genética en los organismos presentes en digestores anaerobios 1. Interacción Microbiana en la Digestión Anaerobia La digestión anaerobia es una serie de procesos únicos, en los cuales diversos grupos de microorganismos interactúan entre sí en una dinámica metabólica compleja, siendo parte importante de los ciclos biogeoquímicos del Carbono, Nitrógeno y Azufre. Además, éste procesos posee relevantes aplicaciones directas para las sociedades modernas como lo son el tratamiento de efluentes y la producción de energía. El proceso de degradación anaerobia ha sido básicamente restringido a tan sólo 3 grupos o etapas principales para su estudio (Pavlostathis y Giraldo-Gómez, 1991; Siegris y col., 1993), 1) hidrólisis y fermentación, 2) acetogénesis y 3) metanogénesis; donde la primera etapa del proceso involucra la hidrólisis de sólidos insolubles, es decir partículas orgánicas (como celulosa o hemicelulosa) o coloides orgánicos (como proteínas), en compuestos solubles simples que pueden ser absorbidos a través de la pared celular, para que posteriormente, dichas moléculas hidrolizadas sean catabolizadas por bacterias fermentativas en alcoholes y ácidos grasos, teniendo como resultado de este proceso, la producción de hidrógeno y dióxido de carbono. Durante la acetogénesis, se produce ácido acético a través de la oxidación de ácidos grasos de cadena corta o alcoholes (Schink, 1997), ó a través de la reducción del CO2, usando hidrógeno como donador de electrones para la reacción (Fischer y col., 1932). En el último paso, la metanogénesis, es llevada a cabo por arqueas las cuales obtienen su energía de la conversión de un número restringido de sustratos a metano (Gujer y Zehnder, 1983; Pavlostathis y Giraldo-Gomez, 1991; Hedderich y Whitman, 2006). A pesar de ser agrupadas en tan sólo 3 etapas, la digestión anaerobia es una serie de procedimientos y reacciones específicas en las que intervienen o interactúan por lo menos 11 grupos microbianos (Figura 1), los cuales serán descritos a detalle en este trabajo, a través de las etapas de la digestión anaerobia. POLÍMEROS COMPLEJOS (Polisacáridos, proteínas, lípidos) 1 ÁCIDOS GRASOS DE CADENA LARGA MONÓMEROS (azúcares, aminoácidos) (láurico, mirístico, palmítico) 2 ÁCIDOS GRASOS VOLÁTILES ALCOHOLES (metanol, etanol) Propionato Butirato e Isobutirato Valeriato e Isovaleriato Caproato 3 HIDRÓGENO Y DIÓXIDO DE CARBONO 4 Acetato NITRATOS Y SULFATOS NO3¯ NH3 CO2 6 SO2¯² Metano 10 H2 5 9 8 N2 7 11 H2S 1. 2. 3. 4. 5. 6. Bacterias Hidrolíticas Bacterias Fermentativas Bacterias Sintróf icas Productoras Obligadas de Hidrógeno Bacterias Homoacetogénicas Bacterias Sintróf icas Oxidadoras de Acetato Arqueas Metanogénicas Acetoclásticas 7. 8. 9. 10. 11. Arqueas Metanogénicas Hidrogenotróf icas Bacterias Sulf ato Reductoras Bacterias Denitrif icantes Bacterias Disimiladoras Reductoras de Nitratos Metanótrof os Anaerobios Fig. 1. Interacciones microbianas en el proceso de degradación anaerobia. http://www.postgradoeinvestigacion.uadec.mx/divulgacionAQM.html 15 2011 Volumen 3, No. 6 Revista Científica de la Universidad Autónoma de Coahuila 1.1. Primera Etapa: Hidrólisis y Fermentación La primera etapa de la digestión anaerobia es algunas veces limitante en el funcionamiento de un digestor anaerobio, ya que a través de la hidrólisis las partículas complejas son solubilizadas para primeramente hacerlas accesibles al interior de la membrana celular de los microorganismos, posteriormente en la etapa de fermentación, la materia orgánica disuelta, proveniente de la hidrólisis es degradada a ácidos orgánicos de cadena corta y alcoholes por una población microbiana heterogénea (Gavala y col., 2003). 1.1.1. Hidrólisis, la solubilización de los compuestos orgánicos La hidrólisis se define tanto como la solubilización de partículas o coloides insolubles, como la descomposición biológica de polímeros orgánicos a monómeros y dímeros, los cuales son capaces de a travesar la membrana celular (Gavala y col., 2003). La hidrólisis de biopolímeros es llevada a cabo regularmente por enzimas extracelulares (hidrolasas) las cuales se encargan de romper enlaces específicos con ayuda de moléculas de agua para obtener moléculas de polímero más pequeñas, dicho proceso ha sido descrito por McCarty y Mosey (como una cinética de primer orden [Ec. (1)]. rs = Kh S (1) donde rs es la velocidad de consumo del sustrato, S es la concentración de sustrato y Kh es la constante de hidrólisis. La solubilización de sustratos insolubles es un proceso complejo que depende de diferentes parámetros tales como el tamaño de partícula, el pH (Veeken, et al., 2000), la temperatura (Veeken y Hamelers, 1999), producción de enzimas, la difusión y adsorción de las enzimas a las partículas. Los primeros datos obtenidos sobre la hidrólisis de polímeros orgánicos provienen de estudios de ecología microbiana en el rumen. Sin embargo, un amplio número de constantes de velocidad de hidrólisis concernientes a la degradación de carbohidratos, proteínas y lípidos han sido reportadas asumiendo cinéticas de primer orden, tal es el caso del trabajo elaborado por Christ y col. (2000) (Tabla 1). Tabla 1. Constantes cinéticas (d-1) para la hidrólisis anaeróbica de carbohidratos, proteínas y lípidos. Sustrato Constante hidrolítica (d-1) Carbohidratos Proteínas Lípidos 0.025-0.200 0.015-0.075 0.005-0.010 Gavala y col., 2003 La importancia de este grupo de bacterias radica en la velocidad con la que estas solubilizan la materia particulada y se ha comprobado que dicha velocidad de hidrólisis del sustrato depende tanto del tipo, origen y de la previa adaptación del inóculo anaerobio (Doyle y col., 1983; Gavala y Lyberatos, 2001), y a su vez, la naturaleza del sustrato determina el tipo y grado de bacterias hidrolíticas presentes. Preeti Rao y col., (1993), observaron que mientras digestores alimentados con estiércol de vaca soportan mayor cantidad de microorganismos amilolíticos, los digestores alimentados con desechos de aves de corral mostraron mayores poblaciones proteolíticas. Ramasamy y col. (1991), reportaron la diferencia existente en las especies de bacterias celulolíticas presentes en el rumen y en un biodigestor; mientras que en el rumen, Ruminococcus sp. representan aproximadamente el 60% del total de este tipo de bacterias, en el biodigestor las especies de los géneros Bacteroides y Clostridium fueron las predominantes. Se ha comprobado que la mayoría de este tipo de bacterias está adherida al sustrato antes de la hidrólisis extensiva. Los principales constituyentes de la biomasa típica alimentada a un digestor anaerobio (estiércol de ganado lechero o engorda, desechos de frutas y verduras, desechos de jardinería), son la celulosa, hemicelulosa y lignina, polímeros distribuidos en una matriz lignocelulósica, en la cual, la lignina juega un papel importante en su http://www.postgradoeinvestigacion.uadec.mx/divulgacionAQM.html 16 2011 Volumen 3, No. 6 Revista Científica de la Universidad Autónoma de Coahuila degradación, ya que por sí sólo este compuesto recalcitrante no tiende a la degradación anaerobia, además que su contenido afecta la degradación de los polímeros de celulosa y hemicelulosa. Se conoce como celulasas, al conjunto de enzimas encargadas de la hidrólisis de la celulosa, los tres principales tipos de celulasas son conocidos como: endocelulasas (encargadas de romper los enlaces 1,4-β glucosídicos internos del polímero de celulosa en polímeros de menor tamaño), exocelulasas (encargadas de romper los enlaces 1,4-β glucosídicos de los polímeros reducidos por las endocelulasas, obteniendo tetrámeros o dímeros de glucosa, como la celobiosa) y la celobiasas o β-glucosidasas (las cuales hidrolizan la celobiosa en dos moléculas de glucosa). La degradación de proteínas (50% de la masa seca celular es proteína), no ha sido estudiada a fondo, sin embargo, se sabe que estas configuraciones tridimensionales de aminoácidos, que pueden o no estar conjugadas a grupos orgánicos o inorgánicos, son degradadas por un conjunto de enzimas proteolíticas, conocidas también como proteasas, las cuales han sido estudiadas tanto en rumen, como en digestores, hallando que las bacterias proteolíticas ruminales predominantes en el rumen de ganado fueron unos bacilos Gram-negativos pertenecientes al género Bacteroides (Hazelwood y Nugent, 1978) en contraste con las bacterias proteolíticas predominantes en los digestores anaerobios, donde se hallaron Clostridium spp. (Siebert y Torien, 1969). 1.1.2. Fermentación o acidogenesis La fermentación es definida como una etapa crucial en la digestión anaerobia, ya que el material orgánico soluble, proveniente de la hidrólisis es convertido principalmente a acetato, ácidos grasos de cadena corta, alcoholes, hidrógeno y dióxido de carbono, es por eso que esta etapa es conocida también como acidogénesis. Una de las principales características de esta etapa de la digestión, es la producción de hidrógeno, debido a que parte de los electrones generados y que no son transferidos a bases de piridinas, son dispuestos vía reducción de protones, formando éste hidrógeno molecular. La disminución del pH en el sistema, es otra característica de la fermentación, y esta se debe a la acumulación de los ácidos orgánicos. Aunque la especie dominante en la digestión anaerobia sean bacterias, se han reportado pequeñas poblaciones de protozoos, hongos y levaduras (Toerien y Hattingh, 1969). Las bacterias anaerobias obligadas y facultativas son las principales que llevan a cabo la fermentación de los sustratos hidrolizados. Las bacterias fermentativas son bacterias anaerobias que utilizan en un inicio las rutas catabólicas de polisacáridos (celulosa, hemicelulosa, fructosa, pectina, sacarosa y almidón), aminoácidos y el glicerol para producir glucosa, posteriormente esta glucosa es transformada vía glucólisis o vía Entner-Doudoroff en piruvato, para seguir alguna de las rutas de la fermentación alcohólica, láctica y acética. Como resultado de esta fermentación, son obtenidos alcoholes, ácidos grasos de cadena corta, adicionalmente, son generados como parte de esta transformación hidrógeno y dióxido de carbono (Tabla 2). http://www.postgradoeinvestigacion.uadec.mx/divulgacionAQM.html 17 2011 Volumen 3, No. 6 Revista Científica de la Universidad Autónoma de Coahuila Tabla 2. Productos Metabólicos liberados durante la fermentación anaerobia de diferentes polisacáridos por cepas puras de bacterias anaerobias Organismo Bacteroides succinogenes Fuente Rumen Bacteroides fibrisolvens Rumen Bacteroides ruminicola Rumen Ruminococcus flavefaciens Neocallimastix frontalis Rumen spirochetes Rumen treponemes Lachnospira multiparus Acetivibrio cellulolyticus Clostridium thermocellum Sustrato Productos* Celulosa F, A, S Celulosa Hemicelulosa Pectina Hemicelulosa Pectina Celulosa Celulosa Pectina Pectina Pectina Celulosa Celulosa F, L, H2, CO2 F, B, L, H2, CO2 F, A, B, L, M, H2, CO2 F, A, P, S F, A, P, B, L, S F, A, S, H2, CO2 F, A, L, E, CO2 F, A, L, S, M F, A, S, M F, A, L, M, E, H2, CO2 A, E, H2, CO2 A, E, H2, CO2 Rumen Rumen Rumen Rumen Rumen Digestor Digestor Sedimentos de Clostridium papyrosolvens Pectina F, A, L, E estuario Clostridium butyricum Sedimentos de lago Pectina A, B, M, E, H2, CO2 *F = formato, A = acetato, P = propionato, B = butirato, S = sucinato, L = lactato, M = metanol, E = etanol. Chynoweth e Isaacson, 1987 Algunos otros microorganismos fermentativos predominantes en el rumen son Clostridium cellobioparum, Ruminococcus albus (Godbole y col., 1981), y en los digestores de estiércol de vaca, Eubacterium cellulosolvens, Clostridium cellulosolvens, Clostridium cellulovorans y Bacteroides cellulosolvens (Ramasamy, 1997). A pesar de la gran variedad de productos derivados de la fermentación bacteriana, los ácidos grasos volátiles (AGV) son uno de los principales productos de la fermentación de carbohidratos, tanto en digestores como en el rumen. El metabolismo de los carbohidratos puede verse influenciado por la presión parcial de hidrógeno derivado de esta misma fermentación (McInerney y Bryant, 1981), inclusive afectar la posterior oxidación de los ácidos orgánicos producidos en acetato. Sin embargo, la presión parcial de hidrógeno puede ser mantenida por medio de las arqueas metanógenas hidrogenotróficas, las bacterias homoacetogénicas y las bacterias sulfato reductoras. Lin (2003) demostró que otro factor que influye sobre la acidogénesis son los metales pesados. Los efectos tóxicos relativos sobre la producción de acetato y butirato fueron Cu > Zn > Cr > Cd > Pb > Ni y Cu > Zn > Cr > Cd > Ni > Pb, respectivamente. Siendo el cobre el metal más tóxico y el plomo el menos tóxico para los microorganismos productores de ácidos grasos volátiles. Los AGV son importantes intermediarios en para la producción de metano, y sus concentraciones afectan la eficiencia tanto de la fermentación, como de la metanogénesis. Wang y col., (2009), estudiaron los efectos de distintos productos de la fermentación en la producción y rendimiento del metano. Cuando se usaron las concentraciones más altas de etanol, ácido acético y ácido butírico (2400, 2400 y 1800 mg/l, respectivamente) no hubo inhibición significativa sobre la actividad de los metanógenos, sin embargo, cuando la concentración de ácido propiónico se incrementó a 900 mg/l, sí se observó una inhibición ya que la concentración microbiana disminuyó de 6 × 107 a 0.6–1 × 107 ml−1. La máxima producción acumulada de metano se obtuvo a una concentración de etanol, ácido acético, ácido propiónico, y ácido butírico de 1600, 1600, 300 y 1800 mg/l, respectivamente, teniendo una concentración máxima de metanógenos de 7.3 × 108 ml−1. Los ácidos grasos se pueden presentar tanto en su forma ionizada, como en la no-ionizada, siendo las formas noionizadas del acetato, propionato y butirato las más tóxicas para las arqueas metanógenicas (Van Lier y col., 1993). La acumulación de AGV conlleva a la disminución del pH en el sistema, dicha caída de pH afecta la metanogénesis. Van Kessel y Russell (1996) no detectaron la formación de metano en una vaca fistulada con un fluido ruminal cuando el pH era menor a 6. http://www.postgradoeinvestigacion.uadec.mx/divulgacionAQM.html 18 2011 Volumen 3, No. 6 Revista Científica de la Universidad Autónoma de Coahuila 1.2. Segunda Etapa: Acetogénesis Durante la segunda etapa de la digestión anaerobia, la acetogénesis, el ácido acético es producido por cualquiera de los dos diferentes mecanismos acetogénicos, la acetogénesis por hidrogenación y la acetogénesis por deshidrogenación (Tabla 3). La acetogénesis por hidrogenación es el mecanismo por el cual se produce acetato como un único producto final de la reducción del dióxido de carbono más hidrógeno, debido a su analogía con la homofermentación láctica, esta vía también es conocida como homoacetogénesis. En digestión anaerobia la etapa de acetogénesis suele referirse a la acetogénesis por deshidrogenación, y específicamente, a la oxidación anaerobia de ácidos grasos de cadena larga y corta (volátiles), las bacterias encargadas de esta vía son las conocidas como bacterias reductoras obligadas de protones o productoras obligadas de hidrógeno. Ellas son inhibidas inclusive por una pequeña presión parcial de hidrógeno, por lo cual sólo pueden sobrevivir en asociaciones sintróficas con microorganismos que consumen hidrógeno, como los metanógenos acetoclásticos e hidrogenotróficos, las bacterias homoacetogénicas y bacterias sulfato reductoras. Tabla 3. Cambios en la energía libre de Gibbs bajo condiciones estándar en reacciones de hidrogenación y deshidrogenación y sus potenciales redox correspondientes Reacción REACCIONES DE HIDROGENACIÓN Alcoholes primarios CH3CH2OH + H2O CH3COO – + H+ + 2H2 Ácidos grasos CH3 CH2CH2COO– + 2H2O 2CH3COO – 2H+ + 2H2 CH3CH2COO– + 2H2O CH3COO – + CO2 + 3H2 CH3COO– + H+ + 2H2O 2CO2 + 4H2 CH3 CH(CH3)CH2COO– + CO2 + 2H2O 3CH3COO– +2H+ +H2 REACCIONES DE HIDROGENACIÓN 4H2 + 2CO2 CH3COO– + H+ + 2H2O H2 + S0 H2S 4H2 + SO4-2 + H+ HS + 4H2O ∆G°' (kJ por mol) No. de pares de electrones E°' de las reacciones redox de liberación de electrones (mV) +9.6 2 -190, -375 +48.3 +76.0 +94.9 +25.2 2 3 4 1 -125, -250 +30, -176, -470 -200, -300, -430, -520 -94.9 -33.9 -151.0 Schink, 1997 1.2.1. Acetogénesis por hidrogenación: Bacterias sintróficas productoras obligadas de hidrógeno. La sintrofia es un caso especial de cooperación simbiótica entre dos tipos de microorganismos metabólicamente diferentes, los cuales dependen el uno del otro para le degradación de un cierto sustrato, la mayoría de las veces por razones de conservación de energía. El término fue acuñado para describir la cercana relación simbiótica de las bacterias fermentadoras oxidadoras de ácidos grasos con metanógenos oxidadores de hidrógeno (Schink, 1997). Sin embargo, también actualmente es empleado para describir la relación de bacterias fermentadoras oxidadoras de alcoholes, como la Thermoanaerobium brockii, la cual oxida etanol (Ben-Bassat et al., 1981) y también se pueden encontrar bacterias oxidadoras de compuestos aromáticos, aminoácidos; Boone y Bryant (1980) aislaron Syntrophobacter wolinii, la cual sólo beta-oxida propionato a acetato. A pesar de ello, un organismo puede tener una amplia afinidad por distintos sustratos, tal como la Syntrophomonas wolfei, la cual beta-oxida ácidos grasos con un tamaño de cadena de 4 a 7 carbonos. 1.2.2. Acetogénesis por deshidrogenación: Bacterias homoacetogénicas. Las bacterias homoacetogénicas son microorganismos estrictamente anaerobios, los cuales catalizan la formación de acetato a partir de hidrógeno y dióxido de carbono, este tipo de acetogénesis fue reportada por primera vez por Fischer y col., (1932) cuando trabajaba con cultivos enriquecidos de lodos de una planta de tratamiento de agua. Sin embargo, fue hasta 1936 cuando Wieringa, usando hidrógeno y dióxido de carbono como sustratos, purificó al acetógeno anaerobio Clostridium aceticum. http://www.postgradoeinvestigacion.uadec.mx/divulgacionAQM.html 19 2011 Volumen 3, No. 6 Revista Científica de la Universidad Autónoma de Coahuila En 1942, Fontaine y col. encontraron que cuando se enriquecía un cultivo de Clostridium thermoaceticum con glucosa a 60° C, éste microorganismo producía por cada unidad de glucosa, 3 unidades de acetato como producto final, llamándosele a este nuevo tipo de fermentación “homoacetogénica” en analogía con la fermentación láctica homofermentativa, y se sugería que el desdoblamiento de la glucosa formaba 3 unidades de C2, las cuales eran convertidas a acetato, o que por lo menos, la síntesis de una molécula de acetato provenía de dos unidades de C1, teoría comprobada por Wood en 1952, cuando empleó moléculas de CO2 marcadas con 13C y análisis de masa para el cultivo enriquecido, sus descubrimientos indicaron claramente que el carbono de ambos grupos (carboxil y metil) en una molécula de acetato, pueden ser derivados del CO2. La ruta de formación de acetato a partir de dióxido de carbono fue descrita en su mayoría con el uso de C. thermoaceticum por los grupos de trabajo de Wood (1991) y Ljungdahl (1986), es por eso que esta ruta de fijación de CO2 a acetato, donde se utiliza hidrógeno como donador de electrones, es llamada Wood-Ljungdahl. La ruta Wood-Ljungdahl también es llamada “ruta del acetil CoA” o la “ruta del monóxido de carbono deshidrogenasa”, y el total de la síntesis se encuentra esquematizado en la Figura 2, además el balance general de la reacción se describe de acuerdo a la reacción (2). 4 H2 + 2 CO2 acetato – + H+ + 2 H2O (∆G°' = - 95 kJ/mol) (2) Algunas de las especies de bacterias homoacetogénicas que han sido mayormente estudiadas son: - Clostridium thermoaceticum - Acetobacterium woodii - Clostridium formicoaceticum - Peptostreptococcus productos - Eubacterium limosum - Clostridium thermoautotrophicum - Acetogenium kivui Fig. 2. Síntesis del acetato a partir del CO2 en las bacterias homoacetogénicas. FH4= tetrahidrofolato, CH3-Co-E= proteína corrinoide metilada/Fe-S proteína. (Dieker y Wohlfarth, 1994). 1.3. Tercera Etapa: Metanogénesis y sus dos vías principales, acetoclástica e hidrogenotrófica. La metanogénesis es la etapa final de la digestión anaerobia. Esta consiste en la formación de metano a partir de dos rutas principales: la acetoclástica y la hidrogenotrófica. Los dos tipos de metanógenos, acetoclásticos e hidrogenotróficos son esenciales para el último paso de la conversión entre materia orgánica y metano, sin embargo, los roles de estas arqueas durante esta fase del proceso son limitados (Demirel y Scherer, 2008). http://www.postgradoeinvestigacion.uadec.mx/divulgacionAQM.html 20 2011 Volumen 3, No. 6 Revista Científica de la Universidad Autónoma de Coahuila Los metanógenos son un grupo especial de microorganismos, pertenecen al dominio Archaea, y entre sus coenzimas únicas pueden citarse la F420 y la F430. En reactores anaerobios el 70% del metano es obtenido por vía acetoclástica (Ferguson y Mah, 1983). Unos de los géneros y especies más conocidas son Methanosarcina barkeri y Methanosaeta thermophila. Los metanógenos se encuentran en casi todos los ambientes anaerobios concebibles, desde el rumen hasta los intestinos humanos, pasando por el fondo del océano, pantanos, digestores, tiraderos y sedimentos marinos. Los metanógenos crecen a temperaturas en rangos de 5 a 110° C y a salinidades correspondientes a las aguas dulces hasta la de la salmuera, descubrimientos que han redefinido los límites ambientales de la vida. Estos microorganismos son parte de del ciclo global del carbono; cada año aproximadamente 400 toneladas métricas de metano son generadas como productos finales en la degradación materia orgánica en los diversos hábitats anaerobios. El metano es oxidado a dióxido de carbono (CO2) en zonas aerobias, pero una indeterminada cantidad escapa dentro de la atmósfera superior (Ferry, 1993). El estudio de los microorganismos de la producción de metano posee un crecimiento sin precedentes en su relativa corta historia. Y este interés se origina del amplio impacto que los metanógenos poseen en el mundo biológico, incluyendo el medio ambiente, la temprana evolución, la bioquímica y la biología molecular. Los primeros estudios en la producción de metano dieron las pistas para la creación de uno de los más grandes taxones de la vida, los metanógenos. Este grupo es el más y por lo tanto, mejor estudiado del grupo Archaea. Éste dominio es un grupo fenotípicamente diverso de microorganismos que comparten una historia evolutiva común. Los cuatro grupos fenotípicos clásicos incluyen además de a los metanógenos a los halófilos extremos, las arqueas sulfato reductoras y a los termofílicos extremos (DeLong, 1992). Así el estudio de los metanógenos ha permitido encontrar la información para aproximarse a los tempranos orígenes de la evolución de los Archaea. El estudio de los metanógenos también ha contribuido al entendimiento del mundo bioquímico. Hay investigaciones que reportan diversos cofactores y metaloenzimas en las rutas de la metanogénesis. Así también, hay avances en el conocimiento de la bioenergía del proceso. Otros estudios bioquímicos han reportado los aparatos de transcripción y traducción de los metanógenos, aún más detalladamente que en los casos de Eucaria y Bacteria (Ferry, 1993). Todas las formas morfológicas de las bacterias (cocos, filamentos, espirilos) se encuentran en los metanógenos. Las propiedades únicas de los metanógenos, la formación de noveles estructuras, componentes y rutas metabólicas en ciertos organismos permitieron hace años la propuesta del dominio Archaea. Este grupo está claramente separado de los demás según las bases de de sus propiedades bioquímicas. Además de los metanógenos, otros organismos han sido colocados en el dominio. Como algunos halófilos, Thermoacidophiles con Sulfolobus y el género Thermoplasma (Dubach y Bachofen, 1985). Los metanógenos son anaerobios estrictos que crecen principalmente en sustratos tales como hidrógeno y dióxido de carbono (hidrogenotróficos), y acetato (acetoclásticos). Sin embargo, otros sustratos compuestos metilados (metilotróficos), como las metilaminas son también sustratos para estos microorganismos (Figura 3). Algunas de las propiedades de estos microorganismos son: a) b) c) d) e) f) g) h) el requerimiento de ambientes con bajos potenciales de reducción, la presencia de coenzimas únicas, como la coenzima M, factores F420 y F430, 7-metilpterin, metanopterin, metil-tetrahidrometanopterin, metanofuran, su16S rRNA es relativamente distante de los otros procariotes, hay diferencias en el brazo del tRNA, su pared celular contiene o D-amino ácidos o ácido murámico, sus lípidos consisten de gliceroles fitanil éter (cíclicos y acíclicos) y escualenos, ellos sólo tienen un genoma pequeño (aproximadamente un tercio del de E. coli), no se han encontrado quinonas y los citocromos están presentes en muy pocos casos, metanofosfagen(ciclo 2,3-difosfoglicerato) está presente como un posible material fosfórico de reserva. Aún que la formación de metano sin hidrógeno es energéticamente favorable, sólo se genera del 27 al 30% de producto por esta ruta y 70% por acetato. Se conocen actualmente tres clases de microorganismos acetoclásticos, http://www.postgradoeinvestigacion.uadec.mx/divulgacionAQM.html 21 2011 Volumen 3, No. 6 Revista Científica de la Universidad Autónoma de Coahuila pero ellos tienen de 2 a 4 veces una tasa de crecimiento menor respecto a los hidrogenotróficos. Estas clases de utilizadores de acetato son: a) b) c) Methanosarcina barkeri (morfología: paquete cuadrado de cuatro células) Methanosaeta (morfología: baciliar, filamentosa) Methanosarcina mazei (morfología: empaquetados o solos) Durante la etapa de la metanogénesis, puede ocurrir un fenómeno de oxidación anaerobia del metano por algunas arqueas o bacterias sulfato reductoras (BSR). Alperin y Hoehler (2009) emplearon un modelo de difusión-reacción en sedimentos marinos para entender la relación sintrófica de los agregados de arqueas y BSR, la termodinámica de la sintrofia, las tasas de reacción, los efectos del agregado en el tamaño y la morfología de las arqueas y las BSR, encontrando que la relación sintrófica para la oxidación anaerobia del metano era termodinámica y físicamente posible debido a una amplia variedad de compuestos intermediarios (incluyendo el hidrógeno, formato y acetato), y la relación directa entre arqueas y BSR proveía una modesta ventaja energética comparada con la no asociación. Fig. 3. Rutas metabólicas de la metanogénesis: hidrogenotróficas (flechas de líneas dobles), acetoclásticos (flechas sólidas) y metilotróficos (flechas de líneas quebradas grises). La ruta hidrogenotrófica puede operar de formato o H2/CO2, obteniéndose un potencial reducido de formato o de H2, respectivamente. La ruta acetoclástica obtiene electrones de la oxidación de CO producido por la división del acetato. La ruta metalotrófica puede operar en dos vías: (1) los compuestos metilados C-1 son fuente del grupo metil así como de electrones (una molécula oxida a CO2 y provee de electrones por cada tres moléculas que se reducen a metano, flechas verdes punteadas). Y (2), los compuestos metilados C-1 se reducen a metano usando electrones del H2. Los nombres de las proteínas estudiadas aquí se indican en la ruta. Para la síntesis de cofactores, mptH juega un rol en la biosíntesis del tetrahidrofolato de http://www.postgradoeinvestigacion.uadec.mx/divulgacionAQM.html 22 2011 Volumen 3, No. 6 Revista Científica de la Universidad Autónoma de Coahuila M. jannashii, y mptN está involucrado en la biosíntesis de metanoproteína. Ambos, el tetrahidrofolato y la metaproteína son intermediarios C-1 acarreadores en la metanogénesis. La biosíntesis de las coenzimas F420 está en las enzimas cof (cofC, cofD, cofE, cofG, cofH). Las enzimas ask (askA, askD, askE y askF) participan en los pasos de elongación del 2-oxiácido de la biosíntesis de coenzima B9 y de la coenzima M en el paso final de la metanogénesis. Esta última coenzima es el cofactor orgánico más pequeño conocido, pero es un esencial metil acarreador terminal en la metanogénesis y es producido por las enzimas com (comA, comB, comC, comD, comE) (Graham and White, 2002). 2. Inhibición en el proceso de degradación anaerobia Una sustancia puede juzgarse inhibitoria cuando la tasa de crecimiento de alguno o algunos de los grupos microbianos involucrados es disminuido o se detiene totalmente, produciéndose un cambio adverso en la población en general (Chen y col., 2008). La inhibición en los tratamientos de degradación anaerobia es usualmente indicada por un decremento en la tasa de producción de metano y la acumulación de ácidos orgánicos (Kroeker y col., 1979). Los microorganismos involucrados en los sistemas de degradación anaerobia trabajan dentro de estrechos niveles de presión de hidrógeno, temperatura, concentración de sustrato, contenido de sulfato, entre otros. Debe tomarse en cuenta que las bacterias formadoras de ácido y los microorganismos productores de metano difieren ampliamente en términos de fisiología, necesidades nutrimentales, cinética de crecimiento y por lo tanto la sensibilidad que cada uno ofrece a las condiciones ambientales es muy variable. De acuerdo a Demirel y Yenügin (2002) las fallas relacionadas al balance entre estos dos grupos de organismos son las principales causas de la inestabilidad de los digestores anaerobios. En este sentido es de suma importancia tomar en consideración las causas de inhibición más comunes, los procesos que desencadenan y las alteraciones que producen. Entre los compuestos inorgánicos que causan inhibición en la producción de metano se citan con frecuencia amoniaco, sulfatos y H2S, y algunos metales ligeros (Na, K, Mg, Ca y Al) y pesados (Cr, Fe, Co, Zn, Cd y Zn) (Grady y col., 1999; Zayed y Winter, 2000; Livera y col., 2011). En cuanto a compuestos orgánicos, se encuentran reportados compuestos bencénicos, fenoles y derivados, otros alcoholes, ácidos de cadena larga y lignina, entre otros (Hwu y Lettinga, 1997; Gavala y Ahring, 2002; Chen y col., 2008; Bekmezci y col., 2011). Sin embargo, la literatura muestra una amplia variación en los niveles tóxicos para la mayoría de estas sustancias. La razón principal de esto es la complejidad del proceso de digestión anaerobia, donde mecanismos como antagonismo, sinergismo, aclimatación, etc., pueden presentar efectos sobre la inhibición. 2.1. Inhibición por amoniaco y sulfuro De entre los compuestos inorgánicos más ampliamente reportados se encuentran el amoniaco y sulfuro. Los desechos que típicamente contienen altas cargas de proteínas y/o urea y de sulfatos (comúnmente estiércoles), específicamente estimulan la actividad de bacterias proteolíticas y BSR durante la digestión anaerobia de tales desechos (Aspé y col., 2001). Las actividades de estas bacterias decrecen la eficiencia del proceso debido a la liberación de NH3 y H2S, respectivamente, y ambos compuestos son ampliamente conocidos como inhibitorios de la actividad metanogénica (Sossa y col., 2004). Investigaciones previas confirman que NH3 posee un poder inhibitorio mayor al del NH4+ sobre la actividad de los metanógenos (Koster y Koomen, 1988). La toxicidad por amoniaco es controlada por el pH y la temperatura (Speece, 1996). Se ha reportado que concentraciones entre 80 y 100 mg/l de amoniaco inhiben totalmente cultivos metanogénicos no adaptados creciendo a pH 7.5 y condiciones mesofílicas (Koster y col., 1986; Aspé y col., 2001). Sin embargo, sistemas de alta carga de residuos, inmovilizados, adaptados o granulares pueden lidiar con concentraciones de amoniaco mayores. Sung y Liu (2003) reportaron la inhibición por amoniaco sobre consorcios metanogénicos termofílicos acetoclásticos sometidos a diferentes estrategias de adaptación y pH; ellos reportaron valores pare el 100% de inhibición cuando la cantidad de amoniaco fue de 8000 mg/l a pH cercano a la neutralidad. Zhou y Qiu (2006) reportaron que la actividad metanogénica específica de un inóculo granular se inhibe al 50% cuando la cantidad de amoniaco oscila entre 2350 mg/l. Por su parte, la producción de metano en las comunidades de filtros anaerobios se han reportado exitosa cuando las concentraciones de amoniaco son de 6000 mg/l (Parkin y col., 1986) e incluso de 7800 mg/l (De Baere, 1984), y en reactores UASB se reportó actividad favorable de los metanógenos a valores de amoniaco oscilantes entre 5000 y 7500 mg/l. En estudios de la inhibición del amoniaco sobre consorcios metanogénicos inmovilizados se observó una disminución del 50% de la actividad metanogénica http://www.postgradoeinvestigacion.uadec.mx/divulgacionAQM.html 23 2011 Volumen 3, No. 6 Revista Científica de la Universidad Autónoma de Coahuila cuando la concentración de amoniaco era de 365.39 mg/l (Sossa y col., 2004). Éstos resultados son comparativamente mayores a estudios realizados también sobre consorcios metanogénicos, pero donde no se utilizó inmovilización, los cuales reportan valores de inhibición del 50% de la actividad metanogénica a 100 mg/l de amoniaco (Aspé y col., 2001). En la búsqueda por entender los efectos del amoniaco sobre la población microbiana en reactores anaerobios y que afectan su rendimiento, los reportes sobre los mecanismos fisiológicos postulan que el amoniaco afecta la metanogénesis en dos maneras; 1) 2) el amoniaco inhibe directamente la síntesis de enzimas necesarias para la producción de metano (Kayhanian, 1999), y la molécula hidrofóbica del amoniaco se difunde pasivamente dentro de la célula, causando un desequilibrio protónico y/o una deficiencia de potasio (Sprott y Patel, 1986). De entre los diferentes grupos de metanógenos involucrados en el proceso de degradación anaerobia reportes previos muestran contradicción al determinar cuáles, los metanógenos acetoclásticos o los hidrogenotróficos son los más sensibles al efecto del amoniaco. Zhou y Qiu (2006) reportaron cepas de metanógenos consumidores de acetato sensibles al amoniaco a concentraciones más bajas en comparación con cepas hidrogenotróficas, la misma observación fue hecha previamente por Koster y Lettinga (1984) y Angelidaki y Ahring (1993). Por el contrario, Zeeman y col., (1985) y Wiegant y Zeeman (1986) demostraron que son los metanógenos hidrogenotróficos los más sensibles. Otro de los compuestos inorgánicos mayormente reportado como inhibitorio es el H2S, producto de la reducción de sulfato, que puede estar presente naturalmente en el sustrato (estiércoles, desechos industriales, entre otros). En ambientes anaerobios las bacterias sulfato-reductoras (BSR) compiten por los electrones disponibles con otros anaerobios, incluyendo las bacterias fermentativas, las bacterias acetogénicas productoras de hidrógeno obligadas y las arques metanogénicas. El rol de los metanógenos, de las bacterias degradadoras de celulosa y las BSR es crítico en la red de la degradación anaerobia, y los pasos catalizados por estos microorganismos son a menudo limitantes del proceso de conversión en general (Raskin, y col., 1996). Para los organismos degradadores de celulosa y para los metanógenos utilizadores de acetato el H2S puede controlar su inhibición. Como no todos los metanógenos exhiben la misma respuesta a la toxicidad al H2S (Kroiss y Webnegg, 1983) la literatura muestra niveles de toxicidad muy variados en forma de sulfuro. Además, la toxicidad del sulfuro no siempre puede describirse en los términos de este compuesto, ya que en inóculos granulares en presencia de sulfatos la toxicidad es dependiente del pH. En un pH alcalino el efecto del H2S es mayor que a valores de pH ácidos y cercanos a la neutralidad. Pero en caso de tratamientos con inóculos dispersos, el efecto inhibitorio del H2S puede determinarse en relación a la cantidad de éste (Speece, 1996). Maillacheruvu y col., (1993) establecieron una inhibición en el 50% de la actividad específica de los metanógenos en filtros anaerobios con acetato y propionato en presencia de 200 mg/l de sulfuro y entre 60 y 75 mg/l en reactores de agitación continua. Por su parte, Isa y col., (1986), observaron el mismo porcentaje de inhibición en metanógenos acetoclásticos con 1200 mg/l en reactores con una elevada carga orgánica y de sulfato luego de un prolongado periodo de adaptación. Sobre metanógenos acetoclásticos también, Koster y col., (1986) observaron una inhibición al 50% con 250 mg/l de H2S. Por su parte, Zaid y col., (1992) encontraron que los metanógenos acetoclásticos e hidrogenotróficos se inhibían a partir de 1000 mg/l del sulfuro en biofilmes 2.2. Inhibición por cationes y metales pesados La biometanización de desechos industriales conlleva el desarrollo y adaptación del proceso de forma que sea posible lidiar con la variada composición de estos materiales y las impurezas que presentan. Para estos desechos, se han reportado numerosos productos químicos causantes de inhibición, dependiendo del tipo de desecho. Por ejemplo, para los desechos de la industria alimenticia, a pesar de que la materia orgánica disponible suele ser elevada, el tratamiento suele ser obstaculizado por la presencia de cationes y aniones como el Na+ (Feijoo y col., 1995), dependiendo de la naturaleza de los desechos. En el caso de los residuos de pescados y mariscos, el contenido de sodio puede alcanzar el mismo que el agua de mar (12 g/l) (Rinzema y col., 1988). Reportes previos señalan diferentes valores de inhibición para algunos cationes, mayormente se reportan Ca2+, K+ y Na+. Los valores inhibitorios reportados son 4600 mg/l, 4800 mg/l y 7400 mg/l, respectivamente (Kulgeman y Chin, http://www.postgradoeinvestigacion.uadec.mx/divulgacionAQM.html 24 2011 Volumen 3, No. 6 Revista Científica de la Universidad Autónoma de Coahuila 1970). Sin embargo, luego de 60 días de aclimatación, se reportan valores de inhibición de hasta 20,000 mg/l de sodio sobre consorcios metanogénicos alimentados primariamente con acetato. En el caso del potasio, el mayor efecto sucede sobre reducción de la máxima especificidad de utilización de sustrato (kmax) (Speece, 1996).Se ha reportado que el K+ es un compuesto inhibitorio para la degradación anaerobia de aguas residuales de la industria del café, pero que la adición del Ca2+ puede actuar con un efecto antagónico a este catión (Daoming y Forster, 1993). Incluso se reporta que para el caso de estos elementos (además de magnesio y aluminio) en cantidades mínimas favorece el proceso de degradación anaerobia (Soto y col., 1993). Por ejemplo, el sodio es esencial a bajas concentraciones, McCarty (1964) reportó una concentración favorable de sodio para el desarrollo de la degradación anaerobia entre 100 y 200 mg/l, mientras que estudios más recientes establecieron que el contenido de sodio necesario para una inhibición en el 50% de la actividad metanogénica es entre 5600 y 8000 mg/l (Vallero y col., 2003). El sodio se reporta más tóxico para los microorganismos metanógenos hidrogenotróficos que para los utilizadores de acetato (Liu y Boone, 1991; Soto y col., 1993). Por su parte, Jackson-Moss y Duncan (1991) reportaron que el aluminio deja de ser tolerado por los anaerobios luego de 2500 mg/l sin aclimatación. Y Yu y col., (2001) reporta valores inhibitorios para el caso del calcio de 8000 mg/l y superiores. Mientras que para el potasio el 50% de inhibición de la actividad de los metanógenos acetoclásticos se estableció en 28,934 mg/l (McCarty, 1964) Casos especiales son los desechos de la industria del papel y de la industria textil, las cuales generan aguas residuales de compleja composición. En estos casos las sustancias inhibitorias más comunes son los sulfatos, tanino, compuestos halogenados, policrilatos o fosfonatos y metales pesados (Ali y Sreekrishnan, 2001; Lee y Pavlostathis, 2004). Dentro de los metales pesados que se reportan como los de mayor preocupación al hierro, cobre, zinc, cadmio y niquel (Jin y col., 1998). La toxicidad de los metales pesados es atribuida mayormente a la interrupción de la función enzimática y a la alteración de la estructura mediante la unión de los metales en los grupos prostéticos (Ulmanu y col., 2003). Las concentraciones de metales pesados que causan el 50% en la disminución de la producción de metano durante la degradación anaerobia de suero indicó que los niveles de toxicidad decrecen en este orden, Cu> Zn> Ni (Lin, 1993, Nies, 1999). Mientras que en la degradación anaerobia de lodos de depuradora se reportó un decremento en los niveles de toxicidad en este orden, Cr >Ni >Cu >Zn (Wong y Cheung, 1995). 2.3. Inhibición por compuestos orgánicos Una gran variedad de compuestos orgánicos se ha reportado que pueden inhibir los procesos anaerobios. Entre los compuestos orgánicos que se han reportado tóxicos a este proceso se incluyen alquilbencenos, nitrobencenos, bencenos halogenados, alcoholes y fenoles, alcanos, éteres, aminas, amidas, y también los ácidos grasos de cadena larga han reportado efectos adversos en la degradación anaerobia. Ungerfeld y col., (2004) reportan el efecto de dos alquilbencenos en la actividad metanogénica durante el tratamiento de aguas residuales en rangos termofílicos. En este trabajo se determinó la disminución total de la producción de metano en una cepa de Methanobrevibacter ruminantium cuando la concentración del 3bromopropanosulfato ascendió hasta 3.47 mg/l. Entre los hidrocarburos clorados, se reporta un 96% de inhibición en metanógenos acetoclásticos y 94% en la actividad de hidrogenotróficos a 100 mg/l de cloruro de metileno; con 100 mg/l de cloruro de etileno se observó 30% y 88% de inhibición en metanógenos acetoclásticos y utilizadores de hidrógeno mientras que con 100 mg/l se observaron valores de inhibición del 100% y del 72% para los metanógenos utilizadores de acetato y los hidrogenotróficos, respectivamente (Colleran y col., 1992). Generalmente se considera que los metanógenos acetoclásticos son la clase más sensible. Similares observaciones reportó Freedman (1991) en los ensayos de toxicidad de ácido bromoetano-sulfónico sobre consorcios metanogénicos. Esto es aplicable también para el ácido benzóico, pues se reporta un 50% de inhibición en acetoclásticos a 4000 mg/l, mientras que el umbral de inhibición para los hidrogenotróficos fue mucho mayor (Golden y col., 1994). En cuanto al fenol, se reporta el 50% de inhibición de los metanógenos acetoclásticos cuando la concentración es de 1500 mg/l, mientras que para los metanógenos reductores de H2/CO2, este valor de inhibición se presenta a los 2000 mg/l. Para el catecol el 50% de inhibición se obtiene a 1500 mg/l para ambos metanógenos, acetato e hidrógeno utilizadores (Golden y col., 1994). http://www.postgradoeinvestigacion.uadec.mx/divulgacionAQM.html 25 2011 Volumen 3, No. 6 Revista Científica de la Universidad Autónoma de Coahuila En el caso de los ácidos grasos, los de cadena larga se reportan inhibitorios sobre la degradación anaerobia (Hwu y Lettinga, 1997). Lalman y Bagley (2001) reportaron efectos inhibitorios del ácido oléico a concentraciones de 30 mg/l y de esteárico a 100 mg/l en la actividad de metanógenos acetoclásticos. Aunque las comunidades microbiológicas anaerobias se han estudiado ampliamente, el entendimiento que los investigadores poseen es aún limitado. La cuantificación e identificación de todas las poblaciones contribuyentes en tales sistemas, así como una descripción profunda de la relación entre ellas, su estructura, función y condiciones ambientales aún no han sido alcanzadas. De acuerdo a Ward y col., (2008), todas las causas de la inhibición pueden ser evitadas en primera instancia cuando se consideran los parámetros operacionales y de diseño y su relación y/o efecto con la microbiología del proceso, ya que los factores ambientales internos del reactor se ven fuertemente influenciados por las características del mismo. Un control sobre estas variables puede asegurar un proceso favorable. 3. Revisión de la expresión genética en comunidades anaerobias 3.1. Usos de la expresión genética La expresión genética se relaciona con la funcionalidad de organismos o comunidades biológicas. Así pues, la expresión genética puede elucidar fenómenos complejos en ecosistemas. Cohlen-Kupiec y col. (1997) fusionaron el promotor para la enzima fijadora de nitrógeno de M. maripaludis (PnifK) con el gen lacZ para elucidar la regulación de la expresión. En el promotor de nifK existen dos grupos de secuencias palindrómicas que sirven como sitios de unión para represores. Ellos reportaron que una mutación direccionada solo en la primera secuencia palindrómica de PnifK resulto en una clara de-represión en cultivos crecidos en NH4Cl. En otro experimento, fusiones de genes probaron que la prolil t-RNA sintetasa (ProRS) de Methanococcus jannaschii podía sintetizar ambos, prolina t-RNA y cisteína t-RNA (Stathopoulos y col., 2000). Este resultado fue inesperado, y ayudo a resolver la discrepancia sobre la carencia de homólogos de cysS, genes para enzimas sintetizadoras de cisteina t-RNA en M. jannaschii y Methanobacterium thermautotrophicus. Por otra parte, análisis de expresión de genes usando Northern blot reveló que el gen 2pgk de 96 pb es transcrito junto con otros dos marcos de lectura (ORFs) en un solo RNA mensajero de 1600 nucleotidos (Lehmacher y Hensel, 1994). El gen 2pgk codifica la proteína 2-fosfoglicerato quinasa, la cual cataliza el primer paso en la síntesis de 2,3difosfoglicerato cíclico (cDPG). Se asume que cDPG juega un papel importante en la termo-adaptación de metanógenos hipertermofilos (Hensel y König, 1988), esto se asume debido a las altas concentraciones de estos compuestos en células de Methanothermus fervidus y Methanopyrus kandleri en sus temperaturas óptimas de crecimiento. En otra perspectiva, debido a la cercana relación entre expresión genética y funcionalidad del sistema, se han hecho intentos para usar expresión diferencial de genes como una herramienta para indicar desbalances en procesos o realizar diagnósticos de procesos. En medicina, Rybaczyk y col. (2008) reportaron que la expresión de monoamino oxidasa-A (MAO-A) es un buen indicador para distinguir entre células cancerígenas y células normales en humanos, roedores y peces usando información extraída de gene expression ómnibus. Ellos encontraron que la expresión de MAO-A disminuye un 95.4% en pacientes humanos y 94.2% en casos de cáncer animal cuando se comparan con controles no cancerosos de casi todo tipo de cánceres. Sin embargo, en cuanto a digestión anaerobia, Freitag y Prosser (2009) mostraron dificultades en relacionar la actividad funcional con la actividad transcriptiva en humus. Ellos concluyeron que la transcripción genética de un solo gen, en su caso Methyl Coenzima M reductasa (mcrA), solo permitió una profundización limitada en la dinámica del proceso. Encontraron una correlación más significativa al relacionar la tasa del proceso con la proporción transcrito/gen en vez de solo la abundancia del transcrito. De este experimento es posible inferir que más investigación se necesita para relacionar directamente el desempeño de un proceso con la transcripción de genes. Sin embargo, la expresión diferencial de genes seria más apta para evaluar las necesidades de una comunidad anaerobia, evaluar el estrás que los organismos están sufriendo y así poder tomar medidas en disminuir el estrés biológico para llegar a mayores rendimientos de metano. Como ejemplo, Spanheimer y col. (2007) observaron un incremento en copias de qRT-PCR de los genes otaA, otaB y otaC debido a estrés osmótico en Methanosarcina mazei Gö1 crecida con 400mM de NaCl comparado con el control de 38.5 mM. El cluster ota codifica las tres subunidades de la enzima transportadora de glicina betaine impulsada por ATP, Ota. La glicina betaine es un soluto acumulado en metanógenos para combatir estrés osmótico (Rossler y Müller, 2001). Toma de solutos será preferida por metanógenos a síntesis de solutos de novo, como el http://www.postgradoeinvestigacion.uadec.mx/divulgacionAQM.html 26 2011 Volumen 3, No. 6 Revista Científica de la Universidad Autónoma de Coahuila glutamoto y Nε-acetil-β-lisina. El análisis de concentraciones internas en extractos de células mostró que el glutamato y Nε-acetil-β-lisina eran los únicos solutos acumulados en células crecidas en 400 y 800mM de NaCl. Sin embargo, cuando los cultivos eran suplementados con 1mM de Glicina betaine, concentraciones de glutamato y Nεacetil-β disminuían en las células acopladas a un incremento en glicina betaine, este fenómeno iba a la par de una pequeña disminución en la expresión del cluster ota. Así pues, este tipo de análisis sobre expresión genética puede ser utilizado para evaluar problemas en reactores anaerobios tratando de aguas residuales de alta salinidad como aquellas provenientes de industrias procesadoras de comida marina (Soto y col., 1993; Chen y col., 2003). Si perfiles de expresión genética en estos reactores muestran una sobre expresión de proteínas sintetizadoras/transportadoras de proteínas entonces inhibición puede ser disminuida adicionando pequeñas cantidades de solutos. Además, Hendrickson y col. (2007) y Xia y col. (2009) estudiaron la expresión gentica de Methanococcus maripalidus bajo diferentes limitaciones de nutrientes. Bajo limitación de hidrógeno Hendrickson y col. (2007), reportaron un incremento de 4 a 22 veces en enzimas de la metanogénesis; principalmente aquellas involucradas en la reducción y oxidación de F420, F420- reductora hidrogenasa (fru), metilenetetrahidrometanopterina deshidrogenasa (mtd) y metilenetetrahidrometanopterina reductasa (mer). En contraste, Xia y col. (2009) reportaron solo un incremento de 1.9 veces en la expresión de mtd usando qRT-PCR, pero decremento e incremento en abundancia de otras 59 y 82 proteinas, respectivamente. Un hecho importante fue la marcada disminución en la deshidrogenasa de metilenetetrahidrometanopterina dependiente de H2, Hmd. En los metanógenos hidrogenotróficos metilenetetrahidrometanopterina puede ser reducida usando metilenetetrahidrometanopterina deshidrogenasa dependiente de F20 (Mtd) o Hmd. La primera ruta requiere la presencia de la hidrogenasa reductora de F420 (Fru o Frc) mientras que la última ruta reduce metilenetetrahidrometanopterina directamente utilizando H2. A pesar de que ambas rutas están presentes simultáneamente, Hmd es preferida cuando hay abundancia de H2 en el medio, pero debido a su baja afinidad por el sustrato los metanógenos deben cambiar a la ruta de Fru y Mtd cuando hidrogeno molecular es escaso. De acuerdo con esto, Kato y col. (2008) usando microarreglos en cultimos de M. thermoautotrophicus bajo limitación de H2, encontraron un aumento en la expresión de frh, mtd, mer y fwd genes que involucran la oxidación y reducción de F420. Bonacker y col. (1992) también observaron un preferencia en la expresión enzimas entre Metil coenzima M reductasa (McrI también conocida como MCRA) y su isoenzima McrII bajo limitación de hidrogeno. MCRII no se expresó con tasas de gas pequeñas (150ml 80%H2/20%CO2) a 70ºC, mientras que MCRI no se expresó bajo altas tasas de gas (650ml 80%H2/20%CO2) a 55ºC. Algunos metanógenos son capaces de utilizar alcoholes secundarios como donadores de electrones usando la secAlcohol deshidrogenasa (sec-ADH). Widdel y Wolfe (1989) mostraron que sec-ADH se expresa bajo limitaciones de H2 en M. thermophilum mientras que en otras tres especies, esto es M. organophilum, M. hungatei y M. bryantii, requerían de la presencia de acetona para expresar sec-ADH. Es de interés medir el efecto sobre el perfil genético en digestores anaerobios con limitación de H2 y suplementar con agua residual de Molino de aceite de palma, que es rica en alcoholes secundarios. 3.2. Interacciones de genes en comunidades anaeróbicas Ecosistemas anaerobios como el rumen pueden ser comprendidos aproximadamente de 1000 unidades taxonimicas operacionales (Hess y col., 2011). Esto vuelve a los ambientes anaerobios altamente dinámicos con fluctuaciones en niveles de nutrientes, pH y contenido de agua, lo cual provoca un dinamismo en la dominancia entre grupos tróficos. Así pues la regulación de redes metabólicas es altamente compleja. La expresión genética puede usarse para relacionar actividades esenciales con factores ambientales y últimamente con desempeño de proceso. Existen diferentes métodos para estudiar la diversidad funcional como Microarreglos, qRT-PCR, FISH y MAR-FISH. Lange y col. (2000) desarrollaron un protocolo para transcripción inversa PCR in situ para monitorear la expresión de Methanosarcina mazei S-6. Concluyeron que técnicas de PCR in situ acopladas con identificación de organismos con FISH es una manera de determinar los organismos que expresan genes específicos bajo ciertas condiciones. 3.2.1. Expresión dependiente de sustratos Como se mencionó en la sección 2, el primer paso en la digestión anerobia es la hidrolisis. Doerner y col. (1992) mostraron que al alimentar con celulosa, transcripción diferencial de cel B y celD era regulada, pero no la transcripción de celA y celC (estos últimos dos genes parecen ser constitutivos) en Ruminococcus flavefaciens FD1. Vercoe y col. (2003) estudiaron después el mecanismo de fosforilacion-defosforilacion de R. flavefaciens FD-1. http://www.postgradoeinvestigacion.uadec.mx/divulgacionAQM.html 27 2011 Volumen 3, No. 6 Revista Científica de la Universidad Autónoma de Coahuila Encontraron que en células cultivadas con celulosa fosforilaban la treonina en proteinas, mientras que células cultivadass con celubiosa fosforilaban serina. Esta diferencia en aminoácidos fosforilados puodría regular la inducción y coordinación de expresión de genes celulolíticos en R. flavifaciens FD-1. Después de la hidrólisis de polímeros compuestos muchos productos de fermentación son generados. El propionato es un importante intermediario en la digestión anaerobia. Ariesyadi y col. (2007) estudiaron la diversidad funcional de lodos granulares bajo diferentes concentraciones de 14C-propionato viz. 0.5, 2.5, 5 y 15 mM. Análisis con MARFISH revelaron que 52-62% de células oxidadoras de propionato eran Smithella sp. SR en todas las concentraciones de propionato. Smithella sp. LR componen 42% de las células MAR-positivas en la concentración de 0.5mM, pero este porcentaje disminuyo drásticamente en las demás concentraciones. Mientras que células de Syntrophobacter componían solo el 6% de las células MAR-positivas en la concentración 0.5mM de propionato e incrementaron entre 16-31% en el resto de los tratamientos. En este mismo estudio, Methanosaeta fue la única arquea que mostró positividad MAR y componían solo del 7-10% de las células MAR-positivas. Oxidación de propionato por bacterias sintróficas lleva a la producción de H2 y formiato estos compuestos en concentraciones elevadas pueden inhibir la oxidación de propionato, lo que llevaría a desbalance del proceso. Así pues, bacterias sintróficas necesitan la presencia de organismos hidrogenotróficos cuando se cultivan con propionato. Sin embargo, aún en la presencia de organismos hidrogenotraficos las concentraciones de formiato pueden elevarse, asi que las bacterias sintróficas expresan deshidrogenasas de formato (FDH), para oxidar formiato. De Bok y col. (2003) purificaron dos enzimas FDH de Sytrophobacter fumaroxidans que también pueden reducir CO2 en mono y co-cultivos creciendo con fumarato y propionato, respectivamente. Los mismos autores también reportaron mayores niveles de ambas enzimas cuando las bacterias sintróficas eran crecidas en la presencia de M. hungateii. Esto puede ser debido a que el formiato es un portador de electrones más obvio que el H2 difusivo por su solubilidad en el agua. Así pues ambas enzimas son necesarias para que los organismos crezcan con propionato (de Bok y col., 2002), una de las FDHs reduce equivalentes de formiato y la otra para fijar CO2 por la vía reversa de rompimiento de acetil-coenzima. En efecto, Kato y col. (2009) que probaron con microarreglos registraron una mayor expresión de FdhII en cultivos de Pelotomaculum thermopropionicum con M. thermautotrophicus creciendo en alcohol y propionato al contrario de monocultivo P. thermopropionicum crecido en fumarato. Adicionalmente, observaron una sobre-regulación en la liasa piruvato:formiato (Pfl) que cataliza la liberación de formiato por la conversión de piruvato a Acetil-CoA en co-cultivos sintróficos. Además, usando cluster jerárquico se mostro una sobre-regulación de 2.4% de los genes y una baja-regulación 1.7% de los clusters de genes en los co-cultivos de todos los que estaban degradando diferentes sustratos, mientras que los porcentajes de expresión genética variaron entre 10.2% y 33.4% en todos los tratamientos. Así pues, solamente la asociación sintrófica falla en explicar la gran diferencia en perfiles de expresión, sugiriendo que estos cambios en regulación son altamente dependientes del sustrato en vez de influencia sintrófica. Una ruta principal en la degradación de propionato es la Metil-Malonil-CoA (MMC). Kato y col. (2009) mostraron la expresión de enzimas pertenecientes a esta ruta (el cluster mmc) en cultivos alimentados con propionato e inesperadamente en aquellos alimentados con lactato. Usualmente los organismos sintróficos exhiben fases lag prolongadas, sin embargo se encontró que el lactato estimulaba la expresión de la ruta para la oxidación de propionato, disminuyendo la fase lag exhibida en tratamientos de propionato. Muchas veces fallas en el digestor son debidas a la acumulación de ácidos grasos, ya que estos inhiben a los metanógenos; el propionato inhibe a los metanógenos en un mayor nivel. Suplementar lactato podría probarse para remediar problemas como la acumulación de propionato. La metanogénesis es considerada el último paso en la digestión anaerobia, llevada a cabo por diferentes grupos de arqueas, denominadas metanógenos. Los metanógenos son susceptibles a cambios externos en el medio. Se han hecho estudios para relacionar la expresión de gen mcrA con la producción de metano (Freitag y Prosser, 2009). Guo y col. (2008), observaron una disminución del 8% en la producción de metano en la fermentación de rumen al adicionar saponinos de té, mientras que la expresión del gen mcrA disminuyo 76%. Ellos atribuyen esta reducción drástica de mcrA a una disminución en la población de protozoarios del 79%, ya que se sabe que los metanógenos realizan endosimbiosis con protozoarios ciliados. A partir de esto podemos concluir que es difícil encontrar una correlación directa entre la expresión de un solo gen con la producción de metano, el cual involucrada múltiples procesos en una comunidad microbiana compleja. En el estudio de Guo y col. (2008) también se observó que una reducción en protozoarios llevó a un incremento molar en propionato de 2.6%. Acumulación de propionato en un http://www.postgradoeinvestigacion.uadec.mx/divulgacionAQM.html 28 2011 Volumen 3, No. 6 Revista Científica de la Universidad Autónoma de Coahuila reactor puede llevar a la inhibición de metanógenos, la cual en cambio, disminuye el consumo de hidrogeno, y esto llevaría a una inhibición en la oxidación de propionato lo que disminuiría la cantidad de hidrogeno siendo generada, esto causaría también un estrés de limitación de hidrogeno en los metanógenos. Kota y col. (2008) reportaron una sobre-regulación de transcritos de enzimas reductoras y oxidadoras de F420 durante limitación de hidrogeno, bajo pH y condiciones estáticas. Por el contrario, en los metanógenos acetoclásticos, M. mazei Go1, Hovey y col. (2005) mostraron una disminución en la expresión de estas enzimas reductoras y oxidadoras de F420. Sin embargo, ellos también observaron un aumento en la expresión de ferrodoxinas y flavoproteinas no caracterizadas las cuales se presumen están involucradas como portadoras de electrones en la ruta acetoclastica. Hovey y col. (2005) también mostraron una expresión elevada de enzimas metiltransferasas viz. mtaA1. mtaB1 y mtaC1 cuando las células fueron cultivadas con metanol. Estas enzimas están involucradas en la metanogénesis a partir de compuestos metilados. Shih y Lai (2010) estudiaron la respuesta de estrés hipo e hiperosmotico en Methanohalophilus portucalensis cultivados con trimetilamina. Ellos observaron un aumento en los transcritos relacionados a la producción metanogénica a partir de productos metilados. Asi pues, M. portucalensis requiere energía para superar estrés osmótico. Muchos metanogenos también podrían tener similares respuestas. CONCLUSIÓN La digestión anaerobia es una biotecnología que ofrece una alternativa prometedora a los problemas energéticos y ambientales actuales. Sin embargo, más investigación es requerida para comprender más detalladamente los mecanismos de interacción entre los grupos microbianos, la producción de metabolitos y los factores ambientales y su relación como los agentes inhibitorios en orden de incrementar los rendimientos de metano, de forma que las necesidades energéticas mundiales puedan ser cubiertas satisfactoriamente. El estudio de la expresión genética surge como una respuesta a dicha necesidad, pudiendo alcanzar un mejor entendimiento de las interacciones tróficas de los microorganismos, sus respuestas a varios tipos de estrés y elucidar fenómenos complejos de las comunidades. Adicionalmente, la expresión genética puede agregar pistas de las necesidades que tienen las comunidades de un digestor anaerobio y así tomar las mejores decisiones para lidiar con las respuestas al estrés, mejorando así los rendimientos de metano. REFERENCIAS Ali M y Sreekrishnan TR. 2001. Aquatic toxicity from pulp and paper mill effluents: a review. Advances in Environmental Research. 5: 175-196. Angelidaki I y Ahring BK. 1993. Thermophilic digestion of livestock waste: the effect of ammonia, Applied Microbiology and Biotechnology. 38: 560-564. Ariesyady HD, Ito T, Yoshiguchi K y Okabe S. 2007. Phylogenetic and functional diversity of propionate-oxidizing bacteria in an anaerobic digester sludge. Applied Microbiology and Biotechnology. 75: 673-683. Aspé E, Martí MC, Jara A y Roeckel M. Ammonia inhibition in the anaerobic treatment of fishery effluents. 2001. Water Environmental Research. 73: 154–64. Bekmezci OK, Ucar D, Haksonen AH y Sahinkaya E. 2011. Sulfidogenic biotreatment of synthetic acid mine drainage and sulphide oxidation in anaerobic baffled reactor. Journal of Hazardous Materials. In Press. doi:10.1016/j.jhazmat.2011.01.087. Ben-Bassat A, Lamed R y Zeikus JG. 1981. Ethanol production by thermophilic bacteria: metabolic control of end product formation in Thermoanaerobium brockii. Journal of Bacteriology. 146:192–199. Bonacker LG, Baudner S y Thauer RK. 1992. Differential expression of the two methyl-coenzyme M reductases in Methanobacterium thermoautotrophicum as determined immunochemically via isoenzyme-specific antisera. European Journal of Biochemistry. 206: 87-92, Boone DR y Bryant MP. 1980. Propionate-degrading bacterium, Syntrophobacter wolinii sp. nov. gen. nov., from methanogenic ecosystems. Applied and Environmental Microbiology. 40: 626–632. http://www.postgradoeinvestigacion.uadec.mx/divulgacionAQM.html 29 2011 Volumen 3, No. 6 Revista Científica de la Universidad Autónoma de Coahuila Chen Y, Cheng JJ y Creamer KS. 2008. Inhibition of anaerobic digestion process: a review. Bioresource Technology. 99: 4044-4064. Cohen-Kupiec R, Blank C y Leigh JA. 1997. Transcriptional regulation in Archaea: in vivo demonstration of a repressor binding site in a methanogen. Proceedings of The National Academy Of Sciences. 94: 1316-1320. Colleran E, Concannon F, Golden T, Geoghegan F, Crumlish B, Killilea E, Henry M y Coates J. 1992. Use of methanogenic activity tests to characterise anaerobic sludges, screen for anaerobic biodegradability and determine toxicity thresholds against individual trophic groups and species. Water Science and Technology. 25: 31-40. Daoming S y Forster CF. 1993. An examination of the start up of a thermophilic upflow sludge blanket reactor treating a synthetic coffee waste. Environmental Technology. 14: 9656-972 De Baere L, Devocht M, Assche P y Verstraete W. 1984. Influence of high sodium chloride and ammonium chloride salt levels on methanogenic association. Water Research. 18: 543-548. De Bok FAM, Hagedoorn PL, Silva PJ, Hagen WR, Schiltz E, Fritsche K y Stams AJM. 2003. Two W-containing formate dehydrogenases (CO2 reductases) involved in syntrophic propionate oxidation by Syntrophobacter fumaroxidans. European Journal of Biochemistry. 270: 2476-2485 Demirel B y Scherer P. 2008. The roles of acetotrophic and hydrogenotrophic methanogens during anaerobic conversion of biomass to methane: a review. Reviews in Environmental Science and Bio/Technology. 7: 173–190. Demirel B y Yenigün O. 2002. Two-phase anaerobic digestion processes: a review. Journal of Chemical Technology and Biotechnology. 77: 743-755. Doyle O, O’Malley J, Clausen E, Gaddy J. 1983. Kinetic improvements in the production of methane from cellulosic residues. Energy Biomass Wastes. 7: 54561. Feijoo G, Vidal G, Moreira MT, Mendez R y Lema JM.1995. Degradation of high molecular weight compounds of kraft pulp mill effluents by a combined treatment with fungi and bacteria. Biotechnology Letters. 17: 1261-1266. Fischer E, Lieske R y Winzer K. 1932. Biologische Gasreaktiohen, lI. Mitteilung: fiber die Bildung yon Essigsfiure bei der biologischen Umsetzung von Kohlenoxyd und Kohlensfiure mit Wasserstoff zu Methan. Biochemistry Z. 245: 212 Freedman DL y Gossett JM. 1991. Biodegradation of dichloromethane and its utilization as growth substrate under methanogenic conditions. Applied and Environmental Microbiology. 57: 2847-2857 Freitag TE y Prosser JI. 2009. Correlation of methane production and functional gene transcriptional activity in a peat soil. Applied and Environmental Microbiology. 75: 6679-6687 Gavala H, Yenal U, Skiadas I, Westermann P y Ahring B. 2003. Mesophilic and thermophilic anaerobic digestion of primary and secondary sludge. Effect of pre-treatment at elevated temperature. Water Research. 37: 4561-4572. Gavala HN y Ahring, BK. 2002. Inhibition of the anaerobic digestion process by linear-alkylbenzene sulfonates. Biodegradation. 13: 201–209. Gavala, H. N. y Lyberatos, G. 2001. Influence of anaerobic culture acclimation on the degradation kinetics of various substrates. Biotechnology and Bioengineering. 74(3): 181-195. Godbole SH, Gore JA, Ranade DR. 1981. Associative Action of the Various Groups of Microorganisms in the Production of Biogas. Biovignyanam. 7: 107–113. Grady Jr CPL, Daigger GT y Lim HC. 1999. Biological Waste Water Treatment. Marcel Dekker, New York. http://www.postgradoeinvestigacion.uadec.mx/divulgacionAQM.html 30 2011 Volumen 3, No. 6 Revista Científica de la Universidad Autónoma de Coahuila Guo YQ, Liu JX, Lu Y, Zhu WY, Denman SE y McSweeney CS. 2008. Effect of tea saponin on methanogenesis, microbial community structure and expression of mcrA gene, in cultures of rumen micro-organisms. Letters in Applied Microbiology. 47: 421-426 Hazelwood GP y Nugent JHA. 1978. Leaf fraction 1 protein as a nitrogen source for the growth of a proteolytic rumen bacterium. Journal of General Microbiology. 106:369-371. Hendrickson EL, Haydock AK, Moore BC, Whitman WB y Leigh JA. 2007. Functionally distinct genes regulated by hydrogen limitation and growth rate in methanogenic Archaea. Proceedings of the National Academy of Sciences of the United States of America. 104: 8930-8934 Hess M, Sczyrba A, Egan R, Kim TW, Chokhawala H, Schroth G, Luo S, Clark DS, Chen F, Zhang T, Mackie RI, Pennacchio LA, Tringe SG, Visel A, Woyke T, Wang Z y Rubin EM. 2011. Metagenomic discovery of biomassdegrading genes and genomes from cow rumen. Science. 331: 463-467. Hovey R, Lentes S, Ehrenreich A, Salmon K, Saba K, Gottschalk G, Gunsalus RP y Deppenmeier U. 2005. DNA microarray analysis of Methanosarcina mazei Gö1 reveals adaptation to different methanogenic substrates. Molecular Genetics and Genomics. 273: 225-39. Hwu CS y Lettinga G. 1997. Acute toxicity of oleate to acetate-utilizing methanogens in mesophilic and thermophilic anaerobic sludges. Enzyme and Microbiology Technology. 21: 297–301. Isa Z, Grusenmeyer S. y Verstraete W. 1986. Sulfate reduction relative to methane production in high-rate anaerobic digestion: microbiological aspects. Applied and Environmental Microbiology. 51: 580-587. Jin P, Bhattacharya SK, Williama CJ y Zhang H. 1998. Effects of sulfide addition on copper inhibition in methanogenic systems. Water Research. 32: 977–988. Kansal A, Rajeshwari KV, Balakrishnan M, Lata K, y Kishore VVN. 2004. Anaerobic digestion technologies for recovery from industrial waste water- a study in Indian context. TERI Information Monitor on Environmental Science.3: 67-75. Kato S, Kosaka T y Watanabe K. 2008. Comparative transcriptome analysis of responses of Methanothermobacter thermautotrophicus to different environmental stimuli. Environmental Microbiology. 10: 893-905 Kato S, Kosaka T y Watanabe K. 2009. Substrate-dependent transcriptomic shifts in Pelotomaculum thermopropionicum grown in syntrophic co-culture with Methanothermobacter thermautotrophicus. Microbial Biotechnology. 2: 575-584 Kayhanian M., 1999. Ammonia inhibition in high-solids biogasification: an overview and practical solutions. Environmental Technology. 20: 355–365. Koster IW y Koomen E. 1988. Ammonia inhibition of the maximum growth rate (µm) of hydrogenotrophic methanogens at various pH-levels and temperatures. Applied Microbiology and Biotechnology. 28: 500–505. Koster IW y Lettinga G. 1984.The influence of ammonium-nitrogen on the specific activity of pelletized methanogenic sludge, Agricultural Wastes. 9: 205-216. Koster IW, Rinzema A, De Vegt AL y Lettinga G. 1986. Sulfide inhibition of the methanogenic activity of granular sludge at various pH levels. Water Research. 20: 1561–1567. Kroeker, EJ, Schulte DD, Sparling AB y Lapp HM. 1979. Anaerobic treatment process stability. Journal of the Water Pollution Control Federation. 51: 718-727. Kroiss H y Plahl-Wabnegg F. 1983. Sulphide toxicity with anaerobic wastewater treatment. Environmental Symposium on Anaerobic Wastewater treatment, The Netherlands, 72-85. http://www.postgradoeinvestigacion.uadec.mx/divulgacionAQM.html 31 2011 Volumen 3, No. 6 Revista Científica de la Universidad Autónoma de Coahuila Kugelman IJ y Chin KK. 1971. Toxicity, synergism, and antagonism in anaerobic waste treatment processes. In: Anaerobic Biological Treatment Processes, American Chemical Society Advances in Chemistry Series 105: 55–90. Lalman J y Bagley DM. 2001. Anaerobic Degradation and Methanogenic Inhibitory Effects of Oleic and Stearic Acids. Water Research. 35: 2975-2983. Lange M, Tolker-Nielsen T, Molin S y Ahring BK. 2000. In situ reverse transcription-PCR for monitoring gene expression in individual Methanosarcina mazei S-6 cells. Applied and Environmental Microbiology. 66: 1796-1800. Lee YH y Pavlostathis SG. 2004. Decolorization and Toxicity of Reactive Anthraquinone Textile Dyes under Methanogenic Conditions. Water Research 38:1838-1852. Lehmacher A y Hensel R. 1994. Cloning, sequencing and expression of the gene encoding 2-phosphoglycerate kinase from Methanothermus fervidus. Molecular and General Genetics. 242: 163-168. Lin CY. 1993. Effect of heavy metals on acidogenesis in anaerobic digestion. Water Research. 27: 147–152. Liu Y y Boone DR. 1991. Effects of salinity on methanogenic decomposition. Bioresource Technology. 35: 271–273. Livera, J, McLaughlin MJ, Hettiarachchi Kirby JK y Beak D. Cadmium solubility in paddy soils: effects of soil oxidation, metal sulfides and competitive ions. Science of the Total Environment. 409: 1489-1497. Ljungdahl LG. 1986. The autotrophic pathway of acetate synthesis in acetogenic bacteria. Annual Review of Microbiology. 40: 415-450. Maillacheruvu, KY, Parkin GF, Peng WC, Kuo WC, Oonge ZI y Lebduschka V. 1993. Sulfide toxicity in anaerobic systems fed sulfate and various organics. Water Environmental Research. 65: 100–109. McCarty PL. 1964. Anaerobic waste treatment fundamentals III. Public Works. 95: 91-94. McInerney MJ y Bryant MP. 1981. Anaerobic degradation of lactate by syntrophic associations of Methanosarcina barkeri and Desulfovibrio and effect of H2 on acetate degradation. Applied and Environmental Microbiology. 41: 346354. Nagamani B, y Ramasamy K. 1999. Biogas Technology: An Indian Perspective. Current Science. 77: 44-55. Nies, D.H. 1999. Microbial heavy-metal resistance. Applied Microbiology and Biotechnology. 51: 730–750 Parkin GF y Owen WF. 1986. Fundamental of anaerobic-digestion of wastewater sludge. Journal of Environmental Engineering. 112: 867–920. Pavlostathis, S.G. and Giraldo-Gomez, E. 1991. Kinetics of anaerobic treatment: a critical review. Critical Reviews in Environmental Control. 21: 411–490. Preeti Rao P, Shivaraj D y Seenayya G. 1993. Succession of microbial population in cow dung and poultry litter waste digesters during methanogenesis. Indian Journal of Microbiology. 33: 185–189. Ramasamy K, Nagamani B y Kalaichelvan G. 1991. 31st Annual Conference of AMI held at TNAU, Coimbatore, 96 p. Ramsamy K. 1997. Proceedings of the National Symposium on Community and Institutional Biogas Complexes held at Punjab Agricultural University, Ludhiana, 4–5 p. Raskin L, Rittmann BE y Stahl DA. 1996. Competition and coexistance of sulfate-reducing and methanogenic populations in anaerobic biofilms. Applied and Environmental Microbiology. 62: 3847-3857. Rinzema, A, van Lier J y Lettinga G. 1988. Sodium inhibition of aceticlastic methanogens in granular sludge from a UASB reactor. Enzyme and Microbial Technology. 10: 24–32. http://www.postgradoeinvestigacion.uadec.mx/divulgacionAQM.html 32 2011 Volumen 3, No. 6 Revista Científica de la Universidad Autónoma de Coahuila Schink B. 1997. Energetics of syntrophic cooperation in methanogenic degradation. Microbiology and Molecular Biology Reviews. 61(2): 262–280. Shih CJ y Lai MC. 2010. Differentially expressed genes after hyper- and hypo-salt stress in the halophilic archaeon Methanohalophilus portucalensis. Canadian Journal of Microbiology. 56: 295-307 Siebert ML y Torien DF. 1969. The proteolytic bacteria present in the anaerobic digestion of raw sewage sludge, Water Research. 3: 241-250. Sossa K, Alarcón M, Aspé y Urrutia H. 2004. Effect of ammonia on the methanogenic activity of methylaminotrophic methane producing archaea enriched biofilm. Anaerobe. 10: 13-18. Soto M, Méndez R y Lema J. Sodium Inhibition and sulfate reduction in the anaerobic treatment of mussel processing waste. 1993. Journal of Chemical Technology and Biotechnology. 58:1. Spanheimer R, Hoffmann M, Kögl S, Schmidt S, Pflüger K y Müller V. 2008. Differential regulation of Ota and Otb, two primary glycine betaine transporters in the methanogenic archaeon Methanosarcina mazei Gö1. Journal of Molecular Microbiology and Biotechnology. 15: 255-263. Speece RE. 1996. Anaerobic biotechnology for industrial wastewaters. Archae Press, Nashville, Tennessee, USA. Sprott GD y Patel GB. 1986. Ammonia toxicity in pure cultures of methanogenic bacteria. Systematic and Applied Microbiology. 7: 358–363. Stathopoulos C, Tong L, Longman R, Vothknecht UC, Becker HD, Ibba M y Soll D. 2000. One Polypeptide with Two Aminoacyl-tRNA Synthetase Activities. Science. 287: 479. Sung S. y Liu T. 2003. Ammonia inhibition on thermophilic digestion. Chemosphere 53: 43–52. Toeri en DF y Hattingh WHJ. 1969. Anaerobic digestion. 1. The microbiology of anaerobic digestion. Water Research. 3:385 Ulmanu M, Maranon, E., Ferna´ndez, Y., Castrillo´n, L., Anger, I., y Dumitriu, D., 2003. Removal of copper and cadmium ions from diluted aqueous solutions by low cost and waste material adsorbents. Water, Air and Soil Pollution. 142: 357–373. Ungerfeld, EM, Rust SR, Boone DR y Liu Y. 2004. Effects of several inhibitors on pure cultures of ruminal methanogens. Journal of Applied Microbiology. 97: 520-526. Vallero MVG, Lettinga G. y Lens PNL. 2003. Long-term adaptation of methanol-fed thermophilic (55° C) sulfatereducing reactors to NaCl. Journal of Industrial Microbiology and Biotechnology. 30: 375–382. van Kessela JAS y Russell JB. The effect of pH on ruminal methanogenesis. Microbial Ecology. 20: 205-210 Veeken A y Hamelers B. 1999. Effect of temperature on hydrolysis rate of selected biowise components. BioresourceTechnology. 69: 249–254. Vercoe PE, Kocherginskaya SA y White BA. 2003. Differential protein phosphorylation–dephosphorylation in response to carbon source in Ruminococcus flavefaciens FD-1. Journal of Applied Microbiology. 94: 974-980 Wang Y, Zhang Y, Wanga J y Menga L. 2009. Effects of volatile fatty acid concentrations on methane yield and methanogenic bacteria. Biomass and Bioenergy. 33: 848-853. Ward AJ, Hobbs PJ, Holliman PJ y Jones DL. 2008. Optimization of the anaerobic digestion of agricultural resources. Bioresource Technology. 99: 7928-7940. http://www.postgradoeinvestigacion.uadec.mx/divulgacionAQM.html 33 2011 Volumen 3, No. 6 Revista Científica de la Universidad Autónoma de Coahuila Widdel F y Wolfe RS. 1989. Expression of secondary alcohol dehydrogenase in methanogenic bacteria and purification of the F420-specific enzyme from Methanogenium thermophilum strain TCI. Archives of Microbiology. 152: 322-328 Wiegant, WM y Zeeman G. 1986. The mechanisms of ammonia inhibition in the thermophilic digestion of livestock wastes. Agricultural Wastes. 16: 243-253. Wieringa, K. T. 1936. Over het verdwijnen van waterstof en koolzuur onder anaerobe voorwarden. Antonie van Leeuwenhoek Journal of Microbiology and Serology. 3:263-273. Wong, MH y Cheung YH. 1995. Gas production and digestion efficiency of sewage sludge containing elevated toxic metals. Bioresource Technology. 54: 261–268. Wood HG. 1991. Life with CO or CO2 and H2 as a source of carbon and energy. Federation of American Societies for Experimental Biology Journal. 5: 156-163 Xia Q, Wang T, Hendrickson EL, Lie TJ, Hackett M y Leigh JA. 2009. Quantitative proteomics of nutrient limitation in the hydrogenotrophic methanogen Methanococcus maripaludis. BMC Microbiology. 9: 149 Zayed, G. y Winter, J. 2000. Inhibition of methane production for whey by heavy metals protective effect of sulfide. Applied Microbiology and Biotechnology, 53: 726–731. Zeeman G, Wiegant WM, Koster-Treffers ME y Lettinga, G., 1985. The influence of the total ammonia concentration on the thermophilic digestion of cow manure. Agricultural Wastes, 14, 19–35. Zhou HB y Qiu, GZ. 2006. Inhibition effect of ammonia nitrogen on specific methanogenic activity of anaerobic granular sludge. Journal of Central South University of Technology, 13, 63-67. http://www.postgradoeinvestigacion.uadec.mx/divulgacionAQM.html 34