Capítulo 24. Replicación, Transcripción y Traducción
Anuncio

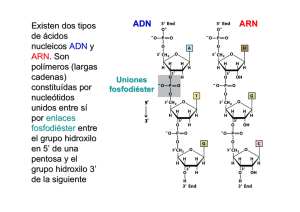
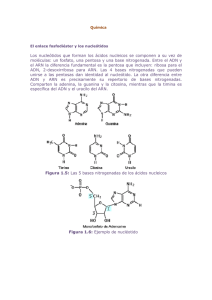
Capítulo 24. Replicación, Transcripción y Traducción El dogma central de la Biología involucra esencialmente la duplicación del ADN, la transcripción de la información contenida en el ADN en forma de ARN y la traducción de esta información del ARN a la proteína. Este principio se puede resumir de la siguiente manera: Figura 1. La información fluye en una única dirección del ADN a las proteínas, pero hay excepciones, como es el caso de la transcripción de ADN a partir de ARN. Transmisión de la información genética Implícito en la estructura de doble hélice del ADN esta el mecanismo por el cual este puede replicarse (duplicarse), es decir hacer copias exactas de sí mismo. La replicación del ADN es un proceso que ocurre sólo una vez en cada generación celular, por lo tanto, las dos células hijas provenientes de la división celular contienen una copia idéntica del ADN de la célula madre que le dio origen. Watson y Crick propusieron un mecanismo de replicación semiconservativa en base al modelo estructural del ADN planteado, según el cual la doble hélice del ADN se abre por el medio y las bases apareadas se separan a nivel de los puentes de hidrógeno. A medida que se separan, las dos cadenas actúan como moldes o guías, cada una dirigiendo la síntesis de una nueva cadena complementaria a lo largo de toda su extensión. Como vimos en el capítulo 23, la complementariedad de las bases sólo permite dos tipos de apareamientos T-A y G-C; las bases se van agregando una a una y la selección de cuál base entra en un sitio específico de la cadena en formación, queda determinada por la base presente en la cadena molde con la que se va a aparear. De esta manera, cada cadena molde forma una copia de su cadena complementaria original y se producen dos replicas exactas de la molécula. Este modelo brindó una respuesta de cómo la información hereditaria se duplica y pasa de generación en generación. Este mecanismo se denomina replicación semiconservativa porque la doble hélice progenitora se replica dando lugar a dos dobles hélices hijas, cada una de las cuales está formada por una cadena progenitora (cadena vieja) y una cadena hija (sintetizada de novo). La unidad que se mantiene de una generación a la siguiente, es una de las dos hebras individuales que formaba parte de la doble hélice progenitora, es por ello que este proceso recibe el nombre de replicación semiconservativa. 259 Figura 2. Mecanismo de replicación, transcripción y traducción. Replicación del ADN Luego del descubrimiento de la estructura del ADN, en 1957 dos biólogos americanos, Matthew Meselson y Franklin Stahl utilizando cultivos bacterianos de Escherichia coli, demostraron que el ADN celular se replica por el mecanismo semiconservativo propuesto por Watson y Crick. En el experimento, aprovecharon la disponibilidad de un isótopo pesado del nitrógeno (15N) y un método extremadamente sensible para separar macromoléculas a base de su densidad. Estos investigadores utilizando el elemento N presente en la molécula de ADN, como el14N y el la incorporación del 15N radiactivo, pudieron demostrar que cuando el ADN se replica, una de sus cadenas pasa a las células hijas sin cambiar (14N) y es la que actúa como de molde o patrón, para así formar una segunda hebra complementaria (15N y completar las dos cadenas del ADN. El inicio de la replicación, tanto en procariotas como en eucariotas, se produce cuando la doble hélice de ADN es desenrollada y abierta en una secuencia específica de nucleótidos denominada origen de replicación. En los procariotas existe un único origen de replicación, localizado dentro de una secuencia específica de nucleótidos cuya longitud es de aproximadamente 300 pares de bases; en los eucariotas, en cambio, hay múltiples orígenes de replicación. Si se observa la figura 2, la zona de síntesis donde se comienzan a separar las cadenas progenitoras, la molecúla de ADN parece formar una estructura en forma de Y, denominada horquilla de replicación. Dentro de esta horquilla, la ADN polimerasa, sintetiza las nuevas cadenas complementarias. La replicación es bidireccional, por lo tanto existen dos horquillas de replicación que se mueven en direcciones opuestas desde el origen de replicación. 260 Para la síntesis de una nueva cadena complementaria de ADN se necesita no solo la presencia de la cadena progenitora (vieja) que sirva de molde, sino también de una secuencia de inicio para la nueva cadena que permita que la ADN polimerasa prolongue la cadena. Esta secuencia de inicio, conocida habitualmente como cebador (primer, en inglés) está formada por nucleótidos de ARN. Los nucleótidos de ARN pueden formar puentes de hidrógeno con los nucleótidos de la cadena de ADN, siguiendo un principio similar de complementariedad (la G se aparea con la C, la A del ARN con la T del ADN y el U del ARN con la A del ADN). La síntesis del cebador es llevada a cabo por una enzima denominada ARN primasa. Con los cebadores de ARN colocados en el lugar correcto, la ADN polimerasa agrega nucleótidos al extremo 3’ de las cadenas en crecimiento y a continuación cataliza la formación del enlace fosfodiéster para ligar este residuo a la nueva cadena que crece. El complejo polimerásico, no formará la unión fosfodiéster, a menos que la base que está entrando a la cadena en formación, sea complementaria a la base existente en la cadena patrón. La frecuencia con la que se inserta una base equivocada es menor a 1 en 100 millones, debido a la capacidad de la ADN polimerasa de corregir errores (eliminación de nucleótidos mal incorporados). Se comprobó que las primeras polimerasas descubiertas sintetizaban nuevas cadenas solamente en la dirección 5´- 3´. Dada la estructura antipararela de la doble hélice de ADN, la replicación de las dos nuevas cadenas de ADN sobre los dos brazos de la horquilla de replicación parecía requerir la síntesis en la dirección 5´- 3´, sino también en la dirección 3´- 5´. Durante varios años los investigadores trataron de identificar otra ADN polimerasa que pudiera funcionar en la dirección 3´-5´, pero no la encontraron. Un científico japonés, Reiji Okazaki encontró que, aunque la cadena 5´-3´ se sintetiza continuamente como una sola unidad, la cadena 3´-5´ se sintetiza de manera discontinua, como una serie de fragmentos, cada uno de los cuales es sintetizado en la dirección 5´-3´. La cadena que crece de manera continua se conoce como cadena adelantada y la cadena que se sintetiza por fragmentos se conoce como cadena retrasada. La síntesis de la cadena adelantada requiere un único cebador en un único sitio, pero la síntesis de los fragmentos que conforman la cadena rezagada, los fragmentos de Okazaki, requieren múltiples cebadores. Una vez que la ADN polimerasa alarga estos cebadores, todos los fragmentos de ARN de la hebra retrasada son degradados y reemplazados por ADN. Luego de que el cebador es degradado, una enzima específica es la encargada de unir todos los fragmentos sintetizados. Síntesis de proteínas Los procesos que se llevan a cabo para la síntesis de proteínas constan de dos pasos fundamentales, la transcripción (1) y la traducción (2), a través de los cuales la información contenida en el ADN se convierte en proteínas (Figura 1). En la transcripción se sintetiza ARN a partir de un molde de ADN y en la traducción, la secuencia de bases del ARNm especifica la secuencia de aminoácidos que se ensamblarán para formar las proteínas. No solamente una, sino las tres clases de ARN, desempeñan funciones como intermediarios en los pasos que llevan del ADN a la proteína. Cabe destacar que la transcripción de genes, aparte de dar lugar a la formación de ARNm, también sintetiza el ARNr (que forma parte de los ribosomas, complejo compuesto por proteínas y ARNr, donde se realiza el proceso de traducción) y el ARNt (moléculas que funcionan como adaptadores en dicho proceso de traducción). 261 1) Transcripción En una célula eucariota, el ARNm, es sintetizado en el núcleo y trasladado a los ribosomas en el citoplasma. El ARNm lleva la información que dicta qué aminoácidos formarán la proteína que se va a sintetizar. Las moléculas de ARNm son copias (transcriptos) de secuencias de ADN, pero a diferencia de las moléculas de ADN, las moléculas de ARN son de cadena única (monocatenaria). En cada evento de transcripción, se transcribe solo una de las dos cadenas del ADN y según el gen en cuestión, se transcribe una cadena o la otra, nunca las dos. Como ocurre en la síntesis de ADN, los ribonucleótidos presentes en la célula como trifosfatos son añadidos por una enzima ARN polimerasa, que se desplaza por la cadena patrón de ADN y va insertando nucleótidos de ARN siguiendo la complementariedad de bases. Por ejemplo: si la secuencia de ADN es: 3'... TACGCT...5', la correspondiente secuencia de ARNm mediada por la ARN polimerasa será: 5'... AUGCGA...3'. Es decir, esta enzima cataliza la adición de ribonucleótidos, uno a uno, al extremo 3´de la cadena de ARN en crecimiento. Es importante señalar que el ARNm, tiene una secuencia complementaria a la cadena molde de ADN, que es igual a la otra cadena del ADN, salvo el remplazo de timina (T) por uracilo (U). Pero, a diferencia de la ADN polimerasa, la ARN polimerasa no necesita un cebador para comenzar la síntesis de ARN e inicia una nueva cadena simplemente uniendo dos ribonucleótidos. ARN Polimerasa Figura 3. La ARN polimerasa reconoce el promotor, una secuencia específica de nucleótidos, que define el sitio de inicio de la transcripción. En una primera etapa, la enzima ARN polimerasa se asocia a secuencias específicas de nucleótidos del ADN, que se conoce como promotoras, las cuales dirigen la transcripción de segmentos adyacentes de ADN (genes) (Figura 3 y 4). Esta secuencia define además el punto exacto de inicio de la transcripción y la dirección hacia la cual avanzará la ARN polimerasa. Una vez unida la ARN polimerasa, abre y desenrolla la doble hélice de manera que queden expuestos algunos nucleótidos. Ésta va añadiendo ribonucleótidos, moviéndose a lo largo de la de la hebra de ADN que se utiliza como patrón, desenrollando la hélice y exponiendo así nuevas regiones para transcribir, hasta que se encuentra con otras secuencias específicas del ADN, llamadas terminadoras, que señalan la detención de la síntesis de ARN. 262 Cuando se ha copiado toda la hebra al final del proceso, las instrucciones genéticas copiadas o transcriptas al ARNm están listas para salir al citoplasma. La cadena de ARN queda libre y el ADN se cierra de nuevo, por apareamiento de sus cadenas complementarias (Figura 4). Esta etapa, se denomina terminación de la transcripción. Secuencia de inicio (Promotor) ADN Secuencia de terminación ARN m inmaduro Figura 4. Inicio y terminación de la transcripción. La transcripción en las células eucariotas es igual, en principio a la de las células procariotas; comienza con la unión de la enzima, una ARN polimerasa, a una secuencia determinada, el promotor, en una cadena de la doble hélice. Esta cadena funciona luego como molde para el ensamble de ribonucleótidos. Las moléculas de ARN transcriptas (ARNr, ARNt y ARNm) desempeñan luego sus distintos papeles en la traducción a proteína de la información genética codificada.A pesar de esa similitud básica, hay algunas diferencias significativas de transcripción, en los procariotas y en los eucariotas. Una diferencia es que los genes eucarióticos no están agrupados en operones (en el cromosoma bacteriano, un segmento de ADN que consiste de un promotor, un operador y un grupo de genes estructurales adyacentes, se le denomina operon) en los cuales dos o más genes estructurales se transcriben a una sola molécula de ARN, como ocurre frecuentemente en procariotas. En los eucariotas cada gen estructural se transcribe por separado. También hay diferencias en las enzimas implicadas en la trascripción, siendo la más notable que en los procariotas una sola ARN polimerasa cataliza la biosíntesis de los tres tipos de ARN. En los eucariotas hay tres polimerasas diferentes: una transcribe los genes que se traducirán a proteínas, una segunda transcribe los genes de los ARN ribosómicos grandes, y una tercera transcribe para una variedad de ARN pequeños, incluyendo los ARNt y los ARN pequeños del ribosoma. 263 Fenómenos postranscripcionales Si bien los pasos básicos son los mismos para la mayoría de los organismos, hay diferencias entre los distintos dominios (Bacteria, Arquea y Eucaria) tanto en la replicación, como en la transcripción y en la traducción. En los procariontes (organismos que carecen de núcleo y orgánulos limitados por membranas), los ribosomas se unen a moléculas de ARNm, sin sufre ninguna modificación y de esta manera su traducción a proteína comienza aún antes de que se haya completado la transcripción. En los eucariontes, sin embargo, la traducción y transcripción ocurren en forma separada tanto en tiempo como en espacio. La transcripción ocurre en el núcleo y la traducción, en el citoplasma, puede ocurrir en minutos, horas o incluso días más tarde. Una molécula de ARN recién sintetizada recibe el nombre de transcripto primario. La modificación más extensiva de los transcriptos primarios tal vez tenga más a lugar en los ARNm que en los ARNt. Por ejemplo, antes de salir del núcleo para ser traducido, el ARNm sufre varias modificaciones. Es decir que, aún antes de haberse completado la transcripción, cuando la cadena de ARNm tiene aproximadamente 25 pares de bases de largo, al extremo 5’ del ARNm se le une un nucleótido inusual, la 7-metil guanina (caperuza). Esta caperuza es necesaria para la unión del ARNm al ribosoma. Completada la transcripción el ARNm se separa de la cadena molde de ADN, y enzimas específicas agregan al extremo 3’ una cadena de nucleótidos de adenina (que se puede denominar poliadenina, cola o poliA) (Figura 5). La longitud de esta cadena puede ser de hasta 200 nucleótidos. 5´ CAPERUZA 3´ Exón Intrón Exón 2 Intrón Exón 3 AAAAAAAAA OH Figura 5. ARNm con caperuza y poliadenina El transcripto primario de un ARNm eucariótico contienen las secuencias que corresponden a un gen, aunque las secuencias que codifican un polipéptido pueden no ser contiguas. Los fragmentos que interrumpen la región codificante del transcripto se denominan intrones y los segmentos codificantes se denominan exones. Antes que las moléculas de ARNm portando la caperuza y la cola poliA, dejen el núcleo, se produce el procesamiento por corte y empalme (splicing, en inglés), en el cual se eliminan las secuencias no codificantes del transcripto primario (intrones) y los exones son unidos para formar una secuencia continua que especifica un polipéptido funcional (Figura 6). Luego de todas estas modificaciones, se obtiene un ARN maduro o trascripto maduro. Varios transcriptos idénticos de ARNm maduro pueden ser procesados de más de una forma. Este empalme alternativo resulta en la formación de más de un polipéptido funcional a partir de moléculas de ARN que originalmente eran idénticas. Los ARNm son finalmente transportados al citoplasma asociados a proteínas (ribonucleoproteínas), que ayudan a transportar las moléculas de ARNm a través de los poros nucleares y además ayudan a unir estos ARNm a los ribosomas. 264 NÚCLEO CAPERUZA AAAAAAAAA OH 5´ 3´ Exón Intrón Exón 2 Intrón Exón 3 Exón Intrón Exón 2 Intrón Exón 3 5´ CAPERUZA ARNm inmaduro 3´ AAAAAAAAA OH ARNm inmaduro Splicing CAPERUZA Exón Exón 2 Exón 3 AAAAAAAAA OH ARNm maduro CITOSOL Figura 6. Procesamiento del ARNm inmaduro por corte y empalme, dando lugar a un ARNm maduro que es transportado al citoplasma. 2) Traducción Como se mencionó anteriormente, las instrucciones para la síntesis de proteínas están codificadas en secuencias de nucleótidos en la molécula de ADN. La replicación semiconservativa del ADN transmite estas instrucciones de la célula madre a la célula hija y de generación en generación. De esta manera cada nueva célula y cada nuevo organismo, hereda la información necesaria para sintetizar las proteínas específicas que determinan su estructura y funciones particulares. La síntesis de proteínas requiere además del ARNm, de los otros dos tipos de ARN: el ARNr y el ARNt. Estas moléculas difieren del ARNm estructuralmente y sobretodo funcionalmente. En la mayoría de las células el ARNr es el tipo más abundante. Los ribosomas consisten de dos subunidades (una pequeña y otra más grande) estando formadas aproximadamente por 2/3 de ARN y 1/3 de proteínas. Estas subunidades son diferentes en procariotas y eucariotas, en cuanto al tipo de ARNr y las proteínas que las conforman. La subunidad pequeña del ARNr, contiene un sitio de unión para el ARNm. Por lo cual, cuando el ARNm se encuentra en el citoplasma, es reconocido mediante secuencias específicas (en bacterias) y por la caperuza (en eucariotas), que están presentes en el extremo 5´de la molécula de ARNm. La subunidad más grande tiene dos tipos de unión para el ARNt. En este momento cobra importancia el ARNt, que funciona como adaptador entre ARNm y los aminoácidos; es decir, es el diccionario por medio del cual se traduce el lenguaje de los ácidos nucleicos al lenguaje de las proteínas. Existen más de 20 tipos diferentes en cada célula, por lo menos uno para casa uno de los tipos de aminoácidos que se encuentran en las proteínas. Los 265 ARNt, tienen dos sitios de unión importantes, uno de ellos se conoce como anticodón, que se acopla al codón (tres nucleótidos continuos en el ARNm que forman el código para un aminoácido específico) de la molécula de ARNm (Figura 7). El otro sitio, en el extremo 3´del ARNt, se acopla a un aminoácido en particular. Este extremo, siempre termina en una secuencia que posee el triplete CCA, donde cada aminoácido se une. La secuencia de los otros nucleótidos, varía de acuerdo con el tipo particular de ARNt. Figura 7. Acoplamiento entre el ARNm y el ARNt, mediado por los ribosomas. El aminoácido correcto es unido a su ARNt por una enzima específica llamada aminoacilARNt sintetasa, con gasto de una molécula de ATP por cada aminoácido que se une a la enzima. Al haber 20 aminoácidos, también hay 20 aminoacil-ARNt sintetasa. Todos los ARNt con el mismo aminoácido son activados por la misma enzima. Primero, se escinde una molécula de ATP desprendiéndose dos fosfatos (PP) y se forma un complejo entre el aminoácido, una molécula de AMP y la enzima. Este complejo permanece intacto hasta que se encuentra con el ARNt apropiado. Cuando esto sucede, la molécula de AMP se desprende de la enzima y se forma un enlace entre el aminoácido y el extremo 3´del ARN y luego, también se desprende el complejo aminoacil-ARNt. Este proceso se llama aminoacilación o "carga" (Figura 8). Cuando la molécula de ARNt se ha unido mediante puentes de hidrógeno al ARNm, anticodón con codón, coloca de esta manera el aminoácido específico en su lugar. Luego, se rompe el enlace entre el ARNt y el aminoácido, cuando se ha formado un nuevo enlace, un enlace petídico. Así este ARNt queda libre para unirse a otro aminoácido y repetir este proceso de carga. 266 Figura 8. Complejo de activación de los ARNt. La etapa de elongación de la cadena polipeptídica, se inicia cuando un segundo codón del ARNm se coloca en la posición opuesta al sitio A (aminoacil) de la subunidad mayor. Un aminoacil-ARNt complementario al segundo codón del ARNm, se posiciona en el sitio A del ribosoma (Figuras 10). Cuando los dos sitios P y A están ocupados, una enzima que contiene la subunidad mayor (peptidil transferasa) forja un enlace peptídico entre los dos aminoácidos. El ribosoma se mueve un codón a lo largo de la cadena de ARNm. Y se vuelve a repetir el mismo proceso. Más de un ribosoma puede traducir un ARNm al mismo tiempo, haciendo posible con esto producir varios polipéptidos simultáneamente a partir de un solo ARNm. Este conjunto de ribosomas, se denominan polisomas. La síntesis del polipéptido se lleva a cabo hasta alcanzar el codón de finalización (alto o stop, Figura 11). El codón de finalización puede ser de tres tipos AUG, UAA y UGA; estos tres codones que no son reconocidos por ningún ARNt (es decir, que no codifican para ningún aminoácido) funcionan como señales de terminación. De esta manera, cuando aparece uno de estos tripletes UAA, UAG, UGA; la proteína recién formada se libera del ribosoma, gracias a que un factor de libramiento lee el triplete y la síntesis del polipéptido termina. De esta manera, la cadena polipeptídica se desprende y las dos subunidades ribosomales se separan. 267 Figura 9. lniciacion de Ia sfntesis proteica 268 Figura 10 . Etapa de elongación de la cadena polipeptídica. 269 Figura 11. Terminaci6n de Ia traducci6n. 270 Procesos de postraducción Una vez traducidas, las proteínas deben adoptar una estructura tridimensional adecuada. La cadena polipeptídica naciente se pliega y es modificada para adquirir su forma biológicamente activa. En algunos casos, el plegamiento se consigue gracias a la ayuda de otras proteínas denominadas chaperonas moleculares. Además pueden ser modificadas mediante la unión de distintas moléculas como azúcares, nucleósidos, o fosfatos y dirigidas a lugares específicos de la célula, como por ejemplo, la membrana celular, el núcleo, etc., de acuerdo con su función. Cuando una proteína cumplió su función o cuando no se plegó correctamente, es degradada. Las proteínas que van a ser degradadas son “marcadas” por la unión de una proteína llamada ubiquitina. Una vez marcadas, un complejo multiproteico denominado proteosoma, degrada las proteínas en aminoácidos por ruptura de los enlaces peptídicos. Aminoácidos Como vimos en el Capítulo 12, las proteínas contienen 20 aminoácidos diferentes, pero el ADN y el ARN contienen solo 4 nucleótidos diferentes. De las 64 combinaciones posibles de codones, 61 combinaciones especifican aminoácidos particulares y 3 son codones de terminación (Tabla 1). Dado que 61 combinaciones codifican 20 aminoácidos, esto denota que existe más de un codón para la mayoría de los aminoácidos, es por ello que se dice que el código genético es degenerado. Tabla 1. El código genético, consiste de 64 combinaciones de tripletes (codones) que determinarán cada uno de los aminoácidos. AMINOÁCIDO 271 CÓDIGO CÓDIGO DE 3 LETRAS DE 1 LETRA CODONES Alanina Ala A GCC - GCU - GCG - GCA Arginina Arg R CGC - CGG - CGU - CGA - AGA - AGC Asparagina Asn N AAU - AAC Ácido Aspártico Asp D GAU - GAC Cisteína Cys C UGU - UGC Acido Glutámico Glu E GAA - GAG Glutamina Gln Q CAA - GAC Glicina Gly G GCU - GGC - CGA - GGG Histidina His H CAU - CAC Isoleucina Ile I AUU - AUC - AUA Leucina Leu L UUA - UUG - CUA - CUG - CUU - CUC Lisina Lys K AAA - AAG Metionina Met M AUG - Fenilalanina Phe F UUC - UUU Prolina Pro P CCU - CCC - CCA - CCG Serina Ser S UCU - UCC - UCA - UCG - AGU - AGC Treonina Thr T ACU - ACC - ACA - ACG Tirosina Tyr Y UAU - UAC Triptofano Trp W UGC Valina Val V GUU - GUC - GUA - GUG