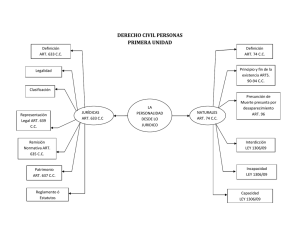
DESARROLLO DE U TEST DE ELISA SA DWICH PARA
Anuncio

Profesor Patrocinante Dr. Alex Romero Z. Instituto de Patología Animal Facultad de Ciencias Veterinarias Profesor Co-Patrocinante Dr. Ricardo Enríquez S. Instituto de Patología Animal Facultad de Ciencias Veterinarias DESARROLLO DE U TEST DE ELISA SADWICH PARA DETECTAR CEPAS DE Flavobacterium psychrophilum Tesis de Grado presentada como parte de los requisitos para optar al grado de Licenciado en Bioquímica y Título Profesional de Bioquímico PAULA ADREA SATAA SEPÚLVEDA VALDIVIA – CHILE 2008 AGRADECIMIENTOS Me faltan palabras para agradecerle Dr. Alex Romero, por recibirme en su laboratorio, sus enseñanzas, apoyo y confianza. Muchas gracias. A Vania, Sra. Mónica y Don Esteban por la paciencia y la gran disponibilidad que han tenido conmigo. A mis compañeros de laboratorio Claudio R, Claudio A y René, por su buena onda y simpatía sobre todo en los momentos más difíciles. No cambien!! A mis pececitos Hans y Constanza, por su gran amor y paciencia. Ustedes son la luz de mi vida y la fuerza que me permite salir adelante cada día. Los amo. A mi mamá y papá por darme la oportunidad de estudiar y confiar en mí. Gracias por su apoyo y por estar siempre conmigo. Los quiero. A mis hermanos Leo y Pedro por todo el cariño, alegría y compañía. Los quiero. Esta tesis fue realizada gracias a apoyo del proyecto FONDECYT 1080571. “Cuando me equivoco me ayudan, cuando dudo me aconsejan y siempre que los llamo están a mi lado” A mis padres. i ÍNDICE DE CONTENIDOS Página 1. RESUMEN 1 SUMMARY 2 2. INTRODUCCIÓN 3. MATERIALES Y MÉTODOS 3.1 MATERIALES 3 19 19 3.1.1 Reactivos 19 3.1.2 Instrumentos 21 3.1.3 Cepas bacterianas 22 3.1.3.1 Cepas de Flavobacterium psychrophilum 22 3.1.3.2 Cepa de Escherichia coli 24 3.1.4 Oligonucleótidos 24 3.1.5 Animales de experimentación 24 3.1.6 Anticuerpos 25 3.1.6.1 Anticuerpo anti-proteínas de membrana de F. psychrophilum 25 3.1.6.2 Anticuerpo anti- F. psychrophilum completa 25 3.1.6.3 Anticuerpo anti- M13 25 3.1.6.4 Anticuerpo anti- F. psychrophilum 25 3.1.6.5 Anticuerpos recombinantes 68-97-125 25 ii 3.2 MÉTODOS 3.2.1 Cultivo de bacterias 26 26 3.2.1.1 Cultivo de F. psychrophilum 26 3.2.1.2 Cultivo de E. coli 26 3.2.2 Extracción de ADN genómico 27 3.2.3 Cuantificación de ADN 28 3.2.4 Reacción de PCR para la identificación de F. psychrophilum 28 3.2.5 Extracción de proteínas de membrana 29 3.2.6 Preparado de células lisadas de F. psychrophilum 29 3.2.7 Cuantificación de proteínas 30 3.2.8 Producción de anticuerpos policlonales anti-F. psychrophilum 30 3.2.9 Electroforesis en geles de poliacrilamida 31 3.2.10 Western blot 32 3.2.11 Ensayos de Dot blot 33 3.2.12 Inmunohistoquímica 34 3.2.13 Inmunofluorescencia 35 3.2.14 Evaluación de fagos 35 3.2.15 Amplificación y concentración de fagos 35 3.2.16 ELISA sandwich 36 3.2.17 Análisis estadístico 37 4. RESULTADOS 4.1 4.2 38 Caracterización de las cepas de F. psychrophilum 38 4.1.1 Identificación de F. psychrophilum mediante PCR 38 Producción de anticuerpos policlonales anti-F. psychrophilum 41 iii 4.2.1 Extracción de proteínas de membrana de F. psychrophilum 41 4.2.2 Titulación de los anticuerpos policlonales anti-F. psychrophilum producidos en conejo 43 4.2.3 Determinación del límite de detección de antígenos utilizando anticuerpos policlonales contra F. psychrophilum 4.3 43 Caracterización de los anticuerpos policlonales anti-proteínas de membrana de F. psychrophilum 46 4.3.1 Análisis de los patrones de proteínas inmunoreactivas de F. psychrophilum 46 4.3.2 Análisis de la reacción cruzada de los anticuerpos policlonales contra F. psychrophilum en órganos de Salmo salar y bacterias patógenas de peces 48 4.3.3 Inmunodetección de proteínas totales de distintos aislados de F. psychrophilum 51 4.3.4 Identificación de F. psychrophilum en peces infectados naturalmente con este patógeno 55 4.3.5 Inmunodetección en extendidos de cultivo del patógeno y lesiones de truchas arcoiris (O. mykiss) infectados naturalmente con F. psychrophilum 4.4 57 4.3.5.1 Inmunohistoquímica (IHQ) 57 4.3.5.2 Inmunofluorescencia (IFAT) 58 Evaluación de los anticuerpos recombinantes 62 iv 4.4.1 Inmunodetección de F. psychrophilum utilizando los anticuerpos recombinantes mediate Dot-blot 62 4.4.2 Inmunodetección de aislados de F. psychrophilum utilizando anticuerpos recombinantes mediante Dot-blot 4.4.3 Titulación anticuerpos recombinantes 4.5 64 64 Desarrollo de una prueba de ELISA sandwich para detectar F. psychrophilum 65 4.5.1 Análisis de la sensibilidad de la prueba de ELISA sandwich 65 4.5.2 Análisis de la especificidad de la prueba de ELISA sándwich 70 4.5.3 Inmunodetección de aislados de F. psychrophilum por el método de ELISA sandwich 73 5. DISCUSIÓN 75 6. BIBLIOGRAFÍA 85 v INDICE DE FIGURAS Página Figura 1. Características morfológicas de Flavobacterium psychrophilum Figura 2. Electroforesis en gel de agarosa al 1% del producto de PCR específico del gen 16S rRNA de Flavobacterium psychrophilum Figura 3. 45 Análisis de Western blot de los patrones de proteínas Inmunoreactivas Figura 7. 44 Análisis de la sensibilidad de los anticuerpos contra Flavobacterium psychrophilum mediante Dot blot Figura 6. 42 Titulación de los anticuerpos policlonales contra Flavobacterium psychrophilum mediante Dot blot Figura 5. 40 Electroforesis en gel de poliacrilamida al 12% de proteínas totales y de membrana de Flavobacterium psychrophilum Figura 4. 39 47 Análisis de Western blot de la reacción cruzada de los anticuerpos contra Flavobacterium psychrophilum con proteínas de órganos de Salmo salar Figura 8. 49 Análisis de Western blot de la reacción cruzada de los anticuerpos contra Flavobacterium psychrophilum con proteínas de bacterias patógenas de peces Figura 9. Electroforesis en gel de poliacrilamida al 12% de proteínas totales de aislados de Flavobacterium psychrophilum Figura 10. 50 Análisis de Western blot de la reacción de los anticuerpos policlonales contra proteínas totales de distintos aislados 53 vi de Flavobacterium psychrophilum Figura 11. Caracterización de Flavobacterium psychrophilum de lesiones de peces infectados naturalmente Figura 12. 54 56 Inmunodetección en extendidos de bacterias en cultivo e improntas de lesiones de peces infectados naturalmente con Flavobacterium psychrophilum mediante IHQ Figura 13. 59 Inmunodetección en extendidos de bacterias en cultivo e improntas de lesiones de peces infectados naturalmente con Flavobacterium psychrophilum mediante IHQ Figura 14. 60 Inmunodetección en extendidos de bacterias en cultivo y lesiones de peces infectados naturalmente con Flavobacterium psychrophilum mediante IFAT Figura 15. Análisis de Dot blot de los anticuerpos recombinantes contra Flavobacterium psychrophilum Figura 16. 61 63 Análisis de Dot blot de proteínas totales de aislados de Flavobacterium psychrophilum utilizando anticuerpos recombinantes 66 Figura 17. Titulación de los anticuerpos recombinantes en presencia de PM-1306 mediante ELISA sandwich Figura 18. 67 Titulación de los anticuerpos recombinantes en presencia de PM- 4605 mediante ELISA sandwich 68 Figura 19. Análisis de la sensibilidad de la prueba de ELISA sandwich 69 Figura 20. Análisis de la especificidad de la prueba de ELISA sandwich contra las proteínas totales de órganos de Salmo salar Figura 21. Análisis de la especificidad de la prueba de ELISA sandwich 71 vii contra las proteínas totales de bacterias patógenas de peces Figura 22. 72 ELISA sandwich para la detección de las proteínas totales de los distintos aislados de Flavobacterium psychrophilum 74 viii ÍNDICE DE TABLAS Página Tabla 1. Registro de enfermedades de salmónidos de importancia económica en Chile Tabla 2. Tabla 3. 4 Putativos precursores de proteasas de secreción de Flavobacterium psychrophilum 8 Origen de los aislados de Flavobacterium psychrophilum 23 ix LISTA DE ABREVIATURAS BSA : Albúmina sérica de bovino DAB : Diaminobencidina. ELISA : Enzimo inmuno ensayo FP : Flavobacterium psychrophilum IHQ : Inmunohistoquímica. IFAT : Inmunofluorescencia indirecta kDa : Kilo Dalton LB : Luria-Bertani PBS : Tampón fosfato salino PCR : Reacción en cadena de la polimerasa PEG : Polietilenglicol PMSF : Fosfometilsulfonilfosfato SDS : Dodecil sulfato de sodio. TEMED : N, N, N´, N´ - tetrametilendiamina Tris : Tris-(hidroximetil)-aminometano Triton X-100 : Octal fenoxi polietoxietanol Tween 20 : Polioxietilen (20) sorbitan monolaurato TYES : Triptona, extracto de levadura y sales TBS : Tampón tris salino TMB : 3,3’,5,5’ tetrametil bencidina 1 1. RESUMEN Flavobacterium psychrophilum es el agente etiológico de la “Enfermedad Bacteriana de Agua Fría” y el “Síndrome del Alevín de la Trucha Arco iris” que causa serias mortalidades de especies salmonídeas y no salmonídeas e importantes pérdidas económicas para la industria salmonera. Por lo tanto, es importante contar con técnicas diagnósticas que permitan la identificación temprana de esta bacteria. El objetivo del presente trabajo fue desarrollar una prueba de ELISA capaz de detectar este patógeno de peces. Se generaron anticuerpos policlonales inoculando conejos con proteínas de membrana y totales de la cepa 1306 y 4605. Estos anticuerpos presentaron una alta especificidad no presentando reacción cruzada con bacterias patógenas ni con órganos de importancia para el diagnóstico. Más aún estos fueron capaces de detectar 9 aislados de F. psychrophilum en estudio. Por otra parte, los anticuerpos recombinantes reconocieron las proteínas de membrana y totales de la mayoría de las cepas (1306, 4605 y los 9 aislados). Ambos anticuerpos, policlonales y recombinantes demostraron en el desarrollo de la prueba de ELISA sándwich, ser capaces de detectar de manera eficaz proteínas de F. psychrophilum. 2 SUMMARY Flavobacterium psychrophilum is the ethiological agent of Bacterial Cold-Water Disease” and “Rainbow Trout Fry Syndrome” that causes serious mortalities of species salmonid and non salmonid and substantial economic losses in salmonid aquaculture. Therefore, it is important to have diagnostic technics that allow the early identification of this bacteria. The aim of this study was to develop of ELISA test able to detect this pathogen of fish. Policlonal antibodies were generated inoculating rabbits with membrane proteins and total proteins of the strains 1306 and 4605. These antibodies presented a high specificity, not presenting cross reaction with other pathogenic bacterias neither with fish tissue of importance in the diagnosis. Even more these were capable to detect the nine isolated of F. psychrophilum studied here. On the other hand, the recombinant antibodies recognized total and membrane proteins and total of the strains (1306, 4605 and the nine isolated). Both policlonal, and recombinant antibodies demonstrated in the development of this ELISA sandwich test, to be able to detect in an effective way proteins of F. psychrophilum. 3 2. INTRODUCCION Hoy en día y considerando el aumento creciente que ha ido adquiriendo la salmonicultura en Chile, es importante poseer las herramientas necesarias para diagnosticar y prevenir a tiempo la aparición y diseminación de enfermedades que son la causa de altas mortalidades e impacto económico para la industria salmonera. Las condiciones intensivas en la producción de los centros de cultivo, los cambios en el medio ambiente marino y la baja rigurosidad en las medidas de prevención y control de enfermedades, permiten que enfermedades originadas por agentes infecciosos, principalmente bacterias y virus, tengan una mayor relevancia, ya que pueden ser transmitidas de un pez a otro. En Chile, el primer incidente documentado de enfermedades que afectaron a truchas y salmones lo realizó Wood en el año 1970, al registrar la presencia de una peligrosa enfermedad característica sólo de salmones, la Enfermedad Bacteriana del Riñón (BKD), la que se diseminó desde Río Blanco (V Región) a las pisciculturas de Polcura y Lautaro y a otras zonas donde se liberaron peces sobrevivientes, constituyendo éste, el primer registro de introducción y diseminación de una enfermedad exótica (Snyder, 1971). A continuación, de acuerdo al orden de aparición se muestran en la tabla 1 las enfermedades más importantes registradas hasta el año 2000 y que han tenido un serio efecto económico para la industria de cultivo de salmones: 4 Tabla 1. Registro de enfermedades de salmónidos de importancia económica en Chile Año de Enfermedades aparición 1970 1984 1989 1992 1993 1995 1995 2000 Enfermedad Bacteriana del Riñón (BKD). Provocada por la bacteria Renibacterium salmoninarum. Necrosis Pancreática Infecciosa (IPN). Autor y año de la publicación Wood, 1970 Mc Allister and Reyes, 1984 Septicemia rickettsial de salmones (SRS). Bravo Provocada por la bacteria Piscirickettsia salmonis. Campos, 1989. Enfermedad Entérica de la Boca Roja (ERMD) Toledo Provocada por la bacteria Yersinia ruckeri. al.,1992. Síndrome del Alevín de la Trucha Arcoiris. Bustos Provocada por la bacteria Flexibacter psychrophilus. 1995 Microsporidiosis Causada por el microsporidio Nucleospora salmonis. Furunculosis atípica. Causada por Aeromonas salmonicida atípica. and et et al., Bravo, 1996 Bravo, 2000 Mixosporidiosis Chacko et al., Provocada por Kudoa thyrsites. 2001 Fuente: FIP Nº 09/2001; “Técnicas de diagnóstico de enfermedades de salmónidos, mitilídos, pectinídos y ostreídos”. 5 Las enfermedades bacterianas son responsables de grandes mortalidades, especialmente las producidas por bacterias Gram negativas, las cuales pueden actuar como patógenos primarios o ser invasores oportunistas y/o secundarios, causando procesos patológicos en peces susceptibles (Monrás et al., 2003). Una de las bacterias más comunes presentes en salmones corresponden al género Flavobacterium. Este género posee un importante grupo de patógenos de peces de agua dulce, ampliamente diseminado en cultivos de salmónidos a través del mundo (Bernardet and Kerouault, 1989). Estas bacterias dan origen a diversas patologías en salmones, desde necrosis ulcerativa de la piel hasta infección sistémica afectando los principales órganos (Holt et al.,1993). Estos organismos presentan un amplio rango de virulencia y han sido denominados patógenos oportunistas de peces (Shotts and Teska, 1989). La habilidad de estos organismos para causar enfermedad aguda y crónica, y su natural presencia en el medio ambiente acuático, los caracteriza como el grupo de bacterias patógenas mas devastadores para los peces (Shotts and Starliper, 1999). Dentro de éstas, la especie de mayor importancia en salmónidos es Flavobacterium psychrophilum. Esta eubacteria Gram negativa afecta a todas las especies de peces salmónidos (Borg, 1960), siendo particularmente susceptibles salmón coho (Oncorhynchus kisutch), y trucha arcoiris (Oncorhynchus mykiss). Sin embargo, infecciones por F. psychrophilum han sido descritas en algunas especies de peces no salmónidos tal como anguila (Anguilla anguilla), carpa común (Cyprinus carpio) (Lehmann et al., 1991), y ayu (Plecoglossus altivelis) (Iida and Mizokami,1996). F. psychrophilum fue inicialmente aislado de riñón y lesiones externas de la enfermedad del salmón Coho juvenil, O. kisutch (Walbaum), en el estado de 6 Washington, USA y fue originalmente asignado como Cytophaga psychrophila (Borg, 1960). La patología de la enfermedad causada por este organismo ha sido descrita por Davis (1946), en trucha arcoiris juveniles O. mykiss (Walbaum), en la cual una característica lesión epidémica ocurrió sobre o cercana al pedúnculo, por lo cual se le denomino “Enfermedad del Pedúnculo”. Por otra parte, en infecciones de salmón coho se refirió a “Enfermedad Bacteriana de Agua Fría” y “Enfermedad a Baja Temperatura”, ya que la epizootología fue más prevalente en invierno y primavera cuando la temperatura del agua estuvo bajo los 10ºC (Borg, 1960; Holt, 1988). Asimismo, Rangdale (1995) la denominó como “Síndrome del alevín de la trucha arcoiris” (Rainbow Trout Fry Síndrome, RTFS), diagnosticándose en truchas arcoiris en incubadoras y balsas jaulas en los lagos de Chile desde el año 1993 (Rangdale, 1995). F. psychrophilum es un bacilo filamentoso, curvo y de bordes redondeados Gram negativo, flexible, débilmente refráctil y aerobio estricto (Holt, 1988; Rangdale, 1995; Matedoja et al., 2001). Tiene un diámetro de 5µm, una longitud entre 1,25 y 6,25µm (Pacha, 1968; Bernardet and Grimont, 1989), y además presentan movimiento deslizante (Bernardet and Kerouault, 1989; Schmidke and Carson, 1995). Para su aislamiento, los medios de cultivos que se utilizan son: agar MAO, agar Cytophaga, agar Shieh y TYES, siendo la temperatura óptima de cultivo de 15ºC por 48-96 horas (Holt et al.,1993). En estos medios de cultivo desarrolla colonias de pigmentación amarilla convexas, circulares y solevantadas, con un diámetro de 1-5 mm. Además no está capacitado para metabolizar carbohidratos (Pacha, 1968; Bernardet and Kerouault, 1989). Los factores más importantes que determinan la severidad de la enfermedad son la temperatura del agua y la virulencia de las cepas de F. psychrohpilum. Sin embargo, 7 los factores que determinan la virulencia de este patógeno, aún no están completamente dilucidados. Holt et al. (1989) demostraron que la temperatura del agua es de suma importancia en la expresión de la patogenicidad de ésta bacteria. En truchas arcoiris (O. mykiss), salmones coho (O. kisutch), y chinook (Oncorhynchus tshawytscha), infectados experimentalmente con F. psychrophilum, se observaron que las más altas mortalidades ocurrieron a bajas temperaturas del agua (3-15ºC). La mortalidad bajó al incrementarse la temperatura por sobre los 15ºC, no observándose mortalidades por sobre los 23ºC. Estos autores, sugieren que el efecto que tiene la temperatura sobre la mortalidad, se debe en parte, a su efecto sobre el crecimiento de la bacteria dentro de los tejidos del huésped. La temperatura además podría tener un efecto sobre la producción de exoproteasas y otros posibles determinantes de virulencia, tal como el factor leucocitolítico ya descrito para otros patógenos bacterianos (Fuller et al., 1977). La virulencia de los diferentes aislados de F. psychrophilum puede variar enormemente (Holt et al., 1993; Madsen and Dalsgaard, 2000; Nematollahi et al., 2003). Varios factores de virulencia incluyendo proteasas, exotoxinas, endotoxinas y adesinas contribuyen a la patogenicidad de F. psychrophilum (Dalsgaard, 1993; Bertolini et al., 1994; Secades et al., 2001, 2003). Dalsgaard (1993) demostró que F. psychrophilum es activamente proteolítico y que esta naturaleza proteolítica juega un importante rol en la patogenicidad de las cepas. Duchaud et al. (2007) secuenciaron el genoma completo de F. psychrophilum. Estos autores indicaron que el genoma de este patógeno de peces, codifica para 13 proteasas putativas de secreción probablemente implicadas en la virulencia y/o en la destrucción de los tejidos del hospedero (Tabla 2). 8 Tabla 2. Putativos precursores de proteasas de secreción de Flavobacterium psychrophilum (Duchaud et al. 2007) Locus Gen FP0081 FP0082 Familia Producto M50 Probable precursor de la familia M50 de una metaloproteasa de zinc asociada a membrana M1 Probable precursor de la familia M1 de una metaloproteasa Probable precursor de la familia M13 de una metaloproteasa Pep0 FP0086 Pep0 M13 FP0231 Fpp1 No asignado FP0232 Fpp2 M43 precursor de una metaloproteasa psicrofilica Fpp1 precursor de una metaloproteasa psicrofilica Fpp2 FP0280 M36 Probable precursor de la familia fungalisina M36 de una metaloproteasa FP0281 M36 Probable precursor de la familia fungalisina M36 de una metaloproteasa FP1024 M43 Probable precursor de la familia citofagalisina de una metaloproteasa FP1619 M43 Probable precursor de la familia citofagalisina de una metaloproteasa FP1763 S8 Probable precursor de la familia subtilisina S8 de una serina endopeptidasa FP1777+ FP1776 M43 Precursor de una colagenasa truncada FP2364 M48 Probable precursor de la familia M48 de una metaloproteasa de zinc asociada a membrana S8 Probable precursor de la familia subtilisina S8 de una serina endopeptidasa FP2396 9 Dos proteasas extracelulares fueron caracterizadas bioquímica e inmunológicamente: la metaloproteasa Fpp1 (FP0231; Secades et al., 2001) y Fpp2 (FP0232; Secades et al., 2003). En el año 2001 Secades caracterizó una metaloproteasa psicrofílica de 55kDa (Fpp1), que depende de las concentraciones de ión calcio para su actividad y estabilidad térmica. Adicionalmente, estroncio y bario también pueden activar esta proteína. Sobre la base de sus características bioquímicas, Fpp1 fue incluida en el grupo de las metaloproteasas que tienen un pH óptimo de 6,5 para su actividad. Además esta enzima demostró ser más activa entre los 25ºC y los 40ºC. Sin embargo, su actividad disminuyó rápidamente a los 45ºC. Esta proteasa hidroliza gelatina, laminina fibronectina, fibrinógeno, colágeno tipo IV, y, en menor grado colágeno tipo I y II. Fpp1 también degrada actina y miosina, elementos básicos del sistema muscular de los peces. Secades (2003), caracterizó otra metaloproteasa, Fpp2, la cual tiene una masa molecular estimada de 62kDa con el ión calcio unido a la molécula, el cual juega un rol importante en la termoestabilidad de la enzima. Su actividad proteolítica es óptima a pH 6,0-7,0 y 24ºC, siendo inactiva a temperaturas por sobre los 42ºC. En condiciones óptimas la enzima es capaz de hidrolizar la matriz extracelular y proteínas musculares. Adicionalmente a estas dos metaloproteasas, Bertolini et al. (1994) encontraron que F. psychrophilum produjo 2 proteasas bacterianas (114 y 152kDa), las que degradaban caseína y gelatina, y otras ocho proteasas (32 a 86kDa), que degradaban gelatina no así caseína. Aunque se encontraron algunas relaciones entre la composición de las proteasas y la virulencia de los aislados, la correlación no fue absoluta. 10 Otros importantes factores de virulencia son las toxinas citolíticas tal como las hemolisinas bacterianas. Lorenzen (1994), sugirió que la anemia desarrollada por alevines de truchas arcoiris infectados con F. psychrophilum se debió en cierto grado a la capacidad que tiene esta bacteria de lisar y aglutinar parcialmente los eritrocitos de truchas arcoiris. Esto fue demostrado más tarde por Lorenzen et al. (1997).Por otra parte, se ha demostrado que en otros patógenos de peces, como Vibrio anguillarum, la toxina VAH5 es capaz de lisar los eritrocitos de trucha arcoiris (Rodkhum et al., 2005). En F. psychrophilum el gen FP0063 codifica una proteína con un 53% de homología a VAH5, la cual podría cumplir un rol similar en la patogenicidad y cooperar con la secreción de proteasas para la destrucción del tejido (Duchaud et al., 2007). Por otra parte, la familia de toxinas formadoras de poros, citolisinas activadas por tiol (thiol-activated cytolysin, TACYs), han sido implicadas en la patogenicidad de varias bacterias Gram positivas. Estas TACYs son capaces de gatillar varias vías de señalización en una varidad de tipos celulares del hospedero, como se muestra para listeriolisina O (LLO), y neumolisina. En F. psychrophilum, el gen FP0097 codifica una proteína que presenta dominios similares a los descritos en TACYs, lo cual sugiere que la proteína FP0097 puede funcionar como una modulina bacteriana, ya que inducen la liberación de citocinas, que a su vez dañan los tejidos (Duchaud et al., 2007). Varias moléculas de la superficie de F. psychrophilum, incluyendo lipopolisacáridos (LPS), han sido implicadas en su patogénesis e identificados como potencial vacuna y/o como macromoléculas candidatos para el diagnóstico (MacLean et al., 2001). Estudios previos han sugerido que el componente O de los LPS de F. psychrophilum es altamente inmunogénico y que puede estar relacionado con la 11 respuesta inmune protectiva en trucha arcoiris (Crump et al., 2001; Rahman et al., 2002; LaFrentz et al., 2004). Crump et al. (2001) mediante análisis de Western blot de células tratadas con proteinasa K, encontraron que F. psychrophilum exhibe LPS que comprenden oligosacáridos de baja masa molecular y polímeros que contienen el antígeno-O de alta masa molecular. Estos autores identificaron un antígeno de 16kDa aproximadamente, que correspondería a un LPS de baja masa molecular el cual es probablemente el componente predominante de la envoltura celular gruesa de esta bacteria, ya que fue encontrado en abundancia en el medio de cultivo. Adicionalmente Crump et al. (2001) identificaron LPS con cadenas de polisacáridos O de alta masa molecular, con una masa molecular de aproximadamente 70 a 200Kda. Análisis de la composición del LPS de F. psychrophilum reveló una cadena O compuesta por una repetición de trisacáridos, conteniendo un inusual azúcar (N-acilado bacillosamina), que podría ser único para esta bacteria y por tanto, ser utilizado como molécula blanco para fines de diagnóstico. Además de las toxinas bacterianas, exoenzimas y macromoléculas asociadas a membrana externa, componentes asociados con la adhesión también deben ser consideradas como importantes factores de virulencia. Un primer requisito para el éxito de la colonización del tejido del hospedero, es la capacidad de adherirse. Componentes de la superficie bacteriana como fimbrias, pili, cápsula y capa superficial han demostrado estar asociadas con la adhesión a la superficie, así como la iniciación y progresión de varias enfermedades infecciosas. En este contexto, se ha demostrado que F. psychrophilum posee una delgada capa (Dalsgaard, 1993; Rangdale, 1995). En consecuencia, la superficie celular bacteriana debe ser considerada como una suave 12 capa polielectrolita que permite el flujo de fluidos a través de una capa de cargas fijas. La presencia de esta capa suave disminuye la barrera de energía a causa de la repulsión electrostática en la interacción de los organismos con las células diana y, por consiguiente, deben desempeñar un papel importante en la adhesión inespecífica (Morisaki et al., 1999). Kondo et al. (2002) reportaron que F. psychrophilum tiene fuertes propiedades adhesivas a la superficie de los peces, branquias y ovas. Duchaud et al. (2007) identificaron en el genoma de F. psychrophilum 27 genes probablemente involucrados en la adhesión bacteriana. Quince de ellos están organizados en tandem y codifican proteínas con repeticiones ricas en leucina similares a BspA y LrrA de la bacteria periodontopatogénica Bacteroides forsythus y Treponema denticola, respectivamente. Estas proteínas inmunogénicas de la superficie celular se fijan fuertemente a los componentes de la matriz extracelular y juegan un rol en la unión al tejido oral humano. Estos autores plantean que probablemente otras proteínas implicadas en la adhesión son las siguientes: (i) FP2413, que contiene cinco dominios fibronectina tipo III que participan en la unión a la superficie celular y un dominio CUB que se encuentran en las proteínas extracelulares de espermadesina y en familias peptidasas eucariotas, y (ii) FP1830, FP0016 y FP0595, las cuales contienen dominios fibronectina tipo III. Las proteínas de la superficie celular de F. psychrophilum son importantes blancos del sistema inmunológico del hospedero. Mediante SDS-PAGE e inmunodetección se identificaron antígenos reconocidos por el hospedero como OmpH/P18 (FP2098; Massias et al., 2004; Dumetz et al., 2006), OmpA/P60 (FP0156; Merle et al., 2003; Dumetz et al., 2007), y la proteína antigénica específica para esta bacteria (FspA), (FP2019; Crump et al., 2005). Todos ellos están probablemente 13 implicados en las interacciones bacteria-hospedero, ya que se incluyen en un conjunto de proteínas de F. psychrophilum antigénicas reconocidas principalmente por antisueros de peces convalescientes que fueron naturalmente infectados con este patógeno. Duchaud et al. (2007) identificaron 35 proteínas que contienen un péptido señal y una región conservada C-terminal de 70 aminoácidos. Numerosas búsquedas en las bases de datos identificaron 170 proteínas que contienen esta región conservada C-terminal exclusivamente en los miembros del phylum Bacteroidetes. Por lo tanto, los miembros de este phylum contienen el motivo C-terminal [YPNPX21-23 (N / D) X2GX18-27GXY] que probablemente direcciona a las proteínas a ser expuestas en la superficie bacteriana. Y algunas de éstas podrían ser de importancia para la virulencia. La identificación de F. psychrophilum y el diagnóstico de la enfermedad que este patógeno causa son tradicionalmente basados en los signos clínicos, seguido por el aislamiento en el clásico cultivo en agar y análisis taxonómicos (Bernardet et al., 1996; Daskalov et al., 1999). Estos métodos consumen tiempo y no diferencian las distintas cepas. Por lo tanto, métodos más sensibles y específicos de identificación son necesarios para posibilitar el diagnóstico exacto y temprano de la enfermedad. Existen métodos para detectar este patógeno los cuales incluyen técnicas moleculares tales como, polimorfismo del largo de los fragmentos de restricción (RFLP), (Nilson et al., 2000), Hibridación in situ (Liu et al., 2001), y la reacción en cadena de la polimerasa (PCR), (Toyama et al., 1994; Izumi and Wakabayashi, 1997; Cepeda and Santos, 2000), las cuales son consideradas particularmente ventajosas para la diferenciación en análisis epizootiológicos, además de ser capaces de detectar bajas cantidades del patógeno. Las técnicas inmunológicas incluyen, inmunofluorescencia la cual ha provisto 14 una efectiva detección del patógeno en improntas de bazo de peces enfermos. Sin embargo, la absorción del antisuero de F. psychrophilum con células de F. columnare puede ser necesaria para evitar reacciones cruzadas con este patógeno estrechamente relacionado (Lorensen and Karas, 1992; Amita et al., 2000). También se encuentran dentro de estas técnicas, el ensayo peroxidasa (Rangdale and Way, 1995; Aikawa, 1998), y el ensayo inmunosorbente acoplado a enzima (ELISA), que tiene una sensibilidad de 1x104 células por ml (Mata and Santos, 2001). La prueba de ELISA es una aplicación básica usada para analizar antígenos solubles (Hornbeck, 1991). Este técnica permite el procesamiento simultáneo de muchas muestras. Asimismo es capaz de detectar y cuantificar sustancias tales como péptidos, hormonas, proteínas y anticuerpos. El elemento crucial de la detección es la interacción antígeno-anticuerpo, en donde se utilizan anticuerpos que pueden ser policlonales o monoclonales. Ambos anticuerpos se obtienen a partir de la inmunización de animales de laboratorio con un antígeno específico. La diferencia entre estos radica en que los anticuerpos policlonales se originan a partir de la síntesis de numerosos clones de linfocitos B, y cada uno con diferente especificidad y sensibilidad a epítopes del antígeno. En cambio, los anticuerpos monoclonales son generados a través de la fusión de las células productoras de anticuerpos, linfocitos B y células de mieloma para formar un hibridoma. Estos últimos a diferencia de los anticuerpos policlonales, tienen la desventaja del tiempo y alto costo para producirlos y mantenerlos (Lipman et al., 2005). Actualmente se están utilizando nuevas tecnologías, como la de ADN recombinante, para la producción de anticuerpos en menor tiempo y costo, sin perder la capacidad de multiplicarse indefinidamente como tampoco la afinidad ni especificidad 15 frente a un antígeno específico. Una de las estrategias que tiene una gran aplicabilidad es la metodología de “Phage display”. El “Phage display” es el término universal que se utiliza para describir una novedosa, poderosa y versátil técnica de bioselección in vitro en la cual un péptido o proteína es fusionada genéticamente a una de las proteínas de superficie de un bacteriófago o fago filamentoso (Smith,1985). Esta fusión resulta en el despliegue del péptido o proteína en el exterior del fago mientras que el ADN que codifica para la fusión reside dentro del mismo fago. Esta fusión da cuenta de la relativa sencillez de la manipulación in vitro e in vivo de los fagos, permiten una rápida generación de moléculas ligando desplegados en los fagos con altas afinidades de unión, a muy bajo costo y en poco tiempo (Smith, 1985; Smith and Petrenko, 1997). Estas importantes características ubican a esta tecnología por sobre otras técnicas para el desarrollo de biomoléculas utilizadas en diagnóstico. En los últimos años la tecnología de obtención de fragmentos de anticuerpos expuestos en la superficie de fagos filamentosos se ha expandido rápidamente, y es muy utilizada como alternativa para obtener estas moléculas en lugar de los anticuerpos monoclonales. Esta tecnología, como se menciono anteriormente, explota la capacidad de los fagos filamentosos de presentar en su superficie proteínas o fragmentos de las mismas, fusionadas a proteínas de la cápside de la particula viral. La fusión no afecta la integridad o propiedades biológicas del fago (por ejemplo, infectividad), y se produce un acoplamiento entre los fagos portadores de la proteína de fusión y el gen codificante para dicha proteína (Vispo, 2004). Los fagos filamentosos principalmente estudiados son el fl, fd y M13, los cuales comparten 98% de homología en su genoma. El fago M13 es el más comúnmente 16 utilizado en los procesos de “Phage display”. El ADN del fago se encuentra empaquetado en un cilindro flexible de proteínas. Estas proteínas han sido denominadas: proteína pVIII (proteína principal de la cápside del fago), las proteínas pVII y pXI (con 5 copias en cada uno de los extremos del fago), y las proteínas pIII y pVI (con 4 o 5 copias cada una expresadas en el otro extremo del fago). Estos fagos necesitan de bacterias que expresen pili sexual, ya que la interacción de esta estructura con la proteína menor de cubierta del fago permite el ingreso de este virus a la célula bacteriana comenzando así la síntesis de los anticuerpos recombinantes (Lubrowski et al., 1999; Nilsson et al., 2000). Posterior a los pasos de infección, replicación, amplificación y purificación, las partículas de fagos son sometidas a un proceso de bioselección o “biopannig” donde se prueba la afinidad de unión y reconocimiento de los fagos hacia moléculas blanco. El uso de anticuerpos recombinantes trae consigo varias ventajas como ser generados económicamente, ya que grandes cantidades de ScFv son producidos en sistemas bacterianos (Kipriyanov et al., 1997), como Escherichia coli (Fuchs et al., 1991; Nilsson et al., 2000), son simples de aislar y no requieren procedimientos complejos de plegamiento (Hashimoto et al., 1999; Sanchez et al., 1999), y por último, la generación de ScFvs sirve para minimizar problemas asociados con la pérdida de especificidad al antígeno debido al incorrecto plegamiento (Jager and Pluckthun, 1997), ya que el reducido tamaño de un ScFv disminuye drásticamente el número de conformaciones de potenciales anticuerpos. En nuestro laboratorio contamos con anticuerpos recombinantes analizados mediante ELISA e IFAT por Buttcovich (2005). Buttcovich, identificó 3 anticuerpos recombinantes específicos para F. psychrophilum 68, 97 y 125. La utilización de estos 17 anticuerpos recombinantes con las técnicas inmunológicas mencionadas mostraron reproducibilidad, especificidad y menores costos que el empleo de técnicas moleculares. Aunque se han descrito en estudios tempranos el desarrollo de pruebas de ELISA sandwich utilizando anticuerpos policlonales (Rangdale and Way, 1995; Lorenzen and Olesen, 1997; Aikawa, 1998; Mata and Santos, 2001), a la fecha no se ha desarrollado un test de ELISA sandwich utilizando anticuerpos recombinantes y policlonales para detectar eficazmente al patógeno de peces F. psychrophilum con el fin de lograr mayor objetividad, sensibilidad y especificidad. 18 HIPÓTESIS Es posible detectar aislados chilenos de Flavobacterium psychrophilum utilizando anticuerpos policlonales y recombinantes por medio de un test de ELISA sandwich. OBJETIVO GENERAL Desarrollar un test de ELISA capaz de detectar aislados chilenos de Flavobacterium psychrophilum. OBJETIVOS ESPECÍFICOS - Producir anticuerpos policlonales en conejos contra las proteínas de membrana y la bacteria completa de F. psychrophilum. - Caracterizar los anticuerpos policlonales producidos mediante Western blot, Inmunohistoquímica e Inmunofluorescencia. - Evaluar los anticuerpos recombinantes mediante Dot blot y ELISA. - Desarrollar un test de ELISA sandwich que permita la identificación de chilenos de F. psychrophilum en salmónidos. aislados 19 3. MATERIALES Y MÉTODOS 3.1 MATERIALES 3.1.1 Reactivos -Aceite de inmersión: Merck -Ácido acético: Equilab -Ácido clorhídrico: Merck -Ácido sulfurico: Merck -Acrilamida-Bisacrilamida: Winkler -Agar Agar: Merck -Agarosa: Winkler -Albúmina de suero bovino: Sigma -Anticuerpo anti IgG conejo: SAPU -Anticuerpo anti IgG ratón: SAPU -Anticuerpo anti IgG conejo, acoplado al fluoróforo Alexa Fluor 488: Molecular probes. -Anticuerpo anti IgG conejo conjugado a peroxidasa: Dako. -Azul de bromofenol: Sigma. -Azul de Coomasie R-250: Bio-rad -Bálsamo de Canadá: Winkler -β-Mercaptoetanol: Merck -Bromuro de etidio: Winkler -Cloruro de sodio: Merck -Cloruro de potasio: J.T. Baker -Cloruro de calcio: Merck 20 -Coadjudante de Freund completo: Sigma -Coadjudante de Freund incompleto: Sigma -Di-sodio dihidrógeno fosfato dihidrato: Merck -DAB: Sigma -Etanol: Merck -Extracto de levadura: BD (Becton, Dickinson and company) -Fluorescent mounting medium: Dako. -Glicerol: Merck -Glicina: Winkler -Glucosa: Merck -Hematoxilina: Sigma -Isopropanol: Merck -Leche descremada: Calo -Marcador de peso molecular rango amplio, preteñido: Winkler -Metanol: Equilab --PMSF: Sigma -Perhidrol: Merck -Persulfato de amonio: Gibco BRL -Potasio dihidrógeno fosfato: Merck -SDS: J.T. Baker -Sulfato de magnesio: Merck -Tampón Carbonato-Bicarbonato: Sigma -Temed: Gibco BRL -TMB-ELISA: Pierce 21 -Triptona: BD (Becton, Dickinson and company) -Tris base: Winkler -Triton x-100: Promega -Tween-20: Sigma -Xilol: Arquimed 3.1.2 Instrumentos Agitador Gyro-Rocker Stuart Scientific Balanza, MP-300 G Chyo Cabina de flujo laminar Captair Cruma Cámara de Bioseguridad clase II tipo A Factomet Centrífuga refrigerada Heraeus Biofuge fresco. Centrífuga Rotina 35R Hettich Espectrofotómetro Spectronic Génesis 8 Filtros de 0,2 µm, Orange Scientific Fuente de poder, Power PAC 1000, Biorad. Incubador con agitación ZHWY-100B Zticheng Lector de microplaca ELx800 Bio-Tek Microscopio CH30RF200 Olympus optical pHmetro Extech Refrigerador incubador FOC 225E Velp Scientifica Sistema de electroforesis Mini Protean II, BioRad Sistema Mini protean II, BioRad. Sistemas de transferencia Transblot Semy dry transfer cell, BioRad 22 Sonicador Microson Ultrasonic Cell Disruptor XL Termocilador Gene amp PCR system 2400 Perkin Elmer Vortex V-1 Boeco Germany 3.1.3 Cepas bacterianas 3.1.3.1 Cepas de Flavobacterium psychrophilum Para la producción de anticuerpos policlonales en conejos, se utilizaron 2 cepas de F. psychrophilum. Ambas cepas fueron confirmadas como F. psychrophilum por serología y PCR. La cepa de campo se denominó 4605 y la cepa de referencia ATCC 49411, se denominó 1306. Ambas cepas fueron aisladas de riñón de alevín de trucha arcoiris (Oncorhynchus mykiss) y proporcionadas por el Laboratorio de Biotecnología y Patología Acuática del Instituto de Patología Animal. Para las pruebas de inmunodetección, se utilizaron 9 aislados de F. psychrophilum mantenidas en el Laboratorio de Biotecnología y Patología Acuática del Instituto de Patología Animal. Estos aislados fueron confirmados por PCR por Figueroa (2008). Estas cepas fueron obtenidas de pisciculturas ubicadas en distintas áreas del sur de Chile. El órgano y hospedero desde donde se aislaron las muestras bacterianas, se presentan en la tabla 3. 23 Tabla 3. Origen de los aislados de Flavobacterium psychrophilum NÚMERO UBICACIÓN HOSPEDERO PROCEDENCIA 14 Piel Trucha Estuario 15 Riñón Trucha Vertiente 16 Bazo Trucha Estuario 25 Riñón Salmón del atlántico Sin información 27 Riñón Salmón del atlántico Sin información 31 Riñón Trucha Estuario 40 Branquia Salmón del atlántico Piscicultura 41 Bazo Trucha Estuario 46 Riñón Trucha Vertiente 24 3.1.3.2 Cepa de Escherichia coli Para la amplificación de los anticuerpos recombinantes 68, 97 y 125 se utilizó la cepa de E. coli ER2537, comercialmente disponible en New England Biolabs, la cual es una robusta cepa F+ con una rápida tasa de crecimiento y es especialmente adecuada para la propagación del bacteriófago M13. 3.1.4 Oligonucleótidos Se utilizaron partidores específicos para la reacción de amplificación del gen ARN ribosomal 16S de F. psychrophilum designados como PSY1 y PSY2, ambos desarrollados por Toyama et al. (1994), los que amplifican un fragmento de 1.100bp aproximadamente. PSY1: 5’- CGATCCTACTTGCGTAG-3’ Forward PSY2: 5’- GTTGGCATCAACACACT-3’ Reverse 3.1.5 Animales de experimentación Para la producción de anticuerpos policlonales, se utilizaron 3 conejos adultos del mismo sexo, los cuales fueron inyectados con proteínas de membrana extraídas de la cepa 13-06 y 46-05 y con la bacteria completa de la cepa 1306. Los conejos fueron mantenidos en un cuarto aislado del contacto con otros patógenos. Esto se llevó a cabo por un período de 3 meses. 25 3.1.6 Anticuerpos 3.1.6.1 Anticuerpo anti-proteínas de membrana de F. psychrophilum Este fue producido en el Laboratorio de Biotecnología y Patología Acuática de acuerdo a procedimientos que se detallan en métodos. 3.1.6.2 Anticuerpo anti-F. psychrophilum completa Este fue producido en el mismo Laboratorio de acuerdo a procedimientos que se detallan en métodos. 3.1.6.3 Anticuerpo anti- M13 Este es un anticuerpo monoclonal anti-M13 pIII (isotipo IgG2a de ratón), adquirido comercialmente de New England Biolab 3.1.6.4 Anticuerpo anti-F. psychrophilum Este es un anticuerpo policlonal anti-F. psychrophilum hecho en conejo, donado por el Dr. Hugo Folch del Instituto de Inmunología, Facultad de Medicina de la Universidad Austral de Chile. 3.1.6.5 Anticuerpos recombinantes 68 - 97 - 125 Son anticuerpos recombinantes caracterizados por Buttcovich (2005). específicos contra F. psychrophilum, 26 3.2 MÉTODOS 3.2.1 Cultivo de bacterias 3.2.1.1 Cultivo de F. psychrophilum Las cepas de F. psychrophilum fueron mantenidas a – 80 ºC. Para su cultivo, se descongelaron y resuspendieron en caldo TYES (triptona 0,4%, extracto de levadura 0,04%, MgSO4 0,05% y CaCl2 0,05%; pH 7,2), suplementado con ramnosa 0,5%. Estas se incubaron durante 72 horas a 18ºC. Para la obtención de colonias aisladas estas fueron sembradas en agar TYES (caldo TYES y agar 1,5%), suplementado con leche 0,25% y ramnosa 0,5% durante el mismo tiempo y temperatura. Se verificó su pureza mediante tinción de Gram. 3.2.1.2 Cultivo de E. coli La cepa de E. coli ER2537 también fue mantenida a – 80 ºC. Para su cultivo, se descongeló y resuspendió en caldo LB (triptona 1%, extracto de levadura 0,5% y NaCl 0,5%; pH 7,4), y se incubó a 37ºC con agitación constante a 200 rpm durante toda la noche. Para la obtención de colonias aisladas, las bacterias fueron sembradas en agar mínimo M9 (Na2HPO4 1,2%, KH2PO4 0,6%, NaCl 0,1% NH4Cl 0,2%, agar 3%, glucosa 20%, MgSO4 1M, CaCl2 1M y tiamina 10mg/ml; pH 7,4), bajo las mismas condiciones anteriores. Se verificó su pureza mediante tinción de Gram. 27 3.2.2 Extracción de ADN genómico El ADN fue extraído de cepas y muestras de tejidos, usando el Kit “DNeasy Blood and Tissue Kit” (Qiagen). Para tal efecto, en el caso de tejido, este sé resuspendió en tampón fosfato salino 1X (PBS 1X: NaH2PO4 50 mM y NaCl 150 mM; pH 7,4), y se homogenizó por vortex. Luego se procedió a centrifugar por 10 minutos a 5000 x g a 4ºC. En el caso de la suspensión bacteriana, este último paso se realizó de inmediato y se siguió con el mismo procedimiento en ambos casos, como se indica a continuación. El sobrenadante fue descartado y el sedimento bacteriano sé resuspendió en 180 µl de tampón ATL y posteriormente se añadió 20 µl de proteinasa K. La mezcla se agitó en vortex y se incubó por 1 hora a 56ºC hasta la lisis total. Ocasionalmente se mezcló con vortex durante la incubación para dispersar la muestra. Luego se mezcló por inversión unas 10 veces. Se añadió 200µl de tampón AL a la muestra y se mezcló con vortex. Luego se añadieron 200µl de etanol absoluto y se mezcló nuevamente con vortex. La mezcla se transfirió a una columna de Mini spin Dneasy (aportadas por el kit), contenida en un tubo colector de 2ml y se centrifugó a 6000 x g por 1 min. El filtrado fue descartado. La columna de Mini spin fue puesta en un nuevo tubo colector de 2ml y el ADN contenido en la matriz se lavó con 500µl de tampón AW1 y AW2 respectivamente. En cada caso se centrifugó a 6000 x g por 1 min. Finalmente, la columna de mini spin Dneasy se colocó en un tubo eppendorf de 1,5ml y se agregó 200µl de tampón AE directamente a la membrana. Se incubó a temperatura ambiente por 1 min. y se centrifugó a 6000 x g por 1 min. para eluir el ADN. El ADN obtenido, se almacenó a -20ºC hasta el momento de su utilización en las reacciones de PCR. 28 3.2.3 Cuantificación de ADN Para determinar la concentración estimada de ADN, se midió la absorbancia a una longitud de onda de 260nm por espectrofotometría. La pureza del ADN fue determinada mediante la razón de los valores de absorbancia medidos a 260nm y 280nm (A260/A280). Una preparación pura de ADN tiene un valor de 1,8. La concentración de las muestras se calculó usando la relación en donde 1 unidad de absorbancia es igual a 50µg/ml para ADN de doble hebra (Sambrook et al., 1989). 3.2.4 Reacción de PCR para la identificación de F. psychrophilum La identificación de F. psychrophilum se realizó siguiendo el protocolo de rutina descrito en el Laboratorio de Biotecnología y Patología Acuática y según lo descrito por Toyama et al. (1994). La reacción de PCR se llevó a cabo en un volumen final de 25µl utilizando: 12.3µl de agua ultra pura, 5 µl de GreenBuffer (Promega) PCR 5x, 3µl MgCl2 (Promega) 25 Mm, 1µl dNTPs 10Mm c/u, 0.5µl primer PSY1 50 pmoles/µl, 0.5µl primer PSY2 50 pmoles/µl, 0.2µl GoTaq polimerasa (Promega) 5U/µl, y 2.5µl DNA templado 100ng aproximadamente. La reacción de amplificación consistió en una denaturación inicial a 94ºC por 5 minutos; 30 ciclos, denaturación a 94º C por 2 minutos, apareamiento a 45ºC por 1 minuto y medio, extensión a 72ºC por 2 minutos y extensión final a 72º C por 5 minutos. Los fragmentos obtenidos fueron visualizados en un gel de agarosa al 1%, con tinción de Bromuro de etidio y exposición a la luz UV 254 nm. 29 3.2.5 Extracción de proteínas de membrana El aislamiento de proteínas de la superficie de F. psychrophilum se realizó según lo descrito por Massias et al. (2004) con algunas modificaciones. Una vez que el cultivo de bacterias alcanzó una A600: 0.6, 100ml de cultivo fueron cosechado por centrifugación a 6000 x g por 10min a 4ºC. Las células cosechadas fueron lavadas dos veces con tampón fosfato salino (PBS1X), y posteriormente, el sedimento bacteriano fue resuspendido en 10ml de Tris-HCl 0,0625 M, pH 6,8 en presencia de 0.4Mm de PMSF y sonicados (Sonicador Microson Ultrasonic Cell Disruptor XL), en hielo (80watt), por 2min. a intervalos de 20seg, con 40 seg. de enfriamiento entre los intervalos. El sonicado crudo se examinó por microscopia para determinar si la lisis celular ocurrió durante este tratamiento. Posteriormente el sonicado fue centrifugado por 6000 x g por 20min a 4ºC. El sobrenadante fue centrifugado a 16.000 x g por 35 min. a 4ºC. El sedimento de esta centrifugación se resuspendió en 100µl de Tris-HCl 0,0625 M, pH 6,8. Esta fracción insoluble o de membrana fue almacenada a –20ºC hasta su utilización. 3.2.6 Preparado de células lisadas de F. psychrophilum El cultivo bacteriano, en la etapa de crecimiento exponencial, fue centrifugado a 6000 x g por 10min a 4ºC. Las células cosechadas fueron resuspendidas en Tris-HCl 0,0625 M, pH 6,8 en presencia de 0.4Mm de PMSF y sonicadas en hielo, como se indicó anteriormente. Luego fueron centrifugadas a 3000 x g por 5min para remover las células intactas. El lisado celular fue almacenado a –20ºC. 30 3.2.7 Cuantificación de proteínas Basado en el método de Bradford (Bradford et al, 1976), se fabricó una curva de calibración de 4 a 20 µg de proteínas por ml de reactivo, utilizando BSA en agua destilada como estándar. La lectura se realizó después de 10 min. en un espectrofotómetro (Spectronic, Génesis 8), a una longitud de onda de 595 nm. Para determinar la concentración de proteínas de las muestras, se realizaron diluciones determinadas y luego se les agregó el reactivo; Las muestras se incubaron por 10 min. a temperatura ambiente. La absorbancia fue leída a la misma longitud de onda y su valor fue interpolado en la curva de calibración. Todas las lecturas se realizaron contra su respectivo blanco, en este caso se utilizó agua destilada, (que también se usó para las diluciones de las muestras), con el reactivo de Bradford. 3.2.8 Producción de anticuerpos policlonales anti- F. psychrophilum Se generaron anticuerpos policlonales contra proteínas de membrana y bacteria completa de F. psychrophilum de la cepa 1306 y 4605. Para esto se utilizaron 3 conejos, los que fueron inmunizados por vía subcutánea en el lomo. Se realizaron sangrías periódicas para evaluar el título de los anticuerpos séricos mediante immunodetección en gota (Dot Blot). El proceso tardó 3 meses. La primera inyección consistió de 0,5mg de antígeno disuelto en 0,5ml de solución salina (PBS 1X), y emulsionada con 0,5ml de coadyuvante completo de Freund. Las tres inyecciones suplementarias se efectuaron después de tres semanas, luego de la primera inmunización con intervalos de tres semanas entre cada una de 31 ellas. Todas se realizaron con 0,5mg de antígeno disuelto en 0,5ml de solución salina y emulsionada con 0,5ml de coadyuvante incompleto de Freund. Antes de comenzar la inmunización se obtuvo el suero para el control preinmune y entre cada período de inyección se realizaron sangrías para obtener el suero inmune a modo de control. Al finalizar el período de inmunización se colectó la sangre de los 3 conejos en tubos Falcon y se dejo por 3 horas a 37ºC para que coagule, luego se desprendió el coagulo y el suero obtenido se centrifugó por 5 min. a 3000 rpm, obteniéndose así un suero libre de células. Este fue alicuotado y almacenado a -20ºC. 3.2.9 Electroforesis en geles de poliacrilamida Se prepararon geles con el sistema de tampón discontinuo en condiciones denaturantes al 12%, de acuerdo al procedimiento descrito por Laemmli (1970). El gel separador se preparó con acrilamida-bisacrilamida 30% (p/v) 0,8% (p/v) a una concentración final de 12% (p/v), tamponado con Tris-HCl 1,5 M, pH 8,8 y SDS 10 %. El gel espaciador, en cambio se preparó a una concentración final de 4% (p/v) tamponado con Tris-HCl 0,5 M, pH 6,8 y SDS 10%. Ambos fueron polimerizados con persulfato de amonio 10% (p/v), y TEMED 0,15% (p/v), de concentración final. Las muestras con tampón de muestra y con un 5% (v/v), de β-mercaptoetanol fueron denaturadas calentándolas por 10 min. en agua a 90ºC, para luego ser cargadas cuidadosamente en los surcos del gel. Al sistema ya montado e inmerso en tampón de corrida 1X (25mM Tris, 192mM glicina y 0,1% SDS, pH 8,3), se aplicó aproximadamente una corriente de 80 volt hasta que el indicador del frente iónico llegó al borde inferior del gel. Finalizada la electroforesis, el gel se fijó durante 1 h., luego se tiñó en solución de 32 teñido por 12 h. Finalmente el gel se destiñó mediante repetidos lavados con una solución de desteñido (30% metanol y 10% ácido acético), hasta que el gel quedara completamente claro y sólo se apreciaran las bandas teñidas. Todas estas etapas se llevaron a cabo con agitación suave y constante en una plataforma rotatoria. 3.2.10 Western Blot Primero se realizó una electroforesis en gel de poliacrilamida al 12% en condiciones denaturantes, de las proteínas de membrana y lisado celular de las distintas cepas de F. psychrophilum, cargando determinadas concentraciones de proteínas por carril, detalladas en cada experimento. Luego de realizada la corrida electroforética, los geles fueron electrotransferidos a una membrana de nitrocelulosa en tampón de transferencia (48mM Tris, 30mM glicina, 20% metanol y 0,375% SDS), se transfirió como mínimo 6 horas a 70 mA. Se tomó la membrana ya transferida y se procedió a bloquear los sitios libres con solución de bloqueo (Tris-HCl 20mM, NaCl 150mM y leche descremada 5%). Luego se incubó con el primer anticuerpo dependiendo del experimento, a la dilución indicada en cada figura, con una incubación mínima de 6 horas, luego se realizaron 3 lavados con TBS 1X (Tris-HCl 20mM y NaCl 150mM), cada uno de 10 minutos con agitación suave. Se incubó con el segundo anticuerpo anti-IgG de conejo unido a biotina a una dilución 1:2000 por 2 hora. Luego se realizaron 3 lavados con TBS 1X, cada uno de 10 minutos con agitación suave. Posteriormente se incubó con estreptavidina-peroxidasa a una dilución de 1:1000 por 1 hora. Luego se realizaron 3 lavados con TBS 1X, cada uno de 10 minutos con agitación suave. Luego se realizaron nuevamente 3 lavados con TBS 1X, cada uno de 10 minutos con agitación suave. Para el revelado de la membrana, se incubó con solución de 33 revelado (9ml de PBS, 1ml DAB 10mg/ml y 50µl de H2O2), por 5 minutos. La reacción se detuvo sumergiendo la membrana en agua. 3.2.11 Ensayos de Dot Blot Cada una de las muestras de proteínas de membrana y totales de cada cepa se aplicaron en cada casillero en forma de spot, se secó la nitrocelulosa y luego se incubó por 1 hora con tampón de bloqueo seguido de una incubación con el primer anticuerpo toda la noche (anticuerpo policlonal o recombinante según corresponda), diluido en tampón de bloqueo. En el caso de tratar con el anticuerpo policlonal, se incubó con el segundo anticuerpo anti-IgG de conejo unido a biotina a una dilución 1:2000 por 2 horas. Se lavó tres veces por 10 minutos con TBS1X. Posteriormente se incubó con estreptavidina-peroxidasa a una dilución de 1:1000 por 1 hora. Se lavó tres veces por 10 minutos con TBS1X. En el caso de tratar con el anticuerpo recombinante, se incubó con el segundo anticuerpo anti-M13 de ratón a una dilución 1:1000 por 6 horas. Se lavó tres veces por 10 minutos con TBS1X. Posteriormente se incubó con el tercer anticuerpo anti-IgG de ratón unido a biotina a una dilución de 1:1000 por 2 horas. Se lavó tres veces por 10 minutos con TBS1X. Posteriormente se incubó con estreptavidina-peroxidasa a una dilución de 1:1000 por 1 hora. En ambos casos se reveló con solución de revelado durante 5 minutos, y se detuvo con agua destilada. 34 3.2.12 Inmunohistoquímica Se realizaron extendidos de cultivo puros e improntas de diferentes órganos de truchas arcoiris para este ensayo. Solo las improntas se trataron con una mezcla de metanol 100% (v/v), perhidrol 3% (v/v), durante 10 min. para inactivar las peroxidas endógenas. Seguidamente, se lavó 3 veces durante 5 min. con PBS 1X. Posteriormente para extendidos de cultivos e improntas, en una cámara húmeda, se cubrieron con una solución de bloqueo (PBS 1X, Albúmina 1%, tritón X-100 0,3% y Leche descremada 5%), por 30 min. Con esto se bloquearon los posibles sitios de unión inespecífica del anticuerpo. Luego se lavó 2 veces durante 10 min. con PBS 1X y se incubó con el primer anticuerpo policlonal diluido en solución de bloqueo durante toda la noche. Se lavó 3 veces durante 10 min. con PBS 1X. Se incubó con un segundo anticuerpo antiinmunoglobulina de conejo (DAKO), durante 20 minutos, se lavó 3 veces durante 5 min. en PBS 1X. Luego se continuó con la incubación de estreptavidina-peroxidasa (DAKO), durante 20 min; se lavó 3 veces durante 5 min. en PBS 1X. Para revelar se adicionó la solución de revelado (40ml de PBS1X, 2ml de DAB 10mg/ml y 30µl de H2O2), por 10 min. en oscuridad, agregando 15 µl más de perhidrol a los 5 minutos de comenzado el revelado. Se lavó 3 veces durante 5 min. con PBS 1X. Se realizó tinción de contraste utilizando hematoxilina y tetraborato de sodio. Finalmente solo las improntas se deshidrataron con una batería de xilol y alcoholes en gradiente descendente (100%, 90%, 80%, 70% y 50% v/v). Ambos, extendidos de cultivo puros e improntas, se montaron con bálsamo de Canadá. 35 3.2.13 Inmunofluorescencia Muestras de extendido de cultivo e improntas fueron fijadas con metanol absoluto por 5 min a –20ºC. Posteriormente se realizó el bloqueo con el mismo tampón indicado anteriormente, durante 30 minutos. Se agregó el primer anticuerpo policlonal diluido en tampón de bloqueo y se incubó toda la noche en una cámara húmeda. Después de 3 lavados con PBS 1X, cada uno de 5 minutos, se incubó con el segundo anticuerpo anticonejo acoplado al fluoróforo Alexa Fluor 488 a una dilución de 1:200, por 1 hora. Luego se lavó 3 veces con PBS 1X y se montó con un medio especial para fluorescencia fabricado por DAKO. 3.2.14 Evaluación de fagos activos Para evaluar si el fago M13 recombiante se encontraba activo, se cultivó E. coli ER2537 en 10 ml de medió LB a 37ºC con agitación constante toda la noche. Posteriormente en una placa de agar LB se agregó una mezcla que contenía agar blando (contiene la mitad de agar agar que el agar LB original), y E. coli ER2537. Una vez que la placa se enfrió se agregó una alícuota de fago recombínate (68, 97 y 125), y de incubó a 37ºC toda la noche. La presencia de un halo de inhibición en la placa de agar LB indicó que el fago lisó la bacteria y con esto se comprobó que el fago se encontraba activo. 3.2.15 Amplificación y concentración de fagos Se incubó una colonia de E. coli ER2537, en 10ml de medio LB toda la noche a 37ºC en agitación constante a 200rpm. Este cultivo fue diluido a una razón de 1:100 en un nuevo medio LB y se incubó a 37ºC en agitación constante a 200rpm hasta obtener 36 una D.O600nm entre 0,3 y 0,5. Este cultivo se alicuotó de a 20ml y posteriormente se agregó 20µl de cada clon positivo para F. psychrophilum incubándose a 37ºC por 4 horas en agitación a 200rpm. Luego cada uno de los cultivos fue trasladado a tubos falcon de 50ml estériles y se centrifugaron a 10.000 rpm a 4ºC por 15 minutos. El sobrenandante fue transferido a un nuevo tubo falcon de 50ml estéril, y se agregó 3,3ml de PEG-NaCl estéril, se incubó a 4ºC toda la noche, para precipitar los fagos. Posteriormente se centrifugaron a 10.000rpm a 4ºC por 15 minutos. Se descartó el sobrenadante y el sedimento se resuspendió en 1ml de TBS 1X pH 7,4 estéril. Esta suspensión se transfirió a un tubo eppendorf de 1,5 ml estéril y se agrego 1/6 de PEGNaCl estéril para posteriormente ser incubado por 1 hora en hielo. Transcurrido el tiempo de incubación, se centrifugó por 10 minutos a 10.000 rpm a 4ºC. Se descartó el sobrenadante. El sedimento fue resuspendido en 200µl de TBS 1X y se centrifugo a 4ºC por 1 minuto a 13.000rpm, para sedimentar cualquier material insoluble. El sobrenadante fue transferido a un nuevo tubo eppendorf de 1,5ml y se agrego 200µl de glicerol 80% estéril. Este fue almacenado a -20ºC. 3.2.16 ELISA sandwich Se activó una placa de ELISA poly-sorpTM surface (Nalge Nunc international), con 100µl de los anticuerpos recombinantes previamente diluidos en tampón carbonatobicarbonato 0.05 M pH 9.6 a un título de 1:10. Se dejó activando toda la noche a 4ºC. Se eliminó la solución de activación y se bloqueó por 2horas a 37ºC con 200µl de solución de bloqueo (PBS 1X y 1% BSA). Se eliminó la solución de bloqueo y se lavó 3 veces con 200 µl de solución de lavado (PBS 1X y 0,05% Tween 20). Se prepararon diluciones del antígeno en solución de bloqueo y se incubó por 1 hora a 37ºC. Se 37 eliminó el antígeno y se lavó 3 veces con 200µl de solución de lavado. Posteriormente se incubó con el primer anticuerpo anti-proteínas de membrana 1306 1/15000 por 1 hora a 37ºC. Se eliminó el anticuerpo y se lavó 3 veces con 200µl de solución de lavado. Luego se incubó con un segundo anticuerpo anti-conejo conjugado a peroxidasa 1/5000 por 1 hora a 37ºC. Se eliminó el anticuerpo y se lavó 3 veces con 200µl de solución de lavado. Finalmente se reveló agregando 100µl de TMB hasta un tiempo máximo de 30 minutos. La reacción se detuvo adicionando 100µl de H2SO4 5N. La lectura se efectuó a 450nm. El valor final de absorbancia para cada muestra, se determinó al restar el valor del pocillo control al del pocillo con el antígeno. 3.3 Análisis estadístico Los análisis de la prueba de ELISA sándwich se llevaron a cabo con el programa Microsoft Excel. Los datos se presentaron como promedio más menos error estándar. Según el método recomendado por Brunell et al. (1983) se determinó el punto de corte. Para ello se calculó el promedio más 3 desviaciones estándar, los valores sobre este punto fueron clasificados como positivos y bajo este valor como negativos. 38 4. RESULTADOS 4.1 Caracterización de las cepas de F. psychrophilum Se utilizaron dos cepas de trabajo, una cepa de referencia 1306 y una cepa de campo 4605. Ambas cepas fueron cultivadas en agar TYES a 18ºC por 3 días, presentando las características colonias circulares, convexas, brillantes de color amarillo claro, como se observa en la Figura 1A. La pureza de cada cultivo se verificó por tinción de Gram en donde se pudo observar los típicos bacilos filamentosos Gram negativos (Figura 1B). 4.1.1 Identificación de F. psychrophilum mediante PCR Asimismo ambas cepas fueron confirmadas mediante PCR utilizando partidores PSY1 y PSY2 específicos que amplifican el gen ribosomal 16S. Como resultado se obtuvo un fragmento de 1.100 pb aproximadamente correspondiente a la amplificación parcial del gen ribosomal 16S de F. psychrophilum como se muestra en la Figura 2. El tamaño de este fragmento fue similar para todos los aislados utilizados en este estudio (14,15,16,25,27,31,40,41 y 46), los cuales fueron caracterizados mediante técnicas moleculares (PCR y RFLP), por Figueroa (2008). 39 A B 100X Figura 1. Características morfológicas de Flavobacterium psychrophilum. En A se muestran las colonias amarillas en agar TYES de F. psychrophilum. En B se muestra los bacilos filamentosos delgados y curvos, Gram-negativos de F. psychrophilum. Imagen tomada a 100X. 40 A pb 1.100 1.500 1.000 500 1 2 B 3 4 pb 1.100 1 2 3 4 1.500 1.000 500 Figura 2. Electroforesis en gel de agarosa al 1% del producto de PCR específico del gen 16S rRNA de Flavobacterium psychrophilum. En la figura se observa un producto amplificado de 1.100pb lo cual se obtiene para ambas cepas de F. psychrophilum en A para la cepa de referencia 1306 y en B para la cepa de campo 4605. Carril 1: estándar de peso molecular de 100pb; carril 2: cepa de F. psychrophilum, carril 3: control positivo y carril 4: control negativo. 41 4.2 Producción de anticuerpos policlonales anti-F. psychrophilum 4.2.1 Extracción de proteínas de membrana de F. psychrophilum Para la producción de anticuerpos policlonales primero se procedió a extraer las proteínas de membrana de las cepas 1306 y 4605 tal como se describió en materiales y métodos. Las proteínas fueron cuantificadas por el método de Bradford y posteriormente fueron resueltas en un gel de poliacrilamida en condiciones denaturantes al 12% teñido con azul de Coomassie. En la Figura 3 se observa el patrón de bandas correspondientes a proteínas totales y de membrana de F. psychrophilum, en A para la cepa 1306 y en B para la cepa 4605, evidenciándose proteínas que van desde los 18 hasta por sobre los 120 Kda. El patrón electroforético de proteínas totales y de membrana de ambas cepas de F. psychrophilum son similares y sólo se observan algunas diferencias en cuanto a intensidad y presencia/ausencia de bandas como se indica en la Figura 3 en A y B (flechas). Por ejemplo al comparar el patrón de las proteínas totales del carril 2 de la Figura 3A y 3B, se puede observar en la Figura 3A una banda muy intensa a los 27 kDa para las proteínas totales de la cepa 1306 no así para la cepa 4605. Asimismo si comparamos las proteínas de membrana del carril 3 de la Figura 3A y 3B se puede observar en esta última, una banda más intensa a los 49kDa para la cepa 4605 que para la cepa 1306. Una vez obtenidas las proteínas de membrana estas fueron almacenadas a -20°C para posteriormente ser inoculadas en conejos y así producir anticuerpos policlonales, tal como se detalló en materiales y métodos. 42 A kDa 117 85 1 PT B PM kDa 1 PT PM 117 85 49 49 34 34 25 25 19 19 Figura 3. Electroforesis en gel de poliacrilamida al 12% de proteínas totales y de membrana de Flavobacterium psychrophilum. Perfil electroforético de proteínas totales y de membrana en A para la cepa 1306 y en B para la cepa 4605. Carril 1: estándar preteñido de proteínas de 18-120 kDa, PT: proteínas totales (10µg) y PM: proteínas de membrana (10µg). 43 4.2.2 Titulación de los anticuerpos policlonales anti-F. psychrophilum producidos en conejo El título de los anticuerpos se determinó por el método de dot blot. Se adicionaron 6µg de proteínas constante a una membrana de nitrocelulosa pierce de 0.45 µm. En la figura 4 se observa una clara diferencia en la intensidad de reacción entre los anticuerpos anti-proteínas de membrana y los anticuerpos anti-bacteria completa conforme van aumentando las diluciones de los anticuerpos, siendo más conservada para las proteínas de membrana. Adicionalmente a un título de 1:50.000 sólo los anticuerpos anti-proteínas de membrana son capaces de reconocer eficientemente las proteínas de F. psychrophilum. Como controles negativos de la prueba se adicionaron los sueros preinmunes de cada uno de los conejos, BSA y proteínas totales de hipófisis de Salmo salar, donde no se observó reacción. 4.2.3 Determinación del límite de detección de antígenos utilizando anticuerpos policlonales contra F. psychrophilum Una vez determinado el título óptimo de los anticuerpos (1:50.000), se analizó la cantidad mínima de proteínas de F. psychrophilum que el anticuerpo podía detectar. En la Figura 5 se puede observar que los anticuerpos anti-bacteria completa solo fueron capaces de detectar hasta 2 µg de los antígenos, a partir de los cuales fueron generados. Sin embargo, los anticuerpos anti-proteínas de membrana fueron más eficientes al detectar hasta 0,5 µg, mostrando tener mayor sensibilidad en este ensayo. Por ende, los anticuerpos policlonales anti-proteínas de membrana serán utilizados para los siguientes ensayos. 44 A PM 4605 B PM 1306 PT 1306 C 1:1000 1:3000 1:5000 1:10.000 1:15.000 1:30.000 1:50.000 SCP BSA PT-Ss Controles negativos Figura 4. Titulación de los anticuerpos policlonales contra Flavobacterium psychrophilum mediante Dot blot. Se utilizaron distintas diluciones de los anticuerpos cuyo título se determinó mediante dot blot. A, B y C corresponden a los ensayos realizados utilizando los anticuerpos anti-proteínas de membrana (cepa 4605); antiproteínas de membrana (cepa 1306) y anticuerpo anti-bacteria completa (cepa 1306) respectivamente. Como control negativo se utilizó el suero control preinmune (SCP), BSA y proteínas totales de hipófisis de Salmo salar (PT-Ss). 45 µg de proteína 0,5 1 2 5 10 A PM 1306 B PM 4605 C PT 1306 Figura 5. Análisis del límite de detección de proteínas utilizando anticuerpos contra Flavobacterium psychrophilum mediante Dot blot. A, B y C corresponden a los ensayos realizados utilizando los anticuerpos anti-proteínas de membrana (cepa 1306); anti-proteínas de membrana (cepa 4605) y anticuerpo anti-proteínas totales (cepa 1306) respectivamente. Como control negativo se utilizó BSA y proteínas de hipófisis de Salmo salar donde no se observó inmunoreacción. 46 4.3 Caracterización de los anticuerpos policlonales anti-proteínas de membrana de F. psychrophilum 4.3.1 Análisis de los patrones de proteínas inmunoreactivas de F. psychrophilum Las inmunoreacciones tanto de las proteínas de membrana como de las proteínas totales de este patógeno, fueron analizadas mediante Western blot utilizando los anticuerpos policlonales, hechos en conejos, específicos para las proteínas de membrana de F. psychrophilum. Los anticuerpos anti-proteína de membrana 1306 y 4605 se utilizaron a una dilución de 1:50.000, en presencia de 5µg de proteínas totales y de membrana de F. psychrophilum. En la figura 6A y 6B se observan los distintos patrones de bandas inmunoreactivas generados al utilizar los anticuerpos anti-proteína de membrana 4605 y 1306, respectivamente. Estos anticuerpos mostraron una fuerte inmunoreacción con al menos 2 proteínas de membrana presentes en la cepa 4605 de 40 y 55kDa. Asimismo estos anticuerpos también detectaron una proteína de 55kDa en la cepa 1306 presente en los extractos proteicos totales y de membrana. Sin embargo, también se pudo detectar diferencias en cuanto a las proteínas antigénicas presentes en ambas cepas. Los anticuerpos anti-PM1306 detectaron una proteína de alrededor de 88kDa en la cepa 1306 tanto en extractos proteicos como de membrana. Al contrario, esta proteína no fue detectada por los anticuerpos anti-PM4605. Estos anticuerpos además detectaron en forma diferencial algunas proteínas de bajo peso molecular. Es así como se pudo observar la presencia de una proteína de 19kDa en el extracto proteico de membrana de la cepa 1306 y la cepa 4605 las cuales no fueron detectadas con el antiPM4605. 47 A B 1 2 3 E 1 2 3 E kDa 117 85 49 34 25 19 anti-PM 4605 anti-PM 1306 Figura 6. Análisis de Western blot de los patrones de proteínas inmunoreactivas. A y B análisis de las proteínas de F. psychrophilum utilizando anticuerpos anti-proteínas de membrana de la cepa 4605 y 1306 respectivamente. En A y B Carril 1: proteínas totales de la cepa 1306; Carril 2: Proteínas de membrana de la cepa 1306; Carril 3: proteínas de membrana de la cepa 4605 y carril E: estándar preteñido de proteínas. En cada carril se cargaron 5 µg de proteínas. Los anticuerpos se utilizaron a una dilución de 1:50.000. 48 4.3.2 Análisis de la reacción cruzada de los anticuerpos policlonales contra F. psychrophilum en órganos de Salmo salar y bacterias patógenas de peces Mediante análisis de Western blot se evaluó si los anticuerpos eran capaces de reaccionar con proteínas de órganos de peces y bacterias de importancia para la salmonicultura. Para esto se utilizo una dilución de 1:50.000 de los anticuerpos antiproteínas de membrana. En todos los análisis se utilizó como control positivo las proteínas de membrana (5µg) de la cepa 4605 y 1306. Para evaluar la reacción cruzada de los anticuerpos policlonales con proteínas de órganos de Salmo salar, se cargaron 10 µg de proteínas. En este caso se puede observar, en la Figura 7, que sólo 1 a 2 proteínas inmunoreactivas de gónada, hígado y corazón reaccionaron con los anticuerpos anti-PM 4605 y anti-PM1306. El análisis de la reacción cruzada con proteínas de patógenos de importancia se realizó cargando 2 µg de proteínas. Como se observa en la figura 8 no hubo reacción cruzada con ninguna de las proteínas de bacterias incluyendo las bacterias de su misma familia como F. maritimus y F. columnare. 49 A 1 2 3 4 5 4605 7 8 9 10 anti-PM 4605 B 1 2 3 4 5 1306 7 8 9 10 anti-PM 1306 Figura 7. Análisis de Western blot de la reacción cruzada de los anticuerpos contra Flavobacterium psychrophilum con proteínas de órganos de Salmo salar. A y B corresponden al análisis de proteínas de órganos de S. salar con los anticuerpos anti-proteínas de membrana 4605 y 1306 respectivamente. Carril 1: gónada, carril 2: corazón, carril 3: riñón, carril 4: bazo, carril 5: ciegos pilóricos, carril 7: hígado, carril 8: músculo, carril 9: branquias y carril 10: cerebro. El carril 6 corresponde a las proteínas de membrana de F. psychrophilum (5 µg) de las cepas 4605 y 1306, respectivamente. 50 A 1 2 3 4 5 4605 7 8 9 10 B 1 2 3 4 5 anti-PM 4605 1306 7 8 9 10 anti-PM 1306 Figura 8. Análisis de Western blot de la reacción cruzada de los anticuerpos contra Flavobacterium psychrophilum con proteínas de bacterias patógenas de peces. A y B corresponden al análisis de proteínas de bacterias patógenas de peces con los anticuerpos anti-proteínas de membrana 4605 y 1306. Carril 1: Piscirickettsia salmonis, carril 2: Escherichia coli, carril 3: Flexibacter maritimus, carril 4: Aeromonas hidrophila, carril 5: Aeromonas salmonicida, carril 7: Vibrio ordalii, carril 8: Flavobacterium columnare, carril 9: Bacillus cereus y carril 10: Renibacterium salmoninarum. El carril 6 corresponde a las proteínas de membrana de F. psychrophilum (5 µg) de las cepas 4605 y 1306, respectivamente. 51 4.3.3 Inmunodetección de proteínas totales de distintos aislados de F. psychrophilum Previó a los ensayos de inmunodetección, se realizó una electroforesis en gel de poliacrilamida en condiciones denaturantes al 12% de las proteínas totales de los aislados de F. psychrophilum. En la figura 9 se pueden apreciar los perfiles electroforéticos de las proteínas totales de 9 cepas de campo, evidenciándose una serie de bandas de distintos tamaños teñidas con azul de Coomassie, junto al estándar de peso molecular (carril E). Posteriormente se evaluó mediante análisis de Western blot si los anticuerpos contra proteínas de membrana de F. psychrophilum eran capaces de reconocer proteínas de los distintos aislados de este patógeno. Para ello se cargaron 5µg de proteínas totales de los 9 aislados previamente caracterizados mediante sus características morfologías (Gram) y genotipificar usando técnicas moleculares como PCR y RFLP (Figueroa, 2008). Los anticuerpos anti-proteínas de membrana 1306 y 4605 se utilizaron a una dilución de 1:50.000, tal como se usaron en ensayos anteriores. Como controles positivos se utilizaron 5µg de proteínas de membrana de las cepas 4605 y 1306. En la Figura 10A, se observa que al tratar con los anticuerpos anti proteínas de membrana 4605 se identifican en todos los aislados bandas de 40 y 55 Kda las cuales varían en intensidad al comparar con el control positivo. Asimismo la mayoría de los aislados presentaron patrones inmunoreactivos por sobre los 80 kDa. Por otra parte sólo los aislados 14, 15 16 y 25 presentaron bandas bajo los 25 Kda. En la Figura 10B se observa el mismo patrón que en A en cuanto a las bandas que se encuentran a los 40 y 55 kDa, siendo más intensas al tratar con el anticuerpo 52 anti proteínas de membrana 1306. Asimismo se observa que la mayoría de los aislados presentan bandas inmunorreactivas por sobre los 80kDa pero en menor intensidad. Además sólo los aislados 14, 27, 31, 40, 41 y 46 presentaron una banda muy características a los 19Kda aproximadamente. 53 kDa 14 15 16 25 27 E 31 40 41 46 117 85 49 34 25 19 Figura 9. Electroforesis en gel de poliacrilamida al 12% de proteínas totales de aislados de Flavobacterium psychrophilum. Perfil electroforético de proteínas totales de los aislados 14, 15, 16, 25, 27, 31, 40, 41 y 46. Se cargaron 10µg de proteínas totales en cada carril. Carril E: estándar preteñido de proteínas de 18-120 kDa. 54 A kDa 14 15 16 25 4605 27 31 40 41 46 117 85 49 34 25 19 B kDa 14 15 16 25 1306 27 31 40 41 46 117 85 49 34 25 19 Figura 10. Análisis de Western blot de la reacción de los anticuerpos policlonales contra proteínas totales de distintos aislados de Flavobacterium psychrophilum. A y B, patrones de proteínas inmunoreactivas de distintos aislados de F. psychrophilum utilizando los anticuerpos anti-proteínas de membrana 4605 y 1306 respectivamente. Cada carril indica el aislado de F. psychrophilum utilizado, los que fueron confirmados mediante Gram y PCR (Figueroa, 2008). Como controles positivos se utilizaron proteínas de membrana de las cepas 4605 y 1306 respectivamente. 55 4.3.4 Identificación de F. psychrophilum en peces infectados naturalmente con este patógeno F. psychrophilum fue detectado en truchas arcoiris (O. mikyss) infectadas naturalmente. Para ello se realizaron improntas tomando muestras de lesión externa en la zona del pedúnculo y de lesión interna de bazo para confirmar mediante aislamiento en agar TYES, tinción de Gram y PCR la presencia del patógeno. En la Figura 11A se observa una impronta de la zona del pedúnculo donde se ve la presencia de bacilos largos filamentosos Gram negativos. Lo mismo se observa en la Figura 11B en una impronta de bazo del mismo pez infectado. En la Figura 11C se muestra una confirmación de la presencia del patógeno en el pez mediante tinción de Gram de las colonias obtenidas. La bacteria presente en el pez fue aislada en agar TYES cultivándola por 3 días a 18ºC, obteniéndose las características colonias amarillas. En C se muestra una confirmación de la presencia del patógeno en el pez. En la Figura 11D se observa la identificación de la bacteria mediante la reacción de PCR, obteniéndose para las muestras de la zona del pedúnculo (Carril 2) y bazo (Carril 3) un fragmento de 1.100pb aproximadamente, que corresponde a parte del gen 16S del ARN ribosomal de F. psychrophilum. 56 A B 100X C 100X D E 1 2 3 4 1100 pb 40X Figura 11. Caracterización de Flavobacterium psychrophilum de lesiones de peces infectados naturalmente. La presencia de F. psychrophilum en las lesiones de peces infectados, se confirmaron por Gram en improntas de lesión. A, impronta de lesión externa, en la zona del pedúnculo (flecha) y B, impronta de lesión interna, bazo(flecha). Imágenes tomadas a 100X. En C, se muestra un cultivo puro de F. psychrophilum obtenida de la lesión del pez. Asimismo se confirmo mediante PCR específico la presencia de F. psychrophilum. Imagen tomada a 40X. Carril E: estándar de peso molecular de 100pb, carril 1: control positivo, carril 2: muestra de la zona del pedúnculo, carril 3: muestra de bazo y carril 4: control negativo. 57 4.3.5 Inmunodetección en extendidos de cultivo del patógeno y lesiones de truchas arcoiris (O. mykiss) infectados naturalmente con F. psychrophilum Para las pruebas de inmunohistoquímica e inmunofluorescencia se estableció una dilución de los anticuerpos anti-proteínas de membrana 1306 y 4605 de 1:15.000. 4.3.5.1 Inmunohistoquímica (IHQ) Se realizaron análisis inmunohistoquímicos de extendidos de cultivo puro de psychrophilum fijados con metanol F. y de improntas de lesión externa (zona del pedúnculo) e interna (bazo) de truchas arcoiris infectadas naturalmente con este patógeno, como se muestran en las figuras 12 y 13. La Figura 12 muestra el análisis inmuhistoquímico utilizando el anticuerpo anti proteínas de membrana 1306 (anti-PM1306). En A se observa la inmundetección de la cepa 1306, observándose bacilos de color café. En B se muestra el control negativo donde no se observó tinción inmunopositiva. En C se observan numerosas bacterias teñidas positivamente presentes en impronta de lesión externa (zona del pedúnculo), de peces infectados naturalmente con el patógeno. En D se muestra un control negativo de impronta de lesión externa. En E se observan pocas bacterias (indicadas con fechas), teñidas positivamente pero presentes en impronta de lesión interna (bazo). Finalmente en F se observa el control negativo de bazo donde no se observa reacción del anticuerpo. La Figura 13 muestra el análisis inmuhistoquímico utilizando el anticuerpo anti proteínas de membrana 4605 (anti-PM 4605). En A se observa la inmundetección de la cepa 4605, observándose bacilos de color café. En B se muestra el control negativo donde no se observó reacción. 58 Para los cuadros C, D, E y F se observaron los mismos resultados que lo descrito anteriormente para la Figura 12. La única diferencia radica en la utilización de un anticuerpo (anti-PM 4605), distinto al de la Figura 12. Tanto en extendidos de cultivo como en improntas de lesión se observa que las bacterias se encuentran bien preservadas conservando su morfología alargada de extremos redondeados. 4.3.5.2 Inmunofluorescencia (IFAT) En la Figura 14 se muestra un análisis mediante inmunofluorescencia de extendidos de bacterias en cultivo y lesiones de peces infectados naturalmente con F. psychrophilum. Los anticuerpos anti-proteínas de membrana 1306 (anti-PM 1306) y 4605 (anti-PM 4605) fueron probados a una dilución de 1:15.000, tal como se indicó anteriormente. En la Figura 14A y 14C, corresponden a extendidos de bacterias en cultivo donde se observa la presencia de bacilos alargados, con morfología característica de F. psychrophilum. En la Figura 14B y 14D, corresponden a lesiones externas (zona del pedúnculo) de peces infectados naturalmente con el patógeno, donde se observa la presencia de bacilos con extremos redondeados correspondientes al patógeno (flechas). No se observó reacción en los controles negativos de extendido de cultivo e improntas de lesión externa. 59 A B 100X 100X D C 100X E 100X F 100X 100X Figura 12. Inmunodetección en extendidos de bacterias en cultivo e improntas de lesiones de peces infectados naturalmente con Flavobacterium psychrophilum mediante IHQ. A y B, extendidos de cultivo puro de F. psychrophilum de la cepa 1306. C y D, improntas de la zona del pedúnculo de trucha arcoiris. E y F, improntas de bazo de trucha arcoiris. B, D y F son los controles negativos de cada ensayo realizado. Imágenes tomadas a 100X. 60 A B 100X C 100X D 100X E 100X F 100X 100X Figura 13. Inmunodetección en extendidos de bacterias en cultivo e improntas de lesiones de peces infectados naturalmente con Flavobacterium psychrophilum mediante IHQ. A y B, extendidos de cultivo puro de F. psychrophilum de la cepa 1306. C y D, improntas de la zona del pedúnculo de trucha arcoiris. E y F, improntas de bazo de trucha arcoiris. B, D y F son los controles negativos de cada ensayo realizado. Imágenes tomadas a 100X 61 A B control control Anti-PM1306 63X C 40X D control control 63X 40X Anti-PM4605 Figura 14. Inmunodetección en extendidos de bacterias en cultivo y lesiones de peces infectados naturalmente con Flavobacterium psychrophilum mediante IFAT. A y C extendidos de cultivo puro de F. psychrophilum de la cepa 1306 y 4605, respectivamente (imágenes tomadas a 63X). B y D, improntas de la zona del pedúnculo de trucha arcoiris (imágenes tomadas a 40X). Los cuadros superiores derechos son los controles negativos de cada ensayo realizado. 62 4.4 Evaluación de los anticuerpos recombinantes En un paso previo a los ensayos de inmunodetección, se evaluó si el fago M13 recombinante se encontraba activo (ver detalle sección 3.2.14). Como resultado se observó en todas las placas (LB-agar), lisis completa de la bacteria E. coli ER2537, confirmando que estos se encontraban activos. 4.4.1 Inmunodetección de F. psychrophilum utilizando los anticuerpos recombinantes mediate Dot-blot Se analizó la reacción de los anticuerpos recombinantes contra las cepas 4605 y 1306, utilizando la bacteria completa y sus respectivas proteínas totales (5µg) y de membrana (5µg). Buttcovich (2005) analizó grupos de anticuerpos recombinantes dentro de los cuales identificó y caracterizó 3 de ellos (68, 97 y 125) mediante IFAT y ELISA. Estos anticuerpos recombinantes mostraron ser específicos para F. psychrophilum al ser analizados por ambas técnicas. Así se escogieron estos 3 anticuerpos, los cuales fueron utilizaron a una dilución de 1:10 (Buttcovich, 2005). En la Figura 15 se puede observar que todos los fagos en estudio reconocen proteínas de membrana de ambas cepas. Sin embargo, éstos no fueron capaces de reconocer las proteínas totales de la cepa 1306 pero sí de la cepa 4605. Además estos clones fueron capaces de reaccionar principalmente con la bacteria completa de la cepa 4605. Como control negativo se utilizó BSA y E. coli, donde no se observa inmunorreacción. 63 68 4605 97 1306 4605 125 1306 4605 1306 F.p. PT PM BSA E.c. Figura 15. Análisis de Dot blot de los anticuerpos recombinantes contra Flavobacterium psychrophilum. En la Figura se muestra la inmunodetección de la bacteria completa (F.p.) y proteínas totales (PT) y de membrana (PM) de F. psychrophilum con los distintos anticuerpos recombinantes 68, 97 y 125. Como control negativo se utilizó BSA y E. coli (E. c.) donde no se observa inmunorreacción. 64 4.4.2 Inmunodetección de aislados de F. psychrophilum utilizando anticuerpos recombinantes mediante Dot-blot Se utilizaron 5 µg de proteínas totales de los aislados 14, 15, 16, 25, 27, 31, 40, 41 y 46 previamente caracterizados por Figueroa (2008). Los anticuerpos recombinantes fueron utilizados a una dilución de 1:10. En la Figura 16 se puede observar que los anticuerpos recombinantes son capaces de reconocer todos los aislados a excepción del aislado 31 el cual fue sólo reconocido por el anticuerpo recombinante 68. 4.4.3 Titulación anticuerpos recombinantes En una primera etapa se procedió a titular los anticuerpos recombinantes mediante esta prueba, los cuales se utilizaron como anticuerpo de captura. Para ello se realizaron distintas diluciones de los anticuerpos recombinantes 68, 97 y 125 partiendo con una dilución de 1:10 hasta una dilución de 1:1.280. La titulación de los anticuerpos recombinantes se realizó utilizando 0,5µg constante de proteínas de membrana de la cepa 1306 y 4605. El anticuerpo policlonal anti-proteínas de membrana 1306 fue utilizado para estas pruebas a una dilución de 1:15.000. En la Figura 17 y 18 se muestran las curvas correspondientes a la titulación de los 3 anticuerpos recombinantes 68, 97 y 125. En ambas Figuras se observa que al aumentar las diluciones disminuye la absorbancia radicalmente. En la Figura 17, en presencia de PM-1306, los 3 anticuerpos recombinante utilizados para capturar el antígeno originaron absorbancias de alrededor de 0,2 al utilizar una dilución de 1:10, la cual decae a 0,08 al aumentar la dilución a 1:20. En la Figura 18, en presencia de PM4605 se observa que sólo el anticuerpo recombinante 68 obtiene una absorbancia 65 alrededor de 0,15 al utilizar una dilución de 1:10 mientras que los clones 97 y 125 dan una absorbancia bajo 0,1. Con estos datos se escogió el anticuerpo recombinante 68 para continuar con la caracterización mediante las pruebas de ELISA. 4.5 Desarrollo de una prueba de ELISA sandwich para detectar F. psychrophilum Para el desarrollo de la prueba de ELISA sandwich, se utilizó como anticuerpo de captura el anticuerpo recombinante 68 a una dilución de 1:10 y como anticuerpo de detección se utilizó el anticuerpo policlonal-anti proteínas de membrana1306 a una dilución de 1:15.000 4.5.1 Análisis de la sensibilidad de la prueba de ELISA sándwich La sensibilidad de esta prueba fue analizada a distintas cantidades de proteínas totales de la cepa 1306 y de membrana de la cepa 1306 y 4605. Se partió con una cantidad de 3,2 µg hasta una cantidad de 0,5 µg de proteína. En la Figura 19 se muestran las curvas correspondientes a la detección de las distintas cantidades de proteínas totales y de membrana, por parte de los anticuerpos de captura y detección. En esta figura se observa que ambos anticuerpos son capaces de detectar eficazmente bajas cantidades de las proteínas de membrana de la cepa 1306 obteniendo una absorbancia alrededor de 0,39. Sin embargo, no se observó lo mismo a bajas cantidades de las proteínas totales (1306) y de membrana (4605). Siendo estas eficientemente detectadas por sobre los 1,5 µg de proteína. 66 14 15 16 25 27 31 40 41 46 68 97 125 Figura 16. Análisis de Dot blot de proteínas totales de aislados de Flavobacterium psychrophilum utilizando anticuerpos recombinantes. En la Figura se muestra las proteínas totales de los distintos aislados de F. psychrophilum inmunodetectadas con los distintos anticuerpos recombinantes 68, 97 y 125. 67 Absorbancia (450nm) 0,25 0,2 68 0,15 97 0,1 125 0,05 0 0 200 400 600 800 1000 1200 1400 Diluciones anticuerpo de captura Figura 17. Titulación de los anticuerpos recombinantes en presencia de PM-1306 mediante ELISA sandwich 68 Absorbancia (450nm) 0,16 0,14 0,12 0,1 68 0,08 97 0,06 125 0,04 0,02 0 0 200 400 600 800 1000 1200 1400 Diluciones anticuerpo de captura Figura 18. Titulación de los anticuerpos recombinantes en presencia de PM-4605 mediante ELISA sándwich 69 Absorbancia (450nm) 0,6 0,5 0,4 PM1306 0,3 PM4605 PT1306 0,2 0,1 0 0 0,5 1 1,5 2 2,5 3 3,5 ug de proteína Figura 19. Análisis de la sensibilidad de la prueba de ELISA sandwich. El gráfico representa la sensibilidad dada por la detección de bajas cantidades de proteínas de F. psychrophilum utilizando los anticuerpos de captura (68) y detección (anti-PM1306) 70 4.5.2 Análisis de la especificidad de la prueba de ELISA sandwich Para evaluar la especificidad de la prueba de ELISA contra F. psychrophilum, se realizaron pruebas de reacción cruzada contra las proteínas totales de órganos de Salmo salar y de bacterias de importancia para la salmonicultura. Para estas pruebas se utilizaron como controles positivos, el anticuerpo policlonal anti-F. psychrophilum donado por el Dr. Hugo Folch y proteínas totales de la cepa 1306 de F. psychrophilum. En la Figura 20 se muestra la prueba de reacción cruzada con órganos de Salmo salar (barra 4 a la 14), en donde se utilizaron 0,5µg de proteínas totales. En esta figura se puede observar que no hubo reacción cruzada con ninguna de las proteínas de órganos de S. salar al comparar con el control negativo y con los controles positivos. En la Figura 21 se muestra la prueba de reacción cruzada con bacterias de importancia para la salmonicultura (barra 4 a la 12), en donde se utilizaron 0,5µg de proteínas totales. En esta figura, como en la figura anterior, no se observó reacción cruzada con ninguna de las proteínas de bacterias al comparar con el control negativo y con los controles positivos. 71 0,3 Absorbancia (450nm) 0,25 0,2 0,15 0,1 0,05 0 1 2 3 4 5 6 7 8 9 10 11 12 13 Antígeno Figura 20. Análisis de la especificidad de la prueba de ELISA sandwich contra las proteínas totales de órganos de Salmo salar. Barra 1: Control positivo, anticuerpo policlonal-anti F. psychrophilum; barra 2: control positivo, proteínas totales de la cepa 1306 de F. psychrophilum; barra 3: control negativo, BSA; barra 4: gónada; barra 5: corazón; barra 6: riñón; barra 7: bazo; barra 8: ciegos pilóricos; barra 9: hígado; barra 10: músculo; barra 11: branquias; barra 12: cerebro y barra13: hipófisis. 72 Absorbancia (450nm) 0,3 0,25 0,2 0,15 0,1 0,05 0 1 2 3 4 5 6 7 8 9 10 11 12 Antígeno Figura 21. Análisis de la especificidad de la prueba de ELISA sandwich contra las proteínas totales de bacterias patógenas de peces. Barra 1: Control positivo, anticuerpo monoclonal-anti F. psychrophilum; barra 2: control positivo, proteínas totales de la cepa 1306 de F. psychrophilum; barra 3: control negativo, BSA; barra 4: Renibacterium salmoninarum; barra 5: Bacillus cereus; barra 6: Flavobacterium columnare; barra 7: Vibrio ordalli; barra 8: Aeromonas salmonicida; barra 9: Aeromonas hidrophila; barra 10: Flexibacter maritimus; barra 11: Escherichia Piscirickettsia salmonis coli y barra 12: 73 4.5.3 Inmunodetección de aislados de F. psychrophilum por el método de ELISA sandwich Para evaluar si la prueba de ELISA fue capaz de reaccionar con diferentes cepas de F. psychrophilum, se utilizaron 10 aislados de este patógeno (14, 15, 16, 25, 27, 31, 40, 41 y 46). De estos aislados, se utilizaron las proteínas totales (0,4µg) previamente cuantificadas por el método de Bradford. Como control negativo se utilizó BSA y como control positivo proteínas totales de la cepa 1306 de F. psychrophilum. Para la estandarización de la técnica de ELISA se siguió la metodología recomendada por Brunell et al. (1983), con la finalidad de obtener un punto de corte para clasificar a las muestras como positivas o negativas. Como control positivo se utilizaron las proteínas totales cepas de F. psychrophilum y como control negativo se utilizó BSA y proteínas totales de bacterias patógenas y órganos de peces. Para el grupo control positivo se obtuvieron los siguientes resultados: media = 0,1933 y la desviación estándar = 0,1174 y para el grupo control negativo se obtuvieron los siguientes resultados: media = 0.0491 y la desviación estándar = 0.0028. El punto de corte se obtuvo sumando el promedio del grupo control negativo y 3 desviaciones estándar. El resultado obtenido de esta sumatoria fue 0.0575. Con este valor de punto de corte se puede concluir que la prueba de ELISA es capaz de detectar 8 de los 9 aislados utilizados para esta prueba. Los aislados reconocidos positivamente fueron 14, 15, 16, 25, 27,31,41 y 46. Sólo el aislado 40 no fue detectado por esta prueba. 74 0,45 Absorbancia (450nm) 0,4 0,35 0,3 0,25 0,2 0,15 0,1 0,05 0 1 2 3 4 5 6 7 8 9 10 11 12 Antígeno Figura 22. ELISA sandwich para la detección de las proteínas totales de los distintos aislados de Flavobacterium psychrophilum. Barra 1: Control positivo, anticuerpo monoclonal-anti F. psychrophilum; barra 2: control positivo, proteínas totales de la cepa 1306 de F. psychrophilum; barra 3: control negativo, BSA; barra 4: aislado 14; barra 5: aislado 15; barra 6: aislado 16; barra 7: aislado 25; barra 8: aislado 27; barra 9: aislado 31; barra 10: aislado 40; barra 11: aislado 41 y barra 12: aislado 46. 75 5. DISCUSIÓN Flavobacterium psychrophilum es una importante bacteria Gram negativa causante de la “Enfermedad Bacteriana de Agua Fría” y el “Síndrome del Alevín de la Trucha arcoiris”. Las proteínas de la membrana externa de este patógeno son consideradas moléculas claves en la interface entre la célula y su ambiente. Estas proteínas de membrana son importantes en la interacción del patógeno con las células y tejido del hospedero en el contexto de su patogénesis e inmunidad a la infección (Dumetz et al., 2008). Varios componentes de la superficie de F. psychrophilum han sido implicados en su patogénesis e identificados como posibles candidatos para el diagnóstico. Entre ellos podemos mencionar a las metaloproteasas Fpp1 y Fpp2 (Secades et al. , 2001 y 2003), lipopolisacáridos de baja y alta masa molecular (Crump et al., 2001) y una glicoproteína de membrana (Merle et al., 2003), los que pueden ser utilizados con fines diagnósticos y de control de la enfermedad que produce este patógeno. Se han descrito con anterioridad estudios sobre el desarrollo de una prueba de ELISA sandwich basada en el uso de células enteras como antígenos y anticuerpos policlonales hechos en conejo para detectar F. psychrophilum (Rangdale and Way, 1995; Lorenzen and Olesen, 1997; Mata and Santos, 2001). En este trabajo, con la obtención de proteínas de membrana de F. psychrophilum, la posterior generación de anticuerpos policlonales contra estas proteínas en conejos y la utilización de anticuerpos recombinantes ya producidos en el laboratorio de Biotecnología y Patología Acuática, se logró desarrollar por primera vez una prueba de ELISA sandwich para detectar cepas de F. psychrophilum. 76 Uno de los primeros objetivos de este trabajo fue producir anticuerpos policlonales en conejos contra las proteínas de membrana y la bacteria completa de F. psychrophilum. Mediante el método descrito por Massias et al. (2004) con algunas modificaciones, se logró extraer proteínas de membrana, las cuales fueron posteriormente resueltas por SDS-PAGE al 12% visualizándose proteínas que van de los 18 a los 120kDa, tal como se observó en la Figura 3. Al comparar las proteínas de membrana y totales de las cepas en estudio (1306 y 4605), sólo se observaron diferencias en cuanto a intensidad y presencia/ausencia de bandas, tal como se indicó en ésta Figura. Los patrones de proteínas obtenidos aquí fueron comparados con los patrones de proteínas de membrana y totales descritos por otros autores (Merle et al., 2003; Massias et al., 2004; Sudheesh et al., 2007; Dumetz et al., 2007), observándose similitud en estos patrones. La obtención de estas proteínas de membrana permitió generar anticuerpos policlonales en conejos, los cuales en el desarrollo de esta tesis fueron caracterizados mediante diversos análisis para comprobar que éstos podían ser utilizados en el desarrollo de una prueba de ELISA sandwich. Para poder determinar la dilución óptima de los anticuerpos policlonales y el limite de detección del antígeno se realizaron análisis de Dot blot. Los anticuerpos policlonales producidos contra las proteínas de membrana (PM) de ambas cepas presentaron altos títulos (1:50.000) frente a una cantidad constante de proteína, como se observó en la Figura 4. Estos anticuerpos además detectaron bajas cantidades de proteínas (0,5µg), en comparación con los anticuerpos policlonales generados contra la bacteria completa. Estos resultados nos indican que los anticuerpos anti-PM son capaces de reconocer fuertemente las proteínas de membrana de F. psychrophilum y 77 por lo tanto, son los más adecuados para ser utilizados en los ensayos de inmunodetección. Posterior a esto se realizó la caracterización de los anticuerpos policlonales contra las proteínas de membrana de F. psychrophilum mediante análisis de Western blot. Estos análisis revelaron que ambos anticuerpos no sólo fueron capaces de reaccionar con las proteínas a partir de los cuales fueron generados, sino que también éstos fueron capaces de reconocer varias proteínas de una cepa distinta a la utilizada para producirlos, como se observó en la Figura 6. Estos resultados muestran diferencias entre los patrones de bandas inmunoreactivas obtenidas al tratar con ambos anticuerpos anti-proteínas de membrana. Cabe destacar que las bandas más predominantes con una masa molecular aproximadamente de 88kDa, 55kDa, 40kDa, 24kDa, 23kDa y 19kDa son altamente inmunogénicas tal como ha sido descrito por Sudheesh et al. (2007). Esto resultados además han sido demostrados por LaFrenz et al. (2004) en estudios de la naturaleza inmunogénica de esta bacteria. Estos autores identificaron 3 regiones inmunogénicas del patógeno correspondiente a 18-28kDa, 41-49kDa, y 70-100 kDa mediante análisis de Western blot usando suero inmune de trucha arcoiris (O. mykiss). Si bien, estas regiones fueron detectadas con un suero de peces, nuestros resultados sugieren que estas regiones también serían detectadas con los anticuerpos hechos en conejo. Respecto de la posible naturaleza de las proteínas inmunodetectadas en este estudio, se sugiere que algunas podrían ser proteasas de acuerdo a lo descrito por algunos autores. Bertolini et al. (1994) encontraron 2 proteasas de F. psychrophilum de 114 y 152kDa las que degradaban caseína y gelatina y otras ocho proteasas desde 32 78 a 86kDa, que degradaban gelatina, no así caseína. Además Secades et al. (2001) caracterizó una metaloproteasa psicrofílica de 55kDa la que denominó como Fpp1. Una vez que se demostró que los anticuerpos policlonales anti-proteínas de membrana eran capaces de reconocer antígenos de superficie de F. psychrophilum, se procedió a evaluar, mediante análisis de Western blot, si estos eran capaces de reconocer otras proteínas de órganos de Salmo salar y de bacterias patógenas de peces. Para evaluar la reacción cruzada de los anticuerpos policlonales anti-PM en órganos de Salmo salar, los análisis de Western blot sólo mostraron 1 a 2 proteínas inmunorreactivas en gónada, corazón e hígado. Sin embargo, estos hallazgos no afectarían la aplicación de estos anticuerpos en métodos de diagnóstico, ya que para la detección de este patógeno se utilizan muestras de bazo y riñón principalmente. Mas aún, en ocasiones se utilizan muestras de cerebro de peces infectados con esta bacteria (Cipriano and Holt, 2005), para realizar el diagnóstico, órgano que no presentó inmunorreacción con nuestros anticuerpos. Al realizar el análisis de la reacción cruzada con bacterias patógenas de peces, no se observaron reacciones positivas para ninguna de las bacterias utilizadas. Mas aún, bacterias de la misma familia de este patógeno como F. maritimus y F. columnare no fueron detectadas con los anticuerpos. Autores como Lorenzen and Karas (1992) y Crump et al. (2001) han generado anticuerpos policlonales hechos en conejo contra F. psychrophilum completa. Estos anticuerpos a diferencia de nuestros anticuerpos, presentaron reacción cruzada con F. maritimus y F. columnare, bacterias de la misma familia de este patógeno. Uno de los objetivos de esta tesis fue además determinar si los anticuerpos obtenidos eran capaces de reconocer los distintos aislados de F. psychrophilum. Por medio de los ensayos de Western blot, ambos anticuerpos policlonales anti-PM 79 inmunodetectaron distintas proteínas en los aislados. De hecho, se pudo observar diferencias en los patrones de proteínas inmunorreactivas en los distintos aislados (Figura 10). Tal como se observó en la Figura 6, ambos anticuerpos policlonales detectaron proteínas inmunorreactivas predominantes alrededor de 19kDa, 40kDa, 55kDa y 88kDa para estos aislados (Figura 10). Además cabe destacar que para todos los aislados se conservaron proteínas muy intensas de masa molecular aproximada a los 40kDa y 55kDa. La conservación de ciertas proteínas inmunorreactivas en distintos aislados de F. psychrophilum ha sido descrita por Crump et al. (2001). En este estudio se describe una proteína de 80kDa aproximadamente, la cual estaría presente también en nuestros aislados pero con una menor intensidad. Cabe destacar la presencia en algunos de nuestros aislados de una proteína inmunoreactiva de baja masa molecular de aproximadamente 19kDa. Crump et al. (2001) identificaron el mismo patrón inmunoreactivo señalando que esta proteína de baja masa molecular correspondería a un lipolisacárido. Si bien este resultado confirmó que nuestros anticuerpos reconocían proteínas en los distintos aislados de F. psychrophilum, también permitirá aplicarlos en estudios de caracterización antigénica de este patógeno. Posteriormente, los anticuerpos policlonales obtenidos fueron aplicados en ensayos de inmuhistoquímica e inmunofluorescencia para evaluar la capacidad de éstos para reconocer los antígenos de F. psychrophilum en la bacteria. Los resultados obtenidos mostraron que F. psychrophilum puede ser detectada en extendidos de cultivo e improntas de peces infectados naturalmente con los anticuerpos anti-PM1306 y anti-PM4605 a una dilución de 1:15.000 (Figura 12, 13 y 14). En todos los ensayos se observó una reacción positiva dada por la presencia de bacilos alargados. Los controles negativos no mostraron reacción. La detección de F. psychrophilum mediante las 80 técnicas de Western blot, IHQ e IFAT confirmaron que los anticuerpos son capaces de detectar antígenos de F. psychrophilum tanto en su forma secuencial y conformacional. Lorenzen and Karas (1992) realizaron análisis de inmunofluorescencia de improntas de bazo de alevines de trucha arcoiris utilizando antisuero de conejo preparado contra F. psychrophilum. Estos autores lograron detectar la bacteria tanto viva como muerta. Adicionalmente Ekman and Norrgren (2003) realizaron análisis inmunohistoquímicos de tejidos de riñón y bazo principalmente de trucha arcoiris y salmón del atlántico. En ambos tejidos se observó fuertemente la presencia de F. psychrophilum. En resumen, se obtuvieron anticuerpos policlonales específicos contra proteínas de membrana de F. psychrophilum, capaces de detectar distintos aislados del patógeno tanto por Western blot, IHQ e IFAT, los que ahora pueden ser aplicados para el desarrollo de una prueba de ELISA. Posterior a esto se evaluó mediante análisis Dot blot y ELISA, si los anticuerpos recombinantes eran capaces de detectar F. psychrophilum. Buttcovich (2005) en su tesis de pregrado, analizó una biblioteca de anticuerpos recombinantes contra F. psychrophilum mediante ELISA e IFAT. Buttcovich logró obtener 2 clones específicos contra este patógeno de peces. Estos clones recombinantes no reaccionaron con las diferentes cepas bacterianas de salmónidos (A. hidrophila, A. salmonicida, A. sobria, R. salmoninarum, V. anguilarum, Y. ruckeri, P. salmonis), como tampoco lo hicieron con las bacterias de la misma familia (F. columnare, F. maritimus). Estos tampoco presentaron reacción cruzada con tejido renal. Demostrando que estos clones son aplicables a métodos de diagnóstico inmunológico como ELISA. 81 Tres anticuerpos recombinantes positivos para la detección de F. psychrophilum fueron utilizados(68, 97 y 125). Previó a los ensayos de inmunodetección se comprobó que éstos se encontraran activos mediante la observación de lisis celular de E. coli ER2537 en placa de agar LB. Mediante análisis de Dot blot, se confirmó que estos anticuerpos eran capaces de detectar la bacteria completa y sus proteínas totales y de membrana, utilizando las cepas 1306 y 4605. Como se observó en la Figura 15 todos los clones fueron capaces de reconocer las distintas proteínas y también la bacteria completa de la cepa 4605, lo que no ocurrió con la cepa 1306 en donde sólo se reconoció, por parte de los 3 clones, sólo las proteínas de membrana. Lo anterior sugiere que existe una diferencia en el reconocimiento de proteínas totales por parte de los anticuerpos recombinantes. Esto no debería influir al momento de desarrollar la prueba de ELISA puesto que estos anticuerpos están dirigidos contra un epítope de la superficie de la bacteria. Además los clones 68 y 97 son capaces de reconocer la bacteria completa de la cepa 1306, lo que demuestra que estos son más adecuados para ser utilizados en los ensayos posteriores. Por otra parte, mediante ensayos de Dot blot, se observó que 2 anticuerpos recombinantes fueron capaces de detectar 8 de los 9 aislados del patógeno y sólo el anticuerpo 68 fue capaz de detectarlos en su totalidad. Estos resultados indican que el clon 68 reconoció un antígeno específico de F. psychrophilum presente en los 9 aislados y en las 2 cepas en estudio (1306 y 4605). Los anticuerpos recombinantes han sido generados, utilizando la tecnología del “Phage display” o despliegue en fago, y empleados por muchos autores. Cao et al. (2000) produjeron anticuerpos recombinantes como inhibidores del crecimiento de 82 Helicobacter pylori. Adicionalmente, Manoutcharian et al. (2004) han producido anticuerpos recombinantes para inmunizaciones y fabricación de vacunas contra Cisticercosis por Taenia solium en cerdos. Además se han utilizado en enfermedades infecciosas. Investigaciones realizadas sobre el ántrax, han utilizado una biblioteca de scFv humana expresada en fagos para ser sometida a una selección directa in vitro contra espora vivas nativas de Bacillus subtilis. Con esto se desarrollo un método para detectar esporas individuales mediante el uso de bibliotecas de anticuerpos acopladas a fluoresceína y expresadas en fago (Santamaría, 2003). Es importante contar con este tipo de tecnología (“Phage display”) sobre todo cuando se quiere desarrollar una prueba de diagnóstico lo más sensible y específica posible para poder detectar patógenos de importancia en la salmonicultura. Cabe destacar que este trabajo describe por primera vez el uso de anticuerpos recombinantes y policlonales específicos para detectar F. psychrophilum. Para el montaje de la prueba de ELISA sandwich se utilizó como anticuerpo de captura el anticuerpo recombinante 68 a una dilución de 1:10 y como anticuerpo de detección el anticuerpo policlonal anti-PM 1306 a una dilución de 1:15.000. Mediante esta prueba se determinó el título óptimo de los anticuerpos recombinantes y además se determinó con cuales de estos 3 clones se obtendría una mayor detección de las proteínas de membrana de las cepas 1306 y 4605. Los resultados muestran que la dilución óptima del anticuerpo de captura para el ensayo fue de 1:10 para todos los clones (Figuras 17 y 18). Sin embargo, sólo con el clon 68 se obtuvieron absorbancias por sobre 0,15 de OD450nm al utilizar PM-1306 y PM-4605, lo que confirmó que este clon es más sensible que los otros dos (97 y125) para ser utilizado en la prueba final. 83 La sensibilidad de la prueba fue analizada utilizando distintas cantidades de proteínas totales de la cepa 1306 y de membrana de la cepa 1306 y 4605. De la Figura 19, se puede concluir que la prueba de ELISA fue capaz de detectar bajas cantidades de proteínas totales y de membrana (0,5-1,0 µg). Lo cual demuestra que estos anticuerpos son capaces de detectar positivamente las proteínas de la superficie de F. psychrophilum. La especificad de la prueba fue analizada mediante pruebas de reacción cruzada con proteínas totales de órganos de peces (Salmo salar) y con bacterias patógenas de importancia para la salmonicultura, como se describió para los anticuerpos policlonales. Los resultados observados en la Figura 20 y 21 no arrojaron valores positivos para ninguna de las proteínas totales de órganos y bacterias utilizadas para esta prueba, lo cual demuestra que ambos anticuerpos son específicos para F. psychrophilum. Posterior a esto se realizó una prueba para evaluar si estos anticuerpos en conjunto eran capaces de detectar antígenos de la membrana de distintos aislados tal como se realizó para cada anticuerpo por separado. Para esto se utilizaron 9 aislados de F. psychrophilum en donde se lograron detectar positivamente 8 de los 9 aislados (Figura 22). Para diferenciar que muestras eran positivas y negativas se obtuvo el punto de corte de nuestra prueba (0,0575) siguiendo el método recomendado por Brunell et al. (1983). Estudios previos se han realizado utilizando la prueba de ELISA sandwich para diferenciar serológicamente entre aislados de F. psychrophilum. Estos estudios se realizaron utilizando anticuerpos policlonales hechos en conejo y preparados de bacteria completa (Lorenzen and Olensen, 1997). Mata y Santos (2001) también realizaron ensayos utilizando la técnica de ELISA sandwich con el fin de mejorar el 84 diagnóstico de enfermedades causadas por F. psychrophilum en salmón y trucha. Estos autores lograron montar un ensayo de ELISA específico, sensible y rápido utilizando anticuerpos policlonales y de bacteria completa. La prueba de ELISA ha sido utilizada ampliamente en el diagnóstico de patógenos de peces. La unión covalente de enzimas a las moléculas de anticuerpos sin alterar las propiedades catalíticas de éstas y la especificidad del anticuerpo produce una herramienta inmunológica que posee alta sensibilidad y alta especificidad. Esta prueba puede ser usada para la detección del antígeno soluble extraído desde el tejido infectado, y es probablemente el método más sensible. Permite analizar un gran número de muestras y está especialmente bien adaptada para los requerimientos de certificación sanitaria. La prueba de ELISA sandwich desarrollada en este estudio a demostrado ser un método de diagnóstico sensible, específico y confiable para la detección de F. psychrophilum. Es por esto, que es importante seguir realizando pruebas con los anticuerpos recombinantes y policlonales y buscar nuevos métodos de detección para aumentar la sensibilidad de la prueba de tal manera de poder detectar cualquier aislado de este patógeno de peces. A futuro se espera poder utilizar esta prueba de manera efectiva para detectar, en etapas temprana de la infección, cantidades bajas de la bacteria completa para así poder controlar y dar tratamiento a tiempo a especies salmónidas afectadas. 85 6. BIBLIOGRAFIA Amita, K., Hoshimo, M., Honma, T., and Wakabayashi H. (2000). An investigation on distribution of Flavobacterium psychrophilum in the Umikava River. Fish Pathol., 35, 193–197. Aikawa, H. (1998). Indirect immunoperoxidase technique for the diagnosis of cold-water disease in cultured ayu, Plecoglossus altivelis. Fish Pathol., 33: 149– 50. Bernardet, J.F. and Grimont P.A.D. (1989). Deoxyribonucleic acid relatedness and phenotypic characterization of Flexibacter columnaris sp. nov., nom. rev., Flexibacter psychrophilus sp. nov., and Flexibacter maritimus Wakabayashi, Hikida and Masumura 1986. Int. J. Syst. Bacteriol., 39, 346–354. Bernardet, J.F. and Kerouault, B. (1989). Phenotypic and genomic studies of Cytophaga psychrophila isolated from diseased rainbow trout (Oncorhynchus mykiss) in France. Appl. Environ. Microbiol., 55, 1796–1800. Bernardet, J.F., Sergers, P., Vancanneyt, M., Berthe, F., Kersters, K. and Vandamme P. (1996). Cutting a Gordian Knot: emended classification and description of the genus Flavobacterium, emended description of the family Flavobacteriaceae, and proposal of Flavobacterium hydatis nom. nov. (basonym, Cytophaga aquatilis Strohl & Tait 1978). Int. J. Syst. Bacteriol., 46, 128–148. 86 Bertolini, J.M., Wakabayashi, H., Watral, V.G., Whipple, M.J. and Rohovec, J.S. (1994). Electrophoretic detection of protease from selected strains of Flexibacter psychrophilus and assessment of their variability. J. Aquat. Anim. Health., 6, 224– 233. Borg, A.F. (1960). Studies on Myxobacteria Associated with Diseases in Salmonid Fishes. American Association for the Advancement of Science, Wildlife Disease No. 8, Washington, DC, USA. Bravo, S. and Campos, M. (1989). Coho salmon syndrome in Chile. FHS/AFS Newsl., 17(3): 3 Bravo, S. (2000). Occurrence of atypical furunculosis in Chile. Bull. Eur-Ass. Fish Pathol., 20 (5): 209-211 Bravo, S. (1996). Enterocytozoon salmonis in Chile. FHS/AFS Newsletter. Vol. 24 (1): 12-13. Bradford, M.M. (1976). A Rapid and Sensitive Method for the Quantitation of Microgram Quantities of Protein Utilizing the Principle of Protein Dye Binding. Analytical Biochem., 72, 248-254. 87 Brunell, P.A., Wingle, K., Murphy, M.D. et al. (1983). Antibody response folowing measles-mumps-rubella vaccine under conditions of customary use. JAMA., 250: 1409-1412. Bustos, P., Calbuyahue. J.; Montaña, J.; Opazo, B.; Entrala, P. and Solervicens, R. (1995). First Isolation of Flexibacter psychrophilus as Causative Agent of Rainbow Trout Fry Syndrome (RTFS) Producing Rainbow Trout Mortality in Chile. Dept of Marine Resources-Fundacion, Chile. Buttcovich, C. L., (2005). Análisis de una biblioteca de anticuerpos recombinantes contra Flavobacterium pshycrophilum mediante inmunoensayo acoplado a enzima (ELISA) y técnica indirecta de anticuerpos inmunofluorescentes (IFAT). Tesis, Escuela de Tecnología Medica, Facultad de Medicina. Univ. Austral de Chile, 58 pp. Cepeda, C. and Santos, Y. (2000). Rapid and low-level toxic PCR based method for routine identification of Flavobacterium psychrophilum. Int. Microbiol., 3, 235–238. Cao, J., Sun, Y., Berglindh, T., Mellgard, B., Li, Z. and Mardh, B. (2000). Helicobacter pylori-antigen-binding fragment expressed on the filamentous M13 phage prevent bacterial growth. Biochim. Biophys. Acta. Mar., 6;1474 (1): 107-113. 88 Chacko, A. J., Hoffman, G.L. and Bravo, S. (2001). Kudoa thyrsites (Myxozoa, Multivalvulida) detected in the muscles of farmed Atlantic Salmon from Chile. Abstract Book: International Conference EAFP 9-14 September, Dublin, Irlanda. Cipriano, R.C. and Holt, R.A. (2005). Flavobacterium psychrophilum, Cause of Bacterial Cold Water Disease in Rainbow Trout Fry Syndrome. Fish Disease Leaflet No. 86. United States Department of the Interior. Kearneysville, WV: US Geological Service, National Fish Health Research Laboratory. Crump, E.M., Perry, M.B., Clouthier, S.C. and Kay, W.W. (2001). Antigenic characterization of the fish pathogen Flavobacterium psychrophilum. Appl. Environ. Microbiol., 67, 750–759. Crump, E.M., Burian, J., Allen, P.D. and Kay, W.W. (2005). Identification and expression of a host-recognized antigen, FspA, from Flavobacterium psychrophilum. Microbiol., 151, 3127–3135. Dalsgaard, I. (1993) Virulence mechanisms in Cytophaga psychrophila and other Cytophaga-like bacteria pathogenic for fish. Annu. Rev. Fish Dis., 1, 127–144. Daskalov, H., Austin D.A. and Austin B. (1999) An improved growth medium for Flavobacterium psychrophilum. Lett. Appl. Microbiol., 28, 297–299. 89 Davis, H. S. (1946). Care and Diseases of Trout. US Department of Interior Research Report, No. 12. US Government Printing Office, Washington, DC, USA. Duchaud, E., Boussaha, M., Loux, V., Bernardet, J-F., Michel, C., Kerouault, B., Mondot, S., Nicolas, P., Bossy, R. And other authors (2007). Complete genome sequence of the fish pathogen Flavobacterium psychrophilum. Nat. Biotechnol., 25, 763–769. Dumetz, F., Duchaud, E., LaPatra, S.-E., Le Marrec, C., Claverol, S., Urdaci, M. C. and Le Hénaff, M. (2006). A protective immune response is generated in rainbow trout by an OmpH-like surface antigen (P18) of Flavobacterium psychrophilum. Appl. Environ. Microbiol., 72, 4845–4852. Dumetz, F., LaPatra, S.-E., Duchaud, E., Claverol, S. and Le Hénaff, M. (2007). The Flavobacterium psychrophilum OmpA, an outer membrane glycoprotein, induces a humoral response in rainbow trout. J. Appl. Microbiol., 103, 1461–1470. Dumetz, F., Duchaud, E., Claverol, S., Orieux, N., Papillon, S., Lapaillerie, D. and Le Hénaff, M. (2008) Analysis of the Flavobacterium psychrophilum outer-membrane subproteome and identification of new antigenic targets for vaccine by immunomics. Microbiology., 154, 1793–1801. 90 Ekman, E. and Norrgren L. (2003). Pathology and immunohistochemistry in three speciesof salmonids after experimental infection with Flavobacterium psychrophilum. J. Fish Dis., 26, 529–538. Figueroa, V.N. (2008). Genotipificación de Flavobacterium psychrophilum aislados de salmónidos cultivados en el sur de chile.Tesis, Escuela de Tecnología Médica, Facultad de Medicina. Univ. Austral de Chile, 38 pp. Fuchs, P., Breitling, F., Dubel, S., Seehaus, T. and recombinant antibodies to the surface of Little, M. (1991). Targeting Escherichia coli : fusion to a peptidoglycan associated lipoprotein. Biotechnol., 9:1369-1372. Fuller, D. W., Pilcher, K.S. and Fryer, J.I. (1977). A leukocytolytic factor isolated from cultures of Aeromonas salmonicida. J. Fish. Res. Board Can., 34: 1118-1125 Hashimoto, Y., Tanigawa, K., Nakashima, M., et al. (1999). Construction of the singlechain Fv from 196-14 antibody toward ovarian cancer-associated antigen CA125. Biol. Pham. Bull., 22: 1068-1072. Holt, R.A. (1988). Cytophaga psychrophila, the Causative Agent of Bacterial Cold-water. Disease in Salmonid Fish. PhD thesis, Oregon State University, Corvallis. 91 Holt, R.A., Amandi, A., Rohovec, J.S. and Fryer, J.L. (1989). Relation of water temperature to bacterial cold-water disease in coho salmon, chinook salmon, and rainbow trout. J. Aquat. Anim. Health.,1, 94–101. Holt, R.A., Rohovec, J.S. and Fryer, J.L. (1993). Bacterial cold-water disease. In: Bacterial Diseases of Fish (ed. by V. Inglis, R.J. Roberts & N.R. Bromage), pp. 3– 23. Blackwell Scientific Publications, Oxford. Hornbeck, P. (1991). Enzyme-linked immunosorbent assay. In Coligan JE, Kruisbeek AM, Margulies DH, Shevack EM, Strober W, eds. Curr. Protoc. Immunol., New York: John Wiley and Sons, Inc. p 2.1.2-2.1.22 Iida, Y. and Mizokami, A. (1996). Outbreaks of coldwater disease in wild ayu and chub. Fish Pathol.,31, 157–164 Izumi, S. and Wakabayashi H. (1997). Use of PCR to detect Cytophaga psychrophila from apparently healthy juvenile ayu and coho salmon eggs. Fish Pathol., 32, 169–173. Jager, M. And Pluckthun, A. (1997). Domain interactions in antibody Fv and scFv fragments:effects on unfolding kinetics and equilibria. FEBS lett., 462: 307-312. 92 Kipriyanov, S.M., Moldenhauer, G. And Little, M. (1997). High level production of soluble single chain antibodies in smal-scale Escherichia coli culture. J. Immunol. Methods., 200:69-77. Kondo, M., Kawai, K., Kurohara, K. and Oshima, S. (2002). Adherence of Flavobacterium psychrophilum on the body surface of the ayu Plecoglossus altivelis. Microbes Infect., 4, 279–283. Laemmli, U. K. (1970). Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature., 227; 680-5 LaFrentz, B.R., LaPatra, S.E., Jones, G.R. and Cain, K.D. (2004). Protective immunity in rainbow trout Oncorhynchus mykiss following immunization with distinct molecular mass fractions isolated from Flavobacterium psychrophilum. Dis. Aquat. Org., 59:17-26. Lehmann, J., Mock D., Stürenberg, F.-J. and Bernardet, J.-F. (1991) First isolation of Cytophaga psychrophila from a systemic disease in eel and cyprinids. Dis. Aquat. Org., 10, 217–220. Lipman, N.S., Jackson, L.J., Trudel, L.J. and Weis-Garcia, F. (2005). Monoclonal versus polyclonal antibodies: distinguishing characteristics, applications, and information resources, ILAR J., 46 pp. 258–268. 93 Liu, H., Izumi S. and Wakabayashi H. (2001). Detection of Flavobacterium psychrophilum in various organs of ayu Plecoglossus altivelis by in situ hybridization. Fish Pathol., 36, 7–11. Lorenzen, E. (1994). Study on Flexibacter psychrophilus in Relation to Rainbow Trout Fry Syndrome (RTFS). PhD thesis, Royal Veterinary and Agricultural University, Copenhagen. Lorenzen, E., Dalsgaard I. and Bernardet J-F. (1997). Characterization of isolates of Flavobacterium psychrophilum associated with coldwater disease or rainbow trout fry syndrome I: phenotypic and genomic studies. Dis. Aquat. Org., 31, 197–208. Lorenzen, E. and Karas N. (1992). Detection of Flexibacter psychrophilus by immunoflurescence in fish suffering from mortality syndrome: a rapid diagnostic method. Dis. Aquat. Org.,13, 231–234. Lorenzen, E. and Olesen, N. J. (1997). Characterization of isolates of Flavobacterium psychrophilum associated with coldwater disease or rainbow trout fry syndrome II: Serological studies. Dis. Aquat. Org., 31: 209–208. Lubkowski, J., Hennecke, F., Plückthum, A. and Wlodawer, A. (1999). Filamentous phage infection: crystal structure of g3p in complex with its coreceptor, the Cterminal domain of ToIA. Structure.,7:711-722. 94 MacLean, L.L., Vinogradov, E., Crump, E.M., Perry, M. B. and Kay, W.W. (2001). The structure of the lipopolysaccharide O-antigen produced by Flavobacterium psychrophilum (259-93). Eur. J. Biochem., 268, 2710±2716 Madetoja, J., Hänninen, M-N., Hirvelä-Koski, V., Dalsgaard, I. and Wiklund, T. (2001). Phenotypic and genotypic characterization of Flavobacterium psychrophilum from Finnish fish farms. J. Fish Dis., 24: 469–479. Madsen, L. and Dalsgaard, I. (2000). Comparative studies of Danish Flavobacterium psychrophilum isolates: ribotypes, plasmid profiles, serotypes and virulence. J. Fish Dis., 23, 211–218. Manoutcharian, K., Díaz, O.A., Gevorkian, G., Fragoso, G., Acero, G. et al. ( 2004). Recombinant bacteriophago- based multiepitope vaccine against Taenia solium pig cysticercosis. Vet. Immunol. Immunpathol., 99(1-2): 11-24 Massias, B., Dumetz, F., Urdaci, M. C. and Le Hénaff, M. (2004). Identification of P18, a surface protein produced by the fish pathogen Flavobacterium psychrophilum. J. Appl. Microbiol., 97, 574–580. Mata M. and Santos, Y. (2001). An improved enzyme-linked immunosorbent assay (ELISA) for detection of Flavobacterium psychrophylum isolated from salmon and rainbow trout. Bull. Eur. Ass. Fish Pathol., 21(5):195-199. 95 McAllister, P.E. and Reyes, X. (1984). Infectius Pancreatic Necrosis Virus: Isolation From Rainbow Trout, Salmo gairdneri Richardson, Imported Into Chile. J. Fish. Dis., 7: 319-322. Merle, C., Faure, D., Urdaci, M. C. and Le Hénaff, M. (2003). Purification and characterization of a membrane glycoprotein from the fish pathogen Flavobacterium psychrophilum. J. Appl. Microbiol., 94, 1120–1127. Monrás M., Quinteros V., Enríquez R. (2003). Furunculosis atípica en salmonídeos de cultivo en Chile. Vetermas, 22-24. Morisaki, H., Nagai, S., Ohshima, H., Ikemoto, E. and Kogure, K.(1999). The effect of motility and cell-surface polymers on bacterial attachment. Microbiology., 145, 2797–2802. Nematollahi, A., Decostere, A., Pasmans, F., Ducatelle, R. and Haesebrouck, F. (2003). Adhesion of high and low virulence Flavobacterium psychrophilum strains to isolated gill arches of rainbow trout Oncorhynchus mykiss. Dis. Aquat. Org., 55: 101–107. Nilson, W.B. and Strom M.S. (2000). The phage infection process: a funcional role for the distal linker region of bacteriophage protein 3. J. Virol., p:4229-4235. 96 Pacha, R.E. (1968). Characterization of Cytophaga psychrophila (Borg) isolated during outbreaks of bacterial cold-water disease. App. Microbiol.,16, 97–101. Rahman, M. H., Kuroda, A., Dijkstra, J.M., Kiryu, I., Nakanishi, T. and Ototake, M. (2002). The outer membrane fraction of Flavobacterium psychrophilum induces protective immunity in rainbow trout and ayu. Fish Shell. Immunol., 12: 169–179. Rangdale R.E., Richards R.H. and Alderman D.J. (1996). Isolation of Cytophaga psychrophila, causal agent of rainbow trout fry syndrome (RTFS) from reproductive fluids and egg surfaces of rainbow trout (Oncorhynchus mykiss). Bull. Eur. Ass. Fish Pathol., 16, 63–67. Rangdale R.E. (1995). Studies on Rainbow Trout Fry Syndrome (RTFS). PhD thesis, University of Stirling, Stirling. Rangdale R.E. and Way K. (1995). Rapid identification of Cytophaga psychrophila from infected spleen tissue using an enzyme-linked immunosorbent assay (ELISA). Bull. Eur. Ass. Fish Pathol., 15, 213–216. Rodkhum, C., Hirono, I., Crosa, J.H. and Aoki, T. (2005). Four novel hemolysin genes of Vibrio anguillarum and their virulence to rainbow trout. Microb. Pathog., 39, 109–119 97 Sambrook, J., Fritsch, E.F. and Maniatis, T.; Hrsg. (1989). Molecular Cloning - A Laboratory Manual, 2nd Edition. Cold Spring Habour Laboratory Press, New York. Sanchez, L., Ayala, M., Freyre, F., et al. (1999). High cytoplasmatic expresion in E. coli, purification, and in.vitro refolding of a single chain Fv antibody fragment against the hepatitis B surface antigen. J. Biotechnol., 72:13-20. Santamaría, H. (2003). Phage display y su aplicación en el diagnóstico clínico. Revista de Investigación Clínica. Vol 55, Núm 6, pp711-718. Schmidtke, l.M. and Carson, J. (1995). Characteristics of Flexibacter psychrophilus isolated from Atlantic salmon in Australia. Dis. Aquat. Org., 21: 157 ± 161. Secades, P., Alvarez B. and Guijarro J.A. (2001). Purification and characterization of a psychrophilic, calcium-induced, growth-phase-dependent metalloprotease from the fish pathogen Flavobacterium psychrophilum. Appl.Environ. Microbiol., 67, 2436–2444. Secades, P., Alvarez, B. and Guijarro, J.A. (2003). Purification and properties of a new psychrophilic metalloprotease (Fpp2) in the fish pathogen Flavobacterium psychrophilum. FEMS Microbiol. Lett. 226, 273–279. 98 Shotts, E.B. and Teska, J.D. (1989).Bacterial pathogens of aquatic vertebrates. In: B. Austin and D.A. Austin, Editors, Methods for the Microbiological Examination of Fish and Shellfish, Ellis Wowood, Chichester (1989), pp. 164–186. Shotts, E.B. and Starliper, C.E. (1999). Flavobacterial diseases: columnaris disease, cold-water disease and bacterial gill disease. In: P.T.K. Woo and D.W. Bruno, Editors, Fish Disease and DisordersViral, Bacterial and Fungal Infections vol. 3, CAB Publishing, New York (1999), pp. 559–576. Smith, G.P. (1985). Filamentous fusion phage: novel expression vectors that display cloned antigens on the virion surface. Science., 228:1315-17. Smith, G.P. and Petrenko, V.A. (1997). Phage display. Chem. Rev., 97: 391-410. Snyder, B. (1971). Supplemental Report on Inland Freshwater Resources of Central Chile. UNDP/FAO. 35 pp. Sudheesh, P. S., LaFrentz, B. R., Call, D. R., Siems, W. F., LaPatra, S.-E., Wiens, G. D. and Cain, K. D. (2007). Identification of potential vaccine target antigens by immunoproteomic analysis of a virulent and a non-virulent strain of the fish pathogen Flavobacterium psychrophilum. Dis. Aquat. Org., 74, 37–47. 99 Toledo, M.S., Troncoso, M., Portell D.P. and Figueroa, G. (1992). Reporte de un brote de yersiniosis asociada a Yersinia ruckeri en salmónidos de cultivo. XV Congreso Chileno de Microbiología, Valdivia, 9 al 12 de octubre, 1992. Toyama, T., Kita-Tsukamoto K. and Wakabayashi H. (1994) Identification of Cytophaga psychrophila by PCR targeted 16S ribosomal RNA. Fish Pathol.,29, 271–275. Vispo, N. S. (2004). Combinatoria Molecular. 413 pp. Wilklund, T., Madsen, L., Bruun, M.S. and Dalsgaard, I. (2000). Detection of Flavobacterium psychrophilum from fish and water samples by PCR amplification. J. Appl. Microbiol., 88:299-307. Wood, W. J. (1970). Introducción del Salmón del Pacífico en Chile, Informe al Servicio Agrícola y Ganadero, División de Pesca y Caza. 8 pp.