BIOSÍNTESIS DE NUCLEÓTIDOS
Anuncio
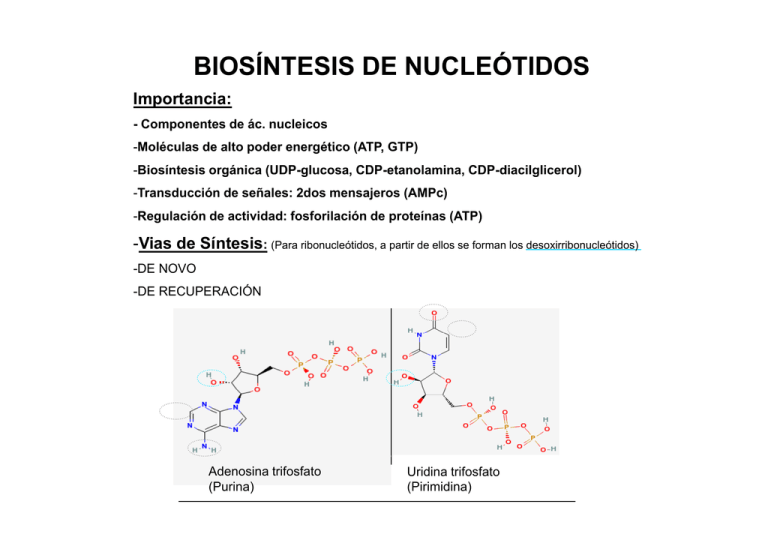
BIOSÍNTESIS DE NUCLEÓTIDOS Importancia: - Componentes de ác. nucleicos -Moléculas de alto poder energético (ATP (ATP, GTP) -Biosíntesis orgánica (UDP-glucosa, CDP-etanolamina, CDP-diacilglicerol) -Transducción de señales: 2dos mensajeros (AMPc) -Regulación de actividad: fosforilación de proteínas (ATP) -Vias de Síntesis: (Para ribonucleótidos, a partir de ellos se forman los desoxirribonucleótidos) -DE DE NOVO -DE RECUPERACIÓN Adenosina trifosfato (Purina) Uridina trifosfato (Pirimidina) Se recuperan las bases y se añaden las ribosas ib Pirimidinas: Se va sintetizando la base y la ribosa es incluida al final Purinas: el anillo de la base crece sobre una estructura que contiene ribosa Síntesis de Novo de PIRIMIDINAS Glutamina + H 2O Carbamilfosfato sintetasa (CPS) Responsable de varias reacciones sucesivas Glutamato Carbamilfosfato sintetasa i t t (CPS) Aspartato p transcarbamilasa Fosforribosil transferasa CO2 H Orotidilato decarboxilasa Formación de UDP y UTP: intercambio de P entre nucleótidos UMP + ATP UDP + ADP UDP + XTP UTP + XDP Nucleosido monofosfato quinasa Nucleosido difosfato quinasa Formación de CTP a partir de UTP Síntesis de Novo de PURINAS NH3 Fosforribosil aminotransferasa PRPP 7 8 Formación de AMP y GMP a partir de IMP Adenilsuccinato sintetasa Guanilato sintetasa Vías de RECUPERACIÓN -Vía alternativa para aprovechar las bases ya formadas y disminuir el gasto de energía intracelular -Requieren R i fosforribosil f f ib il pirofosfato i f f t (PRPP) y enzimas i específicas ífi Adenina + PRPP Adenilato (AMP) + PPi Guanina + PRPP Guanilato (GMP) + PPi Hipoxantina + PRPP Uracilo + PRPP Inositato (IMP) + PPi Uridilato (UMP) + PPi Adenina fosforribosiltransferasa Hipoxantina-guanina fosforribosiltransferasa Pirimidina fosforribosiltransferasa (solo incorpora uracilo) UTP CTP SÍNTESIS DE DESOXIRRIBONUCLEÓTIDOS - Se forman por reducción del C2´ de Ribonucleótidos di o trifosfato. - La reducción es catalizada por la ribonucleótido reductasa - Intervienen radicales que se originan en residuos de la enzima -El reductor final es el NADPH PASOS: 1- Transferencia de un e- del residuo cisteína de R1 al tirosilo de R2 2- El radical de cisteína toma un H del C3´ formando un radical 3- El radical en C3´provoca la eliminación del OH del C2´ 4- Se forma un puente disulfuro entre otros dos residuos cisteina y se dona un H al C2´ 5- El radical del C3´recupera el H de la cisteina y el desoxirribonucleótido se libera 6- El puente disulfuro se reduce por NADPH y la enzima vuelve a estar activa Formación de TMP a partir de dUMP Timidilato sintasa Muchos anticancerígenos bloquean la síntesis de TMP Fluoroacilo (F-dUMP): -No permite la extracción de un H+ del C5 -La actividad de la enzima provoca un complejo covalente: F-dUMP-metilentetrahidrofolato-enzima Inhibición suicida (la enzima misma genera al inhibidor a partir del sustrato) Inhibidores competitivos de la dihidrofolato reductasa Complejo inhibidor formado por la timidilato sintasa en presencia de F-dUMP Control de Biosíntesis por retroinhibición PIRIMIDINAS La enz. aspartato transcarbamilasa es inhibida por CTP (producto final) y es activada por ATP Control de Biosíntesis por retroinhibición PURINAS Síntesis de NAD+ (Nicotinamida adenina dinucleótido) Nicotinato + PRPP adenina nicotinamida Síntesis de FAD (flavina adenina dinucleótido) Riboflavina bo a a + ATP Riboflavina-5-fosfato + ATP Riboflavina-5-fosfato bo a a 5 os ato + ADP Flavina adenina dinucleótido + PPi adenina riboflavina Degradación de purinas a urato - El urato se excreta en la orina - Su nivel elevado provoca gota, que afecta a las articulaciones y a los riñones - La enfermedad se trata con inhibidores de la xantina oxidasa ÁCIDOS NUCLEICOS (Fosfo-ribosilpirofosfato) Adenosina trifosfato (Purina) Uridina trifosfato (Pirimidina) Síntesis de novo: Pirimidinas Purinas (inositato) Pirimidinas: ribosa incluida al final Purinas: el anillo crece sobre una estructura que contiene ribosa REPLICACIÓN DEL ADN Generalidades Estructurales -Doble hélice de hebras antiparalelas -Bases hacia el interior, Ribosas y P hacia el exterior - A-T (2 ptes H) y G-C (3 ptes H) p estructurales: -3 tipos A, B y Z Helicasa - Separa ambas hebras de ADN - Cambios conformacionales de la enzima e hidrólisis de ATP → desestabilizan la doble cadena. ADN Polimerasa -Añade nucleótidos al extremo 3` ((crece de 5`a 3`)) -Necesita un cebador -La base correcta se determina por capacidad de formar puentes H -La p polimerasa p posee un dominio p polimerasa y otro exonucleasa → corrección de errores 5` 3` 5` 3` Topoisomerasas A- topoisomerasa p I: corta una hebra de ADN superenrollado. p Relaja j la tensión g generada por superenrollamientos producidos por la separación de las hebras. B- Topoisomerasa II: introduce superenrollamientos, (ADN girasas) corta 2 hebras de ADN. Topoisomerasa II Topoisomerasa I dnaA: proteína de iniciación - Reconoce el origen de la replicación y comienza el desenrrollamiento. - Reconoce secuencias específicas p ((Ej. j locus ori C de E. coli)) Eucariotas: múltiples orígenes de replicación (cél. Humanas: varios cientos por cromosoma) SSB - Proteína que reconoce y se une a hebras simples → impide el reensamble de ambas hebras Primasa - ARN polimerasa que sintetiza el cebador → 5-6 nucleótidos de ARN - El cebador es luego g eliminado p por un dominio exonucleasa de la ADN p polimerasa. Ligasa - Responsable de la unión de fragmentos de Okazaki: trozos de ADN de la hebra retardada. - Cataliza uniones próximas OH-3`con P-5`. Recombinación del ADN - Intercambio de material genético g - incremento de la diversidad biológica - Intervienen las enzimas Recombinasas: ^ Intervienen 4 moléculas de enzima simultáneamente ^ Se g genera una unión de Holliday y entre 2 moléculas de ADN TRANSCRIPCIÓN Y MADURACIÓN DEL ARN Transcripción: pasaje de la información contenida en ADN hacia ARN Enzimas y Procesos que intervienen en la Transcripción en Procariotas (E.Coli) ARN Polimerasa: - 4 subunidades: αββ`σ - σ : localización de secuencia promotora - αββ`: unidad catalitica “núcleo de la enzima” - Secuencias promotoras: 5-6 bases en posición 10 y 35 antes del punto de iniciación →caracterización por footprinting g - Capaz de desenrollar ambas hebras de ADN (17 bases) - Sin cebador - “Burbuja de Transcripción”: ARN-Pol, ADN molde y cadena ARN creciente. - Sin activ. exonucleasa → mayor probabilidad de error - Señal de terminación: secuencia rica en GC con poli U al medio y al final de la misma. ARNm: no sufre modificaciones → la traducción comienza mientras se va sintetizando. ARNr y ARNt: se modifican por hidrólisis de ciertas secuencias/trozos y por modificación de bases (pseudouridilato en ARNt) Técnica de Footprinting Señal de Terminación Eucariotas -Transcripción y Traducción separadas en espacio y tiempo - 3 tipos de ARN polimerasas según producto de síntesis y localización -ARNm: altamente modificado - Cofia 5` 5 → GTP y Metilación de ribosas - Cola poli (A) (extremo 3`) - Maduración: “espliceosoma” (corte y ensamblado) - Modificaciones post trascripcionales: cambios en la secuencia del ARNm - 1 gen → varios ARNm → varias proteínas } Proteoma más complejo que Genoma ARN polimerasa I: Nucleolo – ARNr (sub. Mayor) ARN Polimerasa II: Nucleoplasma – ARNm ARN Polimerasa III: Nucleoplasma – ARNt y ARNr (sub. Menor)