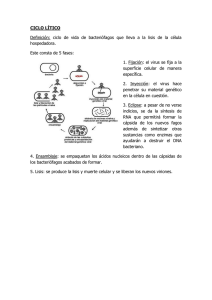
Imputación de datos faltantes en un modelo de tiempo de fallo
Anuncio

IMPUTACIÓN DE DATOS FALTANTES EN UN MODELO DE TIEMPO DE FALLO ACELERADO Autor: Moisés Castro Cacabelos Tutores: Francisco Gude Sampedro y Ana Pérez González Máster en Técnicas Estadı́sticas Julio 2014 El presente documento que tiene como tı́tulo “Imputación de datos faltantes en un modelo de tiempo de fallo acelerado” ha sido realizado por Moisés Castro Cacabelos como Trabajo Fin de Máster de Técnicas Estadı́sticas bajo la dirección de Francisco Gude Sampedro y Ana Pérez González, que autorizan la entrega del mismo. Fdo.: Francisco Gude Sampedro Fdo.: Ana Pérez González Índice general Resumen 7 1. Introducción y objetivo 9 2. Datos faltantes 11 2.1. Modelos de datos faltantes . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 11 2.2. Principales métodos para tratar datos faltantes . . . . . . . . . . . . . . . . . . . . . . 11 2.2.1. Análisis de casos completos . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 11 2.2.2. Análisis de casos disponibles . . . . . . . . . . . . . . . . . . . . . . . . . . . . 12 2.2.3. Métodos de imputación . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 12 2.2.4. Máxima verosimilitud . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 15 2.3. Principales paquetes de R que implementan técnicas de datos faltantes . . . . . . . . . 15 2.4. Utilización de la librerı́a MICE para la imputación múltiple . . . . . . . . . . . . . . . 16 3. Análisis de supervivencia 21 3.1. Introducción . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 21 3.2. Conceptos básicos . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 22 3.3. Censura . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 22 3.4. Estimadores de la función de supervivencia . . . . . . . . . . . . . . . . . . . . . . . . 23 3.4.1. Kaplan-Meier . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 23 3.4.2. Actuarial . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 23 3.4.3. Nelson-Aalen . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 24 3.5. Modelos de supervivencia . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 24 3.5.1. Modelo de riesgos proporcionales de Cox . . . . . . . . . . . . . . . . . . . . . . 24 3.5.2. Modelo de tiempo de fallo acelerado (AFT) . . . . . . . . . . . . . . . . . . . . 25 5 6 ÍNDICE GENERAL 3.5.3. Comparación del modelo AFT respecto al de Cox . . . . . . . . . . . . . . . . . 26 4. Estudio de simulación 29 5. Aplicación a datos reales 39 5.1. Descripción del problema . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 39 5.2. Descripción del conjunto de datos . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 39 5.2.1. Descripción de la población y objetivo del estudio . . . . . . . . . . . . . . . . 39 5.2.2. Descripción de las variables . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 40 5.3. Imputación a los datos reales . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 42 5.4. Resultados del análisis de supervivencia . . . . . . . . . . . . . . . . . . . . . . . . . . 47 6. Conclusiones 49 Bibliografı́a 51 A. Abreviaturas 53 Resumen Es frecuente en estudios de supervivencia, al igual que en otros estudios biomédicos, que nos encontremos con datos faltantes. Este problema ha sido tratado en estudios en los que se han analizado los datos siguiendo modelos de riesgos proporcionales de Cox. Sin embargo, en modelos de supervivencia de tiempo de fallo acelerado, existe escasa información en la literatura en cuanto al tratamiento de este tipo de estudios. Este trabajo aborda el tratamiento de datos faltantes en modelos de tiempo de fallo acelerado con distribución lognormal, mediante una revisión de la literatura, para seguir con estudios de simulación y finalizar con la aplicación a un caso práctico sobre un estudio de supervivencia en pacientes que han sido sometidos a trasplante hepático. En el estudio de simulación se realiza una comparativa del comportamiento de diversos métodos de imputación múltiple. Para ello se utiliza una librerı́a de R denominada “mice”. De los resultados obtenidos se desprende que el comportamiento de los estimadores de los parámetros varı́a en función del mecanismo de imputación utilizado. De la aplicación a datos reales, siguiendo el criterio AIC para valorar los resultados del análisis con datos imputados, las imputaciones en las que se aplicaron los métodos “pmm” para variables continuas y “logreg” para variables binarias son las que han mostrado mejores resultados. Los factores de riesgo que se obtienen tras el análisis de supervivencia pueden cambiar sustancialmente en caso de que se analicen datos con casos completos o con datos imputados por los métodos anteriormente indicados. 7 Capı́tulo 1 Introducción y objetivo Los datos faltantes son un problema que surge con mucha frecuencia cuando un estadı́stico afronta un análisis de datos. Aparecen, por ejemplo, en el campo de la investigación médica, psicologı́a o estudios sociológicos, entre otros. Crean una dificultad añadida en la investigación cientı́fica debido a que la mayor parte de los procedimientos de análisis de datos existentes no están diseñados (o adaptados) para la ausencia de observaciones. Un manejo inadecuado de los datos faltantes puede conducir a un posterior análisis estadı́stico erróneo. Para el análisis de datos en estudios de supervivencia, uno de los modelos más utilizados es el de riesgos proporcionales de Cox. De hecho, podemos encontrar bibliografı́a abundante relativa a la asignación de datos faltantes en modelos de Cox. Sin embargo, en este trabajo estamos interesados en aplicar otros modelos de supervivencia, como es el caso del modelo de tiempo de fallo acelerado (AFT). A diferencia del modelo de Cox, en la literatura son escasas las referencias que nos acercan al tratamiento de datos faltantes cuando se pretende analizar los datos con modelos AFT. El objetivo de este trabajo es investigar los métodos de imputación de datos faltantes que mejor se adecúan al análisis de supervivencia de tiempo de fallo acelerado. Para ello, se realizarán simulaciones con imputación de datos faltantes en un modelo AFT y además, se aplicarán diferentes métodos de imputación de datos faltantes a un caso real. Para ello organizamos la memoria de la siguiente forma: en el capı́tulo 2 se exponen los diferentes métodos de imputación que se utilizan habitualmente, en el capı́tulo 3 se describe el modelo de supervivencia de tiempo de fallo acelerado (AFT), en el capı́tulo 4 se realiza un estudio de simulación para un modelo AFT con distribución lognormal, en el capı́tulo 5 se realiza un análisis descriptivo de los datos e imputación a los datos reales, en el capı́tulo 6 se muestran las principales conclusiones y en el capı́tulo 7 se referencia la bibliografı́a utilizada. 9 10 CAPÍTULO 1. INTRODUCCIÓN Y OBJETIVO Capı́tulo 2 Datos faltantes 2.1. Modelos de datos faltantes Rubin (1976) clasificó los modelos de pérdida de datos en tres tipos diferentes: MCAR, MAR y MNAR. Vamos a describir a continuación brevemente cada uno de ellos [1]. MCAR: una variable es MCAR (missing completely at random) si la probabilidad de pérdida de una observación para todos los individuos es la misma y no depende de las medidas de otras variables [2]. Por ejemplo, un tubo que contiene una muestra de sangre de un individuo es roto por accidente o un cuestionario de individuo se pierde accidentalmente [3]. MAR: una variable es MAR (missing at random) si la probabilidad de pérdida de la observación de un individuo depende de la información observada. Por ejemplo, si se hace un test de aptitud a unos alumnos y a los que superan una nota de corte establecida se les hace otro más difı́cil mientras que a los demás no, por tanto éstos tienen datos perdidos para la segunda variable y se debe a las observaciones de la primera. MNAR: una variable es MNAR (missing not at random) si la probabilidad de que la observación de un individuo esté perdida está relacionada con los valores perdidos. Por ejemplo, un caso MNAR es cuando en un cuestionario le preguntas a alguien por su renta anual y éste no contesta porque es muy alta. 2.2. 2.2.1. Principales métodos para tratar datos faltantes Análisis de casos completos El análisis de casos completos es una estrategia simple que podemos aplicar a cualquier análisis estadı́stico con observaciones faltantes. El analista descarta a todos los individuos que tenga valores faltantes en alguna de las variables seleccionadas inicialmente y luego procede con el análisis utilizando métodos estándar. La primera cuestión que hay que plantearse con este análisis es si la submuestra que se analiza es una muestra aleatoria de la muestra original. Si la pérdida es MCAR, entonces los resultados del análisis resultarán generalmente insesgados pero con la consiguiente pérdida de eficiencia. 11 12 CAPÍTULO 2. DATOS FALTANTES Hay que tener en cuenta que es muy raro que ocurra una pérdida MCAR en los datos del mundo real. Cuando tengamos una pérdida MAR (que la tendremos habitualmente para datos reales) el análisis de las observaciones completas puede producir estimaciones sesgadas [4]. 2.2.2. Análisis de casos disponibles El análisis de casos disponibles intenta mitigar la pérdida de datos eliminando casos en una base análisis por análisis. La aplicación prototipo de este análisis ocurre cuando un investigador utiliza un diferente subconjunto de casos para calcular cada elemento en análisis. Por ejemplo, para el cálculo de una matriz de correlaciones, el tamaño muestral para estimar la varianza de una variable no tiene porqué ser el mismo que el utilizado para otra variable o para alguna de las covarianzas. Sin embargo, este método no está limitado a correlaciones, y es común encontrar artı́culos de investigación publicados que informan de diferentes tamaños de muestra a través de análisis de regresión o una ANOVA. Utilizar tantos datos como sea posible es una buena idea, y es cierto que el análisis de casos disponibles tiende a ser más poderoso que el análisis de casos completos, particularmente cuando las variables en un conjunto de datos tienen bajas correlaciones. Sin embargo, las desventajas del análisis de casos disponibles limitan su utilidad. Consistente con el análisis de casos completos, el principal problema del análisis de casos disponibles es que requiere datos MCAR y puede producir estimaciones de parámetro distorsionadas cuando el supuesto no se sostiene. Sin embargo, el análisis de casos disponibles también tiene un número de problemas únicos. Por ejemplo, utilizar diferentes subconjuntos de casos plantea problemas sutiles con medidas de asociación. Para ilustrarlo, consideramos la siguiente fórmula para la covarianza muestral: bXY = σ X (xi − µ bX )(yi − µ bY ) N −1 . El análisis de casos disponibles utiliza el subconjunto de casos con datos completos en ambas variables X e Y para calcular la covarianza. La mayorı́a de los paquetes software utilizan la misma submuestra bX de los casos que tienen para calcular la media de las variables, pero también es posible calcular µ b datos en X y calcular µY de los casos que tienen datos en Y. Una cuestión similar surge cuando calculamos el denominador del coeficiente de correlación. bXY σ r=q . 2 σ bX bY2 σ Los paquetes software tı́picamente utilizan el subconjunto de casos con datos completos en X e Y para 2 y σ bX bY2 de submuestras separadas (por ejemplo, calcular las varianzas, pero otra opción es calcular σ 2 bX de los casos que tienen datos sólo en X). El último enfoque es problemático porque puede calcular σ producir valores de correlación que exceden de ±1 [5]. 2.2.3. Métodos de imputación Imputación por media incondicional La imputación por media incondicional es una estrategia que consiste en calcular la media muestral para cada una de las variables que tiene datos faltantes, y luego utilizar este valor para sustituir todos los valores faltantes que tiene la variable correspondiente. Esta estrategia no suele funcionar muy bien 2.2. PRINCIPALES MÉTODOS PARA TRATAR DATOS FALTANTES 13 cuando la pérdida de datos depende de otras variables. Sustituyendo estos valores faltantes por la media se reduce la varianza en la variable y causa estragos en las covarianzas y correlaciones. Además, no es fácil estimar los errores estándar. También hay que tener en cuenta que este método no es aplicable a variables cualitativas. Imputación por media condicional (regresión) Como su nombre indica, la imputación por regresión reemplaza valores faltantes con respuestas predichas de un modelo de regresión. En un análisis multivariante, los casos completos son utilizados para estimar un modelo de regresión donde la variable incompleta es la respuesta y las variables explicativas son algunas de las variables completas. El modelo de regresión estimado permite estimar respuestas predichas para los casos incompletos. Aunque la idea de tomar información de las variables completas es buena, la imputación por regresión también produce estimaciones de parámetro sesgadas. Sobre todo si utilizamos modelos paramétricos de regresión, por ejemplo regresión lineal. En tal caso, los valores imputados caerán en una lı́nea recta (en el caso de una sóla covariable) o un hiperplano en el caso d-dimensional. Esto implica que los casos con valores imputados tienen correlación igual a 1, y rellenar los datos con un conjunto de casos perfectamente correlacionados pueden sobreestimar la correlación total. Por ello en los últimos años se han publicado trabajos que realizan imputaciones a partir de modelos de regresión no paramétricos. Imputación por regresión estocástica La imputación por regresión estocástica también utiliza ecuaciones de regresión para predecir las variables incompletas a partir de las variables completas, pero requiere un paso adicional que consiste en aumentar cada predicción con un término residual distribuido mediante la distribución del error, generalmente una distribución normal. Añadir residuos a los valores imputados reestablece la pérdida de variabilidad de los datos y efectivamente elimina el sesgo asociado con los esquemas de imputación de regresión estándar. Con este método de imputación obtenemos estimaciones insesgadas de los parámetros bajo datos MAR. Imputación hot-deck La imputación hot-deck es una técnica que imputa los valores faltantes con puntuaciones de otros encuestados con caracterı́sticas similares. Originalmente se desarrolló para tratar datos faltantes de encuestas poblacionales, y el procedimiento tiene una larga historia en aplicaciones de estudio. La aplicación más tı́pica este método de imputación reemplaza cada valor faltante con un dibujo aleatorio de una submuestra de los encuestados que tienen puntuaciones similares en un conjunto de variables. El procedimiento hot-deck clasifica los encuestados en factores basados en caracterı́sticas demográficas tales como sexo, edad y estado civil. Observar que las variables no necesitan ser categóricas. La imputación hot-deck generalmente preserva las distribuciones univariantes de los datos y no atenúan la variabilidad de los datos rellenados al mismo grado que otros métodos de imputación. Sin embargo, este método de imputación no es muy apropiado para estimar medidas de asociación y puede producir estimaciones sesgadas de las correlaciones y los coeficientes de regresión. 14 CAPÍTULO 2. DATOS FALTANTES Last Observation Carried Forward Last observation carried forward es una técnica que requiere datos longitudinales. Imputa medidas repetidas con la observación que le precede. Esta estrategia se aplica a los casos que tienen datos perdidos permanentemente o de forma intermitente. También asume que los valores no cambian significativamente después de la última medida observada o durante el periodo intermitente donde faltan valores. Imputación múltiple El método de imputación múltiple consiste en realizar varias imputaciones de las observaciones faltantes para luego analizar los conjuntos de datos completados y combinar los resultados obtenidos para obtener una estimación final. El análisis de imputación múltiple está dividido en tres fases: fase de imputación, fase de análisis y fase de puesta en común. La fase de imputación crea múltiples copias de los conjuntos de datos (m), y cada una de ellas contiene diferentes estimaciones de los valores perdidos. Conceptualmente, este paso es una versión iterativa de la imputación por regresión estocástica, aunque sus fundamentos matemáticos se basan en muchas ocasiones en los principios de estimación bayesiana. El objetivo de la fase de análisis, como su nombre indica, es analizar los conjuntos de datos rellenados. Este paso aplica los mismos procedimientos estadı́sticos que un individuo hubiera utilizado si tuviera todos los datos. La única diferencia es que realizamos cada análisis m veces, una para cada conjunto de datos imputados. La fase de análisis nos lleva a m conjuntos de estimaciones de parámetros y errores estándar, con lo que el propósito de la fase de puesta en común es combinar todo en un conjunto simple de resultados. Rubin (1987) perfiló fórmulas relativamente sencillas para poner en común las estimaciones de los parámetros y los errores estándar. Por ejemplo, la estimación del parámetro puesto en común es simplemente la media aritmética de las m estimaciones de la fase de análisis. Combinar los errores estándar es ligeramente más complejo pero sigue la misma lógica. El proceso de analizar conjuntos de datos múltiples y poner en común los resultados parece latoso, pero los paquetes de software de imputación múltiple automatizan completamente el procedimiento. Las m estimaciones son combinadas en una estimación en conjunto y una matriz de varianzascovarianzas utilizando las reglas de Rubin, que están basadas en la teorı́a asintótica en un marco bayesiano [6]. La matriz de varianzas-covarianzas combinada incorpora la variabilidad dentro de la imputación (incertidumbre sobre los resultados de unos conjuntos de datos imputados) y la variabilidad entre las imputaciones (reflejando la incertidumbre debido a la información perdida). Supongamos que θbj es una estimación de una cantidad univariante o multivariante de interés (por ejemplo, un coeficiente de regresión) obtenida de los j-ésimos conjuntos de datos imputados y que Wj es la varianza estimada de θbj . La estimación combinada θb es la media de las estimaciones individuales: θb = m 1 X θbj . m j=1 La varianza total de θb está formada por la suma de la varianza dentro de la imputación W = 1 Pm 1 Pm b b2 j=1 Wj y la varianza entre las imputaciones B = m−1 j=1 (θj − θ) : m b =W + 1+ var(θ) 1 B. m 2.3. PRINCIPALES PAQUETES DE R QUE IMPLEMENTAN TÉCNICAS DE DATOS FALTANTES15 2.2.4. Máxima verosimilitud La idea de utilizar el método de máxima verosimilitud para tratar datos faltantes viene desde hace más de 50 años. Las primeras soluciones de máxima verosimilitud fueron de alcance limitado y tuvieron relativamente pocas aplicaciones prácticas. Muchos de los avances importantes se produjeron en los años 70 cuando se apuntalan las técnicas modernas de manejo de datos faltantes. La estimación por máxima verosimilitud extrae continuamente diferentes combinaciones de valores de los parámetros poblacionales hasta que identifica el particular conjunto de valores que produce el valor más alto del log-verosimilitud (es decir, el mejor ajuste para los datos). Conceptualmente, el proceso de estimación es el mismo con o sin datos faltantes. Sin embargo, los datos faltantes introducen algunos matices adicionales que no son relevantes para los análisis de casos completos. Los registros de los datos incompletos requieren una ligera alteración para el cálculo del log-verosimilitud de los individuos para acomodar el hecho de que los individuos ya no tienen el mismo número de observaciones. Los datos faltantes también necesitan un ajuste de los cálculos de los errores estándar. Finalmente, el análisis de datos faltantes suele requerir algoritmos de optimización iterativos, incluso para problemas de estimación muy simples. El algoritmo EM (Expectation-Maximization) es un algoritmo particularmente importante para el análisis de datos faltantes. Las primeras aplicaciones del algoritmo se enfocaron principalmente a la estimación de un vector de medias y una matriz de covarianzas con datos faltantes, pero se ha extendido el algoritmo para abordar una variedad de complicados problemas de estimación de datos completos. El algoritmo EM es un procedimiento iterativo de dos pasos: el paso de esperanza y el paso de maximización. Una de las aplicaciones más comunes es la estimación del vector de medias y la matriz de covarianzas. En este caso, el proceso iterativo comienza con una estimación inicial del vector de medias (µ) y de la matriz de covarianzas (Σ). El primer paso utiliza los elementos del vector de medias y la matriz de covarianzas para construir un conjunto de ecuaciones de regresión que predicen las variables incompletas de las variables observadas. El propósito de este paso es rellenar los valores faltantes de forma que parezca una imputación por regresión estocástica. El segundo paso aplica fórmulas de datos completos estándar a los datos rellenados para generar estimaciones actualizadas del vector de medias y la matriz de covarianzas. El algoritmo lleva las estimaciones del parámetro actualizadas hacia el primer paso de nuevo, donde se construye un nuevo conjunto de ecuaciones de regresión para predecir los valores faltantes. El segundo paso reestima el vector de medias y la matriz de covarianzas. El b no cambian, punto en el cual el b y Σ algoritmo EM repite los dos pasos hasta que los elementos µ algoritmo ha convergido en las estimaciones de máxima verosimilitud. Es importante reiterar que el algoritmo no imputa ni reemplaza los valores faltantes. Más bien, utiliza todos los datos disponibles para estimar el vector de medias y la matriz de covarianzas. 2.3. Principales paquetes de R que implementan técnicas de datos faltantes MissingDataGUI Este paquete proporciona resúmenes numéricos y gráficos para los datos faltantes de variables categóricas y cuantitativas. Se aplica una variedad de métodos de imputación, incluyendo imputaciones univariantes como valores fijos o aleatorios, imputaciones multivariantes como las vecinanzas más cercanas e imputación múltiple, e imputaciones condicionadas a una variable categórica [7]. 16 CAPÍTULO 2. DATOS FALTANTES Amelia II Amelia II imputa de forma múltiple datos faltantes en una sección cruzada única (como un estudio), de una serie de tiempo (como variables coleccionadas por un año en un paı́s), o de un conjunto de datos de sección cruzada de series de tiempo (tales como variables coleccionadas durante años para varios paı́ses). Amelia II implementa un algoritmo basado en bootstrap, por lo que generalmente es considerablemente más rápido que otros enfoques y puede manejar muchas más variables. A diferencia de Amelia I y otro software de imputación estadı́sticamente riguroso, virtualmente nunca se bloquea. Amelia II también incluye diagnósticos útiles del ajuste de modelos de imputación múltiple [8]. VIM Este paquete introduce nuevas herramientas para la visualización de valores faltantes y/o imputados, que pueden ser utilizados para explorar los datos y la estructura de los valores faltantes y/o imputados. Dependiendo de la estructura de los valores faltantes, los métodos correspondientes pueden ayudar a identificar el mecanismo generando los valores perdidos y permite explorar los datos incluyendo los valores faltantes. Además, la calidad de imputación puede ser visualmente explorada utilizando varios métodos gráficos univariantes, bivariantes y multivariantes. Un interfaz de usuario gráfico disponible en el paquete VIMGUI permite un fácil manejo de los métodos gráficos implementados [9]. MICE Hace imputación múltiple utilizando Fully Conditionally Specification (FCS) implementado por el algoritmo MICE (Multiple Imputation by Chained Equations). Cada variable tiene su propio modelo de imputación. Se proporcionan modelos de imputación incorporados para datos continuos (pmm), datos binarios (regresión logı́stica), datos categóricos no ordenados (regresión logı́stica politómica) y datos categóricos ordenados (odds proporcional). Se puede utilizar imputación pasiva para mantener consistencia entre las variables. Se dispone de varios gráficos de diagnóstico para examinar la calidad de las imputaciones [10]. 2.4. Utilización de la librerı́a MICE para la imputación múltiple Dado que el objetivo de nuestro trabajo es la aplicación de diferentes métodos de imputación múltiple a un conjunto de datos reales, pasamos a indicar las pautas necesarias para llevar a cabo este proceso [11]. La especificación del modelo de imputación es el paso más importante en imputación múltiple. El modelo de imputación deberı́a: Explicar el proceso que creó los datos faltantes Preservar las relaciones en los datos Preservar la incertidumbre sobre estas relaciones La idea es que la adhesión a estos principios producirá imputaciones adecuadas, y ası́ da lugar a inferencias estadı́sticas válidas. Necesitamos seguir los siguientes pasos: 1. Debemos decidir si el supuesto MAR es plausible. Las ecuaciones encadenadas [12] pueden manejar tanto datos MAR como MNAR. La imputación múltiple bajo datos MNAR requiere supuestos de modelado adicionales que influyen en las imputaciones generadas. 2.4. UTILIZACIÓN DE LA LIBRERÍA MICE PARA LA IMPUTACIÓN MÚLTIPLE 17 2. La segunda elección se refiere a la forma de imputación del modelo. La forma abarca la parte estructural y la distribución de error asumido. En el método de imputación de datos multivariantes Fully Conditional Specification (FCS), la forma necesita ser especificada para cada variable incompleta en los datos. La elección dependerá de la escala de la variable que se imputa, y preferiblemente incorpora información sobre la relación entre las variables. Actualmente se dispone de distintos paquetes del software libre R que pueden disponerse en la página web cran.r-project.org/ como MissingDataGUI, Amelia II o VIM. El paquete que nosotros vamos a utilizar para imputar datos faltantes en nuestro conjunto de datos reales es el MICE (Multiple Imputation by Chained Equations). El paquete MICE en R imputa datos multivariantes incompletos mediante ecuaciones encadenadas. El software MICE 1.0 apareció en el año 2000 como una librerı́a S-PLUS, y en 2001 como un paquete de R. MICE 1.0 introdujo selección de predictores, imputación pasiva y puesta en común automática. El MICE 2.9 extiende la funcionalidad del MICE 1.0 de varias formas. En el MICE 2.9, el análisis de los datos imputados está hecho de forma general, mientras el rango de modelos bajo el cual la puesta en común trabaja está sustancialmente extendido. MICE 2.9 añade una nueva funcionalidad para imputar datos con varios niveles, selección de predictores automática, manejo de datos, valores de post-procesamiento imputados, rutinas de puesta en común especializadas, herramientas de selección del modelo y gráficos de diagnóstico. La imputación de datos categóricos está mejorada para problemas derivados causados por la predicción perfecta. Se presta especial atención a las transformaciones, a la suma de las puntuaciones, a los ı́ndices e interacciones utilizando imputación pasiva, y a la configuración apropiada de la matriz predictora. El algoritmo MICE requiere una especificación de un método de imputación univariante separadamente para cada variable incompleta. El nivel de medida determina en gran parte la forma del modelo de imputación univariante. La función mice() en R, distingue variables numéricas, binarias, categóricas ordenadas y categóricas no ordenadas, y establece los valores por defecto. Tabla 2.1. Técnicas de imputación univariantes incorporadas. Método pmm norm norm.nob norm.predict mean logreg polyreg polr lda cart sample Descripción Predictive mean matching Regresión lineal bayesiana Regresión lineal no bayesiana Regresión lineal Imputación por media incondicional Regresión logı́stica Modelo logı́stico multinomial Modelo logı́stico ordenado Análisis lineal discriminante Árboles de clasificación y regresión Muestra aleatoria de los datos observados Tipo de escala Numérico Numérico Numérico Numérico Numérico Factor, 2 niveles Factor, > 2 niveles Ordenado Factor Cualquiera Cualquiera La tabla 2.1 contiene una lista de algunos de los métodos de imputación considerados en la librerı́a mice. El argumento “method” de mice() especifica el método de imputación. La función mice.impute.pmm() implementa predictive mean matching, un método de imputación semiparamétrico. Sus principales ventajas son que los valores imputados coinciden con alguno de los valores observados en la misma variable y que puede preservar relaciones no lineales incluso si la parte estructural del modelo de imputación es incorrecta. Es un buen método de imputación en general. Las funciones mice.impute.norm() y mice.impute.norm.nob() imputan de acuerdo a un 18 CAPÍTULO 2. DATOS FALTANTES modelo de imputación lineal, y son rápidas y eficientes si los residuos del modelo son casi normales. La función mice.impute.norm.predict()aplica una regresión lineal entre las variables. El método mice.impute.mean() simplemente imputa la media de los datos observados. La función mice.impute.logreg() imputa factores con dos niveles mediante el modelo de regresión logı́stica. La función mice.impute.polyreg() imputa factores con dos o más niveles por el modelo multinomial. La función mice.impute.polr() implementa el modelo logı́stico ordenado, también conocido como modelo odds proporcional. La función mice.impute.lda() utiliza el análisis lineal discriminante para calcular la probabilidad posterior de cada caso incompleto, y consecuentemente muestra imputaciones de estas posteriores. La función mice.impute.cart() imputa mediante un árbol de clasificación si la variable es categórica, y si la variable es continua aplica un árbol de regresión. Finalmente, la función mice.impute.sample() coge solamente una muestra aleatoria de los datos observados, e imputa éstos en lugar de los valores perdidos. Esta función no condiciona en ninguna otra variable. A la hora de elegir método de imputación, hay que tener en cuenta que con frecuencia las variables continuas no se distribuyen mediante una normal. El problema de imputar tales variables suponiendo normalidad es que la distribución de los valores imputados no se corresponde con los valores observados en el caso de no normalidad de las observaciones. Una forma de tratar la no normalidad es utilizando el predictive mean matching (nombrado anteriormente). El pmm es un método de imputación para valores perdidos con la propiedad de que los valores imputados obtenidos son valores observados de la variable. 3. Una tercera elección se preocupa sobre el conjunto de variables que se incluyen como predictores en el modelo de imputación. El consejo general es incluir tantas variables relevantes como sea posible, incluyendo sus interacciones. Esto puede, sin embargo, conducir a especificaciones del modelo difı́ciles de manejar. Una caracterı́stica útil del algoritmo MICE es la habilidad para especificar el conjunto de predictores a ser utilizados para cada variable incompleta. La especificación básica está hecha a través del argumento predictorMatrix, que es una matriz cuadrada de tamaño ncol(data) conteniendo ceros y unos. Cada fila en predictorMatrix identifica que predictores se van a utilizar para la variable correspondiente a esa fila. El valor 1 en un elemento de la matriz indica que la variable de la columna es predictora para imputar la variable objetivo (de la fila), y el 0 significa que no es utilizada. La configuración por defecto de predictorMatrix especifica que los datos faltantes de una variable son imputados utilizando el resto de variables del estudio. Condicionado a todos los demás datos suele ser razonable para pequeños o medianos conjuntos de datos, contener hasta 20-30 variables aproximadamente, sin variables derivadas, efectos de interacción y otras complejidades. Como regla general, utilizar toda la información disponible conduce a imputaciones múltiples que tienen sesgo menor y máxima eficiencia. Para conjuntos de datos que contienen cientos o miles de variables, utilizar todos los predictores puede no ser factible (a causa de la multicolinealidad y problemas computacionales). Para la imputación, es conveniente seleccionar un subconjunto apropiado de datos que no contiene más de 15 a 25 variables. Van Buuren et al (2011) proporciona la siguiente estrategia para seleccionar variables predictoras de una base de datos grande: a) Incluir todas las variables que aparecen en el modelo de datos completos, es decir, el modelo que será aplicado a los datos después de la imputación. De no hacerlo puede sesgar el análisis de datos completos, especialmente si el modelo de datos completos contiene fuerte relaciones predictivas. Observar que este paso es algo contrario a la intuición, como puede parecer esa imputación fortalecerı́a artificialmente las relaciones del modelo de datos completos, que serı́a claramente indeseable. Si se hace correctamente, sin embargo, este no es el caso. 2.4. UTILIZACIÓN DE LA LIBRERÍA MICE PARA LA IMPUTACIÓN MÚLTIPLE 19 Observar que las interacciones de interés cientı́fico también necesitan ser incluidas en el modelo de imputación. b) Además, incluir las variables que están relacionadas con la falta de respuesta. Los factores que se conoce que van a influir en la ocurrencia de datos faltantes (estratificación, razones para la falta de respuesta) deben incluirse por razones de fondo. Otras variables de interés son aquellas en las que las distribuciones difieren entre los grupos de respuesta y de no respuesta. Estos pueden ser encontrados inspeccionando sus correlaciones con el indicador de respuesta de la variable a ser imputada. Si la magnitud de esta correlación excede un cierto nivel, entonces la variable deberı́a ser incluida. c) Incluir también variables que explican una considerable proporción de la varianza. Tales predictores ayudan a reducir la incertidumbre de las imputaciones. Son básicamente identificados por sus correlaciones con la variable objetivo. d ) Quitar de las variables seleccionadas en los pasos b y c aquellas variables que tienen demasiados valores faltantes dentro del subgrupo de casos incompletos. Un simple indicador es el porcentaje de casos observados dentro de este subgrupo, el porcentaje de casos utilizables. La mayorı́a de los predictores utilizados para la imputación están incompletos. En principio, uno podrı́a aplicar los pasos de simulación citados para cada predictor incompleto a su vez, pero esto puede dar lugar a una cascada de problemas de imputación auxiliares. Al hacerlo, se corre el riesgo de que cada variable necesite ser incluida después de todo. En la práctica, hay a menudo un pequeño conjunto de variables clave, para las cuales las imputaciones se necesitan, que sugiere que todos los pasos anteriores se realicen sólo para las variables clave. Este fue el enfoque cogido en Van Buuren y Groothuis-Oudshoorn (1999), pero puede perder importantes predictores de los predictores. Una estrategia más seguras y eficiente, aunque más laboriosa, es realizar los pasos del modelo también para los predictores de los predictores de las variables clave. Esto está hecho en Groothuis-Oudshoorn (1999). Es raramente necesario ir más allá de los predictores de los predictores. En el nodo terminal, podemos aplicar un método simple como imputación por un valor aleatorio observado de la propia variable que no necesita predictores para ello. 4. La cuarta elección es si deberı́amos imputar variables que son funciones de otras variables (incompletas). Muchos conjuntos de datos contienen variables derivadas, suma de puntuaciones, variables de interacción, relaciones y ası́ sucesivamente. Puede ser útil incorporar las variables transformadas en el algoritmo de imputación múltiple. Con frecuencia hay una necesidad para las versiones transformadas, combinadas o recodificadas de los datos. En el caso de datos incompletos, uno podrı́a imputar el original y después transformar el original completo, o transformar el original incompleto e imputar la versión transformada. Sin embargo, si ambos (original y transformado) se necesitan dentro del algoritmo de imputación, ninguno de estos métodos trabaja porque no podemos estar seguros de la transformación que se da entre los valores imputados de las versiones originales y las transformadas. La librerı́a MICE implementa un mecanismo especial, llamado imputación pasiva, para tratar con tales situaciones. La imputación pasiva mantiene la consistencia entre diferentes transformaciones de los mismos datos. El método puede ser utilizado para asegurar que la transformación siempre depende de las imputaciones generadas más recientemente en los datos originales sin transformar. La imputación pasiva se invoca especificando una tilde (∼) como primer carácter del método de imputación. Esto proporciona un método simple para especificar una gran variedad de dependencias entre las variables, tales como las variables transformadas, recodificaciones, interacciones, suma de puntuaciones, y ası́ sucesivamente, que pueden ser necesarios en otras partes del algoritmo. 20 CAPÍTULO 2. DATOS FALTANTES 5. La quinta elección se preocupa del orden en que las variables deberı́an ser imputadas. La secuencia de visita puede afectar a la convergencia del algoritmo. El algoritmo MICE imputa por defecto columnas incompletas de datos de izquierda a derecha. Teóricamente, el esquema de visita es irrelevante siempre y cuando cada columna sea visitada lo suficiente, pero algunos esquemas son más eficientes que otros. En particular, para datos que faltan monotónicamente, la convergencia es inmediata si las variables están ordenadas de acuerdo al número de casos faltantes. Más que reordenar los datos, es más conveniente cambiar el esquema de visita del algoritmo mediante el argumento visitSequence. De forma básica, el argumento visitSequence es un vector de enteros de longitud igual al número de variables del conjunto de datos, especificando la secuencia de números de columna para una iteración del algoritmo. Cualquier columna dada puede ser visitada más de una vez dentro de la misma iteración, que puede ser útil para asegurar sincronizaciones propias entre variables. Es obligatorio que todas las columnas con datos faltantes que se utilizan como predictores sean visitadas, o sino el algoritmo se interrumpirá con un error. 6. La sexta elección se preocupa de la configuración de las imputaciones de partida y el número de iteraciones. 7. La séptima elección es m, el número de datos de imputación múltiple. Estableciendo un m demasiado bajo puede dar lugar a grandes errores de simulación e ineficiencia estadı́stica, especialmente si la fracción de información perdida es alta. Para fracciones de información perdida γ=(0.1, 0.3, 0.5, 0.7, 0.9) necesitamos establecer m=(20, 20, 40, 100, >100) imputaciones, respectivamente. Otros autores (Schafer (1997)) dicen que con pocas imputaciones (3-5) para un modelo univariante son suficientes. Tener en cuenta que estas elecciones siempre se necesitan. La imputación necesita hacer elecciones por defecto. Estas elecciones están destinadas a ser útiles a través de una amplia gama de aplicaciones. Sin embargo, las elecciones por defecto no son necesariamente las mejores para los datos. Simplemente, no hay un ajuste mágico que siempre trabaja, por lo que a menudo se necesita alguna adaptación. Capı́tulo 3 Análisis de supervivencia 3.1. Introducción En muchos estudios, especialmente los relacionados con la medicina, la variable principal que queremos valorar es el tiempo que transcurre hasta un suceso. A este tiempo, normalmente se le llama “supervivencia”, aunque el suceso en cuestión no tiene por qué ser la defunción. Cuando el suceso que queremos evaluar ocurre en todos los pacientes, entonces disponemos de varias técnicas estadı́sticas que pueden aplicarse. Sin embargo, lo más habitual es que el suceso en cuestión (la defunción, la recidiva, la reaparición de los sı́ntomas) no se presente en todos los individuos. Además, comprobar la distribución deberı́a ser el primer paso antes de aplicar cualquier prueba, y es muy frecuente que estos datos de supervivencia no sigan la distribución normal, con lo cual la mayorı́a de las pruebas estadı́sticas no son aplicables. En esta situación, hay una serie de técnicas estadı́sticas (análisis de la supervivencia) apropiadas para estudios en los que cada paciente es seguido durante un determinado perı́odo y en los que se recoge el intervalo que transcurre entre el hecho inicial y el hecho final, o hasta que acaba el seguimiento si no ocurre el hecho final. Además, entre estas técnicas, disponemos de pruebas para comparar curvas de supervivencia, y modelos más complejos basados en la regresión que permiten valorar el efecto de un conjunto de valores pronósticos [13]. En ciencias de la salud, el auge de estas técnicas empieza hacia los años setenta. La ventaja que ofrecen estas técnicas y lo que las ha popularizado es que permiten generalizar el análisis de respuestas binarias (sı́/no; fallecido/vivo), incluido el tiempo de seguimiento, es decir, el tiempo que ha transcurrido desde el inicio del seguimiento hasta producirse la respuesta o hasta el final del seguimiento si la respuesta no se ha producido. Además, este tiempo que se analiza se puede valorar en condiciones muy flexibles, porque la duración del perı́odo de observación puede ser muy diferente para cada sujeto. Ası́ pues, el análisis de la supervivencia es una técnica muy apropiada para analizar respuestas binarias en estudios longitudinales o de seguimiento que se caractericen por: 1. Duración variable del seguimiento: los estudios de seguimiento tienen fechas muy bien definidas de inicio y de cierre, pero los sujetos se incorporan al estudio en momentos diferentes. 2. Observaciones incompletas: en la fecha de cierre del estudio aún no se ha producido el evento terminal en ciertos sujetos (sujetos retirados “vivos”). Además, puede haber pérdidas (sujetos perdidos). Estas observaciones incompletas dan lugar a lo que se llama “datos censurados”, y el análisis de supervivencia se caracteriza por incluir la información que aportan estos datos. 21 22 3.2. CAPÍTULO 3. ANÁLISIS DE SUPERVIVENCIA Conceptos básicos Función de supervivencia La función de supervivencia se define como la probabilidad de que una persona sobreviva (no le ocurra el evento de interés) al menos hasta el tiempo t. Una definición más formal puede darse de la siguiente manera: sea T una variable aleatoria positiva (o no negativa) con función de distribución F(t) y función de densidad de probabilidad f(t). La función de supervivencia S(t) es: S(t) = 1 − F (t) = P [T > t] Función de riesgo La función de razón de riesgos o tasa instantánea de fallas λ(t) se define como el cociente entre la función de densidad y la función de supervivencia: λ(t) = f (t) . S(t) Se interpreta como la probabilidad de que a un individuo le ocurra el evento de interés en la siguiente unidad de tiempo ∆t dado que ha sobrevivido hasta el tiempo t [14]. 3.3. Censura En estudios longitudinales, sólo se conoce el tiempo exacto de supervivencia para los individuos que muestran el evento de interés durante el perı́odo de seguimiento. Para los otros (los que están libres de la enfermedad al final del perı́odo de observación o los que se perdieron) todo lo que podemos decir es que no mostraron el evento de interés durante el perı́odo de seguimiento. En esta situación tenemos lo que se llaman observaciones censuradas. Podemos distinguir tres tipos de censura: Censura por la derecha: un sujeto está censurado por la derecha si lo que se sabe es que le ocurre el evento de interés algún tiempo después del perı́odo fijado de seguimiento. Censura por la izquierda: un sujeto está censurado por la izquierda si lo que se sabe es que le ocurre el evento de interés algún tiempo antes del perı́odo fijado de seguimiento. Censura por intervalos: un sujeto está censurado por intervalos si lo que se sabe es que le ocurre el evento de interés entre dos instantes, pero el tiempo de fallo exacto no se conoce. En la figura 3.1 mostramos gráficamente tiempos de vida censurados de las tres formas. La “X” indica muerte y la “O” censura. El primer individuo muere el dı́a 7. El segundo individuo no muere durante el periodo de estudio y hay censura por la derecha el dı́a 12. El tercer individuo no muere durante el periodo de observación y es censurado el dı́a 10. El cuarto individuo es censurado por intervalos: es observado de forma intermitente y muere en algún momento entre los dı́as 6 y 7. El quinto individuo es censurado por la izquierda, porque se ve que en el dı́a 1 ha muerto cuando entra en el estudio [15]. 3.4. ESTIMADORES DE LA FUNCIÓN DE SUPERVIVENCIA 23 Figura 3.1: Representación de los distintos tipos de censura. 3.4. 3.4.1. Estimadores de la función de supervivencia Kaplan-Meier El método de Kaplan-Meier se basa en los tiempos de supervivencia individuales y asume que la censura es independiente del tiempo de supervivencia. El estimador de Kaplan-Meier de supervivencia en el tiempo t se define mediante la siguiente ecuación: b S(t) = Y r(ti ) − d(ti ) ti ≤t r(ti ) . donde ti , i=1,2,...,n es el conjunto total de tiempos de fallo registrados, r(ti ) es el número de individuos en riesgo y d(ti ) el número de muertes (o de ocurrencia del evento de interés) en el momento ti . 3.4.2. Actuarial El método actuarial (también conocido como tabla de la vida) es una aproximación del estimador de Kaplan-Meier. Se basa en tiempos de supervivencia agrupados y es adecuado para conjuntos de datos grandes. El método actuarial supone que los sujetos se extraen aleatoriamente dentro de cada intervalo, por tanto, en promedio, se extraen en mitad del intervalo. Esto no es importante cuando los intervalos de tiempo son cortos, pero puede haber sesgos cuando los intervalos de tiempo son largos. Este método 24 CAPÍTULO 3. ANÁLISIS DE SUPERVIVENCIA también supone que la tasa de fallo dentro de un intervalo es el mismo para todos los sujetos y es independiente de la probabilidad de supervivencia en otros perı́odos de tiempo. Las tablas de la vida se producen de un estudio de la población a grande escala y se utilizan con menos frecuencia en estos dı́as (se prefiere Kaplan-Meier porque es menos propenso al sesgo). 3.4.3. Nelson-Aalen El riesgo instantáneo se define como la proporción de la población presente en el tiempo t que falla por unidad de tiempo. El riesgo acumulativo en el tiempo t, H(t), es riesgo sumado para todos los tiempos hasta t. La relación entre el riesgo acumulativo y la supervivencia es la siguiente: H(t) = −ln[S(t)], S(t) = e−H(t) o El estimador de Nelson-Aalen del riesgo acumulativo en el tiempo t se define como: b H(t) = X d(ti ) ti ≤t r(ti ) . La estimación de Flemington-Harrington de supervivencia se puede calcular utilizando la estimación de Nelson-Aalen del riesgo acumulativo utilizando la relación entre la supervivencia y el riesgo acumulativo descrita anteriormente. 3.5. 3.5.1. Modelos de supervivencia Modelo de riesgos proporcionales de Cox Este modelo es el más utilizado para datos de supervivencia en la medicina. En este modelo, definiremos el riesgo para el i-ésimo individuo de la siguiente forma: 0 λ(t; Zi (t)) = λ0 (t)eβ Zi (t) donde Zi (t) es el vector de covariables para el i-ésimo individuo en el tiempo t, λ0 (t) es la función de riesgo basal y β es el vector de parámetros. El cociente entre el riesgo para dos sujetos con el mismo vector de covariables es constante en el tiempo, es decir: 0 0 λ(t; Zi (t)) λ0 (t)eβ Zi (t) eβ Zi (t) 0 = = = eβ (Zi (t)−Zj (t)) 0 0 λ(t; Zj (t)) λ0 (t)eβ Zj (t) eβ Zj (t) Suponiendo que haya una muerte en el tiempo t*, la verosimilitud de que la muerte le ocurra al individuo i-ésimo y no a otro individuo es: 0 ∗ 0 ∗ λ0 (t∗ )eβ Zi (t ) eβ Zi (t ) Li (β) = P = . P 0 ∗ ∗ ∗ β Zj (t ) ∗ β 0 Zj (t∗ ) j Yj (t )λ0 (t )e j Yj (t )e 3.5. MODELOS DE SUPERVIVENCIA 25 Al producto de los términos de la última expresión L(β) = Q Li (β) se le llama verosimilitud parcial. La maximización de log(L(β)) da una estimación para β sin necesidad de estimar el parámetro de ruido o función de riesgo basal λ0 (t). Una extensión del modelo de Cox permite obtener la estimación de los modelos para distintos grupos disjuntos o estratos. El modelo obtenido se conoce como modelo de Cox estratificado y está definido para el estrato j-ésimo como: 0 λ(t; Zi (t)) = λj (t)eβ Zi (t) Este modelo permite obtener la estimación del modelo en presencia de una variable de estratificación sobre la cual se desean obtener funciones de supervivencia por cada uno de los distintos grupos y probablemente poder estudiar la existencia o no de las funciones de supervivencia entre los grupos. El modelo de Cox estratificado también constituye una de las maneras de corregir el modelo de Cox cuando no se cumple el supuesto de riesgos proporcionales para alguna de las covariables. En este caso suele correrse el modelo estratificando por la covariable que no cumple con el supuesto de riesgo proporcional. Este procedimiento permite corregir el sesgo en la estimación del parámetro que se puede presentar cuando se viola el supuesto de riesgo proporcional. Sin embargo, presenta una desventaja y es que no existe ningún β que permita estimar el efecto de la covariable de estratificación. 3.5.2. Modelo de tiempo de fallo acelerado (AFT) El modelo es el siguiente: log(Ti ) = β0 + β1 xi1 + β2 xi2 + ... + βp xip + σεi donde εi es el término de error aleatorio, β0 ,...,βp los parámetros de regresión y σ el parámetro de escala. Si no hay datos censurados, podemos fácilmente estimar este modelo mediante mı́nimos cuadrados ordinarios (OLS). Simplemente generamos una nueva variable, Y = log T, y utilizamos el modelo de regresión lineal con Y como variable dependiente. Este proceso conduce a mejores estimaciones insesgadas lineales de los coeficientes, sin suponer ninguna distribución en ε. Si ε es normal, las estimaciones OLS serán también estimaciones de máxima verosimilitud (MLE) y tendrán mı́nima varianza entre todos los estimadores, tanto lineales como no lineales. Pero los datos de supervivencia suelen tener observaciones censuradas, y éstas son difı́ciles de manejar con OLS. De forma alternativa, podemos utilizar MLE suponiendo diferentes distribuciones en ε. Para cada una de las distribuciones de ε, hay una distribución correspondiente para T (tabla 3.1). Observar que todos los modelos AFT se nombran para la distribución de T en lugar de la distribución de ε o log(T). La razón de que se permita suponer diferentes distribuciones es que tienen diferentes implicaciones para las formas de la función de riesgo [16]. 26 CAPÍTULO 3. ANÁLISIS DE SUPERVIVENCIA Tabla 3.1. Correspondencias de distribuciones entre ε y T. Distribución de ε Valor extremo (2 parámetros) Valor extremo (1 parámetro) Log-gamma Logı́stica Normal Distribución de T Weibull Exponencial Gamma Log-Logı́stica Log-Normal AFT con datos faltantes En la práctica, suele haber un gran número de posibles factores pronóstico asociados con los resultados. Una forma de reducir el número de factores antes de intentar un análisis multivariante, es examinar la relación entre cada factor individual y la variable dependiente (por ejemplo, tiempo de supervivencia). Del análisis univariante, los factores que tienen poco o ningún efecto en la variable dependiente pueden ser excluidos del análisis multivariante. Sin embargo, serı́a deseable incluir factores que dicen tener valores de pronóstico por otros investigadores y factores que se consideran importantes desde el punto de vista biomédico. Suele ser útil considerar los métodos de selección del modelo para escoger estos factores significantes de entre todos los posibles y determinar un modelo adecuado con tan pocas variables como sea posible. Con frecuencia, una variable con valor de pronóstico significante en un estudio no es importante en otro. Por tanto, la confirmación en un estudio posterior es muy importante para identificar factores de pronóstico. Otro problema frecuente en análisis de regresión es el de datos faltantes. Se puede hacer tres distinciones sobre los datos faltantes: (1) variables dependientes frente a variables independientes, (2) muchos datos faltantes frente a pocos datos faltantes, y (3) pérdida de datos aleatoria frente a la no aleatoria. Si el valor de la variable dependiente (por ejemplo, tiempo de supervivencia) es desconocido, poco más hay que hacer que quitar a ese individuo del análisis y reducir el tamaño muestral. El problema de datos faltantes es de diferente magnitud dependiendo de cómo de grande sea la proporción de datos faltantes, ya sea para variables dependientes o para variables independientes. El problema, obviamente, es menos crı́tico si falta el 1 % de los datos para una variable independiente que si falta el 40 % de los datos para varias variables independientes. Cuando una proporción pequeña de sujetos tiene datos faltantes para una variable, podemos optar simplemente por eliminarlos y realizar el análisis con los otros individuos de la muestra. Es difı́cil especificar cómo de grande o cómo de pequeño, pero eliminar 10 o 15 de cientos de casos no plantearı́a una objeción práctica seria. Sin embargo, si la falta de datos se da en una gran proporción de personas y el tamaño muestral no es amplio, una cuestión de aleatoriedad puede ser elevada. Si la muestra con datos faltantes no muestra diferencias significantes en la variable dependiente, el problema no es serio. Si los datos son MNAR, los resultados obtenidos de los sujetos eliminados serán engañosos. Por lo que eliminar casos no siempre es una solución adecuada al problema de datos faltantes [17]. 3.5.3. Comparación del modelo AFT respecto al de Cox El modelo de riesgos proporcionales de Cox se utiliza principalmente en los campos de la medicina y la bioestadı́stica, mientras que el modelo AFT se utiliza principalmente en fiabilidad y experimentos industriales [18]. El modelo de riesgos proporcionales de Cox tiene la ventaja de que puede estimar y hacer inferencia sobre los parámetros de interés sin asumir ninguna forma para la función de riesgo basal, o lo que 3.5. MODELOS DE SUPERVIVENCIA 27 es lo mismo, no es necesario especificar una distribución de supervivencia para modelar el efecto de las variables explicativas sobre la variable de duración. Sin embargo, este modelo está basado en la suposición de riesgos proporcionales y esto puede no sostenerse en algunos estudios de supervivencia. De ser ası́, el modelo de Cox estándar no se deberı́a utilizar y puede producir un sesgo importante al estimar o hacer inferencia sobre el efecto de un factor de pronóstico dado en la mortalidad. Por otra parte, si consideramos los modelos AFT, pueden ser de interés debido a que pueden ser reescritos especificando una relación directa entre el logaritmo del tiempo de supervivencia y las variables explicativas, al igual que un modelo de regresión lineal múltiple. Sin embargo, su principal desventaja es que habitualmente la estimación de estos modelos se lleva a cabo asumiendo una distribución para la duración, que en la mayorı́a de los casos es desconocida. Además, este método tiene varias ventajas respecto al modelo de Cox: No necesita asumir riesgos proporcionales Modela directamente el efecto de las variables explicativas en la supervivencia, por lo que la interpretación de los resultados es más fácil que en los modelos de riesgos proporcionales, donde modelamos el efecto de las covariables en una probabilidad condicionada. Además, utilizando esta metodologı́a podrı́amos estimar la media residual del tiempo de vida de un paciente que ya ha sobrevivido hasta el tiempo t 28 CAPÍTULO 3. ANÁLISIS DE SUPERVIVENCIA Capı́tulo 4 Estudio de simulación En este capı́tulo hemos simulado el siguiente modelo de supervivencia de tiempo de fallo acelerado con observaciones faltantes en alguna de las covariables: T = exp(β0 + β1 x1 + β2 x2 + β3 x3 + σε) El objetivo del estudio es comparar el comportamiento de los estimadores sobre la submuestra completa y sobre las muestras imputadas por diversos métodos. Para ello, hemos optado por diversos métodos de imputación múltiple utilizando la librerı́a MICE que comentábamos en la sección 2.4. Para poder realizar la comparación vamos a considerar distintas medidas de error: Error cuadrático medio βj = sesgo2 (βj ) + var(βj ) Error relativo absoluto βj = Error global βj = 1 500 P500 1 500 j=1 (βj P500 β̂j −βj j=1 βj − β̂j )2 Generamos 500 muestras de tamaño 100 para un modelo AFT. Consideramos tres covariables que provienen de una normal estándar multivariante. Asignamos distintos porcentajes de censura por la derecha (0 %, 15 % y 25 %) y distintas covarianzas entre la primera y segunda variable predictora (0, 0.25, 0.5 y 0.9). La tercera variable predictora es incorrelada con las dos primeras. En el caso de datos completos, obtenemos los siguientes errores globales, errores cuadráticos medios y errores relativos absolutos asignados a cada estimador (tabla 4.1). Lo que podemos ver aquı́ es que los errores son más altos cuanto mayor sea la censura, como era de esperar. Los errores cuadráticos medios y relativos absolutos asignados a β1 y β2 aumentan considerablemente cuando la covarianza entre las variables correspondientes es muy alta. Para simular la pérdida de datos, generaremos dos funciones donde los valores obtenidos serán la probabilidad de observación de los datos de las covariables. Por simplicidad, hemos supuesto que la pérdida depende sólo de la primera covariable. Sea δ1 la variable indicadora de si la covariable x1 es observada o no, es decir, δ1 = 1 si x1 es observada y δ1 = 0 en otro caso. Los modelos de datos faltantes considerados en este estudio de simulación han sido los siguientes: P1 : P (X1 sea observada) = P (δ1 = 1/x1 , x2 , x3 ) = 29 1 . 1 + exp(−1.5x2 ) 30 CAPÍTULO 4. ESTUDIO DE SIMULACIÓN Tabla 4.1. Errores global, cuadrático medio y relativo absoluto para datos completos. cov 0 0.25 0.5 0.9 0 0.25 0.5 0.9 0 0.25 0.5 0.9 cens 0 0 0 0 0.15 0.15 0.15 0.15 0.25 0.25 0.25 0.25 error 0.042 0.041 0.041 0.042 0.048 0.052 0.053 0.051 0.059 0.066 0.065 0.066 mseβ0 0.011 0.011 0.011 0.011 0.012 0.013 0.013 0.012 0.015 0.017 0.017 0.017 mseβ1 0.010 0.010 0.013 0.053 0.011 0.012 0.015 0.058 0.012 0.015 0.018 0.067 mseβ2 0.010 0.012 0.015 0.056 0.011 0.014 0.017 0.060 0.014 0.017 0.020 0.071 mseβ3 0.010 0.010 0.010 0.011 0.014 0.015 0.015 0.014 0.019 0.020 0.019 0.019 areβ0 0.041 0.041 0.041 0.041 0.043 0.045 0.046 0.044 0.048 0.053 0.052 0.051 P2 : P (X1 sea observada) = P (δ1 = 1/x1 , x2 , x3 ) = areβ1 0.081 0.079 0.089 0.181 0.084 0.087 0.097 0.191 0.086 0.094 0.104 0.205 areβ2 0.041 0.043 0.048 0.094 0.043 0.047 0.052 0.098 0.047 0.052 0.057 0.106 areβ3 0.027 0.027 0.027 0.028 0.031 0.031 0.031 0.031 0.037 0.037 0.036 0.036 1 . 1 + exp(−1.5x22 ) Podemos ver en las figuras 4.1 y 4.2 las representaciones gráficas de los modelos anteriores. 1 La primera función será P (δ1 = 1/xi1 , x2 , x3 ) = p1 (x1 , x2 , x3 ) = 1+exp(−1.5x que aparece repre2) 1 sentada en la figura 4.1, y la segunda es P (δ1 = 1/xi1 , x2 , x3 ) = p2 (x1 , x2 , x3 ) = 1+exp(−1.5x 2 ) que 2 está representada en la figura 4.2. Vamos a ver en las tablas 4.2 y 4.3 los distintos errores obtenidos para el estimador simplificado, utilizando sólo la submuestra completa y descartando aquellas observaciones que tienen algún valor perdido. Los resultados figuran según la función de probabilidad de observación, además de los distintos porcentajes de censura y covarianzas. Tabla 4.2. Errores global, cuadrático medio y relativo absoluto para 1 el estimador simplificado con p1 (x1 , x2 , x3 ) = 1+exp(−1.5x . 2) cov 0 0.25 0.5 0.9 0 0.25 0.5 0.9 0 0.25 0.5 0.9 cens 0 0 0 0 0.15 0.15 0.15 0.15 0.25 0.25 0.25 0.25 error 0.111 0.121 0.122 0.116 0.136 0.163 0.166 0.158 0.174 0.223 0.222 0.224 mseβ0 0.032 0.032 0.033 0.030 0.034 0.036 0.038 0.034 0.040 0.049 0.048 0.045 mseβ1 0.021 0.023 0.027 0.114 0.024 0.034 0.039 0.150 0.029 0.045 0.053 0.205 mseβ2 0.033 0.036 0.043 0.136 0.040 0.049 0.059 0.177 0.053 0.066 0.077 0.239 mseβ3 0.026 0.023 0.024 0.024 0.038 0.038 0.039 0.039 0.052 0.057 0.054 0.057 areβ0 0.070 0.071 0.070 0.068 0.073 0.076 0.075 0.072 0.080 0.087 0.086 0.083 areβ1 0.114 0.122 0.132 0.261 0.121 0.150 0.159 0.306 0.135 0.166 0.181 0.362 areβ2 0.072 0.075 0.082 0.142 0.081 0.088 0.096 0.164 0.092 0.102 0.111 0.191 areβ3 0.041 0.041 0.041 0.042 0.050 0.051 0.052 0.052 0.059 0.063 0.061 0.063 Al igual que para datos completos, lo que podemos ver aquı́ es que los errores son más altos cuanto mayor sea la censura. Los errores cuadráticos medios y relativos absolutos asignados a β1 y β2 son 31 1 1+exp(−1.5x2 ) . Figura 4.1: Función de probabilidad de observación p1 (x1 , x2 , x3 ) = Tabla 4.3. Errores global, cuadrático medio y relativo absoluto para 1 el estimador simplificado con p2 (x1 , x2 , x3 ) = 1+exp(−1.5x 2) . 2 cov 0 0.25 0.5 0.9 0 0.25 0.5 0.9 0 0.25 0.5 0.9 cens 0 0 0 0 0.15 0.15 0.15 0.15 0.25 0.25 0.25 0.25 error 0.059 0.058 0.056 0.057 0.068 0.074 0.073 0.071 0.084 0.093 0.091 0.092 mseβ0 0.016 0.016 0.016 0.016 0.017 0.019 0.019 0.019 0.022 0.026 0.026 0.026 mseβ1 0.014 0.016 0.019 0.075 0.016 0.020 0.024 0.086 0.018 0.025 0.029 0.107 mseβ2 0.012 0.014 0.018 0.076 0.013 0.017 0.021 0.085 0.016 0.021 0.026 0.106 mseβ3 0.018 0.015 0.014 0.015 0.022 0.021 0.020 0.020 0.029 0.026 0.025 0.025 areβ0 0.050 0.050 0.049 0.050 0.051 0.055 0.055 0.054 0.058 0.065 0.065 0.064 areβ1 0.094 0.100 0.110 0.221 0.102 0.114 0.124 0.236 0.109 0.122 0.133 0.256 areβ2 0.044 0.047 0.053 0.109 0.045 0.052 0.058 0.117 0.050 0.057 0.064 0.129 areβ3 0.034 0.033 0.032 0.032 0.039 0.039 0.038 0.037 0.045 0.044 0.042 0.043 considerablemente más altos cuando la covarianza entre las variables correspondientes es muy alta. Obtenemos mejores errores con la segunda función de pérdida. En cualquiera de los dos casos, los errores asignados a cada estimador son superiores si lo comparamos con el caso de datos completos. En el caso de datos imputados, utilizaremos distintos métodos de imputación en la primera variable y veremos cuál es el más efectivo. El primero que vamos a probar es el Predictive Mean Matching (pmm). Bajo los mismos escenarios considerados anteriormente, obtenemos los errores de las tablas 32 CAPÍTULO 4. ESTUDIO DE SIMULACIÓN Figura 4.2: Función de probabilidad de observación p2 (x1 , x2 , x3 ) = 1 . 1+exp(−1.5x22 ) 4.4 y 4.5. Tabla 4.4. Errores global, cuadrático medio y relativo absoluto para 1 datos imputados por PMM y con p1 (x1 , x2 , x3 ) = 1+exp(−1.5x . 2) cov 0 0.25 0.5 0.9 0 0.25 0.5 0.9 0 0.25 0.5 0.9 cens 0 0 0 0 0.15 0.15 0.15 0.15 0.25 0.25 0.25 0.25 error 0.265 0.279 0.238 0.110 0.292 0.319 0.276 0.128 0.322 0.360 0.311 0.153 mseβ0 0.029 0.028 0.022 0.014 0.029 0.030 0.024 0.015 0.032 0.036 0.029 0.020 mseβ1 0.260 0.265 0.275 0.416 0.282 0.299 0.314 0.471 0.305 0.330 0.346 0.528 mseβ2 0.028 0.051 0.114 0.398 0.032 0.061 0.134 0.444 0.036 0.070 0.151 0.496 mseβ3 0.023 0.022 0.019 0.013 0.029 0.031 0.028 0.018 0.039 0.041 0.036 0.024 areβ0 0.055 0.054 0.050 0.045 0.055 0.058 0.054 0.047 0.059 0.065 0.060 0.054 areβ1 0.460 0.468 0.481 0.573 0.479 0.495 0.512 0.606 0.495 0.514 0.533 0.635 areβ2 0.056 0.085 0.145 0.286 0.061 0.093 0.156 0.302 0.064 0.100 0.165 0.318 areβ3 0.035 0.034 0.033 0.029 0.039 0.042 0.039 0.034 0.047 0.048 0.045 0.040 En este caso, parece que los errores son más altos cuanto mayor sea la censura. Los errores cuadráticos medios y relativos absolutos asignados a β1 y β2 son considerablemente más altos cuando la covarianza entre las variables correspondientes es muy alta, mientras que los errores restantes en el mismo caso son más pequeños. Obtenemos mejores errores con la segunda función. Si comparamos este caso con la versión simplificada en la primera función, veremos que los errores asignados a β1 y β2 son más 33 Tabla 4.5. Errores global, cuadrático medio y relativo absoluto para 1 datos imputados por PMM y con p2 (x1 , x2 , x3 ) = 1+exp(−1.5x 2) . 2 cov 0 0.25 0.5 0.9 0 0.25 0.5 0.9 0 0.25 0.5 0.9 cens 0 0 0 0 0.15 0.15 0.15 0.15 0.25 0.25 0.25 0.25 error 0.123 0.130 0.112 0.058 0.134 0.148 0.131 0.070 0.148 0.167 0.147 0.086 mseβ0 0.019 0.018 0.016 0.012 0.020 0.022 0.019 0.014 0.024 0.028 0.025 0.019 mseβ1 0.108 0.114 0.119 0.175 0.114 0.125 0.132 0.195 0.118 0.131 0.139 0.217 mseβ2 0.014 0.021 0.044 0.153 0.016 0.025 0.050 0.168 0.019 0.029 0.056 0.190 mseβ3 0.019 0.019 0.016 0.012 0.024 0.025 0.022 0.016 0.031 0.034 0.028 0.021 areβ0 0.047 0.046 0.045 0.041 0.048 0.052 0.050 0.045 0.052 0.060 0.057 0.052 areβ1 0.280 0.291 0.296 0.322 0.285 0.302 0.309 0.336 0.286 0.304 0.311 0.350 areβ2 0.044 0.055 0.081 0.150 0.047 0.058 0.085 0.154 0.052 0.063 0.089 0.161 areβ3 0.032 0.032 0.030 0.028 0.036 0.037 0.035 0.032 0.042 0.043 0.040 0.037 altos en el caso de imputación, mientras que en los errores asignados a los otros estimadores y los globales, solemos obtener mejores resultados en el caso de imputación cuando la covarianza es muy alta. En la segunda función, los errores globales y los asignados a β1 y β2 obtenidos son mejores en la versión simplificada, mientras que los relativos asignados a los demás son mejores con la imputación y los cuadráticos medios asignados también a los otros sólo son mejores con la imputación en el caso en que la covarianza sea alta. Análogamente, obtenemos otras tablas de errores con los métodos sample (tablas 4.6 y 4.7), mean (tablas 4.8 y 4.9), norm.predict (tablas 4.10 y 4.11), cart (tablas 4.12 y 4.13) y norm (tablas 4.14 y 4.15). Estos dos últimos son métodos no paramétricos. Tabla 4.6. Errores global, cuadrático medio y relativo absoluto para 1 datos imputados por SAMPLE y con p1 (x1 , x2 , x3 ) = 1+exp(−1.5x . 2) cov 0 0.25 0.5 0.9 0 0.25 0.5 0.9 0 0.25 0.5 0.9 cens 0 0 0 0 0.15 0.15 0.15 0.15 0.25 0.25 0.25 0.25 error 0.296 0.325 0.343 0.192 0.326 0.370 0.383 0.206 0.360 0.415 0.420 0.224 mseβ0 0.019 0.023 0.031 0.023 0.019 0.024 0.031 0.024 0.023 0.031 0.034 0.028 mseβ1 0.286 0.303 0.373 0.765 0.310 0.342 0.417 0.795 0.336 0.376 0.451 0.821 mseβ2 0.022 0.065 0.205 0.745 0.026 0.075 0.226 0.764 0.030 0.084 0.243 0.787 mseβ3 0.021 0.021 0.019 0.013 0.027 0.030 0.026 0.018 0.036 0.038 0.034 0.023 areβ0 0.051 0.057 0.068 0.055 0.051 0.059 0.068 0.055 0.056 0.066 0.071 0.060 areβ1 0.501 0.518 0.582 0.857 0.522 0.548 0.613 0.872 0.541 0.572 0.635 0.883 areβ2 0.056 0.109 0.215 0.427 0.060 0.115 0.224 0.431 0.064 0.121 0.230 0.436 areβ3 0.035 0.035 0.034 0.030 0.040 0.042 0.040 0.035 0.048 0.048 0.045 0.040 Parece que ocurre algo muy similar con los errores obtenidos por estos últimos cinco métodos de imputación comparado con el “pmm”, excepto para el error global del método de imputación “mean” probado con la segunda función de probabilidad de observación, que también son más altos los errores cuanto mayor es la covarianza entre las dos primeras variables. Lo interesante aquı́ es ver cuáles son 34 CAPÍTULO 4. ESTUDIO DE SIMULACIÓN Tabla 4.7. Errores global, cuadrático medio y relativo absoluto para 1 datos imputados por SAMPLE y con p2 (x1 , x2 , x3 ) = 1+exp(−1.5x 2) . 2 cov 0 0.25 0.5 0.9 0 0.25 0.5 0.9 0 0.25 0.5 0.9 cens 0 0 0 0 0.15 0.15 0.15 0.15 0.25 0.25 0.25 0.25 error 0.128 0.143 0.162 0.141 0.140 0.163 0.183 0.155 0.154 0.182 0.200 0.170 mseβ0 0.016 0.016 0.015 0.012 0.017 0.020 0.019 0.014 0.021 0.026 0.024 0.019 mseβ1 0.112 0.127 0.186 0.613 0.118 0.139 0.202 0.634 0.122 0.146 0.210 0.646 mseβ2 0.014 0.025 0.074 0.531 0.016 0.029 0.082 0.545 0.020 0.033 0.088 0.560 mseβ3 0.018 0.018 0.017 0.013 0.023 0.025 0.023 0.017 0.030 0.032 0.029 0.023 areβ0 0.045 0.046 0.046 0.043 0.046 0.051 0.051 0.046 0.051 0.060 0.058 0.054 areβ1 0.290 0.314 0.389 0.754 0.296 0.325 0.404 0.765 0.298 0.328 0.406 0.768 areβ2 0.045 0.062 0.115 0.349 0.048 0.065 0.119 0.352 0.053 0.069 0.121 0.354 areβ3 0.032 0.033 0.032 0.030 0.036 0.037 0.036 0.034 0.042 0.043 0.041 0.039 Tabla 4.8. Errores global, cuadrático medio y relativo absoluto para 1 datos imputados por MEAN y con p1 (x1 , x2 , x3 ) = 1+exp(−1.5x . 2) cov 0 0.25 0.5 0.9 0 0.25 0.5 0.9 0 0.25 0.5 0.9 cens 0 0 0 0 0.15 0.15 0.15 0.15 0.25 0.25 0.25 0.25 error 0.078 0.128 0.243 0.230 0.089 0.161 0.285 0.260 0.110 0.196 0.325 0.291 mseβ0 0.021 0.036 0.069 0.046 0.021 0.034 0.063 0.045 0.024 0.038 0.061 0.046 mseβ1 0.021 0.026 0.047 0.486 0.025 0.035 0.056 0.509 0.029 0.046 0.071 0.543 mseβ2 0.018 0.046 0.143 0.649 0.022 0.062 0.178 0.690 0.026 0.075 0.205 0.732 mseβ3 0.017 0.017 0.016 0.013 0.022 0.026 0.023 0.017 0.030 0.034 0.030 0.023 areβ0 0.058 0.078 0.116 0.090 0.058 0.076 0.108 0.088 0.063 0.080 0.105 0.088 areβ1 0.115 0.127 0.174 0.666 0.124 0.150 0.188 0.675 0.137 0.168 0.212 0.692 areβ2 0.054 0.091 0.177 0.397 0.059 0.105 0.196 0.409 0.064 0.116 0.210 0.420 areβ3 0.035 0.034 0.033 0.030 0.039 0.042 0.040 0.035 0.046 0.048 0.045 0.040 los métodos de imputación con los que obtenemos mejores errores. En este caso son el “mean” y el “norm.predict”, pero con el primero en general no obtenemos mejores errores comparado con la versión simplificada mientras que con el segundo sı́. Con lo cual, el “norm.predict” es el mejor método de imputación que podremos aplicar a estos datos. Cabe destacar que cuanto mayor es la covarianza entre las variables, mejores errores obtenemos (como dijimos anteriormente), lo cual es lógico teniendo en cuenta que lo que hace este método es aplicar una regresión lineal. Nótese que el método norm.predict funciona bien en este modelo, en parte porque hemos simulado variables con distribución normal. En muchas ocasiones los datos reales no siguen una distribución Normal, por lo que métodos no paramétricos ofrecen generalmente un mejor comportamiento en ausencia de normalidad. 35 Tabla 4.9. Errores global, cuadrático medio y relativo absoluto para 1 datos imputados por MEAN y con p2 (x1 , x2 , x3 ) = 1+exp(−1.5x 2) . 2 cov 0 0.25 0.5 0.9 0 0.25 0.5 0.9 0 0.25 0.5 0.9 cens 0 0 0 0 0.15 0.15 0.15 0.15 0.25 0.25 0.25 0.25 error 0.054 0.056 0.057 0.070 0.062 0.072 0.073 0.083 0.076 0.091 0.090 0.101 mseβ0 0.014 0.014 0.014 0.012 0.015 0.017 0.017 0.013 0.018 0.023 0.022 0.018 mseβ1 0.014 0.016 0.021 0.181 0.016 0.020 0.026 0.195 0.019 0.025 0.031 0.213 mseβ2 0.012 0.014 0.022 0.189 0.013 0.017 0.026 0.202 0.016 0.020 0.031 0.218 mseβ3 0.014 0.014 0.013 0.012 0.018 0.020 0.018 0.016 0.024 0.027 0.024 0.021 areβ0 0.047 0.047 0.046 0.043 0.048 0.053 0.052 0.046 0.053 0.061 0.059 0.054 areβ1 0.095 0.100 0.117 0.369 0.102 0.114 0.130 0.381 0.108 0.122 0.141 0.394 areβ2 0.043 0.047 0.059 0.190 0.045 0.052 0.064 0.196 0.050 0.056 0.069 0.200 areβ3 0.032 0.032 0.031 0.029 0.035 0.037 0.035 0.033 0.042 0.043 0.041 0.039 Tabla 4.10. Errores global, cuadrático medio y relativo absoluto para 1 datos imputados por NORM.PREDICT y con p1 (x1 , x2 , x3 ) = 1+exp(−1.5x . 2) cov 0 0.25 0.5 0.9 0 0.25 0.5 0.9 0 0.25 0.5 0.9 cens 0 0 0 0 0.15 0.15 0.15 0.15 0.25 0.25 0.25 0.25 error 0.105 0.107 0.095 0.065 0.118 0.139 0.124 0.083 0.142 0.174 0.156 0.108 mseβ0 0.029 0.027 0.023 0.015 0.029 0.030 0.026 0.016 0.032 0.035 0.030 0.020 mseβ1 0.021 0.023 0.027 0.114 0.024 0.034 0.038 0.147 0.029 0.045 0.052 0.199 mseβ2 0.030 0.032 0.034 0.111 0.035 0.042 0.044 0.139 0.042 0.050 0.053 0.180 mseβ3 0.025 0.023 0.020 0.013 0.030 0.034 0.029 0.018 0.038 0.044 0.037 0.023 areβ0 0.067 0.065 0.060 0.048 0.066 0.069 0.063 0.050 0.070 0.075 0.069 0.056 areβ1 0.114 0.122 0.132 0.261 0.123 0.149 0.158 0.302 0.136 0.166 0.180 0.356 areβ2 0.068 0.070 0.073 0.130 0.072 0.081 0.084 0.147 0.079 0.089 0.093 0.169 areβ3 0.042 0.040 0.037 0.030 0.046 0.048 0.044 0.035 0.052 0.055 0.050 0.040 36 CAPÍTULO 4. ESTUDIO DE SIMULACIÓN Tabla 4.11. Errores global, cuadrático medio y relativo absoluto para 1 datos imputados por NORM.PREDICT y con p2 (x1 , x2 , x3 ) = 1+exp(−1.5x 2) . 2 cov 0 0.25 0.5 0.9 0 0.25 0.5 0.9 0 0.25 0.5 0.9 cens 0 0 0 0 0.15 0.15 0.15 0.15 0.25 0.25 0.25 0.25 error 0.055 0.057 0.053 0.047 0.063 0.072 0.068 0.058 0.078 0.092 0.085 0.075 mseβ0 0.014 0.014 0.013 0.011 0.015 0.017 0.016 0.013 0.018 0.023 0.021 0.017 mseβ1 0.014 0.016 0.019 0.075 0.016 0.020 0.024 0.086 0.018 0.025 0.029 0.106 mseβ2 0.012 0.013 0.017 0.073 0.013 0.016 0.020 0.081 0.016 0.020 0.024 0.099 mseβ3 0.016 0.015 0.014 0.011 0.020 0.021 0.019 0.015 0.026 0.029 0.025 0.020 areβ0 0.046 0.047 0.045 0.041 0.048 0.053 0.051 0.045 0.053 0.061 0.058 0.052 areβ1 0.094 0.100 0.110 0.221 0.101 0.113 0.123 0.236 0.108 0.122 0.133 0.256 areβ2 0.043 0.047 0.052 0.108 0.045 0.051 0.056 0.114 0.050 0.055 0.063 0.125 areβ3 0.033 0.033 0.031 0.028 0.037 0.038 0.035 0.032 0.043 0.044 0.041 0.037 Tabla 4.12. Errores global, cuadrático medio y relativo absoluto para 1 datos imputados por CART y con p1 (x1 , x2 , x3 ) = 1+exp(−1.5x . 2) cov 0 0.25 0.5 0.9 0 0.25 0.5 0.9 0 0.25 0.5 0.9 cens 0 0 0 0 0.15 0.15 0.15 0.15 0.25 0.25 0.25 0.25 error 0.289 0.294 0.251 0.127 0.318 0.336 0.291 0.146 0.351 0.380 0.330 0.170 mseβ0 0.021 0.022 0.019 0.014 0.021 0.024 0.021 0.015 0.024 0.030 0.026 0.020 mseβ1 0.267 0.267 0.283 0.489 0.290 0.301 0.324 0.540 0.314 0.334 0.358 0.591 mseβ2 0.025 0.047 0.118 0.470 0.028 0.056 0.139 0.514 0.033 0.066 0.157 0.564 mseβ3 0.021 0.020 0.018 0.013 0.028 0.029 0.026 0.017 0.037 0.039 0.034 0.023 areβ0 0.054 0.054 0.051 0.045 0.054 0.059 0.055 0.047 0.059 0.066 0.061 0.054 areβ1 0.482 0.480 0.494 0.646 0.501 0.508 0.527 0.677 0.519 0.531 0.550 0.703 areβ2 0.059 0.086 0.151 0.322 0.063 0.094 0.163 0.337 0.067 0.101 0.172 0.352 areβ3 0.036 0.035 0.034 0.030 0.041 0.043 0.040 0.034 0.048 0.049 0.046 0.040 37 Tabla 4.13. Errores global, cuadrático medio y relativo absoluto para 1 datos imputados por CART y con p2 (x1 , x2 , x3 ) = 1+exp(−1.5x 2) . 2 cov 0 0.25 0.5 0.9 0 0.25 0.5 0.9 0 0.25 0.5 0.9 cens 0 0 0 0 0.15 0.15 0.15 0.15 0.25 0.25 0.25 0.25 error 0.127 0.133 0.115 0.059 0.138 0.152 0.135 0.071 0.152 0.170 0.151 0.088 mseβ0 0.016 0.017 0.015 0.012 0.018 0.020 0.018 0.013 0.021 0.026 0.023 0.019 mseβ1 0.103 0.110 0.115 0.172 0.108 0.120 0.128 0.190 0.112 0.125 0.134 0.213 mseβ2 0.014 0.020 0.042 0.150 0.016 0.024 0.049 0.162 0.019 0.028 0.054 0.187 mseβ3 0.017 0.017 0.016 0.012 0.022 0.023 0.022 0.016 0.029 0.030 0.028 0.021 areβ0 0.047 0.047 0.046 0.042 0.049 0.053 0.051 0.045 0.055 0.061 0.059 0.053 areβ1 0.283 0.288 0.293 0.320 0.286 0.299 0.307 0.333 0.287 0.301 0.310 0.350 areβ2 0.045 0.054 0.081 0.148 0.048 0.058 0.085 0.152 0.053 0.062 0.089 0.160 areβ3 0.033 0.032 0.032 0.029 0.036 0.037 0.036 0.032 0.042 0.042 0.041 0.038 Tabla 4.14. Errores global, cuadrático medio y relativo absoluto para 1 datos imputados por NORM y con p1 (x1 , x2 , x3 ) = 1+exp(−1.5x . 2) cov 0 0.25 0.5 0.9 0 0.25 0.5 0.9 0 0.25 0.5 0.9 cens 0 0 0 0 0.15 0.15 0.15 0.15 0.25 0.25 0.25 0.25 error 0.316 0.320 0.261 0.093 0.347 0.367 0.305 0.112 0.381 0.413 0.343 0.135 mseβ0 0.027 0.026 0.023 0.014 0.026 0.027 0.024 0.016 0.029 0.033 0.029 0.020 mseβ1 0.314 0.314 0.318 0.393 0.339 0.353 0.362 0.455 0.364 0.386 0.397 0.521 mseβ2 0.029 0.048 0.105 0.334 0.033 0.057 0.122 0.383 0.038 0.065 0.138 0.443 mseβ3 0.023 0.022 0.020 0.014 0.030 0.032 0.028 0.018 0.039 0.042 0.036 0.024 areβ0 0.053 0.051 0.048 0.043 0.053 0.055 0.053 0.046 0.058 0.063 0.060 0.053 areβ1 0.524 0.523 0.524 0.533 0.544 0.553 0.559 0.571 0.562 0.576 0.582 0.606 areβ2 0.054 0.078 0.133 0.239 0.059 0.085 0.142 0.254 0.063 0.093 0.150 0.271 areβ3 0.035 0.034 0.033 0.029 0.039 0.042 0.039 0.033 0.047 0.048 0.045 0.039 38 CAPÍTULO 4. ESTUDIO DE SIMULACIÓN Tabla 4.15. Errores global, cuadrático medio y relativo absoluto para 1 datos imputados por NORM y con p2 (x1 , x2 , x3 ) = 1+exp(−1.5x 2) . 2 cov 0 0.25 0.5 0.9 0 0.25 0.5 0.9 0 0.25 0.5 0.9 cens 0 0 0 0 0.15 0.15 0.15 0.15 0.25 0.25 0.25 0.25 error 0.134 0.138 0.116 0.057 0.146 0.157 0.136 0.069 0.160 0.176 0.152 0.086 mseβ0 0.017 0.017 0.016 0.012 0.018 0.021 0.019 0.014 0.022 0.028 0.025 0.019 mseβ1 0.123 0.125 0.129 0.186 0.129 0.137 0.144 0.208 0.134 0.144 0.151 0.232 mseβ2 0.014 0.022 0.046 0.162 0.016 0.025 0.052 0.179 0.019 0.030 0.058 0.203 mseβ3 0.019 0.019 0.016 0.012 0.024 0.025 0.022 0.016 0.031 0.033 0.029 0.021 areβ0 0.045 0.046 0.044 0.041 0.046 0.051 0.050 0.044 0.052 0.060 0.057 0.052 areβ1 0.304 0.306 0.308 0.324 0.309 0.317 0.322 0.340 0.311 0.320 0.325 0.355 areβ2 0.045 0.056 0.083 0.150 0.047 0.059 0.087 0.156 0.053 0.064 0.090 0.162 areβ3 0.032 0.032 0.030 0.028 0.035 0.037 0.035 0.032 0.042 0.043 0.040 0.037 Capı́tulo 5 Aplicación a datos reales 5.1. Descripción del problema Es frecuente que previo a la realización del análisis estadı́stico en estudios de investigación clı́nica y epidemiológica, nos encontremos con datos faltantes. El caso que presentamos a continuación no se escapa a este problema. En concreto, se trata de un estudio de supervivencia en pacientes que han sido sometidos a trasplante hepático, y cuyo objetivo es conocer aquellas variables que pueden encontrarse asociadas a un peor pronóstico. Es habitual en estudios de supervivencia, la utilización del modelo de riesgos proporcionales de Cox en el análisis de los datos, y es por ello que existen una serie de métodos de imputación de datos faltantes implementados para estos modelos [19]. Sin embargo, en ocasiones deseamos aplicar otros modelos, como por ejemplo, los modelos de tiempo de fallo acelerado (AFT). Los modelos AFT nos proporcionan una alternativa elegante al modelo de riesgos proporcionales en cuanto relacionan el efecto de las variables predictoras con el tiempo de supervivencia en lugar del valor de riesgo como ocurre en el modelo de Cox. Estas caracterı́sticas permiten una interpretación más sencilla de los resultados. Sin embargo, los modelos AFT no se utilizan tanto debido a la ausencia de algoritmos eficientes y fiables que permitan una estimación adecuada de los parámetros y sus errores estándar [20]. Asimismo, en los modelos AFT son escasas las referencias existentes [21, 22] en la literatura en relación con las aproximaciones para tratar datos faltantes. 5.2. 5.2.1. Descripción del conjunto de datos Descripción de la población y objetivo del estudio Para ilustrar nuestra aproximación, analizamos los datos referentes a un estudio de supervivencia en todos los pacientes sometidos a trasplante hepático ortotópico realizado en el Hospital Clı́nico Universitario de Santiago de Compostela (CHUS), entre Julio de 1994 y Julio de 2011. Se excluyeron del estudio a los 22 primeros pacientes trasplantados y otros 8 pacientes que no sobrevivieron más de 7 dı́as posttrasplante. Finalmente, se incluyeron en el análisis 629 pacientes. El estado vital de los pacientes y la fecha de muerte se obtuvieron a través de los registros clı́nicos y el final del seguimiento data de Junio de 2012. La mediana (rango) de seguimiento fue de 67 meses (1 semana - 17,5 años). Durante este tiempo, 414 (65,8 %) personas permanecieron vivas y 215 (34,2 %) fallecieron. 39 40 CAPÍTULO 5. APLICACIÓN A DATOS REALES El objetivo de este estudio es analizar aquellos factores que pueden influir en la supervivencia de los pacientes que han sido sometidos a trasplante hepático. 5.2.2. Descripción de las variables Las variables recogidas para el estudio son aquellas ya descritas en la literatura y que han mostrado que pueden tener relación con la supervivencia: edad, sexo, diagnóstico previo de diabetes, ı́ndice de masa corporal, etiologı́a del trasplante (cáncer de hı́gado, abuso de alcohol, virus de la hepatitis C, enfermedades autoinmunes), tiempo de isquemia frı́a del órgano, transfusión de hematı́es y de plaquetas durante la cirugı́a, existencia de trombosis portal previa al trasplante, nutrición en el postoperatorio inmediato, creatinina y/o filtrado glomerular previa al trasplante, y en los pacientes trasplantados a partir de 2004 el MELD, ya que previamente no se posee el INR, una medida del tiempo de coagulación, para poder calcular este marcador pronóstico. Además, dado que también formaba parte del objetivo del estudio, introducimos la glucosa basal y las medidas de glucosa en los dı́as siguientes a la cirugı́a. Al igual que ocurre en otras bases de datos biomédicas, tenemos datos faltantes en la mayorı́a de las variables descritas anteriormente. La ausencia de datos oscila desde el 0,16 % como ocurre en el virus de la hepatitis C o en la trombosis portal, hasta pérdidas del 57,23 % como ocurre con el MELD. Lo ilustramos en la tabla 5.1. Tabla 5.1. Número de observaciones faltantes en las variables del estudio con su correspondiente porcentaje. Sexo Edad Índice de masa corporal Diabetes mellitus Meld Abuso de alcohol Virus de la hepatitis C Año del trasplante Carcinoma Tiempo de isquemia frı́a 0 (0 %) 0 (0 %) 0 (0 %) 0 (0 %) 360 (57.23 %) 0 (0 %) 1 (0.16 %) 0 (0 %) 0 (0 %) 17 (2.70 %) Trasfusión de hematı́es Trasfusión de plaquetas Trombosis portal Nutrición parenteral Glucosa basal Creatinina basal Insulina media Muerte Tiempo de supervivencia desde el trasplante 27 24 1 9 22 18 27 (4.29 %) (3.82 %) (0.16 %) (1.43 %) (3.50 %) (2.86 %) (4.29 %) 0 (0 %) 0 (0 %) También podemos verlo gráficamente en la figura 5.1. Obsérvese en la primera gráfica como el porcentaje de individuos en la muestra con observaciones faltantes es significativamente superior en la variable meld con respecto a las demás, y en la segunda gráfica se puede apreciar las combinaciones existentes de las observaciones faltantes y las no faltantes. Antes de proceder al análisis, una cuestión importante es discernir los mecanismos que conducen a la pérdida de datos: MCAR, MAR o MNAR. Hemos podido comprobar que la fecha del trasplante tiene gran influencia en la pérdida de datos. Ası́ por ejemplo, antes de 2004 la falta de datos es mucho mayor. Esto es debido a la introducción de la historia clı́nica electrónica (IANUS) que empieza a estar operativa en esta época y, por tanto, a partir de ahı́ no se pierden datos salvo aquellos que no se introduzcan en la historia clı́nica. El caso de la variable MELD merece mención aparte. Es el acrónimo de Model for End-stage Liver Disease, un sistema de puntuación para medir la severidad de la enfermedad hepática crónica. Fue inicialmente desarrollado para predecir la muerte dentro de 3 meses de cirugı́a en pacientes que habı́an sido sometidos a TIPS (transjugular intrahepatic portosystemic shunt) y fue subsecuentemente hallado útil para determinar el pronóstico y para priorizar los pacientes en espera de trasplante. Hemos 5.2. DESCRIPCIÓN DEL CONJUNTO DE DATOS 41 Figura 5.1: Proporción y combinación de datos faltantes en las variables. recogido el MELD como indicador pronóstico previo al trasplante en 269 pacientes (42,8 %), con un rango entre 4 y 42 con una media de 14.1, desviación tı́pica de 6,3 y una mediana de 14. No ha sido posible recoger el MELD en los pacientes trasplantados antes de 2004, ya que en el laboratorio no se realizaba el INR, sino únicamente el tiempo de Quick, por lo que no ha sido posible el cálculo del ı́ndice. En la tabla 5.2 se muestra un análisis descriptivo de las variables. Para describir las variables cuantitativas se utilizará la mediana (primer y tercer cuartil), mientras que para las variables cualitativas indicaremos el porcentaje correspondiente para cada categorı́a. En la figura 5.2, se puede apreciar la curva de supervivencia estimada con todos los individuos en riesgo y su intervalo de confianza al 95 %, obtenida por el estimador de Kaplan-Meier. En las tres figuras que se muestran posteriormente (5.3, 5.4 y 5.5), se puede observar cómo cambian los boxplots de las variables edad, tempo y timee en función de los datos faltantes de las otras. En ellas podemos apreciar diferencias entre los boxplots para casi todas las variables con datos faltantes, sobre todo para la variable meld. El hecho de que haya tanta diferencia entre los boxplots que incluyen datos observados y aquellos a cuyos individuos les falta la medida en esa variable, nos indica que la pérdida de datos no es completamente aleatoria. No podemos suponer MCAR. Por lo tanto la estimación utilizando sólo la submuestra completa no parece la más adecuada en esta situación. Esto ha motivado que consideremos la imputación múltiple como alternativa a la estimación con la submuestra completa. 42 CAPÍTULO 5. APLICACIÓN A DATOS REALES Tabla 5.2. Caracterı́sticas generales de las variables. Sexo Hombre Mujer Edad (años) Diabetes mellitus No Sı́ Índice de masa corporal (Kg/m2 ) Abuso de alcohol No Sı́ Virus de la hepatitis C No Sı́ Carcinoma No Sı́ Trombosis portal No Sı́ Meld Tiempo de isquemia frı́a (horas) Trasfusión de hematı́es (unidades) Trasfusión de plaquetas (unidades) Nutrición parenteral Glucosa basal pretrasplante Creatinina basal pretrasplante Insulina media en los 7 dı́as posttrasplante Tiempo de supervivencia desde el trasplante (años) Muerte No Sı́ 5.3. 471 (74.9 %) 158 (25.1 %) 54 [45, 60] 504 (80.1 %) 125 (19.9 %) 27 [25, 29] 244 (38.8 %) 385 (61.2 %) 498 (79.3 %) 130 (20.7 %) 456 (72.5 %) 173 (27.5 %) 573 (91.2 %) 55 (8.8 %) 14 [9, 17] 7 [6, 9] 6 [2, 10] 0 [0, 1] 4 [3, 6] 105 [90, 137] 0.9 [0.7, 1.1] 39.57 [13.57, 115.50] 5.63 [2.24, 9.79] 414 (65.8 %) 215 (34.2 %) Imputación a los datos reales Después de aplicar imputación a los datos por distintos métodos, se procede a verificar si cumple la proporcionalidad de riesgos de acuerdo a los supuestos exigidos por el modelo de Cox. Primero se prueba con los casos completos e imputando por pmm, cart y sample todas las variables con observaciones faltantes. Luego, se aplica logreg a las variables binarias al mismo tiempo que se aplica pmm, cart, sample, mean, norm y norm.predict a las variables continuas. Los resultados se muestran en la tabla 5.3. Como puede apreciarse en esta tabla, utilizando el método de casos completos se cumple la proporcionalidad de riesgos. Sin embargo, si se utiliza pmm en la imputación de todas las variables o norm.predict para las variables continuas y logreg para las binarias no se cumplen los supuestos de proporcionalidad de riesgos, como ocurre en la mayorı́a de los casos. De ahı́ la conveniencia de utilizar el método AFT para analizar la supervivencia utilizando imputación múltiple. 5.3. IMPUTACIÓN A LOS DATOS REALES 43 Figura 5.2: Curva de Kaplan-Meier. Tabla 5.3. P-valores obtenidos en las pruebas de proporcionalidad de riesgos tras los diferentes métodos de imputación aplicados. Método/s Casos completos Pmm Cart Sample Pmm/logreg Cart/logreg Sample/logreg Mean/logreg Norm/logreg Norm.predict/logreg p-valor 2.88e-01 1.66e-02 4.29e-02 7.36e-02 8.61e-03 3.44e-03 4.71e-02 4.24e-02 1.86e-01 1.12e-02 A continuación, interesa saber qué método de imputación es el más adecuado de todos ellos a nuestros datos. Tras la aplicación del criterio AIC a cada uno de los conjuntos de datos imputados por los distintos métodos aplicados, obtenemos los siguientes resultados (tabla 5.4). Puede apreciarse que los métodos pmm para la imputación de variables continuas y logreg para las binarias, son los que obtienen menores valores de AIC. Cabe recordar que el método pmm es adecuado para datos que no asumen normalidad como frecuentemente ocurre en datos reales. Una vez conocido el método a utilizar en la imputación, falta hallar el número óptimo de imputaciones. Para ello se calcula el AIC global para cada caso. Una forma de hacerlo, es hallar el AIC 44 CAPÍTULO 5. APLICACIÓN A DATOS REALES Figura 5.3: Boxplot de la edad en función de si tenemos los datos observados en las otras variables o no. Tabla 5.4. AIC de los diferentes métodos de imputación. Método/s Pmm Cart Sample Pmm/logreg Cart/logreg Sample/logreg Mean/logreg Norm/logreg Norm.predict/logreg AIC 1510.50 1517.97 1519.09 1508.17 1511.78 1524.04 1519.04 1515.75 1512.13 para cada conjunto de datos imputados de forma separada y luego calcular la media de esos valores [23]. Ası́, se obtienen los resultados que aparecen recogidos en la tabla 5.5. Se obtienen AIC similares para las diferentes imputaciones, encontrándose un menor AIC cuando el número de imputaciones es m=3. Puede apreciarse que con el aumento del número de imputaciones no necesariamente se obtendrán mejores resultados. Tras realizar tres imputaciones (m=3) por el método pmm para variables cuantitativas y por el método logreg para variables cualitativas se obtienen los siguientes factores de riesgo para cada imputación (tabla 5.6). Para hallar las covariables que finalmente entrarán en el modelo de supervivencia, se utilizará pri- 5.3. IMPUTACIÓN A LOS DATOS REALES 45 Figura 5.4: Boxplot del año de trasplante en función de si tenemos los datos observados en las otras variables o no. Tabla 5.5. AIC global para cada número de imputaciones. m 1 2 3 5 10 15 20 25 30 AIC 1508.17 1513.18 1499.80 1508.55 1509.23 1510.24 1510.16 1510.04 1507.96 Tabla 5.6. Factores de riesgo asociados a cada imputación. m 1 2 3 Factores de riesgo TH, sexo, imc, actrm, edad, carc, NPTt, meld, tempo, TIF TH, sexo, imc, actrm, edad, carc, NPTt, meld, tempo TH, sexo, imc, actrm, edad, carc, NPTt, meld, tempo mero la técnica de selección de variables majority, que consiste en seleccionar las variables que aparecen al menos en la mitad de los modelos. En este caso son: TH, sexo, imc, actrm, edad, carc, NPTt, meld y tempo. Posteriormente procederemos a verificar mediante el test de Wald si son necesarias todas estas variables o si se puede quitar alguna. 46 CAPÍTULO 5. APLICACIÓN A DATOS REALES Figura 5.5: Boxplot del tiempo de supervivencia desde el trasplante en función de si tenemos los datos observados en las otras variables o no. En la tabla 5.7 se muestra un breve resumen de todas las variables con sus diferentes estimaciones, errores estándar y p-valores finales asociados a cada parámetro, utilizando casos completos y la imputación por pmm para variables cuantitativas y por logreg para variables cualitativas. Las variables significativas obtenidas habiendo imputado los datos son: edad, tempo, carc, NPTt y actrm. Por otra parte, si tenemos en cuenta únicamente los casos completos, obtenemos que las variables significativas son: carc y TH. De todas formas, nos apoyaremos en el criterio AIC para obtener los factores de riesgo que más influyen en la supervivencia. Además, puede observarse que las estimaciones en algunos de los parámetros varı́an sensiblemente de un método a otro. Ası́ por ejemplo, la variable tempo alcanza significación estadı́stica cuando se realiza la imputación mientras que en el análisis de casos completos no la alcanza. Debemos tener en cuenta que debido al proceso de pérdida de datos el análisis de casos completos se restringe a los últimos años del trasplante. Para averiguar en qué orden debemos chequear las variables para utilizar el test de Wald, introducimos en el modelo todas las variables para ir eliminando “paso a paso” todas aquellas variables que habı́an sido introducidas siguiendo el criterio AIC. Finalmente, se aplica el test de Wald para esas variables en el orden en que fueron eliminadas: meld, imc, sexo, edad, TH, carc, actrm, tempo y NPTt. Dado que las dos primeras variables meld e imc no alcanzan un p-valor<0.05, éstas no se introducen en el modelo final, mientras que las restantes variables sı́ alcanzan un p-valor<0.05, por lo que éstas sı́ se introducen en el modelo final. 5.4. RESULTADOS DEL ANÁLISIS DE SUPERVIVENCIA 47 Tabla 5.7. Estimaciones de los coeficientes y errores estándar en las covariables introducidas en los modelos de supervivencia con casos completos y tras la imputación. Intercepto Sexo Edad Imc Dm Meld Oh Vhc Tempo Carc TIF TH TP TVP NPTt Glu0 Cr0 Actrm 5.4. Datos imputados Estimación Error estándar -3.12e+02 7.94e+01 -5.59e-01 3.27e-01 -3.88e-02 1.34e-02 6.51e-02 3.67e-02 3.62e-01 3.99e-01 -8.39e-02 5.50e-02 1.05e-02 3.16e-01 -2.80e-01 3.39e-01 1.59e-01 3.97e-02 -1.02e+00 3.44e-01 5.46e-02 7.40e-02 -3.25e-02 1.89e-02 -5.28e-03 5.23e-02 1.06e-01 4.55e-01 -8.12e-02 3.40e-02 3.92e-04 2.21e-03 -4.12e-02 2.90e-01 -5.96e-03 1.81e-03 p-valor 1.86e-04 8.88e-02 4.40e-03 7.64e-02 3.65e-01 2.33e-01 9.74e-01 4.10e-01 1.49e-04 4.06e-03 4.66e-01 1.00e-01 9.20e-01 8.16e-01 3.91e-02 8.60e-01 8.88e-01 1.15e-03 Casos completos Estimación Error estándar -8.21e+01 2.15e+02 -6.32e-01 5.33e-01 -2.61e-02 2.47e-02 3.69e-02 5.23e-02 -4.67e-03 5.29e-01 -2.99e-02 4.26e-02 2.97e-01 5.47e-01 -3.22e-01 5.84e-01 4.33e-02 1.07e-01 -1.62e+00 4.86e-01 1.01e-01 1.04e-01 -8.79e-02 4.28e-02 1.08e-01 1.86e-01 2.60e-01 7.98e-01 -5.26e-03 8.54e-02 -2.18e-04 3.52e-03 3.18e-01 6.32e-01 -3.14e-03 2.93e-03 p-valor 7.03e-01 2.36e-01 2.90e-01 4.80e-01 9.93e-01 4.82e-01 5.87e-01 5.82e-01 6.87e-01 8.30e-04 3.32e-01 4.00e-02 5.62e-01 7.44e-01 9.51e-01 9.51e-01 6.16e-01 2.84e-01 Resultados del análisis de supervivencia A continuación se relatan aquellos factores que afectan a la supervivencia de estos individuos en relación a si el análisis se basa en los casos disponibles, en los casos completos o en los datos imputados. Si aplicamos el modelo de supervivencia AFT a los casos disponibles, y utilizamos luego el criterio de selección de variables AIC obtenemos que las variables que más influyen en la supervivencia de los individuos son las siguientes: TIF, NPTt, TH, carc y meld. Sin embargo, si aplicamos el método de imputación de casos completos a estos datos, y luego el modelo de supervivencia AFT, por el criterio AIC obtenemos que las variables que más influyen en la supervivencia son: sexo, TH y carc. Aplicando el método de imputación óptimo a estos datos con el correspondiente número de imputaciones, obtenemos que las variables que más influyen en la supervivencia son: sexo, edad, TH, carc, actrm, tempo y NPTt. Tras la selección de éstas últimas variables en el caso datos imputados, obtenemos las siguientes estimaciones, errores estándar y p-valores para los parámetros (tabla 5.8). 48 CAPÍTULO 5. APLICACIÓN A DATOS REALES Tabla 5.8. Estimaciones finales de los coeficientes y errores estándar de las covariables introducidas en los modelos de supervivencia tras los pasos de imputación. Intercepto Sexo Edad Dm Oh Vhc tempo carc TIF TH TP TVP NPTt Glu0 Cr0 Actrm Estimación -2.86e+02 -6.40e-01 -2.82e-02 3.32e-01 1.70e-01 -2.60e-01 1.46e-01 -8.22e-01 2.97e-02 -3.86e-02 -4.67e-02 1.44e-01 -1.21e-01 3.33e-04 -2.22e-01 -5.30e-03 Error estándar 7.43e+01 3.24e-01 1.25e-02 4.12e-01 3.21e-01 3.46e-01 3.71e-02 3.21e-01 6.55e-02 1.65e-02 4.63e-02 4.70e-01 2.47e-02 2.32e-03 2.71e-01 1.81e-03 p-valor 1.36e-04 4.87e-02 2.51e-02 4.21e-01 5.97e-01 4.53e-01 9.59e-05 1.06e-02 6.51e-01 1.96e-02 3.13e-01 7.60e-01 1.49e-06 8.86e-01 4.14e-01 3.69e-03 Capı́tulo 6 Conclusiones En este trabajo, hemos tratado de abordar el análisis de los modelos de supervivencia AFT cuando existen datos faltantes. Los hallazgos más importantes pueden resumirse en dos: 1. En los estudios de simulación realizados el método de imputación que ofrece mejores resultados es el norm.predict. 2. De la aplicación a datos reales se puede derivar que la selección de las variables predictoras en la supervivencia de los pacientes cambia sensiblemente, ası́ como los coeficientes y errores estándar estimados en las mismas. Entre los métodos de análisis en supervivencia, los más frecuentemente utilizados son el modelo de riesgos proporcionales de Cox y el modelo AFT. Cada uno de ellos ofrece ventajas y desventajas desde el punto de vista comparativo. Ası́, el modelo de Cox tiene la ventaja de que no necesitamos especificar una distribución de supervivencia, mientras que el modelo AFT no necesita asumir proporcionalidad en los riesgos y modela directamente el efecto de las variables explicativas en la supervivencia. En este trabajo hemos desarrollado la imputación de datos faltantes en modelos AFT dadas las escasas referencias en la literatura en relación con la imputación de datos faltantes en modelos AFT. En el estudio de simulación, de entre los distintos escenarios que hemos considerado (porcentajes de censura, covarianzas, métodos de imputación), obtuvimos distintos errores dependiendo del método de imputación utilizado. Los errores siempre aumentan cuanto más grande sea la censura, pero cuanto mayor sea la correlación entre las dos primeras variables en algunos métodos obtenemos errores más altos mientras que en otros disminuyen. Con los resultados obtenidos llegamos a la conclusión de que el mejor método de imputación para esos datos simulados era el norm.predict. De los resultados obtenidos en el estudio de aplicación a datos reales, destacar las diferencias obtenidas en relación con los diferentes métodos de imputación que se utilizan. De entre los métodos de imputación aplicados, se puede extraer, siguiendo el criterio AIC, que los mejores resultados se obtienen utilizando pmm para variables continuas y logreg para las binarias. Si aplicamos el modelo AFT a los datos disponibles obtenemos que los factores de riesgo son: TIF, NPTt, TH, carc y meld. Si lo aplicamos a los casos completos serı́an: sexo, TH y carc. Y si lo aplicamos a los datos imputados obtenemos que los factores de riesgo más importantes son: TH, sexo, NPTt, edad, carc, actrm y tempo. Todas las variables predictoras obtenidas por los diferentes métodos son bien conocidas por ser factores o marcadores de riesgo de supervivencia en pacientes sometidos a trasplante hepático. 49 50 CAPÍTULO 6. CONCLUSIONES La necesidad de mayor cantidad de transfusión de hematı́es, de nutrición parenteral y de tratamiento con insulina y el meld son marcadores que nos indican que estos individuos se encuentran en peores condiciones en su estadı́o de la enfermedad. La presencia de un hepatocarcinoma supone un bien conocido factor riesgo de mayor mortalidad, y resulta evidente que conforme al paso del tiempo, las técnicas quirúrgicas, y especialmente los tratamientos que se administran para evitar el rechazo del hı́gado trasplantado, han mejorado de forma notable la supervivencia de estos pacientes. Bibliografı́a [1] Baraldi A.N., Enders C.K., 2010, An introduction to modern missing data analyses, Journal of School Psychology 48, 5–37. [2] Gelman A., Hill J., 2006, Data Analysis Using Regression and Multilevel/Hierarchical Models. Analytical Methods for Social Research, Cambridge. [3] Donders A.R., van der Heijdenc G.J., Stijnend D., Moons K.G., 2006, Review: A gentle introduction to imputation of missing values, Journal of Clinical Epidemiology 59, 1087-1091. [4] Graham J.W., 2012, Missing Data: Analysis and Design. Springer, New York. [5] Enders C.K., 2010, Applied Missing Data Analysis. Guilford Press, New York. [6] White I. R., Royston P., Wood A.M., 2011, Multiple imputation using chained equations: Issues and guidance for practice, Statistics in Medicine 30, 377–399. [7] Cheng X., Cook D., Hofmann H., 2014, A GUI for Missing Data Exploration. http://cran.rproject.org/web/packages/MissingDataGUI/index.html [Última consulta: 25/06/2014] [8] Honaker J., King G., Blackwell M., 2013, Amelia II: A Program for Missing Data. http://gking.harvard.edu/amelia [Última consulta: 25/06/2014] [9] Templ M., Alfons A., Kowarik A., Prantner B., 2013, Visualization and Imputation of Missing Values. https://github.com/alexkowa/VIM [Última consulta: 25/06/2014] [10] van Buuren S., Groothuis-Oudshoorn K., Robitzsch A., Vink G., Doove L., Jolani S., 2014, Multivariate Imputation by Chained Equations. http://www.multiple-imputation.com [Última consulta: 25/06/2014] [11] van Buuren S., 2012, Flexible Imputation of Missing Data. Chapman & Hall/CRC, New York. [12] van Buuren S., Groothuis-Oudshoorn K., 2011, mice: Multivariate Imputation by Chained Equations in R, Journal of Statistical Software 45. [13] Rebasa P., 2005, Conceptos básicos del análisis de supervivencia, 78, 222-230. [14] Borges R., 2004, Análisis de supervivencia básico utilizando el lenguaje R. [15] Stevenson M., 2009, An Introduction to Survival Analysis. [16] Allison P.D., 1995, Survival Analysis Using the SAS System: A Practical Guide, SAS Institute. [17] Lee E.T., Wenyu Wang J., 2003, Statistical Methods for Survival Data Analysis. WileyInterscience, New Jersey. 51 52 BIBLIOGRAFÍA [18] Orbe J., Ferreira E., Núñez-Antón V., 2002, Comparing proportional hazards and accelerated failure time models for survival analysis, Statistics in Medicine 21, 3493-3510. [19] Marshall A., Altman D., Holder R., 2010, Comparison of imputation methods for handling missing covariate data when fitting a Cox proportional hazards model: a resampling study, BMC Medical Research Methodology 10. [20] Chiou S. H., 2013, Statistical Methods and Computing for Semiparametric Accelerated Failure Time Model with Induced Smoothing, Doctoral Dissertations. [21] Zhang N., Little R.J., 2011, Subsample ignorable likelihood for accelerated failure time models with missing predictors, 95. [22] Nan B., Kalbfleisch J.D., Yu M., 2009, Asymptotic theory for the semiparametric accelerated failure time model with missing data, The Annals of Statistics 37, 2351-2376. [23] Consentino F., Claeskens G., 2010, Order Selection Tests with Multiply-Imputed Data. Belgium. Apéndice A Abreviaturas imc: Índice de masa corporal dm: diabetes mellitus meld: Model for End-stage Liver Disease oh: abuso de alcohol vhc: virus de la hepatitis C tempo: año del trasplante carc: carcinoma TIF: tiempo de isquemia frı́a TH: trasfusión de hematı́es TP: trasfusión de plaquetas TVP: trombosis portal NPTt: nutrición parenteral glu0: glucosa basal pretrasplante cr0: creatinina basal pretrasplante actrm: insulina media administrada en los 7 dı́as posttrasplante exitus: muerte timee: tiempo de supervivencia desde el trasplante INR: International Normalized Ratio 53