estudio de poblaciones naturales españolas de pinus pinaster ait. y
Anuncio

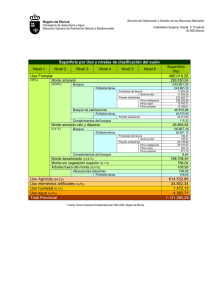
ESTUDIO DE POBLACIONES NATURALES ESPAÑOLAS DE PINUS PINASTER AIT. Y PINUS HALEPENSIS MILL. MEDIANTE ADN MITOCONDRIAL. AGÚNDEZ D.1, BURBAN CH.2, ROBLEDO J.J.3, GONZÁLEZ-MARTÍNEZ S.C. 1, PETIT R.J.2, ALÍA R. 1 1INIA-CIFOR. Dpto de Mejora Genética y Biotecnología. Apdo 8111. 28080 Madrid. España. 2 INRA.BP 45, 33611 Gazinet cedex. Francia. 3Unidad de Anatomía, Fisiología y Genética, ETSIM, Ciudad Universitaria s/n, 28040 Madrid. España. E-mail: [email protected] RESUMEN Diez poblaciones naturales de Pinus halepensis Mill. analizadas han resultado monomórficas en los tres fragmentos de ADN mitocondrial considerados. En el caso del fragmento estudiado en Pinus pinaster Ait., las poblaciones muestreadas revelan el mitotipo occidental europeo (Península Ibérica y Francia), excepto la población de Las Gabarras (Girona), que muestra el mitotipo oriental europeo (Sureste de Francia, Italia, Córcega y Cerdeña). Este resultado parece indicar un origen singular de las poblaciones catalanas en el ámbito ibérico, apoyado por los resultados obtenidos con otros marcadores y que se ha manifestado también en poblaciones de frondosas próximas. PALABRAS CLAVE: PCR-RFLP-SSCP, diversidad genética, coníferas, conservación recursos genéticos SUMMARY: Ten analysed native populations of Pinus halepensis Mill. have turned out to be monomorphic at each one of the three mitochondrial DNA fragments considered. The fragment analysed in Pinus pinaster Ait. shows that all sampled populations share the western-European mitotype (Iberian Peninsula and France), excepting Las Gabarras (Girona), which shows easternEuropean mitotype (south-eastern France, Italy, Corsica and Sardinia). This result suggests a singular origin, in the Iberian Peninsula context, of the catalan population, supported by results provided by other markers and also by analysis of closed broadleaved tree populations. KEY WORDS: PCR-RFLP-SSCP, conifers, genetic diversity, genetic resource conservation INTRODUCCIÓN Pinus pinaster Ait. (pino negral) presenta una distribución fragmentada en la Península Ibérica, habitando en condiciones ecológicas muy variables y manifestando una elevada variabilidad genética entre sus poblaciones (ALÍA, et al., 1996). Los análisis efectuados con terpenos (BARADAT & MARPEAU, 1988) muestran un patrón complejo en la distribución de la variación para el conjunto de la especie, existiendo en la Península Ibérica poblaciones pertenecientes a los tres grupos principales de razas definidos por los autores: atlántico, mediterráneo y magrebí. Los estudios realizados con isoenzimas (GONZÁLEZ MARTÍNEZ et al., 2001; SALVADOR et al., 2000) también apuntan a una estructura genética compleja en la Península Ibérica, con tres grandes grupos de poblaciones: atlántico, meridional y oriental. El estudio de la variación genética de Matsucoccus feytaudi Duc., cochinilla específica del pino negral, mediante ADN mitocondrial, reveló la existencia de tres linajes distintos en la distribución del insecto: Europeo occidental, Andaluz y Marroquí (BURBAN et al., 1999). Los estudios de variación genética de Pinus halepensis Mill. (pino carrasco) también han mostrado una clara estructuración geográfica de la variabilidad en la Península Ibérica. El análisis mediante isoenzimas destaca dos poblaciones de Málaga y dos catalanas diferenciadas del resto (AGÚNDEZ et al., 1997, 1999) y los microsatélites de cloroplasto (GÓMEZ, 1998) han sugerido además la existencia de refugios glaciares de la especie en Málaga y Girona. El ADN mitocondrial se transmite maternalmente en coníferas. La ausencia de recombinación genética derivada de este hecho, unida al lento ritmo evolutivo del ADN mitocondrial en las plantas, permite su utilización en la caracterización de los procesos de migración en especies de coníferas como Pinus sylvestris L. (SORANZO et al., 2000; SINCLAIR et al., 1998, 1999) o Picea abies L. (SPERISEN et al., 1998). Las técnicas de PCR (reacción en cadena de la polimerasa) y RFLP (polimorfismo en la longitud de fragmentos digeridos con enzimas de restricción) combinadas con SSCP (polimorfismo en la conformación de la cadena sencilla) permiten una mayor resolución en la evaluación de polimorfismos debidos a sustituciones que afectan a la conformación de la cadena sencilla (recogido en DUMOLIN-LAPÈGUE et al., 1996). En este trabajo se presentan los resultados obtenidos con Pinus halepensis Mill. y Pinus pinaster Ait. en la Península Ibérica analizando ADN mitocondrial con el fin de comprobar la existencia de posibles centros de migración de estas dos especies. MATERIAL Y MÉTODOS Material vegetal. El material procede de 9 poblaciones naturales de Pinus pinaster y 10 de Pinus halepensis, seleccionadas de manera que representaran principalmente la región mediterránea de su distribución en conexión con las masas del sudeste de Francia y con las del norte de Marruecos (Tabla 1 y Figura 1). Tabla 1. Poblaciones de Pinus pinaster y Pinus halepensis estudiadas Población Oña Codos Riopar Estepona Gaucín Cómpeta Pradell Benicasim Las Gabarras Cabanellas Tivisa Guardiola Zuera Colmenar Cucalon Paterna Carratraca Frigiliana Benicasim Código Especie Región Procedencia Latitud Longitud Altitud BU Ppr S. de Oña 42º 45´ 04´´ N 3° 31’ 10’’ W 700 Z Ppr Sist. Ibérico Central 41º 17´ 35´´ N 1º 25´ 11´´ W 1100-1200 AB Ppr S. Segura-Alcaraz 38º 28´ 05´´ N 2º 27´ 31´´ W 1200 MA1 Ppr S. Bermeja 36º 31´ 05´´ N 5º 07´ 11´´ W 500 MA2 Ppr S. de Ronda 36º 32´ 10´´ N 5º 17´ 56´´ W 500-700 MA3 Ppr S. Almijara-Nevada 36º 51´ 44´´ N 3º 53´ 33´´ W 1000-1500 T Ppr S. del Pradell 41º 10´ 00´´ N 0º 54´ 00´´ E 550-750 CS Ppr Benicasim 40º 05´ 04´´ N 0º 00´ 46´´ E 150-500 G Ppr Litoral Catalán 41º 54´ 51´´ N 3º 03´ 02´´ E 150-250 CAB Php Alta Cataluña 42º 14´ 08´´ N 2º 47´ 24´´ E 210 TIV Php Cataluña Litoral 41º 03´ 25´´ N 0º 45´ 45´´ W 400 GUA Php Cataluña Interior 41º 40´ 04´´ N 1º 45´ 41´´ E 420 ZUE Php Monegros-Ebro 41º 55´ 00´´ N 0º 55´ 04´´ W 575 COL Php Alcarria 40º 05´ 20´´ N 3º 20´ 10´´ W 750 CUC Php Maestrazgo-Serranos 39º 47´ 15´´ N 0º 36´ 44´´ W 575 PAT Php Betica Septentrional 38º 37´ 37´´ N 2º 16´ 37´´ W 1000 CAR Php Sur 36º 50´ 28´´ N 4º 50´ 04´´ W 650 FRI Php Sur 36º 49´ 00´´ N 3º 55´ 18´´ W 570 BIM Php Litoral Levantino 40º 04´ 52´´ N 0º 01´ 14´´ W 430 Ppr = Pinus pinaster ; Php = Pinus halepensis ; n = número de árboles analizados n 10 10 10 10 10 10 10 10 10 5 5 5 5 5 5 5 5 5 5 Se ha recomendado muestrear un número reducido de individuos por población (2 ó 3) y muchas poblaciones como estrategia de muestreo óptima en la estimación de la diferenciación entre poblaciones utilizando marcadores haploides (PONS & PETIT, 1995). En este trabajo se han analizado 10 individuos por población de Pinus pinaster y 5 individuos por población de Pinus halepensis. La extracción de ADN total se realizó a partir de semillas germinadas o plántulas siguiendo el método de DELLAPORTA et al. (1983) o DOYLE & DOYLE (1990), no habiendo encontrando diferencia entre ellos. Los fragmentos de ADN mitocondrial amplificados y su tamaño aparecen en la Tabla 2, junto con las secuencias de los cebadores utilizados. En Pinus pinaster solo se ha utilizado Nad1-intron2, al ser el único fragmento que mostrara polimorfismo en análisis previos de la especie sobre distintos fragmentos de ADN mitocondrial (BURBAN, com. pers.). Para la amplificación se ha seguido con modificaciones a MOREAU et al. (1994). La cantidad de 20ng de ADN molde se incorporan en un volumen total de 25 μl que contienen 0.2 μM de cada cebador, 0.2 U de Taq polimerasa (Red Goldstar de Eugentec), 2mM de MgCl2, 0.4 mM de cada dinucleótido dATP, dGTP, dCTP, dTTP, tampón 1x (Eugentec 10x) y 4.4μg/ml de BSA. La amplificación se realizó en un termociclador Techne PHC3, con el siguiente programa: 1 ciclo de 94º C durante 1’, 35 ciclos de 45’’ a 94º C, 45’’ a la temperatura de duplicación, y una elongación de 2’ o 2’ 15’’ a 72º C. Tabla 2. Fragmentos de ADN mitocondrial y cebadores utilizados. Fragmento nad 1 intron 2 nad 4 intron 3 nad 5 intron 1 Cebador 1 Cebador 2 nad 1 exon 2 F nad 1 exon 3 R nad 4 exon 3 F nad 4 exon 4 R nad 5 exon 1 F nad 5 exon 2 R Secuencia de los cebadores gca tta cga tct gca gct ca gga gct cga tta gtt tct gc gga gct ttc caa aga aat ag gcc atg ttg cac taa gtt ac ttt ttt cgg acg ttt tct ag ttt ggc caa gta tcc tac aa Tamaño (pb) 2200 Tª (ºC) 57 Tiempo de elongación 2’ 15’’ 2100 49 2’ 00’’ 2600 47 2’ 15’’ Referencias DEMESURE et al. (1995) DUMOLIN et al. (1997) DUMOLIN et al. (1997) pb = pares de bases; secuenciado para nad1, tamaño aproximado en nad4 y nad5 (Burban, com. pers.) Tª = temperatura de duplicación. La digestión de 10 μl de producto de PCR con la enzima de restricción Hinf1 se realizó en un volumen total de 25 μl. Los fragmentos resultantes se analizaron mediante la técnica SSCP, descrita en ORITA et al. 1989, BASSAM et al. 1991 y recogida en BODÉNÈS et al. 1996. RESULTADOS Y DISCUSIÓN El análisis de nueve poblaciones ibéricas de Pinus pinaster Ait.en el fragmento NAD1-Intron2 del ADN mitocondrial mediante técnicas RFLP-SSCP ha revelado la existencia de dos haplotipos distintos. Ocho de las poblaciones comparten un único haplotipo, apareciendo el segundo exclusivamente en la población de Las Gabarras, situada en el límite oriental de la distribución de la especie en España (Figura 1). Las diez poblaciones de pino carrasco han resultado monomórficas en los tres fragmentos de ADN mitocondrial compartiendo todas ellas el mismo haplotipo. La menor variación entre poblaciones de Pinus halepensis Mill. encontrada en este estudio en relación con la de Pinus pinaster Ait. confirma observaciones previas sobre la menor variabilidad genética del pino carrasco respecto a otras especies del género (ALÍA et al. 1999). La población de Pinus pinaster de Las Gabarras destaca con un mitotipo único entre todas las poblaciones ibéricas analizadas. Este mitotipo es igual al encontrado en poblaciones de pino negral de Francia suroriental, Italia, Córcega, Cerdeña y Túnez (BURBAN, com. pers.). La diferenciación hallada mediante análisis de isoenzimas de 47 poblaciones naturales de Pinus pinaster, muestra la población de Las Gabarras (Girona) con frecuencias alélicas intermedias entre las poblaciones del este peninsular y las poblaciones del sudeste francés y de Italia, en cuyo grupo queda incluida (BARBA et al., 2001). Figura 1. Distribución de haplotipos de Pinus pinaster y Pinus halepensis en las poblaciones de la Península Ibérica analizadas (los códigos de las poblaciones se definen en la Tabla 1). Las especiales características genéticas de las poblaciones situadas en esta región geográfica se han manifestado igualmente en Pinus halepensis. GÓMEZ (1998) identifica un posible refugio de esta especie en Cabanellas (Girona) tras el análisis de diversidad con microsatélites de cloroplasto. Quercus suber L. también muestra un haplotipo característico y fijado en 3 poblaciones estudiadas en Girona (Begur, Santa Coloma y Figueras) y una en Valencia (Pinet), analizadas mediante PCR-RFLP con ADN de cloroplastos (JIMÉNEZ, 2000). Estos resultados hacen pensar en la existencia en esta zona de una dinámica postglaciar independiente de la de otras regiones peninsulares para distintas especies vegetales. Si se considera además la elevada diversidad de especies forestales en la región catalana (VALLEJO et al. 2000), ésta se manifiesta como un importante elemento a considerar en la estrategia española de conservación de los recursos genéticos forestales. En Pinus pinaster, el mitotipo oriental caracteriza poblaciones sensibles al ataque de Matsucoccus feytaudifuertemente afectadas (sudeste de Francia), recientemente afectadas (norte de Italia y Córcega) o zonas donde el insecto no está presente (Túnez); en la Península Ibérica está presente en todas las masas muestreadas a excepción de la de Córdoba (BURBAN et al., 1999). En este estudio no se incluyeron poblaciones catalanas por lo que no se realizó seguimiento del insecto, si bien sería interesante comprobar su presencia; en el caso de presencia del insecto, se trataría de poblaciones resistentes a la cochinilla cuya utilización como fuente de semilla para las repoblaciones de la parte oriental del área de distribución sería recomendable; si la cochinilla no estuviera aún presente, habría que tener un cuidado especial para no introducirla en esta región, de tal forma que no sufrieran el mismo destino que las masas del sudeste de Francia. REFERENCIAS AGÚNDEZ, D., DEGEN, B., VON WUEHLISCH, G., ALÍA, R.; (1997). Genetic variation of Aleppo pine (Pinus halepensis Mill.) from Spain. Forest Genetics 4(4): 201-209. AGÚNDEZ, D., DEGEN, B., VON WUEHLISCH, G., ALÍA, R.; (1999). Multilocus analysis of Pinus halepensis Mill. from Spain: Genetic diversity and clinal variation. Silvae Genetica 48(3-4): 173-178. ALÍA, R., MARTÍN, S., DE MIGUEL, J., GALERA, R., AGÚNDEZ, D., GORDO, J., SALVADOR, L., CATALÁN, G., GIL, L.; (1996). Regiones de procedencia. Pinus pinaster Aiton. Organismo Autónomo Parques Nacionales. Madrid. ALÍA, R., GALERA, R., MARTÍN, S., AGÚNDEZ, D., DE MIGUEL, J., IGLESIAS, S.; (1999). Mejora genética y masas productoras de semilla de los pinares españoles. Instituto Nacional de Investigación y Tecnología Agraria y Alimentaria (INIA). Madrid. Monografías INIA: Forestal N.1. BARADAT, Ph., MARPEAU, A.; (1988). Le pin maritime Pinus pinaster Ait. Biologie et génétique des terpènes pour la conaissance et lamélioration de l´espèce. Thèse. Université Bordeaux I. Bordeaux. BARBA D., GONZÁLEZ-MARTÍNEZ S.C., RIBEIRO M.M., AGÚNDEZ D., SALVADOR L., ALIA R., GIL L.; (2001). Variación genética de Pinus pinaster Ait.:Aplicación a la identificación y caracterización del Material Forestal de Reproducción (MFR). (en este congreso). BASSAM B.J., CAETENO-ANOLLES G., GRESSOF P.M., (1991). Fast and sensitive silver staining of DNA in polyacrilamyde gels. Annals of Biochemistry 196: 80-83. BODENES C., LAIGRET F., KREMER A.; (1996). Inheritance and molecular variation of PCRSSCP fragments in pedunculate oak (Quercus robur L.). Theoretical and Applied Genetics 93: 348-354. BURBAN C., PETIT R.J., CARCREFF E., JACTEL H.; (1999). Rangewide variation of the maritime pine bast scale Matsococus feytaudi Duc. (Homoptera: Matsucoccidae) in relation to the genetic structure of its host. Molecular ecology 8: 1593-1602. DELLAPORTA, S.L., WOOD J., HICKS J.B.; (1983). A plant minipreparation: Version II. Plant Molecular Biology Reporter 1: 19-21. DEMESURE, B., SODZI, N., PETIT, R.J.; (1995). A set of universal primers for amplification of polymorphic non-coding regions of mitochondrial and chloroplast DNA in plants. Molecular ecology 4: 129-131. DOYLE, J.J., DOYLE, J.L.; (1990). Isolation of plant DNA from fresh tissue. Focus 12: 13-15. DUMOLIN-LAPÈGUE, S., BODÉNÈS, C., PETIT, R.J.; (1996). Detection of rare polymorphisms in mitochondrial DNA of oaks with PCR-RFLP combined to SSCP analysis. Forest Genetics 3(4): 227-230. DUMOLIN-LAPÈGUE, S., PEMONGE, M.H., PETIT R.J.; (1997). An enlarged set of consensus primers for the study of organelle DNA in plants. Molecular ecology 6: 393-397. GÓMEZ, A.; (1998). Análisis de la variabilidad genética mediante marcadores moleculares de ADN en poblaciones españolas de Pinus halepensis Mill. Tesis Doctoral. Universidad Politécnica de Madrid. GONZÁLEZ MARTÍNEZ, S.C., AGÚNDEZ, D., ALÍA, R., SALVADOR, L., GIL, L.; (2001). Geographical variation of gene diversity of Pinus pinaster Ait. in the Iberian Peninsula. En: Müller-Starck, G. (ed.). Genetic response of forest systems to changing environmental conditions. Kluwer Academic Press, Dordrecht. JIMÉNEZ, M.P.; (2000). Análisis de la variabilidad genética de Quercus suber L. mediante marcadores moleculares y su aplicación a la conservación de recursos genéticos. Tesis doctoral. Universidad Politécnica de Madrid. MOREAU F., KLEINSCHMIT J., KREMER A.; (1994). Molecular differentiation between Quercus petraea and Quercus robur assessed by random amplified DNA fragments. Forest Genetics 1: 5164. ORITA M., IWAHANA H., KANAZAWA H., HAYASHI K., SEKIYA T.; (1989). Detection of polymorphisms of human DNA by gel electrophoresis as single-strand conformation polymorphisms. Proceedings of the National Academy of Sciences of the USA 86: 2766-2270. PONS, O., PETIT, R.J.; (1995). Estimation, variance and optimal sampling of gene diversity. I. Haploid locus. Theor Appl Genet 90: 462-470. SALVADOR, L., ALÍA, R., AGÚNDEZ, D., GIL, L.; (2000). Genetic variation and migration pathways of maritime pine (Pinus pinaster Ait) in the Iberian peninsula. Theor Appl Genet 100: 89-95. SINCLAIR, W.T., MORMAN, J.D., ENNOS, R.A.; (1999). The postglacial history of Scots pine (Pinus sylvestris L.) in western Europe: evidence from mitochondrial DNA variation. Molecular Ecology 8: 83-88. SINCLAIR, W.T., MORMAN, J.D., ENNOS, R.A.; (1998). Multiple origins for Scots pine (Pinus sylvestris L.) in Scotland: evidence from mitochondrial DNA variation. Heridity 80: 233-240. SORANZO, N., ALÍA, R., PROVAN, J., POWELL, W.; (2000). Patterns of variation at a mitochondrial sequence-tagged-site locus provides new insights into the postglacial history of European Pinus sylvestris populations. Molecular Ecology 9: 1205-1211. SPERISEN, C., BÜCHLER, U., MÁTYÁS, G.; (1998). Genetic variation of mitochondrial DNA reveals subdivision of norway spruce (Picea abies (L.) Karst.). En: Karp, A., Isaac, P., Ingram, D. (eds.). Molecular tools for screening biodiversity. Pp. 413-417. Chapman & Hall, London. VALLEJO, R., SÁNCHEZ DE RON, D., DE TUERO, M., ALÍA, R.; (2000). Los recursos genéticos forestales en España. Investigación Agraria, Sistemas y Recursos Forestales. Fuera de Serie, número 2: 21-43.