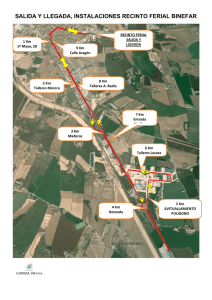
ANALISIS MOLECULAR DE LA LACASA DE Phanerochaete flavido
Anuncio

Universidad de Granada Facultad de Farmacia Departamento de Microbiología ANALISIS MOLECULAR DE LA LACASA DE Phanerochaete flavido-alba: Caracterización del gen y regulación por fenoles y metales. Francisco Rodríguez Rincón TESIS DOCTORAL Granada, España 2006 ii Editor: Editorial de la Universidad de Granada Autor: Francisco Rodríguez Rincón D.L.: Gr. 1026 - 2006 ISBN: 84-338-3850-4 ANALISIS MOLECULAR DE LA LACASA DE Phanerochaete flavido-alba: Caracterización del gen y regulación por fenoles y metales. Memoria presentada por el Licenciado en Biología y Magíster Scientae Francisco Rodríguez Rincón para optar al grado de Doctor en Ciencias por la Universidad de Granada (España) Los Directores: Dr. José Martínez López Prof. Titular de Microbiología Universidad de Granada Dra. Teresa de la Rubia Nieto Prof. Titular de Microbiología Universidad de Granada Dr. Antonio Suárez Prof. Titular de Microbiología Universidad de Granada El Doctorando: Francisco Rodríguez Rincón Granada, Abril de 2006 iii iv “¿Cómo se debe enseñar correcta y completamente?. Exactamente igual que como no se enseñaría correcta y completamente; por tanto, se dice, enseñar correcta y completamente. Como una estrella, una aberración visual, una lámpara, una ilusión, el rocío, una burbuja, un sueño, un relámpago y una nube: considera así todos los fenómenos compuestos”. Lhasa Zhol v vi AGRADECIMIENTOS Este trabajo ha sido realizado en el Departamento de Microbiología de la Facultad de Farmacia de la Universidad de Granada, gracias a una Comisión de estudios otorgada por la Universidad de Pamplona (Colombia) y con fondos del Contrato de la Unión Europea INCO-ICA3-CT2002-10033 para materiales y equipos. Agradecimientos especiales a mis directores por su apoyo, esfuerzo, orientación y dedicación en la realización de esta tesis, por haberme permitido formar parte de su grupo, por su confianza mostrada durante estos años, por poner a mi disposición sus laboratorios y por brindarme la oportunidad de aprender de las lacasas, de los hongos ligninolíticos y de la biología molecular. Agradezco también a todos los compañeros del Departamento de Microbiología y del Departamento de Bioquímica y Biología Molecular, especialmente a Fernando, Inmaculada, Mathias, José María, Gloria, Belén, Carolina, Olga, Raúl, Yolanda Carmen, José Antonio y Teresa por su inestimable ayuda en todo momento. A mi querida esposa, Gloria, por compartir conmigo este tiempo, por su amor y continuo apoyo. A mis padres y hermanos, ya que no hubiera llegado a este punto de la vida sin ellos. A todas aquellas personas que han contribuido en una u otra forma en la elaboración de esta investigación. Gracias a todos. vii viii INDICE Página RESUMEN 1 1. INTRODUCCIÓN 3 1.1. LIGNINA 1.2. DEGRADACIÓN DE LA LIGNINA POR LOS HONGOS DE LA PODREDUMBRE BLANCA DE LA MADERA. 1.2.1. Peroxidasas ligninolíticas 1.2.2. Lacasas EC.1.10.3.2. 1.2.3. Otras oxidasas y reductasas 1.2.4. Metabolitos de bajo peso molecular 1.2.5. Actividad ligninolítica 1.3. LACASAS: UN TIPO DE MULTICOBRE OXIDASAS 1.4. PROCESAMIENTO Y SECRECIÓN DE LAS LACASAS 1.5. EVOLUCIÓN DE LAS LACASAS 1.6. ASPECTOS BIOTECNOLÓGICOS 1.7. GENÉTICA MOLECULAR DE LOS HONGOS DE LA PODREDUMBRE BLANCA DE LA MADERA (WRF) 1.7.1. Hongos modelo 1.7.2. Regulación génica de las lacasas 1.8. PHANEROCHAETE FLAVIDO-ALBA 1.9. OBJETIVOS 5 8 9 11 11 12 12 13 20 20 24 26 26 28 29 34 2. MATERIALES Y MÉTODOS 35 2.1. MICROORGANISMOS Y VECTOR 2.1.1. Origen y mantenimiento del hongo 2.1.2. Cepa de Escherichia coli 2.1.3. Vector 2.2. MEDIOS DE CULTIVO 2.2.1. Medios de cultivo para el hongo 2.2.1.1. Medio YMPG 37 37 37 37 37 37 37 ix 2.2.1.2. Medio BL 2.2.2. Medio de cultivo de bacterias 2.3. CULTIVO Y MANIPULACIÓN DEL HONGO 2.3.1. Obtención de inóculos 2.3.2. Cultivos para aislamiento de DNA 2.3.3. Cultivos inducidos para aislamiento de RNA 2.3.3.1. Inducción por fenoles 2.3.3.2. Inducción por hierro y cobre 2.4. ANALISIS ENZIMÁTICOS 2.4.1. Cuantificación de proteínas 2.4.2. Cuantificación de la actividad lacasa 38 39 40 40 40 40 40 41 42 42 42 2.5. MÉTODOS DE ANÁLISIS DE ÁCIDOS NUCLÉICOS 42 2.5.1. Aislamiento del DNA genómico 2.5.2. Aislamiento del DNA plasmídico de Escherichia coli 2.5.3. Aislamiento de RNA 2.5.4. Amplificación de DNA genómico 2.5.4.1. Amplificación por PCR 2.5.4.2. Amplificación por PCR Inversa 2.6. Síntesis del cDNA mediante RT-PCR 2.6.1. Transcripción Reversa (RT) 2.6.2. Amplificación del cDNA (cPCR) 2.7. Ligación de los productos de amplificación en pGEM®-T 2.8. Transformación de células de Escherichia coli XL1-Blue 2.8.1. Obtención de células electrocompetentes 2.8.2. Transformación de células electrocompetentes 2.9. Secuenciación de ácidos nucleicos 2.10. Cuantificación de la expresión del gen pfaL mediante Real Time Q-PCR 2.10.1. Síntesis del cDNA 2.10.2. PCR 2.11. Métodos bioinformáticos 2.11.1. Identificación de secuencias en bases de datos 2.11.2. Comparación de secuencias 2.11.3. Diseño de cebadores 2.11.4. Identificación del promotor de pfaL 42 44 44 45 45 45 46 46 46 47 47 47 48 48 x 49 49 49 50 50 50 51 52 2.11.5. Identificación de la señal de secreción de PFAL 2.11.6. Análisis filogenético de la proteína deducida. 3. RESULTADOS 52 53 55 3.1. CARACTERIZACIÓN DE LA SECUENCIA GÉNICA pfaL 3.2. AISLAMIENTO DEL cDNA pfaL 3.3. ANÁLISIS DEL cDNA Y DE LA PROTEÍNA DEDUCIDA 3.4. IDENTIFICACIÓN DE INTRONES DEL GEN pfaL 3.5. ANÁLISIS FILOGENÉTICO DE LA LACASA PFAL 3.5.1. Alineamiento con lacasas fúngicas 3.5.2. Alineamiento de PFAL con ferroxidasas fúngicas 3.5.3. Alineamiento con lacasas fúngicas y ferroxidasas 3.5.4. Identificación de Aminoácidos conservados en PFAL 3.5.4.1. Aminoácidos de PFAL conservados en lacasas de ascomicetos 3.5.4.2. Aminoácidos de PFAL relacionados con unión al Fe 3.6. IDENTIFICACIÓN DE LA SEÑAL DE SECRECIÓN 3.7. IDENTIFICACIÓN DEL PROMOTOR DE PFAL 3.8. REGULACIÓN DE LA EXPRESIÓN DE LA LACASA POR FENOLES, HIERRO Y COBRE 3.8.1. Regulación por fenoles 3.8.2. Regulación por Metales 3.8.2.1. Regulación por deficiencia de Fe 3.8.2.2. Regulación por adición de Fe 3.8.2.3. Regulación por Cobre 57 60 64 67 70 70 70 71 74 74 77 79 81 4. 89 DISCUSIÓN 4.1. ESTRUCTURA DEL GEN pfaL 4.2. FILOGENIA DE PFAL 4.3. ESTRUCTURA PRIMARIA DE PFAL 4.3.1. Aminoácidos conservados de PFAL 4.3.2. Glicosilación 4.3.3. Péptido señal 4.4. ELEMENTOS PUTATIVOS DE REGULACIÓN TRANSCRIPCIONAL 4.5. REGULACIÓN TRANSCRIPCIONAL DE pfaL 84 84 86 86 88 88 91 92 93 93 94 94 95 xi POR COMPUESTOS FENÓLICOS 4.6. REGULACIÓN TRADUCCIONAL/POSTRADUCCIONAL DE PFAL POR VAINILLINA, Cu Y Fe. 4.6.1. Regulación por vainillina 4.6.2. Regulación por cobre 4.6.3. Regulación por hierro 97 99 99 102 105 5. CONCLUSIONES 107 6. BIBLIOGRAFÍA 113 RELACIÓN DE FIGURAS Y TABLAS FIGURAS Figura 1. Figura 2. Figura 3. Figura 4 Figura 5. Figura 6. Figura 7. Figura 8. Figura 9. Figura 10. xii Página Enlaces comunes entre las unidades fenilpropano en la lignina de maderas blandas. Esquema de la estructura química parcial de la lignina de coníferas. Organización de los dominios y sitios activos del cobre en MCBPs. .A. Ciclo catalítico de las lacasas. B. . Ciclo catalítico del sistema lacasa-mediador. Esquema de la organización de las MCBPs. Arbol filogenético de MCBPs de tres dominios. Efecto de la adición de compuestos aromáticos sobre el crecimiento y la actividad lacasa de P. flavido-alba. Amplicón pp12 (cDNA y péptido) de la lacasa de P. flavidoalba Secuencia F21 de DNA genómico (248 bp) de P. flavido-alba Amplificación inversa (PCR-I) de productos religados con T4 DNA ligasa, tras digerir el gDNA con XbaI o XhoI. 6 7 15 17 21 23 32 33 57 58 Figura 11. Figura 12. Figura 13. Figura 14. Figura 15. Figura 16. Figura 17. Figura 18. Figura 19. Figura 20. Figura 21. Figure 22. Figura 23. Estrategia para aislar y secuenciar el gen pfaL de Phanerochaete flavido-alba. cDNA de pfaL Amplicón del extremo 3´ cDNA de pfaL Amplicones internos cDNA de pfaL Amplicón del extremo 5´. Secuencia del cDNA (2108pb) del gen pfaL Secuencias del gDNA y cDNA deducidos para el gen de la lacasa de P. flavido-alba (pfaL)y de la proteina codificada Árbol filogenético construido con las secuencias de PfaL, las ferroxidasas y lacasas fúngicas seleccionadas. Sitios de la región reguladora del gen pfaL de Phanerochaete flavido-alba. Actividad lacasa (A) y cantidad de RNAm (B) de pfaL afectadas por adición de compuestos aromáticos. Actividad lacasa (A) y cantidad de RNAm (B) de pfaL afectadas por hierro y cobre. Comparación de la distribución de exones/intrones entre el gen pfaL de P. flavido-alba y los genes mco de P. chrysosporium con mayor número de intrones (mco1 y mco4) Flujo metabólico propuesto en P. chrysosporium en presencia de vainillina. Relación entre la homeostasis del cobre y del hierro en levaduras. 59 60 61 62 63 64 72 82 85 87 92 101 104 TABLAS Tabla 1. Tabla 2. Tabla 3. Tabla 4. Tabla 5. Composición de fenoles de la Polimerina y comparación con la fracción polimérica oscura (PMA) del OMW. Compuestos aromáticos utilizados para inducir la actividad lacasa en cultivos de P. flavido-alba. Cebadores usados para amplificar secuencias del gen de la lacasa de P. flavido-alba, PfaL. Alineamiento (WU-Blastp, EMBL) de la secuencia deducida para la lacasa PfaL con las proteínas de la base de datos UNIPROT. Oligopéptidos de la lacasa de P. flavido-alba purificada y 30 32 51 64 xiii Tabla 6. Tabla 7. Tabla 8. Tabla 9. Tabla 10. Tabla 10A-B. Tabla 10C-D. Tabla 11. Tabla 12. Tabla 13. Tabla 14. xiv similitud con la secuencia aminoacídica deducida del cDNA pfaL Alineamiento de los intrones del gen pfaL. Porcentajes de identidad de los intrones del gen pfaL. Estructura génica de lacasas de hongos de la podredumbre blanca de la madera y las proteínas que codifican Secuencias seleccionadas de lacasas y ferroxidasas para análisis filogenético de PFAL Posiciones conservadas en los sitios del cobre. Comparación de PFAL y MCOs de P. chrysosporium (Pc-MCOs) con lacasas vegetales y lacasas fúngicas. Secuencias L1-L2 Secuencias L3-L4 Aminoácidos de unión al hierro en la ferroxidasa Fet3 (gi2828219) de S. cerevisiae conservados en PfaL, MCOs de P. chrysosporium, otras ferroxidasas y lacasas de ascomicetos Aminoácidos de unión al Fe en Fet3 (gi2828219) de S. cerevisiae conservados en PFAL y MCOs que están ausentes en lacasas. Predicción del péptido señal de PfaL Regiones reguladoras identificadas en el gen pfaL. Localización respecto al inicio de la traducción. 67 68 68 69 73 75 75 76 78 79 80 83 ABREVIATURAS AAO ABTS BCS BL BLAST BPS cDNA Clustal cPCR Ct Cu DHP DMF DNA Fe Fer gDNA IDCB LB Lip MCBPs MCOs MnP mRNA OMW PCR PCR-I pfaL PfaL PMA QPCR rDNA 18S Aril alcohol oxidasa 2.2’-azinobis (3-etil-benzotiazolin-6-sulfonato) Batocuproindisulfonato sódico Medio basal para el cultivo de P. flavido-alba programa de búsqueda de alineamiento básico local Batofenantrolindisulfonato sódico DNA complementario Programa de Alineamiento múltiple de secuencias Amplificación de la primera cadena de cDNA obtenida por RT Ciclos umbral de PCR en tiempo real Cobre Polímero de coniferil alcohol deshidrogenado (Lignina sintética) N,N-Dimetilformamida Ácido desoxirribonucleico Hierro Ácido ferúlico DNA genómico Sitios Interdominios de unión al cobre Medio de cultivo Luria Bertani Lignin peroxidasa Proteínas multicobre azules Proteínas multicobre Manganeso peroxidasa RNA mensajero Olive oil mill wastewater (alpechín) Reacción en cadena de la polimerasa PCR inversa gen de la lacasa de P. flavido-alba Lacasa de Phanerochaete flavido-alba Pigmento polimérico del alpechín PCR cuantitativa en tiempo real Gen del RNA ribosomal 18S xv RNA RT-PCR Tir Van YMPG xvi Ácido ribonucleico Transcripción reversa del mRNA y cPCR Tirosol Vainillina medio de cultivo “Yeast-Malt-Peptone-Glucose” RESUMEN P. flavido-alba elimina la toxicidad del alpechín (OMW) mediante la oxidación de sus componentes aromáticos monoméricos y poliméricos. Varias enzimas ligninolíticas fueron identificadas como responsables de dicha transformación. Entre ellas la lacasa fue la única enzima detectada en cultivos añadidos de compuestos aromáticos monoméricos del alpechín, sugiriendo un papel importante en su detoxificación. En este trabajo se identificó y caracterizó el gen de lacasa de P. flavido-alba (pfaL). Tiene un tamaño de 2755 pb, Está formado por una secuencia de codificación de 1704 pb, 19 intrones y por una región reguladora UTR-3’ de 244 pb. Aguas arriba del gen se identificaron secuencias consenso putativas de regulación general y típicas de lacasas, tales como una caja TATA, una caja CAAT y elementos de respuesta a metales (MRE), a antioxidantes (ARE), a xenobióticos (XRE), a regulación por nitrógeno (NIT2) y carbono (Mig1p). La estructura del gen y de la proteína son similares a las de multicobre oxidasas de P. chrysosporium, formando juntas una rama filogenética independiente de las ferroxidasas y de las lacasas. Sin embargo, PfaL comparte con estas enzimas, residuos característicos de unión al hierro y al cobre que la hacen particularmente interesante. En la proteína deducida (567 amino ácidos) se identifica una señal de secreción de 43 amino ácidos. El péptido extracelular debe tener un alto grado de glicosilación para justificar el tamaño de la proteína purificada previamente. Mediante QPCR en tiempo real se estableció que la regulación de la expresión es compleja y puede estar determinada por múltiples mecanismos relacionados con estrés químico y oxidativo. La expresión de pfaL se induce manera diferencial por el pigmento polimérico del alpechín, el ácido ferúlico o la dimetilformamida. A pesar de aumentar la cantidad de transcrito ninguna de estas sustancias modificó significativamente la actividad lacasa extracelular. En cambio, la vainillina que proporcionó cultivos con mayor actividad lacasa, disminuyó la cantidad de mRNA acumulado, sugiriendo una regulación de tipo traduccional/postraduccional. De manera similar, se encontró que el cobre y el hierro regulan la producción de lacasa traduccional/postraduccionalmente, quizás a través de mecanismos de la homeóstasis del cobre-hierro. 1. INTRODUCCIÓN 1. Introducción 4 1. Introducción 1.1. LIGNINA El término lignina deriva del latin “lignum” que significa madera. Es uno de los tres componentes escenciales de la madera junto con los carbohidratos y los extraibles (Sjostrom, 1981) La lignina es un polímero heterogéneo compuesto por unidades metoxiladas de fenilpropano que se encuentra formando parte de los tejidos vasculares de las plantas superiores, a las que proporciona soporte estructural, impermeabilidad y resistencia frente al ataque microbiano y al estrés mecánico (Higuchi, 1990). Por su abundancia, representa una de las reservas más importantes de carbono orgánico en los sistemas terrestres. Aunque la estructura exacta de la lignina se desconoce, los nuevos métodos analíticos de identificación de productos de hidrólisis y métodos espectroscópicos han permitido conocer muchas características estructurales de este polímero. En la Figura 1 se muestran algunas de las más comunes uniones encontradas en la lignina de maderas blandas. De todas ellas la unión más abundante es la unión βO- 4 (Adler, 1977). La síntesis de este polímero se produce por la unión de radicales fenoxi que se originan en la oxidación enzimática de alcoholes precursores. Se han implicado numerosas enzimas oxidativas en la síntesis de la lignina, fundamentalmente peroxidasas y fenol oxidasas. La reactividad de los radicales fenoxi ha llevado a asumir que la polimerización de estos precursores es un proceso que ocurre al azar (Kirk y Farrell, 1987), sin embargo, una proteína puede dirigir esta polimerización (Davin et al., 1997). La Figura 2 muestra un esquema de un modelo de la lignina de confieras en donde se muestran diversos tipos de subestructuras entre ellas la recientemente descubierta en lignina de maderas blandas por Karhunen et al. (1995a,b) y conocida como dibenzodioxocina. 5 1. Introducción OCH 3 H 3C O O O OCH 3 H 3 CO O O OCH 3 OCH 3 O O β-O-4 [45-50] α−Ο−4 [6−8%] H 3CO 5-5 [18-25%] OCH 3 O O H 3C O O OCH 3 H 3 CO O O OCH 3 O β−β [3%] 4-O-5 [4-8%] β-5 [9-12%] O H 3 CO OCH 3 o H 3 CO o o O OCH 3 Dibenzodioxocina [18-25%] β-1[7-10%] Figura 1. Enlaces comunes entre las unidades fenilpropano en la lignina de maderas blandas. Entre paréntesis la proporción aproximada de cada una de ellos. 6 1. Introducción OH HO L ig n in -O OH L ig n in OMe OMe O HO O O OMe M eO O HO OH HO OMe OH M eO OH OMe O O OMe OH OMe O M eO HO OMe HO OH O HO O HO HO OH O OMe HO OH O HO OH HO HO HO O OH M eO OH O OMe HO OMe HO O O HO HO O CHO OMe OMe O HO O OH OH OH M eO HO O OMe O OH HO OH O OMe HO HO OMe O M eO O O OMe OH OM e OH Figura 2. Esquema de la estructura química parcial de la lignina de coníferas. Las unidades fenilpropano no se unen directamente sino a través de diferentes tipos de enlaces que se muestran en la Figura 1. 7 1. Introducción Aunque existen varios grupos de organismos capaces de degradar la lignina, son los hongos ligninolíticos pertenecientes al grupo “de la podredumbre blanca de la madera” ("white rot fungi", WRF), los principales responsables de la mineralización de este polímero. Tras la degradación de la lignina se acumulan celulosa y hemicelulosa que son las responsables del color blanquecino característico que aparece durante la degradación de la madera por estos hongos (Cheng y Chang, 1985). Aunque Phanerochaete chrysosporium es el hongo mejor conocido del grupo, no es el más selectivo en la degradación de la lignina (Broda et al., 1996). 1.2. DEGRADACIÓN DE LA LIGNINA POR LOS HONGOS DE LA PODREDUMBRE BLANCA DE LA MADERA. La degradación de la lignina por estos hongos es un proceso oxidativo, multienzimático e inespecífico, en el que participan enzimas ligninolíticas y otras enzimas, junto con metabolitos aromáticos sintetizados por los hongos (Higuchi, 1990; 2004). Se han identificado dos familias de enzimas ligninolíticas extracelulares, las peroxidasas y las lacasas. Estas enzimas son capaces de oxidar una gran variedad de compuestos aromáticos y se diferencian en sus requerimientos catalíticos, potencial de oxido-reducción, y en su modo de acción que puede ser directo o indirecto (mediante la cooperación de intermediarios; Daniel et al., 1989; Sutherland et al., 1991). 8 1. Introducción 1.2.1. Peroxidasas ligninolíticas Lignina peroxidasa (LiP) EC 1.11.1.14 Esta enzima fue descrita por primera vez en P. chrysosporium (Tien y Kirk, 1983; Glenn et al., 1983). Es una glucoproteína monomérica que contiene un grupo hemo. El ciclo catalítico de esta peroxidasa comienza con la oxidación de la enzima por peróxidos, dando lugar al compuesto I, que contiene un complejo oxo-Fe4+ radical catiónico de la porfirina (Schoemaker et al., 1994) (1).. El compuesto I es reducido en dos pasos consecutivos por compuestos aromáticos, para formar el Compuesto II y la enzima nativa (2) (3). El exceso de peróxido da lugar a una reacción irreversible en la que se forma el Compuesto III que es inactivo(4).. LiP Fe 3+ + H2O2 LiP C-I Fe4+ = O (P).+ + H2O LiP C-I Fe4+ = O (P).+ + RH LiP C-II Fe4+ = O (P) + R* + H+ LiP C-II Fe4+ = O (P) + RH + H+ LiP Fe3+ + R* + H2O LiP C-II Fe4+ = O + H2O2 LiP C-III Fe3+O2.+ (1) (2) (3) (4) La LiP se diferencia de otras peroxidasas por su alto potencial de oxido-reducción, lo que le permite oxidar directamente compuestos aromáticos no fenólicos, y por tanto, la mayor parte de las unidades de la lignina (Tien, 1987). En P. chrysosporium se han encontrado al menos 8 isoenzimas de esta enzima, siendo la H8 la mejor caracterizada (Tien y Kirk, 1988). Tras clonar los genes de varias isoenzimas (Pribnow et al., 1989), se ha comprobado que no responden de la misma manera a los factores reguladores. Peroxidasas que oxidan el manganeso (MnP) EC 1.11.1.13 Mas frecuente que la producción de LiP es la producción de peroxidasas que oxidan el Mn2+ a Mn3+ en presencia de peróxidos. Estas peroxidasas manganeso dependientes se han descrito en la mayoría de los hongos ligninolíticos de la podredumbre blanca de la madera (Hatakka, 1994). 9 1. Introducción El Mn3+ formado en la reacción es un fuerte oxidante, que estabilizado con ácidos dicarboxílicos secretados por el hongo, actúa como intermediario en la oxidación de compuestos fenólicos y no fenólicos (Hammel et al., 1989). El ciclo catalítico de la MnP es semejante al de otras peroxidasas incluidas la LiP y transcurre a través de la formación de intermediarios del enzima MnP C-I y MnP C-II. MnP Fe 3+ + H2O2 MnP C-I Fe4+ = O (P).+ + H2O MnP C-1 Fe4+ = O (P).+ + Mn2+ MnP C-II Fe4+ = O (P) + Mn3+ MnP C-1 Fe4+ = O (P).+ + RH MnP C-II Fe4+ = O (P) + R* MnP C-II Fe4+ = O (P) + Mn2+ MnP Fe3+ + Mn3+ + + H2O MnP C-II Fe4+ = O + H2O2 MnP C-III Fe3+O2.+ (1) (2a) (2b) (3) (4) El primer producto de la reacción de la MnP nativa con el peroxido de hidrógeno es el compuesto I (1). La reducción monoelectrónica del compuesto I por Mn 2+ conduce al compuesto II que de nuevo se reduce por Mn2+ para generar el enzima nativo. Mientras que el compuesto I puede ser reducido además de por el Mn2+ (2a), por otros donadores como los fenoles (2b), el compuesto II de la MnP solo puede ser reducido por Mn2+ por lo que depende de este catión para cerrar su ciclo (3). Al igual que la LiP la MnP es sensible a altas concentraciones de H2O2 que provocan la inactivación reversible del enzima al compuesto III que es un estado de oxidación inactivo catalíticamente (4). La mayoría de estas peroxidasas se caracterizan por depender del Mn2+ para cerrar su ciclo catalítico (reducción del compuesto II de la enzima, Schoemaker et al., 1994). Son enzimas que solo actúan de manera indirecta sobre la lignina. En P. chrysosporium se han detectado varias isoenzimas codificadas por tres genes (Pease y Tein, 1992). Además de estas peroxidasas dependientes de Mn2+, se han encontrado más recientemente en varios hongos del género Pleurotus (Martínez et al., 1996; Sarkar et al., 1997) y en Bjerkandera adusta (Heinfling et al., 1998) otras peroxidasas que oxidan eficazmente el Mn2+ y que no dependen de él para cerrar su ciclo catalítico. 10 1. Introducción Son peroxidasas que pueden oxidar compuestos aromáticos de forma directa (independientes de Mn2+) e indirectamente (con Mn2+). En Pleurotus eryngii se ha purificado una de estas enzimas y se ha comprobado que comparte propiedades catalíticas de la LiP y de la MnP de otros basidiomicetos. Además es capaz de oxidar fenoles y colorantes que no pueden oxidar directamente las LiPs ni las MnPs “convencionales” (Camarero et al., 1999). 1.2.2. Lacasas EC.1.10.3.2. Las lacasas son fenoloxidasas ampliamente distribuidas en plantas, hongos de diferentes clases (Gianfreda et al., 1999) y algunas bacterias (Diamantidis et al., 2000). Las lacasas se descubrieron en el látex de la planta Rhus vernicifera (Thurston, 1994), como la sustancia termolábil responsable del endurecimiento de la resina. En las plantas la lacasa tiene una función protectora frente a lesiones sufridas por el vegetal, pues la oxidación de fenoles del latex por la lacasa provoca radicales libres que tienden a polimerizar lo que origina una cubierta protectora en la lesión. La función de la lacasa en los hongos no está clara, se ha relacionado con la morfogénesis, con la pigmentación de los conidios, con la formación de rizomorfos, con el desarrollo de cuerpos fructificantes (Thurston, 1994) con la patogénesis (Lewis y Yamamoto, 1990) y en la protección frente a compuestos fenólicos tóxicos liberados durante la degradación de la lignina (Bollag et al., 1988). 1.2.3. Otras oxidasas y reductasas En los WRF se han encontrado al menos dos oxidasas extracelulares: la glioxal oxidasa (Kersten, 1990) y la aril alcohol oxidasa (AAO) (Asada et al., 1995). El papel de estas enzimas en la degradación de la lignina es el de generar el peróxido de hidrógeno necesario para la actuación de las peroxidasas. Además ciertas reductasas de compuestos aromáticos, y oxidoreductasa de la celobiosa, parecen estar involucradas en la degradación de la lignina (Constam et al., 1991; Muhein y Leisola, 1991) . Sus funciones parecen ser la reducción de 11 1. Introducción quinonas y radicales fenoxilos evitando su toxicidad y la tendencia a la repolimerización, además de que al ser reducidos estos compuestos aromáticos, se convierten en sustratos de las enzimas ligninolíticas con lo que continua la degradación (Schoemaker et al., 1989). 1.2.4. Metabolitos de bajo peso molecular Los hongos ligninolíticos se caracterizan por producir gran cantidad y variedad de metabolitos extracelulares (Shimada e Higuchi, 1991). Algunos de estos compuestos participan en la degradación de la lignina; como mediadores de las enzimas ligninolíticas como el alcohol veratrílico o el ácido hidroxiantranílico mediadores de la LiP (Cancel et al., 1993) y la lacasa (Eggert et al., 1996a); otros como sustratos de las enzimas (como el glioxal o metilglioxal) para la glioxal oxidasa o los ácidos dicarboxílicos que estabilizan al Mn3+ producido por la MnP (Kishi et al., 1994). Además de estos metabolitos aromáticos estos hongos producen también especies reducidas del oxigeno como H2O2, anión superóxido (O2·-) y radical hidroxilo (OH.) que es oxidante fuerte (Backa et al., 1993). 1.2.5. Actividad ligninolítica P. chrysosporium produce el sistema enzimático ligninolítico durante el metabolismo secundario, en respuesta a la limitación en nitrógeno, carbono o azufre (Dosoretz y Grethelein, 1991; Faison y Kirk, 1985; Jeffries et al., 1981; Kirk et al., 1978). Sin embargo, en otros hongos de la podredumbre blanca de la madera, la degradación de la lignina se produce bajo condiciones de exceso en carbono y nitrógeno (Périé y Gold, 1991). De forma general en los laboratorios, la producción de enzimas ligninolíticas y la propia ligninolisis se producen bajo elevadas tensiones de O2 (Faison y Kirk, 1985) aunque en cultivos de Ganoderma australis la degradación selectiva de la lignina es estimulada por la limitación en nitrógeno y la baja tensión de O2 (Ríos y Eyzaguirre, 1992). Otros factores que afectan a la ligninolisis son la agitación de los cultivos, de forma diferente dependiendo del hongo (Waldner et al., 1988), y la presencia de un surfactante. 12 1. Introducción En los WRF, la acumulación de los enzimas extracelulares ligninolíticos puede variar dependiendo no sólo de la especie de hongo, sino también de las condiciones ambientales (Tuor et al., 1995). Además, aunque en ciertos hongos se detectan secuencias génicas que hibridan con sondas de la LiP de P. chrysosporium, no se detecta LiP en sus cultivos (Rüttimann et al., 1992). La complejidad en la producción de las enzimas ligninolíticas se encuentra ilustrada por la lacasa de P. chrysosporium. . Este hongo se consideró como una excepción entre los hongos de la podredumbre blanca de la madera por su incapacidad de producir lacasa (Gold y Alic, 1993). Sin embargo, en 1995, se detectó lacasa en cultivos con celulosa como fuente de carbono (Srinivasan et al., 1995) y más recientemente se purificaron parcialmente varias fracciones proteicas con actividad lacasa (Dittmer et al., 1997). Dado que en el genoma de P. chrysosporium no se han encontrado genes de lacasas, es probable que estas actividades enzimáticas pudieran corresponder a alguna de las multicobre oxidasas (MCOs) descritas mas recientemente (Larrondo et al 2003 y 2004) En otras especies del mismo género, como en P. sordida, no se ha detectado lacasa (Rüttimann-Johnson et al., 1994); en P. laevis no se detectó LiP, pero en cambio se detectaron elevados niveles de MnP y lacasa (Bogan y Lamar, 1996). En P. flavido-alba, hemos purificado una lacasa (Pérez et al., 1996) y descrito varias MnPs y LiPs (Ben Hamman et al., 1997). Este trabajo describe por primera vez el gen de la lacasa de P. flavido-alba y su regulación por fenoles y metales. Así mismo incluye la posición filogenética de la proteína en el contexto de la familia de las multicobre oxidasas (MCOs). Por ello es necesario introducir algunos aspectos de estas proteínas. 1.3. LACASAS: UN TIPO DE MULTICOBRE OXIDASAS Características estructurales y sitios de unión al cobre Las lacasas (p-difenol:dioxígeno oxidoreductasa, EC 1.10.3.2) junto con las ferroxidasas, ascorbato oxidasas, ceruplasmina y otras enzimas constituyen las proteínas multicobre oxidasas (MCOs). Las MCOs poseen secuencias y estructuras similares que intervienen en los sistemas redox relacionados con el ión cobre 13 1. Introducción (Messerschimdt A, 1997). También se denominan proteínas multicobre azules (MCBPs) por el hecho de que el ión cobre de tipo 1 (ver mas adelante), tiene un máximo de absorción a 610 nm, y da el color azul a estas proteínas. Históricamente, los átomos de cobre en proteínas multicobre han sido clasificados en tres tipos (tipo 1, tipo 2 y tipo 3) por sus propiedades espectroscópicas (Solomon et al., 1996). En el espectro UV–visible, el cobre tipo 1 (azul) muestra máxima absorción cerca de 610 nm y el cobre tipo 3 muestra máxima absorción cerca de 330 nm. Los cobre tipo 1 y tipo 2 son detectables por resonancia electrónica paramagnética (EPR), mientras el cobre dinuclear tipo 3 no es detectable. Los aminoácidos de estos centros de unión al cobre están distribuidos en cuatro regiones altamente conservadas (L1, L2, L3 y L4) típicas de lacasas que se extienden desde 8 hasta 24 residuos a lo largo de la secuencia de la proteína e incluyen tanto los aminoácidos específicos de unión al cobre como otros no específicos (Kumar et al., 2003) ) (Figura 3). Los aminoácidos de unión del cobre tipo 1 son dos histidinas, una cisteína y una metionina. Los primeros tres residuos son esenciales para la unión del cobre azul, formando un trígono fuerte con el ión cobre, mientras el enlace con el cuarto residuo (una metionina axial) es más distante y débil, pudiendo ser reemplazado por otros aminoácidos, tales como leucina o fenilalanina (Figura 3). Generalmente, los sitios de unión del cobre tipo 1 se encuentran dentro de un dominio. Pero las MCBPs poseen otros sitios de unión del cobre interdominios (IDCB), diferentes de los del tipo 1, que consisten solo de residuos de histidinas. En las MCOs, como las lacasas y las ceruplasminas, el sitio interdominio es trinuclear, compuesto de un cobre tipo 2 y dos cobres tipo 3 coordinados por ocho histidinas (cuatro de cada dominio) (Figura 3). En el caso de las nitrito reductasas, el sitio interdominio es un cobre tipo 2 mononuclear coordinado por tres histidinas (dos del primer dominio y uno del segundo dominio). 14 1. Introducción D1 D2 D3 Dominio 3 Dominio 1 1 2 3 AoZu 57 VVIHWHGILQ LcAb 60 VSIHWHGFFQ MvBO 91 NSVHLHGSFS DHGO 114 TAVHWHGIRL YD56 97 TALHFHGVVP YAK8 82 TSLHSHGLFQ CopA 96 TSIHWHGIIL CumA 102 TTIHWHGIRL Fet3 78 TSMHFHGLFQ Fet5 76 TSLHFHGLFQ L-1 99 103 128 156 140 124 136 142 121 123 321 -1-1GTFFYHGHLGMQRSAGLYGSL GTFWYHSHLSTQYCDGLRGAF RTLWYHDHAMHITAENAYRGQ GTSWYHSHFSLQYSNGLYGPL GTFWYHSHSSVQYGDGMRGVL GTYWVHSHDMSQYPDGLRTPF GTYWYHSHSGFQEQVGVVYGP GSYWYHPHVSSSEELGRGLVP GTYWYHSHTDGQYEDGMKGLF GTFWYHAHMGAQYGDGMRGAF 325 274 249 307 291 272 365 235 271 274 445 398 398 484 452 417 522 398 413 418 L-2 1 2 3 HPWHLHGHDF HPFHLHGHNF HPIHIHLVDF HPIHLHGHDF HPWHMHGHHF HPFHLHGHTF HPIHLHGMWS HPIHLHGMSF HPFHLHGHAF HPFHLHGHNF L-3 501 446 451 538 525 475 565 445 478 491 321 1 1 GVNAFHCHIEPHLHMGMGVVF GAWFLHCHIDWHLEAGLAIVF GVYMFHCHNLIHEDHDMMAAF GAWLLHCHLQYHASEGMALQY GKWWLHCHVEWHMMKGLGIVF GAWVIHCHIEWHMESGLLATF GRWAYHCHLLYHMEMGMFREV GTWMFHCHVIDHMETGLMAAI GVWFFHCHIEWHLLQGLGLVL GVWYFHCHVDWHLQQGLASGF L-4 Sitio mononuclear cobre tipo 1 (azul) Sitio interdominio trinuclear cobre tipo2/tipo3 Figura 3. Organización de los dominios y sitios activos del cobre en MCBPs. Se muestra un alineamiento de varias secuencias MCBPs alrededor de los sitios de unión al cobre en el primero y último dominio. Estos sitio corresponden con las designaciones de L1-L4 de Kumar et al. (2003). Los números 1, 2 y 3, arriba de los alineamientos indican posiciones consenso de los residuos de unión al cobre tipo 1, tipo 2 y tipo 3, respectivamente. Los residuos en rojo son del sitio del cobre tipo 1(azul). Los residuos en verde son los del sitio IDCB tipo 2/3. El número amarillo en la columna de la mitad es el número de residuos entre el Segundo y tercer fragmento de las secuencias. La secuencia ID (SWISSPROT) o numero de acceso (NCBI), el nombre común (si lo hay), y el origen de cada secuencia son: AoZu: ASO_CUCPM, ascorbato oxidasa de Cucurbita pepo var. melopepo; LcAb: LAC1_AGABI, lacasa de Agaricus bisporus; MvBO: BLRO_MYRVE, bilirubin oxidasa de Myrothecium verrucaria; DHGO: BAA08486, dihidrogeodin oxidasa de Aspergillus terreus; YD56: YD56_YEAST, YD56 de Saccharomyces cerevisiae; YAK8: FIO1_SCHPO, YAK8 de Schizosaccharomyces pombe; CopA: CPA1_PSESM, CopA de Pseudomonas syringae; CumA: NP_743195, CumA de Pseudomonas putida; Fet3: FET3_YEAST, ferric reductasa de Saccharomyces cerevisiae; Fet5: P43561, ferric reductasa de Saccharomyces cerevisiae. (Modificado de Nakamura et al, 2005). 15 1. Introducción La lacasa fúngica mejor estudiada es la de Trametes versicolor (Bollag y Leonowicz, 1984). Es una p-difenol O2 oxidoreductasa que se diferencia de la mayoría de fenoloxidasas en que produce agua en lugar de peróxido en la reducción del oxígeno. Son glucoproteínas con 4 átomos de cobre y de peso molecular variable (D´Souza et al., 1996). Los átomos de cobre están clasificados en tres tipos (1, 2 y 3) con diferentes propiedades (Shin et al., 1996; Yaropolov et al., 1994): Cobre tipo 1 (T1): es responsable del color azul de la proteína. Tiene una alta absorbancia en la región del visible (605 nm) provocada por la unión covalente cobre-cisteína. Debido al alto potencial redox de este cobre es el sitio donde ocurre la oxidación del sustrato. Cobre tipo 2 (T2): se caracteriza por no presentar absorbancia detectable en la región del visible y por tener una alta afinidad por aniones (F-, CN-...) que actúan como inhibidores de la actividad del enzima. Cobre tipo 3 (T3): es un complejo binario formado por un par de iones Cu2+-Cu2+ unidos por puente hidróxido con un máximo de absorbancia a 330 nm y un espectro de fluorescencia característico. La reacción que cataliza la lacasa es la siguiente: 4 AH + O2 4 A + 2 H2O [AH: Dador de electrones] Los cobres T2 y T3 forman un cluster trinuclear que es el lugar donde se reduce el oxígeno (Thurston, 1994). El cobre del centro T1 es el primer aceptor de electrones del sustrato. Después los electrones se transfieren secuencialmente al centro T2-T3 que tras recibir cuatro electrones reducen una molécula de oxígeno a agua. Así la oxidación monoelectrónica del sustrato va acoplada a la reducción por cuatro electrones del oxígeno molecular (Figura 4A). 16 1. Introducción A H2O Lacasaox Medred Substratoox O2 Lacasared Medox Substratored B Figura 4. A. Ciclo catalítico de las lacasas (Yaropolov et al. 1994). B. Ciclo catalítico del sistema lacasa-mediador. 17 1. Introducción La lacasa presenta una amplia especificidad de sustrato puesto que cataliza la oxidación de orto y para difenoles, amino fenoles, dímeros de lignina fenólicos e incluso no fenólicos en presencia de algunos intermediarios, (Kawai et al., 1987; Hatakka et al., 1991). La oxidación puede estar controlada por las diferencias en potencial redox entre los sustratos reductores y el cobre tipo 1 de la lacasa (Thurston, 1994). En general las lacasas tienen baja especificidad por estos sustratos en relación al oxígeno. La oxidación monoelectrónica de un sustrato por la lacasa implica la formación de un radical libre. Estos radicales posteriormente pueden ser oxidados a quinonas por el enzima o bien sufrir reacciones no enzimáticas variadas. La oxidación de fenoles frecuentemente produce reacciones de acoplamiento carbono-carbono y carbono-oxígeno entre los radicales lo que origina productos de mayor peso molecular que los sustratos es decir reacciones de polimerización. La tendencia de esta enzima de provocar polimerización no es una desventaja en biorremediación ya que la polimerización oxidativa de los contaminantes puede ser un método aceptable para su eliminación. Mientras que las lacasas solo pueden oxidar directamente subunidades fenólicas de la lignina (más fáciles de oxidar pero menos abundantes), pueden reaccionar indirectamente con componentes no fenólicos de la lignina mediante la participación de mediadores redox que son compuestos de bajo peso molecular fácilmente oxidados por estas produciendo radicales muy inestables y muy reactivos que pueden a su vez oxidar sustratos más complejos (Figura 4B). Se han citado mediadores sintéticos como el [2.2’-azinobis (3-etil-benzotiazolin-6sulfonato)] (ABTS) ó 1-hidroxibenzotriazol (Bourbonnais et al., 1997; Pickard et al., 1999) y naturales como 4-hidroxibenzóico, 4-hidroxibenzol y 3hidroxiantranilato (Bourbonnais y Paice, 1990, Eggert et al. 1996, 1997). En Panus tigrinus, se ha aislado una forma amarilla de la lacasa, probablemente formada como consecuencia de la unión de la proteína a moléculas derivadas de la lignina. Esta enzima, cataliza la oxidación del alcohol veratrílico y de compuestos diméricos no fenólicos sin la participación de mediador (Leontievsky et al., 1997). En cultivos de Pleurotus ostreatus se ha aislado una lacasa que no posee el color azul característico debido a la falta del cobre tipo 1 (azul) (Palmieri et al., 1997). Curiosamente, en lugar de los cuatro átomos de cobre por cadena, esta lacasa blanca contiene dos iones de zinc, un ión de hierro y un ión de cobre, y a pesar de ello exhibe actividad lacasa. Sin embargo, el análisis de la secuencia de 18 1. Introducción aminoácidos reveló que todos los residuos de unión al cobre están conservados. Otra lacasa blanca, extraída de Phellinus ribis, lleva dos iones de zinc y un ión manganeso (Min et al., 2001). También otra lacasa blanca fue encontrada en Volvariella volvacea (Chen et al., 2004). La presencia de lacasa blanca plantea muchas dudas. Por ejemplo, es posible cambiar los iones cobre por otros metales?; también, el cambio de ión cobre por otro metal, haría perder la función de la enzima MCBP?. Para responder la primera pregunta, Nakamura et al, (2005), propusieron que el cobre tipo 1 puede ser reemplazado por Fe/Mn, así perdiendo el color azul; los cobres del tipo 3 serían reemplazados por iones zinc, y el de tipo 2 permanecería igual. La actividad es inhibida por interrupción de la transferencia electrónica (por haluros, azida, cianuro e hidróxidos) o por quelación del cobre y por modificación de los aminoácidos por cambios conformacionales (Gianfreda et al., 1999). Diferentes lacasas tienen diferente tolerancia hacia estos inhibidores (Xu, 1996). Para la utilización de la lacasa con fines biotecnológicos y medioambientales, así como para el conocimiento de las propiedades de la enzima se requieren grandes cantidades de la proteína, por lo que se han realizado muchos estudios para identificar los mejores inductores y métodos de aislamiento y purificación. La producción enzimática depende no solo del tipo de hongo sino de las condiciones nutricionales empleadas, de la presencia o ausencia de inductor, tiempo de inducción, naturaleza y composición del medio de cultivo y sobre todo de la manipulación genética. Los principales inductores de esta actividad incluyen compuestos fenólicos relacionados con la lignina (Gianfreda et al., 1999) aunque también compuestos no derivados de la lignina y diferentes extractos vegetales de diversa naturaleza han demostrado ser buenos inductores (Salas et al., 1995). Pérez et al. (1998) y Ruiz (1999) han demostrado la inducción de la lacasa de Phanerochaete flavido-alba por alpechín. 19 1. Introducción 1.4. PROCESAMIENTO Y SECRECIÓN DE LAS LACASAS Los genes de lacasas presentan secuencias en su extremo N-terminal que codifican los llamados péptidos señal. Estos péptidos señal dirigen a las lacasas hasta el espacio extracelular siguiendo una ruta de secreción en la que se suceden diferentes eventos: un plegamiento cotraduccional en el retículo endoplásmico donde se forman los puentes disulfuro (Freeman, 1989), la incorporación de una secuencia precursora (Glucosa3-Manosa9-Glucosa-N-acetilglucosamina2) que se une a la asparagina de la mayoría de las secuencias NXT/S de la proteína (Gavel y Von Heijne, 1990), y la unión de iones calcio que estabilizan la estructura de la apolacasa resultante. Luego, en el aparato de Golgi, tiene lugar la adición de los iones cobre (Taylor, 2005) y un procesamiento adicional de los carbohidratos antes de ser secretadas. En ascomicetos, como Saccharomyces cerevisiae y Melanocarpus albomyces, se han detectado procesamientos C-terminal (propéptidos), previos al procesamiento N-terminal que parecen influir en la activación de la lacasa (Bulter et al., 2003; Kiiskinen y Saloheimo, 2004). 1.5. EVOLUCIÓN DE LAS LACASAS Ryden y Hunt (1993) propusieron una hipótesis evolutiva de las MCBPs, con base en análisis de secuencias y de las estructuras cristalinas. Estos autores señalan que las MCPBs poseen multidominios de unión al cobre correspondientes a secuencias repetidas en tandem, que se originaron a partir de un solo dominio de unión al cobre tipo 1. Las rusticianinas de la familia de cupredoxinas son las enzimas más relacionadas con las MCBPs que poseen un solo dominio (Kanbi et al., 2002). Ello ha permitido sugerir que el dominio de estas cupredoxinas se ha multiplicado y modificado en el tiempo, creando sitios interdominios de unión al cobre (IDCB: tipo 2 y tipo 3) y sitios de unión a sustratos, tal como se encuentran en las MCBPs contemporáneas (Figura 5). 20 1 1. Introducción 2 1 2 1 1 2 1 Cupredoxina 1 1 de dominio Sitio BCB perdido en el segundo dominio 2 2 Creación sitio tipo 2 2 2 Duplicación [X] a id rd Pé o 3 tip [Y] 1 Creación sitio tipo 3 io sit Nitrito reductasa 2 2 1 [C] 1 1 2 Cobre tipo 1 (azul) 1 2 2 Si el tio B se C gu B p nd er o d do ido m in en io 1 1 [A] [B] 2 Cobre tipo 3 1 2 io in m do 3 un ,2, de 1 Ascorbato n tios ó ci si Lacasa e r id a s In rd Pé oxidasa 3 Cobre tipo 2 2 Triplicación pérdida sitio tipo 2,3 2 4 1 5 1 2 2 1 2 o io id in m rd do pe B do BC un eg tio s Si el en 6 Ceruplasmina Figura 5. Esquema de la organización de las MCBPs. Los óvalos indican dominios BCB (union de cobre azul). Puntos azules y verdes indican los átomos de cobre del tipo 1 y tipo2/tipo3, respectivamente. Flechas sólidas indican la vía evolutiva propuesta y las flechas entrecortadas indican vías evolutivas alternativas. Las MCBPs actuales se muestran a la derecha. Cinco proteínas hipotéticas de dos dominios: [X], [Y] y [A] tienen sitios BCB en ambos dominios, mientras [B] y [C] tienen un solo sitio BCB en el primero y segundo dominio, respectivamente. Las MCBP hipotéticas [A], [B], y [C] tienen sitios IDCB trinucleares, mientras [Y] tiene un sitio IDCB mononuclear. Esta figura es tomada de Nakamura et al. (2005). 21 1. Introducción En esta hipótesis, un par de dominios de la cupredoxina inicial se duplicó hasta generar MCBPs de seis dominios, como en las ceruplasminas, y la adición de un dominio para generar MCBPs de tres dominios, como en las ascorbato oxidasas y lacasas. Debido a la similitud en estructura y función de los sitios IDCB del tipo 2 y del tipo 3 (sitio trinuclear) en las ceruplasminas, ascorbato oxidasas y lacasas, Murphy et al. (1997) propuso una evolución dependiente. Así por ejemplo, las ceruplasminas al perder tres dominios formaron proteínas tipo lacasas; o viceversa, las lacasas al duplicar/adicionar dominios formaron las ceruplasminas. Para explicar ambas relaciones filogenéticas, se propone la existencia de un intermediario evolutivo de MCBPs con dos dominios que incluyen residuos IDCB trinucleares de unión al cobre (Nakamura et al., 2003). Surge así, tres tipos de MCBPs intermediarios con dos dominios (A, B y C en la Figura 5), cuya diferencia radica en la localización de los residuos del dominio de unión al cobre azul (BCB). El intermediario A tiene residuos en ambos dominios, mientras el B tiene los residuos solo en el segundo dominio, y el intermediario C los tiene solo en el primer dominio. Estas diferencias significan que los sitios de unión al cobre azul evolucionaron de manera regresiva; es decir, el tipo A sería el más antiguo, pues contiene residuos en ambos dominios; el tipo B y el tipo C perdieron residuos BCB del primero y segundo dominio, respectivamente. En la figura 5, el intermediario tipo C puede ser un ancestro directo de la nitrito reductasa, mientras que el intermediario tipo B, lo sería de lacasas/ascorbato-oxidasas y ceruloplasminas. Entre las MCBPs, las de tres dominios forman el grupo más diverso en término de secuencias, funciones y distribución. Se encuentran en hongos, plantas, insectos (Dittmer et al., 2004; Parkinson et al., 2003) y en bacterias (Alexandre y Zhulin, 2000; Claus, 2003). El árbol filogenético de las MCBPs de tres dominios se divide en tres regiones, cada región corresponde a un taxón representativo de bacterias, plantas y hongos (incluye insectos) (Figura 6). 22 1. Introducción PLANTAS Ascorbato oxidasa, Lacasa Sin cobre azul INSECTOS Lacasa BACTERIA Lacasa, CueO, PcoA , PpoA, CotA, CumA Hongos Lacasa Fet3, Fet5 Figura 6. Arbol filogenético de MCBPs de tres dominios. Las ramas están separadas de acuerdo a los grupos de organismos: plantas, hongos, bacterias e insectos. Las regiones cuyas secuencias no poseen los residuos de unión al cobre azul tipo 1 están sombreadas. Fet3 y Fet5 son ferroxidasas de Saccharomyces cerevisiae; CueO y PcoA son proteínas de la homeóstasis del cobre en E. coli; PpoA es una polifenol oxidasa con actividad tirosinasa de Marinomonas mediterranea; CotA es un componente de la endospora de Bacillus subtilis; CumA es una proteína de la cubierta de la espora de Pseudomonas con actividad Mn-oxidativa. El alineamiento de secuencias multiples y la construcción del árbol filogenético fueron hechos usando el programa ClustalW (http://www.ebi. ac.uk/clustalw/). Tomado de Nakamura et al (2005). En el árbol filogenético de las MCBPs de tres dominios, las lacasas de insectos forman un grupo independiente en medio de la región de lacasas de hongos (Fig. 2). Hasta el momento, han sido clonadas y caracterizadas dos lacasas del gusano del tabaco (Manduca sexta) y una del mosquito de la malaria (Anopheles gambiae) 23 1. Introducción (Dittmer et al., 2004). La principal diferencia entre estas lacasas y las de los hongos y plantas es la extensión de la secuencia amino-terminal con varios residuos conservados de cisteína, aromáticos y polares. La principal función de las lacasas de insectos es la esclerotización de la cutícula en la epidermis (Binnington y Barrett, 1988), donde se ha observado actividad oxidativa de estas enzimas sobre sustratos catecólicos (Sugumaran et al., 1992; Sugumaran y Nellaiappan, 1990). Estas enzimas también han sido encontradas en el intestino, en túbulos de Malpighio y en la glándula colleterial, donde, sin embargo, su función es desconocida. Recientemente, también se ha identificado una lacasa en el veneno de avispas parasitoides (Parkinson et al., 2003). 1.6 ASPECTOS BIOTECNOLÓGICOS Los organismos productores de lacasas han evolucionado para adaptarse a diferentes condiciones ambientales. En tales condiciones cuando las lacasas son secretadas, funcionan de manera inusual. Esta habilidad adaptativa es importante en términos de aplicaciones industriales, como por ejemplo, en blanqueo de textiles (Novozymes A/S, Dinamarca), y de pulpa de papel (Addleman y Archibald, 1993; Reid y Paice, 1994), síntesis orgánica, bioremediación y detergentes de lavado. También para desarrollar métodos con el fin de introducir características deseables a otras enzimas, como lo puede ser la estabilidad térmica a altas temperaturas (Suzuki et al., 2003) o en la durabilidad a altas condiciones ácido/básicas (Xu et al., 1998; Shirai et al., 2001). Las principales aplicaciones de las lacasas se encuentran en la industria del papel, concretamente en el blanqueo biológico y en la detoxificación de efluentes. Existen distintas patentes a base de lacasas inmovilizadas solas o con mediadores como Pulp-Zyme®,Novo-Nordisk (que utiliza lacasa-ABTS). En relación a la detoxificación de efluentes, existen procesos que se basan en la decoloración y detoxificación en continuo del efluente con hongos inmovilizados, tal como el cultivo inmovilizado en alginato de T. versicolor productor de lacasa (Archibald et al., 1990). 24 1. Introducción Otra aplicación de estas enzimas y de los hongos productores es la degradación de contaminantes medioambientales. Los cultivos de T. versicolor inducidos para la producción de lacasa, oxidan el antraceno y el benzo-[α]-pireno. Además, la lacasa purificada oxida el benzo-[α]-pireno tras la adición de ABTS (Collins et al., 1996). Las lacasas naturales no siempre son adaptables para aplicaciones industriales. Las principales limitantes son: (i) la necesidad de usar un mediador redox para oxidación indirecta de sustratos diferentes a fenoles y arilaminas, incluyendo lignina. Los mediadores pueden actuar como agentes donadores de electrones debido a los radicales libres formados con la lacasa, expandiendo el rango de sustratos para la enzima, tales como compuestos no-fenólicos e hidrocarburos aromáticos policíclicos. Y (ii) su actividad óptima es generalmente a pH ácido, lo que impide su uso en ambientes neutros. Técnicas de ingeniería genética permitirán obtener lacasas recombinantes modificadas con alto potencial de oxidación, amplio rango de sustratos, o pH neutro. Ello implicaría realizar mutagénesis dirigida en lacasas cuyas estructuras cristalinas hayan sido estudiadas, o DNA “shuffling” (mutagénesis al azar in vitro, seguida de selección). Sin embargo, estas metodologías requieren un sistema de expresión con alta eficiencia de transformación y alta capacidad de secreción de metaloenzimas activas. Aunque todas las lacasas fúngicas poseen una estructura conservada en los sitios activos, exhiben una alta diversidad en el resto de la proteína y en su parte polisacárida, además de las isoformas producidas por muchos hongos (Mayer y Staples, 2002). Esta compleja situación, junto con la falta de un método eficiente de transformación para basidiomicetos, ha estimulado la búsqueda de organismos heterólogos de expresión para incrementar la producción de lacasas y estudiar su estructura y propiedades catalíticas. Algunas lacasas han sido expresadas en Saccharomyces cerevisiae (Cassland y Jonson, 1999; Kojima et al., 1990; Larsson et al., 2001), Pichia pastoris (Gelo-Pujic et al., 1999; Jonsson et al., 1997; Otterbein et al., 2000), Trichoderma reesei (Saloheimo y Niku-Paavola, 1991), Aspergillus oryzae (Berka et al., 1997; Ducros et al., 1997; Xu et al., 1999; Yaver et al., 1996 y 1999), Aspergillus niger (Record et al., 2002) y Yarrowia lipolytica (Madzak et al., 2005). En la actualidad se han obtenido las estructuras cristalinas de: (i) 25 lacasa inactiva (desglicosilada y sin cobre) de C. cinereus (Ducros et al., 1998 y 2001); 1. Introducción (ii) (iii) (iv) lacasas activas de los basidiomicetes Trametes versicolor y P. cinnabarinus (Antorini et al., 2001 y 2002; Piontek et al., 2002); lacasa activa glicosilada de T. versicolor conjugada con un sustrato (Bertrand et al., 2002); lacasa del ascomicete Melanocarpus albomyces (Hakulinen et al., 2002). Para el diseño de lacasas con fines de aplicación industrial, el desarrollo de eficientes sistemas de expresión permiten llevar a cabo estudios de mutagénesis dirigida (Gelo-Pujic et al., 1999; Xu et al., 1998 y 1999) o evolución in vitro. Además, un experimento DNA shuffling del gen de una lacasa requiere el análisis de al menos 104 transformantes para obtener un mutante con las propiedades de interés (Madzak et al., 2005). Por otra parte, con el fin de comparar los efectos de varias mutaciones es necesario seleccionar transformantes con el mismo número de copias y loci integrados. 1.7. GENÉTICA MOLECULAR DE LOS HONGOS PODREDUMBRE BLANCA DE LA MADERA (WRF) DE LA 1.7.1. Hongos modelo P. chrysosporium ha servido de organismo modelo para estudios en genética molecular de los hongos de la podredumbre blanca. Ello ha permitido establecer metodologías para el estudio de su biología y maquinaria ligninolítica, tales como producción de auxótrofos (Gold et al. 1982), análisis de recombinantes (Alic y Gold, 1985; Raeder et al., 1989b; Krejci y Homolka, 1991; Gaskell et al., 1994), purificación rápida de DNA y RNA (Haylock et al., 1985; Raeder y Broda, 1985), expresión diferencial (Birch 1998; Kurihara et al., 2002; Assmann et al., 2003), cariotipificación mediante electroforesis en campo pulsado (Gaskell et al., 1991; D’Souza et al., 1993; Orth et al., 1994), y transformación genética mediante complementación de auxótrofos (Alic et al., 1989,1990,1991; Alic, 1990; Randall et al., 1991; Akileswaran et al., 1993; Zapanta et al., 1998) y por resistencia a antibióticos (Randall et al., 1989,1991; Randall y Reddy, 1992; Gessner y Raeder, 1994; Ma et al., 2003). La eficiencia de transformación es relativamente baja y la 26 1. Introducción ruptura de genes es difícil (Alic et al., 1993), aunque se han descrito algunos estudios de expresión génica (Gettemy et al. 1997; Birch et al. 1998; Ma et al., 2001). Algunos aspectos de la biología molecular de P. chrysosporium han sido descritos en las revisiones de Alic y Gold (1991), Pease y Tien (1991), Gold y Alic (1993), Cullen y Kersten (1996) y Cullen (1997). P. ostreatus es otro organismo modelo de WRF utilizado para establecer protocolos de transformación (Yanai et al., 1996; Honda et al., 2000; Irie et al., 2001; Sunagawa y Magae, 2002) y metodologías de mapeo físico (Larraya et al., 1999) y genético (Eichlerova y Homolka, 1999; Eichlerova- Volakova y Homolka, 1997; Larraya et al., 2000, 2002). T. versicolor ha sido también transformado con vectores de resistencia a antibióticos (Bartholomew et al., 2001; Kim et al., 2002a) y ha permitido demostrar mutación génica (Dumonceaux et al., 2001). El principal avance de investigación fue el obtenido por el US Department of Energy’s Joint Genome Institute (JGI), quien ha sequenciado el genoma total de P. chrysosporium. El ensamble de las secuencias aleatorias (“shotgun sequencing”) ha dado un tamaño de 30 Mpb del genoma, el cual se encuentra disponible libremente en la web (www.jgi.doe.gov/ whiterot). La cepa utilizada para la secuenciación fue un derivado homocariótico (Stewart et al., 2000) del dicarion BKM-F-1767. La comparación con otros genomas de hongos (por ejemplo, Neurospora crassa; Kupfer et al., 1997) predijo aproximadamente 8500 proteínas con significativos alineamientos Smith-Waterman. Tales proteínas solo podrán ser verificadas mediante el análisis del cDNA. De este modo, junto con el ascomycete N. crassa (Galagan et al., 2003), P. chrysosporium es uno de los primeros genomas disponibles de hongos filamentosos. La importancia de conocer la genética molecular de estos genomas eucarióticos abre nuevas áreas de exploración relacionadas con la degradación de la lignocelulosa y con los procesos evolutivos implicados. Como se mencionó anteriormente, el genoma de P. chrysosporium no contiene secuencias codificantes de lacasas. Sin embargo, alguna evidencia bioquímica ha permitido detectar actividad lacasa (Podgornik et al., 2001). Recientemente, se han descrito varias secuencias levemente relacionadas con ferroxidasas transportadoras de hierro (Fet3), con ascorbato oxidasas y con lacasas. Una de esas secuencias, la 27 1. Introducción mco1, ha sido caracterizada como una ferroxidasa extracelular (Larrondo et al., 2003) pero su función no ha sido aclarada con certeza. La ausencia de lacasas convencionales en P. chrysosporium no descarta su papel en la degradación de lignina por otros hongos. Además de su actividad oxidativa sobre unidades fenólicas de lignina a radicales fenoxi, se ha demostrado que en presencia de ciertos mediadores, la lacasa puede despolimerizar lignina sintética (Kawai et al., 1999) y deslignificar pulpa de madera (Bourbonnais et al. 1997; Call y Muncke, 1997), confirmando de este modo su actividad ligninolítica. De otra parte, frecuentemente los genes de lacasas se presentan como familias multigénicas (Cullen 1997) y se distribuyen ampliamente entre los hongos degradadores de lignina (Thurston 1994; Youn et al. 1995; Mayer y Staples, 2002). Los WRF, tales como Pycnoporus cinnabarinus degradan eficientemente la lignina, y al contrario de P. chrysosporium, secreta lacasas pero no peroxidasas. Dos genes de lacasas han sido identificados y caracterizados en este hongo (Eggert et al. 1998; Temp et al. 1999). Además, un mutante “lac- ” de P.cinnabarinus fue incapaz de degradar DHP marcado con C14. Estos datos confirman la importancia degradativa de la lacasa (Eggert et al. 1997). 1.7.2. Regulación génica de las lacasas La actividad lacasa en los basidiomicetos en general está codificada por varios genes (Jönsson et al., 1995; Saloheimo et al., 1991). Los estudios de secuenciación génica y del cDNA han puesto de manifiesto que la mayoría de las lacasas muestran secuencias que suelen codificar para 520-550 aminoácidos incluido el péptido de secreción y que existen secuencias muy conservadas como las correspondientes a la unión del cobre (Thurston, 1994). Generalmente, se ha descrito que los genes de lacasas son regulados diferencialmente y los patrones de regulación difieren substancialmente entre especies (Wahleithmer et al. 1995; Yaver y Golightly 1996; Yaver et al. 1996; Smith et al. 1998; Palmieri et al. 2000; Soden y Dobson, 2001). Los transcritos de la lacasa de P. radiata son rápidamente detectados en condiciones limitadas en nitrógeno (Saloheimo y Niku-Paavola, 1991). En Trametes villosa, lcc1 es fuertemente inducido en cultivos añadidos de 2,5-xylidina, mientras los niveles de 28 1. Introducción transcritos de lcc2 no cambian. Por el contrario, experimentos Northern blots no detectaron transcritos lcc3, lcc4, y lcc5 en todas las condiciones (Yaver y Golightly, 1996; Yaver et al. 1996). Tres lacasas de Rhizoctonia solani (lcc1, lcc2, 1cc3) son transcritas en bajos niveles constitutivos y pueden ser reprimidas en cultivos adicionados de p-anisidina. Sin embargo, la lcc4 de R. solani es expresado en niveles muy altos, inducibles por adición de p-anisidina. En R. solani, los genes lcc1, lcc2, lcc3 están agrupados, separados de lcc4, lo que sugiere una relación entre la organización genómica y la regulación transcripcional (Wahleithmer et al. 1995). También se ha establecido una regulación transcripcional de las lacasas inducibles por cobre y por otros metales (Karahanian et al., 1998; Palmieri et al., 2000; Soden y Dobson, 2001; Galhaup et al., 2002). 1.8. PHANEROCHAETE FLAVIDO-ALBA De acuerdo a la página web del National Center for Biotechnology Information, National Library of Medicine, Bethesda, MD, USA, P. flavido-alba es un microorganismo del Reino Fungi, Phylum Basidiomycota, Clase Hymenomycetes, Orden Aphyllophorales, Familia Corticiaceae. P. flavido-alba fue seleccionado por nuestro grupo de investigación de entre otros WRF por su capacidad de decolorar el 90% del color soluble en residuos de la industria papelera frente al 80% de decoloración que alcanzó P. chrysosporium (Pérez et al., 1997). Hasta el momento se han detectado en cultivos de P. flavido-alba actividades peroxidasas (LiP y MnP) y lacasa. Al menos se producen 5 isoenzimas de LiP de pIs entre 3.2 y 3.9 y una masa molecular comprendida entre 30 y 40 kDa, muy parecidas a las descritas en P. chrysosporium (Ben Hamman et al., 1997 y 1999a). La lacasa resulta especialmente interesante por su masa molecular de 98,5 kDa y pI menor de 3.5 (Pérez et al., 1996). En cuanto a las aplicaciones biotecnológicas nuestro grupo ha demostrado la capacidad de P. flavido-alba para eliminar la toxicidad del alpechín (Martínez et al., 1998), transformar las mezclas aromáticas típicas del alpechín (Madrid et al., 29 1. Introducción 1995) y decolorar tanto el pigmento mayoritario del alpechín como el propio alpechín (Tabla 1) (Ben Hamman, 1998). Tabla 1. Composición de fenoles de la Polimerina (Capasso et al., 2002) y comparación con la fracción polimérica oscura (PMA) del OMW (Pérez et al., 1987), determinados por oxidación con CuO y HPLC. Fenoles 30 Polimerina PMA Hidroxitirosol - - Acido 3,5-dihidroxibenzoico + - Acido 3,4-dihidroxifenilacético - - Catecol + - p-tirosol - + Acido p-hidroxibenzoico + + Acido vainíllico + + p-hidroxibenzaldehido + + Acido siringico - + Acido 2,3-dihidroxibenzoico - - Vainillina + + Acido p-hidroxipropiónico - + Metilcatecol - - Siringaldehido + + Acido p-cumárico + + Acido ferúlico + + Acido salicílico + - 1. Introducción Igualmente, se demostró que P. flavido-alba transforma eficazmente la mezcla de ácidos fenólicos del alpechín. En la degradación de estos compuestos parecen estar implicadas las peroxidasas ligninolíticas, ya que la transformación es más eficaz en cultivos inducidos para la producción de LiP y en menor medida en los cultivos que estimulan la producción de MnP. Sin embargo, los concentrados de LiP, muestran un insignificante efecto degradador de esta mezcla, sugiriendo que además de éstas pueden estar implicadas otras enzimas o metabolitos producidos por el hongo (Madrid et al., 1995). La decoloración del alpechín por P. flavido-alba está asociada a modificaciones en la distribución de tamaños moleculares de los componentes del pigmento, concretamente provoca la pérdida del componente o componentes de mayor tamaño molecular (Ben Hamman et al., 1999b). Esto sugiere que la decoloración por P. flavido-alba es un proceso en el que ocurre, bien la despolimerización de los componentes de mayor tamaño o la repolimerización de los de menor tamaño molecular, lo que a su vez provoca la acumulación de moléculas de tamaños intermedios. También se comprobó que la decoloración del alpechín va acompañada de una disminución del 90% en su contenido fenólico y de su destoxificación (Martínez et al., 1998; Ben Hamman, 1998). Pérez et al. (1998) han caracterizado las enzimas ligninolíticas presentes en los sobrenadantes de cultivos de alpechín decolorados. Al comparar los patrones de enzimas extracelulares en estos cultivos decolorados con los cultivos control no adicionados del residuo, comprobaron que el alpechín modifica significativamente las MnPs producidas. Así mismo, se puso de manifiesto que la lacasa es fuertemente inducida por el residuo. En ningún caso detectaron actividad LiP ni ninguna proteína de masa molecular entre 39-40 kDa, que es el tamaño molecular aparente de las LiPs de P. flavido-alba. Parece por tanto, que las MnPs y la lacasa podrían ser las enzimas ligninolíticas implicadas en este proceso de transformación del alpechín. La producción de lacasa de P. flavido-alba es afectada por compuestos aromáticos (Figura 7, Tabla 2). Altas producciones de lacasa se obtuvieron en cultivos añadidos de p-hidrobenzaldehido o vainillina, mientras que con otros inductores (por ejemplo, veratril alcohol, guaicol o ácido ferúlico) los niveles de actividad no fueron significativamente diferentes a los del cultivo control. 31 4H3MBA 4HBNZA 3,4DMB 4H3MB 4HBENZ 4H3MCIN 2-MPHE 3,4-DHCIN 3,4-DMO BENZ 3,4,5-TMB 3,4-DHB CONT 3,4,5-THB 4HCIN Crecimiento Actividad Lacasa 1. Introducción Figura 7. Efecto de la adición de compuestos aromáticos sobre el crecimiento y la actividad lacasa de P. flavido-alba. Crecimiento (columnas blancas) se expresa en % de peso seco respecto al cultivo control (CONT). Actividad específica (columnas grises) se expresa en proporción respecto al cultivo control. (Tomado de De la Rubia et al., 2002). Tabla 2. Compuestos aromáticos utilizados para inducir la actividad lacasa (Fig. 7) en cultivos de P. flavido-alba. Símbolo 4HCIN Símbolo 2MPHE Aromático guaiacol 4H3MCIN ácido ferúlico 4HBENZ 4H3MB ácido p-hidroxibenzoico ácido vainíllico BENZ Acido protocatéquico 3,4,5-ácido trimetoxibenzoico ácido benzoico 3,4DMB ácido verátrico 3,4DMO veratril alcohol 4HBNZA p-hidroxibenzaldehido 3,4DHCIN ácido caféico 4H3MBA vainillina 3,4,5THB 3,4DHB 3,4,5TMB 32 Aromático Acido p- cumárico ácido gallito 1. Introducción La proteína purificada mostró un espectro de absorción típico de proteínas tipo multicobre y un rango de actividad oxidativa en ABTS a un pH óptimo común a otras lacasas. Oxidó o-y p- difenoles (catecol, hidroquinona) y fenoxi-ácidos, pero no oxidó tirosina; también mostró mayor afinidad a metilhidroquinona y derivados dimetoxi (DMPs), no tanto para los monometoxilados. Las características físicoquímicas de la lacasa inducida son similares a la constitutiva en cuanto a pI (2.8– 3.5), secuencia N-terminal y masa molecular (98.7 kDa y 81.3 kDa desglicosilada), pero poco similares a lacasas típicas, especialmente en su secuencia N-terminal, alta masa molecular, alto porcentaje de glicosilación y altos Km. Igualmente, algunas particularidades de esta lacasa han sido encontradas, tales como oxidación de Mn II (De la Rubia et al., 2002) y oxidación de Fe II (Lucas et al., 2005), lo que sugiere que esta enzima posee otras características especiales para su estudio y aplicación biotecnológica. En experimentos previos a la realización de este trabajo, nuestro grupo de investigación obtuvo una sonda de cDNA de 141 pb (llamada sonda “pp12”). (Figura 8). La secuencia “pp12” contiene los sitios de unión al cobre L3 y L4 de P. flavido-alba que corresponden con los mismos sitios conservados de lacasas de diferentes hongos basidiomicetos y que están ubicados cerca al dominio Cterminal. Con esta sonda se detectó por hibridación un fragmento XbaI de 4 kb de gDNA de P. flavido-alba. 5`ACTAGTGATTCATCCTTTCCATTTTCATGTCATAGCCCCTGGATCCTCGGCTTCGACTCGACAACATA CCAAGGCCAGACTCCTAACCCGACCAACCCGTTGAGGCGTGACACCTTCGTCGCCGAGCGTGGATGGCTT ATGCTGCGCTACATCTCCGACAACCCCGGTCCCTGGATCCTGCACTGCCACATAGAATCCCGCGGCCATG GCGGCGGAGCATGCGACGTCGGGCCCAATTCGCCCTATAGTGAGTCGTATTACAATT-3` HPLHFHGHSPWILGFDSTTYQGQTPNPTNPLRRDTFVVPSGGWLMLRYISDNPGPWILHCHI Figura 8. Amplicón pp12 (cDNA y péptido) obtenido por RT-PCR de cultivos de P. flavido-alba utilizando los “primers” correspondientes a los sitios consenso L3 y L4 de las lacasas (Kumar et al 2003; aminoácidos subrayados). 33 1. Introducción 1.9. OBJETIVOS En virtud del conocimiento actual de la lacasa de P. flavido-alba y dada su potencial aplicación ambiental, el presente trabajo se propuso identificar y caracterizar la secuencia génica que codifica dicha enzima. Dentro de este amplio objetivo se planteó: 1. Caracterizar el gen de la lacasa tanto a nivel del genoma como del transcriptoma del hongo; 2. Analizar la regulación de la expresión del gen en cultivos in vitro por moléculas de interés medioambiental: el pigmento polimérico del alpechín, fenoles monoméricos (vainillina, ácido ferúlico y tirosol) y metales (Fe y Cu). Además la lacasa de P. flavido-alba y las proteínas mas semejantes (las MCOs de P. chrysosporium) se han diferenciado filogenéticamente de lacasas y ferroxidasas fúngicas. 34 2. Materiales y Métodos 2. Materiales y Métodos 36 2. Materiales y Métodos 2.1. MICROORGANISMOS Y VECTOR 2.1.1. Origen y mantenimiento del hongo En este trabajo se ha utilizado el hongo Phanerochaete flavido-alba FPL 106507, cepa procedente de la colección del Forest Products Laboratory de Madison, WI, USA. El hongo fue cultivado a 30ºC durante 7 días en agar YMPG (“Yeast-MaltPeptone-Glucose) (Bonnarme y Jeffries, 1990) y conservado a 4ºC con resiembras periódicas. 2.1.2. Cepa de Escherichia coli La cepa de Escherichia coli utilizada en este trabajo fue XL1-Blue (Stratagene). Esta cepa es de alta eficiencia para la transformación de DNA plasmídico y permite la selección de clones recombinantes mediante la detección de actividad βgalactosidasa. 2.1.3. Vector pGEM®-T (Promega): Plásmido de pequeño tamaño y elevado número de copias, diseñado para clonar fragmentos de ADN procedentes de reacciones de PCR. Este vector posee el gen que confiere resistencia al antibiótico ampicilina así como el gen que codifica la β-galactosidasa de E.coli, la cual permite la identificación de clones recombinantes. 2.2. MEDIOS DE CULTIVO 2.2.1. Medios de cultivo para el hongo 2.2.1.1. Medio YMPG (Bonnarme y Jeffries, 1990) Este medio se usó para el crecimiento del hongo en medio sólido, en la preparación de inóculos y para aislamiento de DNA genómico. Su composición por litro es la siguiente: 37 2. Materiales y Métodos Glucosa 10 g Extracto de malta 10 g Peptona 2g Extracto de levadura 2g Asparagina 1g KH2PO4 2g MgSO4 7H20 1g Tiamina hidrocloruro 1 mg Agar 20 g Ajustar el pH a 4,5 con HCl concentrado. Autoclavar a 121ºC 20 minutos. 2.2.1.2. Medio BL El medio basal para la lacasa o medio BL es una modificación del medio basal descrito por Kirk et al. (1986) en la concentración de la fuente de nitrógeno: tartrato amónico 22mM; no se adicionaron aditivos como el Tween 20 y el alcohol veratrílico, y se omitió el MnSO4 de la solución de elementos traza. La composición por litro es la siguiente: Glucosa 10 g Tartrato amónico 2 g (22 mM) Tartrato de sodio 20 mM (pH 4.5) KH2PO4 2g MgSO4.7H20 0,5 g CaCl2 . 2H20 0,1 g Tiamina hidrocloruro 1 mg Solución de elementos traza sin MnSO4 70 ml 38 2. Materiales y Métodos La composición de la solución de elementos traza (por litro): Acido nitriloacético 1,5 g (pH 6,5) MgSO4 .7H20 3,0 g NaCl 1,0 g FeSO4. 7H20 0,1 g CoSO4 0,1 g CaCl2. 2H20 0,1 g ZnSO4 . 7H20 0,1 g CuSO4 . 5H20 0,01 g AlK(SO4)2 . 12H20 0,01 g H3BO3 0,01 g Na2MoO4 . 2H20 0,01 g Las dos soluciones se autoclavaron por separado a 121ºC durante 20 minutos, posteriormente se mezclaron en las cantidades señaladas. Finalmente se adicionó 1ml de solución de tiamina (stock de 1mg/ml) previamente esterilizada por filtración. 2.2.2. Medio de cultivo de bacterias Medio Luria-Bertani (LB) (Sambrook y Russell, 2001). Se utilizó como medio de cultivo rutinario para el crecimiento de Escherichia coli tras el proceso de transformación. La composición por litro: Triptona Extracto de levadura 10 g 5g NaCl 10 g Agar (para medio sólido) 15 g El medio líquido y sólido se esterilizó en autoclave a 121ºC durante 20 minutos. 39 2. Materiales y Métodos 2.3. CULTIVO Y MANIPULACIÓN DEL HONGO 2.3.1. Obtención de inóculos Para la preparación de los inóculos, se utilizaron matraces Erlenmeyer de 1000 ml, cada uno con 100 ml de medio YMPG líquido inoculado con micelio crecido en YMPG sólido. Los cultivos fueron incubados en reposo a 30ºC. Tras 15 días de crecimiento el micelio obtenido se trituró en esterilidad con una batidora (Waring Blender) durante 10 seg. La suspensión obtenida fue utilizada como inóculo al 5% en el volumen del medio a inocular. 2.3.2. Cultivos para aislamiento de DNA Para el aislamiento del DNA genómico, se prepararon cultivos de 25 ml de YMPG en matraces Erlenmeyer de 250 ml. inoculados como se describió en el apartado anterior. Los cultivos se incubaron durante 7 días a 30 °C. El micelio de cada uno de los matraces se obtuvo por filtración (filtros Nº 1238), se congeló rápidamente en nitrógeno líquido y se almacenó a -80ºC hasta su uso. 2.3.3. Cultivos inducidos para aislamiento de RNA 2.3.3.1. Inducción por fenoles Se prepararon cultivos en matraces de 250 ml con 25 ml de medio BL con los inductores de la lacasa - aromáticos fenólicos simples y pigmento polimérico de los residuos de las almazaras (PMA)- que fueron inoculados como en el apartado anterior e incubados a 30ºC en reposo. A los tres días de incubación se adicionaron los inductores a los cultivos. De acuerdo con Ben Hamman (1998), el PMA (pigmento polimérico del alpechín) se disolvió en agua destilada y se adicionó a una concentración final de 8,3 g/l (5 réplicas). Los inductores fenólicos monoméricos usados fueron vainillina, ácido ferúlico y tirosol (Fluka). Su elección se basó en estudios previos sobre la oxidación de componentes fenólicos del alpechín por Phanerochaete flavido-alba (Ben Hamman 40 2. Materiales y Métodos et al. 1999a y 1999b ; Pérez et al. 1996; de la Rubia, et al. 2002). Los fenoles monoméricos se disolvieron en N,N-dimetilformamida: agua (3:7 v/v), se esterilizaron en autoclave y se adicionaron separadamente a una concentración final de 1mM (5 réplicas por solución). Igualmente, se prepararon dos grupos de cultivos control (cultivos sin y con N,Ndimetilformamida) según los solventes usados en la preparación de las soluciones del PMA y de inductores aromáticos. Los cultivos con los inductores añadidos se incubaron a 30°C durante otros tres días más. El micelio de cada uno de los cultivos se obtuvo por filtración rápida (filtros Nº 1238), se congeló en nitrógeno líquido y se almacenó a -80ºC hasta su uso. La actividad lacasa se determinó en los fluídos extracelulares en cada uno de los cultivos. 2.3.3.2. Inducción por hierro y cobre Para este estudio se utilizaron tres tipos de medios de cultivo BL (control de metales), BL sin FeSO4 y BL sin CuSO4. La inoculación de estos cultivos fue similar a la del ensayo anterior. Al tercer día de crecimiento del hongo, tres cultivos en el medio BL sin FeSO4 se les añadió quelante del hierro, ”BPS” (batofenantrolinadisulfonato sódico) a 1 mM y a otros tres se les añadió sulfato ferroso amónico: (SO4)2Fe(NH4)2 a 25 µM. Un lote de tres cultivos en medio BL sin CuSO4 se les añadió quelante de cobre “BCS” (batocuproinadisulfonato sódico) a 1mM. Otro lote de tres cultivos se le añadió CuSO4 24 µM. Después de incubar con las sales a 30ºC durante tres días adicionales, el micelio de cada uno de los cultivos se recuperó por filtración, se congeló en nitrógeno líquido y se almacenó a -80ºC hasta su uso. La actividad lacasa se midió en los fluídos extracelulares de cada uno de los cultivos. 41 2. Materiales y Métodos 2.4. ANALISIS ENZIMÁTICOS 2.4.1. Cuantificación de proteínas La concentración de proteínas de los sobrenadantes de los cultivos se midió por el método de Bradford (1976) utilizando el reactivo y el procedimiento de Bio-Rad Protein Asssay en un espectrofotómetro Cintra 10e a 595 nm, teniendo en cuenta la capacidad de unión del colorante Azul de Coomassie a dichas moléculas. La absorción a 595 nm se transformó en µg/ml de sobrenadante, utilizando la extrapolación en una ecuación de regresión lineal estandarizada con albúmina de suero bovino (Sigma). 2.4.2. Cuantificación de la actividad lacasa La actividad lacasa se determinó mediante la oxidación del sustrato ABTS [2,2´azino-bis(3-etilbenzotiazolina-6-sulfonato)]. La mezcla de reacción contenía 4,6 mM ABTS, tampón glicina/NaCl (50 mM /20 mM, pH 3,0), una alícuota de 200 µl de la muestra y agua destilada hasta 1 ml. La oxidación del ABTS (CЄ = 29,3 mM-1 cm-1) se midió mediante el incremento en Absorbancia a 436 nm (Niku-Paavola et al., 1988) Una mU de actividad lacasa se definió como la cantidad de enzima requerida para la oxidación de 1 nmol de ABTS por minuto a 30°C. La actividad específica se determinó como mU`s de actividad por mg proteína o por mg de peso seco de micelio. Las medidas de peso seco fueron obtenidas de tres micelios que se secaron durante 24 h a 105ºC. 2.5. MÉTODOS DE ANÁLISIS DE ÁCIDOS NUCLÉICOS 2.5.1. Aislamiento del DNA genómico Siguiendo la metodología de Sambrook y Russell (2001) y González et al., (1992), el DNA genómico de P. flavido-alba se aisló y purificó del siguiente modo: 400 mg de micelio de cultivos en medio YMPG congelado a -80ºC, se trituraron con mortero y pistilo en nitrógeno líquido. 42 2. Materiales y Métodos Adición de tampón de lisis HSE1 y 0,1 volúmenes de SDS 10%. La mezcla se incubó a 65ºC durante 15 minutos. Adición de 1 volumen de tampón TE2. El lisado se mezcló con un volumen de fenol:cloroformo:alcohol isoamílico (25:24:1) (Sigma), se centrifugó a 5000 rpm y se recuperó la fase acuosa. El tratamiento con fenol se repitió hasta obtener una fase acuosa limpia. Adición de 0,1 volúmenes de acetato sódico 3M pH 5,2 y 2 volúmenes de etanol absoluto. Precipitación del DNA a -20ºC durante 30 minutos. Centrifugación en frío a 15000 rpm durante 15 minutos. Lavado del precipitado con 500 µl de etanol 70% y centrifugación a 15000 rpm durante 5 minutos. Secado al vacío y resuspensión en 100 µl de agua MilliQ estéril. Para eliminar restos de RNA se adicionó 1 µl de RNasa libre de DNasa de una solución stock (10 mg/ml) y se incubó a 37ºC durante 20 min. El DNA genómico se cuantificó espectrofotométricamente midiendo la absorbancia (A260) de una dilución de la muestra en TE. La concentración se calculó teniendo en cuenta que una unidad de absorbancia equivale a 50 µg/ml de DNA de doble cadena. Valores entre 1,7 y 2 de la relación A260/A280 permitieron determinar la pureza de la muestra. El DNAg se separo mediante electroforesis horizontal en gel de agarosa al 0,7%. La muestra se mezcló con el tampón de carga3 en una relación 5:1 y el potencial aplicado fue de 50-90 voltios. Tras tinción del gel con una solución de bromuro de etidio (0,5 µg/ml) durante 20 minutos, la banda del DNAg intacto se visualizó y fotografió en un procesador de imágenes (Bio Rad) con luz ultravioleta conectado a un ordenador, usando el programa Quantity One de Bio Rad. Se utilizaron los marcadores de peso molecular de Roche DNA λ digerido con Hind III (marcador II) y DNA λ digerido con Eco RI y Hind III (marcador III). 1 HSE: Hepes 10mM pH 6,9, Sacarosa 0,5M y EDTA 20mM TE: Tris-HCl 50mM pH 8 y EDTA 20mM 3 Tampón de carga: azul de bromofenol 0.25%, sacarosa 40% 2 43 2. Materiales y Métodos 2.5.2. Aislamiento del DNA plasmídico de Escherichia coli Cultivos de células crecidas en medio LB durante 16 horas, se precipitaron por centrifugación a 13000 rpm, desechando el sobrenadante. El plásmido se aisló mediante el kit “Quantum Prep® Plasmid Miniprep Kit” (Bio Rad), siguiendo las indicaciones del fabricante. Tras electroforesis el plásmido se identificó por comparación con el tamaño de los marcadores II y III (Roche) en un gel de agarosa al 0,7%. La cuantificación y la pureza del DNA se determinaron como en el apartado anterior. 2.5.3. Aislamiento de RNA El RNA total fue extraído y purificado del micelio crecido en medio BL mediante el “RNeasy Plant mini kit” (Qiagen), siguiendo las indicaciones del fabricante, que se describen brevemente. 100 mg de micelio se congeló en nitrógeno líquido y se maceró hasta un polvo fino con mortero y pistilo previamente tratados con NaOH (0,1 M), EDTA (1 mM) y H2O libre de RNasas. Adición de tampón lisis RLC®/β-mercaptoetanol (450 µl/5µl) y agitación vigorosa. Homogenización de la muestra a través de una columna “QIAshredder”y centrifugación 2 minutos a velocidad máxima. El filtrado se mezcló con 0,5 volúmenes de etanol. Unos 700 µl del filtrado se colocaron en una minicolumna “RNeasy” y se centrifugó 15 seg a 10000 rpm para adherir el RNA a la membrana de silica-gel. Lavado de la membrana dos veces con tampón RW1® (500 µl) y centrifugación a 10000 rpm durante 15 segundos. Lavado de la membrana con tampón RPE® (500 µl) y centrifugación a velocidad máxima durante 2 minutos. Una vez seca la membrana, el RNA se eluyó dos veces con 50 µl de agua MilliQ libre de RNasas. El RNA total se cuantificó por espectrofotometría a 260 nm y la concentración se calculó teniendo en cuenta que 40 µg/ml equivalen a una unidad de absorbancia. La integridad del RNA se verificó por electroforesis en gel de agarosa al 0,8% seguida de tinción con bromuro de etidio. El RNA total se usó para la síntesis de 44 2. Materiales y Métodos cDNA. El sistema de revelado fue el mismo que el utilizado para el DNA genómico. 2.5.4. Amplificación de DNA genómico 2.5.4.1. Amplificación por PCR Esta técnica se utilizó para la amplificación del DNA genómico (F21).Las reacciones se realizaron en un termociclador Perkin Elmer 9600. Se preparó una mezcla de reacción que contenía 1 µl de DNA genómico (10 ng/ µl), 5 µl de tampón de PCR 10x (Biotools), 2 µl de MgCl2 (50 mM), 1 µl de cada uno de los desoxinucleósidos trifosfatos (10 mM), 1 µl de cada uno de los cebadores (20 pM) franLac1 y franLac3R (Tabla 3) diseñados a partir de la secuencia pp12 (Fig. 8), 1 µl de DNA polimerasa (1 U/ µl) (Biotools) y agua MilliQ hasta un volumen de 50 µl. Las condiciones fueron las siguientes: un ciclo de desnaturalización a 95ºC durante 5 minutos, hibridación de los cebadores a 55ºC durante 30 segundos y elongación a 72ºC durante 20 segundos; 35 ciclos de desnaturalización a 94ºC durante 30 segundos, hibridación de cebadores a 55ºC durante 30 segundos y elongación a 72ºC durante 20 segundos. La amplificación se concluyó con una incubación final de 5 min a 72ºC. Una alícuota de 5 µl de la reacción de PCR y 3 µl de un marcador de DNA de 100 pb de tamaño (Biotools) se sometieron a una electroforesis en gel de agarosa al 3%. La electroforesis se desarrolló a 80 V en tampón TAE4 durante 45 min. Los amplicones se visualizaron con luz UV tras su tinción con bromuro de etidio. 2.5.4.2. Amplificación por PCR Inversa Ochman et al. (1990) El DNA genómico (300 ng) se digirió con una de las enzimas de restricción previamente seleccionadas (Xba I y Xho I, apartado 1.8) durante 3 h a 37°C en un 4 TAE: Tris-acetato 40mM pH 8 y EDTA 1mM 45 2. Materiales y Métodos volumen de 25 µl. La enzima se inactivó a 65ºC durante 15 min. La muestra digerida se circularizó en 100 µl de tampón 10x y T4 DNA ligasa según las indicaciones del fabricante (New England Biolabs). La mezcla obtenida se dializó en membranas de nitrocelulosa de 0,025 µm durante 15 minutos en agua MilliQ. Para la amplificación se usó “GeneAmp XL PCR kit” (Applied Biosystems). Siguiendo sus recomendaciones, en un tubo de 200 µl se agregó: 1 µl de DNA religado, 15 µl de tampón de PCR 3,3x, 4µl de la mezcla de dNTPs 10 mM, 0,4 µl de cada cebador 100 µM (AFPINV1 y AFPINV2) diseñados a partir del fragmento F21 (Tabla 3, apartado 2.11.3) , 4 µl de acetato de Magnesio 25 mM, 1 µl de DNA polimerasa rTth (2U/µl) (Applied Biosystems) y agua MilliQ hasta un volumen de 50 µl. La amplificación se realizó en un termociclador Mastercycler (Eppendorff) con el siguiente programa: desnaturalización inicial a 95ºC durante 2 minutos, 16 ciclos de desnaturalización a 95ºC durante 30 segundos, hibridación y extensión de cebadores a 68ºC durante 4 minutos; 12 ciclos de desnaturalización a 95ºC durante 30 segundos, hibridación y extensión de cebadores a 68ºC durante 4 minutos con aumentos de 15 segundos en cada ciclo; y una extensión final a 72ºC durante 10 minutos. El resultado de la amplificación se comprobó por electroforesis de una alícuota de 5 µl en agarosa al 0,7%, se tiñó con bromuro de etidio y se visualizó con luz ultravioleta. El resto de la muestra se desalinizó en una columna Microcon-100 (Amicon®). 2.6. Síntesis del cDNA mediante RT-PCR 2.6.1. Transcripción Reversa (RT) La RT se realizó con 25 U de “AccuScript RT” (Stratagene) a 42ºC durante 1 h en 20 µl de una mezcla con 1 µg de RNA total, 1x de tampón RT, 10 mM de ditiotreitol (DTT), 1 mM de cada dNTP y 6 ng/µl de oligo-dT. 2.6.2. Amplificación del cDNA (cPCR) La primera cadena de cDNA fue amplificada en alícuotas de 50 µl mediante tres reacciones consecutivas de PCR con la DNA polimerasa Pfu Ultra HF® (Stratagene), usando cebadores diseñados del gDNA (Tabla 3, apartado 2.11.3) y en condiciones establecidas en el protocolo del “kit AccuScript High Fidelity RTPCR System” (Stratagene). 46 2. Materiales y Métodos Los tres fragmentos amplificados fueron: cDNA -3`: desde el sitio consenso L3 de la lacasa (Kumar et al., 2003) hasta la cola poliA con los cebadores y Lacfin1 (directo) y dTCA (reverso). cDNA-5`: desde 5`-UTR hasta N-terminal de la proteína extracelular con los cebadores In1 (directo) y Nup1 (reverso) cDNA interno: desde el sitio N-terminal hasta el sitio L3 con los cebadores LacN3 (directo) y Lac3up (reverso). Los productos de amplificación se comprobaron en una electroforesis en agarosa al 2.0% usando alícuotas de 5 µl y se visualizaron como en el apartado 2.5.1. El resto de los productos de cPCR se purificaron en columnas Microcon 100 (Amicon®). 2.7. Ligación de los productos de amplificación en pGEM®-T Alícuotas de los fragmentos de cDNA purificados se incubaron por separado en una mezcla de ligación (Promega) con el vector de clonación pGEM®-T durante 12 horas a 4ºC. La mezcla contenía tampón de ligación 2x, 50ng de Vector pGEM®-T, producto de cPCR, 1 µl de T4 DNA ligasa (Promega) y agua MilliQ en un volumen final de 10 µl. 2.8. Transformación de células de Escherichia coli XL1-Blue 2.8.1. Obtención de células electrocompetentes El inóculo se obtuvo de un cultivo de la cepa crecido en 5 ml de LB adicionado con 10 µl de una solución de tetraciclina (10mg/ml). Tras 24 horas, 300 µl de este cultivo se usó de inóculo a 30 ml de LB. Se incubó a 37º en agitación hasta que la densidad óptica del cultivo a 600nm fue de 0,6. Las células se recuperaron por centrifugación a 4000 rpm durante 15 minutos a 4º C. Se desechó el sobrenadante y las células fueron resuspendidas en 30 ml de agua MilliQ estéril y fría. Se repitió la centrifugación y el lavado. 47 2. Materiales y Métodos Se recuperaron las células por centrifugación y se resuspendieron en 15 ml de agua MilliQ, estéril y fría. Se centrifugaron y se desechó el sobrenandante. Las células se resuspendieron en 600 µl de glicerol (10%) estéril y frío y se centrifugaron nuevamente. Finalmente, las células se resuspendieron en 100 µl de glicerol (10%) y se transformaron. 2.8.2. Transformación de células electrocompetentes 100 µl de células electrocompetentes se mezclaron con 10 µl del vector en una cubeta de eletroporación (Genotronics-F-620), la cual se colocó en un electroporador (Bio-Rad) y se aplicó un pulso de 2500 voltios. Se sacó la muestra y se colocó en un tubo eppendorff de 1,5 ml, donde se adicionó 890 µl de medio LB. El minicultivo se incubó a 37°C durante 2 horas. Posteriormente, se tomaron alícuotas de 20, 50 y 100 µl de las células transformadas, las cuales se sembraron en placas de agar-LB añadido con 40 µl de ampicilina (100 mg/ml), 4 µl de IPTG5 (200mg/ml) y 40 µl de X-Gal (2%). Las placas se incubaron a 37°C durante 12 horas. Los clones transformados fueron identificados por coloración y resistencia a la ampicilina, y se seleccionaron siguiendo los procedimientos de Sambrook y Russell (2001). 2.9. Secuenciación de ácidos nucleicos Los productos de amplificación o de clonación, previamente purificados y cuantificados, se secuenciaron utilizando el producto comercial ABI PRISMTM (Perkin-Elmer) que utiliza en la reacción de extensión la enzima AmpliTaq FS en un secuenciador automático Applied Biosystems modelo 373 STRECHT por el Servicio de Secuenciación del Instituto de Parasitología y Biomedicina López.Neyra (CSIC, Granada). La determinación de la secuencia se realizó mediante el 5 IPTG: isopropil tio-β-D-galactósido 48 2. Materiales y Métodos método de terminadores de cadena marcados con fluorocromos (Sanger et al. 1997). La extensión de secuencias clonadas en pGEM®-T se realizó en ambos sentidos con los cebadores universales SP6 (5´-ATTTAGGTGACACTATAGAA-3´) y T7 (5´TAATACGACTCACTATAGGG-3´). Para el caso de productos de amplificación, la extensión del DNA a secuenciar se realizó con el cebador específico (Tabla 3). La muestra a secuenciar se añadió con 6,4 pMol del cebador y agua MilliQ (volumen final de 12 µl). 2.10. Cuantificación de la expresión del gen pfaL mediante QPCR en Tiempo Real 2.10.1. Síntesis del cDNA La síntesis del cDNA se realizó a partir de 0,5 µg de RNA total usando la reversotranscriptasa “Multiscribe RT” (Applied Biosystems) según las condiciones del fabricante, que se detallan a continuación: Mezclar en un tubo de microcentrífuga 5 µl de tampón RT 10x, 2 µl de dNTPs 25x, 5 µl de cebadores aleatorios 10x, 2,5 µl de enzima 50 U/µl, 0,5 µg de RNA total y agua DEPC hasta 50 µl. La reacción se incubó 10 min a 25ºC y 120 min a 37ºC. 2.10.2. PCR Las PCRs cuantitativas a tiempo real se realizaron en el termociclador ABI PRISM 7700 Sequence Detection System. Cada reacción contenía 1 µl de cDNA (dilución de 5ng/µl), 10 µl TaqMan Universal PCR Master Mix 2x, 1 µl de la mezcla de cebadores específicos y sonda TaqMan marcada con FAM 20x, y agua DEPC hasta un volumen de 20 µl. El programa de PCR consistió de 50ºC/2 min, 95ºC/10 min, 40 ciclos de 95ºC/15 seg y 60º/1 min. La eficiencia de la amplificación se determinó mediante correlación lineal entre diluciones seriales del molde desde 0,1 ng hasta 20 ng versus ciclos umbral de PCRs, lo cual pudo representarse en una curva de regresión. Se consideran válidas aquellas correlaciones por encima del 90% y valores E (E=10-1/pendiente) cercanos a 2. 49 2. Materiales y Métodos Las cuantificaciones se realizaron a partir de cuatro reacciones de PCR independientes. El ciclo umbral (Ct) es el ciclo en el que comienza a detectarse el amplicón y fue calculado por el software de Applied Biosystems (MACRO) que acompaña al equipo de PCR en tiempo real. Los resultados se normalizaron con los niveles de Ct obtenidos para el gen control endógeno (rDNA 18S de Applied Biosystems). Los niveles de expresión relativa de transcrito pfaL se calcularon usando el método Delta-Delta (Livak y Schmittgen, 2001), que se resume en la siguiente expresión matemática: Niveles de expresión = Donde: Exp. gen/Exp.basal gen Exp.endógeno/Exp.basal endógeno = 2 -∆∆ Ct ∆ Ct = Ct (diana) – Ct (referencia) ∆∆ Ct = ∆ Ct – ∆ Ct (calibrador) El calibrador fue la muestra que se usó para comparar. 2.11. Métodos bioinformáticos 2.11.1. Identificación de secuencias en bases de datos Para la búsqueda de secuencias homólogas a las de la lacasa de P. flavido-alba, depositadas en las bases de datos del Genbank, se usó el algoritmo de alineamientos Blast, a través del servidor del NCBI (http://www.ncbi.nlm.nih.gov/) y del EMBL (http://www.ebi.ac.uk/), y la base de datos del genoma de Phanerochaete chrysosporium (http://genome.jgi-psf.org/whiterot). 2.11.2. Comparación de secuencias Los productos de secuenciación se analizaron con el programa Chromas (www.technelysium.com.au) y FASTA y se alinearon con múltiples secuencias de las bases de datos mediante el software ClustalW (www.ebi.ac.uk/clustalw). 50 2. Materiales y Métodos 2.11.3. Diseño de cebadores Los cebadores específicos (Tabla 3) para amplificación y secuenciación del gen de la lacasa se diseñaron manualmente. Los cálculos de Tm, %GC, dimerización y estructuras secundarias fueron determinados en el analizador de oligonucleótidos de Sigma-genosys (http://www.sigma-genosys.com/calc/DNACalc.asp). Tabla 3. Cebadores usados para amplificar secuencias del gen de la lacasa de P. flavido-alba, PfaL Cebador Secuencia Tm (ºC) Franlac1 5`-CGGCTTCGACTCGACAAC-3` 64.7 Franclac3 5`-AGTGCAGGATCCAGGGAC-3` 62.8 Lacfin1 5`-GACTCGACAACATACCAAG-3` 55.3 dTCA 5`-TAATACGACTCACTATAGGGTTTTTTTTTTTTCA-3` 66.9 Lac-N3 5`-ACACTGTCTCGCTTCCATC-3` 60.4 Lac3up 5`-AAGGTGTCACGCCTCAAC-3` 61.3 Hwhdwn 5`-ACTTTCCATTGGCACGGT-3` 63.0 Hwhup 5`-CCGTGCCAATGGAAAGT-3` 62.2 Pico17dwn 5`-AGACCCTGGTGTTCCTGAC-3` 61.6 In1 5`-GCCAGCGACCGCTTTGGTAT-3` 70.2 Nup1 5`-GATGGAAGCGAGACAGTGT-3` 60.4 AFPINV1 5`-TCAACGGGTTGGTCGGGTTAGGAGTC-3` 75.3 AFPINV2 5`-ATGGCTTATGCTGCGCTACATCTCCGAC-3` 75.7 AFP3 5`-CATTGGTACCGTATCTGTGG-3` 60.2 51 2. Materiales y Métodos AFP4 5`-GTATGCGACTCTCCCTGGAT-3` 62.9 AFP5 5`-GCAGACGGGTGGATGATTAG-3` 64.3 AFP6 5`-CAAGGAGAGGGAATGCGGTA-3` 66.1 2.11.4. Identificación del promotor de pfaL Con el fin de predecir la ubicación del promotor y de sitios generales de unión de factores de transcripción y algunos motivos putativos de lacasas en las regiones no codificantes hacia el extremo 5’ del gen pfaL, se emplearon los siguientes programas: Programa Dirección de Internet GenScan 1.0 http://genes.mit.edu/GENSCAN.html FRUITFLY http://www.fruitfly.org/cgi-bin/seq_tools/promoter.pl DRAGON 1.5 http://research.i2r.a-star.edu.sg/promoter/promoter1_5/DPF.htm Proscan 1.7 http://bimas.cit.nih.gov/molbio/proscan/ 2.11.5. Identificación de la señal de secreción de PfaL La identificación de la señal de secreción se realizó mediante el uso de los algoritmos SignalP-NN (neural networks model) (Nielsen et al. 1997 a,b) y SignalP-HMM (hidden Markov model) (Nielsen y Krogh 1998), a través del servidor http://www.cbs.dtu.dk/services/SignalP/. Los métodos incorporan una predicción de los sitios de ruptura y la presencia o no de péptido señal. 52 2. Materiales y Métodos 2.11.6. Análisis filogenético de la proteína deducida. Se reunieron por separado las secuencias de lacasas y de ferroxidasas disponibles a través del NCBI (http://www.ncbi.nlm.nih.gov). Lacasas y ferroxidasas se analizaron filogenéticamente por separado con el paquete informático MEGA (v 3.0). Luego de varios ciclos de alineamientos múltiples y análisis preliminares de distancias filogenéticas, estas secuencias fueron depuradas. Se eliminaron las lacasas con identidad mayor del 95% y las secuencias menores de 486 aminoácidos. Las ferroxidasas que tuvieran identidad mayor del 90 % también se eliminaron. Al final se obtuvo una colección de 41 secuencias de multicobre oxidasas, con las que se definieron las relaciones filogenéticas de PfaL con MCOs de P. chrysosporium, con lacasas y con ferroxidasas fúngicas. Tras alinear tales secuencias, se generó un árbol filogenético con el modelo Neighborn-joining de análisis de distancias evolutivas, usando el paquete MEGA versión 3.0 (Kumar et al., 2004). La fiabilidad de la predicción fue evaluada mediante 1000 muestreos al azar de los alineamientos (“bootstrap”). Finalmente, la presentación y selección de las secuencias consenso fueron elaboradas manualmente a partir de los alineamientos. 53 3. RESULTADOS 3.Resultados 56 3. Resultados 3.1. CARACTERIZACIÓN DE LA SECUENCIA GÉNICA pfaL En experimentos previos, utilizando la secuencia de aminoácidos de unión al cobre (L3-L4, Kumar, 2003) se diseñaron cebadores degenerados. Con ellos se había obtenido un fragmento de cDNA de P. flavido-alba (pp12 de 141 pb). Con pp12 se obtuvo una sonda marcada con digoxigenina. Esta sonda hibridó (Southern) con un fragmento (4,2 kb) del gDNA digerida con XbaI y con dos fragmentos XhoI (de 2 kb y de 4kb) (Comunicación personal de José Romera López y Antonio Suárez). A partir de la secuencia de cDNA pp12 de P.flavido-alba de 141 pb se diseñaron dos cebadores, “franLAC1” y “franLAC3r” (A y B en la Figura 11), con los que se amplificó el correspondiente fragmento de gDNA. Después de su clonación, el análisis de la secuencia “F21” de 248 pb, confirmó la presencia de la secuencia pp12 y de dos intrones (Figura 9). 5`TCGGCTTCGACTCGACAACATACCAAGGCCAGACTCCTAACCCGACCAACCCG TTGAGgtatgtgtgccagtcgcgagctgttaccaatttcattctcatacattgat gcagGCGTGACACCTTCGTCGTTCCGAGCGGTGGATGGCTTATGCTGCGCTACAT CTCCGACAACCgtgagtctacccataccatagtttgattcacactcatgatcgtt cgcccacagCCGGTCCCTGGATCCTGCACT-3 Figura 9. Secuencia F21 de DNA genómico (248 pb) de P. flavido-alba. La secuencia contiene pp12 (en mayúscula) y dos intrones. Los cebadores utilizados corresponden a las secuencias subrayadas. La secuencia “F21” permitió diseñar cebadores en orientaciones opuestas, AFPINV1 y AFPINV2, dirigidos hacia “arriba” de su extremo 5` y hacia “abajo” de su extremo 3` respectivamente, para amplificar el gen pfaL mediante PCR Inversa (PCR-I). El gDNA fue digerido con las enzimas de restricción Xba I y Xho I. Estas endonucleasas fueron escogidas en ensayos previos de hibridación Southern. 57 3.Resultados Los productos de la digestión del gDNA con tales enzimas se circularizaron con la enzima T4 DNA ligasa. Los fragmentos circulares formados (Ochman, 1990) se usaron como moldes en la amplificación inversa. El tamaño de los amplicones obtenidos (Figura 10) correspondió al tamaño de las señales detectadas en la hibridación Southern del gDNA. m M gDNA Dig PCR-I Lig 4,2 kb XbaI M PCR-I 4 kb XhoI 2 kb XhoI Figura 10. Amplificación inversa (PCR-I) de productos religados con T4 DNA ligasa, tras digerir el gDNA con XbaI o XhoI. Amplicones XbaI de 4,2 kb (arriba a la derecha) y XhoI de 4 kb y 2 kb (abajo a la derecha). gDNA: DNA genómico. Dig: perfil de digestión del gDNA con endonucleasas de restricción. Lig: religación del digerido. M: Marcador de peso molecular λ EcoRI/ HindIII. m: marcador de peso molecular de 1kb. 58 3. Resultados La secuencia completa del gen de la lacasa de P. flavido-alba (pfaL) se obtuvo mediante secuenciaciones parciales sucesivas de zonas solapadas del amplicón XbaI y del amplicón XhoI de 2 kb (esquematizado en la figura 11). En la secuencia reconstruida del gDNA (4,58 kb), se identificó el gen de la lacasa. . Extremo-5` - 219 pb Interno – 1390 pb H G E F Extremo-3` - 522 pb D C L3 pp12 -141 pb F21 - 248 pb L4 A B XbaI - 4,2 kb p1 p1 p2 p4 p2 p2 p3 XhoI - 2 Kb p5 p6 p1 p2 p2 Figura 11. Estrategia para aislar y secuenciar el gen pfaL de Phanerochaete flavidoalba. Barras en azul corresponden al gDNA de los fragmentos de restricción XbaI y XhoI amplificados por PCR-I con los cebadores AFPINV1 (p1) y AFPINV2 (p2), deducidos del fragmento F21. Los amplicones XbaI y XhoI se secuenciaron con los oligonucleótidos p1p6 (AFPINV1, AFPINV2, AFP3, AFP4, AFP5 y AFP6 de la Tabla 3). Las barras en blanco corresponden al cDNA amplificado en tres etapas mediante RT-PCR con los cebadores (Tabla 3) Lacfin1 (C), dTCA (D), LacN3 (E), Lac3up (F), In1 (G) y Nup1 (H). pp12: secuencia cDNA. La caracterización del alelo del gen pfaL se describe a continuación en varias etapas: aislamiento del cDNA total, identificación de los intrones, análisis de la proteína deducida y predicción de los sitios de regulación. 59 3.Resultados 3.2. AISLAMIENTO DEL cDNA pfaL El cDNA de la lacasa fue obtenido mediante tres amplificaciones consecutivas (RT-PCR), como se detalla en las figuras 11-15, en las que se utiliza la misma nomenclatura que en la figura 11. El fragmento CD (522pb correspondiente al extremo-3`se amplificó con los cebadores dTCA-Lacfin1; el fragmento EF (1390pb) del cDNA interno con los cebadores LacN3-Lac3up; y el fragmento GH (219pb) del extremo-5` con los cebadores In1-Nup1. Los amplicones obtenidos se clonaron en el vector pGEM-T y se secuenciaron. M 1 2 3 4 5 6 7 8 9 10 11 12 13 C Carril Directo Reverso 1 2 3 4 5 Lacfin1 Lacfin1 Lacfin1 Lacfin1 Lacfin1 dTCC dTCG dTA dTCA dTG Tamaño amplicón 0 0 0 522pb 0 6 7 8 9 10 11 “ “ “ “ “ “ dTCC dTCG dTA dTCA dTG dTCC 0 0 0 0 0 0 12 13 “ “ dTCG dTA 0 0 Figura 12. cDNA de pfaL Amplicón del extremo 3´: el carril 4 muestra el amplicón (CD en Fig. 11) de 522 pb obtenido con los cebadores Lacfin1/ dTCA. Carriles 1-5 mezcla de PCR con 2 ul de cDNA; carriles 6-10 mezcla de PCR con 5 ul de cDNA; carriles 11 y 12 mezcla de PCR sin cDNA; M: marcador de 100 pb ; C: control positivo con el cDNA (500pb) de la subunidad γ del receptor ácido nicotinico-acetilcolina de músculo de ratón (Stratagene). 60 3. Resultados A B M 1 1 2 2 3 4 3 5 4 6 7 5 8 9 10 11 m M 6 7 Carril Directo Reverso Tamaño del amplicón 1390pb (1 µl cDNA) 1 Lac-N3 Lac3up 2-3 “ “ 0 4 Lac-N3 dTCA 0 (1 µl cDNA) 5 “ “ 300pb 6 Hwhdwn Lac3up 0 (1 µl cDNA) 7-8 “ “ 1160 9-10 Hwhdwn dTCA 0 (1 µl cDNA) 11 Hwhdwn Lac3up 1160 (=8; 2 µl Polim) m Carril Directo Reverso Tamaño del amplicón 1 Pico17 Lac3up 429pb (1 µl cDNA) 2 “ “ 0 (2 µ l) 3 “ “ 0 (3 µ l) 4 “ “ 0 (4 µ l) 5 “ “ 0 (5 µ l) 6 “ “ 0 (6 µ l) 7 “ “ 0 (7 µ l) Figura 13. cDNA de pfaL Amplicones internos. A. Carril 1: amplicón de 1390 pb delimitado por el extremo N-terminal y la secuencia consenso L3 *; Carriles 7, 8 y 11: B. Carril 1: amplicón de 1160 pb delimitado por las secuencias consenso L3* y L4*. amplicón de 429 p delimitado por el oligopéptido interno P17 (Tabla 5). Marcadores : M (1kb); m (100pb). *Nomenclatura de Kumar et al. (2003). 61 3.Resultados 1 2 3 4 5 M 6 7 8 9 M 10 11 12 C carril directo reverso Amplicón de interés RT con Nup1 1 In1 Nup1 220 pb + 1 µl 2 “ “ 220 pb + 2 µl 3 “ “ 220 pb + 5 µl 4 “ “ 0 - 1 µl 5 “ “ 0 - 2 µl 6 “ “ 0 - 5 µl reverso Amplicón 7 In1 Nup1 220 pb + 1ul 8 “ “ 220 pb + 2 ul 9 “ “ 220 pb + 5 ul 10 “ “ 0 - 1 ul 11 “ “ 0 - 2 ul 12 “ “ 0 - 5 ul carril directo RT con Hwhup Figura 14. cDNA de pfaL Amplicón del extremo 5´. Carriles 1-6: amplicones obtenidos a partir de reverso transcripción con el cebador Nup1 (Tabla 3). Carriles 7-12 : amplicones obtenidos a partir de reverso transcripción con el cebador Hwhup (Tabla 3). El amplicón de 220pb fue purificado y clonado en pGEM-T. Control negativo: Carriles 4-6 y 10-12 amplificaciones a partir de muestras de reverso transcripción negativa (control sin reversotranscriptasa). C: control positivo: fragmento de cDNA 500pb de la subunidad γ del receptor ácido nicotínico-acetilcolina de músculo de ratón (Stratagene). 62 3. Resultados In1 GCCAGCGACCGCTTTGGTGTCCTACACTCCTTCCTTCCTCAAGGACATGGTTCTCCACTCCTTCACTCGCATGCG TATTGCGCTCGCTGCACTCCCCGCCTTGCTCACCGTCATGGTTGGGCTGCATGGTGGCGAAGTGCACGC LacN3 o Nup1 TGGACCGCACGGCCAGTATGCAGGTGTCTCTGCTAGGTCCGAGCTCCACGCTCGCGACACTGTCTCGCTTCCAT CCAGCTCCGACATTATCCTGAACGGCCTCCAAGGCCAGGCTCCTCAGACAAGGAACTACGACTTCGTCGTCTCC GAAATGACCGGTGCTCCCGACGGGTTCTCCAAGTCCATGCTCGTCGTGAACGGTCAGTTCCCTGGACCAACCAT CGAGGCCAACCAGGGCGACCGCCTTGTCATCAAGGTCACCAAC Hwhdwn o Hwhup CAGCTCACAACTAACCGCACCACTATCCATTGGCACGGTTTGTATCAGAACGGCACCGTCTGGTACGACGGTAC CGCTTCGGTCACGGAGTGCGGTATTCCGCCgGGCGAGTCGCTTACCTATGATTTCGAGCCGGGGAGCTTCTCAG GAACGACCTGGTGGCATTCTCATTACGATACGCAATACACGGACGGCGTtACTGGTGCCCTAATCATCCACCCGT CTGCTGACCCTCCGGCCGATTTCCCGACTTACGACGAAGATtTTGTCGTCCAGCTGACCGACATTTATCACACCC TCAGCCCGGCAATTGTGCAAAACTACTTGTCTGGGACCGGCCCAACTGCACCCATTATCCTCGAGACTCCGGAT AGTGGCTCAATCAACGGTGTGGGGCAATACGGTGGGGAAGGCGATTACTTCAACTTCACTCTGGAGGCAAACA AGACCTACCGTCTCCGTCTCATCCACACCGGCTCCGCCGCTCAGATCAGGTTCTCTGTTGATTATCACGCGTTGA CCGTTATCGAAGCCGATGGTACCCTCGTCGAGCCGTACTCCGTCACCGGTGTCAACCTGAACGTCGCGCAGAGG TACTCGGTGCTTCTCACCACGAACCAGACGGAAGGAAATGGAACGTACTGGATGAGGGCTGGGCTTACCTCCG TCGTTGCTGTGGAAGGTAcTACCAGCGATAtccGTGGCGTCATCAGATACGGTACCAATGATACCAG Pico17dwn CCTCCCCACTGCCGACACAGACCCTGGTGTTCCTGACTCTGGCCTCGCGGACATGGATGTGTCATTGCTCACCC CAGCCATTGTTGACACTCCTCCCGACCGCTCCAGGTTTTATCAGGTTGGTTTCAACATCTCGACTACCTCTACCG GAGGAACCATTGCCTACGTGAATGGCACTAGCTGGGCGCCGTTGGTGGGCACAAACAGCTTGCTGCAAATCAA CCAAAACAGCAGCTATGCTGGAGAGGGTGAGAGCGTGGACTCCGGAGATCAGTTCATCATCACAGAGGACACA ATCGAGACTGTTGATATTCTCTTGATCAACCAGGGTG Lacfin1 CCGGAGACCACCCATTCCACCTGCACGGCCATAGCCCCTGGATCCTCGGCTTCGACTCGACAACAT Lac3up ACCAAGgCCAGACTCCTAACCCGACCAACCCGTTGAGGCGTGACACCTTCGTCGTTCCGAGCGGTGGATGGCTT ATGCTGCGCTACATCTCCGACAACCCCGGTCcCTGGACtATGCACTGCCACATTGCCTGGCACATGGCCGCTGGG CTCCTTATGCAGGTCAACAGCTTGCCCTCGGTGGTTGCGGGCTGGACCATTCCTCAGGATGTCGTGGATCAATG CTCGGCCTGAGCGTGTTTCAGCGAACAATTTTATCTGCTCCCAGATTTTTCTTCTCCCTCTTGTTACAACTTCTAT CAGCCAATATTTTTTACTAtTATTATCTCCCTCGCCTCTTGTCGAAAGTAAAAAGCAAGTACATATGTTTTGGGG ATTAGGATTGTTCGTTTGTTCACTTGGGCTTCAGTACTTAATAAATCTTCGCTAGCCAGTATTTACGCGAAATCT TCGATCAAATTATACGATATTTGTTGTGAAAAAAAAAAAACCCTATAGTGAGTCGTATTA dTCA(rc) Figura 15. Secuencia del cDNA (2108pb) del gen pfaL deducida de los amplicones (de 522pb, 1390pb y 219 pb (Figuras 12-14) obtenidos luego de tres amplificaciones consecutivas (RT-PCR) con las parejas de primers dTCA/Lacfin1, LacN3/Lac3up, In1/Nup1 respectivamente (Tabla 3). En rojo posición de los intrones (19). 63 3.Resultados 3.3. ANÁLISIS DEL cDNA Y DE LA PROTEÍNA DEDUCIDA ATGCGTATTGCGCTCGCTGCACTCCCCGCCTTGCTCACCGTCATGGTTGGGCTGCATGGTGGCGAAGTGCACGCTGGACCGCACGGC M R I A L A A L P A L L T V M V G L H G G E V H A G P H G CAGTATGCAGGTGTCTCTGCTAGGTCCGAGCTCCACGCTCGCGACACTGTCTCGCTTCCATCCAGCTCCGACATTATCCTGAACGGCCTCCAAGGCCAGGCTCCTCAGACAAGGAACTACGACTTCGTC Q Y A G V S A R S E L H A R D T V S L P S S S D I I L N G L Q G Q A P Q T R N Y D F V GTCTCCGAAATGACCGGTGCTCCCGACGGGTTCTCCAAGTCCATGCTCGTCGTGAACGGTGTGTCCATCGACGCAACGATACAACTTGTATTGATGTTCACACAGGTCAGTTCCCTGGACCAACCATCG V S E M T G A P D G F S K S M L V V N G I1 Q F P G P T I E AGGCCAACCAGGGCGACCGCCTTGTCATCAAGGTCACCAACCAGCTCACAACTAACCGCACGTAAGTGCTATGCCCGTCGCAAGACCGTATAAGCAGCACCTAACTTAACCCGCATGGCTGACAGCACT A N Q G D R L V I K V T N Q L T T N R T I2 T ATCCATTGGCACGGTTTGGTGGGTGTTTCGATGACTTGATGCGTCCGGACATTCTAACGCCTCAACGTCAACAGTATCAGAACGGCACCGTCTGGTACGACGGTACCGCTTCGGTCACGGAGTGCGGTA I H W H G L I3 Y Q N G T V W Y D G T A S V T E C G I TTCCGCCGGGCGAGTCGCTTACCTATGAGTAAGTAGAGGTCGAGATGTCGAATGCATCACGATTCTTATTCGATATTAGTTTCGAGCCGGGGAGCTTCTCAGGAACGACCTGGTGGCAGTGAGTAAAAT P P G E S L T Y D I4 F E P G S F S G T T W W H TGAGCCGCTTGTAGAACTTACACCACTAACATACCCCTCCTAGTTCTCGTAAGTATACAACGGTTCCATGATAGCTTTGGCACTGATCATCTGATGATCGTGTTGAACAGATTGTGAGGCATTCATTTT I5 S H I6 Y TTTCTCAATATTAGCTCTCCCCTCATCTTCGACTTTGACAGACGATACGCAATACACGGACGGCGTTACTGGTGCCCTAATCATCCACCCGTCTGCTGACCCTCCGGCCGATTTCCCGACTTACGACGA I7 D T Q Y T D G V T G A L I I H P S A D P P A D F P T Y D E AGATTTTGTCGTCCAGCTGACCGACATTTATCACACCCTCAGCCCGGCAATTGTGCAAAACTACTTGTCTGTGAGTAGCGAACCGTTCGTCTGTTACCTTCGTTTTCTGAAAGCATCATTAGGGGACCG D F V V Q L T D I Y H T L S P A I V Q N Y L S I8 G T G GCCCAACTGCACCCATTATCCTCGAGACTCCGGATAGTGGCGTGCGTACCTCTGTTTGTTAGCCGTGCATAGGCTCACTAATTCATCAATGTATTCTAGTCAATCAACGGTGTGGGGCAATACGGTGGG P T A P I I L E T P D S G I9 S I N G V G Q Y G G GAAGGCGATTACTTCAACGTGAGCCTTGCGTCATTGAGCCGTCTCATCTTGTCTCACGCTTGTCTCTTTACAGTTCACTCTGGAGGCAAACAAGACGTGAGTACGATCCTTATGAATAAGCGCGCCCGG E G D Y F N I10 F T L E A N K T I11 TCTGAATCTCGCCCGCCAGCTACCGTCTCCGTCTCATCCACACCGGCTCCGCCGCTCAGATCAGGTTCTCTGTTGATTATCACGCGTTGACCGTTATCGAAGCCGATGGTACCCTCGTCGAGCCGTACT Y R L R L I H T G S A A Q I R F S V D Y H A L T V I E A D G T L V E P Y S CCGTCACCGGTGTCAACCTGAACGTCGCGCAGAGGTACTCGGTGCTTCTCACCACGAACCAGACGGAAGGAAATGGAACGTACTGGATGAGGGCTGGGCTTACCTCCGTCGTTGCTGTGGAAGGTACTA V T G V N L N V A Q R Y S V L L T T N Q T E G N G T Y W M R A G L T S V V A V E G T T CCAGCGATATCCGTGGCGTCATCAGGTTTGCTCTCAAGTATCATGCGGTGAATGAGGGGCTTACGTCTTTGTACCCACAGATACGGTACCAATGATACCAGCCTCCCCACTGCCGACACAGACCCTGGT S D I R G V I R I12 Y G T N D T S L P T A D T D P G GTTCCTGACTCTGGCCTCGCGGACATGGATGTGTCATTGCTCACCCCAGCCATTGTTGACACTCCTCCCGACCGCTCCAGGTGAGTCAACGCTTTAGACTAACTCGAGTCGCGTGGCTAATGGAATACA V P D S G L A D M D V S L L T P A I V D T P P D R S R I13 GGTTTTATCAGGTTGGTTTCAACATCTCGACTACCTCTACCGGAGGAACCATTGCCTACGTGAATGGCACTGTGAGTAAAGACTGCACATCCTCGATGTATTCTGGTGCTAACAACAACCCATTTTATC F Y Q V G F N I S T T S T G G T I A Y V N G T I14 AGAGCTGGGCGCCGTTGGTGGGCACAAACAGCTTGCTGCAAATCAACCAAAACAGCAGCTATGCTGGAGAGGGTGAGAGCGTGGACTCCGGAGATCAGTTCATCATCACAGAGGACACAATCGAGACTG S W A P L V G T N S L L Q I N Q N S S Y A G E G E S V D S G D Q F I I T E D T I E T V TTGATATTCTCTTGGTGCGTACATCATATATTAATTGTCCTCTGATATCTGAGCCTAATACTGGCTCTCAGATCAACCAGGGTGCCGGAGACCACCCATTCCACCTGCACGGCCATAGCCCCTGGATGT D I L L I15 I N Q G A G D H P F H L H G H S P W I GAGTGGTCCTGGTTTCTATTATAGCGTGTGGGAGTGCTTACCCCATGTCTTAGCCTCGGCTTCGACTCGACAACATACCAAGGCCAGACTCCTAACCCGACCAACCCGTTGAGGTATGTGTGCCAGTCG I16 L G F D S T T Y Q G Q T P N P T N P L R CGAGCTGTTACCAATTTCATTCTCATACATTGATGCAGGCGTGACACCTTCGTCGTTCCGAGCGGTGGATGGCTTATGCTGCGCTACATCTCCGACAACCGTGAGTCTACCCATACCATAGTTTGATTC I17 R D T F V V P S G G W L M L R Y I S D N P I18 ACACTCATGATCGTTCGCCCACAGCCGGTCCCTGGACTATGCACTGCCGTAAGCACCTTACTTACTATTGTATCTCAAAGGCGAAATCTCTGACTTGTGCTTTCTAGACATTGCCTGGCACATGGCCGC G P W T M H C H I19 I A W H M A A TGGGCTCCTTATGCAGGTCAACAGCTTGCCCTCGGTGGTTGCGGGCTGGACCATTCCTCAGGATGTCGTGGATCAATGCTCGGCCTGAGCGTGTTTCAGCGAACAATTTTATCTGCTCCCAGATTTTTC G L L M Q V N S L P S V V A G W T I P Q D V V D Q C S A TTCTCCCTCTTGTTACAACTTCTATCAGCCAATATTTTTTACTATTATTATCTCCCTCGCCTCTTGTCGAAAGTAAAAAGCAAGTACATATGTTTTGGGGATTAGGATTGTTCGTTTGTTCACTTGGGC TTCAGTACTTAATAAATCTTCGCTAGCCAGTATTTACGCGAAATCTTCGATCAAATTATACGATATTTGTTGTG Figura 16. Secuencias del gDNA y cDNA deducidos para el gen de la lacasa de P. flavido-alba (pfaL) y de la proteína codificada. Intrones (I1-I19) secuencias marcadas en gris. En azul se destacan los oligopéptidos identificados en la lacasa pura de P. flavido-alba (Tabla 5), en verde los amino ácidos envueltos en la unión al Fe (E, Y, D) en las ferroxidasas fúngicas (Bonaccorsi di Patti et al., 2000) y en amarillo la posible secuencia señal de secreción (25 residuos ) identificada con el programa SignalP3.0. AATAAA señal de poli adenilación. Secuencia de 567 amino ácidos codificados por la ORF (desde ATG a TGA) según GeneScan 1.0 (http://genes.mit.edu/GENSCAN.html). 64 3. Resultados La secuencia completa del cDNA fue deducida por solapamiento de las tres secuencias obtenidas y alineamiento con el gDNA esquematizado en la Fig 11. Se identificó un marco abierto de lectura (ORF, 1704 pb desde el codón de iniciación hasta el de terminación (ATG/TAA) de la traducción, en la secuencia de cDNA resultante (2018 pb) El tamaño deducido corresponde a un polipéptido de 567 aminoácidos (Fig. 16). Mediante un análisis BLASTp (Tabla 4) en la base de datos del EMBL, se encontró que la secuencia deducida para la lacasa PfaL es muy semejante (identidad del 59%) a las proteínas putativas MCO4A y MCO4B de Phanerochaete chrysosporium, y a lacasas de hongos basidiomicetos (identidades cercanas al 35%). Estos resultados indican que efectivamente el cDNA aislado de Phanerochaete flavido-alba correspondería al de una proteína de la familia multicobre-oxidasas Las multicobre oxidasas contienen los cuatro secuencias conservadas de unión al cobre (L1-L4) descritas por Kumar et al. (2003) para las lacasas. Las subsecuencias de aminoácidos L1-L4 en la proteína PfaL alineadas con proteínas recogidas en el BLAST (Tabla 4) mostraron la mayor identidad con las proteínas MCOs de Phanerochaete chrysosporium, y en menor grado con las lacasas y las ferroxidasas (Tablas 4 y 10). Los péptidos identificados de la lacasa de P. flavido-alba purificada (De La Rubia et al 2002; Lucas et al 2005) coinciden con la proteína PfaL de 567 aminoácidos (Fig. 16) deducida del cDNA. Tales oligopéptidos se muestran en la Tabla 5. 65 3.Resultados Tabla 4. Alineamiento (WU-Blastp, EMBL) de la secuencia deducida para la lacasa PfaL con las proteínas de la base de datos UNIPROT. Nº Identificación Organismo Proteína 1 2 3 4 5 6 7 8 9 Q6QNN1 Q6QNN2 Q6QNP2 Q875H3 Q6QNN9 Q6QNM9 Q6RYA3 Q6QNN4 Q6CGD4 Phanerochete chrysosporium Phanerochete chrysosporium Phanerochete chrysosporium Phanerochete chrysosporium Phanerochete chrysosporium Phanerochete chrysosporium Auricularia auricula-judae Phanerochete chrysosporium Yarrowia lipolitica 10 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 Q4P261 Q4HWQ4 Q5ARB0 Q6VMB9 Q3KRP1 LAC2 Q7S4C0 LAC2 Q8TFE4 LAC4 Q6VMB7 Q96WM9I Q00292 LAC2 Q6E124 Q8TFE1 28 30 31 Q6E0Y2 Q6VMB8 Q8TFD9 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 49 50 Q5A503 Q8X1W3 Q8TFE3 Q9ZR59 Q8TG93 COPA1 Q68LM4 Q8WZI0 Q9P861 O13456 LAC3 Q4HZG2 Q5B233 Q7SDL8 Q8J0L7 FIO1 Q5I7J0 Q50JG3 Ustilago maidis Giberella zeae Aspergillus nidullans Coprinopsis cinerea Criphonectria parasitica Agaricus bisporus Neurospora crassa Thanathephorus cucumeris Gaeumannomyces graminis-tritici Thanathephorus cucumeris Coprinopsis cinerea Botrytis cinerea Aspergillus terreus Podospora anserina Auricularia polytricha Gaeumanomyces graminisgraminis Auricularia polytricha Coprinopsis cinerea Gaeumanomyces graminisgraminis Candida albicans Lentinula edodes G. graminis var. tritici Cucumis melo Trametes pubescens Trametes villosa Lentinula edodes Lentinula edodes Arxulla adeninivorans Trametes versicolor Thanathephorus cucumeris Gibberella zeae Aspergillus nidulans Neurospora crassa Claviceps purpurea Schizosaccharomyces pombe Trametes sp AH28-2 Trametes versicolor 66 %Identidad Multicobre oxidasa 4B. (MCO4B) MCO4A MCO2A. MCO Extracelular. MCO3B. Variante splice MCO4B-I13 MCO Variante splice MCO3B-I10 Similar a ferroxidasa de membrane YMR058w FET3 de Saccharomyces cerevisiae sp|P38993 Proteina hipotetica Proteina hipotetica Proteina hipotetica Lacasa 5 (EC 1.10.3.2). Lacasa 3. precursor Lacasa II (EC 1.10.3.2) Proteina hipotetica precursor Lacasa 2 (EC 1.10.3.2) precursor Lacasa (EC 1.10.3.2). precursor Lacasa 4 (EC 1.10.3.2) Lacasa 7 (EC 1.10.3.2). Lacasa 2 (EC 1.10.3.2). Dihydrogeodin oxidasa precursor Lacasa II (Laccase C). Lacasa. lac1 precursor Lacasa (EC 1.10.3.2). Aminoác idos 591 591 617 559 613 444 595 462 695 59 59 55 57 54 59 44 54 39 695 566 570 533 567 520 588 599 608 531 549 581 605 621 619 607 34 34 38 34 33 35 35 41 34 36 33 34 37 34 35 33 Lacasa. Lacasa 6 (EC 1.10.3.2). precursor Lacasa (EC 1.10.3.2). 620 532 578 36 33 34 Potencial MCO ferro-O2-oxidoreductasa. Lacasa. precursor Lacasa (EC 1.10.3.2). L-ascorbate oxidase (EC 1.10.3.3). Lacasa 1A (EC 1.10.3.2). precursor Lacasa 5 (EC 1.10.3.2) Lacasa 1 BVT (EC 1.10.3.2). Lacasa 2. precursor Ferro-O2-oxidoreductasa. precursor Lacasa (EC 1.10.3.2). precursor Lacasa 3 Proteina hipotetica Proteina hipotetica Proteina hipotetica Putativa MCO precursor fio1-MCO de transporte de Fe Lacasa B (EC 1.10.3.2). Lacasa4. 620 533 578 687 526 527 533 533 615 526 572 572 664 549 623 622 525 527 32 34 34 34 34 34 34 34 32 34 40 33 33 32 33 31 34 32 3. Resultados Tabla 5. Secuencias contenidas en la proteína PfaL deducida que coinciden con las de los péptidos de la lacasa de P. flavido-alba purificada y similitud entre ellas con la secuencia aminoacídica deducida del cDNA pfaL Secuencia de aminoácidos Oligopéptido PfaLop-Nt PfaLop-m82 (N-terminal a C-terminal) Método DTVSLPSSSDIILNGLQGQAP Degradación QTRNYDFV Edman EGTTSDIR Espectrometría % Identidad de 100 de 100 de 100 de 100 de 91 de 100 masas Maldi- tof PfaLop-P17 PfaLop- YGTNDTSLPTADTDPGVPDSG Degradación LADM Edman NPLR Espectrometría m24b PfaLop-P18 masas Maldi- tof * FVVPSGGWI ML Degradación Edman PfaLop-23I1 VDQ Espectrometría masas Maldi- tof * EL aminoácido identificado en el oligopéptido (I) no corresponde al deducido en la proteína (L) 3.4. IDENTIFICACIÓN DE INTRONES DEL GEN pfaL Tras el alineamiento manual del gDNA con el cDNA del gen pfaL se identificaron un mayor número de intrones (19 intrones) que en los genes de las lacasas (Tabla 8). Su longitud promedio es de unos 58±4 nucleótidos. Al compararlos mediante ClustalW, presentaron secuencias consenso al inicio y al final de los sitios de “splice”, 5`-GTA/G...y ...T/CAG-3` (Tabla 6). Estas secuencias conservadas para los sitios donador y receptor se corresponden con el patrón general de los hongos, así como con los identificados en los intrones de varios genes de lacasas fúngicas, como por ejemplo en Coprinopsis cinerea (Seitz et al. ,1996 y Hoegger et al., 67 3.Resultados 2004). Las secuencias de los intrones de pfaL presentan alta variabilidad, aunque identidades entre 19% y 38% (mostrada en la Tabla 7) podría indicar la presencia de elementos conservados, importantes para la maquinaria de “splicing”. Tabla 6. Alineamiento de los intrones del gen pfaL. Se usó el programa MegAlign del DNAstar + Majority Majority G T G A G T T A T G C G T C G T T C T T A A T G T T G T A T T T G C G T C C T A A C G C C T A A T C - - C - - - T T T - - C A G T T T T T T T T T T T T T T T T T T T G G A G A G A G G G G G T G G A G A G C T A G A A A A A C A A T A A A A T A G G G G G G G G G G G G G G G G G G G T T T T T G T C C T T T T T C G G A A T C A T C T T C A C T G C A C C T G A A A G A T A C A A A G T C A T T A A T T C C T C T A A C G - A T A T G A T G C G G C C G C T A T C T C A C C A T C A A G A T C T C G G G T A A A C G A C C C G - C A C A T A T C T T T G T T T T C C A C C T G C T C G C C T T G A G C C T G C G A G T G T A C A T C C G A A A G G G G T T T T A A T T G - T C T A T C T T T T T T T G C T T T T A A C C C C T T C G T A C A A A T C A T A G T G G C T G T G T A C T C C G C T C C T A C C C T T A G T T C T T C C A G A G G T A T A G A G A C A A G A A A A A A T T C G C A C A T T T A T T T G T T G G A C C T G C C T T A T A A G G T A A C G A G T G G A G G G C C C T A T T C G T A T A T T C T T C C G A A T T G C T A A T - C A G G G A G G G C C G G G T G G T C A T T A A C C T A A T C C T T C A C A C T A C A G A T G G C T T C T T T T T T T T C T T - T A G T T A A T C T A G T A G A G A T T A G T G G C A T A T A G A G C G A C G C G A C T A G C T T T C C C A G G C C T C G G G G G C C A G A A A C C C T T T G G T G G G A A T T G T T C A T T C G C G G T C A A C C A C T C A C C C A T C G G G G G T A T T A C A C T G C C C C C C A T T C G T C T C T G T T T G T T T A G T T A G C A G A A T A C G T A A A C C C 60 50 40 30 20 10 I15-pfaL.SEQG I1-pfaL.SEQ G I2-pfaL.SEQ G I3-pafL.SEQ G I4-pafL.SEQ G I5-pfaL.SEQ G I6-pafL.SEQ G I7-pafL.SEQ G I8-pfaL.SEQ G I9-pfaL.SEQ G I10-pfaL.SEQG I11-pfaL.SEQG I12-pfaL.SEQG I13-pfaL.SEQG I14-pfaL.SEQG I19-pfaL.SEQG I16-pfaL.SEQG I17-pfaL.SEQG I18-pfaL.SEQG G A T A A A T T A A T A A A T T A A C T A C T C C C T T C C C T C C T T T G A G T A A C T G G G A A T G C T C C C T T C C T C T T G A T C C A T T C T C C T C T C A C C C T T T C T T T T T G T G T A A T C C C A A C A A G C A G A T A G C A G A A A T C A A A A T A T C A A T T T A C A T C T T T C T G C C T T A C C C C C G C C T C T C T C C C G C A T A A T A T - G A T T T G A T G A - T C T C C C C T C T T C - G A A G G A G G G G A - G T T T A T T C C T T - C G G C C A C T C T - T G G T T A T C C A T T G G C C T C T T T T T G C T T T A C T T C T G T T C T C C T T C T C Tabla 7. Porcentajes de identidad de los intrones del gen pfaL. I15 *** 68 I1 27.7 *** I2 28.1 29.8 *** I3 23.2 34.0 26.8 *** I4 29.4 31.9 33.3 25.5 *** I5 20.4 23.4 31.5 20.4 19.6 *** I6 24.6 34.0 24.2 25.0 29.4 20.4 *** I7 35.1 19.1 28.1 30.4 27.5 25.9 33.3 *** I8 26.9 25.5 25.0 23.1 31.4 23.1 30.8 28.8 *** I9 28.1 27.7 24.1 26.8 31.4 25.9 27.6 29.8 21.2 *** I10 25.5 27.7 25.5 29.1 25.5 24.1 25.5 30.9 28.8 25.5 *** I11 38.5 19.1 30.8 28.8 27.5 26.9 30.8 23.1 21.2 28.8 26.9 *** I12 25.5 29.8 27.3 27.3 25.5 24.1 25.5 23.6 21.2 25.5 34.5 23.1 *** I13 28.0 27.7 30.0 26.0 32.0 30.0 24.0 26.0 24.0 26.0 24.0 26.0 22.0 *** I14 31.6 34.0 21.7 25.0 25.5 29.6 30.0 21.1 30.8 31.0 23.6 23.1 18.2 32.0 *** I19 35.1 27.7 22.0 23.2 21.6 22.2 25.4 28.1 26.9 32.8 27.3 36.5 27.3 30.0 27.1 *** I16 29.1 29.8 27.3 30.9 21.6 29.6 32.7 29.1 23.1 30.9 29.1 28.8 25.5 30.0 27.3 20.0 *** I17 27.8 34.0 33.3 24.1 29.4 24.1 29.6 25.9 28.8 29.6 25.9 19.2 24.1 32.0 25.9 22.2 24.1 *** I18 24.5 34.0 28.3 26.4 31.4 26.4 34.0 24.5 25.0 26.4 35.8 28.8 28.3 28.0 24.5 30.2 24.5 24.5 *** I15 I1 I2 I3 I4 I5 I6 I7 I8 I9 I10 I11 I12 I13 I14 I19 I16 I17 I18 G A A A G T C A A G C A A A A A A A T A C C C C T C C T C C C C C T C C A A A A A A A A A A A A A A A A A A G G G G G G G G G G G G G G G G G G G 3. Resultados Tabla 8. Estructura génica de lacasas de hongos de la podredumbre blanca de la madera y las proteínas que codifican Especie de Basidiomiceto gen gDNA cDNA Nº de intrones Aminoácidos del Peptido señal Número de aminoácidos Referencia Trametes sp. I-62 lcc1 ND 1563 ND ND 520 Gonzalez T, et al 2003. Mycol Res. 2003 ;107(Pt 6):727-35.) Trametes sp. I-62 lcc2 ND 1563 ND ND 520 Gonzalez T, et al 2003. Mycol Res. 2003 ;107(Pt 6):727-35.) Trametes sp. I-62 lcc3 ND 1575 ND ND 524 Gonzalez T, et al 2003. Mycol Res. 2003 ;107(Pt 6):727-35.) Trametes versicolor X84683 lcc1 2800 1560 10 22 498 Guo M, et al 2005. Appl Microbiol Biotechnol. 2005 Jonsson L, et al. Biochim Biophys Acta. 1995 Sep 6;1251(2):210-5 Pycnoporus cinnabarinus I-937 lac1 3331 1557 10 21 518 Otterbein L, et al Eur J Biochem. 2000 Mar;267(6):1619 Trametes trogii precursor protein Lcc1 2431 1554 10 21 517 Colao MCh, et al 2003 Appl Microbiol Biotechnol. 2003 Dec;63(2):153-8 Trametes pubescens lap2 3578 1563 8 ND 520 Galhaup C, et al 2002 Microbiology. 2002 Jul;148(Pt 7):2159-69. Trametes pubescens Lap1 A 2554 1581 11 ND 526 Galhaup C, et al 2002 Microbiology. 2002 Jul;148(Pt 7):2159-69. C. cinereus lcc1 2243 1620 7 ND 539 Yaver DS, et al 1999.Appl Environ Microbiol. 1999 Nov;65(11):4943-8. C. cinereus lcc2 2993 1555 13 ND 517 Yaver DS, et al 1999.Appl Environ Microbiol. 1999 Nov;65(11):4943-8. C. cinereus lcc3 2801 1551 13 ND 516 Yaver DS, et al 1999.Appl Environ Microbiol. 1999 Nov;65(11):4943-8. Volvariella volvacea lac4 1689 ND ND ND 562 Chen S, et al 2004.. FEMS Microbiol Lett. 2004 Jan 30;230(2):171-6 Marasmius quercophilus lac1 ND ND ND ND 517 Dedeyan B, et aal . 2000. Appl Environ Microbiol. 66(3):925-9 Fome lignosus Lcc 2201 1557 11 21 518 Liu W, et al 2003 Appl Microbiol Biotechnol. 2003 Dec;63(2):174-81. Epub 2003 Jul 24. ND : No determinado 69 3.Resultados 3.5. ANÁLISIS FILOGENÉTICO DE LA LACASA PFAL La conservación del sitio activo de las diversas oxidasas que contienen cobre sugiere que la actividad enzimática, relacionada con cuatro sitios del cobre en la misma molécula, puede ser un evento biológico temprano (Solomón et al 1996). Por lo tanto, las lacasas son consideradas enzimas antiguas desde el punto de vista evolutivo (Messerchmidt et al 1990). Con objeto de definir la relación de la secuencia deducida para la lacasa de P. flavido-alba (PfaL), se realizaron sucesivos alineamientos de la secuencia de PfaL con las de las proteínas multicobre (incluyendo a lacasas y ferroxidasas) de las bases de datos. Se construyó un arbol filogenético utilizando el programa MEGA3 sobre las secuencias fúngicas alineadas. Se demuestra que PfaL y las MCOs de P. chrysosporium forman un linaje separado entre las multicobre oxidasas, separado de ferroxidasas y lacasas fúngicas. A esta conclusión se ha llegado tras comparar la secuencia de PfaL con: a) lacasas fúngicas (de basidiomicetos y de ascomicetos); b) ferroxidasas fúngicas; y c) con las lacasas y ferroxidasas más próximas filogenéticamente detectadas en a) y b). 3.5.1. Alineamiento de PfaL con lacasas fúngicas El alineamiento (Clustal) de PfaL con las lacasas obtenidas del NCBI (búsqueda por lacasas) mostró un filograma (construido con MEGA 3), donde PFAL queda incluida en un “clade” independiente (no mostrado). Después de eliminar las secuencias con identidad mayor del 95%, el análisis filogenético de PfaL con las lacasas fúngicas mostró cinco grupos principales de lacasas. Sorprendentemente, PfaL fue alineada con lacasas de ascomicetos y no con las de basidiomicetos. 3.5.2. Alineamiento de PfaL con ferroxidasas fúngicas Siguiendo el mismo proceso se construyó un nuevo árbol filogenético (MEGA 3) con las ferroxidasas, que se refinó eliminando las secuencias con identidad superior al 90%. 70 3. Resultados Aproximadamente la mitad de las ferroxidasas formaron un grupo claramente diferenciado del resto de secuencias comparadas. Este grupo, mostró dos ramas principales por el número de proteínas que las forman. Una rama incluyó las ferroxidasas animales. La otra rama incluyó a PfaL entre las ferroxidasas procarióticas y fúngicas. En este agrupamiento sólo se encontraron tres proteínas de basidiomicetos: PfaL, la MCO1 de P. chrysosporium y una proteína de Cryptococcus noeformans (NCBI gi57226018). En esta rama filogenética PfaL se alineó en un grupo diferenciado del resto de ferroxidasas fúngicas junto a la proteína MCO1 de P. chrysosporium y dos proteínas putativas (gi|50545481| y gi|49646143|) de un ascomiceto (Yaworria lipolytica), similares a la ferroxidasa de membrana FET3 (YMR058w) de Saccharomyces cerevisiae. 3.5.3. Alineamiento de PfaL con lacasas y ferroxidasas fúngicas Después de alinear PfaL con las lacasas fúngicas y las ferroxidasas se hizo un nuevo análisis filogenético (Fig. 17). PfaL, las MCOs de P. chrysosporium, una proteína putativa de Yarrowia lipolytica y otra proteína de Cryptococcus neoformans, se incluyeron en un grupo filogenético separado tanto de ferroxidasas como de lacasas. En conclusión, las multicobre oxidasas de las dos especies de Phanerochaete forman un grupo separado de las lacasas fúngicas y de las ferroxidasas. Por otro lado, en este árbol filogenético algunas multicobre oxidasas identificadas como lacasas en las bases de datos, por ejemplo la de una cepa de Stagonospora (gi23305799, Lyons et al., 2003) fueron así mismo ubicadas en el grupo de ferroxidasas. 71 3.Resultados Podosan-lac-gi|1729781| 98 Melalbo-Lac-gi|22218674| 63 87 Ncras-lac-gi|168828| Cryphopar-lac-gi|167469| Neucras-lac-gi|28881397| 93 Collelag-lac-gi|12862766| Gaeugrat-lac-gi|19171198| 58 lac-marine-gi|23305795| Mycos-lac-gi|23305809| Buersparti-lac-gi|23305813| 99 76 Botryf uc-lac-gi|15787446| 100 Phaehal-lac-gi|23305805| Phaespar-lac-gi|23305801| 85 Phae-hal-lac-gi|23305793| 100 Lacasas Phaespar-lacgi|23305789| Hortac-lacIII-gi|66969928| Fuspro-lac3-gi|14718439| 99 77 59 Hortaci-lacI-gi|63146072| Gaeu-Gram-lac2-gi|19309740| 50 98 Glomercin-lac-gi|57232530| Crypar-Lac3-gi|69047730| Hortaci-lacII-gi|67773582| 89 89 Gaeu-Gram-lac1-gi|19309738| 76 Botryf uc-Lac2-gi|15022489| 61 Botci-LAC1-gi|2833229| 100 Botry-f uc-Lac1-gi|15022487| Fuspro-lacRT1-gi|14581397| Stag-lac-gi|23305799| Ncras-f err-gi|18376329| 76 A spef um-Feo-gi|50845196| A rx-f eO2-gi|7798835| 100 Deba-han-gi|50422577| Fet3-Canal-pre-gi|2493323| 72 Fet3-Flula-prec-gi|62901518| 71 Ferroxidasas Cryneo-Feo-gi|57226018| 99 f et3-pre-Canga-gi|62901519| 100 95 Fet3-yeast-prec-gi|2828219| Cryneof -lac-gi|57228040| Y arli-gi|49646143| 69 Pf aL 56 100 Pchry-MCO1-gi|28416421| MCOs Phanerochaete 0 .2 Figura 17. Árbol filogenético construido (“Neighborn-joining”) con las secuencias de PfaL y de las ferroxidasas y lacasas fúngicas seleccionadas (Tabla 9). PfaL, las MCOs de P. chrysosporium y la proteína putativa de Y. lipolytica forman un grupo filogenético independiente de ferroxidasas y lacasas fúngicas. Se indican los valores “boostrap” >50 72 3. Resultados Tabla 9. Secuencias seleccionadas de lacasas y ferroxidasas para análisis filogenético de PFAL Código Cryneof-lac Ncras-lac Neucras-lac Podosan-lac Melalbo-Lac Cryphopar-lac Gaeugrat-lac Nº gi gi|57228040| gi|168828| gi|28881397| gi|1729781| gi|22218674 gi|167469| gi|19171198| GaeuGramlac2 Gaeu-Gramlac1 Lac-marine Collelag-lac Buersparti-lac Botryfuc-lac Mycos-lac Phaehal-lac Phae-hal-lac Phaespar-lac gi|19309740| gi|19309738| gi|23305795| gi|12862766| gi|23305813| gi|15787446| gi|23305809| gi|23305805| gi|23305793| gi|23305801| Phaespar-lac gi|23305789| Stag-lac Glomercin-lac Crypar-Lac3Hortaci-lacII Hortaci-lacI Hortac-lacIII Botci-LAC1Botryfuc-Lac2 Botryfuc-Lac1 FusprolacRT1 Fuspro-lac3 PfaL-V8 Pchry-MCO1fet3-preCangaFet3-yeastprecFet3-FlulaprecFet3-CanalpreDeba-han Ncras-ferr Aspefum-Feo Arx-feO2 Cryneo-Feo gi|23305799| gi|57232530| gi|69047730| gi|67773582| gi|63146072| gi|66969928| gi|2833229| gi|15022489| gi|15022487| gi|14581397| Yarli- gi|49646143| gi|14718439| Especie Crytococcus neoformans Neurospora crassa-TS Neurospora crassa Podospora anserina Melanocarpus albomyces Cryphonectria parasitica Gaeumannomyces graminisvar.tritici Gaeumannomyces-graminisvar graminis Gaeumannomyces graminis var. graminis Ascomicete marino SAP162 Colletotrichum lagenarium Buergenerula spartinae Botriotinis fuckeliana Mycosphaerella sp.2 SAP136 Phaeosphaeria halima SAP159 Phaeosphaeria halima SAP140 Phaeosphaeria spartinicola SAP151 Phaeosphaeria spartinicola SAP149 Stagonospora sp. SAP143 Glomerella cingulata Cryphonectria parasitica Hortaea acidophila Hortaea acidophila Hortaea acidophila Botrytis cinerea Botryotinia fuckeliana Botryotinia fuckeliana Fusarium proliferatum Base de datos gb|AAW44497.1| gb|AAA33592.1|emb|CAD70438.1| emb|CAA70061.1| pdb|1GW0|B gb|AAA33105.1| |emb|CAD10749.1 emb|CAD24842.1 emb|CAD24841.1| gb|AAN17287.1| dbj|BAB32575.1| gb|AAN17296.1| gb|AAL06114.1| gb|AAN17294.1| gb|AAN17292.1| gb|AAN17286.1 gb|AAN17290.1 gb|AAN17284.1| gb|AAN17289.1| gb|AAW47924.1| gb|AAY99671.1 gb|AAY33971.2 gb|AAY33970.1 gb|AAY60098.1| sp|Q12570 gb|AAK77953.1| gb|AAK77952.1| gb|AAG23872.1| gb|AAK72901.1| gi|28416421| gi|62901519| Fusarium proliferatum Phanerochaete flavida-alba Phanerochaete chrysosporium Candida glabrata gb|AAO42609.1| sp|Q96WT3| gi|2828219| Saccharomyces cerevisiae sp|P38993| gi|62901518| Kluyveromyces lactis sp|Q6CII3| gi|2493323| Candida albicans sp|P78591| gi|50422577| gi|18376329| gi|50845196| gi|7798835| gi|57226018| Debaryomyces hansenii Neurospora crassa Aspergillus fumigatus Arxula adeninivorans Cryptococcus neoformans var. neoformans JEC21 Yarrowia lipolytica ref|XP_459860.1| emb|CAD21075.1 gb|AAT84595.1| emb|CAB90817.1| gb|AAW42479.1| emb|CAG84216.1| 73 3.Resultados 3.5.4. Identificación de aminoácidos conservados en PfaL Para definir posibles secuencias conservadas en las multicobre oxidasas de las dos especies de Phanerochaete (P. chrysosporium y P. flavido-alba), se realizaron alineamientos de secuencias específicas: 1) con lacasas de ascomicetos, y 2) con ferroxidasas y lacasas de ascomicetos. 3.5.4.1. Aminoácidos de PfaL conservados en lacasas de ascomicetos Se compararon las posiciones de PfaL y MCOs de P. chrysosporium conservadas en las secuencias L1-L4 (Kumar et al., 2003) de lacasas fúngicas y lacasas vegetales (Tabla 10). Cuando se alinearon las secuencias consenso de unión al cobre (L1-L4 , Kumar et al. 2003) de las proteínas de mayor identidad con PfaL seleccionadas (Tabla 4), la mayor identidad de PfaL se obtuvo para las MCOs de P. chrysosporium(Tabla 10A-D). 74 3. Resultados Tabla 10. Posiciones conservadas en los sitios de union al cobre. Comparación de PFAL y MCOs de P. chrysosporium (Pc-MCOs) con lacasas vegetales y lacasas fúngicas. Tabla 10A. Secuencia L1 Enzima Inicio * Longitud Lacasas vegetales Lacasas fúngicas 96 24 64 24 Mco4 PfAL MCO1B MCO2A MCO3B Consenso H-W-H-G- (X)9- D-G-P(X)3-TQCPI H-W-H-G- (X)9- D-G-(X)5 H-W-H-GLYQNSTNYYD-G H-W-H-GLYQNGTVWYD-G H-W-H-GIPQNGTAYYD-G H-W-H-GLFQNQTNYYD-G H-W-H-GLFQNGTNYYD-G * * * ** **:* ** * TAGVT TASVT TAGIT TAGIT TAAIT ** * QCPI ECGI ECGI ECGI ECGI ECGI **** 166 146 125 178 172 Tabla 10B. Secuencia L2 Lacasas 136 vegetales 8 G-T-L-X-W---H-A-H Lacasas 104 22 G-T-(X)-W-Y-H-S-H-(X)3-Q-Y-C-X-D-G-L-X-G-X-(FLIM) fúngicas Mco4A PFAL MCO1B MCO2A MCO3B G G G G G * T T T T T * T T T T T * W-W-H W W H W W H W W H W W H * * * A S S A A H H H H H * YDT YDT YDT YST YST *:* Q Q Q Q Q * Y Y Y Y Y * T T TTT* D D D D D * G G G G G * V V V I I T T T T T * G G G G G * A A A A A * L L L L L * 203 183 163 215 209 *La posición de inicio de la subsecuencia se muestra con relación a la Secuencia de la proteina de P. taeda para lacasas vegetales y C. cinereus para lacasas fúngicas. La secuencia consenso se muestra como una expresión regular según el patrón de PROSITE (Bucher and Bairoch, 1994; Hoffmann et al., 1999). 75 3.Resultados Tabla 10. Posiciones conservadas en los sitios del cobre. Comparación de PFAL y MCOs de P. chrysosporium (Pc-MCOs) con lacasas vegetales y lacasas fúngicas Tabla 10C. Secuencia L3 Lacasas vegetales Lacasas fúngicas Mco4A PFAL MCO1B MCO2A MCO3B 478 8 396 8 H-P-X-H-L-H-G-(FYH) H-P-X-H-L-H-G-H H P F H L H G H H P F H L H G H H P F H L H G Y H P F H L H G H H P F H L H G H * * * * * * * * 503 479 468 521 516 Tabla 10D. Secuencia L4 Inicio:535 Longitud:21 Lacasas vegetales G-V-W-(FLI)-(FML)-HCH-(FMLI)-(DE)-X-H-X2-W-G-L-X-M-X-(WF Inicio:446 Longitud:23 Lacasas fúngicas G-X-W-(FIL)-(FLM)-HCH-I DAE-X-H-X3-G(LMF)X3-(LFM) Mco4A PFAL MCO1B MCO2A MCO3B 76 G G G G G * I P A L L W W W W W * T T T A A L HCH I AWM HCH I AWL HCH I SWF HCH L AWF HCH L AW*** * - H H H H H * MAAG MAAG MSAG MAAG MAAG *:** LMMQ LLMQ LLMQ LLMQ MLMQ ::** ISSLPSKAAQLDIPRDIVRQCSA- 591 VNSLPSVVAGWTIPQDVVDQCSA- 567 FVTLPSVAATLPIPQVIANMCSAA 559 INSLPSKAAQLDIPQSIVSQCAATVDGAGAR- 617 VNSLPTRAAQLDIPQTIVDQCQVDVGGAGARR 613 .:**: .* **: :. * . 3. Resultados 3.5.4.2. Aminoácidos de unión al Fe en las ferroxidasas conservados en PfaL y en las MCOs de P. chrysosporium Las lacasas y ferroxidasas (excepto MCOs de P chrysosporium) más próximas a PfaL en los alineamientos previos, fueron depuradas de nuevo eliminando lacasas menores de 486 aminoácidos. Los aminoácidos de unión al Fe conservados en PfaL, en las MCOs de P. chrysosporium y en lacasas y ferroxidasas de ascomicetos se muestran en la Tabla 11, tomando como referencia las posiciones en la ferroxidasa Fet3 (gi2828219) de Saccharomyces cerevisiae (Bonaccorsi di Patti et al., 2000). 77 3.Resultados Tabla 11. Aminoácidos de unión al hierro en la ferroxidasa Fet3 (gi2828219) de S. cerevisiae conservados en PfaL, MCOs de P. chrysosporium, otras ferroxidasas y lacasas de ascomicetos A-Sitio E 185 de Fet3 B-Sitio Y354 de Fet3 C- Sitio D 409 de Fet 3 Fet3-yeast-prec Fet3-pre-Canga Fet3-Flula-prec Fet3-Canal-pre Arx-feO2 Deba-han Ncras-ferr Asp-fum-Feo Yarli- -----PTGAEPIPQN -----PTGAEPIPQN -----PTGAEPIPQN -----PTGAEPIPQN -----PTGAEPVPQA -----PTGAEPIPQN -----PTGAEPVPKS -----PTGAEPVPKA ------ENDEPVPQA 190 190 195 190 116 188 187 190 316 ---VNYAFFNNITYTAPKVPTLMTVLSSG------VNYAFFNNITYTTPKVPTLLTVLSAG------VNYAFFNNLTFTAPKVPTLMTALSAG------INYAFFNNISYKAPKVPTLLTVLSAG------VNYAFFNNITYTAPKVPTLMTALSAG------VNYAFFNNISYVLPKVPTLMTVLSSG------ANYAFFNDITYVPPRVPTLYTVLSSPNSSS ---ANYAFFNGITYVMPKVPTLYSVLTTG------IKRGVFNATTWNPAKEATLSQFLR----EK 377 377 382 377 306 382 377 376 542 DLIVNNQDTGK--------------------EIVLNNQDTGT--------------------EIVLNNNDTGK--------------------DIVLNNFDTGK--------------------EIVVNNNDAGF--------------------EIVLNNMDDGK--------------------QIVLNNLDSGR--------------------DIVLNNDDAGK--------------------DLVINNLDDGA--------------------- 412 412 417 412 341 417 416 411 581 PfaL Pchry-MCO4A Pchry-MCO1 >Pchry-MCO2A Pchry-MCO3B -PTAPIIL-ET-PDS GPLTGLQL-ET-PDS -PIGGSAGDEPVPDS --IDGTAGDEPVPDA --IDGVPGDEPVPDA 238 258 219 271 265 ---GTIAYVNGTSWAPLVGTNSLLQIN----QN ---GAFATMNGTSWQPLQGTTTLLQIVDAARNG ---ASRAYMNGTSWDPLPKTNILLEIQRAYNAG ---LFLGFMNGTSWTPLQGTTTLLQARAAFQGG ---VMLGFLNTTSWEPLRGTTTLLRVREAFRAG 434 458 423 476 470 DILLINQGAGD--------------------DVLLVNTGPGD--------------------DLVLENNDNGD--------------------DLLISNLDDGD--------------------DLVIDNIDDGD--------------------- 471 495 460 513 508 Podosan-lac Melalbo-Lac Ncras-lac Crypho-lac Neucras-lac Gaeugrat-lac Collelag-lac Botryfuc-lac GaeuGram-lac2 Crypar-Lac3 Gaeu-Gram-lac1 Botryfuc-Lac2 Botci-LAC1Botryfuc-Lac1- --------GPPPSDT --------APPFSDN --------GPPPSNN --------APPASDN -FLKGVPGSPPPSNN ------PGVPPPSDN -------GPPPDSDN ------QAGPPSGDN -------GP-PTLEN -------GP-PTMAN -------GAGPLPAN -------GAPPALEN -------GAPPTLLT -------GAPPTLLT 242 197 248 230 235 222 232 134 232 220 238 228 125 222 ---LFVWKVNGSSINVDWDKPIVDYVIA-------LFVWKVNGSDINVDWGKPIIDYILT--------FVWRVNGTAININWNKPVLEYVMT---------KWTINGSTLDVDWGHPITQYVIN----GISKVLWSVDGSAIDVQWDKPTLEYVVE----GRQRVYWEINGSDMNITWDEPTLEYLVK-----EKIYRWRVNGSSMDVQWDKPTLQYIAE------PIVTWGINLSAIDVDWKKPILQYVLD----GNGVFRWYLNSTTMELDWSNPTVSQLASN---LAGRVDWAMGNTTFINEWGYPSIQQVYD----GQNWFQWTLNNSSLVLDWHDPTLERIFD------NYFTWTINSSSLLLDWSSPTTLKIFN---------QWLFNGSSLLLNWTDPTLLTVLN---------QWLFNGSSLLLNWTDPTLLTVLN----- 439 394 440 425 437 422 429 332 429 420 446 426 312 409 YWLIENDPTGP--------------------YWLIENDPEGP--------------------YWLIENDPDGA--------------------YWLIENDPTATG-------------------FWVIQNNSPSP--------------------FWIIQNPTVAP--------------------WWVIENTGALP--------------------YWVIQEVPGNVNG---------------NPVS YVIIQTDFG----------------------YWIVQTTMG----------------------QAMVHTGDSEQRPPTDPLPS-----------EWVVYV--------IEDLTG-----------GWAVLAISGPNGXAFYHYPPILLISNHTDNLF GWAVLAISGPNG-------------------- 471 426 472 458 469 454 461 370 459 450 487 459 365 442 La Tabla 12 resume los aminoácidos implicados en la unión al Fe en Fet3 de S.cerevisiae que ocupan posiciones conservadas en PFAL y Pc-MCOs (de la Tabla 11). Estas posiciones no están conservadas en lacasas. Estos aminoácidos son: • 78 E-234, ácido glutámico en la posición 234 de PFAL, conservado en PcMCOs; 3. Resultados • • Y-413 (tirosina en la posición 413), conservado también en MCO1B (Y398); G-468 (glicina)/D471 (ácido aspártico), G presente también en MCO4, pero ausente en MCO1, MCO2 y MCO3. D, conservado en todas las PcMCOs. Uno de los dos (G o D) podría sustituir la función de D-409 de Fet3. Tabla 12. Aminoácidos de unión al Fe en Fet3 (gi2828219) de S. cerevisiae conservados en PFAL y MCOs que están ausentes en lacasas. Proteína Residuos análogos Referencia PFAL E234 Y413 G468 o D471 Este trabajo Fet3 de E185 Y354 D409 Bonacorssi di Patti S. cerevisiae et al., 2000 3.6. IDENTIFICACIÓN DE LA SEÑAL DE SECRECIÓN Consistente con la función extracelular de la lacasa PfaL y establecida para otras lacasas (Yaver et al. 1999), se trató de identificar la secuencia señal de secreción de PfaL con SignalP versión 3.0. El análisis propuso dos posibles péptidos señal de secreción (A y B , en Tabla 13). Nos parece más probable que la señal de secreción sea la correspondiente a la predicción B. El péptido que contiene los primeros 70 aminoácidos (predicción A) produjo diferentes sitios de corte dependiendo del modelo usado por SignalP: el modelo “neural networks” predijo un sitio de corte en G28 y el modelo “hidden Markov” en A33. 79 3.Resultados Tabla 13. Predicción del péptido señal de PfaL (Predicciones A y B), por SignalP versión 3.0 (http://www.cbs.dtu.dk/cgi-bin/nph-webface?jobid=signalp). NN: “neural networks model”; HMM: “hidden Markov model”. Predicción A Predicción B MVLHSFTRMRIALAALPALLTVMVGLHG-GEVHA-GPHGQYAGVSARSELHAR MRIALAALPALLTVMVGLHG GEVHA-GPHGQYAGVSARSELHAR Predicción B>Sequence SignalP-NN SignalP-HMM length = 62 # Measure position Value >Sequence length = 62 Cutoff signal Prediction: Signal peptide ………………………………………………………………………………………….peptide? max. C 26 0.478 0.32 YES Signal max. Y 26 0.522 0.33 YES Signal max. S 3 0.990 0.87 YES mean S 1-25 0.754 0.48 YES D 1-25 0.638 0.43 YES peptide probability: anchor probability: site probability: 1.000 0.000 # cleavage site between pos. 25/26: VHA-GP cleavage 0.685 between 25/26: Predicción A- SignalP-NN >Sequence # Measure VHA-GP SignalP-HMM length = 70 Position Value Cutoff signal >Sequence length = 70 ………………………………………………………………………………………….peptide? max. C 34 0.256 0.32 NO Prediction: Signal peptide max. Y 29 0.416 0.33 YES Signal max. S 9 0.991 0.87 YES Signal peptide probability: anchor probability: site probability: 0.996 0.003 mean S 1-28 0.892 0.48 YES D 1-28 0.654 0.43 YES # cleavage site between 28/29: LHG-GE cleavage 0.610 between 33/34:VHA-GP 80 3. Resultados La predicción B (62 residuos) es la obtenida eliminando los primeros 8 residuos de la predicción A. La predicción B indica el mismo punto de corte en ambos modelos (A25). Este sitio es el mismo que ofrece el modelo SignalP-HMM en la predicción A. Por lo tanto, la señal de secreción tendría una longitud de 25 residuos (Fig. 16 y Tabla 13) y un sitio de ruptura de la peptidasa señal en VHA-GP. La secuencia amino terminal DTVSLPSSSDIILNGLQGQAPQTRNYDFV de la proteína madura purificada (Pérez et al. 1998 y De La Rubia et al. 2002), se encontraría 18 aminoácidos corriente abajo de la secuencia de secreción extracelular. Para explicar este hecho sería necesario asumir que la secreción de PFAL en el fluido extracelular involucra al menos dos procesos proteolíticos: el primero, liberando la señal de secreción y, el segundo, liberando los 18 aminoácidos de la secuencia de intervención. La predicción B de la secuencia señal (Tabla 13) define el extremo N- terminal del producto de traducción de PfaL como : MRIALAALPALLTVMVGLHGGEVHAGPHGQYAGVSARSELHARDTVSLP. El programa PeptideCutter (http://www.expasy.org/tools/peptidecutter/peptidecutter_enzymes.html) identificó un único sitio de corte (posición 43/44, HAR-DTV) para una Asp-N endopeptidasa (Peptidil-Asp metalo-endopeptidasa). Esto sugeriría que el segundo procesamiento proteolítico produciría la secuencia N-terminal identificada en la lacasa de P. flavido-alba purificada. De este modo, parece más que posible que el mecanismo de secreción extracelular de PfaL, sea a través de dos procesamientos proteolíticos en el extremo N-terminal. 3.7. IDENTIFICACIÓN DEL PROMOTOR DE pfaL En la secuencia genómica caracterizada se han identificado un promotor putativo y sitios de unión de factores de transcripción, especialmente algunos motivos putativos de lacasas aguas arriba del extremo 5’ del gen pfaL (Fig. 18 y Tabla 14). 81 3.Resultados -1591 tctagacatgcagtcgtggatggcacgctttcgtttacgacattgcaacgaatccacttg -1531 tttgctttggcgatcccgagctgccgagtagcagactaacccccaaccggatgctcttgg -1471 cctgaggagaggactctctcttgcatgtcctttcactttcaacatgtcaagagtccgcct MRE -1411 taacgcaccttcatgcacttccgggcgtggtaacgagttgcatgcacaccgctcggggct NIT2 -1351 cacagcaccgcgcagctcccatttcgacgatattcattcctccttctaattgttgatgta -1291 ggttagcatttttctccccgacggtctgatgctgtccgaaatggaccgctgtgcactgcc -1231 gctgcgaaggccgacgcgacgcgaacgaagcgaagcggtaacccttcgccagctcgcgaa Ap1 -1171 ttcgtccagccccgtcgcagcgaaccgggatgccatggtgggcgatgcatcctcatgagt -1111 aatgtccttcggtcatttaccgagcgtatgcgactctccctggattcggctccgggagat PRE NIT2 -1051 gctgctctgggtgagtatcataccgggtttataactttcattcccaagggaccataatca -991 ctcatactgattaccagttcttgtcaggccttgtattggaggtggcgcacgattcgcggc PRE ARE NIT2 -931 tccgccaaacccaagtgacccgcgaacagttcggatagaaggctcgaaaatgcctctccg -871 gtggtgatgcgttcatcgtgcgaatggggctgctgcggtaacagaaattgatagaccgcc -811 tctgtttatgtaagttttgagccgcttcttcaacagaaagctctgttactgcttagagag -751 gagttatcaggcctcaccgctccgatgaaggatgccggcactaagctttcctcctaatca -691 gcgggtaccttacctccatgtgcccgggttgatcagcgtacccctccgccgcgtcgtgtg -631 agtttaggttttgttcgtccacaaagggatgcgctaatgtgatcccaggtgaagtggacg -571 cctaccagaacacgttctacgacgccgcctgacgctacccctcggcctcgcagtccacgg MRE XRE -511 ctattgaacgtgggaggccgtacttacgcacgcaagcggctgcgtccctatcccgcagtt Mig1p -451 agcgcccttccataccctgtcagttaggcggagacggcctatgaataccatggttaacgg -391 gttcgatttgttgcagtgcggcacggcacagggcactcgtcagcaatgcagtacgcgtcc ARE -331 tgacaaggagagggaatgcggtatcctcaggccctcgcgcacggttccatattgggccac -271 acttactgctctatgcgactaattccaattccataaaccagatggaagaccatgcgaaga MRE PRE -211 gccgacataccggcaagatcttattgctacgtgcgtacccaaatgtgcgcatctctacat -151 gggtccgcgccgtgtagtccaggccagcgaccgctttggtatgtgcgaatggtataaaac PRE -91 ccttggtttcttgactaacatttcttcacaacccaggtgtcctacactccttccttcctc -31 aaggacatggttctccactccttcactcgcATGCGT +1 Figura 18. Sitios de la región reguladora del gen pfaL de Phanerochaete flavido-alba. El promotor putativo en cuadro gris y las cajas TATA y CCATT están subrayadas. El codón de inicio ATG en mayúscula (sitio +1). Varios elementos de respuesta están identificados para metales (MRE), xenobióticos (XRE), antioxidantes (ARE y Ap1), putativo (PRE), nitrógeno (NIT2) y carbono (Mig1p). El inicio de la transcripción dentro del promotor putativo es “C”, de acuerdo a GenScan 1.0 (apartado 2.11.4). 82 3. Resultados El promotor se ubica en el sitio -113 respecto al codón ATG de inicio de la traducción. Tiene un tamaño de 49 pb, según GenScan1.0 (http://genes.mit.edu/GENSCAN.html). La caja TATA (TATAAAA) y un posible inicio de la transcripción (TSS) se localizan en las posiciones -99 y -68 respectivamente. Tabla 14. Regiones reguladoras identificadas en el gen pfaL. Localización respecto al inicio de la traducción. Regiones identificadas TATA box CCATT box PRE MRE XRE ARE Mig1p NIT2 Ap1 Secuencia consenso TATAAAA CCAATT, ATTGG ACCCA, TGGGT TGCACAC, ACGTGGG, ACGTGCG CACGCA TGACCCGC, TGACAAGG GCGGAG CGGATA, TATCAT, ACGATA TGAGTAA Localización -99 -247, -281, 957 -61, -175, -923, 1044 -183, -504, -1369 -483 -331, -916 -424 -900, -1036, -1325 -1116 Además, se identificaron varias secuencias consenso de elementos reguladores de la transcripción de lacasas de basidiomicetos (Soden y Dobson, 2003). En concreto se identificaron: • • • • • Cuatro elementos PRE (TGGGT y ACCCG), elementos de respuesta putativos de lacasas; Tres cajas CCATT, de regulación constitutiva de lacasas de basidiomicetos; Tres elementos de respuesta a metales (MRE), Un elemento de respuesta a xenobióticos (XRE), Dos elementos de respuesta a antioxidantes (ARE) y un elemento putativo a antioxidantes (Ap1); 83 3.Resultados • • Un elemento de respuesta al carbono, GCGGAG (Mig1p), homólogo a CreA (elemento de respuesta a cAMP); y Tres elementos de respuesta a nitrógeno (NIT2). No se encontraron sitios consenso conservados en otros genes de lacasas, como son los elementos de respuesta a choque térmico (HSE) ni el elemento potenciador de la transcripción de mamíferos (Sp1). 3.8. REGULACIÓN DE LA EXPRESIÓN DE LA LACASA POR FENOLES, HIERRO Y COBRE En esta sección se presentan resultados que ilustran los mecanismos moleculares que regulan la producción de lacasa PFAL en P. flavido-alba. Se muestra cómo compuestos aromáticos y cationes metálicos divalentes pueden regular la expresión del gen pfaL, con o sin aumento de la actividad extracelular. Los inductores mencionados (fenólicos y metálicos- apartado 2.3.3.) se añadieron a cultivos de P. flavido-alba de tres días. Tras 72 horas con los inductores se midió la actividad lacasa y se cuantificó (QPCR en tiempo real) el mRNA pfaL y el rDNA 18S, que se utilizó como control. 3.8.1. Regulación por fenoles Los resultados muestran que el transcrito pfaL es sobre-expresado en cultivos suplementados con el pigmento polimérico del alpechín (PMA) (Figura 19). Sin embargo, la adición de esta mezcla compleja de aromáticos no incrementó significativamente la actividad lacasa en los sobrenadantes (Fig. 19A), lo que sugiere la traducción ineficiente del mRNA pfaL, cambios postraduccionales deficientes de la lacasa PFAL, o simplemente la inactivación de la lacasa extracelular producida. Antes de contemplar la inducción por aromáticos simples (ácido ferúlico, tirosol y vainillina) conviene estudiar el efecto del disolvente empleado para incorporarlos al cultivo (1 ml de una solución 3:7 de DMF/agua en cada 25 ml de medio de 84 3. Resultados Actividad lacasa 20000 A mU / mg de proteina 18000 16508 16000 14000 12000 10000 8000 6000 4000 1803 2000 1283 810 BL PMA 1425 967 0 DMF Fer Van Tir mRNA pfa L cuantificado 4,5 B Niveles de expresión 4,0 3,5 3,0 3,6 2,5 3,3 2,8 2,0 1,5 1,8 1,0 0,5 1,0 0,2 0,0 BL PMA DMF Fer Van Tir Figura 19. Actividad lacasa (A) y cantidad de RNAm (B) de pfaL afectadas por adición de compuestos aromáticos. BL: medio basal; PMA: BL con pigmento polimérico del alpechin (8,3 g/l); DMF: BL con dimetilformamida; Fer: BL con ácido ferúlico (1mM); Van: BL con vainillina (1mM); Tir: BL con tirosol (1mM).; Los resultados representan valores promedio y desviación estándar de cinco repeticiones para la actividad y de 4 repeticiones para cuantificación del mRNA. 85 3.Resultados cultivo). A diferencia del PMA, la DMF aumentó la expresión génica y la actividad lacasa extracelular (Fig. 19). El ácido ferúlico, disuelto en la solución de DMF, incrementó la transcripción, demostrando un efecto aditivo de la mezcla. Por otro lado, el ácido ferúlico no modificó significativamente la actividad extracelular. En conclusión el ácido ferúlico, como el PMA, aumentan la transcripción de pfaL, pero no la actividad extracelular. Por su parte el tirosol, que no modificó la actividad lacasa pero sí disminuyó la cantidad de mRNA, podría reprimir la transcripción o activar la degradación del mRNA. En cambio, los cultivos suplementados con vainillina proporcionaron el máximo incremento en la actividad lacasa total y por mg de proteína (12.9 veces la actividad de los cultivos control). Sorprendentemente, el micelio contenía la cantidad más baja de mRNA pfaL. Esto sugiere que, al contrario de PMA y ácido ferúlico, la vainillina facilitó la traducción de mRNA pfaL y/o provocó posibles modificaciones post-traduccionales del péptido PFAL. Los resultados obtenidos en los cultivos con adición de ácido ferúlico o vainillina permiten concluir que la actividad extracelular de la lacasa de P. flavido-alba está regulada tanto a nivel transcripcional como traduccional/postraduccional. 3.8.2. Regulación por Metales 3.8.2.1. Regulación por deficiencia de Fe La ausencia de Fe II, o que no esté disponible en los cultivos al adicionar el quelante del Fe (Fig. 20), hizo que la actividad extracelular de la enzima PFAL disminuyera, sin afectar los niveles de transcrito. Esto indica que la disponibilidad de hierro regula la traducción del mRNA o la activación de la proteína y sugiere, por tanto, una regulación traduccional o postraduccional de la proteína. 86 3. Resultados Actividad lacasa 700 A mU / mg de proteína 600 500 588 400 300 200 279 293 219 Fe BCS Cu 0,4 0,9 0,1 Fe BCS Cu 100 69 0 BL BPS mRNA pfa L cuantificado 1,8 B 1,6 Nivel de Expresión 1,4 1,2 1,0 0,8 1,3 1,2 0,6 0,4 0,2 0,0 BL BPS Figura 20. Actividad lacasa (A) y cantidad de RNAm (B) de pfaL afectadas por hierro y cobre. BL: medio basal; BPS: medio basal sin FeII con batofenantrolindisulfonato-sódico (1mM); Fe: medio basal sin FeII con sulfato de hierro amónico (25 mM); BCS: medio basal sin CuII con batocuproinadisulfonato-sódico (1 mM); Cu: medio basal sin CuII con sulfato de cobre (24 mM). Valores de actividad son el promedio de tres réplicas; los niveles de expresión son el promedio de cuatro qPCR en tiempo real. 87 3.Resultados 3.8.2.2. Regulación por adición de Fe Al adicionar a los cultivos, desarrollados con déficit de Fe II, concentraciones de Fe similares a las del cultivo control (25 µM), PFAL recuperó su actividad y, al mismo tiempo, disminuyó los niveles de mRNA. Es posible, por tanto, una regulación a varios niveles: 1) traduccional, pues el hierro puede hacer más disponible el mRNA para la síntesis de PFAL, 2) transcripcional, reduciéndolo, 3) aumentado la inestabilidad del mRNA de pfaL. 3.8.2.3. Regulación por Cobre Como en los cultivos a los cuales se les añadió vainillina, la suplementación con Cu II (8,5 veces la concentración del medio BL) aumentó la actividad lacasa extracelular y disminuyó la cantidad de mRNA. El déficit de Cu II no modificó significativamente ninguno de los dos parámetros. En conclusión, los dos metales estudiados regulan la producción de la lacasa en P. flavido-alba en forma análoga como lo hace la vainillina. La indisponibilidad de Fe probablemente impide los cambios postraduccionales que permiten la acumulación extracelular de la lacasa activa. De esta forma, como se ha explicado, el restablecimiento de los niveles de Fe restablecen la actividad extracelular (aumenta) y disminuye la cantidad de mRNA (como en el caso de la vainillina). 88 4. DISCUSIÓN 4. Discusión 90 4. Discusión P. flavido-alba produce las tres enzimas oxidativas características de los hongos ligninolíticos Lip, MnP y lacasa (Ben Hamman, 1998). P. flavido-alba degrada los componentes recalcitrantes y tóxicos del alpechín. La lacasa de P. flavido-alba oxida varios de sus fenoles monoméricos y algunos inducen su producción. Varias de las propiedades de esta lacasa se han deducido de la proteína inducida por 4-hidroxi-3-metoxibenzaldehido (vainillina) (De La Rubia et al., 2002; Lucas et al., 2005). Este trabajo ha permitido conocer otros aspectos de la lacasa de P. flavido alba: la secuencia génica que la codifica, la regulación de su expresión por componentes orgánicos e inorgánicos y su parentesco filogenético con las lacasas y otras multicobre oxidasas fúngicas . La lacasa de P. flavido-alba posee los atributos típicos de una enzima de este tipo (De la Rubia et al., 2002, Lucas et al 2005, Pérez et al., 1996). Sin embargo la organización genética de pfaL y la secuencia peptídica deducida (PfaL) se corresponden mas a los de la ferroxidasa MCO1 de P. chrysosporium (Larrondo et al., 2003). 4.1. ESTRUCTURA DEL GEN pfaL En el presente trabajo, se ha identificado un gen (pfaL) de la lacasa de P. flavidoalba) PfaL en una región de 4589 pb. El gen tiene un tamaño de 2755 pb, incluyendo 19 intrones. En general, el número de intrones en las lacasas de hongos es muy variable, pero inferior al encontrados en el gen pfaL (Tabla 8). Por otra parte, y dada la alta identidad de PfaL (Tabla 4) con las proteínas codificadas por los genes mco de P. chrysosporium, es presumible también una gran similitud en la organización genética. Efectivamente el gen pfaL de P. flavido-alba posee un número de intrones muy similar a los genes mco de P. chrysosporium, (18 intrones en mco4, 19 en mco1 y 14 en mco2 y en mco3; Larrondo et al. 2003 y 2004). Además, también es muy similar la posición de los intrones, en especial en zonas conservadas tales como los exones que codifican las histidinas de los tres centros del cobre en mco1, exones V, VI, VII, XVI, XIX y XX (Fig.21). No obstante, en la 91 4. Discusión expresión de pfaL no hemos encontrado las variantes de “splicing” descritas en la transcripción de los genes mco de P. chrysosporium (Larrondo et al 2004). PfaL está delimitado por una región reguladora de 1590 pb en 5´ y por una UTR-3’ de 244 pb. I II III IV V VI VII VIII IX X XI XII XIII XIV XV XVI XVII XVIII XIX XX mco1 2704 pb pfaL 2755 pb I II III IV V VI VII VIII IX X XI XII XIII XIV XV XVI XVII XVIII XIX mco4 2768 pb Figura 21. Comparación en la distribución de exones/intrones entre los genes pfaL de P. flavido-alba (descritos en este trabajo) y mcos de P. chrysosporium (descritos en Larrondo et al., 2004). Los exones se representan desde I hasta XX. El mRNA (1704 nucleótidos) codifica un polipéptido de 567 aminoácidos, que incluye una señal de secreción de 43 aminoácidos. La secuencia peptídica deducida (PfaL) contiene los seis oligopéptidos ya caracterizados en la lacasa purificada (De la Rubia et al. 2002; Lucas et al. 2005), por lo que se concluye que el gen identificado corresponde efectivamente a la lacasa de P. flavido-alba. 4.2. FILOGENIA DE PFAL La mayoría de las lacasas fúngicas se distribuyen filogeneticamente en dos “clades” principales según el tipo de hongo: lacasas de ascomicetos y lacasas de basidiomicetos (Valderrama et al., 2003). PfaL no se alinea filogenéticamente con las lacasas de los basidiomicetos, sino con las de los ascomicetos. Además una lacasa de un basidiomiceto (Cryptococcus neoformans, gi 627028) y otra de un ascomiceto (Aspergillus nidulans gi1346405) forman un tercer grupo 92 4. Discusión independiente (Valderrama et al., 2003) Actualmente esta lacasa de A. nidulans no se encuentra disponible, pero sí lo esta la de Cryptococcus neoformans. PfaL, MCO1 de P. chrysosporium , una MCO de Yarrowia lipolytica y una lacasa de C. neoformans (que muestra una identidad del 99% con la utilizada por Valderrama et al., 2003, la de C. neoformans gi627028) formaron un grupo filogenético independiente de ferroxidasas y lacasas. Así, además de robustecer el significado de la subfamilia propuesta por Larrondo et al. (2003) para alojar las MCOs de P. chrysosporium, estos resultados refuerzan el interés por el “tercer” grupo de lacasas descrito por Valderrama et al. (2003). 4.3. ESTRUCTURA PRIMARIA DE PfaL 4.3.1. Aminoácidos conservados de PfaL En la secuencia deducida para PfaL se encuentran los residuos típicos que participan en la unión al cobre en las lacasas, diez histidinas y una cisteína (Kumar et al., 2003).PfaL tiene varias características que son únicas para MCOs. Algunas secuencias tienen residuos conservados de ferroxidasas, pero no de lacasas. Otras son más propias de lacasas que de ferroxidasas. Los aminoácidos de PfaL que forman los motivos L1-L4 de unión al cobre (Kumar et al., 2003) están conservados, más en las MCOs de P. chrysosporium, menos en las ferroxidasas y menos aún en lacasas fúngicas. Posiblemente, por extensión y por número de sustituciones, L-3 es el motivo de unión al cobre más conservado entre todas las multicobre oxidasas analizadas. La mayor variación se encuentra en los motivos L-1 y L-4. Quizás es por eso que PFAL sea la única multicobre oxidasa analizada que en L1 posea valina, triptófano y serina. Val y Trp son más hidrófobos que los aminoácidos que ocupan esta posición en las PcMCOs (Asp y Tyr). En las ferroxidasas el Trp está sustituido por la Met, otro aminoácido hidrofóbico. La ferroxidasa Fet3 de S. cerevisiae participa en la homeóstasis del hierro. Curiosamente, a la lacasa de C. neoformans (gi57228040) se le atribuye cierta actividad y rasgos estructurales de ferroxidasas (Liu et al., 1999; Williamson, 1994), que se correlacionan con la posición filogenética intermedia que ocupa junto 93 4. Discusión a PfaL, entre los linajes de lacasas y ferroxidasas, en el árbol mostrado en la Fig.. 17). Se han identificado varias zonas de las ferroxidasas responsables de la oxidación del FeII. (Askwith and Kaplan, 1998; Bonaccorsi di Patti et al. 2000 y 2001). Tres amino ácidos participan en la unión al FeII en la Fet3 de S. cerevisiae: Glu-185, Tyr-354 y Asp-409.Estos residuos están conservados en ferroxidasas y ausentes en lacasas. Sin embargo, PfaL presenta Glu-234 y Tyr-413 como los equivalente a Glu-185 y Tyr-354 de Fet3 de S.cerevisiae. La localización de Asp-409 en PFAL es más dudosa, puesto que es sustituido por Gly-468, aunque quizá su función podría ser asumida por Asp-471. Estos aminoácidos de PfaL se conservan en PcMCOs, aunque Larrondo et al. (2003) describió solo el equivalente de Glu-185 en MCO1, sugiriendo que es el residuo esencial para la oxidación de FeII. 4.3.2. Glicosilación El análisis de la proteína deducida permitió determinar varias características particulares de PFAL. En primer lugar, la masa molecular deducida del péptido PfaL maduro (sin la secuencia señal) es de 56,5 kDa, un 70% de la masa molecular experimental (81,3 kDa) de la lacasa de P. flavido-alba desglicosilada (De la Rubia et al, 2002). Asumiendo que la digestión de N-glucanos fuera completa la diferencia en masa molecular sería atribuida a O-glucanos presentes en la proteína madura. Por lo tanto, PfaL sería una proteína más glicosilada que el común de las lacasas fúngicas (Farnet et al. 2000; Muñoz et al. 1997). 4.3.3. Péptido señal El extremo amino de la lacasa pura (De La Rubia et al., 2002) se corresponde con la secuencia de PfaL a partir del aminoácido 44. De acuerdo a SignalP3.0, pfaL presenta una secuencia señal de secreción de 25 aminoácidos. Por tanto la señal de secreción está incluida en un pre-péptido de 43 aminoácidos. El péptido codificado por pfaL, debería sufrir al menos dos procesamientos proteolíticos en el extremo N-terminal antes de producir la lacasa extracelular. Este mecanismo es diferente al que ocurre generalmente en la secreción de las lacasas. 94 4. Discusión En varias lacasas de ascomicetos se ha reconocido un procesamiento posttraduccional complejo (Bulter T, et al . 2003; Kiiskinen y Saloheimo 2004). Por ejemplo, la lacasa de Myceliophthora thermophila (MtL) tiene una pre-prosecuencia que necesita ser procesada en el extremo amino y en el extremo carboxilo para ser secretada (Alcalde et al., 2005). En otras proteínas ocurre algo similar, como en el precursor del factor sexual α de S. cerevisiae. Dicho factor sufre tres procesamientos proteolíticos antes de su exportación: uno en el extremo carboxilo y dos en el extremo amino. El primer procesamiento proteolítico en el extremo N-terminal (Fujimura-Kamada, et al., 1997) lo ejerce una Zn-metaloproteasa de membrana (STE24). En las lacasas de basidiomicetos, son menos frecuentes estos mecanismos: Yaver et al. (1999) describió por vez primera la necesidad de un procesamiento similares en la biosíntesis de la lacasa de C. cinereus. Nuestros resultados, sugieren un doble procesamiento en el extremo N-terminal de PfaL, en el que se ubican al menos dos sitios putativos de corte por Nendopeptidasas. Actualmente, no es muy abundante la información sobre endopeptidasas de hongos filamentosos, especialmente las involucradas en la secreción de lacasas en hongos de la podredumbre blanca: solo dos familias de proteasas han sido reconocidas en el genoma de P. chrysosporium (base de datos MEROPS http://merops.sanger.ac.uk/). 4.4. ELEMENTOS PUTATIVOS DE REGULACIÓN TRANSCRIPCIONAL El análisis de la región promotora de pfaL, amplificada con el gen mediante PCR inversa, permitió identificar varios elementos consenso de potencial regulación transcripcional, los cuales pueden afectar la producción de la lacasa de P. flavidoalba. Los elementos encontrados son: elementos de respuesta putativos (PRE), elementos de respuesta a metales (MRE), a xenobióticos (XRE), a antioxidantes (ARE y Ap1), a nitrógeno (NIT2) y a carbono (Mig1p) (Figura 18, Tabla 14). MRE (TGC(G/A)CNC) y XRE (TCACGC) han sido descritos en regiones promotoras de diferentes genes de lacasa y de manganeso peroxidasa de hongos WRF (Coll et al., 1993; Gold y Alic, 1993; Giardina et al., 1995). Inicialmente, los MRE fueron identificados en promotores de genes de metalotioneinas en diferentes 95 4. Discusión eucariotes superiores (Imbert et al. 1990). Se sabe que tales genes son inducidos por metales pesados como Cd, Cu y Zn (Hamer, 1995). En Pleurotus sajor-caju, cuatro genes de lacasa fueron expresados diferencialmente por la presencia o no de elementos de regulación. Así por ejemplo, los niveles de mRNA del gen lac4, inducidos por adición de MnSO4, se correlacionan con la presencia de una secuencia MRE (Soden y Dobson 2001). Los genes lac1 y lac4, que poseen secuencias XRE, son regulados transcripcionalmente por adición 2,5-xilidina y ácido ferúlico, mientras los genes lac2 y lac3, sin secuencias XRE, son constitutivamente expresados bajo las mismas condiciones (Soden y Dobson 2001). De este modo, los elementos MRE y XRE pueden estar involucrados en la inducción de la expresión del gen lac4 por manganeso y 2,5xilidina, respectivamente. Además, la presencia de elementos ARE (TGACN NNGC) en el gen lac4, identificados inicialmente en células de mamíferos (Rushmore et al. 1991), indican una función potencial en su expresión por antioxidantes fenólicos. De otra parte, elementos reguladores tales como Mig y NIT2, involucrados en la regulación del carbono y del nitrógeno en la expresión de genes fúngicos, se encuentran también en regiones promotoras de los mismos genes de P. sajor-caju (Soden y Dobson, 2001) y en otros de diferentes especies de hongos. Mig1p es un regulador clave en la represión por glucosa en S. cerevisiae (Trumbly, 1992); en A. nidulans, se ha descrito un homólogo de Mig1p llamado CreA; en Trichoderma harzianum, T. reesei (Strauss et al. 1995; Takashima et al. 1996) y en Metarhizium anisopliae (Screen et al. 1997) se han identificado proteínas cre1 y crr1 que actúan sobre elementos reguladores similares a CreA. Estos hallazgos han sugerido que el mecanismo regulador ejercido por Mig o similares está conservado entre los hongos. La unión de los represores de levaduras y de hongos Mig1p, Mig2p y CRE requieren una caja GC con secuencias consenso 5`-SYGGGG-3` y 5`SYGGRG-3` (S¼C o G, Y¼C o T y R¼A o G), respectivamente (Cubero y Scazzochio 1994; Lundin et al. 1994). Sólo una de estas secuencias fue identificada en la región promotora del gen pfaL, indicando su posible represión por glucosa a través de proteínas reguladoras en P. flavido-alba. La potencial regulación de la transcripción del gen de la lacasa por nitrógeno depende de motivos similares a NIT2 presentes a 900, 1036 y 1325 nucleótidos aguas arriba del gen pfaL (Fig. 18). Un gran número de genes estructurales 96 4. Discusión involucrados en la asimilación de nitrato, metabolismo de purinas, metabolismo de aminoácidos, catabolismo de proteínas y utilización de acetamida requieren una proteína NIT2 (areA o gln3) para su expresión (Marzluf, 1997). De esta forma, es posible que homólogos de NIT2 regulen la expresión de pfaL en respuesta a nitrógeno en P. flavido-alba. En conclusión, los mecanismos por los cuales el carbono, nitrógeno, cobre, hierro y compuestos aromáticos regulan la transcripción de genes de la lacasa PfaL se desconocen. Tal como se ha demostrado en otros genes, las secuencias consenso putativas PRE, MRE, XRE, ARE, Mig y NIT2 identificadas aguas arriba de este gen pueden estar involucradas en dicho mecanismo. La ausencia o presencia de estos elementos puede indicar que un gen en particular responda o sea inducido por la presencia o ausencia de diferentes condiciones fisiológicas, metales o compuestos aromáticos. 4.5. REGULACIÓN TRANSCRIPCIONAL DE pfaL POR COMPUESTOS FENÓLICOS Trabajos previos sobre la degradación del alpechín (OMW), han determinado que el PMA es un complejo de compuestos fenólicos de alto peso molecular, que constituyen el color marrón oscuro del OMW. Su decoloración (cerca del 70%) por P. flavido-alba, está asociada a disminución en contenido fenólico total y a disminución de los efectos fitotóxicos y antibacterianos. Concretamente la decoloración se atribuye a manganeso peroxidasa y a lacasa, que despolimerizan los componentes de alto peso molecular del PMA y repolimerizan y/o mineralizan componentes de bajo peso molecular (Ben Hammam, 1998). Recientemente, se determinó que la inducción de la lacasa de P. flavido-alba por el alpechín, se debe más probablemente a los fenoles monoméricos que al PMA del OMW (Ruiz et al. 2002). La vainillina y el p-hidroxibenzaldehído son los inductores más eficaces (De la Rubia et al, 2002). Nuestros resultados confirman la inducción de la lacasa de P. falvido-alba por vainillina. Además, muestran que la regulación de la expresión de la lacasa pfaL es compleja y depende del inductor. En basidiomicetos la acumulación de lacasa extracelular está asociada a la acumulación del mRNA intracelular, como se ha 97 4. Discusión demostrado por ejemplo en Volvariella volvacea (Chen et al., 2003) o en Trametes (Terrón et al., 2004). La situación es diferente para la lacasa de P. flavido-alba: hay sustancias, como la vainillina, que aumentan la actividad lacasa de los cultivos, pero no aumentan la cantidad de transcrito acumulado intracelularmente, sino que lo disminuyen. Por otro lado el PMA, el ácido ferúlico y la DMF, que no aumentan la actividad lacasa, parecen inducir la transcripción del gen pfaL de P. flavido-alba puesto que los cultivos aumentan casi tres veces la cantidad de mRNA. Parece pues que en estos casos la traducción y/o postraducción quedaron bloqueadas, pues la actividad lacasa extracelular en los tres casos fue semejante a la de los cultivos control sin inductores (Figura 19). Como se observa en los resultados, paradójicamente la vainillina parece regular la lacasa a nivel de la traducción/postraducción. Las lacasas de Trametes sp. I-62 son inducidas de manera diferencial por compuestos aromáticos. El ácido ferúlico induce la expresión de lcc3, el guaiacol y el ácido p-cumárico la de lcc1 y de lcc2, mientras que el ácido 3,5dihidroxibenzoico no altera la transcripción de ningún gen de lacasas (Terrón et al., 2004). El incremento de la cantidad de transcrito de pfaL observado con algunas sustancias, puede resultar del incremento de la transcripción de pfaL y/o del incremento de su estabilidad. Frecuentemente la inducción de la expresión de los genes de lacasa de basidiomicetos se ha relacionado con las secuencias reguladoras ubicadas en 5´. Cerca del promotor de pfaL, a -483 pb del codón ATG, se ha localizado un elemento putativo de respuesta a xenobióticos (XRE) y dos elementos putativos de respuesta a antioxidantes (ARE), a -331 y -916 desde ATG (Fig. 18). Ambos podrían estar relacionados con la inducción de la expresión por PMA, ácido ferúlico y DMF. XRE y ARE han sido encontrados en promotores de genes que degradan xenobióticos o hidrocarburos aromáticos, tales como genes de lacasas fúngicas (Soden y Dobson, 2003), genes de citocromos P450, glutation-Stranferasas y NAD(P)H:quinona oxidoreductasas de eucariotas (Kuramoto et al., 2002; Nguyen et al., 2003). Las secuencias XRE y ARE aumentan (en cis) la transcripción de genes relacionados con el estrés oxidativo . Los genes regulados por XRE y ARE codifican proteínas que ayudan a controlar el estado redox de la célula y la defienden contra el daño oxidativo (Nguyen et al., 2003) 98 4. Discusión En conclusión, el PMA, el ácido ferúlico, o la DMF, pueden cambiar el estado redox celular de P. flavido-alba e inducir la expresión diferencial de mRNA pfaL, a través de factores transcripcionales que se unen a secuencias consenso XRE y/o ARE. También es posible la presencia o ausencia de otros factores desconocidos que estabilizan el mRNA, evitando la traducción y procesamiento de la proteína, lo que explicaría por qué no se detectó actividad lacasa PfaL a pesar de la sobreexpresión génica. Estos mecanismos serán aclarados en futuros estudios de regulación de pfaL. 4.6. REGULACIÓN TRADUCCIONAL/POSTRADUCCIONAL DE PfaL POR VAINILLINA, Fe Y Cu. 4.6.1. Regulación por vainillina La vainillina es un metoxifenol de tipo aldehídico (4-hidroxi-3metoxibenzaldehído) que se encuentra en la naturaleza como intermediario de la degradación de la lignina (Tai et al, 1990) y como componente del alpechín (Martínez-Nieto et al, 1992). Se sabe que induce la actividad lacasa de P. flavidoalba, pero se desconocen los mecanismos moleculares de degradación por esta enzima. En varias lacasas fúngicas, se ha descrito que la inducción de la actividad se corresponde con el aumento de la transcripción del mRNA por compuestos fenólicos (Chen et al, 2003; Collins y Dobson, 1997). En nuestro caso, la adición exógena de vainillina a cultivos de P. flavido-alba incrementó la actividad lacasa de PfaL, pero disminuyó aparentemente la transcripción del gen. Estos resultados sugieren que la vainillina regula la producción de lacasa a nivel traduccional y/o postraduccional (Figura 19). No se conoce el efecto de la vainillina sobre las MCOs de P. chrysosporium, pero sí sobre otras enzimas relacionadas con la oxidación de moléculas aromáticas. En P. chrysosporium, la vainillina produce cambios en las enzimas de la biosíntesis del grupo hemo a nivel mitocondríal (Figura 22). El resultado es la acumulación de hemoproteínas citoplasmáticas y extracelulares como peroxidasas, citocromo P450 oxigenasas y catalasas (Shimizu et al, 2005). Estas peroxidasas fueron relacionadas con la degradación de aromáticos (Kirk y Farell, 1987; Gold et al, 1989; Ichinose et al, 2002a y 2002b; Hiratsuka et al., 2001; Masaphy et al., 1996). 99 4. Discusión También se ha descrito que P. chrysosporium metaboliza la vainillina en vainillinalcohol y ácido vainíllico a través de aril-alcohol deshidrogenasa y aldehido deshidrogenasa (Shimizu et al., 2005). El ácido vainíllico a su vez se descarboxila a metoxihidroquinona y posteriormente es convertido a 1,2,4-trihidroxibenzeno (Rieble et al, 1994). La ruptura del anillo puede ser catalizada por 1,2,4trihidroxibenzeno-1,2-dioxigenasa (Rieble et al, 1994) u homogentisato 1,2dioxigenasa (Shimizu et al., 2005). Aunque la 1,4-benzoquinona reductasa (una flavoproteina) puede mantener las quinonas, derivadas de la vainillina, en sus correspondientes hidroquinonas (Shimizu et al., 2005). Estudios posteriores determinarán el papel de la lacasa de P. flavido-alba y su relación con estas enzimas de P. chrysosporium en la degradación de la vainillina. De otra parte, la adición de vainillina también puede sobre-expresar proteínas antioxidantes y proteínas de choque térmico, pues la oxidación fúngica de compuestos relacionados con la lignina o hidrocarburos aromáticos incrementa la formación de radicales superóxido (Guillen et al, 2000) y expresan respuestas a estrés químico (Kim et al., 2002b). En cultivos de P. chrysosporium con vainillina, se han identificado proteínas de estrés oxidativo, tales como Mn-superóxido dismutasa, glutatión S-transferasa, tiorredoxina peroxidasa, tiorredoxina reductasa, glutatión peroxidasa, glutatión reductasa y catalasa; y proteínas de choque térmico como HSP70 y HSP80 (Shimizu et al., 2005). 100 4. Discusión Figure 22. Flujo metabólico propuesto en P. chrysosporium en presencia de vainillina. Las enzimas sobre-expresadas se muestran en cuadros (ACO aconitasa; ALA5 sintetasa del ácido 5-aminolevulinico; CIT citrato sintetasa; ENO enolasa; GPD gliceraldehido-3-fosfato deshidrogenasa; ICL isocitrato liasa; IDH isocitrato deshidrogenasa; ODH 2-oxoglutarato deshidrogenasa; TPI triosa-fosfato isomerasa). (Tomado de Shimizu et al, 2005). La regulación traduccional/postraduccional de la lacasa PfaL por vainillina puede requerir la acción de uno o varios metabolitos de la vainillina. También es posible que la biosíntesis de la lacasa esté relacionada con la biosíntesis de enzimas del 101 4. Discusión grupo hemo (regulado a su vez por la disponibilidad de Fe), con la inducción de enzimas antioxidativas o con la inducción de enzimas de estrés químico (proteínas de choque térmico). Estudios futuros podrán definir cómo la vainillina o sus metabolitos permiten que el mRNA pfaL esté disponible para la traducción en la proteína y cómo intervienen en el procesamiento, secreción y/o activación de la lacasa PfaL. 4.6.2. Regulación por cobre La lacasa de P. flavido-alba no parece estar regulada transcripcionalmente por cobre, puesto que la suplementación en Cu disminuye la cantidad de transcrito (Fig. 20B). Dos argumentos apoyan esta hipótesis: En condiciones de exceso de cobre, el aumento de actividad observado en cultivos de varios hongos se debe a la sobre-expresión de los genes acumulando mRNA (Soden y Dobson, 2003; Karahanian et al, 1998; Coll et al. 1993); sin embargo, el mRNA pfaL disminuyó al añadir Cu. Por lo que deducimos que el aumento de actividad lacasa que provoca la suplementación de Cu estaría relacionado con eventos traduccionales y postraduccionales. La expresión de los genes de varias metaloenzimas fúngicas, incluyendo lacasas, en presencia de exceso de cobre está regulada por un factor de transcripción (Ace1p, Buchman et al, 1989; Welch et al, 1989; Soden y Dobson, 2003), cuyo consenso de unión no se ha localizado en pfaL (Fig. 18), reforzando la hipótesis de la regulación traduccional/postraduccional de PfaL por cobre. El cobre podría aumentar la síntesis de la proteína (no cuantificada), que podría reflejarse en el aumento de la actividad lacasa y en la disminución de la cantidad de mRNA (Fig. 20). Es la primera vez que se describe este evento de tipo traduccional en lacasas y desconocemos los mecanismos que regulan la traducción del mRNA de pfaL. Futuros estudios permitirán aclarar el efecto del cobre en la traducción de PFAL. Por el contrario, existen fundamentos para la regulación postraduccional de la biosíntesis de MCOs que se pueden proponer para la biosíntesis de la lacasa de P. flavido-alba. La inserción de los cuatro iones de cobre en las MCOs es un 102 4. Discusión fenómeno de postraducción (Taylor, 2005; Guo et al., 2005). Este hecho refuerza nuestra hipótesis de regulación postraduccional por cobre: El aumento de la actividad lacasa que se produce al añadir cobre (Fig. 20). puede deberse a la incorporación de éste a la apoproteína acumulada. Por lo tanto, la actividad lacasa dependería de la disponibilidad de cobre y de la presencia de apoproteina inactiva. En cuanto a la quelación de cobre (por adición de BCS) no se observaron cambios en la actividad lacasa de PfaL (ni en los niveles de mRNA) respecto a los del control (Fig. 20). Una posibilidad es que las células posean mecanismos de regulación controlada del cobre (evento postraduccional), en los que el metal sea transportado y esté disponible de acuerdo a las necesidades del hongo. Durante el crecimiento de levaduras con escasez de cobre, las células lo importan por transportadores de alta afinidad Ctr1p y Ctr3p en estado reducido Cu(I) (Kampfenkel et al., 1995). En condiciones normales de cobre –menos de un átomo de cobre libre por célula (Rae et al., 1999)-, éstos transportadores de membrana requieren la acción conjunta de reductasas férrico/cúpricas Fre1p y Fre2p (Dancis et al., 1992 y 1994; Georgatsou et al., 1994 y 1997). En el citoplasma las metalochaperonas del cobre facilitan su transporte a diferentes destinos (O'Halloran y Culotta, 2000). En exceso de cobre, se expresan metalotioneinas Cup1p y Crs5p, las cuales unen el cobre libre en el citoplasma (Winge et al, 1985;Jensen et al, 1996), y también se expresa la superóxido dismutasa Sod1p, la cual está involucrada en el secuestro de los radicales libres generados en las reacciones redox (Gralla et al, 1991). En S. cerevisiae existe un solapamiento entre homeóstasis del cobre y metabolismo de radicales de oxígeno (Culotta et al., 1995; Tamai et al., 1993). El gen que codifica la metalochaperona del cobre Atx1p (anti-oxidante) fue originalmene descrito como un gen que confiere protección contra estrés oxidativo (Lin y Culotta, 1995). Atx1p es una chaperona de cobre citoplasmática que une cobre y lo libera a la ATPasa tipo-P de cobre “Ccc2p” ubicada en la Red Trans de Golgi de la vía secretora (Pufahl et al., 1997). Ccc2p bombea cobre al lumen de Golgi, donde el cobre es luego insertado a enzimas dependientes de cobre que van a ser secretadas, como lacasas, o donde se activa la forma apo de la Fet3 (Fig. 23). Los factores involucrados en la homeóstasis del cobre están conservados entre eucariotas: metalochaperonas homólogas a Atx1p y a ATPasas tipo-P que 103 4. Discusión Figura 23. Relación entre la homeóstasis del cobre y del hierro en levaduras. Cu(II) y Fe(III) son substratos de la metaloreductasa Fre1p. Cu(I) entra a la célula vía Ctr1p y es transportado a post-Golgi por la chaperona Atx1p. Este Cu(I) es transferido a la ATPasa tipo-P Ccc2p, la cual lo transporta al lumen de Golgi donde activa la forma apo de Fet3p. En asociación con la permeasa de alta afinidad al hierro Ftr1p, Fet3p pasa a la membrana plasmática. Ahí, su actividad ferroxidasa absorbe Fe via Ftr1p. A las células que les falta cobre también les falta hierro debido a la ausencia de Fet3p activo. Tomado de Taylor et al (2005). 104 4. Discusión transportan cobre han sido descritas también en plantas, nemátodos, mamíferos y recientemente en basidiomicetos (Himelblau et al., 1998; Hung et al., 1997; Klomp et al., 1997; Payne y Gitlin, 1998; Wakabayashi, 1998; Uldschmid et al, 2002). En conclusión, hay una relación recíproca entre PfaL y el cobre: PfaL está relacionada en la homeóstasis del cobre, siendo este metal determinante en su actividad lacasa y ferroxidasa. 4.6.3. Regulación por hierro La regulación traduccional/postraduccional es similar a la observada con el cobre (Fig 20), es decir, que la adición del hierro aumentó la actividad lacasa (por aumento de la traducción, o por activación de la proteína o por activación de factores de la homeóstasis del Fe), mientras que los niveles de mRNA pfaL disminuyeron. Los resultados de este trabajo sugieren que, al igual que el cobre, el hierro no induce la transcripción de pfaL, pero interviene en la regulación traduccional y postraduccional de PfaL. Ello indicaría que PfaL estaría involucrada en la homeóstasis del cobre y del hierro en P. flavido-alba. De hecho, estos dos metales están interrelacionados en el metabolismo celular de eucariotas, tal como se detalló anteriormente en la homeóstasis del cobre (Fig. 23). En S. cerevisiae la ferroxidasa Fet3 se activa en el aparato de Golgi por inserción de los cuatro átomos de Cu (cedidos por dos metalochaperonas), antes de participar en el transporte de Fe en la membrana citoplasmáticas. Se sabe que la lacasa de P. flavido-alba tiene actividad ferroxidasa. Puede que su activación postraduccional por cobre también dependa del Fe a través de mecanismos específicos de la homeóstasis de Cu-Fe, que no necesariamente son comunes para diferentes organismos. Por ejemplo, Atx1p es activada transcripcionalmente por hierro en levaduras, pero por cobre en Trametes versicolor y es reprimida por Cu en Arabidospsis thaliana (Uldsmid et al., 2002). Por otro lado en P. chrysosporium se ha identificado una proteína de membrana, ortóloga de Fet3 de levaduras (Larrondo et al., 2004). PfaL y MCO1 de P. chrysosporium son proteínas extracelulares, no proteínas de membrana, por lo que difícilmente participarían en el transporte de Fe como ferroxidasas típicas. Mas bien se ha propuesto el papel detoxificador de MCO1 de P. chrysosporium, evitando la formación de radicales hidroxilo (Larrondo et al 2003): al oxidar FeII a 105 4. Discusión FeIII, el hierro presente no puede participar en la reacción de Fenton, origen de los radicales oxidantes. Sea como fuere, este análisis confirma la estrecha relación entre las MCOs de las dos especies de Phanerochaete, que como se mencionó anteriormente constituyen una nueva rama filogenética entre las multicobre oxidasas. 106 5. CONCLUSIONES 5. Conclusiones 108 5. Conclusiones 5.1. Se ha caracterizado el gen de la lacasa de P. flavido-alba (pfaL) y una región reguladora de su expresión 5.1.1. Se identificó y caracterizó un locus del gen de la lacasa de P. flavido- alba (pfaL). Su tamaño es de 2755 pb, de los cuales 1704 pb codifican un polipéptido de 567 aminoácidos. Este polipéptido contiene los seis oligopéptidos caracterizados en la lacasa pura, confirmando la autenticidad del gen. El resto son secuencias no codificantes: 19 intrones, una región reguladora UTR-3’ de 244 pb y la región 5` que incluye al promotor. 5.1.2. Junto con la secuencia génica, se ha amplificado una región “upstream”- 5` de 1590 pb que contiene un promotor tipico de eucariotas con una caja TATA y sitios potenciales de regulación propios de lacasas, como son secuencias consenso para .PRE, elementos de respuesta a metales (MRE), a antioxidantes (ARE), a xenobióticos (XRE), a nitrógeno (NIT2) y a carbono (Mig). 5.2. El gen de la lacasa de P. flavido-alba y su proteína parecen estar filogenéticamente menos emparentado con los de lacasas de basidiomicetos que con los de lacasasa de ascomicetos. Aún mas, filogenéticamente están mas próximos a los genes de multicobre oxidasas (MCOs) de ascomicetos que a sus lacasas. 5.2.1. El gen pfaL es similar en tamaño y organización de intrones a los genes multicobre oxidasas de P. chrysosporium: pfaL tiene la misma distribución y número de intrones que mco1B; es destacable especialmente la coincidencia en las secuencias que codifican los aminoácidos de unión al cobre. 5.2.2 Los intrones de pfaL poseen sitios conservados de corte/unión, (donador y aceptor), típicos de hongos. Las secuencias internas de los intrones de pfaL tienen poca identidad con la de los genes mco de P. chrysosporium. 109 5. Conclusiones 5.2.3 La secuencia deducida para PfaL presenta un 59% de identidad para las proteínas deducidas MCO4A y MCO4B de P. chrysosporium. 5.2.4. PfaL es evolutivamente cercana a las MCOs de P.chrysosporium, y junto a dos lacasa de C. neoformans (basidiomiceto) y una ferroxidasa de Y. lipolytica (ascomiceto) forman una rama independiente de las multicobre oxidasas, separada de ferroxidasas y lacasas. 5.3. La secuencia deducida para la lacasa de P. flavido-alba conserva posiciones características de lacasas y de ferroxidasas de ascomicetos. 5.3.1. Como lacasa típica, PFAL posee los residuos de unión al cobre tipo1 mononuclear y tipo 2/3 trinuclear distribuidos en las regiones conservadas L1-L4. 5.3.2. A diferencia de las lacasas de ascomicetos, PfaL conserva el Glu equivalente al Glu-185 de la ferroxidasa Fet3 de S. cerevisiae, que se ha implicado en la unión al FeII. Ni las lacasas de ascomicetos, ni PfaL, ni las MCOs de P.. chrysosporium consevan la Tyr equivalente a la Tyr-354 que también se une al FeII en Fet 3 de S. cerevisiae. Sin embargo, a diferencia de las lacasas, PfaL y MCO1B conservan esta Tyr, dos posiciones mas adelante (Tyr-413 en Pfal y Tyr-398 en MCO1). La presencia de estos aminoácidos de unión al hierro justificarían la actividad ferroxidasa de PfaL y de MCO1 de P. chrysosporium y se correlacionan con el análisis filogenético obtenido. 5.4. PfaL se sintetizaría como una preproteína que requiere al menos dos procesamientos proteolíticos para su secreción. 5.4.1. Probablemente, se sintetiza como una pre-proteína con una secuencia señal de secreción, (de extensión poco común, 43 aminoácidos) seguida de la proteína madura de 524 aminoácidos. 5.4.2. Los resultados sugieren que la pre-proteína PfaL sufriría dos procesamientos proteolíticos en el extremo N-terminal, poco frecuente en las lacasas de 110 5. Conclusiones basidiomicetos. Además, PfaL sufririá mayor glicosilación que el común de estas lacasas. 5.5. La regulación de la producción de la lacasa de P. flavido-alba es diferente a la aceptada para las lacasas de los basidiomicetos. En presencia de inductores no hay correlación entre actividad extracelular y cantidad de mRNA intracelular. Probablemente el cobre y el hierro regulan la producción de la lacasa a nivel traduccional/postraduccional. 5.5.1. La complejidad de la regulación de la expresión de pfaL (transcripcional, traduccional y/o postraduccional) puede justificar la falta de correlación entre actividad lacasa y cantidad de transcrito observada en cultivos añadidos del PMA, del ácido ferúlico o de la vainillina. Esto se reflejó cuando el pigmento PMA del alpechín y el ácido ferúlico indujeron la expresión del mRNA pfaL, pero la actividad lacasa no cambió. Mientras que la vainillina indujo la traducción/postraducción del mRNA solamente, determinada en un aumento de actividad y en disminución del mRNA respecto al control sin inductor. 5.5.2. Los inductores aromáticos ensayados (PMA, ácido ferúlico., tirosol y vanillina), al igual que la dimetilformamida, podrían regular positivamente la expresión del gen pfaL, a través de alguna de las regiones reguladoras identificadas que participan en la respuesta al estrés oxidativo (XRE y ARE). Aunque los mecanismos celulares se desconocen, el mRNA pfaL se expresa diferencialmente, dependiendo del tipo de inductor adicionado: la más alta inducción de la transcripción se produce con la mezcla polimérica PMA y con la dimetilformamida, menos con el ácido ferúlico y probablemente inhibida con el tirosol. 5.5.3. En P. chrysosporium, la degradación de vainillina implica a enzimas del estrés oxidativo. La extrapolación de este sistema a P. flavido-alba, conecta la regulación de la traducción y/o postraducción de PfaL, tanto con la degradación de la vainillina como con el proceso mas general de la respuesta al estrés oxidativo. 111 5. Conclusiones 5.5.4. El cobre y el hierro parecen no inducir la transcripción del mRNA pfaL. Es más probable que la regulación sea a nivel traduccional/postraduccional, bien por aumento de la síntesis de PFAL, o por activación de la proteína o por activación de factores involucrados en los mecanismos homeostáticos del Cu /Fe. 5.5.4.1. A nivel postraduccional la actividad lacasa dependería de la disponibilidad de cobre y de la presencia de apoproteina inactiva. 5.5.4.2. A nivel traduccional, se desconocen los mecanismos de la regulación por estos metales. 5.5.4.3. PfaL carece de dominios transmembrana y por tanto no participa en el transporte de hierro a nivel de membrana, papel de la ferroxidasa Fet3 de levaduras. Sin embargo, son varias las características que la definen como ferroxidasa: la cercanía filogenética, la conservación de residuos de unión al hierro y la regulación por hierro. 112 6. BIBLIOGRAFÍA 6.Bibliografía 114 6. Bibliografía Addleman, K., Archibald, F. 1993. Kraft pulp bleaching and delignification by dikaryons and monokaryons of Trametes versicolor. Appl. Environ. Microbiol. 59: 266-273. Adler, E. 1957. Newer views of lignin formation. Tappi 40: 294-301. Aisemberg, G., Grorewold, E., Taccioli, G., Judewicz, N., Major, A. 1989. Transcript in the response of Neurospora crassa to protein synthesis inhibition by cycloheximide. Exp. Mycol. 13: 121-128. Akileswaran, L., Alic, M., Clark, E., Hornick, J., Gold, M. 1993. Isolation and transformation of uracil auxotrophs of the lignin-degrading basidiomycete Phanerochaete chrysosporium. Curr. Genet. 23: 351-356. Alcalde, M., Bulter, T., Zumarraga, M., García-Arellano, H., Mencia, M., Plou, F., Ballesteros, A. 2005. Screening mutant libraries of fungal laccases in the presence of organic solvents. J. Biomol. Screen. 10 (6): 624-31. Alexandre, G., Zhulin, I. 2000. Laccases are widespread in bacteria. Trends Biotechnol. 18: 41-42. Alic, M., Gold, M. 1985. Genetic recombination in the lignin-degrading basidiomycete Phanerochaete chrysosporium. Appl. Environ. Microbiol. 50: 27-30 Alic, M., Kornegay, J., Pribnow, D., Gold, M. 1989. Transformation by complementation of an adenine auxotroph of the lignin-degrading basidiomycete Phanerochaete chrysosporium. Appl. Environ. Microbiol. 55: 406-411. Alic, M. 1990. Mating system and DNA transformation of the lignin-degrading basidiomycete Phanerochaete chrysosporium. Diss. Abstr. Int. B 51: 3681. Alic, M., Clark, E., Kornegay, J., Gold, M. 1990. Transformation of Phanerochaete chrysosporium and Neurospora crassa with adenine biosynthetic genes from Schizophyllum commune. Curr. Genet. 17: 305-311. 115 6.Bibliografía Alic, M., Gold, M. 1991. Genetics and molecular biology of the lignin-degrading Basidiomycete Phanerochaete chrysosporium. En: Bennett, J., Lasure, L. (eds). More gene manipulations in fungi. Academic Press, New York. pp 319-341. Alic, M., Mayfield, M., Akileswaran, L., Gold, M. 1991. Homologous transformation of the lignin-degrading basidiomycete Phanerochaete chrysosporium. Curr. Genet. 19: 491-494. Alic, M., Akileswaran, L., Gold, M. 1993. Gene replacement in the lignin-degrading basidiomycete Phanerochaete chrysosporium. Gene. 136: 307-311. Altschul, S., Madden, T., Schaffer, A., Zhang, J., Zhang, Z., Miller, W., Lipman, D. 1997. Gapped BLAST and PSI-BLAST: a new generation of protein database search programs. Nucl. Acids Res. 25: 3389-3402. Antorini, M., Herpoël-Gimbert, I., Choinowski, T., Sigoillot, J-C., Asther, M., Piontek, K. 2001. Crystal structures of fully functional laccases from two ligninolytic fungi. J. Inorganic Biochem. 1: 86. Antorini, M., Herpoël-Gimbert, I., Choinowski, T., Sigoillot, J-C., Asther, M., Winterhalter, K., Piontek, K. 2002. Purification, crystallisation and X-ray difraction study of fully functional laccases from two ligninolytic fungi. Biochim. Biophys. Acta 1594 (1): 109-114. Aramayo, R., Timberlake, W. 1990. Sequence and molecular structure of the Aspergillus nidulans y A (laccase I) gene. Nucleic Acids Res. 18: 3415. Archibald, F., Paice M., Jurasek L.. 1990. Decolorization of kraft bleachery effluent chromophores by Coriolus (Trametes) versicolor. Enzyme Microb. Technol. 12: 846-853. Asada, Y., Watanabe A., Ohtsu Y., Kuwahara M.. 1995. Purification and characterization of an aryl-alcohol oxidase from the lignin-degrading basidiomycete Phanerochaete chrysosporium. Biosci. Biotech. Biochem. 59: 1339-1341. 116 6. Bibliografía Askwith, C., Kaplan, J. 1998. Site-directed mutagenesis of the yeast multicopper oxidase Fet3p. J. Biol. Chem. 273: 22415-22419. Assmann, E., Ottoboni, L., Ferraz, A., Rodríguez, J., De Mello, M. 2003. Iron-responsive gene of Phanerochaete chrysosporium isolated by differential display reverse transcription polymerase chain reaction. Environ. Microbiol. 5: 777-786. Backa, S., Gierer J., Reitberger T., Nilsson T. 1993. Hydroxyl radical activity associated with the growth of white-rot fungi. Holzforschung 47: 181-187. Bao, W., Omalley D., Whetten R., Sederoff R. 1993. A laccase associated with lignification in Loblolly Pine Xylem. Science 260: 672-674. Bartholomew, K., Dos Santos, G., Dumonceaux, T., Charles, T., Archibald, F. 2001. Genetic transformation of Trametes versicolor to phleomycin resistance with the dominant selectable marker shble. Appl. Microbiol. Biotechnol. 56: 201-204. Basumallick, L., Szilagyi, R., Zhao, Y., Shapleigh, J., Scholes, C., Solomon, E. 2003. Spectroscopic studies of the Met182Thr mutant of nitrite reductase: role of the axial ligand in the geometric and electronic structure of blue and green copper sites. J. Am. Chem. Soc. 125: 14784-14792. Ben Hamman, O., De la Rubia, T:, Martínez, J. 1997. Effect of carbon and nitrogen limitation on lignin peroxidases and manganese peroxidases production by Phanerochaete flavido-alba, J. Appl. Microbiol. 83: 751-757. Ben Hammam, O. 1998. Peroxidasas ligninolíticas de Phanerochaete flavido-alba y aplicación de este hongo al tratamiento del alpechín. Tesis doctoral, Universidad de Granada. 164 pp. Ben Hamman, O., De la Rubia T., Martínez J. 1999a. The effect of manganese on the production of Phanerochaete flavido-alba ligninolytic peroxidases in nitrogen limited cultures. FEMS Microbiol. Lett. 177: 137-142. 117 6.Bibliografía Ben Hamman, O., De la Rubia, T., Martínez, J. 1999b. Decolorization of olive oil mill wastewaters by Phanerochaete flavido-alba, Environ. Toxicol. Chem. 18: 24102415. Berka, R., Schneider, P., Golightly, E., Brown, S., Madden, M., Brown, K. M., Halkier, T., Mondorf, K., Xu, F. 1997. Characterization of the gene encoding an extracellular laccase of Myceliophthora thermophila and analysis of the recombinant enzyme expressed in Aspergillus oryzae. Appl. Environ. Microbiol. 63 (8): 3151-3157. Bertrand, G., 1985. Sur la laccase et sur le pouvoir oxydant de cette diastase. CR Acad. Sci. (Paris). 120 : 266-269. Bertrand, T., Jolivalt, C., Briozzo, P., Caminade, E., Joly, N., Madzak, C., Mougin, C. 2002. Crystal structure of a four copper laccase complexed with an arylamine: insights into substrate recognition and correlation with kinetics. Biochemistry. 41: 7325-7333. Binnington, K., Barrett F. 1988. Ultrastructural localization of phenoloxidases in cuticle and haemopoietic tissue of the blowfly Lucilia cuprina. Tissue Cell. 20: 405-419. Birch, P. 1998. Targeted differential display of abundantly expressed sequences from the basidiomycete Phanerochaete chrysosporium which contain regions coding for fungal cellulose-binding domains. Curr. Genet. 33: 70-76. Bogan, B. W., Lamar R. T. 1996. Polycyclic aromatic hydrocarbon-degrading capabilities of Phanerochaete laevis HHB-1625 and its extracellular ligninolytic enzymes. Appl. Environ. Microbiol. 62: 1597-1603. Bollag, J-M., Leonowicz A. 1984. Comparative studies of extracellular fungal laccases. Appl. Environ. Microbiol. 48: 849-854. Bollag, J-M., Shuttleworth K., Anderson D. 1988. Laccase/mediated detoxification of phenolic compounds. Appl. Environ. Microbiol. 54: 3086-3091. 118 6. Bibliografía Bonaccorsi di Patti, M., Felice, M., Camuti, A., Lania, A., Musci, G. 2000. The essential role of Glu-185 and Tyr-354 residues in the ferroxidase activity of Saccharomyces cerevisiae Fet3. FEBS Lett. 472: 283-286. Bonaccorsi di Patti, M., Paronetto, M., Dolci, V., Felice, M., Lania, A., Musci, G. 2001. Mutational analysis of the iron binding site of Saccharomyces cerevisiae ferroxidase Fet3. An in vivo study. FEBS Lett. 508: 475-478. Bonarme, P., Jeffries, T. 1990. MnII regulation of lignin peroxidases and manganesodependent peroxidases from lignin-degrading white rot fungi. Appl. Environ. Microbiol. 56: 210-217. Bourbonnais, R., Paice, M. 1990. Oxidation of non-phenolic substrates. An expanded role for laccase in lignin biodegradation. FEBS Lett. 267: 99-102. Bourbonnais, R., Paice, M., Reid, I., Lanthier, P., Yaguchi, M. 1995. Lignin oxidation by laccase isozymes from Trametes versicolor and role of the mediator 2,2'azinobis(3-ethylbenzthiazoline-6-sulfonate) in kraft lignin depolymerization. Appl. Environ. Microbiol. 61: 1876-1880. Bourbonnais, R., Paice, M., Freiermuth, B., Bodie, E., Borneman, S. 1997. Reactivities of various mediators and laccases with kraft pulp and lignin model compounds. Appl. Environ. Microbiol. 63: 4627-4632. Bradford, M. 1976. A rapid and sensitive method for the quantification of microgram quantities of protein utilizing the principle of protein-dye binding. Anal. Biochem. 72: 248-254. Broda, P., Birch, P., Brooks, P., Sims, P. 1996. Lignocellulose degradation by Phanerochaete chrysosporium gene families and gene expression for a complex process. Mol. Microbiol. 19: 923-932. Bucher, P., Bairoch, A. 1994. A generalized profile syntax for biomolecular sequence motifs and its function in automatic sequence interpretation. Proc Int Conf Intell Syst Mol Biol 2:53–61. 119 6.Bibliografía Buchman, C., Skroch, P., Welch, J., Fogel, S., Karin, M. 1989. The CUP2 gene product, regulator of yeast metallothionein expression, is a copper-activated DNA-binding protein. Mol. Cell. Biol. 9: 4091-4095. Bulter, T., Alcalde, M., Sieber, V., Meinhold, P., Schlachtbauer, C., Arnold, F. 2003. Functional expression of a fungal laccase in Saccharomyces cerevisiae by directed evolution. Appl. Environ. Microbiol. 69: 987-995. Burke, R., Cairney, J. 2002. Laccases and other polyphenol oxidases in ecto- and ericoid mycorrhizal fungi. Mycorrhiza. 12: 105-116. Call, H., Muncke, I. 1997. History, overview and applications of mediated lignolytic systems, especially laccase mediator systems (lignozyme(R)-process). J. Biotechnol. 53: 163-202. Camarero, S., Sarkar S., Ruiz-Dueñas F., Martínez M., Martínez A. 1999. Description of a versatile peroxidase involved in the natural degradation of lignin that has both manganese-peroxidase and lignin-peroxidase substrate interaction sites. J. Biol. Chem. 274: 10324-10330. Cancel, A., Orth A., Tien M. 1993. Lignin and veratryl alcohol are not inducers of the ligninolytic system of Phanerochaete chrysosporium. Appl. Environ. Microbiol. 59: 2909-2913. Capasso, R., 1997. The chemistry, biotechnology and ecotoxicology of the polyphenols naturally occurring in vegetable wastes. Curr. Topics Phytochem. 1: 145-155. Capasso, R., De Martino, A., Arienzo, M. 2002. Recovery and characterization of the metal polymeric organic fraction (polymerin) from olive oil mill waste waters. J. Agric. Food Chem. 50 (10): 2846-2855. Cassland, P., Jönsson, L. 1999. Characterization of a gene encoding Trametes versicolor laccase A and improved heterologous expression in Saccharomyces cerevisiae by decreased cultivation temperature. Appl. Microbiol. Biotechnol. 52 (3): 393-400. 120 6. Bibliografía Chen, S., Chang, H. 1985. Chemistry of lignin biodegradation. p.p. 535-555. En: T. Higuchi (ed.) "Biosynthesis and biodegradation of wood components". Academic Press, Orlando. Chen, S., Ma, D., Ge, W., Buswell, J. 2003. Induction of laccase activity in the edible straw mushroom, Volvariella volvacea. FEMS Microbiol. Lett. 218: 143-148. Chen, S., Ge, W., Buswell, J. 2004. Biochemical and molecular characterization of a laccase from the edible straw mushroom, Volvariella volvacea. Eur. J. Biochem. 271: 318-328. Choi, G., Larson, T., Nuss, D. 1992. Molecular analysis of the laccase gene from the chestnut blight fungus and selective suppression of its expression in an isogenic hypovirulent strain. Mol. Plant-Microbe Interact. 5: 119-128. Claus, H. 2003. Laccases and their occurrence in prokaryotes. Arch. Microbiol. 179: 145150. Colao, M., Garzillo, A.M., Buonocore, V., Schiesser, A., Ruzzi, M. 2003. Primary structure and transcription analysis of a laccase-encoding gene from the basidiomycete Trametes trogii. Appl Microbiol Biotechnol 63: 153–158. Coll, P., Tabernero, C., Santamaría, R., Pérez, P. 1993. Characterization and structural analysis of the laccase I gene from the newly isolated ligninolytic basidiomycete PM1 (CECT 2971). Appl. Environ. Microbiol. 59: 4129-4135. Collins, P., Kotterman M., Field J., Dobson A. 1996. Oxidation of anthracene an benzo[α]pyrene by laccases from Trametes versicolor. Appl. Environ. Microbiol. 62: 4563-4567. Collins, P., Dobson, A. 1997. Regulation of laccase gene transcription in Trametes versicolor. Appl. Environ. Microbiol. 63: 3444-3450. Constam, D., Muheim A., Zimmermann W., Fiechter A. 1991. Purification and parcial characterization of an intracellular NADH: quinone oxidoreductase from Phanerochaete chrysosporium. J. Gen. Microbiol 137: 2209-2214. 121 6.Bibliografía Cubero, B., Scazzochio, C. 1994. Two different, adjacent and divergent zinc finger binding sites are necessary for Cre A-mediated carbon catabolite repression in the proline gene cluster of Aspergillus nidulans. EMBO Journal. 13: 407-415. Cullen, D., Kersten, P. 1996. Enzymology and molecular biology of lignin degradation. En: Bramble R, Marzluf G (eds) The Mycota III. Springer, Berlin Heidelberg New York. pp 297-314. Cullen, D. 1997. Recent advances on the molecular genetics of ligninolytic fungi. J. Biotechnol. 53: 273-289. Culotta, V., Joh, H., Lin, S., Slekar, K., Strain, J. 1995. Physiological role for Saccharomyces cerevisiae copper zinc superoxide dismutase in copper buffering. J. Biol. Chem. 270: 29991-29997. Dancis, A., Roman, D., Anderson, G., Hinnebusch, A., Klausner, R. 1992. Ferric reductase of Saccharomyces cerevisiae: molecular characterization, role in iron uptake, and transcriptional control by iron. Proc. Natl. Acad. Sci. 89: 3869-3873. Dancis, A., Yuan, D., Haile, D., Askwith, C., Eide, D., Moehle, C., Kaplan, J., Klausner, R. 1994. Molecular characterization of a copper transport protein in S. cerevisiae: an unexpected role for copper in iron transport. Cell. 76: 393-402. Daniel, G., Nilsson, T., Pettersson, B. 1989. Intra and extracellular localization of lignin peroxidase during the degradation of solid wood and wood fragments by Phanerochaete chrysosporium by using transmission electron microscopy and immuno-gold labeling. Appl. Environ. Microbiol. 55: 871-881. Davin, L., Wang, H., Crowell, A., Bedgar, D., Martin, D., Sarkanen S., Lewis, N. 1997. Steroselectiva biomolecular phenoxy radical coupling by an auxiliary (dirigent) protein without an active center. Science 275: 362-366. Dedeyan, B., Klonowska, A., Tagger, S., Tron, T., Iacazio, G., Gil, G., Le Petit, J., 2000. Biochemical and molecular characterization of a laccase from Marasmius quercophilus. Appl. Environ. Microbiol. 66: 925–929. 122 6. Bibliografía De Jong, E., De Vries F., Field, J., Zwan, R., De Bont J. 1992. Isolation and screening of basidiomycetes with high peroxidative activity. Mycol. Res. 96: 1098-1104. De La Rubia, T., Ruiz, E., Pérez, J., Martínez, J. 2002. Properties of a laccase produced by Phanerochaete flavido-alba induced by vanillin. Arch. Microbiol. 179: 70-73. Diamantidis, G., Effosse, A., Potier, P., Bally, R. 2000. Purification and characterization of the first bacterial laccase in the rhizospheric bacterium Azospirillum lipoferum. Soil Biol. Biochem. 32: 919-927. Dittmer, J., Patel N., Dhawale S., Dhawale, S. 1997. Production of multiple laccase isoforms by Phanerochaete chrysosporium grown under nutrient sufficiency. FEMS Microbiol. Lett. 149: 65-70. Dittmer, N., Suderman, R., Jiang, H., Zhu, Y., Gorman, M., Kramer, K. 2004. Characterization of cDNAs encoding putative laccase-like multicopper oxidases and developmental expression in the tobacco hornworm, Manduca sexta, and the malaria mosquito, Anopheles gambiae. Insect Biochem. Mol. Biol. 34: 29-41. Dosoretz, C., Grethelein, H. 1991. Physiological aspects of the regulation of extracellular enzymes of Phanerochaete chrysosporium. Appl. Biochem. Biotech. 28: 253-265. D’Souza, T., Dass, S., Rasooly, A., Reddy, C. 1993. Electrophoretic karyotyping of the lignin-degrading basidiomycete Phanerochaete chrysosporium. Mol. Microbiol. 8: 803-807. D’Souza, T., Boominathan, K., Reddy, C. 1996. Isolation of laccase gene-specific sequences from white rot and brown rot fungi by PCR. Appl. Environ. Microbiol. 62: 3739-3744. Ducros, V., Davies, G., Lawson, D., Wilson, K., Brown, S., Ostergaard, P., Pedersen, A., Schneider, P., Yaver, D., Brzozowski, A. 1997. Crystallization and preliminary Xray analysis of the laccase from Coprinus cinereus. Acta Crystallogr. D.: Biol. Crystallogr. 53: 605-607. 123 6.Bibliografía Ducros, V., Brzozowski, A., Wilson, K., Brown, S., Ostergaard, P., Schneider, P., Yaver, D., Pedersen, A., Davies, G. 1998. Crystal structure of the type-2 Cu depleted laccase from Coprinus cinereus at 2.2 A resolution. Nat. Struct. Biol. 5: 310-316. Ducros, V., Brzozowski, A., Wilson, K., Ostergaard, P., Schneider, P., Svendson, A., Davies, G. 2001. Structure of the laccase from Coprinus cinereus at 1.68 A resolution: evidence for different type 2 Cu-depleted isoforms. Acta Crystallogr. D. Biol. Crystallogr. 57: 333-336. Dumonceaux, T., Bartholomew, K., Valeanu, L., Charles, T., Archibald, F. 2001. Cellobiose dehydrogenase is essential for wood invasion and nonessential for kraft pulp delignification by Trametes versicolor. Enzyme Microb. Technol. 29: 478489. Eggert, C., Temp U., Dean J., Eriksson K. 1996a. A fungal metabolite mediates degradation of non-phenolic lignin structures and synthetic lignin by laccase. FEBS Lett. 391: 144-148. Eggert, C., Temp, U., Eriksson, K. 1996b. The ligninolytic system of the white rot fungus Pycnoporus cinnabarinus: purification and characterization of the laccase. Appl. Environ. Microbiol. 62: 1151-1158. Eggert, C., Temp, U. Eriksson, K. 1997. Laccase is essential for lignin degradation by the white-rot fungus Pycnoporus cinnabarinus. FEBS Lett. 407: 89-92. Eggert, C., LaFayette, P., Temp, U., Eriksson, K., Dean, J. 1998. Molecular analysis of a laccase gene from the white rot fungus Pycnoporus cinnabarinus. Appl. Environ. Microbiol. 64: 1766-1772. Eichlerova-Volakova, I., Homolka, L. 1997. Variability of ligninolytic enzyme activities in basidiospore isolates of the fungus Pleurotus ostreatus in comparison with that of protoplast-derived isolates. Folia Microbiol. 42: 583-588. Eichlerova, I., Homolka, L. 1999. Preparation and crossing of basidiospore derived monokaryons a useful tool for obtaining laccase and other ligninolytic enzyme higher-producing dikaryotic strains of Pleurotus ostreatus. Antonie Van Leeuwenhoek. 75: 321-327. 124 6. Bibliografía Faison, B., Kirk T. 1985. Factors involved in the regulation of a ligninase activity in Phanerochaete chrysosporium. Appl. Environ. Microbiol. 49: 299-304. Farnet, A., Criquet, S., Tagger, S., Gil, G., Le Petit, J. 2000. Purification, partial characterization, and reactivity with aromatic compounds of two laccases from Marasmius quercophilus strain-17. Can. J. Microbiol. 46: 189-194. Fiestas Ros de Ursinos, J., 1981. Differentes utilisations des margines. En: Proc. Seminaire international sur la valorisation des sous produits de l`olivier. Organisation des Nations Unies pour l`alimentation et l`agriculture. Tunisie. pp. 93-110. Forss, K., Framer, K., Stenlund, B. 1966. Spruce lignin and its reactions in sulfite cooking. I. The structure of lignin. Paperi Puu. 48: 565-574. Freeman, R. 1989. Protein disulfide isomerasa: Multiple roles in the modification of nascent secretory proteins. Cell. 57: 1069-1072. Fujimura-Kamada, K., Nouvet, F. J, Michaelis, S. 1997. Novel membrane-associated metalloprotease, Ste24p, is required for the first step of NH2-terminal processing of the yeast a-factor precursor. J. Cell. Biol. 136 (2): 271-85. Galagan, J., et al. 2003. The genome sequence of the filamentous fungus Neurospora crassa. Nature. 422: 859-868. Galhaup, C., Goller, S., Peterbauer, C., Strauss, J., Haltrich, D. 2002. Characterization of the major laccase isoenzyme from Trametes pubescens and regulation of its synthesis by metal ions. Microbiology. 148: 2159-2169. Gaskell, J., Dieperink, E., Cullen, D. 1991. Genomic organization of lignin peroxidase genes of Phanerochaete chrysosporium. Nucleic Acids Res. 19: 599-603. Gaskell, J., Stewart, P., Kersten, P., Covert, S., Reiser, J., Cullen, D. 1994. Establishment of genetic linkage by allele-specific polymerase chain reaction: application to the lignin peroxidase gene family of Phanerochaete chrysosporium. Bio/Technology. 12: 1372-1375. 125 6.Bibliografía Gavel, V., Von Heijne, G. 1990. Sequence differences between glycosylated and nonglycosilated Asn-X-Thr/Ser aceptor sites: implications for protein engineering. Protein Eng. 3: 433-442. Gelo-Pujic, M., Kim, H., Butlin, N., Palmore, G. 1999. Electrochemical studies of a truncated laccase produced in Pichia pastoris. Appl. Environ. Microbiol. 65 (12): 5515-5521. Georgatsou, E., Alexandraki, D. 1994. Two distinctly regulated genes are required for ferric reduction, the first step of iron uptake in Saccharomyces cerevisiae. Mol. Cell. Biol. 14: 3065-3073. Georgatsou, E., Mavrogiannis, L., Fragiadakis, G., Alexandraki, D. 1997. The yeast Fre1p/Fre2p cupric reductases facilitate copper uptake and are regulated by the copper-modulated Mac1p activator. J. Biol. Chem. 272: 13786-13792. German, U., Müller, G., Hunziker, G., Lerch, K. 1988. Characterization of two allelic forms of Neurospora crassa laccase. J. Biol. Chem. 263: 885-896. Gessner, M., Raeder, U. 1994. A histone promoter for expression of a phleomycinresistance gene in Phanerochaete chrysosporium. Gene. 142: 237-241. Gettemy, J., Li, D., Alic, M., Gold, M. 1997. Truncated-gene reporter system for studying the regulation of manganese peroxidase expression. Curr. Genet. 31: 519-524. Gianfreda, L., Xu, F., Bollag, J-M. 1999. Laccases: a useful group of oxidoreductive enzymes. Bioremed. J. 3: 1-25. Giardina, P., Cannio, R., Martirani, L., Marzullo, L., Palmieri, G., Sannia, G. 1995. Cloning and sequencing of a laccase gene from the lignin-degrading basidiomycete Pleurotus ostreatus. Applied and Environmental Microbiology. 61: 2408-2413. Giardina, P., Palmieri, G., Scaloni, A., Fontanella, B., Faraco, V., Cennamo, G., Sannia, G. 1999. Protein and gene structure of a blue laccase from Pleurotus ostreatus. Biochem. J. 341: 655-663. 126 6. Bibliografía Glenn, J., Morgan M., Mayfield M., Kuwahara M., Gold M. 1983. An extracellular H2O2requiring enzyme preparation involved in lignin biodegradation by the white-rot basidiomycete Phanerochaete chrysosporium. Biochem. Biophys. Res. Commun. 14: 1077-1083. Gold, M., Alic, M. 1993. Molecular biology of the lignin degrading basidiomycete Phanerochaete chrysosporium. Microbiological Reviews. 57: 605-622. Gold, M.H., Cheng, T.M., Mayfield, M.B. 1982. Isolation and complementation studies of auxotrophic mutants of the lignin-degrading basidiomycete Phanerochaete chrysosporium. Appl Environ Microbiol, 44:996-1000. Gold, M., Wariishi, H., Valli, K. 1989. Extracellular peroxidases involved in lignin degradation by the white rot basidiomycete Phanerochaete chrysosporium ACS Symp. Ser. 389: 127-140. González R., Ramón, D., Pérez González, J. 1992. Cloning, sequencing, analisis and yeast expression of the egl1 gene from Trichoderma longibranchiatum. Appl. Microbiol. Biotechnol. 38: 370-378. González, T., Terrón, M.C., Zapico, E., Yagûe, S., Téllez, A., Junca, H., González, A.E., 2003. Identification of a new laccase gene and con.rmation of genomic predictions by cDNA sequences of Trametes sp. I-62 laccase family. Mycol. Res. 107: 727– 735. Gralla, E., Thiele, D., Silar, P., Valentine, J. 1991. ACE1, a copper dependent transcription factor, activates expression of the yeast copper, zinc superoxide dismutase gene. Proc. Natl. Acad. Sci. 88: 8558-8562. Guillen, F., Martínez, M., Muñoz, C., Martínez, A. 1997. Quinone redox in the ligninolytic fungus Pleurotus eryngii leading to extracellular production of superoxide anion radical. Arch. Biochem. Biophys. 339: 190-199. Guillen, F., Muñoz, C., Gómez, V., Martínez, A., Martínez, M. 2000. Oxygen activation during the oxidation of methoxyhydroquinones by laccase from Pleurotus eryngii Appl. Environ. Microbiol. 66: 170-175. 127 6.Bibliografía Guo, M., Lu, F., Du, L., Pu, J., Bai, D. 2005. Optimization of the expression of a laccase gene from Trametes versicolor in Pichia methanolica. Appl. Microbiol. Biotechnol. 15: 1-5 Haber, F., Weiss, J. 1934. The catalytic decomposition of hydrogen peroxide by iron salts. Proc. R. Soc. Lond. (A) 147: 332-351. Hakulinen, N., Kiiskinen, L., Kruus, K., Saloheimo, M., Paananen, A., Koivula, A., Rouvinen, J. 2002. Crystal structure of a laccase from Melanocarpus albomyces with an intact trinuclear copper site. Nat. Struct. Biol. 9: 601-605. Hamer, D. 1995. Metallothionein. Annual Review of Biochemistry. 55: 913-951. Hammel, K., Tardone P., Moen M., Price J. A. 1989. Biomimetic oxidation of nonphenolic lignin models by Mn(II): New observations on the oxidability of guaiacyl and syringyl substructures. Arch. Biochem. Biophys. 270: 404-409. Hatakka, A., Lundell T., Tervilawilo A., Brunow, G. 1991. Metabolism of non-phenolic βO-4 lignin model compounds by the white-rot fungus Phlebia radiata. Appl. Microbiol. Biotechnol. 36: 270-277. Hatakka, A. 1994. Lignin-modifying enzymes from selected white-rot fungi: production and role in lignin degradation. FEMS Microbiol. Rev. 13: 125-135. Hatamoto, O., Sekine, H., Nakano, E., Abe, K. 1999. Cloning and expression of a cDNA encoding the laccase from Schizophyllum commune. Bioscience, Biotechnology, and Biochemistry. 63 (1): 58-64. Haylock, R., Liwicki, R., Broda, P. 1985. The isolation of mRNA from the basidiomycete fungi Phanerochaete chrysosporium and Coprinus cinereus and its in vitro translation. Appl. Environ. Microbiol. 46: 260-263. Heinfling, A., Martínez M., Martínez A., Bergbauer M., Szewzyk U. 1998. Purification and characterization of peroxidases from the dye-decolorizing fungus Bjerkandera adusta. FEMS Microbiol. Lett. 165: 43-50. 128 6. Bibliografía Higuchi, T. 1990. Lignin biochemistry: biosynthesis and biodegradation. Wood. Sci. Technol 24: 23-63. Higuchi, T. 2004. Microbial degradation of lignin: Role of lignin peroxidase, manganese peroxidase, and laccase. Proceedings of the Japan Academy, Series B: Physical and Biological Sciences, 80(5): 204-214. Himelblau, E., Mira, H., Lin, S., Culotta, V., Penarrubia, L., Amasino, R. 1998. Identification of a functional homolog of the yeast copper homeostasis gene atx1 from Arabidopsis. Plant Physiol. 117: 1227-1234. Hiratsuka, N., Wariishi, H., Tanaka, H. 2001. Degradation of diphenyl ether herbicides by the lignin-degrading basidiomycete Coriolus versicolor. Appl. Microbiol. Biotechnol. 57: 563-571. Hoegger, P., Navarro-González, M., Kilaru, S., Hoffmann, M., Westbrook, E., Kues, U. 2004. The laccase gene family in Coprinopsis cinerea (Coprinus cinereus). Current genetics. 45 (1): 9-18. Hofmann, K., Bucher, P., Falquet, L., Bairoch, A. 1999. The PROSITE database, its status in 1999. Nucleic Acids Res 27:215–219. Honda, Y., Matsuyama, T., Irie, T., Watanabe, T., Kuwahara, M. 2000. Carboxin resistance transformation of the homobasidiomycete fungus Pleurotus ostreatus. Curr. Genet. 37: 209-212. Hullo, M-F., Moszer, I., Danchin, A., Martin-Verstraete, I. 2001. CotA of Bacillus subtilis is a copper-dependent enzyme. Bacteriol. 183: 5426-5430. Hung, I., Suzuki, M., Yamaguchi, Y., Yuan, D., Klausner, R., Gitlin, J. 1997. Biochemical characterization of the Wilson disease protein and functional expression in the yeast Saccharomyces cerevisiae. J. Biol. Chem. 272: 21461-21466. Ichinose, H., Wariishi, H., Tanaka, H. 2002a. Complete degradation of Yperite, a chemical warfare agent, by basidiomycetes. Appl. Microbiol. Biotechnol. 58: 97-105. 129 6.Bibliografía Ichinose, H., Wariishi, H., Tanaka, H. 2002b. Identification and heterologous expression of the cytochrome P450 oxidoreductase from the white-rot basidiomycete Coriolus versicolor. Appl. Microbiol. Biotechnol. 59: 658-664. Iimura, Y., Takenouchi, K., Nakamura, M., Kawai, S., Katayama, Y., Morohoshi, N. 1992. Cloning and sequenc analysis of laccase genes and its use for an expression vector in Coriolus versicolor. En: Proc. 5th Int. Conf. Biotechnol. Pulp Paper Ind. pp. 145-146. Kyoto, Japan. Imbert, J., Culotta, V., Furst, P., Gedamu, L., Hammer, D. 1990. Regulation of metallothionein gene transcription by metals. En: Metal-ion induced regulation of gene expression ed. Eichorn, G. L., Marzilli, L.G. pp. 139-164, Vol. 8. New York, NY: Elsevier Science Publishing Inc. Irie, T., Honda, Y., Watanabe, T., Kuwahara, M. 2001. Efficient transformation of filamentous fungus Pleurotus ostreatus using single-strand carrier DNA. Appl. Microbiol. Biotechnol. 55: 563-565. Jeffries, T., Choi S., Kirk T. 1981. Nutritional regulation of lignin degradation by Phanerochaete chrysosporium. Appl. Environ. Microbiol. 42: 290-296. Jensen, L., Howard, W., Strain, J., Winge, D., Culotta, V. 1996. Enhanced effectiveness of copper ion buffering by CUP1 metallothionein compared with CRS5 metallothionein in Saccharomyces cerevisiae. J. Biol. Chem. 271: 18514-18519. Jönsson, L., Saloheimo, M., Penttila, M. 1997. Laccase from the white-rot fungus Trametes versicolor: cDNA cloning of lcc1 and expression in Pichia pastoris. Curr. Genet. 32: 425-430. Jönsson, L., Sjöström, K., Häggström, I., Nyman, P. 1995. Characterization of a laccase gene from the white-rot fungus Trametes versicolor and structural features of basidiomycete laccases. Biochim. Biophys. Acta 1251: 210-215. Kampfenkel, K., Kushnir, S., Babiychuk, E., Inze, D., Van Montagu, M. 1995. Molecular characterization of a putative Arabidopsis thaliana copper transporter and its yeast homologue. J. Biol. Chem. 270: 28479-28486. 130 6. Bibliografía Kanbi, L., Antonyuk, S., Hough, M., Hall, J., Dodd , F., Hasnain, S. 2002. Crystal structures of the Met148Leu and Ser86Asp mutants of rusticyanin from Thiobacillus ferrooxidans: insights into the structural relationship with the cupredoxins and the multi copper proteins. J. Mol. Biol. 320: 263-275. Karahanian, E., Corsini, G., Lobos, S., Vicuña, R. 1998. Structure and expression of a laccase gene from the ligninolytic basidiomycete Ceriporiopsis subvermispora. Biochim. Biophys. Acta. 1443: 65-74. Karhunen, P., Rummakko, P., Sipila, J., Brunow, G., Kilpelainen, I. 1995a. The formation of dibenzodioxocin structures by oxidative coupling. A model reaction for lignin biosynthesis. Tetrahedron Letters, 36(25): 4501-4. Karhunen, P., Rummakko, P., Sipila, J., Brunow, G. 1995b.ibenzodioxocins; a novel type of linkage in softwood lignins. Tetrahedron Letters, 36(1): 169-70. Kawai, S., Umezawa T., Higuchi T.. 1987. p-Benzoquinone monoketals, novel degradation products of β-O-4 lignin model compounds by Coriolus versicolor and lignin peroxidase of Phanerochaete chrysosporium. FEBS Lett. 210: 61-87. Kawai, S., Umezawa, T., Shimada, M., Higuchi, T. 1988. Aromatic ring cleavage of 4,6di(tert-butyl)guaiacol, a phenolic lignin model compound, by laccase of Coriolus versicolor. FEBS Lett. 236: 309-311. Kawai, S., Asukai, M., Ohya, N., Okita, K., Ito, T., Ohashi, H. 1999. Degradation of nonphenolic ß-O-4 substructure and of polymeric lignin model compounds by laccase of Coriolus versicolor in the presence of 1-hydroxyben-zotriazole. FEMS Microbiol. Lett. 170: 51-57. Kersten, P. 1990. Glyoxal oxidase of Phanerochaete chryusosporium: Its characterization and activation by lignin peroxidase. Proc. Natl. Acad. Sci. U. S. A. 87: 2936-2940. 131 6.Bibliografía Kersten, P., Cullen, D. 1993. Cloning and characterization of cDNA encoding glyoxal oxidase, a H2O2-producing enzyme from the lignin-degrading basidiomycete Phanerochaete chrysosporium. Proc. Natl. Acad. Sci. 90: 7411-7413. Kiiskinen, LL., Saloheimo, M. 2004. Molecular cloning and expression in Saccharomyces cerevisiae of a laccase gene from the ascomycete Melanocarpus albomyces. Appl. Environ. Microbiol. 70:137-144. Kim, K., Leem, Y., Choi, H. 2002a. Transformation of the medicinal basidiomycete Trametes versicolor to hygromycin B resistance by restriction enzyme mediated integration. FEMS Microbiol. Lett. 209: 273-276. Kim, S., Kim, S., Nam, M., Ha, K. 2002b. Proteome Analysis of Aniline-Induced Proteins in Acinetobacter lwoffii K24. Curr. Microbiol. 44: 61-66. Kim, S., Leem, Y., Kim, K., Choi, H. 2001. Cloning of an acidic laccase gene (clac2) from Coprinus congregatus and its expression by external pH. FEMS Microbiology Letters. 195 (2): 151-156. Kirk, T., Croan, S., Tien, M., Murtagh, K., Farrel, R. 1986. Production of multiple ligninases by Phanerochaete chrysosporium: effect of selected growth conditions and use of a mutant strain. Enzyme Microbiol. Technol. 8: 27-32. Kirk, T., Farrell, R. 1987. Enzymatic "combustion": the microbial degradation of lignin. Annu. Rev. Microbiol. 41: 465-505. Kirk, T., Schultz, E., Connors, W., Lorenz, L., Zeikus, J. 1978. Influence of culture parameters on lignin metabolism by Phanerochaete chrysosporium. Arch. Microbiol. 117: 277-285. Kishi, K., Wariishi, H., Márquez, L., Dunford, H., Gold, M. 1994. Mechanism of manganese peroxidase compound II reduction. Effect of organic acid chelators and pH. Biochemistry 33: 8694-8701. 132 6. Bibliografía Klomp, L., Lin, S., Yuan, D., Klausner, R., Culotta, V., Gitlin, J. 1997. Identification and functional expression of HAH1, a novel human gene involved in copper homeostasis. J. Biol. Chem. 272: 9221-9226. Kojima, Y., Tsukuda, Y., Kawai, Y., Tsukamoto, A., Sugiura, J., Sakaino, M., Kita, Y. 1990. Cloning, sequence analysis and expression of ligninolytic polyphenoloxidase genes of the white rot basidiomycete Coriolus hirsutus. J. Biol. Chem. 265: 1522415230. Krejci, R., Homolka, L. 1991. Genetic mapping in the lignin-degrading basidiomycete Phanerochaete chrysosporium. Appl. Environ. Microbiol. 57: 151-156. Kumar, S., Phale, P., Durani, S., Wangikar, P. 2003. Combined sequence and structure analysis of the fungal laccase family. Biotechnology And Bioengineering. 83 (4): 386-394. Kumar, S., Tamura, K., Nei, M. 2004. MEGA3: Integrated software for Molecular Evolutionary Genetics Analysis and sequence alignment. Briefings in Bioinformatics. 5: 150-163. Kupfer, D., Reece, C., Clifton, S., Roe, B., Prade, R. 1997. Multicellular ascomycetous fungal genomes contain more than 8000 genes. Fungal Genet. Biol. 21: 364-372. Kuramoto, N., Goto, E., Masamune, Y., Gion, K., Yoneda, Y. 2002. Existence of xenobiotic response element binding in Dictyostelium. Biochimica et Biophysica Acta. 1578: 1-11. Kurihara, H., Wariishi, H., Tanaka, H. 2002. Chemical stress-responsive genes from the lignin-degrading fungus Phanerochaete chrysosporium exposed to dibenzo-pdioxin. FEMS Microbiol Lett. 212: 217-220. Larraya, L., Pérez, G., Penas, M., Baars, J., Mikosch, T., Pisabarro, A., Ramírez, L. 1999. Molecular karyotype of the white rot fungus Pleurotus ostreatus. Appl. Environ. Microbiol. 65: 3413-3417. 133 6.Bibliografía Larraya, L., Pérez, G., Ritter, E., Pisabarro, A., Ramírez, L. 2000. Genetic linkage map of the edible basidiomycete Pleurotus ostreatus. Appl. Environ. Microbiol. 66: 52905300. Larraya, L., Idareta, E., Arana, D., Ritter, E., Pisabarro, A., Ramírez, L. 2002. Quantitative trait loci controlling vegetative growth rate in the edible basidiomycete Pleurotus ostreatus. Appl. Environ. Microbiol. 68: 1109-1114. Larrondo, L., Salas, L., Melo, F., Vicuña, R., Cullen, D. 2003. A novel extracellular multicopper oxidase from Phanerochaete chrysosporium with ferroxidase activity. Applied And Environmental Microbiology. 69 (10): 6257-6263. Larrondo, L., González, B., Cullen, D., Vicuña, R. 2004. Characterization of a multicopper oxidase gene cluster in Phanerochaete chrysosporium and evidence of altered splicing of the mco transcripts. Microbiology. 150: 2775-2783. Larsson, S., Cassland, P., Jönsson, L. 2001. Development of a Saccharomyces cerevisiae strain with enhanced resistance to phenolic fermentation inhibitors in lignocellulose hydrolysates by heterologous expression of laccase. Appl. Environ. Microbiol. 67 (3): 1163-1170. Leatham, G., Stahmann, M., 1981. Studies on laccase of Lentinus edodes: Specificity, localization and association with the development of fruiting bodies. J. Gen. Microbiol. 125: 147-157. Lehmann, Y., Meile, L., Teuber, M. 1996. Rubrerythrin from Clostridium perfringens: Cloning of the gene, purification of the protein, and characterization of its superoxide dismutase function. J. Bacteriol. 178: 7152-7158. Leonowicz, A., Rogalski, J., Szczodrak, J., Fiedurek, J. 1986. The possible key role of glucose oxidase in transformation of lignocellulose. En: Proc. 3rd Int. Conf. Biotechnol. Pulp Paper Ind. pp. 160-162. Stockholm, Sweden. Leonowicz, A., Matuszewska, A., Luterek, J., Ziegenhagen, D., Wojtas-Wasilewska, M., Cho, N-S, Hofrichter, M., Rogalsk J. 1999. Biodegradation of lignin by white rot fungi. Fungal Genetics and Biology. 27: 175-185. 134 6. Bibliografía Leonowicz, A., Cho, N-S., Luterek, J., Wilkolazka, A., Wojtas-Wasilewska, M., Matuszewska, A., Hofrichter, M., Wesenberg, D., Rogalski, J. 2001. Fungal laccase: properties and activity on lignin. J. Basic. Microbiol. 41: 185-227. Leontievsky, A., Myasoedova, N., Pozdnyakova, N., Golovleva, L. 1997. "Yellow" laccases of Panus tigrinus oxidizes non-phenolic substrates without electrontransfer mediators. FEBS Lett. 413: 446-448. Lewis, N., Yamamoto E. 1990. Lignin: occurrence, biogenesis and biodegradation. Ann. Rev. Plant. Physiol. Plant. Mol. Biol 41: 455-496. Lin, S., Culotta, V. 1995. The atx1 gene of Saccharomyces cerevisiae encodes a small metal homeostasis factor that protects cells against reactive oxygen toxicity. Proc Natl. Acad. Sci. 92: 3784-3788. Liu, W., Chao, Y., Liu, S., Bao, H., Qian, S. 2003. Molecular cloning and characterization of a laccase gene from the basidiomycete Fome lignosusand expression in Pichia pastoris. Appl. Microbiol. Biotechnol. 63:174–181. Liu, L., Tewari, R., Williamson, P. 1999. Laccase protects Cryptococcus neoformans from antifungal activity of alveolar macrophages. Infect. Immun. 67: 6034-6039. Livak, K., Schmittgen, T. 2001. Analysis of relative gene expression data using real-time quantitative PCR and 2-∆∆Ct method. Methods. 25: 402-408. Lucas, M., De la Rubia, T., Larrondo, L., Martínez, J. 2005. Amino acids sequence and enzymatic similitude between Phanerochaete flavido-alba laccase and P. chrysosporium MCO1 ferroxydase. 13st International Biodeterioreation and Biodegradation Symposium. Sept 2005, Madrid (Spain). Lundin, M., Nehlin, J., Ronne, H. 1994. Importance of a flanking AT-rich region in target site recognition by the GC box binding zinc finger protein MIG1. Molecular and Cellular Biology. 14: 1979-1985. Lyons, J., Newell, S., Buchan, A., Moran, M. 2003. Diversity of Ascomycete Laccase gene sequences in a southeastern US salt marsh. Microb. Ecol. 45 (3): 270-281. 135 6.Bibliografía Ma, B., Mayfield, M., Gold, M. 2001. The green fluorescent protein gene functions as a reporter of gene expression in Phanerochaete chrysosporium. Appl. Environ. Microbiol. 67: 948-955. Ma, B., Mayfield, M., Gold, M. 2003. Homologous expression of Phanerochaete chrysosporium manganese peroxidase using bialaphos resistance as a dominant selectable marker. Curr. Genet. 43: 407-414. Madrid, F., De la Rubia, T., Martínez, J. 1995. Effect of Phanerochaete flavido-alba on aromatic acids present in olive oil mill waste waters. Toxicol. Environ. Chem. 51: 161-168. Madzak, C., Otterbein, L., Chamkha, M., Moukha, S., Asther, M., Gaillardin, C., Beckerich, J-M. 2005. Heterologous production of a laccase from the basidiomycete Pycnoporus cinnabarinus in the dimorphic yeast Yarrowia lipolytica. FEMS Yeast Research 5: 635-646. Malarczyk, E., Nowak, G., Nowak, M., Kochman´ ska-Rdest, J., Fukuzumi, T., Leonowicz, A. 1995. Relations between SOD, laccase and other enzymes during the fruiting process of Pleurotus specia growing on a ligninocellulosic medium. En: Proc. 6th Intern. Conf. Biotechnol. Pulp Paper Ind. (E. Srebotnik and K. Messner, Eds). pp 641-644, Facultas- Universita¨tsverlag Vienna, Austria. Mansur, M., Súarez, T., Fernández-Larrea, J., Brizuela, M. A., González A . E. 1997. Identification of a laccase gene family in new lignin-degrading basidiomycete CECT 20197. Appl. Environ. Microbiol. 63: 2637-2646. Martínez, J., Pérez J., De la Rubia T. 1998. Olive oil mill waste water degradation by ligninolytic fungi. Recent Res. Devel. Microbiol. 2: 373-403. Martínez, M., Bockle B., Camarero S., Guillen F., Martínez A. 1996. MnP isoenzymes produced by two Pleurotus species in liquid culture and during wheat-straw solidstate fermentation. p.p. 183-196. En: Jeffries, T., Viiikari, L. (eds.) "Enzymes for pulp and paper processing”. ACS. Washington. USA. 136 6. Bibliografía Martínez-Nieto, L., Ramos-Cormenzana, A., García-Pareja, M., Garrido-Hoyos, S. 1992. Biodegradación de compuestos fenólicos del alpechín con Aspergillus terreus. Grasas y Aceites. 43: 75-81. Martirani, L., Giardina, P., Maezullo, L., Sannia, G., 1996. Reduction of phenol content and toxicity in olive oil mill waste waters with the ligninolytic fungus Pleurotus ostreatus. Water Res. 30: 1914-1918. Marzluf, G. 1997. Genetic regulation of nitrogen metabolism in the fungi. Microbiology and Molecular Biology Reviews. 61: 17-32. Masaphy, S., Levanon, D., Henis, Y., Venkateswarlu, K., Kelly, S. 1996. Evidence for cytochrome P450-mediated benzo-α-pyrene hydroxylation in the white rot fungus Phanerochaete chrysosporium FEMS Microbiol. Lett. 135: 51-55. Mayer, A., Staples, R. 2002. Laccase: new functions for an old enzyme. Phytochemistry 60: 551-565. McCormick, M., Buettner, G., Britigan, B. 1998. Endogenous superoxide dismutase levels regulate iron-dependent hydroxyl radical formation in Escherichia coli exposed to hydrogen peroxide. J. Bacteriol. 180: 622-625. Messerschmidt, A., Huber, R. 1990. The blue oxidases, ascorbate oxidase, laccase and ceruloplasmin. Modelling and structural relationships. Eur. J. Biochem. 187: 341352. Messerschmidt A. (ed). 1997. Multi-Copper Oxidases. World Scientific, Singapore. Min, K., Kim, Y., Kim, Y., Jung, H., Hah, Y. 2001. Characterization of a novel laccase produced by the wood-rotting fungus Phellinus ribis. Arch. Biochem. Biophys. 392: 279-286. Muheim, A., Leisola M. 1991. Aryl-aldehyde reductase from the white-rot-fungus Phanerochaete chrysosporium. Bioconversion Plant Raw Materials-Biotechnol. Adv. (VTT Symp. ) 122: 30-36. 137 6.Bibliografía Muñoz, C., Guillén, F., Martínez, A., Martínez, M. 1997. Laccase isoenzymes of Pleurotus eryngii. characterization, catalytic properties, and participation in activation of molecular-oxygen and Mn2+ oxidation. Appl. Environ. Microbiol. 63: 2166-2174. Murphy, M., Lindley, P., Adman E. 1997. Structural comparison of cupredoxin domains: domain recycling to construct proteins with novel functions. Protein Sci. 6: 761770. Nakamura, K., Go, N. 2005. Function and molecular evolution of multicopper blue proteins. CMLS, Cell. Mol. Life Sci. Review. www.Birkhauser.ch Nakamura, K., Kawabata, T., Yura, K., Go, N. 2003. Novel types of two-domain multicopper oxidases: possible missing links in the evolution. FEBS Lett. 553: 239-244. Nguyen, T., Sherratt, P., Pickett, C. 2003. Regulatory mechanisms controlling gene expression mediated by the antioxidant response element. Annu. Rev. Pharmacol. Toxicol. 43: 233-260. Nielsen, H., Engelbrecht, J., Brunak, S., Von Heijne, G. 1997a. Identification of prokaryotic and eukaryotic signal peptides and prediction of their cleavage sites. Protein Eng. 10: 1-6. Nielsen, H., Engelbrecht, J., Brunak, S., Von Heijne, G. 1997b. A neural network method for identification of prokaryotic and eukaryotic signal peptides and prediction of their cleavage sites. Int. J. Neural Syst. 8: 581-599. Nielsen, H., Krogh, A. 1998. Prediction of signal peptides and signal anchors by a hidden Markov model. Proc. Int. Conf. Intell. Syst. Mol. Biol. 6: 122-130. Niku-Paavola, M., Karhunen, E., Salola, P., Raunio, V. 1988. Ligninolytic enzymes of the white-rot fungus Phlebia radiata. Biochem. J. 254: 877-884. O’Halloran, T., Culotta, V. 2000. Metallochaperones, an intracellular shuttle service for metal ions. J. Biol. Chem. 275: 25057-25060. Ochman, H., Medhora, M., Garza, D., Hartl, D. 1990. Amplification of flanking sequences by inverse PCR. pp. 219-227. En: Innis, M.A.; Gelfand, D. H., Sninsky, J. J., 138 6. Bibliografía White, T. J. (Edit.). PCR Protocols: A guide to methods and applications. Academic Press, Inc., pp. 482. Orth, A., Rzhetskaya, M., Cullen, D., Tien, M. 1994. Characterization of cDNA encoding a manganese peroxidase from Phanerochaete chrysosporium: Genomic organization of lignin and manganese peroxidase-encoding genes. Gene. 148: 161165. Otterbein, L., Record, E., Longhi, S., Asther, M., Moukha, S. 2000. Molecular cloning of the cDNA encoding laccase from Pycnoporus cinnabarinus I-937 and expression in Pichia pastoris. Eur. J. Biochem. 267: 1619-1625. Palmer, A., Randall, D., Xu, F., Solomon E. 1999. Spectroscopic studies and electronic structure description of the high potential type 1 copper site in fungal laccase: insight into the effect of the axial ligand. J. Am. Chem. Soc. 121: 7138-7149. Palmieri, G., Giardina, P., Bianco, C., Fontanella, B., Sannia, G. 2000. Copper induction of laccase isoenzymes in the ligninolytic fungus Pleurotus ostreatus. Appl. Environ. Microbiol. 66: 920-924. Palmieri, G., Giardina, P., Bianco, C., Scaloni, A., Capasso, A., Sannia, G. 1997. A novel white laccase from Pleurotus ostreatus. J. Biol. Chem. 272: 31301-31307. Park, J., Wood, P., Davies, M., Gilbert, B., Whitwood, A. 1997. A kinetic and ESR investigation of iron II oxalate oxidation by hydrogen peroxide and dioxygen as a source of hydroxyl radicals. Free Rad. Res. 27: 447-458. Parkinson, N., Conyers, C., Keen, J., MacNicoll, A., Smith, I., Weaver, R. 2003. cDNAs encoding large venom proteins from the parasitoid wasp Pimpla hypochondriaca identified by random sequence analysis. Comp. Biochem. Physiol. C. Toxicol. Pharmacol. 134: 513-520. Payne, A., Gitlin, J. 1998. Functional expression of the Menkes disease protein reveals common biochemical mechanisms among the copper-transporting P-type ATPases. J. Biol. Chem. 273: 3765-3770. 139 6.Bibliografía Pease, E., Tien, M. 1991. Lignin-degrading enzymes from the filamentous fungus Phanerochaete chrysosporium. En: Dordick, J. S. (ed) Biocatalysts for industry. Plenum Press, New York. pp 115-135. Pease, E., Tien M.. 1992. Heterogeneity and regulation of manganese peroxidases from Phanerochaete chrysosporium. J. Bacteriol. 174: 3532-3540. Pérez, I., Hernández, M., Ramos Cormenzana, A., Martínez, S. 1987. Caracterización de fenoles del pigmento del alphechín y transformación por Phanerochaete chrysosporium. Grasas y Aceites. 38: 367-371. Pérez, J., Martínez, J., De la Rubia, T. 1996. Purification and partial characterization of a laccase from the white rot fungus Phanerochaete flavido-alba. Appl. Environ. Microbiol. 62: 4263-4267. Pérez, J., Saez, L., De la Rubia, T., Martínez, J. 1997. Phanerochaete flavido-alba ligninolytic activities and decolorization of partially biodepured paper mill wastes. Water Res. 31: 495-502. Pérez, J., De La Rubia, T., Ben Hamman, O., Martínez J. 1998. Phanerochaete flavido-alba Laccase induction and modification of manganese peroxidase isoenzyme pattern in decolorized olive oil mill waste waters. Applied And Environmental Microbiology. 64 (7): 2726-2729. Périé, F., Gold M. H. 1991. Manganese regulation of manganese peroxidase expression and lignin degradation by the white rot fungus Dichomitus squalens. Appl. Environ. Microbiol. 57: 2240-2245. Perry, C., Smith, M., Britnell, C., Wood, D., Thurston, C. 1993. Identification of two laccase genes in the cultivated muschroom Agaricus bisporus. J. Gen. Microbiol. 139: 1209-1218. Pickard, M., Roman, R., Tinoco, R., Vazquez-Duhalt, R. 1999. Polycyclic aromatic hydrocarbon metabolism by white rot fungi and oxidation by Coriolopsis gallica UAMH 8260 Laccase. Appl. Environ. Microbiol. 65: 3805–3809 140 6. Bibliografía Piontek, K., Antorini, M., Choinowski, T. 2002. Crystal structure of a laccase from the fungus Trametes versicolor at 1.90 A resolution containing a full complement of coppers. J. Biol. Chem. 277: 37663-37669. Podgornik, H., Stegu, M., Zibert, E., Perdih, A. 2001. Laccase production by Phanerochaete chrysosporium an artifact caused by Mn III. Lett Appl Microbiol. 32: 407-411. Potthast, A., Rosenau, T., Chen, C-L., Gratzl, J. 1995. Selective enzymatic oxidation of aromatic methyl groups to aldehydes. J. Org. Chem. 60: 4320-4321. Pratap, K., Lemley, A. 1998. Fenton electrochemical treatment of aqueous atrazine and metachlor. J. Agric. Food Chem. 46: 3285-3291. Pribnow, D., Mayfield M., Nipper V., Brown J., Gold M. 1989. Characterization of a cDNA encoding a manganese peroxidase, from the lignin-degradaing basidiomycete Phanerochaete chrysosporium. J. Biol. Chem. 264: 5036-5040. Pufahl, R., Singer, C., Peariso, K., Lin, S., Schmidt, P., Fahrni, C., Culotta, V., PennerHahn, J., O’Halloran, T. 1997. Metal ion chaperone function of the soluble Cu I receptor Atx1. Science. 278: 853-856. Rae, T., Schmidt, P., Pufahl, R., Culotta, V., O’Halloran, T. 1999. Undetectable intracellular free copper: the requirement of a copper chaperone for superoxide dismutase. Science. 284: 805-808. Raeder, U., Broda, P. 1985. Rapid preparation of DNA from filamentous fungi. Lett. Appl. Microbiol. 1: 17-20. Raeder, U., Thompson, W., Broda, P. 1989b. RFLP-based genetic map of Phanerochaete chrysosporium ME446: lignin peroxidase genes occur in clusters. Mol. Micro- boil. 3: 911-918. Randall, T., Rao, T., Reddy, C. 1989. Use of a shuttle vector for the transformation of the white-rot basidiomycete, Phanerochaete chrysosporium. Biochem. Biophys. Res. Commun. 161: 720-725. 141 6.Bibliografía Randall, T., Reddy, C., Boominathan, K. 1991. A novel extra-chromosomally maintained transformation vector for the lignin-degrading basidiomycete Phanerochaete chrysosporium. J Bacteriol. 173: 776-782. Randall, T., Reddy, C. 1992. The nature of extra-chromosomal maintenance of transforming plasmids in the filamentous basidiomycete Phanerochaete chrysosporium. Curr. Genet. 21: 255-260. Record, E., Punt, P., Chamkha, M., Labat, M., Van den Hondel, C., Asther, M. 2002. Expression of the Pycnoporus cinnabarinus laccase gene in Aspergillus niger and characterization of the recombinant enzyme. Eur. J. Biochem. 269 (2): 602-609. Reid, I., Paice, M. 1994. Biological bleaching of kraft pulps by white-rot fungi and their enzymes. FEMS Microbiol. Rev. 13: 369-375. Rieble, S., Joshi, D., Gold, M., 1994. Purification and characterization of a 1,2,4trihydroxybenzene 1,2-dioxygenase from the basidiomycete Phanerochaete chrysosporium. J. Bacteriol. 176: 4838-4844. Ríos, S., Eyzaguirre J. 1992. Conditions for selective degradation of lignin by the fungus Ganoderma australis. Appl. Microbiol. Biotechnol. 37: 667-669. Rogalski, J., Wojtas´-Wasilewska, M., Bialy, B., Luterek, J., Leonowicz, A. 1996. A study of aromatic ring cleavage enzymes in Phlebia radiata. En: Proc. 6th Intern. Conf. Biotechnol. Pulp Paper Ind. (Srebotnik, E., Messner, K. Eds). pp. 447-450, Facultas-Universitätsverlag Vienna, Austria. Ruiz, E., Pérez J., De La Rubia T., Martínez J. 1998. Hacia la caracterización de la lacasa de Phanerochaete flavido-alba. IV Congreso Nacional de Micología. Cádiz. Ruiz, J. 1999. Enzimas ligninolíticas producidas por Phanerochaete flavido-alba durante la degradación del alpechín. Tesina de Licenciatura. Universidad de Granada. Ruiz, J., De la Rubia, T., Pérez, J., Martínez, J. 2002. Efect of olive oil mill waste water on extracellular ligninolytic enzymes produced by Phanerochaete flavido-alba. FEMS Microbiology Letters. 212: 41-45. 142 6. Bibliografía Rushmore, T., Morton, M., Pickett, C. 1991. The antioxidant response element: activation by oxidative stress and identification of the DNA consensus sequence required for functional activity. Journal of Biological Chemistry. 266: 11632-11639. Rüttimann, C., Schwember E., Salas L., Cullen D., Vicuña R.. 1992. Ligninolytic enzymes of the white rot basidiomycetes Phlebia brevispora and Ceriporiopsis subvermispora. Biotechnol. Appl. Biochem. 16: 64-76. Rüttimann-Johnson, C., Cullen D., Lamar R. 1994. Manganese peroxidases of the white rot fungus Phanerochaete sordida. Appl. Environ. Microbiol. 60: 599-605. Ryden, L., Hunt, L. 1993. Evolution of protein complexity: the blue copper-containing oxidases and related proteins. J. Mol. Evol. 36: 41-66. Salas, C., Lobos S., Larrain J., Salas L., Cullen D., Vicuna R. 1995. Properties of laccase isoenzymes produced by the basidiomycete Ceriporiopsis subvermispora. Biotechnol. Appl. Biochem. 21: 323-333. Saloheimo, M., Niku-Paavola, M. 1991a. Heterologous production of a ligninolytic enzyme: expression of the Phlebia radiata laccase gene in Trichoderma reesei. Bio/Technology. 9: 987-990. Saloheimo, M., Niku-Paavola, M-L., Knowles, J. 1991b. Isolation and structural analysis of the laccase gene from the lignin degrading fungus Phlebia radiata. J. Gen. Microbiol. 137: 1537-1544. Sambrock, J., Russel, D. 2001. Molecular cloning: A laboratory manual. Cold Spring Harbor Laboratory Press, New York. Sanger, F., Nicklen, S., Coulson, A. 1977. DNA sequencing with chain-terminating inhibitors. Proc. Natl. Acad. Sci. 74 (12): 5463-5467. Sanjust, E., Pompei, R., Rinaldi, A., Ballero, M., 1991. Olive milling waste water as a medium for growth of four Pleurotus species. Appl. Microbiol. Biotechnol. 31: 223-235. 143 6.Bibliografía Sarkar, S., Martínez A., Martinez M. 1997. Biochemical and molecular characterization of a manganese peroxidase isoenzyme from Pleurotus ostreatus. Biochim. Biopohys. Acta. 1339: 23-30. Schoemaker, H., Meijer E., Leisola M., Haemmerli S., Waldner R., Sanglard D., Schmidt H. 1989. Oxidation and reduction in lignin biodegradation. p.p. 454-471. En: N. G. Lewis and M. G. Paice (eds.) "Plant cell wall polymers". American Chemical Society, Washington. Schoemaker, H., Lundell T., Hatakka A., Piontek K. 1994. The oxidation of veratryl alcohol, dimeric lignin models and lignin by lignin peroxidase - the redox cycle revisited. FEMS Microbiol. Rev. 13: 321-332. Screen, S., Bailey, A., Charnley, K., Cooper, R., Clarkson, J. 1997. Carbon regulation of the cuticle-degrading enzyme PR1 from Metarhizium anisopliae may involve a trans-acting DNA-binding protein CRR1, a functional equivalent of the Aspergillus nidulans CREA protein. Current Genetics. 31: 511-518. Seitz, L., Tang, K., Cummings, W., Zolan, M. 1996. The rad9 gene of Coprinus cinereus encodes a proline-rich protein required for meiotic chromosome condensation and synapsis. Genetics. 142: 1105–1117. Shimada, M., Higuchi T. 1991. Microbial, enzymatic and biomimetic degradation of lignin. p.p. 557-619. En: D. N. S. Hons and N. Shiraiski (eds.). "Wood and cellulosic chemistry". Marcel Dekker. N.Y. Shimizu, M., Yuda, N., Nakamura, T., Tanaka, H., Wariishi, H. 2005. Metabolic regulation at the tricarboxylic acid and glyoxylate cycles of the lignin-degrading basidiomycete Phanerochaete chrysosporium against exogenous addition of vanillin. Proteomics. 5: 3919-3931. Shin, W., Sundaran U., Cole J., Zhang M., Hedman B., Hodgson K., Solomon E. 1996. Chemical and spectroscopic definition of the peroxide level intermediate in the 144 6. Bibliografía multicopper oxidases. Relevance to the catalytic mechanism of dioxygen reduction of water. J. Am. Chem. Society 118: 3202-3215. Shirai, T., Ishida, H., Noda, J., Yamane, T., Ozaki, K., Hakamada, Y. 2001. Crystal structure of alkaline cellulase K: insight into the alkaline adaptation of an industrial enzyme. J. Mol. Biol. 310: 1079-1087. Sjöström, E. 1981. Wood chemistry. Academic Press, Inc. 223 p. Smith, M., Shnyreva, A., Wood, D., Thurston, C. 1998. Tandem organization and highly disparate expression of the two laccase genes lcc1 and lcc2 in the cultivated mushroom Agaricus bisporus. Microbiology. 144: 1063-1069. Soden, D., Dobson, A. 2001. Differential regulation of laccase gene expression in Pleurotus sajor-caju. Microbiology. 147: 1755-1763. Soden, D., Dobson, A. 2003. The use of amplified flanking region-PCR in the isolation of laccase promoter sequences from the edible fungus Pleurotus sajor-caju. Journal of Applied Microbiology. 95: 553-562. Solomon, E., Sundaram, U., Machonkin, T. 1996. Multicopper oxidases and oxygenases. Chem. Rev. 96: 2563-2605. Srinivasan, C., D'Souza T., Boominathan K., Reddy, C. 1995. Demonstration of laccase in the white rot basidiomycete Phanerochaete chrysosporium BKM-F1767. Appl. Environ. Microbiol. 61: 4274-4277. Stewart, P., Gaskell, J., Cullen, D. 2000. A homokaryotic derivative of a Phanerochaete chrysosporium strain and its use in genomic analysis of repetitive elements. Appl. Environ. Microbiol. 66: 1629-1633. Strauss, J., Mach, R., Zerlinger, S., Marcher, G., Stoffler, G., Wolschek, M., Kubicek, C. 1995. Cre1, the carbon catabolite repressor protein from Trichoderma reesei. FEBS Letters 376: 103-107. 145 6.Bibliografía Sugumaran, M., Nellaiappan, K. 1990. On the latency and nature of phenoloxidase present in the left colleterial gland of the cockroach Periplaneta americana. Arch. Insect Biochem. Physiol. 15: 165-181. Sugumaran, M., Giglio, L., Kundzicz, H., Saul, S., Semensi, V. 1992. Studies on the enzymes involved in puparial cuticle sclerotization in Drosophila melanogaster. Arch. Insect Biochem. Physiol. 19: 271-283. Sun, L., Bulter, T., Alcalde, M., Petrounia, I., Arnold, F. 2002. Modification of galactose oxidase to introduce glucose 6-oxidase activity. Chem. Bio. Chem. 8: 781-783. Sunagawa, M., Magae, Y. 2002. Transformation of the edible mushroom Pleurotus ostreatus by particle bombardment. FEMS Microbiol Lett. 211: 143-146. Sutherland, J., Selby, A., Freeman, J., Evans, F., Cerniglia, C. 1991. Metabolism of phenanthrene by Phanerochaete chrysosporium. Appl. Environ. Microbiol. 57: 3310-3316. Suzuki, T., Endo, K., Ito, M., Tsujibo, H., Miyamoto, K., Inamori, Y. 2003. A thermostable laccase from Streptomyces lavendulae REN-7: purification, characterization, nucleotide sequence, and expression. Biosci. Biotechnol. Biochem. 67: 2167-2175. Tai, D., Terazawa, M., Chen, C., Chang, H. 1990. Produkte des biologischen Abbaus von Birkenholz-Lignin. Holzforschung. 44 : 257-262. Takashima, S., Nakamura, A., Iikura, H., Masaki, H., Uozumi, J. 1996. Cloning of a gene encoding a putative carbon catabolite repressor from Trichoderma reesei. Bioscience, Biotechnology and Biochemistry. 60: 173-176. Tamai, K., Gralla, E., Ellerby, L., Valentine, J., Thiele, D. 1993. Yeast and mammalian metallothioneins functionally substitute for yeast copper-zinc superoxide dismutase. Proc. Natl. Acad. Sci. 90: 8013-8017. Taylor, A., Stoj, C., Ziegler, L., Kosman, D., Hart, P. 2005. The copper-iron connection in biology: Structure of the metallo-oxidase Fet3p. P.N.A.S. 102 (43) : 15459-15464. 146 6. Bibliografía Temp, U., Zierold, U., Eggert, C. 1999. Cloning and characterization of a second laccase gene from the lignin-degrading basidiomycete Pycnoporus cinnabarinus. Gene. 236: 169-177. Terrón, M., González, T., Carbajo, J., Yagüe, S., Arana-Cuenca, A., Téllez, A., D. W. Dobson, A., González, A. 2004. Structural close-related aromatic compounds have diferent efects on laccase activity and on lcc gene expression in the ligninolytic fungus Trametes sp. I-62. Fungal Genetics and Biology. 41: 954-962. Thompson, D., Hames, B., Reddy, A., Grethlein, H. 1998. In vitro degradation of natural insoluble lignin in aqueous media by the extracellular peroxidases of Phanerochate chrysoporium. Biotechnol. Bioeng. 57: 704-717. Thurston, C. 1994. The structure and function of fungal laccases. Microbiology. 140: 1926. Tien, M. 1987. Properties of ligninase from Phanerochaete chrysosporium and their possible applications. CRC. Crit. Rev. Microbiol. 15: 141-168. Tien, M., Kirk T. 1983. Lignin-degrading enzyme from the hymenomycete Phanerochaete chrysosporiumn Burds. Science 221: 661-663. Tien, M., Kirk T. 1988. Lignin peroxidase of Phanerochaete chrysosporiumn. Methods Enzymol. 161: 238-249. Trumbly, R. 1992. Glucose repression in the yeast Saccharomyces cerevisiae. Molecular Microbiology. 6: 15-21. Tuor, U., Winterhalter, K., Fietcher, A. 1995. Enzyme of white-rot fungi involved in lignin degradation and ecological determinants or wood decay. J. Biotechnol. 41: 1-17. Uldschmid, A., Engel, M., Dombi, R., Marbach, K. 2002. Identification and functional expression of tahA, a filamentous fungal gene involved in copper trafficking to the secretory pathway in Trametes versicolor. Microbiology. 148: 4049-4058. 147 6.Bibliografía Valderrama, B., Oliver, P., Medrano-Soto, A., Vazquez-Duhalt, R. 2003. Evolutionary and structural diversity of fungal laccases. Antonie van Leeuwenhoek, 84: 289–299. Wahleithmer, J., Xu, F., Brown, K., Brown, S., Golightly, E., Halkier, T., Kauppinen, S., Pederson, A., Schneider, P. 1995. The identification and characterization of four laccase genes from the plant pathogenic fungus Rhizoctonia solani. Curr. Genet. 29: 395-403. Wakabayashi, T., Nakamura, N., Sambongi, Y., Wada, Y., Oka, T., Futai, M. 1998. Identification of the copper chaperone, CUC- 1, in Caenorhabditis elegans: tissue specific co-expression with the copper transporting ATPase, CUA-1. FEBS Lett. 440: 141-146. Waldner, R., Leisola M., Fietcher A. 1988. Comparison of ligninolytic activities of selected white-rot fungi. Appl. Microbiol. Biotechnol. 29: 400-407. Welch, J., Fogel, S., Buchman, C., Karin, M. 1989. The CUP2 gene product regulates the expression of the CUP1 gene, coding for yeast metallothionein. EMBO J. 8: 255260. Williamson, P. 1994. Biochemical and molecular characterization of the diphenol oxidase of Cryptococcus neoformans: identification as a laccase. J. Bacteriol. 176: 656-664. Winge, D., Nielson, K., Gray, W., Hamer, D. 1985. Yeast metallothionein sequence and metal-binding properties. J. Biol. Chem. 260: 14464-14470. Worrall, J., Chet, I., Huttermann, A. 1986. Association of rhizomorph formation with laccase activity in Armillariella spp. J. Gen. Microbiol. 132: 2527-2533. Xu, F. 1996. Oxidation of phenols, anilines, and benzenethiols by fungal laccases: correlation between activity and redox potentials as well as halide inhibition. Biochem. 35: 7608-7614. Xu, F., Berka, R., Wahleithner, J., Nelson, B., Shuster, J., Brown, S. 1998. Site-directed mutations in fungal laccase: effect on redox potential, activity and pH profile. Biochem. J. 334: 63-70. 148 6. Bibliografía Xu, F., Palmer, A., Yaver, D., Berka, R., Gambetta, G., Brown, S., Solomon, E. 1999. Targeted mutations in a Trametes villosa laccase. Axial perturbations of the T1 copper. J. Biol. Chem. 274 (18): 12372-12375. Yanagisawa, S., Dennison, C. 2003. Loop-contraction mutagenesis of a type 1 copper site. J. Am. Chem. Soc. 125: 4974-4975. Yanagisawa, S., Dennison, C. 2004. Loop-contraction mutagenesis of type 1 copper sites. J. Am. Chem. Soc. 126: 15711-15719. Yanai, K., Yonekura, K., Usami, H., Hirayama, M., Kajiwara, S., Yamazaki, T., Shishido, K., Adachi, T. 1996. The integrative transformation of Pleurotus ostreatus using bialaphos resistance as a dominant selectable marker. Biosci. Biotechnol. Biochem. 60: 472-475. Yaropolov, A., Skorobogatko O., Vartanov S., Varfolomeyev S. 1994. Laccase-properties, catalytic mechanism, and applicability. Appl. Biochem. Biotech. 49: 257-280. Yaver, D., Golightly, E. 1996. Cloning and characterization of three laccase genes from the white-rot basidiomycete Trametes villosa: genomic organization of the laccase gene family. Gene. 181: 95-102. Yaver, D., Overjero, M., Xu, F., Nelson, B., Brown, K., Halkier, T., Bernauer, S., Brown, S., Kauppinen, S. 1999. Molecular characterization of laccase genes from the basidiomycete Coprinus cinereus and heterologous expression of the laccase lcc1. Appl. Environ. Microbiol. 65 (11): 4943-4948. Yaver, D., Xu, F., Golightly, E., Brown, K., Brown, S., Rey, M., Schneider, P., Halkier, T., Mondorf, K., Dalboge, H. 1996. The purification, characterization, molecular cloning and expression of two laccase genes from the white-rot basidiomycete Trametes villosa. Appl. Environ. Microbiol. 62: 834-841. Yoshida, H. 1883. Chemistry of lacquer (urushi). J. Chem. Soc. 43: 472-486. 149 6.Bibliografía Youn, H., Hah, Y., Kang, S. 1995. Role of laccase in lignin degradation by white-rot fungi. FEMS Microbiol. Lett. 132: 183-188. Zapanta, L., Hattori, T., Rzetskaya, M., Tien, M. 1998. Cloning of Phanerochaete chrysosporium leu2 by complementation of bacterial auxotrophs and transformation of fungal auxotrophs. Appl. Environ. Microbiol. 64: 2624-2629. 150