Replicación del ADN
Anuncio

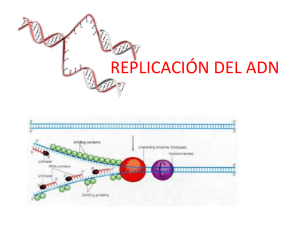
Replicación del ADN EL ácido desoxirribonucleico (ADN), contiene la información genética de todos los seres vivos. Esta información genética debe ser transmitida a la descendencia, así cuando una célula se divide necesita transmitir su información genética contenida en forma de código genético en el ADN. Este ADN tiene características y estructura bien definida que le permiten ser transmitido de manera fidedigna es decir, transmitir a las células hijas copias fieles de su material genético. Como Watson y Crick demostraron, el ADN es una doble hélice con estricta complementariedad. Permitiendo así que cada una de las dos hélices pueda servir de molde para la formación de una nueva hélice complementaria. A este mecanismo, se le conoce como replicación semi conservativa del ADN, donde cada una de las dobles hélices hijas contiene una de las dos cadenas de la hélice original. En la década de los años 60, el científico John Cairns y sus colaboradores, demostraron que la replicación del ADN es un proceso ordenado y secuencial que comienza en un punto específico en el que se forma un bucle o burbuja gracias al desenrrollamiendo de la cadena para así producir la nueva cadena. La reacción de replicación del ADN como todas las reacciones de síntesis en las células requiere la participación de proteínas no enzimáticas y de proteínas con actividad catalítica (enzimas) que regulen este proceso. I. Proteínas implicadas en la replicación del ADN 1. Definiciones de las proteínas implicadas DNA polimerasas: La función de estas enzimas es de añadir nucleótidos en dirección 5’→3’ a la nueva cadena hija que será complementaria 3’→5’ a la cadena utilizada como molde. Hay varios tipos de DNA polimerasas. En los procariotas se han aislado tres DNA polimerasas (I, II, III). En los eucariotas también, se han identificado varias pero la característica más importante de 1 todas estas enzimas ya sea en los procariotas y eucariotas es que ellas no pueden iniciar la síntesis del ADN, ellas solo pueden prolongar la síntesis es decir, se necesita de otra molécula la cual llamaremos cebador para poder iniciar la síntesis. Exonucleasas: Enzimas que cortan la cadena del ADN. Topoisomerasas: Enzimas que impiden que el ADN se superenrrolle como consecuencia de la separación de las hélices durante el proceso de replicación. Helicasa: Enzima que permite el avance de la burbuja desenrollando las dos cadenas del ADN a través de la ruptura de los puentes de hidrógeno entre las bases nitrogenadas. Proteinas SSB: Moléculas que estabilizan las cadenas sencillas del ADN evitando así el cierre de la burbuja de replicación. Primasas: Enzimas que sintetizan los cebadores (pequeñas moléculas de ARN que le indican a la ADN polimerasa donde unirse para comenzar la síntesis del ADN). DNA Ligasas: Enzimas que permiten la unión de los nucleótidos que se encuentran en fragmentos diferentes de las cadenas de ADN. II. Replicación del ADN en células procariotas La replicación del ADN es un proceso continuo, semi conservativo y complejo donde actúan un gran conjunto de enzimas y moléculas. La replicación del ADN es ligeramente diferente entre células procariotas y eucariotas. En células procariotas, el proceso de replicación lo podemos dividir en tres fases: iniciación; elongación y terminación. 1. La fase de iniciación Como su nombre lo indica, es la fase donde comienza la replicación y esta ocurre en un sitio especifico llamado el origen de la replicación (oriC), que se caracteriza por tener una secuencia de nucleótidos rica en pares de A-T 2 facilitando así la apertura de la doble hélice. La fase de iniciación comienza cuando la proteína DNA A se fija a la secuencia OriC, esta proteína reconoce secuencias específicas repetidas de 9 pares de bases de longitud dentro del tramo, una vez fijada esta proteína comienzan a fijarse más proteínas DNA A hasta formar un complejo núcleo-proteico con la finalidad de formar el bucle o burbuja, provocar la desestabilización de la doble hélice y por consecuencia la apertura, facilitando así la entrada de otras proteínas implicadas en el proceso de iniciación. Una vez que la burbuja está abierta comienzan a participar las helicasas, permitiendo así el desenrollamiento o agrandamiento de la burbuja de replicación así a medida que la burbuja de replicación se va haciendo más grande van quedando tramos de ADN de cadena sencilla y a estos tramos se van unir las proteínas SSB que tienen como función principal estabilizar los tramos de cadena sencilla evitando así la degradación de estas por la nucleasas y al mismo tiempo evitan que se reconstruya la doble hélice cerrándose con ello la propia burbuja de replicación. Luego se fija una primasa que es la encargada de sintetizar una pequeña molécula de aproximadamente 12 nucleótidos de longitud que se conoce con el nombre de cebador para indicar a la ADN polimerasa donde unirse para comenzar la síntesis del ADN, es decir la DNA polimerasa podrá añadir nucleótidos en la fase de elongación. Estas primasas y helicasas forman una unidad funcional que es necesaria para que la fase de iniciación se lleve a cabo correctamente. 2. La fase de elongación Comienza con la acción combinada de las helicasas, girasas, topoisomerasas y la DNA polimerasa. En esta fase la DNA polimerasa añade nucleótidos en dirección 5’ a 3’ mientras que recorre la cadena molde en dirección contraria es decir 3’ a 5’, la síntesis de DNA sólo puede llevarse a cabo durante el avance de la horquilla de replicación en una sola de las cadenas. 3 Figura 1. Replicación del ADN. Debido a la unidireccionalidad de la DNA polimerasa la replicación es continua en una de las cadenas y es llamada cadena conductora; nótese que es a partir de la que se sintetiza sobre el extremo 3’ libre (figura 1: cuadro verde-fragmento de color rojo) mientras que en su cadena antiparalela la síntesis es discontinua y es conocida como cadena rezagada o discontinua(figura 1: cuadro rojo), fue el científico japonés R Okasaki que demostró que las DNA polimerasas incorporan nuevos nucleótidos en dirección contraria a la de avance de la burbuja es decir, el inicio de cada fragmento debe aguardar a que la burbuja avance la longitud correspondiente para luego ir creciendo en dirección contraria a dicho avance generando unos fragmentos de DNA de 1.000 a 2.000 nucleótidos de longitud, denominados fragmentos de Okazaki. 4 Figura 2. Dímero de la holoenzima ADN Pol III durante la elongación de la replicación del ADN. La mitad del dímero de la holoenzima ADN Pol III sintetiza la hebra adelantada y la otra mitad sintetiza la hebra rezagada (Figura 2). Para iniciar la síntesis de cada fragmento de Okazaki se necesita el ARN cebador que deja un extremo 3´ libre, que es usado por la ADN polimerasa para añadir nuevos nucleótidos y sintetizar la nueva cadena y cuando la ADN polimerasa tiene contacto con otro extremo de los fragmentos de okazaki el cebador de ARN de éste es eliminado y los dos fragmentos de okazaki son unidos. Por último, la ADN ligasa liga las roturas a través de la catalización de la reacción de condensación entre el grupo fosfato y el OH de la desoxirribosa del nucleótido contiguo. 3. La fase de terminación Como en las células procariotas el cromosoma es circular, las dos burbujas de la replicación se encuentran en el extremo contrario al origen (oriC). Finalmente, la replicación termina cuando se encuentra con una secuencia específica llamada (Ter) a la que se fija una proteína que bloquea el avance de la helicasa terminando así la replicación (Figura 3). Figura 3. Fase de terminación de la replicación del ADN en los procariotas 5 III. Replicación en células eucariotas La replicación del DNA eucariota, de manera general es similar a la replicación en procariotas. En ambos casos, el proceso de replicación es semi conservativo, secuencial, bidireccional, existe una hebra conductora y una hebra rezagada y es llevada a cabo por un equipo de enzimas proteicas catalíticas y no catalíticas como en los procariotas. Sin embargo la replicación del ADN en eucariotas tienes otras particularidades: El ADN en los eucariotas son mucho más complejos y el proceso de replicación es más complicado. En los eucariotas la velocidad de desplazamiento de la burbuja de replicación es de unos 50 nucleótidos por segundo, una velocidad inferior a la de los procariotas, sin embargo para incrementar la velocidad de replicación en los eucariotas hay varios sitos de origen de replicación que se presentan en varios puntos del cromosoma, estando separados entre 30.000 y 300.000 pares de bases (Figura 4). La presencia de múltiples burbujas de replicación acelera el proceso, y permite que la replicación en eucariotas se desarrolle a velocidades mayores que en los procariotas. Figure 4. Múltiples orígenes de replicación. Cada unidad replicativa, incluyendo su origen de replicación y delimitada por las correspondientes secuencias de terminación, recibe el nombre de replicón Otra particularidad es que los fragmentos de Okazaki en el ADN eucariota son más cortos, generalmente contienen 135 nucleótidos, debido a que la horquilla de replicación se mueve más despacio. Por último, el ADN eucariota está unido a las histonas y empaquetado en los nucleosomas. El proceso de replicación ha de ir acompañado por el proceso de síntesis de histonas, ya que en cada ciclo de replicación no sólo se ha de duplicar el ADN sino también las histonas. Las histonas recién 6 sintetizadas se incorporan a la molécula de ADN que lleva la hebra retrasada, mientras que las viejas histonas permanecen en el dúplex que lleva la hebra conductora. Otra gran diferencia es que en los eucariotas el final de la replicación es un poco diferente puesto que ésta se lleva a cabo en los extremos de los cromosomas conocidos como telómeros mientras que en los procariotas como tienen cromosomas circulares carecen de extremos. En los telómeros de la hebra conductora la replicación termina sin problemas ya que las DNA polimerasas finalizan la síntesis en el lugar exacto, mientras que en la hebra rezagada la replicación queda incompleta en el extremo 5’ puesto que el cebador del último fragmento de okazaki es colocado exactamente en el extremo 5’ que es luego eliminado por la exonucleasa con actividad 5’ a 3’ por ende ninguna DNA polimerasa podrá remplazar este espacio perdiéndose así un tramo de la cadena polinucleotídica. Figura 5. Proceso final de la replicación del ADN en eucariotas Sin embargo para resolver este problema en los eucariotas existe una enzima llamada telomerasa que se une al extremo 3’que sobresale del telómero y añade nucleótidos al extremo 3’ utilizando su RNA como molde, evitando así el acortamiento de los telómeros en cada ciclo de división celular. 7 8 Test 1¿Qué es la replicación del DNA? a) Es el proceso de síntesis de RNA b) Es el proceso de síntesis de proteínas c) Es el proceso por el cual el ADN se duplica d) Es el proceso de conservación del ADN 2. ¿Cuál es la función de las DNA polimerasas? a) Anadir nucleótidos en dirección 5’ a 3’ b) Anadir nucleótidos en dirección 3’ a 5’ c) Anadir nucleótidos en ambas direcciones d) Sintetizar el ARN mensajero 3. Las DNA polimerasas en procariotas y eucariotas pueden: a) Iniciar las síntesis del ADN b) Prolongar la síntesis del ADN c) Parar las síntesis del ADN d) Transferir el ARN al citoplasma 4. El proceso por el cual el ADN se duplica es: (respuesta múltiple) a) Semiconservativo, secuencial y unidireccional b) Secuencial y unidireccional c) Semiconservativo, secuencial y bidireccional d) Secuencial y bidireccional 5. El proceso de replicación del ADN se divide en tres fases: a) Iniciación, elongación y terminación b) Elongación, iniciación y terminación c) Terminación, elongación e iniciación d) Fase I, fase 2 y fase 3 6. ¿En qué sitio específico comienza la replicación del ADN? a) En el sitio Ter b) En el sitio oriC c) En el sitio 3’ 9 d) En el sitio 5’ 7. Las primasas y helicasas forman una unidad funcional que es necesaria para: a) Que la fase de iniciación del ADN se lleve a cabo correctamente. b) El proceso de elongación del ADN c) La terminación de la replicación del ADN d) La fase de maduración 8. El proceso de replicación del ADN: a) Es bidireccional en todas las células pero solo posee múltiples sitios de origen en eucariotas b) Es bidireccional solo en eucariotas y posee un solo sitio de origen en todos los tipos celulares c) Tanto en procariotas como en eucariotas es bidireccional y posee un sitio de origen d) Es unidireccional y posee un sitio de origen 9. La ausencia de la enzima ADN ligasa afecta: a) El apareamiento de los nucleótidos b) La ruptura de los puentes de hidrógeno c) La unión de los fragmentos de Okasaki d) El apareamiento de los aminoácidos 10 La DNA polimerasa: (respuesta múltiple) a) Utiliza ribonucleótidos. b) Repara el RNA dañado c) Sintetiza DNA en el sentido 5′ a 3′ d) Siempre requiere cebador Respuestas: 1 c/2 d/3 b/4 c - d/5 a/6 a/7 a/8 a/9 c/10 c-d 10