Tema 2
Anuncio

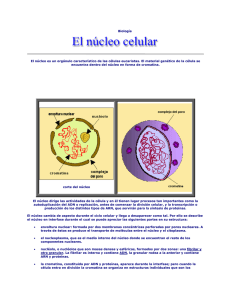
Tema 2. El Genoma Humano en acción La cromatina durante el ciclo celular. La secuencia influye en el estado funcional de la cromatina. Modificaciones epigenéticas y su importancia en la regulación del estado funcional de la cromatina. Relación entre secuencia, estructura y función de la cromatina: territorios cromosómicos. La cromatina durante el ciclo celular En las células somáticas que tienen núcleo, la molécula de ADN está presente en una forma peculiar llamada originalmente cromatina. Por la estructura de la doble hélice del ADN, sabemos que la distancia entre nucleótidos es de 0,34 nm; si el genoma humano haploide tiene 3x109 pares de bases, la longitud total del genoma en forma de doble hélice lineal sería algo superior a 1 metro, y además cada núcleo contiene dos copias del genoma. Todo este material debe entrar en el núcleo de una célula eucariota, cuyo diámetro medio es de 5 m. Esto significa que el ADN ha de adoptar un alto grado de empaquetamiento para poder alojarse dentro del núcleo. Este empaquetamiento se lleva a cabo mediante la unión de la doble hélice con varios tipos de proteínas para dar lugar a una estructura que es, precisamente, la cromatina. Las células eucariotas, al proliferar, siguen una serie de etapas en las que se llevan a cabo los procesos necesarios para dar lugar a dos células hijas: duplicar los componentes celulares, segregarlos espacialmente y dividir la célula de modo que las dos células resultantes lleven todos los ingredientes necesarios para su correcto funcionamiento. Estas etapas deben completarse en orden, de un modo altamente regulado, y constituyen lo que se llama ciclo celular. En cada ciclo celular se distinguen por tanto varias fases: la interfase (etapa en la que la célula duplica su contenido), la mitosis (etapa en la que los componentes se separan a polos opuestos de la célula) y citoquinesis (separación física de las dos células hijas). Probablemente sea la cromatina el componente celular en el que es más importante la duplicación y segregación correctas, ya que esto va a asegurar que la información genética se transmita sin alteraciones. Por tanto, es importante saber cómo se comporta la cromatina en las distintas etapas del ciclo celular. La Figura 2.1 muestra las distintas fases del ciclo celular de una célula eucariota y los cambios que sufre la cromatina. El ciclo celular de una célula eucariota se puede esquematizar como muestra la figura. La INTERFASE está constituida por las fases G1, S y G2; durante la fase S se replica la cromatina para dar lugar a dos complementos cromosómicos completos. En la MITOSIS se separan los componentes nucleares y celulares para dar lugar a dos células hijas. Durante la interfase, que es la etapa más larga del ciclo, la cromatina está sujeta a un grado de empaquetamiento de unas 2000 veces, es decir, lo que en su estado natural ocuparía un tamaño de 2000 m se reduce a un tamaño de 1 m. Esto se consigue por la unión de la doble hebra de ADN con unas proteínas básicas llamadas histonas, cuyos grupos positivos interaccionan con los grupos negativos del esqueleto fosfato del ADN. Hay 5 tipos principales de histonas, llamadas H1, H2A, H2B, H3 y H4, que se asocian entre sí para formar un octámero: dos moléculas de H3 junto con dos moléculas de H4 forman un tetrámero, y dos dímeros H2A/H2B forman otro tetrámero; ambos tetrámeros se asocian para formar un núcleo proteico (octámero) alrededor del cual se enrolla la molécula de ADN. En concreto, 146 pares de bases de ADN dan 1,65 vueltas alrededor del octámero, y sobre este complejo se une la histona H1. Esta estructura, de unos 10 nm de diámetro, es lo que se conoce con el nombre de nucleosoma, y es la unidad básica de organización de la cromatina. Los nucleosomas están unidos entre sí por el filamento de ADN que se va enrrollando a su alrededor, como bolas en una cuerda, y esto da lugar a la fibra de cromatina de 10 nm. En esta estructura, unos 200 pares de bases ocupan 10 nm, lo que significa un grado de empaquetamiento de unas 6 veces respecto al tamaño lineal que ocuparía un fragmento de ADN de esa longitud (200 X 0,34 nm = 64 nm). Figura 2.2. Esquema de un nucleosoma, con el ADN rodeando el octámero de histonas y la histona H1 en el origen del bucle. Imagen obtenida de http://themedicalbiochemistrypage.org/dna.html En condiciones fisiológicas, la fibra de 10 nm sufre un segundo grado de enrollamiento sobre sí misma para dar lugar a una estructura en forma de solenoide, con 6 nucleosomas por vuelta. Esta configuración constituye la fibra de 30 nm, en la que el grado de empaquetamiento del ADN es de unas 40 veces. La fibra de 30 nm sufre diferentes grados de empaquetamiento durante interfase y, especialmente, en la mitosis, en la que la cromatina alcanza su empaquetamiento máximo (unas 10.000 veces) y da lugar a las estructuras visibles que llamamos cromosomas. Estos tipos de alto grado de enrollamiento se consiguen porque la fibra de 30 nm forma asas que se unen por su base a una estructura proteica que sirve como andamio. El andamio ("scaffold" en inglés) está constituido por proteínas no histonas, de las que las principales son la Sc1 (idéntica a la topoisomerasa II) y la Sc2 ó SMC2, que pertenece a una familia de proteínas llamada SMC (Structural Maintenance of Chromosomes, en inglés). Estas proteínas cumplen también un papel importante en el mantenimiento de la condensación de la cromatina (de ahí que también se les llame condensinas). Las asas de la fibra de 30 nm se unen al andamio mediante unas secuencias ricas en Adeninas y Timinas llamadas SAR (Scaffold Attachment Region en inglés), que tienen gran afinidad por las proteínas del andamio. La estructura que resulta de este enrollamiento da lugar a una fibra de unos 300 nm de grosor, en la que el grado total de empaquetamiento del ADN es de unas 2.000 veces. Las SAR son regiones importantes, dispersas por el genoma, que flanquean genes y a menudo se asocian con los orígenes de replicación que veremos más adelante, por lo que se piensa que pueden jugar un papel importante en la función y estructura de la cromatina. Finalmente, el empaquetamiento máximo de la cromatina durante la mitosis se consigue al espiralizarse la fibra de 300 nm para dar lugar a una estructura de 600 nm de grosor (una cromátide) en la que el ADN alcanza ya un grado de empaquetamiento de 10.000 veces. Figura 2.3 video que muestra los distintos grados de empaquetamiento de la cromatina. La secuencia influye en el estado funcional de la cromatina Como ya se ha dicho, el grado de empaquetamiento no es uniforme a lo largo de todo el genoma. De hecho, en el núcleo en interfase siempre se distinguió una cromatina denominada eucromatina, poco condensada, y otra llamada heterocromatina, con un mayor grado de condensación. Hoy sabemos que estas categorías morfológicas tienen un correlato funcional, ya que la eucromatina es transcripcionalmente más activa mientras que los genes incluidos en heterocromatina tienen un bajo nivel de expresión. La heterocromatina, a su vez, puede ser de dos tipos: constitutiva, que nunca se transcribe y se localiza en los centrómeros y en la constricción secundaria de algunos cromosomas; ó facultativa, que en el fondo es un tipo de eucromatina que se heterocromatiniza en situaciones concretas, como es el caso del cromosoma X inactivo en las mujeres. Además de estas dos grandes categorías de cromatina, es importante comprender que dentro de las regiones eucromáticas la cromatina tampoco es totalmente homogénea, como queda reflejado, por ejemplo, en su distinta repuesta a varias tinciones. Este comportamiento es precisamente la base del patrón de bandas característico de cada cromosoma y que permite la creación del cariotipo convencional. Como veremos en un capítulo posterior, la tinción con un colorante llamado Giemsa produce unas bandas oscuras (bandas G) separadas por bandas claras (bandas R). La tinción con otra sustancia llamada Quinacrina (que se une específicamente a regiones ricas en adeninas y timinas) produce un patrón de bandas similar a las bandas G, mientras que la tinción con Cromomicina-A (que se une preferentemente a regiones ricas en guaninas y citosinas) genera un patrón similar a las bandas R. Por tanto, las peculiaridades morfológicas de la cromatina son debidas, al menos en parte, a diferencias en la secuencia de nucleótidos del genoma; el modelo del andamio (scaffold) que vimos en el punto anterior permite explicar esta relación, ya que las bandas G representan regiones con un mayor grado de empaquetamiento del ADN, lo que implica una mayor densidad de las regiones de unión al andamio (llamadas SAR). Como las SAR son relativamente ricas en adenina y timina, esto explica que estas regiones genómicas se tiñan más intensamente por Quinacrina y Giemsa, mientras las bandas R, más ricas en guanina y citosina, tienen un menor grado de empaquetamiento, menor densidad de SAR y se tiñen débilmente por estos colorantes. Por tanto, las distintas bandas del cariotipo reflejan no sólo diferencias en la condensación de la cromatina, sino también diferencias en su composición: ya hemos visto que el genoma humano tiene un contenido medio en G+C del 41%, pero que esto no se distribuye de manera uniforme por todo el genoma. A su vez, las diferencias en condensación y en composición de las distintas regiones de cromatina se correlacionan con otra variable importante: la riqueza en genes y su actividad transcripcional. Por ejemplo, se estima que un 80% de los genes se localizan en bandas R (las más ricas en nucleótidos G+C), que son las menos condensadas. También se ha observado que, en general, las bandas R son de replicación temprana, mientras que las bandas G son de replicación más tardía. La replicación del ADN en eucariotas comienza en muchos orígenes de replicación durante la fase S del ciclo celular, pero no todos los orígenes de replicación comienzan a funcionar a la vez: hay unos orígenes que siempre comienzan al principio de la fase S (replicación temprana) y otros que van comenzando a funcionar más tarde (replicación tardía). Asimismo, se sabe que los genes constitutivos (housekeeping en inglés), que son los que se expresan en todos los tejidos, se replican temprano; por el contrario, los genes que están en regiones heterocromáticas (el cromosoma X inactivo, por ejemplo) son de replicación más tardía. Parece —por tanto— que regiones con alta densidad de genes, ricas en C+G y descondensadas se replican antes que regiones silenciadas ó con baja densidad de genes. Otro aspecto que diferencia los distintos tipos de bandas es la acetilación de las histonas. Como se verá a continuación, se ha podido comprobar que la acetilación de las histonas que forman los nucleosomas lleva a la formación de una cromatina más abierta, que permite mejor el acceso de factores de transcripción y la expresión de los genes contenidos en esas regiones. Esto se debe a que las colas amino-terminales de las histonas interaccionan con más fuerza entre sí y con el ADN cuando los grupos épsilon-amino de las lisinas están en su forma des-acetilada; la acetilación relaja esas interacciones y descondensa parcialmente la fibra de 30 nm y la unión del ADN con los nucleososmas. Curiosamente, se ha comprobado que las histonas de las bandas G están menos acetiladas que las histonas de las bandas R. Esto ayudaría también a explicar la menor condensación de la cromatina en las bandas R, y nos proporciona un mecanismo para entender por qué las bandas R se replican antes y son más activas transcripcionalmente: por un lado, no necesitan descondensarse para que se lleve a cabo la replicación, y por otro constituyen unos dominios en los que el ADN es más accesible a los factores de transcripción. Figura 2.4 Este video muestra el proceso de acetilación de la lisina, y las modificaciones covalentes que sufren algunas lisinas de los extremos Nterminales de las histonas H3 y H4. También se explica esquemáticamente cómo estas modificaciones contribuyen compactación de una región de cromatina. a modificar el grado de En resumen, es importante darse cuenta de que los mecanismos básicos de regulación de la expresión génica están sometidos a un nivel superior de regulación, dependiente de la situación de un gen dentro del genoma y del estado funcional de la cromatina en que se encuentra, que a su vez está influido por el tipo de secuencias que la componen. Generalizando, podemos resumir las principales características de los dos estados de cromatina más frecuentes, representados por los dos tipos principales de bandas citogenéticas (banda G, oscuras, y bandas R, claras): Densidad en genes Porcentaje de G+C Replicación Composición Bandas G Bandas R Baja Alta Bajo Alto Tardía Temprana Ricas en A+T Ricas en G+C Acetilación Histonas Baja Alta Repeticiones predominantes LINE SINE Modificaciones epigenéticas y su importancia en la regulación del estado funcional de la cromatina Es muy importante comprender que los aspectos estructurales y funcionales se integran en un modelo "dinámico", según el cual una región de cromatina estará en un estado más o menos favorable a la expresión génica dependiendo del tipo de secuencias que la forman y también de las modificaciones epigenéticas a ese nivel. Por modificaciones epigenéticas se entienden todos aquellos cambios que sufre la cromatina pero que no afectan a la secuencia de nucleótidos: por eso no son modificaciones "genéticas", sino que están "por encima" (que es lo que significa epi en griego). En los últimos años se ha comprobado experimentalmente la gran importancia que tienen los mecanismos que regulan la actividad de la cromatina mediante cambios epigenéticos. Para entender esto, es importante recordar que la actividad transcripcional basal en eucariotas es esencialmente restrictiva, en el sentido de que los promotores están en estado inactivo hasta que se ponen en marcha por la acción de los elementos llamados ―activadores‖ y el ensamblaje del complejo basal de transcripción. Para que estos factores proteicos se unan a sus dianas en el ADN es necesario que la cromatina de esa región esté relativamente descondensada, para exponer más fácilmente la doble hélice y permitir el acceso de factores proteicos. De hecho, los genes que son transcripcionalmente activos se asocian habitualmente con sitios hipersensibles a DNAsa I, regiones cortas (unos pocos cientos de pares de bases) formadas por ADN que no está asociado a nucleosomas, precisamente porque se encuentra unido a factores de transcripción. Figura 2.5 Las modificaciones de las histonas influyen en las interacciones entre nucleosomas y, por tanto, en el grado de condensación de la cromatina. En esta figura se muestra una región abierta (con nucleosomas más separados) flanqueada por regiones más condensadas. Estas regiones menos condensadas pueden reconocerse porque constituyen sitios hipersensibles a ADNasa I, ya que permiten el acceso de esta nucleasa a la molécula de ADN. También se ha visto que la unión al ADN de factores proteicos, como factores de transcripción, se ve favorecida en estas regiones. Los nucleosomas son, por tanto, un elemento fundamental para mantener este estado basal restrictivo, al impedir el acceso de la maquinaria transcripcional a los promotores. La represión transcripcional mediada por nucleosomas se debe tanto a las interacciones directas del octámero de histonas con el ADN, como a las interacciones que se originan entre nucleosomas vecinos para dar lugar a la fibra de cromatina. Como hemos visto más arriba, las colas N-terminales de las histonas de los nucleosomas quedan libres hacia fuera, y son las regiones más susceptibles de ser modificadas químicamente para variar su carga y alterar las interacciones, tanto entre histonas y ADN como entre nucleosomas vecinos. Por tanto, si conseguimos relajar alguna de estas interacciones conseguiremos favorecer la transcripción de los genes que están incluidos en esas regiones. Esta relajación se puede conseguir gracias a que las colas N-terminales de las histonas tienen una serie de aminoácidos (lisinas y serinas, principalmente) que pueden sufrir modificaciones covalentes del tipo acetilación, metilación ó fosforilación, y que van a ser cruciales en la regulación de estos fenómenos. A su vez, estos cambios se coordinan con otras modificaciones epigenéticas que sufre la propia cadena de ADN en forma de metilación de algunos nucleótidos concretos, y en su conjunto ambos procesos constituyen un importante mecanismo de regulación de la actividad transcripcional. A continuación veremos por separado las modificaciones epigenéticas que sufre la cromatina, por una parte, y el ADN por otra. A. Modificación de la cromatina. Para superar la represión basal debida a la presencia de nucleosomas y facilitar la expresión de un gen, los activadores de la transcripción pueden potenciar la actividad transcripcional alterando, en primer lugar, la propia estructura de la cromatina: aunque el activador no se una directamente al promotor de un gen, puede reclutar distintas actividades modificadoras de la cromatina cuya acción sea conferir a esa región un estado que facilite la transcripción. Estas actividades pueden ser de varios tipos: complejos proteicos implicados en el remodelamiento nucleosomal: son capaces de mover los nucleosomas de su posición para dejar expuesta una región promotora y permitir la expresión génica. Este tipo de actividad está mediada por complejos multiproteicos con actividad ATPasa, de los cuales el primero en ser aislado fue el complejo SWI/SNF (primero se identificó en levaduras, después en humanos). Este complejo desestabiliza el nucleosoma al romper los contactos del ADN con el octámero, que queda libre para moverse y asociarse con una región vecina de la molécula de ADN. Todos los remodeladotes de nucleosomas pertenecen a la familia SNF2 de ATPasas, y pueden dividirse en 7 subgrupos dependiendo de los dominios proteicos que contienen. Figura 2.6 Los complejos "remodeladores" de la cromatina tienen actividad ATPasa y son capaces de mover nucleosomas para descubrir regiones del ADN necesarias para la transcripción. En este video se ilustra la acción de un remodelador típico. Las cuatro familias más importantes y mejor estudiadas de estos remodeladores son SWI2, ISWI, CHD e Ino80. complejos implicados en la acetilación de histonas: el factor Gcn5 de levadura fue el primer activador transcripcional con actividad acetilasa de histonas descubierto, en 1996. Posteriormente se vio que otros co-activadores de la transcripción de mamíferos también poseían actividad acetiltransferasa, factores tales como p300, CBP (CREB-BP) y P/CAF, de ahí que hoy en conozcan en conjunto con el nombre de HAT (Histone AcetylTransferase). Éstos actúan como factores de transcripción integradores de diversas vías de transducción de señales implicadas en el control del ciclo celular y de los procesos de diferenciación, reparación y apoptosis. Otros activadores transcripcionales con actividad HAT son el factor de transcripción TAFII250 (que forma parte del complejo TFIID), SRC-1 y ACTR (coactivadores de receptores nucleares). Al igual que la acetilación de las histonas conduce a la activación transcripcional, la des-acetilación de las histonas crea una estructura cromatínica más condensada que favorece el silenciamiento de la transcripción de los genes incluidos en la esa región genómica. En este sentido, se ha comprobado que los factores HDAC1 y HDAC2 (Histone De-Acetylase), cuya función es des-acetilar las histonas, están presentes en el complejo Sin3-NcoR, un co-represor transcripcional que regula la expresión de genes importantes en el ciclo celular y en el desarrollo embrionario. En conjunto, acetilasas y desacetilasas actúan sobre los mismos aminoácidos de las colas amino-terminales de las histonas H3 y H4, especialmente las lisinas 9, 14, 18 y 23 de la histona H3 y las lisinas 5, 8, 12 y 16 de la histona H4. En la figura 2.4 ya se ha visto el posible mecanismo por el que la des-acetilación de las histonas favorece la compactación de nucleosomas vecinos e impide la transcripción en esa región. De hecho, se ha comprobado que la des-acetilación de la lisina 16 en la histona H4 provoca la condensación de la fibra de 10 nm, mientras que la acetilación de este residuo impide la formación de la fibra de 30 nm y permite la interacción con co-activadores de la transcripción. otras modificaciones muy importantes son la fosforilación de la histona H3 en la serina 10 y la metilación de las lisinas 4, 9, 27, 36 y 79 en la histona H3 y de la lisina 20 de la histona H4, metilación catalizada por unos complejos proteicos que tienen actividad metil-transferasa. Se ha comprobado que estas modificaciones actúan en coordinación con la acetilación, estableciendo lo que ahora se conoce como el "Código de Histonas". Según este código, una región se comportará como eucromatina ó como heterocromatina dependiendo de las modificaciones epigenéticas de las histonas que conforman los nucleosomas de la región; las regiones limítrofes, en las que se da una transición de un tipo de cromatina al otro, muestran características propias de ambos tipos de cromatina, y son capaces de unir complejos proteicos llamados aisladores (en inglés "insulator"), que forman una especie de barrera física e impiden que un tipo de cromatina se extienda más allá del límite de la región e invada la región vecina. De modo general, el código de histonas establece que una región de eucromatina se caracteriza por tener la lisina 4 de la histona H3 metilada (con uno, dos o tres grupos metilo), y las lisinas 9 y 14 de la misma histona acetiladas. Se ha comprobado que esta configuración tiene la propiedad de unirse a un dominio proteico llamado "bromodominio", que está presente en muchos activadores de la transcripción. Una región con estas características puede heterocromatinizarse si sufre una cascada de alteraciones: la pérdida de la metilación de la lisina 4, la desacetilación progresiva de las lisinas 9 y 14, y finalmente la metilación de la lisina 9 constituyen una marca de heterocromatina, a la que se unen proteínas que contienen un dominio de silenciamiento llamado "cromodominio" que recluta factores de silenciamiento como la proteína HP1 (proteína de heterocromatina-1). Estas interacciones están influidas también por la fosforilación de la serina 10 de la histona H3; por ejemplo, dicha fosforilación –generada por la quinasa Aurora B— inhibe la unión de HP1 con la lisina 9 metilada durante la mitosis. Figura 2.7 El "Código de histonas" permite saber el tipo de cromatina que formará una región dependiendo de las modificaciones covalentes de las histonas en esos nucleosomas. En este video se muestran las distintas modificaciones de una región de la histona H3, y se esquematiza el tipo de modificaciones típicas de regiones de heterocromatina y de eucromatina. Estudios recientes han aportado nuevos datos sobre las modificaciones que sufren las histonas. En lo que se refiere a las metilaciones, diversos estudios de inmunoprecipitación de cromatina han podido determinar la posición de dichas modificaciones a lo largo de todo el genoma. Esto ha permitido comparar el estado de la cromatina que rodea al promotor frente al de otras regiones génicas. Además, combinando estos datos con los niveles de expresión de cada gen, se ha podido definir mejor el código de metilaciones de histonas que caracteriza los promotores de genes activos y de genes silenciados. Esta información se resume en la siguiente figura, que muestra un gen activo típico (la flecha hacia la derecha indica transcripción activa a partir de ese punto). Las líneas de colores indican la intensidad de unión de anticuerpos frente a ARN polimerasa II (verde), tri-metilación de la lisina 4 de la histona H3 (azul), acetilación de las lisinas 9 y 14 de la histona H3 (marrón) y tri-metilación de la lisina 36 de la histona H3 (naranja). Puede verse que ésta última se encuentra sobre todo en los nucleosomas que incluyen el cuerpo y el final del gen, mientras que las otras modificaciones son más intensas en los nucleosomas correspondientes al sitio de inicio de la transcripción. La Figura 2.8 muestra las modificaciones de histonas de regiones génicas según la actividad transcripcional. Los genes no activos, en cambio, están caracterizados por la ausencia de estas modificaciones y la presencia de metilación de la lisina 9 de la histona H3 (H3K9me1) y la tri-metilación de la lisina 27 de la histona H3 (H3K27me3) en la zona del inicio de la transcripción, y la tri-metilación de la lisina 79 de la histona H3 (H3K79me3) a lo largo de todo el cuerpo del gen. B. La metilación del ADN juega un papel fundamental en el mantenimiento del silenciamiento transcripcional. El ADN de vertebrados se metila en el carbono 5 de las citosinas que están en el dinucleótido CpG (la "p" indica el grupo fosfato que une una citosina con una guanina, en dirección 5' 3'). Curiosamente, la abundancia de este dinucleótido en el genoma de vertebrados es sólo un 25% de lo esperado, es decir, el porcentaje normalizado de este dinucleótido es de 0,25. En efecto, si el contenido en C+G del genoma es del 41%, la probabilidad esperada de encontrar un dinucleótido CpG es (0,205 x 0,205) = 0,042 (o sea, 4,2%); en cambio, la frecuencia observada de este dinucleótido está en torno al 1% en el genoma humano, de ahí que el porcentaje normalizado sea 1/4 = 0,25. Además, los dinucleótidos CpG no están distribuidos homogéneamente a lo largo del genoma, sino que son más abundantes en los genes, tanto en los exones como, sobre todo, alrededor del inicio de la transcripción. Pues bien, los dinucleótidos CpG que tienen metilada la citosina son los que están distribuidos a lo largo de la secuencia de genes. Por el contrario, los dinucleótidos CpG que no están metilados que tienden a concentrarse en regiones que se denominan islas CpG. Aunque estas islas se han definido tradicionalmente como las regiones de un tamaño igual o superior a 500 pb, con un contenido total de G+C superior a 50% y con un cociente de dinucleótidos CpG observados frente a esperados superior a 0,6, hoy en día se pueden definir por su porcentaje normalizado de CpG. De hecho, el análisis de todos los promotores del genoma humano identifica dos tipos de genes: los que tienen un promotor con un porcentaje normalizado de CpG en torno al 0,61 y los que tienen un promotor con un porcentaje normalizado de CpG en torno al 0,23. Los primeros constituyen un 70% de todos los promotores, y corresponden a los genes constitutivos ó ―domésticos‖ ("housekeeping" en inglés). Por el contrario, los promotores con bajo porcentaje normalizado de CpG constituyen un 30% del total de los promotores, y corresponden a los genes que se expresan en tejidos específicos. Los promotores que contienen una isla CpG están habitualmente hipometilados en la línea germinal, lo cual les protege frente a las transiciones CT que sufren las citosinas metiladas. Figura 2.9 En este video se puede ver el concepto de "isla CpG", al examinar la distribución de dinucleótidos CpG a lo largo de un gen y ver su estado de metilación. La metilación del ADN está catalizada por unas metil-transferasas específicas de las citosinas que forman parte de dinucleótidos CpG, y se conocen básicamente dos tipos de estas metil-transferasas de ADN. Las DNMT3A y DNMT3B son responsables de la metilación de novo, es decir, la metilación de dinucleótidos CpG que no estaban previamente metilados. En cambio, la DNMT1 (ADN-metiltransferasa-1) es responsable de mantener la metilación durante la replicación. En efecto, dado que en la doble cadena de ADN un dinucleótido CpG tiene realmente dos citosinas metilables (ya que existe un CpG en cada una de las cadenas de la doble hélice), un sitio totalmente metilado tendrá realmente dos citosinas metiladas. Tras la replicación del ADN, ambas dobles hélices conservarán una cadena con el CpG metilado (la proveniente de la molécula original), mientras que la cadena de nueva síntesis estará sin metilar. Esto genera dinucleótidos CpG hemi-metilados, es decir, metilados en una de las citosinas pero no en la otra; la DNMT1 tiene la función de re-metilar precisamente estos dinucleótidos para restablecer el estado inicial de metilación completa que tenía la molécula original. Figura 2.10 Un dinucleótido CpG totalmente metilado quedará hemimetilado tras la replicación del ADN de esa región. La metilasa de mantenimiento DNMT1 se encarga de re-metilar estas regiones, como muestra este video. Numerosos estudios han mostrado el significado biológico de la metilación del ADN: 1) es un importante mecanismo epigenético de silenciamiento génico a nivel transcripcional, y permite mantener el silenciamiento de ciertos genes durante los procesos de diferenciación celular; 2) la metilación es un mecanismo de defensa frente a elementos móviles del genoma, cuyos promotores están habitualmente silenciados por metilación; y 3) la metilación es un mecanismo estabilizador de la cromatina, especialmente de la heterocromatina pericentromérica, ya que la des-metilación de este tipo de cromatina conduce a la aparición de reordenamientos cromosómicos severos. Los mecanismos moleculares por los que la metilación conduce al silenciamiento transcripcional pueden ser múltiples: impidiendo la unión de factores de transcripción al promotor. Por ejemplo, algunos factores de transcripción generales como Sp1, CREB, E2F ó NF-kB se unen a dominios que contienen CpG, y esta unión disminuye cuando estos dominios están metilados. Sin embargo, éste mecanismo no se puede aplicar de modo general a todos los genes. mediante la unión específica de represores transcripcionales al ADN metilado. Existen varias proteínas de unión a los dinucleótidos CpG metilados, denominadas genéricamente MeCP ("MethylCytosine binding Protein") ó MDB ("Methyl Binding Domain"), que forman parte de complejos con actividad represora de la transcripción. Por ejemplo, MeCP-2 es una proteína que contiene un dominio de unión a dinucleótidos CpG metilados, así como otro dominio por el que se une a un represor transcripcional llamado Sin3. Curiosamente, Sin3 reprime la transcripción a través de su unión con HDAC2 (que, como ya hemos visto, des-acetila las lisinas de la histona H3). De esta manera, la represión transcripcional debida a la metilación de los dinucleótidos CpG se produce gracias a la asociación entre metilación del ADN y acetilación de la cromatina, ya que el dominio de represión transcripcional de MeCP-2 es capaz de reclutar el complejo Sin3-NCoR-HDAC2 y así iniciar un foco de cromatina hipoacetilada en una región específica del genoma. Esto explica que las regiones en las que el ADN está metilado sean capaces de silenciar genes cercanos (aunque éstos no tengan sus dinucleótidos CpG metilados), al englobarlos en una región genómica silenciada por des-acetilación. Figura 2.11 La regulación epigenética de la transcripción génica incluye tanto la metilación del ADN como las modificaciones covalentes de las histonas. Como se muestra en este video, es precisamente la interacción entre ambos procesos lo que permite modificar la cromatina para facilitar la transcripción o para silenciar una región. De todas formas, uno de los hallazgos novedosos del proyecto ENCODE (mencionado en el capítulo 1) ha sido que la relación entre metilación y unión de factores de transcripción es, a menudo, contraria a lo que se intuía. La visión clásica es que lo primero es la metilación del ADN, y esto impide (o permite) la unión de factores de transcripción y así regula la expresión de un gen. Ahora, en cambio, sabemos que en muchos casos es al revés: la unión de factores de transcripción impide la metilación de esa región de ADN. Dado que se trata de procesos reversibles, en un equilibrio dinámico, esto permite una regulación muy fina de la expresión génica. Como ya se ha mencionado, una propiedad muy importante del silenciamiento por metilación es que es reversible: se ha comprobado que los promotores que han sido silenciados por metilación pueden reactivarse si se des-metila esa región. Un mecanismo posible para explicar esta reactivación es la pérdida de la metilación durante la replicación: esto sucedería si ciertos factores de transcripción ó activadores nucleares consiguieran unirse a sus promotores inmediatamente después de la replicación y antes de que actúe la DNMT1 de mantenimiento en los sitios hemi-metilados. Una vez que se estabiliza el complejo basal de transcripción sobre esa región, éste atraería algunos componentes con actividad HAT, que a su vez actuarían manteniendo la cromatina abierta y permitiendo la transcripción. Esto, además, impediría la acción de la DNMT1 y terminaría por eliminar la metilación en esa región tras varios ciclos de replicación. Este mecanismo se conoce como des-metilación pasiva. Otro mecanismo alternativo para explicar la re-expresión de genes silenciados por metilación, sobre todo en células diferenciadas que ya no se replican, es la desmetilación activa, aunque todavía no se ha aislado de modo concluyente ninguna des-metilasa de ADN. Estudios recientes han demostrado que una proteína llamada TET1 tiene actividad 5-metilcitosina (5mC)-hidroxilasa, convirtiendo las 5mC en 5-hidroxi-mC en células de mamíferos. Estas 5-hidroxi-mC son después desaminadas a 5-hidroxi-mU por unas desaminasas llamadas AID o APOBEC, y los 5-hidroxi-mU son convertidos en citosinas por el sistema de reparación BER (que se estudiará en detalle en el Tema 3) a través de una ADN-glicosilasa llamada TDG. Figura 2.12 Proceso de desaminación activa de citosinas metiladas en mamíferos. En el año 2009 se descubrió que algunas células humanas llevan un nuevo tipo de metilación del ADN (previamente descrito sólo en plantas), que no afecta a las citosinas de dinucleótidos CpG sino a las citosinas de los trinucleótidos CHG ó CHH (siendo ―H‖ cualquier nucleótido excepto G). En humanos, esta modalidad de metilación sólo se da en células pluripotenciales, llegando a constituir un 25% de todas las citosinas metiladas en células madre embrionarias. Al contrario que la metilación de CpGs, esta metilación ―no-CpG‖ es más intensa en el cuerpo de los genes y su relación con la expresión génica no es directa. Se piensa que este nuevo tipo de metilación juega un papel importante en el mantenimiento del estado pluripotencial, ya que desaparece durante la diferenciación celular. En resumen, la metilación del ADN constituye un importantísimo mecanismo epigenético de regulación de la expresión génica, debido a su interacción con los mecanismos que modifican la estructura de la cromatina. El mantenimiento de los patrones de metilación explica también que las modificaciones epigenéticas sean heredadas establemente tras la replicación del ADN. Además, como ya se ha mencionado, la metilación tiene importantes implicaciones biológicas, y por tanto la alteración de los procesos normales de metilación de la cromatina puede perturbar procesos fisiológicos importantes. En primer lugar, por ser un mecanismo fundamental en el silenciamiento de retrovirus endógenos, transposones y ADN satélite, las alteraciones en la metilación de estos elementos puede provocar numerosas anomalías genéticas. Además, la metilación es la base molecular de los fenómenos de impronta genómica (imprinting) que se estudiarán más adelante, y es un mecanismo fundamental en la regulación de la expresión génica durante el desarrollo embrionario. De hecho, el embrión sufre una onda de des-metilación genómica global en la fase de mórula de 8 células, seguida por la re-metilación gradual que vuelve a fijar los patrones de metilación en la fase de blastocisto. También la inactivación del cromosoma X en mujeres es un proceso básicamente controlado por procesos de metilación, acetilación y silenciamiento. Por tanto, cada vez está más claro que la des-regulación de las modificaciones epigenéticas de la cromatina está en la base de algunas enfermedades humanas. El caso más claro es el de una enfermedad neurológica hereditaria llamada Síndrome de Rett, en el que se han identificado mutaciones en el gen que codifica la proteína de unión a metil-citosinas MECP2 dando lugar a un exceso de ―ruido transcripcional‖ por falta de silenciamiento global de muchos genes, con efectos más marcados en el cerebro que en otros órganos. Además, se ha visto que las células de estos pacientes tienen desmetilación de los elementos LINE1 (que, por tanto, muestran mayor actividad de retrotransposición en algunos tejidos). Por otra parte, el papel de la metilación en cáncer también está cobrando cada vez más importancia, ya que se ha visto que muchos tipos de tumores tienen alteraciones importantes en los procesos de metilación de la cromatina: la desaminación de citosinas metiladas (debida habitualmente a la acción de una citidíndesaminasa, ó bien por desaminación hidrolítica espontánea) provoca un cambio de Citosina por Timina, haciendo que los dinucleótidos CpG metilados sean puntos calientes (hotspots) para la generación de mutaciones de este tipo. De hecho, se ha estimado que el 30% de las mutaciones encontradas en tumores en el gen TP53 (que codifica la proteína p53, un supresor tumoral importante) son transiciones CT que tienen lugar dentro de dinucleótidos CpG. se ha comprobado que durante el proceso de iniciación tumoral hay una hipometilación global del genoma de la célula pre-maligna, lo que provoca un aumento en la inestabilidad genómica y la aparición de anomalías cromosómicas. Por ejemplo, los ratones en los que el gen DNMT1 ha sido inactivado muestran una elevada tasa de deleciones o duplicaciones de regiones cromosómicas. Además, algunos oncogenes concretos se sobreexpresan en algunos tumores por des-metilación de sus promotores (por ejemplo el oncogén BCL2). Del mismo modo, se ha identificado una enfermedad (Síndrome ICF, siglas de Inmunodeficiencia, inestabilidad Centromérica y defectos Faciales) en la que los pacientes tienen mutaciones en el gen DNMT3B, que codifica una metiltransferasa que metila específicamente los satélites centroméricos 2 y 3; la desmetilación de esas regiones de heterocromatina provoca fusiones entre cromosomas. En conjunto, estos datos sugieren que la metilación es un mecanismo muy importante para mantener la estabilidad del genoma. al mismo tiempo, la hipermetilación puntual de algunos genes supresores tumorales, con el consiguiente silenciamiento y pérdida de función, es un mecanismo muy frecuente de generación de distintos tipos de cáncer. Por ejemplo, el gen RB1 está frecuentemente metilado en un tipo de tumores llamados retinoblastomas esporádicos. También es frecuente la hipermetilación (y silenciamiento) del gen supresor tumoral p16 en varios tipos de tumores; la hipermetilación del gen VHL (Von Hippel-Lindau) en 20% de los carcinomas de células renales esporádicos; la inactivación, por hipermetilación, del gen de reparación de desemparejamientos hMLH1 en tumores de colon esporádicos que muestran inestabilidad de microsatélites. En conjunto, se estima que en tejido tumoral hasta un 10% de las islas CpG están metiladas, en contraste con células normales en las que este porcentaje es mucho más bajo. por su parte, algunos complejos con actividad modificadora de la cromatina se han asociado también con el desarrollo de cáncer. Por ejemplo, el gen AIB1, que tiene actividad acetilasa de histonas, está amplificado en cáncer de mama y por tanto se expresa a niveles más altos de lo normal. Por otro lado, el co-activador transcripcional CBP, que también funciona como una acetilasa de histonas, está fusionado con el gen MOZ ó con el gen MLL en pacientes con leucemia mieloide aguda; esta fusión hace que la actividad acetilasa esté aumentada en determinadas células de la médula ósea y esto desencadena la leucemia. Además, otro co-activador transcripcional con actividad acetilasa de histonas (la proteína p300) está mutado en cáncer colorrectal y delecionado en el 80% de los glioblastomas, un tipo de tumor cerebral. Lo mismo puede decirse de los complejos con actividad des-acetilasa de histonas (HDAC): ya hemos visto que la supresión de Myc y la unión de Rb con el factor de transcripción E2F dependen en gran medida de su unión con complejos que tienen actividad des-acetilasa. Además, se ha demostrado que algunas oncoproteinas virales como HPV E7 ó el antígeno T de SV40 inactivan el gen RB1 al impedir su unión con complejos que tienen actividad HDAC. Finalmente, las translocaciones asociadas con la leucemia promielocítica producen fusiones entre factores de transcripción que interaccionan con diversas acetilasas y des-acetilasas de histonas, con complejos remodeladores de la cromatina y con proteínas que tienen actividad metil-transferasa de histonas. Figura 2.13 La regulación de la transcripción de los genes que responden al ácido retinoico se lleva a cabo mediante la actuación de complejos con actividad acetilasa o des-acetilasa de histonas. Este video muestra la regulación normal de la expresión de un gen en respuesta a la presencia de ácido retinoico, y la alteración de este mecanismo en la leucemia promielocítica aguda con la translocación cromosómica 15;17. Considerando todos los ejemplos citados en los párrafos anteriores, podemos concluir que los procesos que regulan la metilación de la cromatina están implicados en la iniciación y progresión tumoral; esto, además, abre nuevas posibilidades terapéuticas en muchos tipos de cáncer, ya que el estado de metilación es potencialmente modificable mediante fármacos. Relación entre secuencia, estructura y función de la cromatina: territorios cromosómicos Al ocuparnos de los mecanismos que regulan la función del genoma a nivel global, es importante tener en cuenta el nivel superior de organización de un genoma dentro del núcleo. En los últimos años se ha puesto en evidencia que distintas regiones de la fibra de cromatina tienden a ocupar posiciones concretas en el núcleo, dependiendo de la secuencia subyacente, del estado transcripcional de los genes de esa región y de sus modificaciones epigenéticas, del tiempo de replicación del ADN implicado, etc. Por tanto, cada vez está más claro que si queremos tener una imagen completa de cómo se regula la función del genoma, no podemos ignorar aspectos estructurales tales como la composición en nucleótidos o la posición dentro del núcleo. Uno de los aspectos más intrigantes de la biología genómica de los últimos años es la organización espacial de la cromatina dentro del núcleo de la célula eucariota, dando lugar a lo que se han denominado "territorios cromosómicos". Este concepto ha venido a completar la imagen del núcleo eucariota como una estructura compartimentalizada, en la que distintas regiones contienen maquinarias específicas que llevan a cabo funciones concretas. Esta noción debe integrarse con la presencia de dominios cromatínicos definidos y más o menos estables a lo largo de la vida de una célula. Diversos estudios han mostrado de forma convincente que la cromatina correspondiente a cada uno de los cromosomas tiende a ocupar unas posiciones concretas en el núcleo en interfase, y que esas posiciones tienden a mantenerse durante el ciclo celular. Por ejemplo, la posición de un cromosoma concreto dentro del núcleo está relacionada con el tamaño del cromosoma y con su riqueza en genes, que ―como hemos visto― se correlaciona también con la secuencia de nucleótidos subyacente. En general, se considera que los cromosomas pequeños tienden a localizarse hacia el interior del núcleo; igualmente, cromosomas ricos en genes tienden a ocupar posiciones centrales. También existen datos que muestran que las regiones pobres en genes y en secuencias Alu se asocian con la periferia del núcleo, dejando la cromatina rica en genes en el interior. De todas formas, todavía se desconoce cómo se establecen estos territorios y cómo se mantienen tras la mitosis. Figura 2.14 En este enlace se pueden ver imágenes que ilustran el concepto de “territorio cromosómico”. Aquí hay una buena revisión en inglés. La replicación del ADN y la transcripción génica son dos procesos que tienen lugar dentro del núcleo y que exigen una remodelación importante de la fibra de cromatina para permitir el acceso de las maquinarias proteicas que los llevan a cabo. Además, se ha visto que ambos procesos no tienen lugar al azar, en cualquier lugar del núcleo, sino en compartimentos especializados a los que acuden las fibras de cromatina que están en la vecindad. Se habla así de "fábricas de replicación" ó de "fabricas de transcripción", regiones nucleares ocupadas por regiones genómicas que se replican o transcriben simultáneamente aunque pertenezcan a territorios cromosómicos distintos. Lógicamente, las asas de cromatina que ocupan una fábrica determinada tendrán una configuración similar, en cuanto al grado de condensación de la cromatina, la secuencia de nucleótidos, la riqueza en genes, etc; esto explica que el genoma se organice como un mosaico de regiones que comparten características similares, ya que esas regiones podrían interaccionar dentro del núcleo al situarse en una misma fábrica de replicación o de transcripción. Por ejemplo, se ha visto que en el genoma humano existen unas regiones llamadas RIDGES (Regions of IncreaseD Gene Expression, en inglés) en las que son más abundantes los genes que se transcriben activamente, y son regiones ricas en G+C, pobres en repeticiones tipo LINE y con densidad génica alta. Junto a éstas, se observan también regiones con poca densidad en genes, bajo nivel transcripcional y relativamente pobres en G+C, que se llaman por tanto anti-RIDGES. Algo similar detectó el proyecto ENCODE, del que se ha hablado en el capítulo anterior, al analizar una gran cantidad de modificaciones epigenéticas a lo largo de todo el genoma. Según los datos de ENCODE, se pueden definir dos tipos de regiones genómicas: a) dominios “activos”, que serían las regiones definidas por niveles altos de transcripción, replicación temprana, acetilación de la histona H3 y des-metilación de la lisina 27 de la histona H3 (H3K27); b) dominios “inactivos”, correspondientes a regiones de replicación tardía con poca actividad transcripcional, baja acetilación de H3 y metilación de H3K27. Los dominios activos son muy ricos en sitios de inicio de la transcripción, islas CpG y repeticiones Alu, mientras que las repeticiones tipo LINE1 y LTR están sobre-representadas en los dominios inactivos. Pues bien, los genes presentes en RIDGES (equivalentes a los dominios activos de ENCODE) tienden a localizarse, dentro del núcleo, en torno a las fábricas de transcripción. Lo mismo sucede con los genes de replicación temprana, que se asocian significativamente con las fábricas de replicación nucleares. Por tanto, los modelos actuales postulan que los genes que son replicados o transcritos residen en regiones cromatínicas relativamente descondensadas, las cuales salen de sus propios territorios cromosómicos hacia fábricas de replicación o transcripción vecinas. En esas fábricas, por tanto, pueden interaccionar regiones alejadas de un mismo cromosoma, o incluso asas de cromatina de territorios cromosómicos distintos; esto permite explicar, en el caso de la transcripción, la acción de potenciadores de la expresión que actúan a larga distancia ó incluso en trans. Una de las aportaciones del proyecto ENCODE ha sido la de analizar, a nivel global en todo el genoma, interacciones físicas entre regiones distantes (interacciones de promotores con enhancers y otros elementos reguladores relativamente alejados o incluso situados en otros cromosomas). Esto da lugar a un cuadro bastante complejo en el que los promotores interaccionan con muchos sitios, la mayoría de los cuales están a bastante distancia (>120 kilobases). De hecho, sólo el 7% de las interacciones se producen entre sitios reguladores y el gen más cercano. Además, se observaron interacciones entre múltiples elementos a la vez, lo que da lugar a redes de interacción que permiten una primera visión del genoma en tres dimensiones. Como es lógico, en todas estas interacciones son importantes las modificaciones epigenéticas de la cromatina y del ADN que hemos estudiado en este Capítulo, que en el fondo constituyen la base molecular que permite los fenómemos de condensación y descondensación necesarios para la replicación, transcripción y desplazamiento físico de las asas de cromatina. Conjugando todos estos elementos, obtenemos una visión dinámica de la cromatina nuclear en la que la estructura del núcleo se integra con la función y secuencia del genoma. Esta visión es mucho más rica que los modelos anteriores, que no tenían en cuenta todos estos factores, y además permite explicar mucho mejor la regulación fina de la expresión génica durante el desarrollo embrionario ó la diferenciación celular. La otra cara de la moneda es que se trata de procesos tremendamente complicados y, por ello, potencialmente frágiles, de modo que pequeñas perturbaciones en estos mecanismos pueden dar lugar a alteraciones genéticas y enfermedades humanas. En este sentido, el cáncer es un ejemplo típico de proceso complejo de des-regulación de la diferenciación celular, en el que se observan importantes alteraciones epigenéticas y una fuerte desorganización de la estructura nuclear.