Unidad 3.
Anuncio

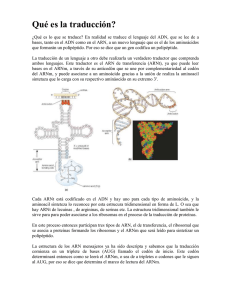
3 Replicación, trascripción traducción del material genético. y La expresión génica se concretiza por la transformación de la información genética desde moléculas de ADN a moléculas de ARN y desde estas hasta los polipéptidos correspondientes. Objetivo: Describir como una célula duplica su material genético precisamente a partir del ADN y explicar como decodifica y utiliza la información contenida en su genoma. 3.2 Mecanismos de transcripción. Los precursores en la síntesis de ARN (unidades estructurales) son cuatro ribonucleótidos trifosfatados: rATP; rCTP; rGTP; rUTP. Las moléculas de ARN son sintetizadas usando como molde a segmentos específicos de ADN, en la reacción de polimerización que es catalizada por la enzima conocida como ARN polimerasa. La secuencia de bases, del ARN a polimerizar, esta determinada por medio de la secuencia de bases de la molécula de ADN utilizada como molde para su síntesis. He ahí que se la denomine Cadena Molde de ADN (o cadena templada). La cadena de ARN crece en dirección 5’ – 3’. Esta dirección coincide con la síntesis de ADN. La ARN polimerasa, a diferencia de la ADN polimerasa, es capaz de iniciar su síntesis sin la presencia de ningún tipo de molécula cebadora. En eucariotas, los nucelosomas deben ser removidos y reestablecidos una vez que la lectura se ha realizado. 1 Características de la ARN polimerasa procariota. La ARN polimerasa es una de las enzimas más grandes que se conoce. Consta de varias sub-unidades: dos alfa (α), beta (β), beta prima (β’) y sigma (σ). 1° etapa- UNIÓN de la ARN polimerasa al ADN La enzima completa es 2° etapa- INICIACIÓN de la síntesis 3° etapa- ELONGACIÓN de la cadena de ARN mensajero denominada Holoenzima y se divide en dos componentes principales: • La enzima central, denominada Core, formada por las sub unidades 2 α, β y β’ • El Factor Sigma ( el polipéptido σ) 4° etapa- TERMINACIÓN de la síntesis y liberación de la cadena de ARNm. 2 Los factores Sigma suelen estar organizados en cascadas • La cascada de factores sigma se genera cuando un factor sigma es necesario para transcribir el gen que codifica para el siguiente factor sigma. • Los genes tempranos del faho SPO1 son transcritos por la RNA polimerasa. • Uno de estos genes tempranos codifica para un factor sigma que causa que la RNA polimerasa transcriba los genes medios. • Dos de los genes medios codifican las subunidades del factor sigma que hace que la RNA polimerasa transcriba los genes tardíos. Secuencia terminadora Todas contienen secuencias Palindrómicas justo antes del punto de terminación. El palíndromo está caracterizado por poseer repeticiones inversas conteniendo un segmento central no repetido (ej. ATCATCGACTA). La secuencia de Uracilos suministra la señal que permite a la ARN polimerasa disociarse del ADN molde. La secuencia palindrómica continúa con una secuencia de pares A-T, las cuales producen en el ARN mensajero una secuencia de 6 a 8 Uracilos en el extremo transcrito. Antiterminación es un evento regulador. Se usa como mecanismo de control en operones de fagos y bacterias. Rho se enlaza al RNA y utiliza su actividad ATPasica para proveer energía para traslocarse a lo largo del RNA hasta que alcanza la región helical RNA-DNA, desenrollando la estructura. • La Terminación se evita cuando proteínas antiterminación actúan sobre la RNA polimerasa ocasionando que lea a través de un terminador o terminadores específicos. 3 ESTRUCTURA DEL ARN mensajero en PROCARIOTAS En células Procariotas es común hallar ARNm que codifican para varias cadenas polipeptídicas diferentes, en este caso esta molécula se denomina ARNm Policistrónico. Todo ARNm contiene dos tipos de regiones, No codificante. Son los segmentos Líder, Trailer. ESTRUCTURA DEL ARN mensajero en EUCARIOTAS La estructura de los ARNm y el proceso de transcripción en las células eucariotas, es similar a lo expresado anteriormente para las células procariotas. Sin embargo, debemos considerar las siguientes diferencias: Codificante- Son los cistrones o segmentos con codones que determinan la serie de aminoácidos de la proteína. Esta región se extiende desde un codón de inicio (usualmente AUG) hasta un codón stop (UAA, UAG, UGA). Raramente el codón de inicio se encuentra en el extremo 5’ del ARNm, ya que a éste lo preceden centenares de bases que conforman una de las regiones no codificantes. El sector comprendido entre el extremo 5’ y el codón de inicio se denomina Lider. Tampoco se traduce la secuencia comprendida entre el codón sin sentido y el extremo 3’ del ARNm y, a esta región, se la denomina extremo Trailer. Los ARNm Policistrónicos presentan secuencias de longitud variable que separan las regiones codificantes o Cistrones, estas se denominan regiones espaciadoras, usualmente de 10 pb. de longitud. Cada Cistrón posee un codón de inicio y un stop. Por lo general las proteínas codificadas por un ARNm Policistrónico, participan en una misma vía metabólica (ver Operón Lac). a) Las células eucariotas poseen tres clases de ARN polimerasas (I, II y III), las cuales se utilizan para sintetizar los distintos tipos de ARN existentes. La RNA polimerasa no puede iniciar la síntesis sola, requiere factores de trascripción específicos para cada sitio. b) Los extremos 3’ y 5´ de los ARNm están modificados. En el caso del extremo 5’ encontraremos una estructura denominada CAP y, en el correspondiente extremo 3’, se encuentra adherido una larga secuencia de nucleótidos cuya base nitrogenada es la Adenina (Cola Poly A). c) Las moléculas de ARNm, luego de ser sintetizas, son modificadas. Los “transcritos primarios” eucariotas sufren un proceso por el cual determinadas secuencias (Intrones) son eliminadas. d) Los ARNm eucariotas son Monocistrónicos - codifica para una cadena Inicio Stop Inicio Stop ARNm policistrónico polipeptídica simple. e) TATA box localizada a -25 Pb del punto de inicio. f) La presencia del promotor esta aumentada por un Exaltador o Enhancer. Principales Diferencias en la Transcripción y Traducción entre Procariotas y Eucariotas 4 3 Replicación, trascripción traducción del material genético. y Objetivo: Describir como una célula duplica su material genético precisamente a partir del ADN y explicar como decodifica y utiliza la información contenida en su genoma. 3.3 Síntesis de Proteínas. Traducción- La información del ADN transcrita a mRNA es interpretada por el rRNA, lo que permite el transporte (tRNA) e incorporación de los aa específicos de la proteína codificada. Esta estrechamente acoplado a la trascripción. QuickTime™ and a decompressor are needed to see this picture. - Molécula mRNA policistrónico. Regiones codificantes para varias proteínas (generalmente de una misma ruta metabólica, Operón Lac). Región codificante: serie de codones que determinan los a.a de una proteína (codón de inicio (AUG) y un codón sin sentido)ÎCistrón. RNA mensajero (mRNA) RNA ribosomal (rRNA) •El rRNA es el más abundante (> 80% del RNA celular). •El RNA menos abundante. 5-10% del RNA celular. •Porta el mensaje genético del DNA. Secuencias de 3 nucleótidos codifican para los aminoácidos de una cadena polipéptida. •Su tamaño es muy variable y depende de la longitud del polipéptido que codifica. •Componente mayoritario de los ribosomas (rRNA 60-65%, proteínas 35-40%). •Algunos de ellos son RNA muy largos y con muchos apareamientos internos que generan una estructura secundario con horquillas y lazos muy compleja. •Interacciona con los otros RNAs (mRNA y tRNA) en cada paso de las síntesis de proteínas. rRNA Î Ribosomas RNA de transferencia (tRNA) •Hay al menos un tRNA por cada aminoácido. •Cadena sencilla de RNA de 75-85 nucleótidos con bases modificadas. •Estructura secundaria en hoja de trébol debido a apareamientos internos que dan lugar a horquillas y lazos. Estructura terciaria en forma de L. •Dos motivos estructurales esenciales: 9Extremo 3’que forma enlace covalente con el aminoácido correspondientes. 9Secuencia anticodón complementaria de los tripletes que codifican aminoácidos. 1 El proceso dinámico de la traducción del mRNA consta de tres etapas: 1-Iniciación 2-Elongación 3-Terminación 1-Iniciación representa todos los procesos requeridos para ensamblar el ribosoma con el codon de inicio. 2-Durante la elongación se lleva a cabo la síntesis de polipéptidos en la sub-unidad ribosomal grande. 3-Cuando el ribosoma alcanza la señal de “stop” se libera con el péptido. Durante todo el proceso el ribosoma requiere factores de traducción que le auxilian a enlazarse al mRNA, a seleccionar los aa y a mediar la terminación. Proceso particularmente complejo en eucariotas. 1. Unión de la subunidad pequeña ribosomal al codón de inicio. 1. Reunir los elementos necesarios 2. Reconocer el sitio de inicio (AUG) 3. Proveer sitios para que se inicie la elongación Iniciación P R O C A R I O T A S .. 1 Unión de la sub-unidad pequeña ribosomal al codón de inicio. 2 Unión del complejo aminoácido, el tRNA al ribosoma. 3 Establecer el complejo de iniciación con sub-unidad grande. 4 Inicia fase de elongación. Factores proteicos requeridos para el inicio de la traducción en bacterias • • • IF‐1 Evita la unión prematura del tRNA al sitio A IF‐2 Facilita la unión de fMet‐tRNAfMet a la subunidad ribosómica 30S IF‐3 Se une a la subunidad ribosómica 30S; impide la asociación prematura de la subunidad 50S; mejora la especificidad del sitio P hacia fMet‐tRNAfMet Reconocimiento Secuencia de Shine-Delgarno: 5 a 10 primeros nucleótidos antes del codón de inicio complementaria a los nucleótidos cerca del extremo 3’ de la sub-unidad 16s rRNA. Secuencias complementarias que permiten que llegue la sub-unidad 30s y se pegue. Elongación 2. Unión del tRNA al ribosoma. Requerimientos - mRNA - tRNA (N-formil-metionina) - codón de inicio - 2 subunidades ribosomales - factores de iniciación (IF1, IF2 y IF3) - GTP, Mg 2+ 3. Establecer el complejo de iniciación. Etapas: 5- Selección del amino-acil-tRNA. 6- Formación del enlace peptídico. 7- Translocación y liberación del tRNA deacilado. 2 Translocación Cambio en la posición de las dos subunidades del ribosoma para dar paso al tercer aminoacido. Formación del enlace se logra cuando el Nitrógeno del grupo amino del aminoácido 1 se une al grupo carbonilo del aminoácido 2 Factor de elongación requerido Enzima involucrada: Peptidil-transferasa • Terminación • • 3 codones Î terminación de la adición de a.a • UAA, UAG o UGA Î detener la elongación y liberar el polipéptido asociado al último tRNA. • Reconoce que aquí ha terminado la proteína 1. Complejo covalente con el codón. 2. Cambio en la conformación del ribosoma, se une al RF. 3. Hidrólisis CONTROL DE LA TRADUCCIÓN Factores de liberación (RF:release factors) – – – • • Los genes que codifican para los factores de liberación en procariotas, RF1 y RF2, se han identificado como prfA y prfB, respectivamente. Mutaciones en estos genes pueden provocar la falta de lectura a las señales de terminación, provocando una terminación ineficiente y una lectura continua. El proceso de terminación es de crítica importancia en la síntesis de proteínas. Mala interpretación del STOP Î adición no funcional e incluso dañina a la cadena de polipéptidos Î pérdida de la función real de la proteína. FR1: UAA y UAG FR2: UAA y UGA FR3: no específico, incrementa la actividad de FR1 y FR2. Riboswitches RF Î sitio A. – sRNA Tripéptido interactúa con el codón STOP, hidroliza el enlace éster del tRNA y libera el a.a. Inhibidores Poliribosomas Operón La iniciación de la síntesis de proteínas en el citoplasma de los eucariotas es parecido al de las bacterias. Diferencia entre la expresión génica de procariotas y eucariotas pero el orden de los sucesos es diferente y el numero de factores accesorios es mayor. •La metionina inicial (AUG) en presenta el grupo formil (formilmetionil-tRNA). •Los factores de iniciación (IEF 1, IEF2 y IEF 3) son menos abundantes que en eucariota (al menos 12). •La traducción y la trascripción están acopladas. •La proteína producto se madura directamente en el citosol. • En Eucariotas, la subunidad pequeña reconoce primero el extremo 5’ del ARNm, y se desplaza al punto de iniciacion, donde se unen las subunidaes grandes. • Casi todos los ARNm de los eucariotas son monocistronicos, pero cada ARNm es por lo menos sustancialmente mas largo que lo estrictamente necesario para codificar su proteína. • El ARNm medio de un citoplasma eucariótico es de unas 1000-2000 bases de longitud. • Tiene un cap metilado en el extremo 5’ y transporta entre 100 y 200 bases de Poli(A) en el extremo 3’ . 3 Se necesitan varios factores de iniciación en eucariotas para desenrollar el ARNm, unir la subunidad del complejo de iniciación y colaborar con la unión de la subunidad mayor. • El codon de iniciacio AUG esta situado a menos de 40 bases del extremo 5’ del ARNm, de manera que tanto el cap como el AUG estan unidos simultaneamente al ribosoma. • Pero en muchos ARNm, el cap y el AUG están mas separados, en casos extremos a ~1000 bases de distancia (5’-ACCAUGG-3´). • Aun así la presencia del cap sigue siendo necesaria para que se forme un complejo estable en el codon de iniciación. Control regulatorio por sRNA • Como puede el ribosoma depender de dos puntos tan separados ? • La migración se detiene cuando la unidad 40S encuentra el codon AUG. El triplete AUG detiene la migración cuando consta de la secuencia GCCªCCCAUGG. Las tres bases purinicas ( A o G ) previas al codon AUG y la G que le sigue inmediatamente son de máxima importancia, e influyen en la eficacia de la traducción. • El modelo de “busqueda”, supone que la subunidad 40S reconoce inicialmente el cap 5’ y “migra” entonces a lo largo del ARNm. • • La unión se estabiliza en el punto de inicio cuando la unidad 40S se une a la subunidad 60S. La búsqueda a partir del extremo 5’ es un proceso lineal. • La metionina del ARNt iniciador no se formila, se llama ARNti Met. • Tiene una estructura terciaria poco habitual, esta modificado por fosforilación 2’ribosa en la base 64 (si se impide esta modificacion, el iniciador se puede utilizar para la elongacion ). La unión de las subunidades 60S con el complejo de iniciación no se puede producir hasta que elF2 y elF3 se han liberado de este complejo de iniciación. Como reconoce el ribosoma el ARMm ? elF4G se une a elF3, que se asocia con la unidad ribosómica pequeña. Esto hace que se unan la subunidad ribosómica 40S al elF4F y así es reclutada para el complejo. En efecto, el IF4F coloca el F4G en su lugar de forma que pueda atraer a la subunidad pequeña 40S. El IF6 (que tiene la mayor parte de actividad anti asociacion ) Se une a la unidad ribosómica grande. Se libera cuando la subunidad mayor se une al complejo de iniciación. 4 Una vez que se ha formado el ribosoma completo en el codon de iniciación, el escenario esta dispuesto para un ciclo en el que el aminoacil -ARNt entra en el lugar A de un ribosoma cuyo lugar P esta ocupado por el peptidil -ARNt • Cualquier aminoacil –ARNt excepto el iniciador puede entrar en el sitio A. • Su entrada esta mediada por un factor de elongación ( EFF-Tu en las bacterias) en los eucariotas el proceso es parecido. • Una vez que el aminoacil-ARNt esta en su lugar, EF-Tu abandona el ribosoma, para trabajar de nuevo con otro aminoacil- ARNt. • Así que muestra la asociacion cíclica con el ribosoma que es la característica de los factores accesorios. • Se carga el aminoacil - ARNt en el sitio A en dos fases. • Primero: el extremo del anticodon se une al lugar A de las subunidades 30S. Entonces, el reconocimiento codon -anticodosn dispara un cambio en la conformación del EF-Tu. • Se rompe el GTP. El extremo CCA del ARNt se mueve hasta el lugar A de la subunidad 50S. • Se libera el complejo binario EF-Tu-GDP. Esta forma de EF-Tu es inactiva y no liga de forma efectiva el aminoacil- ARNt. Vía para que el aminoacil- ARNt entre en el sitio A • El EF- Ts es desplazado a su vez por el GTP, volviendo a formar EF- GTP. • El complejo binario activo se une al aminoacil -ARNt; y el EF- Ts liberado se puede reciclar. • Esto implica que la mayoría de los aminoaciacil -ARNt probablemente están presentes en forma de complejos ternarios. • Por lo tanto , la presencia de GTP es necesaria para que el aminoacil -ARNt se una al lugar A, pero la hidrólisis no es necesaria hasta mas tarde. • El Principio básico es que las reacciones en que participa EF- Tu ocurran con suficiente lentitud para permitir que los aminoacil –ANRt incorrectos se disocien antes de que queden atrapados en la sintesis de proteinas. • En Eucariotas, el factor EF- 1ά es responsible de llevar al aminoacil –ARNt al Ribosoma 5 La Translocacion mueve al ribosoma • El Ribosoma permanece en su lugar mientras la cadena polipeptídica se alarga mediante la transferencia del polipéptido unido al ARNt en el sitio P al aminoacil –ARNt que esta presente en el sitio A. • La actividad responsable de la síntesis de la unión peptidica se llama peptidil transferasa. • La translocacion necesita de GTP y de otro factor de elongación, EF –G este factor es un constituyente principal de la celula. Dominios que se parecen ! La estructura del complejo ternario de aminoacil –ARNt . EF –Tu. GTP (izquierda ) se semeja a la estructura del EF –G ( derecha ) . Los Dominios estructuralmente conservados de EF –Tu y EF –G son de color rojo y verde; el ARNt y el dominio que se le parece el el EF –G Son de color violeta. La capacidad de cada de cada factor de elongación para excluir al otro descansa sobre un efecto alosterico en la conformación global del ribosoma o en la competición directa para los sitios de unión que se superponen. Terminación Mapa de las rutas secretoras bioactivas y endocitoticas. Las proteínas son transportadas del ER a la membrana vía endosomas o lisosomas o bien pueden ser dirigidas al aparato de Golgi o al ER. 6 Videos sobre síntesis de Proteínas 7