Mapa Genético de Caracteres Medelianos Mapeo Genético
Anuncio

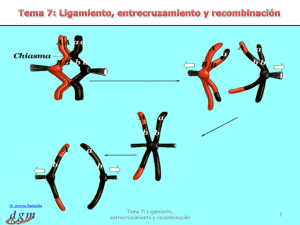
Mapa Genético de Caracteres Medelianos Cap 11 – Hum Mol Gen Mapeo Genético • Objetivo: determinar la frecuencia con que 2 loci son separados por recombinación meiótica. – Fracción de recombinación: es la proporción de recombinantes entre 2 loci. • Genes localizados en distintos cromosomas segregan independientemente >>> 50% de recombinantes >>> 0.5 • Genes sinténicos: genes localizados en el mismo cromosoma. – Haplotipo: conjunto de alelos localizados en una pequeña región que tienden a ser transmitidos en bloque. • La fracción de recombinación es una medida de la distancia entre 2 loci >>> distancia genética se mide en cM, centimorgan) >>> 1% de recombinación = 1 cM » No es igual a distancia física 1 Recombinantes y no recombinantes La fracción de recombinación nunca excede de 0.5 2 La relación entre el mapa físico y el genético no es constante en el genoma • El nro de quiasmas en la meiosis del hombre es de aprox. 49 por célula >>> 2450 cM • La frecuencia de entrecruzamientos es mayor en la mujer >>> 4296 cM. – 1cM aprox. 1 Mb Relación entre el mapa físico y genético Todos los cromosomas tienen un mapa genético ? •En gral hay más recombinación cerca de los telómeros •En la mujer hay mayor frecuencia de recombinación cerca de los centrómeros •En gral la frecuencia de recombinación es variable dependiendo de la región y el cromosomoma Preguntas 3 Hot Spots de Recombinación en el Cromosoma 22 Marcadores Genéticos • Se necesitan doble heterocigotas • Marcadores: – Polimórficos. Porque ? – Fácil de determinar – Se necesita como mínimo 1 cada 20 cM • >>> no menos de 300-500 marcadores/genoma 4 Desarrollo de marcadores genéticos Meiosis informativas vs no informativas 5 Determinación de recombinantes • Como sabemos cuando encontramos ligamiento entre un determinado marcador y la enfermedad que estamos estudiando?? – 1. Como determinamos la fracción de recombinación ?? – Fig 11.1 – 2. Que test podemos usar para evaluar si es significativamente diferente de 0.5 (hipotesis nula)?? Un caso fácil •El individuo II1 (doble heterocitota) podemos saber que alelo fue heredado de cada padre (phase-known) por lo que podemos clasificar cada meiosis como recombinante o no recombinante). •2 recombinantes en 7 meiosis >>> la fracción de recombinación es 0.28 6 Reconociendo recombinantes •Phase-known (II1) Phase-unknown (II1) A1 ó A2? 1 NR 2 NR 3 4 NR NR 5 NR 6 R 1 2 3 4 5 R o NR ?? 6 LOD Score 1. Fig 11.4 (B): Recombinantes o No Recombinantes ? Que hacemos ? Los descartamos ? Estimar la probabilidad total de ligamiento: A) probabilidad de ligamiento (suponiendo que estan ligados) B) probabilidad de ligamiento (suponiendo que no estan ligados) A/B : es la probabilidad de ligamiento el logaritmo de A/B es el LOD Score 2. Si analizamos varias familias. Como podemos determinar la probabilidad total de ligamiento?. Se multiplica las probabilidades para cada flia o suman los LOD Scores 7 Calculando LOD Scores LOD Scores: • Ligamiento: = ó > de 3 • No ligamiento: = ó < de –2 • Valores intermedios: indeterminado Corresponde a una probabilidad de 1000 a 1 porque es muy poco probable que 2 loci elegidos al azar puedan estar ligados. Si algo • Cual es la fraccion de es muy poco probable entonces necesitamos fuertes evidencias recombinacion de la flia A de para convencernos de que es verdad. No?? la fig anterior ? 0.167= 1/6 • De donde sale el valor de LOD score 3 para aceptar ligamiento ? • Intervalo de confianza: 1 unidas LOD LOD scores menores de –2 son utiles ? Yes. En la curva 3 excluye linkage en un intervalo de 12 cM alrededor de marcador. Si se excluyen muchas regiones, entonces pueden quedar unas pocas regiones posibles. 8 Búsquedas globales en el Genoma • Se buscan simultáneamente un nro muy grande de marcadores. • LOD score de 3 es para 1 marcador. • Si se usa mas de un marcador hay que usar una p mayor (3+log (n marcadores). • Pero en gral se acepta un valor de 3.3 • En la práctica, de cualquier forma, si es < de 5 se lo toma como provisional. Mapas de Marcadores Genéticos • El mapeo de marcadores se hace en flias seleccionadas muy grandes (CHEPH flias). – Hay colecciones de líneas celulares inmortalizadas de cada individuo – Tipeo de 8 flias con 8325 microsátelites. • Lo ideal es tener un mapa integrado: – Genético (separado por sexos) y Físico. 9 Tipificación de espermatozoides • Para reducir la distancia entre marcadores se puede usar la tipificación en espermatozoides. – Whole genome amplification • Se puede usar para mapear enfermedades? Problemas en genotipificación • Técnicos – Mala lectura de geles en marcadores polimórficos. – Mezcla de muestras. – No paternidad. • Diagnóstico erróneo – Introduce recombinantes erróneos. • Heterogeneidad de locus – Fenotipo similar causado por mutaciones en genes no relacionados 10 • Los análisis de ligamiento terminan cuando se encuentra un marcador que satisface todos los criterios. • En gral el blanco es de 1 Mb o más. Linkage disequilibrium • Se puede usar para reducir la región candidata en mapeos de enfermedad vs marcador – La idea es tipificar pacientes no relacionados por marcadores en la región de interés. • Si una proporción de los genes “enfermos” derivan de un ancestro común, se puede buscar un haplotipo ancestral compartido que defina una región menor del genoma. 11 Mapeo en Autocigotas • Autocigotas: homocigotas para marcadores heredados recientemente de un antepasado común. • Individuos con enfermedades recesivas raras en familias con alta consanguinidad es probable que sean autocigotas para marcadores ligados al gen responsable de la enfermedad Mapeo en autocigotas 12