Tema 7
Anuncio

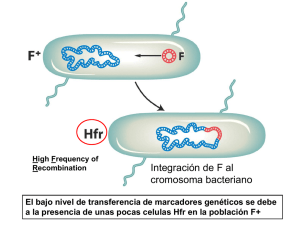
CARTOGRAFÍA EN BACTERIAS Y BACTERIÓFAGOS Las bacterias pueden intercambiar genes, aunque rara vez se aparean dos cromosomas completos. 3 Procesos: Protótrofo = capaz de vivir en medio mínimo (Ej: leu+) Auxótrofo = necesita suplementos al medio mínimo para vivir (ej: leu-) Nos permiten detectar recombinantes que están en muy baja frecuencia CONJUGACIÓN Bacterias F+ ó Hfr (donantes) y F- (receptoras) Bacterias F+ pueden transmitir el plásmido a las F-, transformándolas en F+ Bacterias F+ se transforman en F- por pérdida del plásmido El factor F se integra en el cromosoma bacteriano y la célula se convierte en Hfr Al desintegrarse células F’ (= F+ y algún segmento cromosómicosexducción CONJUGACIÓN (Hfr) : Mapas de tiempo Célula Hfr Integración Célula Hfr Célula Hfr Célula F- Célula F- Interrupción de la transferencia antes de que entre el factor F Corte en F e inicio de la transmisión (O) Célula Hfr Célula FTransferencia El punto de inserción varía distintos orígenes de transferencia Diversas cepas Hfr mapa circular completo 100 min. TRANSFORMACIÓN Bacteria competente Nº par de entrecruzamientos Entrada de ADN D A B C d c a b DNA extracelular se une al receptor de la célula competente Tras la replicación se forman dos células, una transformada y otra no DNA penetra y se separan las cadenas Heteroduplex: 2 cadenas distintas Una cadena actúa como transformante y se recombina con el cromosoma huésped, reemplazando a su región homóloga Recombinante dABc Sólo recombinan juntos los genes que están próximos TRANSFORMACIÓN- Frecuencias de recombinación Genes ligados: Para que haya DT sólo son necesarios 2 entrecruzamientos Genes independientes: Para que haya DT es necesaria una doble transformación, es decir, 4 entrecruzamientos Distancia de transformación = ST /(ST+DT) Transformantes simples/ total de transformantes Estas distancias no son aditivas ab + bc ≠ ac Cruce de tres puntos 4 regiones donde pueden ocurrir entrecruzamientos El menos frecuente (4X) nos indica cuál es el locus central TRANSDUCCIÓN GENERALIZADA El fago es adsorbido por la célula bacteriana Se lisa la célula huésped; se liberan los fagos Se ensamblan los fagos maduros Ocasionalmente un fago puede encapsular un fragmento de ADN = fago transductor Se inyecta el DNA del fago; se degrada el DNA del huésped Se replica el ADN del fago; se sintetizan los componentes proteicos del fago. Si este fago infecta a otra bacteria, este DNA puede, por recombinación, incorporarse al cromosoma bacteriano. TRANSDUCCIÓN GENERALIZADA DISTANCIA: PASOS: 1) Crecemos el fago en la cepa dadora 2) Infectamos con el lisado una cepa receptora 3) Seleccionamos para uno de los alelos donados 4) Se cuantifica el porcentaje de cotransductantes A mayor cotransducción, menor distancia ORDEN GÉNICO: Ej: Donante leu+, thr+, azir Experimento Marcador seleccionado Marcador no seleccionado 1 leu+ 50% azir: 2% thr+ 2 thr+ 3% leu+; 0% azir 3 leu+ y thr+ 0% azir Receptor: leu-, thr-, azis leu cerca de azi y lejos de thr thr -------- leu -- azi ó thr -------azi -- leu thr más cerca de leu que de azi thr -------- leu -- azi azi-thr muy lejos, no cotransducen No pueden ser encapsulados en el mismo fragmento TRANSDUCCIÓN ESPECIALIZADA - Algunos fagos (lisogénicos) pueden integrarse en sitios concretos del ADN bacteriano. - Fallo en la desinserción: lleva un fago defectivo y genes bacterianos situados a los lados del sitio de inserción. MAPAS EN VIRUS Calvas: lugares donde un solo virus a infectado a una bacteria Analizamos las características de las calvas: forma, tipo de huésped, tamaño, etc… Grandes vs pequeñas Claras vs turbias Usos: - Detección de genes muy próximos (estudios de complementación) - Mapas MAPAS EN VIRUS: Complementación Originalmente se denominaron cistrones; realmente son dos genes Si no son alélicas, cada fago produce uno de los productos se complementan y crecen. Si son alélicas, carecen de uno de los productos y no pueden crecer MAPAS EN VIRUS Como se recogen todos los productos, usamos los mismos métodos que para los eucariontes Número de progenie recombinante FR= Número total de la progenie FR% = FR *100 MAPAS EN VIRUS Comparando P y DR conocemos cuál es el gen central FRI%= FRAB%= recombinantes I *100 /total = (RI + DR)*100/total FRII%= FRCB%= recombinantes II *100 /total = (RII + DR)*100/total FRI,II% = FRAC%= recombinantes (I+II)*100/total = (RI + RII + 2*DR)*100/total MAPAS EN VIRUS Al poder analizar tantísimos descendientes, permitió detectar recombinantes dentro de un gen estructura fina del gen Se cruzan dos mutantes rII alélicas Fagos rII sólo infectaban a E.coli B, no pueden infectar a E coli K12 (λ) FR = 2 * rec rII+ / (total) Recombinantes rII+ Total Benzer demostró en 1955 que un gen no es una partícula indivisible, sino que consta de unidades de mutación y de recombinación que están dispuestas en un orden concreto. Hoy sabemos que son los nucleótidos. MAPAS EN VIRUS Mapas por deleción: Si la mutante solapa con la deleción, no puede recombinar y no crecen