Respuesta a señales ambientales
Anuncio

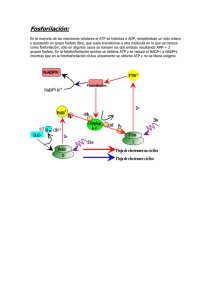
Respuesta a señales ambientales Las bacterias pueden –alterar su morfología -la producción del flagelo -el metabolismo celular - la transcripción de genes -comportamiento celular En respuesta a los cambios ambientales Las bacterias se adaptan y sobreviven ante cada cambio ambiental sofisticados mecanismos de detección con los cuáles las bacterias monitorean el ambiente y transmiten la señal Cambio transcripcional Enzimas o componentes celulares Sistema de detección: Sistemas de dos componentes(TCS) Monitorea el medio HK sensor Transmite la señal Regulador de respuesta Blancos específicos Destino de la señal TCS regulan diversas respuestas en organismos diferentes (Eubacteria, Arquea , Eucariotas) Adquisición de nutrientes: P, N C Metabolismo energético Virulencia: producción de toxinas, adherencia, transferencia de plásmidos Adaptación a cambios físicos o químicos del medio ambiente: pH, osmolaridad, temperatura Vías complejas de desarrollo: esporulación, fructificación, Distribución genómica: una sola célula puede tener mas de un TCS Ejemplos de sistemas de dos componentes oEnvZ/OmpR – expresión de porinas in E. coli oCheA/CheY, CheB – rotación del flagelo en E. coli oArcB/ArcA- estado redox oNarR/NarQ- metabolismo de nitrato y nitrito oNtrB/NtrC – asimilación de nitrógeno en distintas bacterias oVanS/VanR – resistencia a Vancomicina oDesK/DesR- adaptación de las membranas a T en B. subtilis oKinA, KinB/SpoOF, SpoOA – esporulación en Bacillus subtitlis oQuorum sensing oVirA/VirG- virulencia en Agrobacterium tumefaciens TCS en dos patógenos. Photorhabdus luminescens y Yersinia enterocolitica Ambos patógenos comparten una superposición de TCS que juegan un papel en la interacción con el huésped. Transducción de la señal en el TCS Sistema de transducción de señales que utiliza la transferencia de un grupo fosfato para controlar la transcripción de genes o una actividad proteica 1-Histidin –quinasa o quinasa sensora que posee HK a) Dominio sensor: monitorea el estímulo ambiental. Variable en longitud y sec. de AA confiriendo especificidad para diferentes estimulos. b) Dominio transmisor: Se autofosforila seguido de la detección del estímulo. Altamente conservado. Contiene un residuo His que es fosforilado de manera ATP-dependiente y segmentos cortos de aminoácidos conservados , en particular dos motivos ricos en glicina involucrados en la unión a (NG1FG2 ). 2. Regulador de respuesta: contiene un N-terminal conservado ( dominio receptor) que es fosforilado por la quinasa en un residuo de aspartato, activando el dominio de salida en el C- terminal 3.El sistema incluye una fosfatasa que desfosforila el RR: puede ser la HK, el RR o una proteína separada. 4. Es altamente específico si bien existen algunos ejemplos de “cross talk” Arquitectura modular de TCS con más detalles Dhp CA REC Arquitectura del módulo transmisor DHp D=Dimerización H=his P=fosfotransfer Histidin and phosphotransfer domain CA High Temperature UFAs (disordered) Low Temperature (ordered) Adaptation (disordered) Chemical structure and Physical Properties of Lipids Bacilli O O R O2 + 2 H+ desaturase 2 H2O O O R Topology model of B. subtilis desaturase coded by the des gene Aguilar et al. (1998) J. Bacteriol. 180:2194 Diaz et al. (2002) J. Biol. Chem. 277:48099 37ºC 25ºC 25ºC ½ O2 H20 pdes lacZ OD(525nm) 100 1.0 50 37ºC 20ºC (MU) b-galactosidase (MU) b-galactosidase 150 1.5 37ºC 0.5 20ºC 0 0 2 4 6 Time (h) 8 10 37ºC 20ºC des is transiently induced 0 15 30 60 90 180 360 540 des mRNA levels des mRNA 40 30 20 10 0 0 60 120 180 240 300 360 420 480 540 Time (min) min DOS COMPONENTES Señal PO3 H H PO3 ATP ATP D Signal transduction pathway for regulation of membrane fluidity in Bacillus subtilis. DesK H H ATP ATP DesKC DesKC 154 184 195 H 289 293 N 318 324 G1 334 339 355 359 G2 G3 370 DesKC enzymatic activities 10” 30” 1’ 5’ 15’ 30’ DesKC-P H H 0 10” 30” 1’ 5’ AUTOKINASE 15’ GST-DesR-P PHOSPHOTRANSFERASE DesKC-P ATP ATP DesKC 0 10” 30” 1’ 5’ 15’ 30’ GST-DesR-P DesKC-P PHOSPHATASE His188 is not essential for DesKC phosphatase activity 0 10” 30” 1’ 5’ 15’ 30’ V V GST-DesR-P ATP DesKCV ATP DesKCV188 0 10” 30” 1’ 5’ 15’ 30’ 120 P % radioactivity 100 80 DesRP 60 Pi 40 20 0 0 100 200 300 400 500 600 700 800 1800 Time (s) Protein-P The catalytic domain of DesK CA H DHp H DHp V V ATP ATP CA ATP ATP DesKC HK domain DesKCV188 HK domain Three distinct conformational states of DesKC DesKCD174 DesKCH188V DesKC-P 2-helix parallel coiled-coil in the N-terminal linker of the phosphatase state L160 L164 A167 I171 R170 L174 L177 V188 I183 parallel coiled-coil L187 four-helix bundle Mutation induces helical shift in four-helix bundle DesKCD174 DesKCH188V DesKCD174 DesKCV188 Quimiotaxis Los microorganismos son capaces de sensar compuestos químicos en su medio ambiente y moverse hacia ellos QTX (+) o alejarse de ellos QTX(-) Es fundamental 1-Movimiento aleatorio 2-Adaptación (memoria celular) El movimiento no es continuo…tumble and run” Las bacterias nadan y viran gracias a que el flagelo forma una banda propelente detrás de la célula Movimientos en ausencia y presencia de quimioefectores Mov. aleatorio en ausencia de estímulos Gradiente de atractante. Se altera la frecuencia de tumbos, mov. aleatorio pero en dirección del atractante Esto dura un tiempo limitado hasta que la bacteria se adapta al estímulo Tumble Attractant Tumble Run Run No attractant present: Random movement © 2015 Pearson Education, Inc. Attractant present: Directed movement Figure 2.57 © 2015 Pearson Education, Inc. Figure 2.58a-d © 2015 Pearson Education, Inc. Figure 2.58e La quimiotaxia se pone en marcha cuando: 1- el quimioefector se une a 2-receptor específico 3-y se desencadena un proceso de transducción intracelular de la señal 4-la señal llega al conmutador flagelar que determina el sentido de rotación (CW) 5-La bacteria capta el gradiente espacial como si fuera un gradiente temporal 6-La bacteria compara mediante el mismo receptor la diferencia de concentraciones 7-la bacteria vuelve al patrón aleatorio de movimiento inicial-ADAPTACION Sistema sensorial de la bacteria MCPs: quimioreceptor o proteína aceptora de metilos CheA: quinasa sensora CheY: Regulador de respuesta Chew: Proteína accesoria CheR: MCP-metil trasferasa específica CheB: MCP-metil estearasa específica CheZ: fosfatasa Slide 2 MCP Flagellar motor CheR CheW CheA CheY CheZ CheB Cytoplasm Flagellum © 2015 Pearson Education, Inc. Figure 7.19 Slide 3 Repellents bind to MCP and trigger phosphorylation of CheA-CheW complex. MCP Flagellar motor CheR CheW CheA ATP ADP CheY CheZ CheB Cytoplasm Flagellum © 2015 Pearson Education, Inc. Figure 7.19 Slide 4 Repellents bind to MCP and trigger phosphorylation of CheA-CheW complex. MCP CheR CheW CheA CheB P ATP CheA-CheW phosphorylate CheY and CheB. Flagellar motor ADP CheY CheY P CheZ CheB Cytoplasm Flagellum © 2015 Pearson Education, Inc. Figure 7.19 Slide 5 Repellents bind to MCP and trigger phosphorylation of CheA-CheW complex. MCP CheR CheW CheA CheB P ATP CheA-CheW phosphorylate CheY and CheB. ADP CheY CheY Flagellar motor CheY-P binds to flagellar switch. P CheZ CheB Cytoplasm Flagellum © 2015 Pearson Education, Inc. Figure 7.19 Slide 6 Repellents bind to MCP and trigger phosphorylation of CheA-CheW complex. MCP CheR CheW CheA CheB P ATP CheA-CheW phosphorylate CheY and CheB. ADP CheY CheY Flagellar motor CheY-P binds to flagellar switch. P CheZ CheB Cytoplasm CheZ dephosphorylates CheY-P. Flagellum © 2015 Pearson Education, Inc. Figure 7.19 Slide 7 Repellents bind to MCP and trigger phosphorylation of CheA-CheW complex. MCP CheR MCP is both methylated and demethylated. CheB P CheW CheA ATP CheA-CheW phosphorylate CheY and CheB. ADP CheY CheY Flagellar motor CheY-P binds to flagellar switch. P CheZ CheB Cytoplasm CheZ dephosphorylates CheY-P. Flagellum © 2015 Pearson Education, Inc. Figure 7.19 Estado basal: equilibrio, sin efector. Hay una cantidad suficiente de CheA-P que Posibilita el movimiento aleatorio El grado de ocupación de los G del MCP es baj Constitutivo Activado efector Grupos metilos Inactivo Grupos fosfatos Balance entre la actividad de CheA y CheZ Nado corto, viraje orientativo Módulo de conversión Flexion efector En presencia del efector, se induce el cambio conformacional, se inactiva quinasa, disminuye CheY-P y CheB-P, el rotor disminuye la frecuencia de tumbos. efector Inactivo Constitutivo Grupos metilos Inactivo Nado corto suave, viraje menos frecuente El receptor metilado Adaptación Inactivo Constitutivo Activado Grupos metilos Grupos fosfatos Activado Inactivo Constitutivo Grupos metilos Grupos fosfatos Activado Inactivo Constitutivo Grupos metilos Grupos fosfatos