Clonagem molecular - Biologia Molecular e Genética
Anuncio
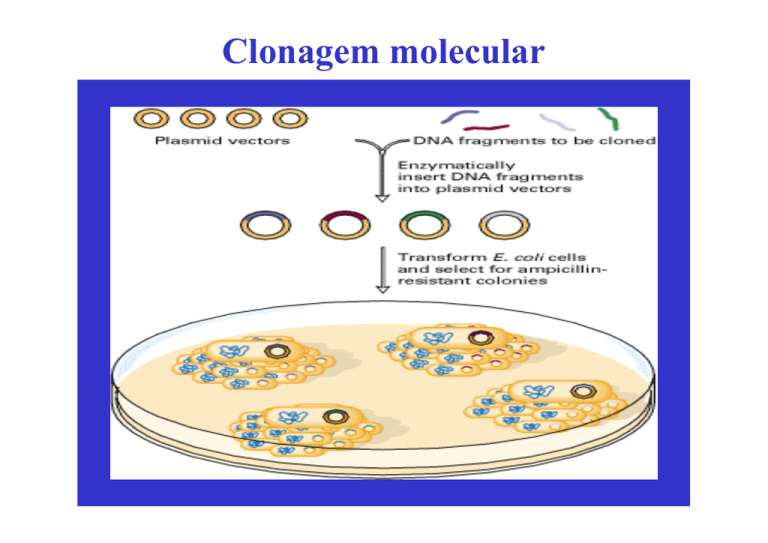
Clonagem molecular Tópicos Significado e objectivos da clonagem molecular Etapas da clonagem molecular Sistemas de clonagem em E. coli de fragmentos de diferentes dimensões Sistemas de clonagem em E. coli de produção de DNA de cadeia simples Preparação de bibliotecas genómicas Preparação de bibliotecas de cDNA Desenvolvimento da clonagem molecular An Introduction to Genetic Engineering Definição de clonagem molecular A clonagem molecular é o processo de construção de moléculas de DNA recombinante e da sua propagação em hospedeiros apropriados que possibilitam a selecção do DNA recombinante. Esta tecnologia permite estudar os genes e os seus produtos, obter organismos transgénicos e realizar terapia génica. Expressões utilizadas com o mesmo significado: clonagem molecular engenharia genética manipulação génica clonagem génica tecnologia do DNA recombinante modificação genética nova genética Representação esquemática da clonagem molecular Genetics a molecular approach Objectivos gerais da clonagem molecular (1) Recombinant DNA. Principles and Methodologies Objectivos gerais da clonagem molecular (2) A clonagem molecular permite isolar e amplificar uma sequência particular de DNA a partir de uma mistura complexa de fragmentos, porque se formam moléculas de DNA recombinante independentes que replicam numerosas vezes no hospedeiro. Papel dos ácidos nucleicos na clonagem molecular Os ácidos nucleicos são as moléculas centrais da clonagem molecular: • O DNA é utilizado como vector de clonagem ou como inserto • O cDNA, obtido a partir de mRNA, é utilizado como inserto • DNA e RNA obtidos por clonagem molecular são utilizados como sondas de hibridação na identificação, quantificação e caracterização de outros ácidos nucleicos Principais etapas da clonagem molecular (1) Construção de DNA recombinante Transformação Principais etapas da clonagem molecular (2) Amplificação Isolamento de clones de DNA recombinante Especificação das etapas da clonagem molecular Preparação do DNA vector Preparação do DNA dador Ligação de DNA vector e DNA dador Selecção do sistema vector/hospedeiro Introdução de DNA recombinante no hospedeiro Selecção de clones recombinantes Identificação de clones recombinantes Preparação de DNA vector e DNA dador Preparação de DNA vector Digestão com enzima(s) de restrição de tipo II Métodos de produção de fragmentos de DNA dador Digestão com enzimas de restrição de tipo II Digestão parcial com DNase I Quebra mecânica controlada Síntese enzimática (cDNA, PCR) Síntese química de oligodesoxirribonucleótidos Métodos de modificação das extremidades do DNA Preenchimento parcial de extremidades 3’ recuadas Preenchimento total de extremidades 3’ recuadas Remoção de extremidades 3’ projectadas Adição de linkers Adição de adaptadores Adição de caudas de homopolímeros Desfosforilação Nomenclatura das enzimas de restrição Nomenclatura das enzimas: As enzimas são denominadas por abreviaturas das estirpes bacterianas a partir das quais foram isoladas. A primeira letra maiúscula corresponde à inicial do nome genérico e as duas letras minúsculas às inicias do restritivo específico. A numeração romana indica a ordem cronológica de isolamento da enzima na estirpe produtora. Exemplo: AluI foi a primeira enzima de restrição a ser isolada de Arthrobacter luteus. As sequências de reconhecimento são escritas de 5’ → 3’ (pode estar representada apenas uma das cadeias) e os locais de clivagem são indicados por setas. R. J. Roberts, 1988, Nucl. Acids Res. 16(suppl):271. Digestão enzimática As enzimas de restrição catalisam a hidrólise das ligações fosfodiéster do DNA, originando extremidades 5’P e 3’OH (excepto NciI que origina extremidades 3’P e 5’OH). A ligação que é clivada está representada a rosa. Especificidades das enzimas de restrição de tipo II Tipos de extremidades: BamHI, EcoRI e XhoI clivam a 5’ do eixo de simetria, originando extremidades coesivas, 5’P projectadas. HaeIII cliva segundo o eixo de simetria, originando extremidades cegas (sem projecções). HhaI cliva a 3’ do eixo de simetria, originando extremidades coesivas, 3’OH projectadas. As sequências de reconhecimento são, em geral, constituídas por quatro a seis pares de nucleótidos, e palindrómicas, isto é, quando lidas na mesma orientação em ambas as cadeias, 5’→3’ ou 3’→5’, são idênticas. A clivagem ocorre dentro da sequência de reconhecimento ou num local próximo. As setas indicam os locais de corte e os símbolos a verde assinalam o eixo de simetria. EcoRI e metilase EcoRI As bactérias que produzem endonucleases de restrição também contêm as enzimas de modificação correspondentes que metilam bases no local de reconhecimento das endonucleases. A metilase EcoRI (M.EcoRI) catalisa a adição de um grupo metilo a duas adeninas da sequência reconhecida por EcoRI. A sequência metilada não é clivada por EcoRI, assegurando que o DNA da célula que produz esta enzima de restrição não é destruído. Isosquisómeros Reconhecem a mesma sequência e clivam nos mesmos locais AccIII T↓CCGGA AGGCC↑T e BspE1 T↓CCGGA AGGCC↑T Estas enzimas geram extremidades compatíveis com XmaI C↓CCGGG GGGCC↑C Reconhecem a mesma sequência e clivam em locais diferentes Acc65I G↓GTACC CCATG↑G e KpnI GGTAC↓C C↑CATGG XmaI C↓CCGGG GGGCC↑C e SmaI CCC↓GGG GGG↑CCC Reconhecem a mesma sequência e têm sensibilidades diferentes à metilação DpnII e MboI ↓GATC CTAG↑ Sau3A1 ↓GATC CTAG↑ DpnI e ↓Gm6ATC CTAm6G↑ ↓Gm6ATC CTAm6G↑ Isosquisómeros são enzimas que reconhecem a mesma sequência de nucleótidos. Métodos de modificação das extremidades do DNA (1) 1. Preenchimento parcial de extremidades 3’ recuadas dATP 5’_________ OH 3’ dGTP 3’_________TCGA p 5’ → Hind III Klenow 5’ _________ AG OH 3’ 3’ _________ TCGA p 5’ dCTP 5’_________ OH 3’ dTTP 3’_________GATC p 5’ → XbaI Klenow 5’ _________ CT OH 3’ 3’ _________ GATC p 5’ A extremidade Hind III modificada é compatível com a extremidade XbaI modificada Métodos de modificação das extremidades do DNA (2) 2. Preenchimento total de extremidades 3’ recuadas dATP 5’_________G OH 3’ dTTP 5’ _________ GAATT OH3’ 3’ _________ CTTAA p 5’ 3’_________CTTAA p 5’ → Klenow dATP dCTP dGTP 5’_________A OH 3’ dTTP 5’ _________ AAGCT OH 3’ 3’ _________ TTCGA p 5’ 3’_________TTCGA p 5’ → Klenow As extremidades coesivas são convertidas em extremidades cegas Métodos de modificação das extremidades do DNA (3) 3. Remoção de extremidades 3’ projectadas As enzimas utilizadas são: • Polimerase de DNA do fago T4 • Polimerase Klenow • Nuclease S1 • Nuclease mung bean As extremidades coesivas são convertidas em extremidades cegas 4. Adição de linkers ↓ AatII GGACGTCC CCTGCAGG ↑ Linker fosforilado 5’-d (pGGACGTCC) -3’ Linker não fosforilado 5’-d (GGACGTCC) -3’ As extremidades cegas são convertidas em extremidades coesivas Métodos de modificação das extremidades do DNA (4) 5. Adição de adaptadores - Adaptador com extremidades coesivas: XmnI 5’ .......AATTCGAACCCCTTCG...................3’ 3’ .....................GCTTGGGGAAGCCTAG.......5’ EcoRI BamHI Uma extremidade coesiva é convertida numa extremidade coesiva diferente - Adaptador com uma extremidade cega: XmnI 5’ .......AATTCGAACCCCTTCG.......3’ 3’ .....................GCTTGGGGAAGCp..........5’ EcoRI Uma extremidade coesiva é convertida em extremidade cega ou vice-versa 6. Adição de caudas de homopolímeros As extremidades cegas são convertidas em extremidades coesivas pela transferase terminal Métodos de modificação das extremidades do DNA (5) 7. Desfosforilação Remoção dos grupos 5’P das extremidades do DNA pela fosfatase alcalina para impedir a recircularização do vector (aumenta a eficiência de clonagem) ou a ligação intermolecular do DNA dador, em particular, a ligação de fragmentos de DNA de regiões genómicas diferentes. Ligação de DNA vector e DNA dador Construção de DNA recombinante Estratégias de ligação de DNA vector e DNA dador Clonagem não dirigida (DNA vector EcoRI + DNA dador EcoRI) Clonagem dirigida (DNA vector EcoRI/PstI + DNA dador EcoRI/PstI) Construção de novos locais de clivagem Construção de DNA recombinante A ligase de DNA é a enzima que liga extremidades 3’ OH e 5’ P adjacentes, produzindo uma molécula de DNA recombinante. Ligação intermolecular do DNA dador e intramolecular do DNA vector A tendência para a circularização de moléculas individuais é mais acentuada quando a concentração de DNA é baixa e as possibilidades de colisão entre diferentes moléculas com extremidades coesivas complementares é reduzida. Selecção do sistema vector/hospedeiro Vectores procarióticos de clonagem em E. coli Características dos vectores de clonagem Tipos de vectores e suas aplicações Plasmídios Vectores derivados do fago lambda Cosmídios Vectores derivados do fago M13 Fagemídios Cromossomas artificiais derivados do fago P1 (PACs) Cromossomas artificiais bacterianos (BACs) Vectores shuttle Estirpes de E. coli Características dos vectores de clonagem Os vectores de clonagem são moléculas de DNA que permitem a propagação do inserto no hospedeiro. Características fundamentais - São moléculas de DNA de cadeia dupla - Têm capacidade de replicação autónoma no hospedeiro - Possuem locais de restrição únicos para clonagem - Possuem pelo menos uma marca genética de selecção Características adicionais vantajosas - Baixa massa molecular - Mais do que um sistema de selecção Polylinker ou MCS Este polylinker inclui uma cópia dos locais de reconhecimento, indicados por chavetas, de cada uma das 10 enzimas de restrição assinaladas. Está representada apenas uma cadeia. Os polylinkers são sintetizados quimicamente e inseridos nos vectores de clonagem. Em condições de reacção apropriadas, a inserção de um único fragmento de restrição é maximizada. Na clonagem de fragmentos EcoRI num vector clivado com EcoRI, são reconstituídos dois locais EcoRI no DNA recombinante. MCS (multiple cloning site) – local múltiplo de clonagem Vector plasmídico pUC19 A origem de replicação (ori) deriva do plasmídio semelhante a ColE1, pMB1. A porção do gene lacZ incluída no vector codifica o fragmento amino-terminal da β-galactosidase. O MCS está inserido de modo a preservar a grelha de leitura e a sua correcta expressão. Clonagem de DNA num vector plasmídico (1) Clonagem de DNA num vector plasmídico (2) Os clones recombinantes são colónias bacterianas. Ciclo de vida do fago λ Mapa simplificado do genoma do fago λ O genoma do fago λ tem cerca de 50 kb. Na extremidade esquerda estão localizados os genes que codificam as proteínas da cabeça e cauda; os genes que codificam proteínas envolvidas no ciclo lítico mapeiam na extremidade direita. A região central do genoma pode ser removida ou substituída por DNA exógeno sem afectar a capacidade de replicação do fago, permitindo a inserção de DNA exógeno até ≈25 kb. Encapsidação in vivo de DNA do fago λ Durante o estádio tardio da infecção fágica formam-se longas moléculas de DNA, denominadas concâtemeros; estas moléculas multiméricas consistem de cópias do genoma do fago λ ligadas pelas extremidades e separadas por locais cos (vermelho). Figure 7-11. © 2000 by W. H. Freeman and Company. Encapsidação in vitro de DNA de λ recombinante Sistema baseado em extractos celulares de duas estirpes de E. coli lisogénicas de fagos λ mutantes. Proteína E – proteína principal da cápside Proteína D – proteína secundária da cápside 38-53 kb Sistema baseado numa única estirpe, E. coli C λ cI857 cos2 Sam7, lisogénica do fago λ mutante cos2 (deleção de 22 pb de cosN que impede a formação de placas fágicas de DNA endógeno). Vector λ de inserção Clonagem de DNA num vector λ de substituição As enzimas Sau3A e BamHI geram extremidades coesivas compatíveis. Os fagos recombinantes infectam E. coli, originando em meio sólido placas fágicas (clones recombinantes). Figure 7-12. © 2000 by W. H. Freeman and Company Cosmídio O cosmídio é um plasmídio que contém um local cos. MboI e BamHI geram extremidades coesivas compatíveis A clonagem em cosmídios conjuga a elevada eficiência de infecção associada à clonagem no fago λ com a facilidade de manipulação de DNA plasmídico, dado que o DNA recombinante replica como um plasmídio. Clonagem de DNA num cosmídio Da infecção de E. coli com os viriões recombinantes resultariam quatro tipos diferentes de colónias, embora esteja representada apenas uma. Figure 7-16. © 2000 by W. H. Freeman and Company Características do fago M13 - M13 é um fago filamentoso - Tem um genoma circular de DNA de cadeia simples (cadeia +) de 6,4 kb - Infecta E. coli F’ - O ciclo de vida permite a sua utilização como vector de clonagem - As partículas virais são libertadas no meio de cultura sem lise de E. coli (≈ 200 partículas/célula/geração) - Forma placas fágicas análogas às do fago λ - 90% do genoma codifica proteínas indispensáveis à realização do ciclo de vida Características dos vectores M13 (série mp) - É utilizada a forma replicativa (RF) do DNA vector - O DNA recombinante é introduzido por transformação em células de E. coli F’ - Existem dois tipos de vector com o polylinker nas duas orientações - Adjacente ao polylinker está clonada a região 5’ do operão lac - A selecção de recombinantes é baseada na α-complementação - O DNA recombinante de cadeia simples é extraído das partículas fágicas e utilizado como molde para sequenciação, na obtenção de sondas de hibridação e em mutagénese sítio-dirigida - A capacidade de clonagem oscila entre 300-400 nucleótidos (sequências clonadas maiores do que 1000 nucleótidos são instáveis, originando deleções durante a propagação dos fagos) Clonagem de DNA num vector M13 Transformação RF – Forma replicativa, de cadeia dupla Clonagem de DNA num fagemídio O fagemídio é um vector híbrido que possui uma origem de replicação plasmídica e uma origem de replicação fágica (M13, f1). Capacidade de clonagem dos vectores de E. coli Tipo de vector DNA clonado (kb) Plasmídio Fago λ Cosmídio PAC (cromossoma artificial derivado do fago P1) BAC (cromossoma artificial bacteriano) 10 25 45 100 300 Vector shuttle Genótipos de estirpes de E. coli All genes in the bacterium are presumed to be in the wild-type state, except for those listed, which are mutant alleles carried by that bacterium. Genes listed on the F’ episome, however, represent wild-type alleles unless specified otherwise. Strains are λunless specified otherwise. BL21(DE3) F-, ompT, hsdSB, (rB-, mB-), dcm, gal, λ(DE3) BL21(DE3)pLysS F-, ompT, hsdSB, (rB-, mB-), dcm, gal, λ(DE3), pLysS (Cmr) DH5α φ8OdlacZ∆M15, recA1, endA1, gyrA96, thi-1, hsdRl7 (rK-, mK+), supE44, relA1, deoR, ∆(lacZYA-argF)U169 Significado das marcas genéticas de estirpes de E. coli (1) Symbol Description Effect dam Adenine methylase mutation Blocks methylation of adenine residues in the sequence 5’…GmATC…3’. dcm Cytosine methylase mutation Blocks methylation of cytosine in the sequence 5’…CmCAGG...3’or 5’...CmCTGG...3’. endA1 Endonuclease mutation Improves quality of plasmid DNA isolations. hsdR (rK- , mK+) Restriction minus, modification positive Allows cloning without cleavage of transformed DNA by endogenous restriction endonucleases. DNA prepared from this strain can be used to transform rK+ E. coli strains. hsdS20 (rB-, mB-) Restriction minus, modification minus lacIq Allows cloning without cleavage of transformed DNA by endogenous restriction endonucleases. DNA prepared from this strain is unmethylated by the hsdS2O melhylases. Overproduction of the lac repressor protein Leads to high levels of the lac repressor protein, inhibiting transcription from the lac promoter. Significado das marcas genéticas de estirpes de E. coli (2) Symbol Description Effect lon Deletion of lon protease Reduces proteolysis of expressed fusion proteins. mcrA Mutation in restriction system Blocks restriction of DNA methylated at the sequence 5’..GmCGC.,3’. mcrB Mutation in restriction system Blocks restriction of DNA methylated at the sequence 5’..AGm CT. .3’. ompT Mutation of protease VII, an outer membrane protein Reduces proteolysis of expressed fusion protein. P2 P2 bacteriophage lysogen present in host λ phages containing the red and gam genes of λ are growth inhibited by P2 lysogens . recA1, recAl3 Mutation in recombination Prevents recombination of introduced DNA with host DNA, ensuring stability of inserts. Inserts are more stable in recAl than recAl3 hosts. Características do hospedeiro do fago M13 GENOMA: ∆(lac-proAB)[F’ traD36, proAB, lacIqlacZ∆M15] ∆ (lac-proAB) Deleção cromossómica de um segmento de DNA que inclui o operão lac e os genes vizinhos que codificam enzimas envolvidas na biossíntese da prolina. proAB Genes proAB existentes no epissoma F’, podendo complementar a auxotrofia para a prolina do hospedeiro ∆(lac-proAB). A manutenção de F’ no hospedeiro tem que ser garantida isolando a estirpe em MM/Glu. lacZ∆M15 Deleção parcial no gene lacZ, correspondente aos aa 11-41 da β-galactosidase. Permite a α-complementação. traD36 Mutaçäo no factor de transferência que evita a transferência do epissoma F’ (por conjugacäo). Métodos de introdução de DNA no hospedeiro (1) Transformação bacteriana Transfecção Electroporação Microinjecção Bombardeamento com microprojécteis Infecção viral Métodos de introdução de DNA no hospedeiro (2) Métodos de selecção de clones recombinantes (1) Selecção indirecta Inactivação de genes de resistência a antibióticos Selecção directa Gene lacZ Selecção pela cor - α-complementação Fenótipo Spi- Métodos de selecção de clones recombinantes (2) A inserção de DNA em pBR322 é detectada por inactivação de um gene de resistência a um antibiótico (tetR), indicada pelo fenótipo sensível TetS. Em pUC18 ocorre inactivação da βgalactosidase (lacZ’), resultando na incapacidade de converter o substrato artificial X-Gal num corante azul. Métodos de identificação de clones recombinantes Análise do padrão de restrição Sequenciação de DNA Hibridação de ácidos nucleicos Complementação génica de estirpes mutantes PCR Análise de produtos génicos (reconhecimento imunológico) Identificação do clone recombinante relevante Análise do padrão de restrição Identificação de clones com fragmentos de DNA inseridos em orientações opostas. Identificação de clones recombinantes por hibridação de colónias Identificação de clones recombinantes por hibridação de placas fágicas Hibridação Southern Construção de sondas degeneradas Uma curta sequência de aminoácidos da proteína relevante é utilizada para construir um conjunto de sondas oligonucleotídicas que servem para identificar o gene que codifica a proteína. Uma das sondas do conjunto de sondas degeneradas terá um emparelhamento perfeito com o gene. Figure 12-11. From H. Lodish, D. Baltimore, A. Berk, S. L. Zipursky, P. Matsudaira, and J. Darnell, Molecular Cell Biology, 3d ed. Copyright © 1995 by Scientific American Books, Inc.. Triagem de bibliotecas YAC por PCR Identificação de clones recombinantes com anticorpos Figure 12-12. Recombinant DNA, 2d ed. Copyright © 1992 Estratégias de clonagem Bibliotecas genómicas Chromosome walking Bibliotecas de cDNA Preparação de uma biblioteca de DNA genómico A digestão enzimática parcial do DNA dador origina fragmentos sobreponíveis, isto é, com sequências de DNA comuns. Chromosome walking (1) Esta técnica é utilizada para isolar fragmentos de DNA sobreponíveis partindo de um fragmento de DNA previamente clonado que mapeia próximo do gene de interesse (representado a vermelho), permitindo identificar o clone que contém o gene. Figure 12-15. From J. D. Watson et al., 1992, Recombinant DNA, 2d ed., W. H. Freeman and Company, p. 128. Chromosome walking (2) Neste exemplo, os fragmentos de DNA cromossómico estão clonados no fago λ. O clone inicial da biblioteca genómica, representado a verde, é utilizado como sonda na triagem de sequências sobreponíveis presentes noutros clones da biblioteca. O walk é conduzido em ambas as direcções, dado que em geral não se sabe a que distância o gene de interesse está do clone inicial, ou se está para a esquerda ou para a direita. Para simplificar está representado um walk unidireccional. Os clones isolados no walk são utilizados como sondas em hibridações Southern com DNA genómico de mutantes. Esta análise permite detectar deleções cromossómicas e rearranjos no DNA, mas normalmente não detecta mutações pontuais, a não ser que alterem as sequências reconhecidas pelas enzimas de restrição utilizadas na análise. Deste modo, o clone de DNA contendo a região correspondente ao gene de interesse é identificado. Normalmente, um walk envolve mais do que as três etapas representadas na figura. Nalguns casos, parte do gene pretendido pode já estar clonado, sendo o walking de DNA necessário para isolar o gene completo. Iniciar walks em paralelo a partir de vários clones diferentes (estes walks serão eventualmente ligados), acelera o processo de isolamento de clones sobreponíveis em longas extensões contíguas de DNA. Isolamento de mRNA (1) Isolamento de mRNA (2) A cromatografia de afinidade em coluna oligo-dT é utilizada no isolamento de mRNA eucariótico. O RNA citoplásmico é constituído fundamentalmente por RNAs ribossómicos (rRNAs) e RNAs de transferência (tRNAs). Os RNAs mensageiros (mRNAs), são muito menos abundantes, mas têm caudas poli-A que hibridam com oligo-dTs covalentemente ligados à matriz da coluna. Após hibridação, os rRNAs e tRNAs são removidos da coluna por lavagem, sendo os mRNAs depois eluídos com um tampão de baixa concentração salina. A preparação de mRNA purificado resultante contém muitas moléculas de mRNA diferentes que codificam diferentes proteínas. Preparação de cDNA (1) Para se obter uma representação mais normal das sequências, em vez do primer oligo(dT), são utilizadas misturas de primers oligonucleotídicos aleatórios. Preparação de cDNA (2) Preparação de uma biblioteca de cDNA num vector λ (1) Preparação de uma biblioteca de cDNA num vector λ (2) Uma mistura de mRNAs é utilizada para produzir cDNAs correspondentes a todos os mRNAs celulares (etapas 1 a 3). Os cDNAs de cadeia simples (verde claro) são depois convertidos em cDNAs de cadeia dupla, que são tratados com metilase EcoRI para impedir posterior digestão com EcoRI (etapas 4 a 6). Os cDNAs de cadeia dupla previamente protegidos são ligados a linkers EcoRI de cadeia dupla sintéticos, em ambas as extremidades, e depois clivados com a enzima de restrição correspondente, produzindo cDNAs com extremidades coesivas (letras vermelhas). Os cDNAs com extremidades coesivas são inseridos em vectores λ de clonagem, e os viriões recombinantes resultantes são utilizados para infectar E. coli que é depois plaqueada em meio sólido nutritivo (etapas 7 a 9). Obtenção de clones genómicos e de cDNA por PCR e RT-PCR (1) Obtenção de clones genómicos e de cDNA por PCR e RT-PCR (2) (A) Os clones genómicos podem ser obtidos por PCR a partir de DNA cromossómico extraído das células. São adicionados primers que ladeiam a região de DNA que se pretende clonar, sendo amplificado apenas o DNA entre os primers (incluindo os primers). Esta técnica permite obter selectivamente uma curta extensão de DNA cromossómico. (B) Os clones de cDNA podem ser obtidos por RT-PCR a partir de mRNA extraído das células, e purificado por cromatografia de afinidade em coluna oligo-dT. O primeiro primer (oligo-dT) é depois adicionado à população de mRNAs, sendo utilizada a transcritase reversa para produzir a cadeia de cDNA complementar. Após adição do segundo primer, a molécula de DNA de cadeia simples é amplificada através de muitos ciclos de PCR, originando cDNA de cadeia dupla. Em ambos os tipos de clonagem, a sequência de nucleótidos de pelo menos parte da região que se pretende clonar deve ser previamente conhecida. Diferenças entre clones de DNA genómico e clones de cDNA (1)