Reacción de Marcaje
Anuncio

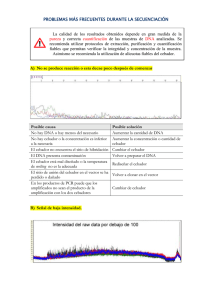
ACTIVIDADES PRÁCTICAS SECUENCIACIÓN DEL DNA MEDIANTE LA TÉCNICA DE TERMINACIÓN DE LAS CADENAS POR DIDESOXINUCLEOTIDOS. ANÁLISIS DE SECUENCIAS. Objetivos: Conocer de manera teórica la teçnica de secuenciación con didesoxinucleótidos. Adquirir los principios básicos para el análisis de las secuencias obtenidas. INTRODUCCION Hace alrededor de quince años, no existia ningún medio simple para determinar la secuencia de cadenas cortas de DNA de solo algunos nucleótidos. En la actualidad las secuencias de fragmentos de DNA de varias centenas de nucléotidos pueden ser obtenidas en dos o tres dias gracias a las enzimas de restricción y mas recientemente a la téçnica de reacción en cadena de la polimerasa (PCR) que permite disponer de las cantidades necesarias del DNA de interés. Estos progresos espectaculares se deben a dos técnicas fundamentales, una desarrollada en Harvard por Maxam y Gilbert y la otra puesta a punto en Cambridge, Inglaterra, por Sanger. Sobre esta última, la mas utilizada en la actualidad, trataremos en esta práctica. Técnica de Secuenciación de Sanger (terminación de las cadenas con didesoxinucleótidos) La técnica se basa en la utilización de una DNA polimerasa, en sus inicios el fragmento mayor (Klenow) de la DNA polimerasa I de E. coli la cual sintetiza in vitro fragmentos de diferetes tamaños que al ser posteriormente separados en un gel de poliacrilamida permiten la lectura directa del orden de los nucleótidos en la cadena. En esta técnica el DNA monocatenario es utilizado como matriz para la sintesis de cadenas complementarias las cuales son alargadas por la DNA pol a partir de un punto único donde se encuentra el oligonucleótido que sirve de cebador para la enzima. En la reacción se emplean además de los cuatro nucleótidos precursores normales , análogos de nucleótidos (los 2 3didesoxinucleosidos 5-trifosfatos (ddNTPs), que impiden la continuidad de la polimerización, de ahí el nombre de la técnica. Estos ddNTPs no poseen el grupo 3-OH y por lo tanto no permiten la formación del enlace fosfodiester con los otros precursores. Un didesoxinucleótido adicionado al medio de reacción es incorporado eficientemente y de manera aleatoria a las cadenas en crecimiento. De esta forma se producirán una serie de cadenas de DNA incompletas terminadas por el residuo didesoxi especifico que fue adicionado. Para determinar una secuencia (Fig. 1) se efectuan cuatro reacciones de sintesis separadas, incluyendo en cada una un nucleótido de terminación diferente. Los productos de las reacciones son sometidos a una electroforesis en paralelo, sobre un gel de poliacrilamida. Las bandas separadas en el gel, leídas desde la base del mismo y en dirección al punto de aplicación nos permitirán identificar el orden de los nucléotidos en el fragmento estudiado. DNA polimerasas empleadas en la actualidad. La enzima que tiene mayor popularidad actualmente es la T7 DNA polimerasa del fago T7 y fundamentalmente una versión modificada genéticamente conocida comercialmente como Sequenase (Unite States Biochemical Corporation) Esta enzima posee caracteristicas muy favorables como la gran procesatividad, la baja actividad exonucleasa 3 a 5 y la incorporación eficiente de los análogos de nucleótidos. Estas características redundan en una mayor calidad y resolución de los productos de la reacción. Con el desarrollo de otra técnica revolucionaria, la PCR y el empleo de enzimas termoestables, se han desarrollado nuevos protocolos de secuenciación ciclica que vinculan la potencia de la PCR con el principio de la técnica inventada por Sanger. Pasos de la técnica de terminación por didesoxinucleotidos DNA matriz En un inicio para la obtención de secuencias nucleotidicas era necesaria la obtención a toda costa de DNA simple cadena, lo cual requiere la inserción del fragmento a estudiar en un vector (Ej. M13 o sus derivados) capaz de producir simple cadena en condiciones adecuadas. En la actualidad, aunque aun se emplea este método por la calidad de las secuencias obtenidas, se han desarrollado protocolos mucho mas rápidos, en los que se desnaturaliza previamente el DNA doble cadena con el empleo de alcalis o altas temperaturas (95 oC. - 100 oC). El DNA matriz debe obtenerse con la mayor pureza posible. Unión del Oligonucleótido (Cebador) a la matriz El cebador es un fragmento de DNA de 15-20 nucleótidos, en la mayoria de los casos, que es sintetizado con una secuencia complementaria a una previamente conocida de la matriz sobre el vector en el que ha sido insertado el fragmento de DNA. El proceso de cebado se realiza mediante calentamiento de la solución DNA-cebador, hasta aproximadamente 75 gdos.C. con un descenso progresivo de la temperatura hasta la ambiental. En este proceso se produce el apareamiento de las secuencias complementarias. Reacción de Marcaje Para poder detectar con posterioridad los fragmentos sintetizados por la polimerasa es necesaria la incorporación a estos, de un marcador que facilite su detección. Los marcadores mayormente empleados son los isótopos radioactivos fundamentalmente el P 32, P 33 y el S35 incorporados en la posición del dATP o dCTP. Por otra parte, se han desarrollado sistemas novedosos de secuenciación automática que permiten la utilización de compuestos fluorescentes, capaces de excitarse al ser expuestos a la luz UV y emitir en una longitud de onda especifica. También se emplean métodos químicos de detección, como la tinción con plata, que posee un nivel de resolución compatible al de los mencionados anteriormente. La reacción de marcaje se efectúa al adicionar a la mezcla DNA-cebador los cuatro nucléotidos normales, el nucléotido marcado y la DNA polimerasa. En esta etapa, las concentraciones de los precursores es limitante de manera que los productos polimerizados sean en todos los casos lo mas pequeños posibles. Reacciones de terminación Una vez realizada la reacción de marcaje se procede a depositar en cuatro tubos rotulados A, G, T, C, las mezclas con los cuatro nucleótidos normales y las correspondientes didesoxinucleótidos. La mezcla inicial se reparte en cuatro alicuotas, en dichos tubos y la reacción se deja continuar. En la etapa de terminación se producirá la polimerización de las cadenas cuyas longitud dependerá de la incorporación al azar de los terminadores. Las reacciones son detenidas de forma cronometrada con el empleo de un tampón-colorante que posee EDTA como agente quelante e inhibidor de la polimerasa, formamida para favorecer la desnaturalización de las dobles cadenas y colorantes indicadores de la corrida electroforética. También posee un agente para aumentar la densidad de la solución a la hora de cargar el gel de electroforesis (fundamentalmente ficol). Electroforesis Las corridas de electroforesis se realizan sobre geles verticales de poliacrilamida con urea como agente desnaturalizante. Las dimensiones mas corrientes son 40cm de alto por 35cm de ancho y un espesor de 0.2 a 1mm. Estos geles permiten separar fragmentos que difieren unos de otros en solo un nucleótido En geles con las dimensiones antes mencionadas se pueden aplicar hasta 40 reacciones de secuencia, es decir 10 secuencias completas. En los sistemas automatizados en cada pocillo se puede aplicar toda una secuencia debido a la fluorescencia diferencial de los análogos de nucleótidos utilizados, lo que aumenta la capacidad de trabajo considerablemente. Sistemas de este tipo, se emplean corrientemente en muchos laboratorios como los vinculados a los grandes proyectos de secuenciación del genoma humano, levadura y otros. La corrida de manera general se realiza entre los 50 y 60 Watts de potencia, permitiendo una temperatura de alrededor de 600C. Esto último es determinante en el éxito de la corrida. Detección de los fragmentos Los fragmentos separados durante la electroforesis son detectados mediante técnicas autoradiográficas comunes, en las que se emplean filmes para rayos X. No obstante, se han desarrollado filmes con emulsiones especiales para aumentar la sensibilidad de la detección. Lectura y análisis Estos dos aspectos finales y en última instancia los mas importantes, serán abordados durante la actividad a desarrollar en el laboratorio Ejercicio de entrenamiento D.Freifelder (1983) "Problems for Molecular Biology" Editorial Cientico-Técnica, 299 páginas Ejercicio 479, pag. 44