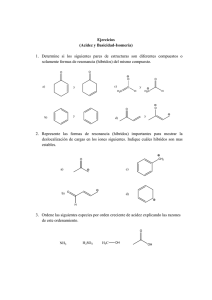
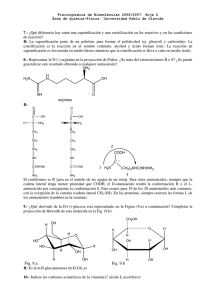
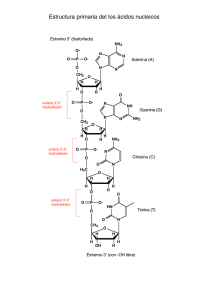
TEMA 6. ACIDOS NUCLEICOS: QUIMICA DE LOS ACIDOS NUCLEICOS. ESTRUCTURA DE ADN. 5’ 3’ Hidrólisis alcalina suave de ARN O N O NH2 N NH NH O N CH2 O N O NH2 N H2C H2C O O P O O O O ONH2 O P O N O CH2 O N O N P O O - CH2 O O N OH - O O O O P O- N O O O O HN N NH2 N O P OH O O N P O- O O- NH2 N O CH2 O N OH O O- N OH O HN O HO O O N H O O O CH2 NH2 P O- N HO N CH2 O N N O P O O- HO P O O OO P OO- NH2 N N CH2 O O O O N P OO- O OH - O O P N CH2 O O- N NH OH HN CH2 N O P O O HO HO N O O N O O O P OO O O NH2 N O CH2 O- O CH2 O O P HO- O NH OH CH2 O N O O O N H HN O OH O O OH O - N N CH2 N O O CH2 O OH NH2 O O P O O OH NH2 N NH2 N OH N N N NH2 Hidrólisis ácida suave de ADN O O NH NH N O N O O O H O H H O H P O NH2 H H N N O ON N O O H H O H P H O- O OH O H O H H O H P O NH2 H+ H N O- N H suave O O NH2 H H O H P N O- N O O O O O H O H H H O H O H H N NH O O- P N O N NH2 H H O H H O- P O OH O O H O H H O H P O O- H H O H H O H P O O- H 5’ 3’ 3’ 5’ Conformación de desoxirribosa Apareamiento de bases Giro a derechas Conformación del enlace glicosídico anti Conformación de desoxirribosa Apareamiento de bases Giro a derechas Conformación del enlace glicosídico anti Conformación de desoxirribosa Pirimidinas Purinas Conformación del enlace glicosídico: sym purinas anti pirimidinas Apareamiento de bases Giro a izquierdas Secuencias que adoptan estructuras inusuales Secuencias palindrómicas: regiones de ADN de doble hebra con secuencias de bases que se repiten en forma invertida entre ambas cadenas presentando doble simetría Son potenciales formadoras de: Horquillas (ADN simple hebra y ARN) Estructuras cruciformes (ADN duplex) Secuencias que adoptan estructuras inusuales Repeticiones espejadas: regiones de ADN de doble hebra que no tienen secuencias complementarias en la misma cadena. Se encuentran en moléculas de ADN largas (varios miles e pares de bases) Estructuras de 3 o 4 hebras: ADN triplex y ADN cuadruplex ADN triplex: se presenta en segmentos polipirimidinicos y polipurunicos Apareamientos de bases Las hélices triplex son más estables a pH ácido porque C tiene que estar protonada C+ (pKa en la hebra 7,5, pKa normal 4,2) Paralelas Antiparalelas H-ADN: Hélice triplex en trayectos polipirimidina o polipurina Con repeticiones espejadas Repetición espejada (pertenece a la misma hebra) ADN cuadruplex En secuencias con alta proporción de Guanina Posibles orientaciones de las hebras Desnaturalización Las soluciones de ADN nativo son muy viscosas a pH 7 y 25ºC rápido lento Al aumentar temperatura o variar el pH a situaciones extremas la viscosidad decrece abruptamente Densidad de flotación Efecto hipercrómico: Aumento de absorbancia a 260 nm que manifiesta una estructura bicatenaria al perder su estructura nativa o plectonémica Estimación del porcentaje de pares de bases en función de la magnitud del efecto hipercrómico Temperatura media de fusión: Temperatura a la cual se alcanza el 50 % de desnaturalización Tm y complejidad del ADN ADN parcialmente desnaturalizado Las flechas indican regiones ricas en A=T Estimación del porcentaje de pares de bases en función de Tm TEMA 6. ACIDOS NUCLEICOS: ESTRUCTURA DE RNA. SECUENCIAMIENTO DE ADN. Estructura secundaria de ARN Otras bases nitrogenadas presentes en ARN Las bases son modificadas en reacciones postranscripcionales ARNm 5’cap del ARNm ARNt ARNt para Ala de levaduras Estructura tridimensional de un ARNt Estructura general y forma de unión un aminoácido al extremo 3’ terminal de un ARNt Aminoacil ARNt sintetasas Mecanismo de aminoacilación de ARNt Catalizado por ARNtsintetasas ARNr Estructura secundaria de ARNr bacterianos Algunos apareamientos encontrados en ARNr Mutaciones: su naturaleza molecular Deleciones e inserciones Mutaciones: su naturaleza molecular Transiciones y transversiones Ejemplos Transversión Transversión Transición Agentes mutagénicos : Radiaciones Formación de dímeros de timina Mecanismo general de reparación catalizado por la fotoliasa Agentes alquilantes Reparación Metiltransferasas Dioxigenasas Desaminación de bases: ácido nitroso Derivados de bases con actividad farmacológica Derivados de nucleósidos con actividad farmacológica Secuenciamiento de ADN A- Método de Maxam-Gilbert 1- Clivaje con enzimas de restricción Sitios de corte de diferentes enzimas Gel de un mismo fragmento digerido con diferentes enzimas de restricción 2- Marcaje con 31P del grupo 5’-terminal de cada fragmento (enzimaticamente) y separación de ambas hebras Ejemplo de pasos 1 y 2 3- Clivaje químico selectivo Dividir en 4 alícuotas la muestra correspondiente a una cadena marcada y realizar en paralelo reacciones químicas de forma de obtener: a) Clivajes a nivel de G b) Clivajes a nivel de G y A c) Clivajes a nivel de C d) Clivajes a nivel de T y C a) Metilación y clivaje de la cadena a nivel de G CH O O CH33 N N N NH O CH3 O N NH NH NH N N O O P N NH2 O O O (CH3)2SO4 O P P O NH2 O O O- H O O O O O P P O O O O H O- O OH O OH O O P O P O OH O O- H2N H H - O O P O O O O H O P OH O H H H - O O P O- H H O- N O O O- OH P N O NH2 N N O O O N O O- HN O P O O- H O- NH2 b) Hidrólisis ácida suave y clivaje a nivel de A y G O O O NH NH N O N O O O H O H H O H P H N N O- O N N O O N O O NH2 H NH H H O H P H O H H O H O P O NH2 H O H+ H N suave - N H O O H O H H O O H N O- N NH2 O H O H H O H H N NH O- P O N O N NH2 H O H O O H H O P O O- H H O H H O H P O O O OH H N O O H O- H O- P O H P H N OH O O O H O OH O O O- NH2 H P O P H N O OH O O H O- O O- O O P H OOH O H O OH O H P O O- c y d) Reacción con hidracina y clivaje de la cadena a nivel de T y C OH OH O HN HN HN O O - O P O O O O N -O O N N O O O O P O- NN CH CH33 OO PP OO OO H H HH O O-- P P O O OO OO- O O H O OO -- O O-H H H NHNH NHNH22 OO PP O NH2NH2 - O O O HH NN NH NH22 HN HN HH OO OO PP OO- - O O O OO N H N O 3-ceto-4-metilpirazol O - O P CH3 O O - OH O O- P O OH -O OO P O P O O- N H H O - P O NH2 OH N OO OH H O P H O O O -O O OOH O O O P H O H O - O H O H2O P O- O - O O El clivaje selectivo en C se leva a cabo realizando la reacción con hidracina en solución de NaCl 5M. 4- Separación electroforética en (gel de poliacrilamida) de cada mezcla de productos de reacción. La técnica presenta sensibilidad para diferenciar hasta 600 fragmentos que difieran en solo un nucleótido. Revelado por autorradiografía dónde solo son visibles los fragmentos que contienen 32P 5-Interpretación de resultados (Los primeros nucleótidos a partir del extremo 5’terminal no pueden ser identificados, T en el ejemplo) Comparación y superposición con otros fragmentos Método de Sanger o de los didesoxinucleótidos Frederick Sanger Premio Nobel 1958 (Determinación de la estructura de insulina) y 1980 (Método de secuenciación de ADN) Variante utilizando ddNTPs marcados fluorescentes BIBLIOGRAFÍA -BIOCHEMISTRY. Lubert STRYER 3 Edition 1998. Ed. Freemon -BIOORGANIC CHEMISTRY Hermann Dugas. 3rd Edition 1996. Springer Verlang -BIOQUÍMICA. D.y J. Voet 1992. Ed. Omega -BIOQUIMICA. Albert L. LEHNINGER. 2da. Ed. Ediciones Omega -PRINCIPIOS DE BIOQUIMICA. Albert L. LEHNINGER. Ediciones Omega -BIOQUIMICA. Mathews, C and Van Holde, K E. Ed. McGraw- Hill Interamericana. Segunda edición. 1998. -ORGANIC CHEMISTRY. 2da. Edition.G.Marc LOUDON. Editorial Benjamin -ORGANIC CHEMISTRY. J. Mc MURRY. 3ra Edición 1994. Ed Interamericana -QUIMICA ORGANICA. Francis Carey ·3 Edicion 1999. Ed. Mc Graw Hill. -ORGANIC CHEMISTRY J. Clayden, N. Greeves, S. Warren, and P. Wothers, 2000.Oxford University Press. -QUIMICA ORGANICA. ESTRUCTURA Y FUNCION. Vollhardt, P and Schore, N. Ed. Omega. 3ra Edición, 2000.