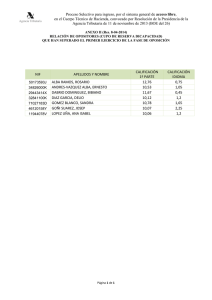
(Mill.) NE Brown (Verbenaceae) - Universidad Nacional de Colombia
Anuncio

Estructura genética y fitogeografía de
poblaciones colombianas de Lippia alba
(Mill.) N.E. Brown (Verbenaceae)
José Omar Cardona Montoya
Universidad Nacional de Colombia
Facultad de Ciencias Agropecuarias, Escuela de posgrados en Ciencias Agrarias
Palmira, Colombia
2014
Estructura genética y fitogeografía de
poblaciones colombianas de Lippia alba
(Mill.) N.E. Brown (Verbenaceae)
José Omar Cardona Montoya
Tesis o trabajo de investigación presentada(o) como requisito parcial para optar al título
de:
Doctor en Ciencias Agrarias Línea de investigación Mejoramiento Genético Vegetal
Director (a):
Director:
PhD. Jaime Eduardo Muñoz Flórez
Co-director:
Joel Tupac Otero Ospina
Línea de investigación:
Mejoramiento Genético Vegetal
Grupo de investigación en diversidad biológica
Universidad Nacional de Colombia
Facultad de Ciencias Agropecuarias, Escuela de posgrados en Ciencias Agrarias
Palmira, Colombia
2014
A los PhD que hicieron posible este logro
El error es exclusivo de los que construyen,
porque solo los que hacen se equivocan
Agradecimientos
El autor expresa su agradecimiento a(l):
MSc. Paula Rugeles Silva
PhD Jaime Eduardo Muñoz Flórez director de tesis.
Ministerio de Agricultura y Desarrollo Rural de Colombia por su aporte en recursos al
proyecto.
Grupo de investigación en diversidad biológica de la Universidad Nacional de ColombiaSede Palmira, e integrantes de los laboratorios de Biología molecular y Fitoquímica de
la Universidad Nacional de Colombia Sede Palmira:
Resumen y Abstract
IX
Resumen
Prontoalivio -Lippia alba, Verbenaceae- es un recurso fitogenético propio, promisorio por
sus entidades químicas bioactivas. Objetivo Principal: usar y sustentar marcadores que
combinen el significado adaptativo con la capacidad de discriminación entre individuos.
Se relacionaron variables climáticas de origen y de sitio de muestreo (ecomarcadores)
con marcadores genéticos (moleculares-RAM y fitoquímicos), usando SAS/STAT9.2,
DIVA-GIS5.2, WARDMLM y GenAlEx6.5. Los resultados mostraron la existencia de
variabilidad inter/intraespecífica y diferencias en el control genético para caracteres,
posiblemente adaptativos. La DG intrapoblacional (WP) fue significativamente mayor que
entre poblaciones (AP), detectando un proceso común el cual está estructurando las
poblaciones estudiadas. La prueba de diferenciación entre poblaciones mostró la menor
variación haplotípica entre poblaciones (Est.Var.AP=0,141) con remanente (95%)
distribuida dentro de la población (Est.Var.WP=2,46). El valor FST fue 0,054 (p<0,001),
indicando diferenciación genética pequeña entre regiones. La variabilidad química entre
poblaciones [AP=0.033] fue baja con un FST=0.023 P(rand≥data=0.070) y proporción 2%.
Los valores obtenidos de Fst muestra un estructura moderada entre las subpoblaciones
(FST=0.023 p≤0.07), sustentando el flujo génico mantenido por la especie en las dos
regiones estudiadas.
Palabras clave: estimativos de Comunalidad final-We, Patrones espaciales, diversidad
genética de Nei, variabilidad química, Estrategia WARDMLM.SAS, RAM
X
Título de la tesis o trabajo de investigación
Abstract
Bushy Matgrass (Lippia alba, Verbenaceae) is a plant genetic resource itself, promising
for their bioactive chemical entities. Main Objective: use and support markers that
combine adaptive significance with the ability to discriminate between individuals.
Climatic variables source and sampling site (ecomarcadores) with genetic markers
(molecular-RAM and phytochemicals) were correlated using SAS/STAT9.2, DIVA-GIS5.2,
WARDMLM and GenAlEx6.5. The results show the existence of inter / intra variability and
differences in the genetic control characters, possibly adaptive. Intrapopulational DG
(WP) was significantly higher than among populations (AP), detecting a common process
which is structuring the populations studied. Differentiation test populations haplotype
showed the least variation among populations (Est.Var.AP = 0.141) with the remaining
(95%) distributed within the population (Est.Var.WP = 2.46). The FST value was 0.054 (p
<0.001), indicating little genetic differentiation between regions. The chemical variability
between populations [AP = 0.033] was low FST = 0.023 with P (rand ≥ data = 0.070) and
proportion 2%. The values of Fst shows a moderate structure among subpopulations
(FST = 0.023 p ≤ 0.07), supporting gene flow maintained by the species in the two
regions studied.
Keywords: Final estimates Commonality, Spatial patterns, Nei's genetic diversity,
chemical variability, Strategy WARDMLM.SAS, RAM
Contenido
XI
Contenido
Pág.
Resumen ......................................................................................................................... IX
Lista de figuras ............................................................................................................. XIII
Lista de tablas .............................................................................................................. XV
Introducción .................................................................................................................. 17
1.
Capítulo 1 ................................................................................................................ 21
1.1
Marcadores ecológicos tipificantes de morfotipos de Lippia alba (Mill.) N.E.
Brown en dos ecosistemas colombianos .................................................................. 21
1.1.1
Introducción ........................................................................................ 21
1.1.2
Metodología ........................................................................................ 24
1.1.3
Resultados .......................................................................................... 29
2.
Capítulo 2 ................................................................................................................ 41
2.1
Diversidad molecular de accesiones colombianas de Lippia alba (Mill.) N.E.
Brown 41
2.1.1
Introducción ........................................................................................ 41
2.1.2
Metodología ........................................................................................ 45
2.1.3
Resultados .......................................................................................... 48
3.
Capítulo 3 ................................................................................................................ 65
3.1
Variabilidad fitoquímica entre accesiones colombianas de Lippia alba (Mill.)
N.E. Brown ............................................................................................................... 65
3.1.1
Introducción ........................................................................................ 65
3.1.2
Metodología ........................................................................................ 68
3.1.3
Resultados .......................................................................................... 69
4.
Conclusiones y recomendaciones ........................................................................ 79
4.1
Conclusiones ................................................................................................. 79
4.2
Recomendaciones ......................................................................................... 83
A. Anexo A: matriz de factores de criterio para 40 variables ecoambientales
tomadas en 59 accesiones colectadas en dos zonas ecológicas contrastantes BhT
y BsT .............................................................................................................................. 84
A.
Anexo A: (Continuación)....................................................................................... 85
A.
Anexo A: (Continuación)....................................................................................... 85
XII
Título de la tesis o trabajo de investigación
A.
Anexo A: (Continuación). ......................................................................................86
A.
Anexo A: (Continuación). ......................................................................................87
C. Anexo C: lista de accesiones con datos de colecta .............................................92
D. Anexo D: frecuencias alélicas y genotípicas por cebador y por locus ...............95
E.
Anexo E: bandas por individuo para siete cebadores RAM ..............................111
E. Anexo E: (continuación)..........................................................................................121
F. Anexo F: matriz de distancias genéticas para a) región Chicamocha, b) región
Sumapaz… ...................................................................................................................124
G. Anexo G: protocolo usado para cualificación de metabolitos secundarios.
Laboratorio fitoquímica Universidad Nacional-Palmira ............................................127
Bibliografía ...................................................................................................................129
Contenido
XIII
Lista de figuras
Pág.
Figura 1-1: Respuesta biológica de la planta, utilizable como biomarcadorbioindicador......…………...............................................……………………………………..23
Figura 1-2: Grupo de variables con valor de peso (Estimativos
de Comunalidad Final-ECF), usadas por el procedimiento FACTOR
para conformar la matriz de Marcadores Ecológicos-ME…………………………………..31
Figura 1-3: Dendograma generado por 11 factores considerados
determinantes de la estructura ecológica de L. alba en dos regiones colombianas……..32
Figura 1-4: Autocorrelaciones para 14 Lags (=distancia entre clases)
y residuales usando el modelo ARIMA AR(1)………………………………………………35
Figura 1-5: Variograma ajustado experimental, con valores: experimental
teórico y robusto…………………………………………………………………………….…37
Figura 1-6:
Semivariograma ajustado (línea) para la región Sumapaz…………….…38
Figura 1-7: Semivariograma experimental ajustado (línea).
para la región Chicamocha……………………………………………………………….…..40
Figura 2-1:
Patrón de bandas generadas por el cebador ACA_...................................51
Figura 2-2:
Patrón de bandas generadas por el cebador CA_.....................................52
Figura 2-3: Diversidad genética (hj=1-p2-q2) para 19 loci del cebador
(RAM) ACA_.....................................................................................................................56
Figura 2-4: Diversidad genética (hj=1-p2-q2) individual para 14 loci del
cebador (RAM) AG_.........................................................................................................56
Figura 2-5: Diversidad genética (hj=1-p2-q2) individual para 19 loci del
cebador (RAM) CA_.........................................................................................................57
Figura 2-6: Diversidad genética (hj=1-p2-q2) individual para 9 loci
del cebador (RAM) CGA_.................................................................................................57
XIV
Título de la tesis o trabajo de investigación
Figura 2-7: Diversidad genética (hj=1-p2-q2) individual para 8 loci del
cebador (RAM) CCA_.......................................................................................................58
Figura 2-8: Diversidad genética (hj=1-p2-q2) individual para 11 loci
del cebador (RAM) TG_...................................................................................................58
Figura 2-9: Diversidad genética (hj=1-p2-q2) individual para 14 loci del
cebador (RAM) GT_..........................................................................................................59
Figura 2-10: Diversidad genética promedio general (Hi=0,2467) y por
cebador RAM en 59 accesiones de Lippia alba colectadas en dos zonas
agroecológicas colombianas………………………………………………………………...60
Figura 2-11: Estructura jerárquica de dos poblaciones colombianas de
L. alba con base en polimorfismos generados por siete cebadores RAM………………62
Figura 2-12: Región Chicamocha. Análisis de coordenadas principales-PCoAbasado en polimorfismos RAM y distancia genética de Nei&Li………………………….62
Figura 2-13: Región Sumapaz. Análisis de coordenadas principales-PCoAbasado en polimorfismos RAM y distancia genética de Nei&Li………………………….63
Figura 2-14: Estructura espacial combinada para las dos subpoblaciones
(Chicamocha y Sumapaz)…………………………………………………………………..63
Figura 2-15: Estructura espacial para cada población, Chicamocha y Sumapaz……64
Figura 2-16: Correlograma (Omega=97.136) para la estructura espacial
de la población Sumapaz, [0.001 P(Omega-rand≥Omega-data]………………………..64
Figura 2-17: Correlograma (Omega=28.488) para la estructura espacial
de la población Chicamocha mostrando una población no estructurada……………….64
Figura 3-1: Biplot para presencia/ausencia de ms en 59 accesiones
de Lippia alba…………………………………………………………………………………76
Figura 3-2: Grupos fitoquímicos con base en AFPM de SAS/STATv9.0
para datos netos de laboratorio……………………………………………………….…….77
Figura 3-3: Biplot para factores con valores de probabilidad
máxima [FPM] de presencia de ms en 59 accesiones colombianas
de Lippia alba…………………………………………………………………………………78
Figura 3-4: Clúster para presencia de siete metabolitos secundarios
en una muestra de 59 accesiones colombianas de Lippia alba…………………………81
Contenido
XV
Lista de tablas
Pág.
Tabla 1-1: Autocorrelación espacial (Índices: Global de Moran y Geary)………………..28
Tabla 1-2: Varianza explicada por cada Factor, correspondiente al
Valor Propio (o Eigenvalue) para variables………………………………………………….29
Tabla 1-3: Estimativo de Comunalidad Final-ECF- total y para variables……………… 30
Tabla 1-4: Salida (Output) de autocorrelaciones para 14 Lags…………………………….34
Tabla 1-5: Valores de los variogramas teórico y ajustado (robusto)
para 14 observaciones…………………………………………………………………….…..36
Tabla 1-6:
Índices y estimativos de diversidad……………………………………..……40
Tabla 2-1: Pasos del termociclador……………………………………………………..……47
Tabla 2-2: Parámetros (número efectivo de alelos-Ae y
Heterocigocidad Esperada-He) y variables (Polimorfismos-Pj
y alelos por locus)………………………………………………………………………… ….48
Tabla 2-3: Valores de Coeficiente de Simpson determinantes
de la variabilidad genética………………………………………………………………… ….49
Tabla 2-4: Valores de diversidad genética intraespecífica…………………………… …..50
Tabla 2-5: Alelos únicos y alelos raros hallados en la muestra de 59 accesiones
de L. alba colectadas en dos zonas (Chicamocha y Sumapaz-Colombia)…………… …53
Tabla 2-6: Estadígrafos para siete cebadores RAM para 59 colectas de L. alba…… ….55
Tabla 2-7: Número de loci, polimorfismos y valores de Diversidad Genética-DG………55
Tabla 2-8: Análisis molecular de varianza-AMOVA- y valor de FPT para 58
accesiones colombianas de L. alba………………………………………………………….61
Tabla 2-9: Valores porcentuales………………………………………………………………61
XVI
Título de la tesis o trabajo de investigación
Tabla 3-1: Estimativos de Varianza [Est.Var.] para el tamizaje fitoquímico
de 58 accesiones colombianas de L. alba, usando el Programa GenAlex v6.5…………72
Tabla 3-2: Número final de grupos y de individuos por grupo [Peso-Proporción]
obtenidos con la estrategia WARDMLM.SAS………………………………………………73
Tabla 3-3: Proporción de individuos con valor [1] de presencia de siete tipos de
metabolito secundario en dos tipos de solvente, ordenados por tipo de solvente (a)
Cloroformo y (b) Etanol......................................................................................................74
Introducción
La biodiversidad de especies de plantas tropicales, junto con la diversidad química
encontrada en cada individuo, lleva a la conclusión de que las especies tropicales son
quizá la fuente más valiosa de nuevas entidades químicas bioactivas (Berier et al., s.f.).
Determinando la diversidad genética, química, y por factores ambientales de la población
colombiana de Lippia alba (Prontoalivio) y cuantificándola, es posible proponer planes y
programas de conservación de la variabilidad actual y/o incrementarla (con regeneración
por semilla, somaclonalmente, introducciones, otros), si es requerida. Los
biomarcadores/bioindicadores ecológicos, con base en factores edafoclimáticas, índices
y estimativos de diversidad; cumplen un rol definitivo en la valoración de comunidades y
poblaciones de L. alba. El uso combinado de marcadores moleculares, fitoquímicos y
ecológicos permitirá estructurar, con alto nivel de confiabilidad, la(s) población(es)
actuale(s) de L. alba en Colombia. Este documento sustenta el diseño y uso de
marcadores ecológicos que combinen el significado adaptativo con la capacidad de
discriminación entre individuos.
La tribu Verbenaceae se compone de los complejos: a) sensu Verbena/Glandularia con
170 especies, y b) Lantana/Lippia/Aloysia con 200–400 especies (Yuan et al., 2009).
Inferencias filogenéticas sugieren a Suramérica como lugar de origen de las
Verbenaceae con aproximadamente seis eventos de colonización, los cuales dieron
origen a las especies presentes en el viejo mundo (Marx et al., 2010). El análisis
filogenético realizado por Marx et al. (2010) mostró ocho clades reconocidos como tribus
(Casselieae, Citharexyleae, Duranteae, Lantaneae, Nerosportoneae, Petreae, Priveae,
Verbeneae). Lantaneae y Verbeneae forman un clade, con aproximadamente 2/3 de las
especies en el clade Verbeneae.
Marcadores Ecológicos-ME
Son
vectores con base en variables sintéticas (obtenidos como Componentes
Principales-CP o Factores) de respuestas generadas a partir de tres componentes
básicos: Biomarcadores, Bioinidicadores e indicadores ecológicos. Biomarcador:
cualquier respuesta biológica frente a un factor ambiental al nivel sub-individual, medida
dentro del organismo o en sus productos (pubescencia, espinas, ceras, aceites
esenciales, etc.) indicando una alteración respecto a su estado normal que no puede ser
detectado a partir del organismo en su conjunto. De este modo, los biomarcadores se
restringen a determinaciones bioquímicas, fisiológicas, histológicas, morfológicas
(incluyendo aspecto, pigmentación, malformaciones, etc.) del estado general excluyendo
los efectos sobre el comportamiento (Kosakivska, 2008). De hecho, los patrones de
adaptación fisiológica pueden ser clasificados dentro del tema de estrategias ecológicas
(Boschker y Middelburg, 2002; Vestergaard et al., 2003; Shugart, 2006; Kosakivska,
2008). Bioindicador: es un organismo que aporta información sobre las condiciones
ambientales de su hábitat mediante su presencia, ausencia y comportamiento. Los
18
Título de la tesis o trabajo de investigación
efectos fisiológicos no se incluyen en la definición de bioindicador (Boschker y
Middelburg, 2002; Vestergaard et al., 2003; Shugart, 2006; Kosakivska, 2008). Indicador
ecológico: trata parámetros que describen la estructura y la función de los ecosistemas:
diversidad de especies, dinámica de poblaciones, cinética del ciclo de nutrientes, tasa de
respiración/productividad, etc. (Shugart, 2006).
Diversidad genética y estructura de poblaciones naturales de Lippia spp.
La estructura genética de poblaciones naturales del género Lippia spp. ha sido
escasamente estudiada (Montanari et al., 2004; Viccini et al., 2004-2006; Dias et al.,
2005-2006; Suárez et al., 2007-2008; Yamamoto et al., 2008; Martínez et al., 2008).
Análisis previos utilizando marcadores RAPD fueron efectivos para comprender la
diversidad genética de especies de Lippia spp. y contribuyeron a entender su adaptación
al ambiente, conservación e implicaciones taxonómicas (Viccini et al., 2004; Pereira et
al., 2006). Viccini et al., (2004), evaluaron el grado de diversidad genética en nueve
especies de Lippia spp. (L. corymbosa, L. diamantinensis, L. filifolia, L. florida, L.
hermannioides, L. lupulina, L. rotundifolia, L. rosella y L. sidoides) del sudoeste de Brasil.
La distancia genética interespecífica promedio fue similar para todas las especies y más
alta que las distancias intraespecíficas. Las especies con presencia limitada, mostraron
diversidad interespecífica más baja. El dendograma por el método UPGMA mostró
grupos mayores con una clara diferenciación entre especies (Viccini et al., 2004). En
2012, Santos et al., aislaron y caracterizaron ocho loci de microsatélites (SSR)
polimórficos en dos poblaciones brasileras de Lippia alba, los cuales mostraron potencial
informativo de moderado alto para estudios genéticos. Siete de ocho secuencias SSR
revelaron múltiples bandas típicas de especies poliploides sugiriendo genomas
tetraploides en Lippia alba. El índice de diversidad de Shannon (H) fueron 2.013 y 1.989,
respectivamente. La heterocigocidad esperada (He) tuvo rango entre 0.407 y 0.718, con
media de 0.564 para la población A; y de 0.234 a 0.704 con media 0.503 en la población
B.
Suárez et al. (2007) llevaron a cabo un estudio piloto en Lippia origanoides, L. alba, y L.
citriodora para observar polimorfismos intra e inter-específicos en cuatro regiones del
cpDNA, de ITS del ADN ribosomal y 50 loci ISSR. Tres de cuatro regiones del cpDNA
(petA-psbE, tnrL-trnF and trnL intron) y la región ITS mostraron mayormente variación
interespecífica. Diez y siete regiones ISSR de 50 loci analizados mostraron
polimorfismos, inter e intraespecíficos. La divergencia de secuencias entre pares de
especies en la región del plastidio, mostró rangos de 0.4% a 1%, mientras que la
divergencia en secuencias de regiones ITS fue cerca del 5%. Los resultados obtenidos
sugieren que las regiones ITS, tal como han sido reportadas para otros géneros de
plantas, puede convertirse en una región de elección para estudios de genética de
poblaciones en este tipo de especies. Los valores de diversidad genética descrita por
Suárez et al. (2008) en Lippia origanoides (plantas alógama Verbenaceae relacionada)
fueron: P=86.21%, para porcentaje de loci polimórficos; I=0.453, para índice de
diversidad de Shannon; y HB=0.484 para Heterocigocidad panmitica promedio.
Variabilidad química de aceites esenciales en Lippia alba
La composición química del aceite esencial de L. alba depende sensiblemente del origen
geográfico de la planta, las condiciones de su cultivo, la edad y la parte de la planta
empleada para la extracción, y de algunos otros factores geobotánicos (Stashenko et al.,
Capítulo 2
19
2003). La mayoría de estudios en L. alba son determinaciones genotípicas (Montanari et
al., 2004; Viccini et al., 2004-2006; Dias et al., 2005-2006; Suárez et al., 2007-2008;
Yamamoto et al., 2008; Martínez et al., 2008; Santos et al., 2012), caracterizaciones
bioquímicas [quimiotipificación] (Fischer et al., 2004; Hennebelle et al., 2006) y de
morfología externa (Montanari et al., 2004; Hennebelle, 2007). Individuos de L. alba de la
región nororiental de Colombia mostró ser un quimiotipo nuevo, no descrito previamente
en la literatura, conteniendo la carvona [40-57%] como componente mayoritario en los
aceites, seguido de limoneno [24-37%], biciclosesquifelandreno [2-22%] piperitenona [12%], piperitona [0.8-1.2%] y el β-burboneno [0.6-1.5%]. El componente principal (en las
fracciones volátiles) fue limoneno [27-77%], seguido por la carvona [14-30%],
biciclosesquifelandreno [1-33%] y el β-burboneno [0.5-6.5%] (Stashenko et al., 2003). El
análisis de aceites esenciales obtenidos de hojas de muestras de Guayana Francesa,
Martinica y Guadalupe, mostraron siete quimiotipos diferentes (Hennebelle et al., 2006),
existiendo una posible conexión entre quimiotipos y morfotipos (Hennebelle et al., 2006).
La interacción GxE de 10 genotipos de cuatro composiciones químicas (quimiotipos) de
L. alba de tres regiones del estado de Sao Paulo (SP), Brasil, tuvieron un rango amplio
de producción de hoja, mientras que el rendimiento de aceites esencial tuvo una alta y
uniforme determinación genotípica. Variación no cualitativa fue detectada para
composición química, y la variación cuantitativa fue de baja magnitud. Genotipos Linalol y
limoneno/la carvona fueron invariablemente más productivos para rendimiento de aceite
que los genotipos citral y mirceno/canfor (Yamamoto et al., 2008). La variación de aceite
esencial de Lippia junelliana de 16 sitios diferentes de medio oeste Argentino mostró
ocho compuestos como componente importante en los aceites (Juliani et al., 2002). De
acuerdo con estos componentes, cuatro quimiotipos silvestres se han detectado. El
quimiotipo primero contenía altos niveles de ocimenone (54-76%) con cantidades más
bajas de mirceno (10%). El segundo se componía de grandes cantidades de
dihidrocarvona (59-80%). Los principales componentes se encuentran en la tercero,
quimiotipo limoneno (10-40%) y piperitenona (10-40%) y la cuarta contenía quimiotipo
limoneno (41%) y óxido de piperitenona (26%) (Riccardi et al., 2000; Juliani et al., 2002).
Las especies de L. alba (Miller) N.E. Brown se caracteriza por una gran variabilidad en
morfología y composición química del aceite esencial (Oliveira et al., 2006). Tavares et al.
(2005), presentan datos sobre la variación cuantitativa de los principales componentes
volátiles de la producción de linalol en un quimiotipo de L. alba. El contenido de [alpha]pineno, (Z)-3-hexenilo y [alpha]-gurjunene fue mayor en plantas madre cultivadas en el
suelo que en las plántulas crecieron en medio MS, mientras que el contenido de
sabineno, mirceno, 1,8 -cineol y p-menta-l, 5,8-trieno fue menor. La adición de 0.2.3 μM
de IAA en el medio fue significativamente mayor para sabineno y el contenido mirceno.
La adición de 0,92 μM de cinetina aumentó significativamente el 3 (S) - (+)-nivel linalol
(Tavares et al., 2005). En Colombia, Camargo y López (2008) y Palacio y López (2008)
evaluaron la respuesta de L. alba a la disponibilidad de agua; y nitrógeno (Hernández et
al., s.f.; Antolinez y López, 2008). Las variaciones cuantitativas de los principales
componentes volátiles de la producción de linalol en quimiotipo L. alba están asociadas
con factores nutricionales y ambientales (Hernández et al., s.f.; Tavares et al., 2005;
Camargo y López, 2008; Palacio y López, 2008; Antolinez y López, 2008).
La composición química y la actividad antimicrobiana de los aceites esenciales en L.
alba las investigó Oliveira et al. (2006) con en el fin de relacionarlas a sus usos
tradicionales. El análisis de los aceites esenciales que permitió la identificación de dos
especies de Lippia alba, una como quimiotipo mirceno-citral (15% y 37,1%,
20
Título de la tesis o trabajo de investigación
respectivamente) y Lippia alba f. intermedia como un quimiotipo citral (22,1%). Los
aceites esenciales de ambas especies fueron activos contra todos los microorganismos
probados (bacterias y hongos) mediante el ensayo de halos de inhibición con rango
desde 1,1 hasta 5,0 cm; probablemente debido a su alto contenido de monoterpenos
oxigenados (51,0% y 40,1%, respectivamente), representada principalmente por
aldehídos y alcoholes. Los datos químicos y farmacológicos de L. alba obtenido por
Oliveira et al. (2006) estuvieron de acuerdo con la encuesta etnobotánica. En Colombia,
EL Centro de Investigaciones en Biomoléculas-CIBIMOL- de la Universidad Industrial de
Santander ha realizado entre otros, estudios valorativos en enfermedades tropicales, de
bioactividad y principios activos de Lippia alba como antimicrobial (Bueno y Stashenko,
2009); antiviral [Ocazionez et al. (2010) en virus del dengue; Meneses et al., (2009) en
virus de la fiebre amarilla], antifúngica (Mesa et al., 2009) en Candida y Aspergillus
(Montiel et al., 2007), en bacterias [Bueno et al., (2009) en M. tuberculosis]; antiprotozoario (Escobar et al., 2010). Una lista completa de reportes la ubica en
http://tux.uis.edu.co/quimica/ investigacion /centros/cibimol.
La apuesta en la presente investigación fue determinar la estructura genética y
fitogeográfica de Lippia alba de tal forma que permitiera una aproximación a la
estructuración poblacional de la especie en Colombia. Esta se logró mediante: a.
Marcadores ecológicos los cuales combinaron el significado adaptativo con la capacidad
de discriminación entre individuos, b. Marcadores moleculares RAM, determinantes de la
estructura y diversidad genética de la muestra de trabajo, y c. Estimativos de la
variabilidad química de las 59 accesiones de Lippia alba incluidas en la prueba.
Capítulo 2
21
1. Capítulo 1
1.1 Marcadores ecológicos tipificantes de morfotipos de
Lippia alba (Mill.) N.E. Brown en dos ecosistemas
colombianos
1.1.1 Introducción
Familia Verbenaceae. Comprende cerca de 1035 especies y 36 géneros con distribución
pantrópica (Gupta et al., 2001; Dias et al., 2007; Lou-Nita Le Roux On: http://www.
plantzafrica.com/index html). El género Lippia (Verbenaceae) incluye cerca de 200
especies la mayoría distribuidas en la región neotropical (Gupta et al., 2001; Jarvis et al.,
2006; Suárez et al., 2007; Manica-Cattani et al., 2009), principalmente presente en las
Antillas mayores (Hennebelle et al., 2006; 2007; 2008). De hecho, la zona geográfica de
su origen es desconocido (En: http://www.ars-grin.gov/cgi-bin/npgs /html/taxon.pl?22367).
Su área más probable de origen es de México a Argentina y en las Antillas. La especie se
distribuye desde Norte América semiseptemtrional (Gupta et al., 2001), México, y por el
Caribe (Haití, Cuba, República Dominicana, Jamaica e islas Leeward) hasta Argentina y
la Amazonia, también se encuentra en Suráfrica, India (Gupta et al., 2001) y Australia. Se
presenta en regiones secas de América Latina desde México hasta Paraguay, Brasil,
Uruguay, Argentina y Colombia (Vit et al., 2002; Arango, 2004; UNCTAD, 2005).
Lippia alba [prontoalivio (Colombia), erva-cidreira (Brasil), juanilama (Costa Rica), salvia
morada (Argentina), bushy matgrass, Bushy Lippia Oaxaca lemon verbana, melissa] es
una de las más importantes plantas medicinales usadas, entre otros, por pobladores
brasileros (Castro et al., 2002; Zoghbi et al., 1998; UNCTAD, 2005). Es mayormente
aprovechada por sus propiedades somáticas, sedativas, antidepresivas y analgésicas
(Gupta et al., 2001; Schroeder et al., 2004; UNCTAD, 2005). El aceite esencial de L. alba
tiene además usos como estomático, antiespasmódico, digestivo, anti-hemorroides y
anti-asmático (Vit et al., 2002; Schroeder et al., 2004; UNCTAD, 2005) y en el control de
insectos en granos almacenados (Gupta et al., 2001). Como sinónimos para L. alba (Mill.)
N.E.Br. están: Lantana alba Mill., Lippia geminata Kunth., Lantana germinata (Kunth.)
Spreng. (Seaforth y Tikasingh, s.f.).
Lippia alba es una especie promisoria de importancia económica y con posible uso
sustentable por calidad de sus aceites esenciales (Gupta et al., 2001). La potencialidad
de este recurso ha sido valorado ampliamente por investigadores locales (Duque, 1985;
22
Título de la tesis o trabajo de investigación
Duran et al., 2007; Linares, 2007; Martínez, 2008; Antolinez, 2008; Stashenko et al.,
2004; Suárez et al., 2007) y extranjeros (Vieira y Skolupa, 1993; Vieira, 1999; Zoghbi et
al., 1998; Gupta et al., 2001; Jaramillo y Manos, 2001; Castro et al., 2002; Fischer et al.,
2004; Schroeder et al., 2004; Montanari, 2004; Viccini et al., 2004; Tavares et al., 2005;
Dias et al., 2005; Zucchi et al., 2005; Hennebelle et al., 2006; Oliveira et al., 2006; Pereira
et al., 2006; Ramalho et al., 2006; Dias et al., 2007; Hennebelle et al., 2007; Godínez y
Volpato, 2008; Hennebelle et al., 2008; Yamamoto et al., 2008; Figueiredo et al., 2008;
Manica et al., 2009; Sousa et al., 2009; Vera et al., 2010; Pierre et al., 2011).
Marcadores Ecológicos-ME. Son vectores con base en variables sintéticas (obtenidos
como Componentes Principales-CP o Factores) de respuestas generadas a partir de tres
componentes básicos: Biomarcadores, Bioindicadores e indicadores ecológicos.
Biomarcador: cualquier respuesta biológica frente a un factor ambiental al nivel subindividual, medida dentro del organismo o en sus productos (pubescencia, espinas,
ceras, aceites esenciales, etc.) indicando una alteración respecto a su estado normal que
no puede ser detectado a partir del organismo en su conjunto. De este modo, los
biomarcadores se restringen a determinaciones bioquímicas, fisiológicas, histológicas,
morfológicas (incluyendo aspecto, pigmentación, malformaciones, etc.) del estado
general excluyendo los efectos sobre el comportamiento (Kosakivska, 2008). De hecho,
los patrones de adaptación fisiológica pueden ser clasificados dentro del tema de
estrategias ecológicas (Boschker y Middelburg, 2002; Vestergaard et al., 2003; Shugart,
2006; Kosakivska, 2008). Bioindicador: es un organismo que aporta información sobre las
condiciones ambientales de su hábitat mediante su presencia, ausencia y
comportamiento. Los efectos fisiológicos no se incluyen en la definición de bioindicador
(Boschker y Middelburg, 2002; Vestergaard et al., 2003; Shugart, 2006; Kosakivska,
2008). Indicador ecológico: trata parámetros que describen la estructura y la función de
los ecosistemas: diversidad de especies, dinámica de poblaciones, cinética del ciclo de
nutrientes, tasa de respiración/productividad, etc. (Shugart, 2006).
Biomarcadores-BM (respuestas biológicas). Entendido como medidas funcionales de
exposición al estrés ambiental normalmente se expresan a niveles de organización
biológica inferiores al de organismo (Shugart, 2006), son una variación xenobiótica en
componentes o procesos estructurales o funcionales celulares o bioquímicos medibles en
un sistema biológico o una muestra (Shugart, 2006). Los tiempos de respuesta y niveles
de organización biológica (Figura 1-1) están directamente correlacionados para factores
genéticos > bioquímicos > histopatológicos > inmunológicos > bioenergéticos. Estos
pueden ser conformados mediante: (i) bioensayos (DL50, Bioacumulación), (ii) análisis
bioquímicos (SPME), marcadores moleculares (RAPD, AFLP, otros posibles), (iii) análisis
fisiológicos (Tasas e índices), (iv) pruebas genéticas (Aloenzimas, microsatélites), (v)
respuesta inmunológica (ELISA), (vi) histopatología (neoplasmas), (vii) anormalidades
morfológicas (asimetrías fluctuantes), (viii) comportamiento (actividad) y (ix)
bioenergética (crecimiento, reproducción) (En: https://www.researchgate.net/publication/
24147541Biomarkersinaquaticplantsselectionandutility). En estudios de ecología
microbiana (rizósfera, filósfera) los biomarcadores pueden proveer información tanto
microbiana (identificando microorganismos específicos) como de biomasa, en los que el
marcador puede presentarse en cantidad detectable y relativamente constante en el
organismo de interés. Para Boschker y Middelburg (2002) los biomarcadores son
compuestos que tienen especificidad biológica en el sentido que son producidos
únicamente por un limitado grupo de organismos. Vestergaard et al. (2003) han
Capítulo 2
23
implementado el uso de marcadores moleculares, celulares, genéticos y ecológicos para
comprender la decoloración del coral en cuatro océanos del planeta.
Bioindicadores-BI (indicadores biológicos). El principal objetivo del uso de los BI es
permitir una comparación entre sitios dentro y entre polígonos, zonas agroecológicas y
Zonobiomas. Un bioindicador comparativamente más alto, evidencia condiciones
extremas o de estrés biótico o abiótico. Los Bioinidicadores son importantes (Doran y
Parkin, 1994; Zhang y Zhang, 2009) ya que la expresión fenotípica (F=G+A+GA) y
respuesta química de la biota a un ambiente está íntimamente influenciada por los
factores bióticos y abióticos.
Figura 1-1: Respuesta biológica de la planta, utilizable como biomarcadorbioindicador. Fuente: https://www.researchgate.net/publication/24147541Biomar
kersinaquaticplantsselectionandut ility.
Es importante identificar componentes que den respuesta rápida a cambios en el
comportamiento de un individuo. Los Indicadores Biológicos-IB- o Bioindicadores
muestran un gran potencial como tipificadores de un individuo, debido a su carácter
integrador, y su mayor sensibilidad y velocidad de respuesta frente a cambios en el
sistema. Con ello se consigue anticiparse a cambios y perturbaciones irreversibles que
se pueden dar en ambiente dado, ya que éste no muestra su verdadero estado de
degradación o cambio hasta que alcanza un nivel importante de afección (a diferencia del
agua y el aire) (http://www.cricyt.edu.ar/ enciclopedia/terminos/Bioindic.htm). Para la
Selección de los Bioindicadores se debe tener en cuenta: Tipo de estudio, se requiere de
un BI de exposición o de efecto general o específico o su combinación, especie y
velocidad de respuesta requerida (Vgr. Especies amenazadas).
24
Título de la tesis o trabajo de investigación
El presente caso de estudio requirió de un tipo adecuado de bioindicadores para evaluar
el efecto ambiental en la expresión-respuesta fenotípica y química de los individuos de L.
alba presentes en cada área de estudio. Actualmente, metodologías analíticas y
programas (software) especializados permiten crear Marcadores Ecológicos-ME- a partir
de Estimativos de Comunalidad. Con base en las hipótesis planteadas, el presente
estudio hace un análisis determinístico consistente en encontrar, usar y sustentar
(determinar) Marcadores Ecológicos que combinen el significado adaptativo con la
capacidad de discriminación entre individuos; en el presente caso fueron valorados los
contenidos de aceites esenciales-ae- marcadores RAM y factores ambientales, los cuales
se asumen, están implícitos en los indicadores de biodiversidad.
1.1.2 Metodología
El presente documento propone, usa y sustenta parámetros a considerar como
Marcadores Ecológicos-ME- a un solo nivel de organización biológica no usualmente
considerado en estudios de biomarcadores para evaluar el efecto ambiental sobre las
características, presencia y distribución en Colombia de la especie Lippia alba. Los
biomarcadores utilizados fueron: Bioquímicos. Siete metabolitos secundarios.
Moleculares. Expresión génica (alelos comunes a una población) a partir de marcadores
moleculares RAM, otros autores han usado AFLP y regiones ITS (Suárez et al., 2007).
Del suelo. El nitrógeno potencialmente mineralizable, pH, N, P y K extraíbles, capacidad
intercambio catiónico, granulometría, tipo de arcillas. Ambientales. Tmax. (Temperatura
Máxima), Tmin. (Temperatura Mínima), HR%, Presión (hPa), UV, DU; y zonificación
agroecológica (En: http://humboldt.org.co/using/ecosistemas /colombia/ ecosistemas.php.
Se propone el uso de marcadores ecológicos, dado que los biomarcadores ecológicos
definen/determinan cambios morfofisiológicos y en composición y contenido de
compuestos fitoquímicos mostrando respuestas por biodisponibilidad (exposición),
mecanismo de acción (efecto) y respuesta (susceptibilidad) detectados en plantas por
efectos antrópicos / antropogénicos (Ernst & Peterson, 1994; Ernst, 1999).
Los Biomarcadores-Bioindicadores tienen la capacidad de predecir cambios a niveles
altos de complejidad biológica (población, comunidad, ecosistema), se han propuesto
como herramientas de diagnóstico precoz de la salud del medio ambiente. Permite
cuantificar cambios (bioquímicos, fisiológicos; respuestas histológicas, morfológicas y de
comportamiento), a nivel individual o sub-individual asociados a la exposición a un factor
de estrés biótico/abiótico (estrés climático, herbivoría, otros). Su principal valor radica en
evidenciar efectos tempranos de un factor de estrés, situaciones extremas o simplemente
contrastar valores de variabilidad biológica (Kurian et al., 2007; Pérez et al., 2008; Bozo
et al., 2007).
Zona de estudio. La selección de la zona de estudio se hizo con base en: a) una
caracterización preliminar para seleccionar los Zonobiomas problema y los sitios de
referencia. Colecta de datos históricos referentes a aspectos socioeconómicos y de
parámetros ambientales (físicos químicos y biológicos y b) Se realizó un estudio piloto y/o
búsqueda de reportes bibliográficos, estableciendo la línea base en función de datos
Capítulo 2
25
existentes en la literatura; principalmente en relación a Lippia alba como potencial
especie centinela y sus respuestas biológicas.
Características de la zona de estudio. La Región Andina,-las más representativa y
densamente poblada de Colombia,-contiene ~30000 especies de plantas vasculares
relativamente desconocidas incluyendo subespecies endémicas, equivalente a >60% de
las especies reportadas para Colombia e incluye especies reportadas en el borde étono
amazónico. Las características más importantes de la región Andina es que más del 60%
de los hábitats naturales originales de la Región han sido destruidos por la agricultura y el
sobrepastoreo. Es la región con mayor intercambio y uso de biomasa y el mayor número
de estudios y colectas de L. alba. Contiene el mayor número de Zonobiomas y
agroecosistemas dentro de Zonobioma; incluyendo y los ecosistemas de Bs-T y Bh-T.
Para efectos de selección de la Región de estudio, se consideraron los ecosistemas Bs-T
y Bh-T, dado que los remanentes de Bs-T contienen familias de plantas similares a los
hallados en Bh-T (IAVH, 1998).
Zonobioma Tropical Alterhídrico Bs-T colombiano. En Colombia el Bosque seco
Tropical se distribuía originalmente en las regiones de la llanura Caribe y valles
interandinos de los ríos Magdalena y Cauca entre los 0 y 1000 metros de altitud y en
jurisdicción de los departamentos del Valle del Cauca, Tolima, Huila, Cundinamarca,
Antioquía, Sucre, Bolívar, Cesar, Magdalena, Atlántico y sur de la Guajira (IAVH, 1998).
También se encontraban enclaves de menor extensión con esta vegetación en las Islas
de San Andrés y Providencia, en la región norte de la península de la Guajira, Santa
Marta (Magdalena), en Gamarra (Cesar), Cañón de río Chicamocha (Santander),
Convención y Ocaña, alrededores de Cúcuta (Norte de Santander), Cañón del Dagua
(Valle del Cauca), Villa Vieja (Huila) y Valle del río Patía (Cauca) (IAVH, 1998). Aunque
no se dispone de información exacta de la extensión de la cobertura original del Bosque
seco Tropical en Colombia, se estima que cubre la mayor parte de las todas las regiones
y localidades anteriormente nombradas y las cuales abarcan una extensión de más de
8’146.000 hectáreas. Esta información fue generada a partir de mapas de formaciones
vegetales de Colombia. Según la clasificación propuesta por Hernández (1990), para las
condiciones de Colombia, el Bosque seco Tropical corresponde al zonobioma Tropical
Alterhídrico que se desarrolla en tierras baja. Este zonobioma abarca siete provincias
biogeográficas de acuerdo a la clasificación dada por Hernández et al. (1992) (IAVH,
1998).
Especie vegetal centinela=Lippia alba (En: www.monografias.com › Biología). En
teoría toda las taxas son Bioindicadores. Las características deseables son:
Especies con línea base de estudios previos e importancia económica o ecológica
Abundancia relativa de la especie no necesariamente diferente entre zonas
agroecológicas. Debe ser tal que permita analizar la variabilidad sin provocar
efectos deletéreos en la población. Uso de BI no destructivos
Características biológicas. Tipo de propagación, ciclo biológico etapas de
desarrollo fenológico, otros posibles como estacionalidad.
Permanencia alta refleja las condiciones ambientales de su hábitat restringido. Se
requiere conocer los patones de migración, mutación, deriva otros. Efecto
fundador.
Requerimientos nutricionales y ambientales
Estado fisiológico. Nutricional hormonal y de contenidos de ms bioprotectantes.
26
Título de la tesis o trabajo de investigación
Ecosistemas presentes en el sitio de muestreo
Identificación del ecosistema a partir del sitio de muestreo localizado con GPS. La fuente
de información de los ecosistemas se hace a partir de http://humboldt.org.co/using/
ecosistemas /colombia/ecosistemas.php. con datos homologados con los sistemas de
clasificación de: Etter (1998), Hernández y Sánchez (1992), Holdrige (1957), Ideam
(1996), Stutz et al., (1996) y UNESCO (1972). El libro de datos debe contener: Código
del ecosistema y zonobioma al que pertenece, Nombre del ecosistema, Descripción (del
ecosistema), Flora predominante y Bioma (En: http://humboldt.org.co/using/ ecosistemas
/colombia/ ecosistemas.php.
Bases de datos. Tres sub-matrices forman parte de la matriz general que conforma el
libro de campo y laboratorio de accesiones de Lippia alba (de esta investigación).
1. Factores climáticos de sitios de colecta, variables físico-químicas de suelos y de
ecosistemas Andinos colombianos,
2. Matriz binaria de presencia/ausencia de metabolitos secundarios-ms- por
accesión,
3. Matriz binaria de presencia/ausencia de bandas de ADN de accesiones de L. alba
a partir de marcadores RAM.
Universo/Población/Muestra. Para el estudio se dispuso de ~80 muestras, con al
menos dos replicaciones de individuos de L. alba colectados, que incluye afectados y no
afectados por herbivoría y/o enfermedades colectados en dos zonas ecológicas
contrastantes Bh-T. y Bs-T.; con base en mapas y polígonos de búsqueda usando el
método de Patrones Espaciales de Jarvis et. al., (2006). El muestreo incluye una base
con datos de campo conteniendo; datos del individuo, de localidad y muestras (botánica,
ADN, química y propagación). Para el muestreo se ha considerado: Listado de
marcadores, especificidad y sensibilidad del biomarcador, dificultad de muestreo, cinética
de la formación del biomarcador y estabilidad del biomarcador.
Metodología analítica usada. En cuanto a las metodologías analíticas habitualmente
utilizadas en la cuantificación de estos (Biomarcadores) se destacan aquellos
procedimientos que no implican tiempos de análisis muy largos. En general, toda
propuesta metodológica generadora de información sustentable (En: http://www.berrilur.
net/index.php?option=comcontent&view=article&id=17&Itemid=17)
es
válida.
Los
Marcadores Ecológicos fueron creados mediante Patrones Espaciales (Jarvis et al.,
2007) usando los procedimientos: CLUSTER, TREE, PRINCOMP, MIXED, DISTANCE,
ARIMA, VARIOGRAM y FACTOR de SAS/STATv9.2; y DIVA-GIS. El enfoque temporal
(En: disi.unal.edu.co/~lctorress /PSist/ PenSis05.pdf), consistió en muestreo de los sitios
una sola vez, determinando que factores pueden modular el Bioindicador-BI y amplificar
o reducir las respuestas; mediante combinación de enfoque espacial y respuestas
específicas. Si el BI muestra un patrón de variación continua que corresponda a
gradientes hay una fuerte evidencia de relación causal (Altieri y Nicholls, 2007). La
temporalidad (de algunas variables) fue ajustada usando el programa DIVA-GIS. Para el
análisis de Componentes Principales-CP- se tomó como base la matriz de correlaciones
dado que disponía de variables multi-escala. Para la selección de CP, se tomó el criterio
de valor propio mayor a 1. De hecho, existen dos criterios de selección de CP; uno con
base en la proporción de variabilidad expresada en la prueba con valores aceptables
Capítulo 2
27
≥0.05; y a otra con base en el peso del valor propio (Eigenvalue) el cual debe ser ≥1. Las
variables sintéticas (con base en la matriz de correlaciones) generadas por el análisis de
CP, fueron usados por la estrategia WARD-MLM para la conformación final de grupos;
así como también para los análisis con SAS/GI y, DIVA-GIS.
Variables usadas para crear los Marcadores Ecológicos-ME. Las variables tomadas
para generar los marcadores ecológicos fueron: 1) Edáficos. Materia orgánica del suelo,
el nitrógeno potencialmente mineralizable, pH, N, P y K extraíbles, Capacidad
intercambio catiónico, Granulometría, otros reportados en la metodología; 2) De
localización. Región, altitud y coordenadas decimal norte/oeste (Georreferenciación); 3)
Climáticos. HRmax/min, Temp. Max/Min. Velocidad del viento máx./min, hPa máx./min,
UVmax/min, DUmax/min; 4) Ecosistema(s). Clasificación de zonas agroecológicas
(Código, Nombre, Descripción, Bioma, modelo climático, cobertura vegetal) tomadas de:
http://hermes. humboldt.org. co// ecosistemas /andes/ y DIVA-GIS. La matriz de datos de
campo (del Anexo B) estuvo conformada por: 1) datos de localización (región, altitud,
CDN y CDO), 2) ecosistemas (código, Bioma, modelo climático, cobertura vegetal), 3)
datos de clima (Tmax/min, HRmax/min, Vel.Vient.max/min, hPamax/min, UVmax/min,
DUmax/min) y 4) datos de suelo (pH, CO, N, Ca, K, Mg, Na Al, CCE, CIC, P Cu, Fe, Mn,
Zn, B, Ar, L, A, Textura) y estimativos e índices de diversidad (Brillouin, Menhinick,
Shannon, Simpson, Chao-1, Chao-2; Jacknife-1, Jacknife-2, ACE).
Variograma para variables espaciales. Los semivariogramas experimentales se
construyeron mediante el procedimiento ‘VARIOGRAM’ del paquete estadístico SAS
(SAS-Institute, 2002). Variograma es la varianza menos la covarianza en función de la
distancia entre dos puntos. El Variograma al estar definido únicamente por la varianza no
tiene ninguna dependencia del tiempo; únicamente del incremento sobre la distancia en
el espacio (h) de los puntos de medición. Es decir, el conocimiento del Variograma
permite siempre evaluar la varianza del estimador. Esta condición es privilegiada cuando
se analizan datos espaciales variados. El Variograma es la herramienta matemática
usada tradicionalmente para representar, en este caso, la variabilidad ambiental de una
medición. Los parámetros de los semivariogramas se muestran en la Tabla 1-6. Los
semivariogramas teóricos fueron ajustados a los semivariogramas experimentales a
través de la transformación logarítmica de las propiedades medidas, lo cual sugiere que
este orden de magnitud es más apropiado que con los valores reales de las propiedades
Con los variogramas ajustados a un modelo matemático (Santalla et. al., En: http:// www.
secforestales.org/buscador/pdf/4CFE05-324.pdf), es posible elaborar
mapas de
gradiente continuo de ocupación para la especie. Estos (mapas) se obtienen con el
método de Kriging (Olea, 1991), herramienta Geoestadística que utiliza la estructura
espacial de los datos para estimar los valores de las variables en lugares no
muestreados. Los estimadores de Kriging se caracterizan por generar una predicción
insesgada donde la varianza del error de predicción es la mínima posible (En:
http://www.secforestales.org/buscador/pdf/4CFE05-324.pdf).
Índices de Moran (I)- y Geary (C). La herramienta Autocorrelación espacial (I de Moran
global) mide la autocorrelación espacial basada en las ubicaciones y los valores de las
entidades simultáneamente. Dado un conjunto de entidades y un atributo asociado,
evalúa si el patrón expresado está agrupado, disperso o es aleatorio. La herramienta
calcula el valor del Índice I de Moran y una puntuación z y un valor P para evaluar la
significancia de ese índice. Los valores P son aproximaciones numéricas del área debajo
28
Título de la tesis o trabajo de investigación
de la curva de una distribución conocida, limitada por la estadística de prueba (En:
http://help.arcgis.com/es/arcgisdesktop/ 10.0/help/index. html#//005p0000000t000000).
Ver Tabla 1-1.
Autocorrelación (AE), Dependencia (DE) y Heterogeneidad Espacial (HE). La
autocorrelación espacial (AE) es la concentración o dispersión de los valores de una
variable en un mapa (Mur, 1992; Vilalta, 2004). Es decir, refleja el grado en que objetos
en una unidad geográfica son similares a otros –objetos-- en unidades geográficas
próximas. La dependencia espacial (DE) se produce cuando “el valor de la variable
dependiente en una unidad espacial es parcialmente función del valor de la misma
variable en unidades vecinas”. Esto significa que, la autocorrelación es sustantiva y no
existe un factor de aleatoriedad. En el análisis de datos agregados geográficamente es
frecuente encontrar que los valores de las variables estén autocorrelacionados
espacialmente o sean espacialmente dependientes. En síntesis, la AE hace
simultáneamente referencia a un fenómeno y técnica estadística y la DE a la explicación
teórica (Vilalta, 2004). La HE se refiere a la variación de las relaciones entre las variables
en el espacio. En términos teóricos, la heterogeneidad espacial se debe una variación
real y substantiva que evidencia la existencia y la validez del contexto geográfico en la
definición de un comportamiento social. Un ejemplo sería cuando la población de cierta
clase social apoyara a un partido político en una ciudad o región, mientras que en otra
ciudad o región la población de la misma o muy similar clase social apoyara a un partido
opuesto. La HE se puede presentar debido a (1) simplemente un problema estadístico
por consecuencia de la heterocedasticidad en un modelo de regresión, o bien (2) al igual
que la DE por la existencia de una variación espacial sustantiva de la variable en
cuestión; en este último caso igualmente el problema de la heterocedasticidad estará
presente (Vilalta, 2004).
Tabla 1-1:
Autocorrelación espacial (Índices: global de Moran y Geary).
Geary
0<c<1
c=1
c>1
Moran
i>0
i=0
i<0
Interpretación
Existe autocorrelación, los datos están agrupados
Los datos están agrupados
Autocorrelación negativa
Diversidad de la especie. Se realiza usando GIS Arc-View 3.2 sobre los mapas
continuos obtenidos para la especie en aplicación de los variogramas. Se entiende por
diversidad (de la especie) la cuantificación numérica del grado de riqueza y abundancia
de una especie en un territorio. Para el cálculo se usó el índice de: Brillouin, Menhinick,
Shannon, Simpson; y los estimativos: Chao-1, Chao-2; Jacknife-1, Jacknife-2, ACE,
usando DIVA-GIS.
Características del producto. El Estimativo Final de Comunalidad-EFC (o Estimativos
de Comunalidad Final-ECF) es la suma de cuadrados promedio de n factores de criterio
Ecuación 1-1. La estrategia: -Transforma variables heterogéneas en (otras) continuas
neutras, -Genera un valor (de peso) diferencial a cada incluida en el análisis y -Calcula
ECF o EFC total y por variable de respuesta. A mayor número de variables, menor
número de Factores de criterio.
EFC=1/nFactores *Σ Factori2
(1-1)
Capítulo 2
29
1.1.3 Resultados
Análisis de Componentes Principales-ACP conformante de la matriz de Marcadores
Ecológicos-ME.
Once vectores propios (Eigenvectors) con valor propio (Eigenvalue) ≥1 permitieron
ecotipificar los 59 morfotipos de Lippia alba conformantes de la muestra final de trabajo.
Los valores propios generados por el análisis mostraron 11 Componentes PrincipalesCP-explicando el 87.56% de la varianza total hallada en la muestra de trabajo. La
varianza explicada por cada factor estuvo en el rango 1.08- 15.08 (Tabla 1-2). Los
Estimativos de Comunalidad final variaron entre 0.55 y 0.99 y el valor final fue 42.9
(Tabla 1-2). Los R2 de las variables con cada Factor lograron el valor máximo de 1. Los
coeficientes estimados por regresión, los valores de R2 de las variables con cada Factor
y los respectivos coeficientes estandarizados para cada variable dentro de cada Factor
se muestran en el Anexo A. La varianza explicada por cada factor o varianza del factor,
expresado como la proporción entre la suma de cuadrados de los pesos de cada factor y
el número de Factores, se muestran en la Tabla 1-2.
Tabla 1-2:
Varianza explicada por cada Factor, correspondiente al Valor Propio (o
Eigenvalue) para cada variable incluida en la prueba.
Proportion
Cumulative
1
15.0850356
Eigenvalue
9.2837434
Difference
0.3079
0.3079
2
5.8012922
1.4231263
0.1184
0.4263
3
4.3781659
0.5384658
0.0894
0.5156
4
3.8397002
0.2952782
0.0784
0.5940
5
3.5444219
0.7541372
0.0723
0.6663
6
2.7902847
0.8761632
0.0569
0.7232
7
1.9141216
0.3145248
0.0391
0.7623
8
1.5995967
0.1197086
0.0326
0.7950
9
1.4798882
0.0944603
0.0302
0.8252
10
1.3854279
0.2993993
0.0283
0.8534
11
1.0860286
0.1579186
0.0222
0.8756
Cuarenta de 46 variables fueron retenidas por los 11 Factores para crear los marcadores
ecológicos (Tabla 1-2 y Figura 1-2). Los valores de Comunalidad Final permite la
conformación de al menos tres grupos de variables: uno de 0.90 a 1.0, otro de 0.81 a
0.899 y un tercero de 0.53 a 0.798. Región y los índices y estimativos de diversidad
destacaron los valores más altos. Las únicas variables agrupadas fueron las de
diversidad. Las demás variables estuvieron repartidas por todo el rango. Zn, Mn, Na y Cu
y Textura destacan por sus valores más bajos de 0.7. Las demás variables tuvieron
valores entre 0.8 y 0.977 (Tabla 1-3).
30
Título de la tesis o trabajo de investigación
La Tabla 1-3 y la Figura 1-2 presentan las comunalidades obtenidas para cada una de
las variables. Variables con valores altos de comunalidad >0.90, explican en mayor
proporción la varianza según su participación en los factores o componentes resultantes
en el análisis, mientras que variables que presenten valores bajos (< 0.60) participan en
menor medida en la explicación de dicha varianza. Los criterios de clasificación de los
valores de comunalidad son arbitrarios y su rango de escala debe sustentarse. Aquí,
proponemos tres grupos.
Tabla 1-3:
Estimativo de Comunalidad Final-ECF- total y para variables.
No.
Variable
1
Región (Sumapaz-Chicamocha)
42
Índice de Margalef
44
Índice de Shannon
45
Índice de Simpson
46
Índice de Brillouin
47
Estimativo de Chao-1
48
Estimativo de Chao-2
49
Estimativo de Jacknife-1
50
Estimativo de Jacknife-2
43
Estimativo de Menhinick
EFC
No.
Variable
(ECF)
0.9977 8 Cobertura vegetal
Ar=contenido de arcilla en
0.9977 38
suelo
CIC=Capacidad de
0.9977 31
intercambio catiónico
hPa máx.=Presión
0.9977 15
atmosférica máxima
0.9977 6 Bioma=Tipo de bioma
hPa min= Presión
0.9977 16
atmosférica mínima
0.9977 7 Modelo climático
Mg=contenido de magnesio
0.9977 27
en suelo
B=contenido de boro en
0.9977 37
suelo
Temp. Máx.=Temperatura
0.9973 9
ambiental máxima
K=contenido de potasio en
0.9922 26
suelo
EFC
(ECF)
0.9077
0.8985
0.8954
0.8856
0.8831
0.8823
0.8782
0.8676
0.8622
0.8539
CDN=Coordenada Decimal
Norte
CDO=Coordenada Decimal
0.9858
Oeste
CICE=Capacidad de Intercambio
0.9548
efectivo de cationes en suelo
22
24
Nitrógeno
0.9449
29
40
Arena
0.9418
20
23
CO=Carbono orgánico
HR min=Humedad relativa
mínima
0.9361
39
0.9338
34
5
Código de clima
0.9313
32
25
Ca=Calcio en suelo
0.9236
14
Altitud= altitud sobre el nivel
del mar
Al=contenido de Aluminio
en suelo
Du min.=Columna de ozono
mínima
L=fracción de limo
Fe=fracción de hierro en
suelo
P=fracción de fósforo en
suelo
Vel.Viento min.
10
Temp. Min=Temperatura mínima 0.9231
36
Zn=fracción de cinc
0.6784
18
UV min=Radiación ultravioleta
Vel. Viento máx.=Velocidad del
viento máxima
HR máx.=Humedad relativa
máxima
UV máx.=Radiación ultravioleta
máxima
DU máx.=Columna de ozono
máxima
0.9207
35
0.6424
0.9195
41
0.9168
28
0.9147
33
Mn=fracción de manganeso
Textura=textura de la
muestra de suelo
Na=fracción de sodio en
suelo
Cu=fracción de cobre en
suelo
3
4
30
12
13
11
17
19
0.9081
2
pH= acidez del suelo
0.8368
0.8097
0.7979
0.7963
0.7793
0.7793
0.77
0.7495
0.7067
0.6365
0.5514
0.5301
Capítulo 2
31
Figura 1-2: Grupo de variables con valor de peso (Estimativos de Comunalidad FinalECF), usadas por el procedimiento FACTOR para conformar la matriz de Marcadores
Ecológicos-ME.
EFC (ECF)
Matrices de Marcadores Ecológicos-ME. Los Anexos A y B muestran las matrices de
ME agrupado con base en Vectores Propios (Eigenvalue) y Factores. Los valores fueron
estimados usando el procedimiento FACTOR de SAS/STATv9.2; y las variables con una
solo tipo de respuesta reportadas por el LOG, fueron eliminadas del editor para
maximizar la potencia del análisis. Los valores de la tabla representan el peso de cada
variable dentro del factor. En síntesis, estas dos matrices representan la línea base (de
datos) de los Marcadores Ecológicos.
Agrupamiento. La Figura 1-3 muestra el agrupamiento generado por los 11 Factores.
Se usó el Método Ward (Ward, 1963; Franco et al., 2010) con el procedimiento Cluster de
SAS/STATv9.2. Dos grandes grupos conforman el conglomerado final. Agrupamiento por
variables ambientales; faltando agregar las variables derivadas de los marcadores
genéticos (moleculares y fitoquímicos). Aparecen dos grandes grupos con accesiones
intercaladas de las dos regiones. No hay una separación por región. Hay accesiones muy
cercanas entre si y otras muy distantes, un indicador de variabilidad entre accesiones. Se
sustenta el método Ward (Hu et al., 2000; Grun & Atieno, 2007) ya que este forma parte
de la estrategia WARD-MLM de Franco et al. (2007; 2010) usado para la conformación
final de grupos (Franco et al., 2010). Adicionalmente los agrupamientos conformados por
la metodología, están más cerca de la realidad, ya que los individuos están muy
relacionados ya que su distribución en Colombia es de carácter antropológico por madres
y abuelas quienes lo han distribuido y llevado a casi todas las regiones colombianas. Se
32
Título de la tesis o trabajo de investigación
denotan máximo cuatro subgrupos, los cuales deben ser coincidentes con los
agrupamientos generados por los factores genéticos (moleculares y fitoquímicos) y por la
gran matriz final que incluye todos los marcadores.
Figura 1-3: Dendograma generado por 11 factores considerados determinantes de la
estructura ecológica de L. alba en dos regiones colombianas.
Discusión principal
Un número relativamente pequeño de Factores (11 de 40), explicaron la variabilidad
presente en la muestra de trabajo. Todas las variables ambientales acompañantes de
cada accesión (datos crudos) fueron traducidas a variables sintéticas (Factores), dado su
neutralidad y carácter cuantitativo continuo, lo cual optimiza los procedimientos de
clasificación. Se usaron dos vías de análisis los procedimientos PRINCOMP y FACTOR
de SASv9, estimado CP y Factores respectivamente, con base en matrices de
correlaciones (para datos multiunidades) y con matrices de varianza-covarianza para
datos con igual unidad de medición. Las pruebas de calidad para la metodología analítica
usadas sustentan la calidad del procedimiento empleado (potencia de la prueba). Para
mayor confiabilidad de los datos climáticos, los valores máximos y mínimos fueron
ajustados usando el paquete DIVA-GIS. La matriz resultante fue usada por la estrategia
WARD-MLM/SAS-STATv9.2 para conformar los posibles grupos. La salida (output) del
ACP del Anexo A reporta la matriz de Factores (Factor Pattern) y la varianza explicada
por cada Factor de criterio.
Respecto al número de variables efectivas, teóricamente se espera un numero bajo de
variables predictoras pobres. Con excepción del valor para Sodio y Cobre en suelo, las
proporciones presentadas por cada variable, no son valores críticos pues están por
encima de 0.50. El análisis sugiere buscar nuevas variables y probar su efectividad. Las
variables pobremente explicadas (por los 11 factores) debe ser sustituidas por otras más
potentes o simplemente excluirlas de futuras pruebas. Y las variables potentes, pueden
Capítulo 2
33
conformar a futuro el grupo de variables canónicas. Los análisis canónicos, no incluidos
en este estudio, son determinantes de variables predictoras potentes.
Los valores reportados para variables climáticas fueron ajustados en espacio y tiempo
usando DIVA-GIS. Muchas otras variables ambientales pueden conformar la matriz de
marcadores ecológicos; de hecho, es importante incluir variables de tipo fisiológico,
microbiológico de suelo, de fauna y demás posibles. Para estas (nuevas variables), los
análisis de CP las valoran y define su inclusión/exclusión como marcadores ecológicos.
Los factores obtenidos en la prueba son válidos dado que están compuestos por valores
propios acorde a la cantidad y calidad del vector propio resultante.
Los estimativos de comunalidad final explican la variabilidad de una variable X dentro
del grupo de Factores comunes. Por tanto, sus valores oscilarán entre cero y uno,
es decir, entre la posibilidad de que los factores comunes no expliquen nada de la
variabilidad de una variable o que por el contrario ésta quede totalmente
explicada por los factores comunes. Los Criterios de selección de número de
Factores se explican en la metodología.
Patrones de variación espacial de L. alba
Autocorrelación-AE, Dependencia-DE- y Heterogeneidad Espacial-HE
Índices Ecológicos-IE. El coeficiente I de Morán-AE- para todas las observaciones
mostró un patrón espacial completamente aleatorio con una muy leve tendencia a la
dispersión (I=-0.0086799 y Neihborg Lag Distance=0.2798). En cada unidad geográfica
(o región) el I de Morán (AE) mostró patrones completamente aleatorios con una muy
leve tendencia a la concentración (I=0.020662478) en la Región del Sumapaz; y
tendencia muy leve a la dispersión (I=-0.0042829) en (la Región del) Chicamocha. Los
procedimientos VARIOGRAM y ARIMA de SAS/STATv9.2 de las Figuras 1-4 y 1-5
muestran los valores de autocorrelación, verificando el patrón completamente aleatorio
del Índice Morán (0.0086799) e índice de Geary (1.19833). Tabla 1-4.
Discusión. Para la estadística I de Moran, la hipótesis nula establece que el atributo que
se analiza está distribuido en forma aleatoria entre las entidades del área de estudio. Es
decir, los procesos espaciales que promueven el patrón de valores observado
constituyen una opción aleatoria.
34
Título de la tesis o trabajo de investigación
Tabla 1-4:
Salida (output) de autocorrelaciones para 14 Lags (=distancia entre clases)
y residuales usando el modelo ARIMA AR(1).
Lag Covariance Correlation -1 9 8 7 6 5 4 3 2 1 0 1 2 3 4 5 6 7 8 9 1
Std Error
0
1.942956
1.00000 |
|***********************|
1
0.528674
0.27210 |
. |*****
|
0.130189
2
0.027139
0.01397 |
.
|
.
|
0.139495
3 -0.044471
-.02289 |
.
|
.
|
0.139519
4
0.081036
0.04171 |
.
|* .
|
0.139582
5
0.210145
0.10816 |
.
|** .
|
0.139794
6
0.231860
0.11933 |
.
|** .
|
0.141205
7
0.244222
0.12570 |
.
|*** .
|
0.142904
8 -0.090658
-.04666 |
. *|
|
0.144766
.
0.000000
9
0.102379
0.05269 |
.
|* .
|
0.145020
10
0.057306
0.02949 |
.
|* .
|
0.145344
11
0.314054
0.16164 |
.
|*** .
|
0.145446
12
0.074161
0.03817 |
.
|* .
|
0.148459
13
0.199074
0.10246 |
.
|** .
|
0.148625
14 -0.023888
-.01229 |
.
|
|
0.149818
.
Autocorrelation Check for White Noise
Lag
6
12
Square
DF
To
ChiSq
ChiPr >
--------------------Autocorrelations--------------------
6.50
6 0.3695 0.272
0.014 -0.023
10.08 12 0.6088 0.126 -0.047 0.053
0.042
0.029
0.108
0.162
Maximum Likelihood Estimation
Parameter
MU
AR1,1
Standard
Estimate
2.04177
0.43659
Error
Approx
t Value
0.30719
0.15887
6.65
2.75
Pr > |t|
<.0001
0.0060
Lag
0
1
0.119
0.038
Capítulo 2
35
Figura 1-4: Autocorrelaciones para 14 Lags (=distancia entre clases) y residuales
usando el modelo ARIMA AR(1).
3
For
L i mi t s
n=1:
7. 5
R
e
s
i
d
u
a
l
5. 0
UCL = 3 . 0 2
2. 5
X= . 0 4
0
- 2. 5
L CL = - 2 . 9 3
- 5. 0
100
F
o
r
e
c
a
s
t
75
50
25
0
0
10
20
30
40
50
60
70
80
90
Ob s e r v a c i o n
Discusión
Cada colecta mostró valores residuales puntuales individuales (Figura 1-4). Alrededor
del 90% de las observaciones están dentro del rango de no diferencias entre ellas para
los marcadores ecológicos con diferencias no significativas o dentro de un rango normal
aceptable (UCL=3.02/LCL=2.93). Las observaciones cercanas a los límites superior e
inferior indican la presencia de al menos 4 observaciones con características muy
particulares. Este resultado demuestra una variabilidad ecológica relativamente baja pero
significativa; la cual puede verificarse determinando índices ecológicos los cuales
sustentarán los resultados del presente análisis.
Usando información ecológica y geográfica es posible predecir patrones explicando la
estructura genética espacial de poblaciones naturales. Los procedimientos Variogram y
Kriging reportan patrones completamente aleatorios (SAS/STATv9.2). Existe
autocorrelación cuando los valores de objetos cercanamente geográficos son más
similares que los objetos lejanos. Por ejemplo, una cuadrícula de altitud tiene una
elevada autocorrelación espacial. La opción de autocorrelación en DIVA-GIS calcula esta
relación mediante el uso de dos estadísticas comunes, Geary y Moran. Se puede calcular
la autocorrelación de un archivo de cuadrículas o de un archivo de puntos (DIVA-GIS
Manual, 2004 En: www.diva-gis.org/docs/DIVA-GIS5_manual.pdf.). La funcionalidad de
autocorrelación espacial implementada en DIVA-GIS está basada en el software
“Rookcase” creado por Sawada (1999).
El conocimiento de la estructura del patrón espacial en una población es de gran utilidad
para la formulación de los modelos ajustados de análisis espacial (Zas et al., 2008). En
estudios genéticos poblacionales, los efectos de la autocorrelación espacial afectan la
estimación de los componentes de varianza y parámetros genéticos derivados. La
36
Título de la tesis o trabajo de investigación
gravedad de estos efectos varía en función de la intensidad y escala del patrón espacial
(Zas et al., 2008).
Estructura espacial de la variación genética de la muestra de L. alba
La estructura espacial de la variación genética de la muestra de L. alba en Colombia se
muestra en la Figura 1-5. Se presenta su estructura espacial con base en 40 variables
ambientales tomadas en 59 accesiones de Lippia alba en dos regiones colombianas
(Sumapaz o Región 0 y Chicamocha o Región 1). La estructura espacial se analizó a
partir de los semivariogramas residuales, los cuales sintetizan la dependencia espacial
de los residuos del modelo estadístico, mediante la representación de la variación de la
similitud entre individuos (o accesiones) en función de distancias conocidas (Tabla 1-5).
Tabla 1-5:
Salida (output) original de valores de los variogramas teórico y ajustado
(robusto) para 14 observaciones.
Lag Class Value(in number of Average Lag)
Distance
Robust
Observación Units Class
Variogram
for class
Variogram
1
2
3
4
5
6
7
8
9
10
11
12
13
14
1
0
1
2
3
4
5
6
7
8
9
10
11
12
59
39
71
54
69
94
50
60
61
38
35
39
43
22
0.00000
0.00656
0.02826
0.06142
0.09173
0.11954
0.15111
0.18182
0.21094
0.23914
0.27190
0.30351
0.33079
0.35944
0.00000
0.27378
0.59868
1,08926
1,096653
0.47256
1,48825
0.57958
0.70233
0.70327
0.93320
1,82804
0.83882
3,22711
0.00000
0.18000
0.32153
0.75056
1,34748
0.35351
0.86429
0.61786
0.55948
0.40211
0.74633
1,22908
0.66747
2,06658
Capítulo 2
Figura 1-5:
robusto.
37
Variograma ajustado experimental, con valores: experimental teórico y
Los rangos (distancias) son similares para todas las localidades, dentro del tamaño
modal de las áreas de estudio. Esto destaca la importancia de los suelos, el cual es
normalmente homogéneo dentro de una serie de suelos: la variabilidad espacial de los
sitios de muestreo es dependiente del espacio dentro de los límites de la fuente de
variación identificada en este caso como es el tipo de suelo. Entre regiones, el patrón de
variación espacial es demasiado complejo, hasta el punto de no encontrarse correlación
espacial entre los puntos y las distancias muestreadas. Sus semivariogramas
experimentales fueron ajustados al semivariograma esférico el cual ajusta mejor los
datos a distancias relativamente cortas entre 200 y 400 metros aproximadamente, los
cuales corresponden con los rangos ajustados de los semivariogramas de la tabla 1.
Aunque estas distancias son más cortas que la máxima distancia muestreada (1000 m)
es posible que la variabilidad espacial a distancias cortas haya sido subestimada.
Los bajos valores de pepita nos indican que una fracción de la varianza ha sido
detectada por el modelo, debido a errores de medida. El análisis del Variograma permitió
estimar las distancias óptimas de muestreo para las propiedades medidas tanto en la
Región 0 como en la Región 1, ver Tabla 2. Los variogramas muestran la relación entre
la varianza del conjunto de medidas a cada distancia medida y la distancia de muestreo.
El cambio de la varianza de una distancia (nivel de muestreo) a otra nos indica la
distancia de muestreo más eficiente (distancia óptima de muestreo).
Región 0 (Sumapaz)
La distancia del Rango o Alcance a0 fue 0.302, el patrón Nuggets o efecto pepita
(cn=0.15) fue pequeño, la varianza del parche (co) y la varianza total fueron 1.80 y 1.95
38
Título de la tesis o trabajo de investigación
respectivamente. En la Figura 1-6 se muestran los tres parámetros que caracterizan la
estructura espacial: el rango (ao=0.3005) que es la distancia a la que se pierde la
dependencia espacial e indica la escala del patrón, el Nuggets (cn) o semivarianza a
distancia cero que representa la varianza debida a errores de muestreo y/o ocasionada
por la dependencia espacial a escalas inferiores a las muestreadas, y la varianza del
parche (co) que refleja la intensidad de la dependencia espacial. La asíntota o sill
(cn+co) es un estimador de la varianza total de la variable. La intensidad del patrón
espacial viene dada por el cociente entre la varianza del parche y la asíntota
(co/(co+cn))= 0.92307692.
Figura 1-6: Semivariograma
ajustado (línea). Se indican los tres parámetros
caracterizantes de la estructura espacial: el rango (ao=0.305); nugget (cn); Varianza
mayor (co) y la intensidad del patrón espacial Sill= (cn+co) o varianza total, para la región
Sumapaz.
Rango
Pr i n c i p a l
a0
Va r i o g r a m
2. 0
Co mp o n e n t
An a l y s i s
1. 9
1. 8
1. 7
1. 6
1. 5
1. 4
1. 3
1. 2
1. 1
Varianza del parche
1. 0
co
0. 9
0. 8
Sill
(cn+co)
0. 7
0. 6
0. 5
0. 4
0. 3
Nugget
cn
0. 2
0. 1
0. 02
0. 04
0. 06
0. 08
0. 10
0. 12
0. 14
0. 16
0. 18
0. 20
0. 22
Av e r a g e
0. 24
Lag
0. 26
Di s t a n c e
0. 28
0. 30
f or
Cl a s s
0. 32
0. 34
0. 36
0. 38
0. 40
0. 42
0. 44
0. 46
0. 48
Discusión. Para la Región del Sumapaz o Región 0, el Variograma sugiere que los
puntos (de registro) separados por distancias cortas están correlacionados y la
correlación decrece a medida que la distancia entre puntos se incrementa. Esto puede
verse en el incremento en el Variograma hasta una distancia residual cercana a 0.3005.
En el presente estudio las variables (incluida en la prueba) están correlacionadas
espacialmente, debido a que el Variograma aparece decreciente a lo largo de las
distancias residuales (Lag) posterior al Rango a0, indicando una variabilidad espacial alta.
Región 1 (Chicamocha)
La distancia a0 fue 0.2, el patrón nugget (cn=0.1) fue pequeño, la varianza de parche (co)
fue de 0.3 y la varianza total de 0.4. En la Figura 1-7 se muestran los tres parámetros
que caracterizan la estructura espacial: el rango (ao=0.68) que es la distancia a la que se
pierde la dependencia espacial e indica la escala del patrón, el nugget (cn) o
semivarianza a distancia cero que representa la varianza debida a errores de muestreo
y/o ocasionada por la dependencia espacial a escalas inferiores a las muestreadas, y la
varianza del parche (co) que refleja la intensidad de la dependencia espacial. La asíntota
o sill (cn+co) es un estimador de la varianza total de la variable. La intensidad del patrón
Capítulo 2
39
espacial dada por la relación entre la varianza del parche y la asíntota (co/(co+cn)) fue
igual a 0.75.
Figura 1-7: Semivariograma
experimental ajustado (línea). Se indican los tres
parámetros caracterizantes de la estructura espacial: el rango (ao=0.2); nugget (cn=0.1);
Varianza de parche (co=0.4) y la intensidad del patrón espacial Sill=(cn+co), para la
región Chicamocha.
Pr i n c i p a l
Co mp o n e n t
An a l y s i s
Va r i o g r a m
3
2
1
0
0. 0
0. 1
0. 2
0. 3
0. 4
Av e r a g e
0. 5
Lag
0. 6
Di s t a n c e
f or
0. 7
0. 8
0. 9
1. 0
1. 1
Cl a s s
Discusión
La meseta tuvo un valor relativamente bajo de a0=0.2 comparado con la Región 0
(Sumapaz). El alcance fue de 0.2 y la varianza neta fue de 0.3. La intensidad del Patrón
espacial fue de 0.75. La forma general de la línea sugiere variabilidad ambiental alta, es
decir, la presencia de dos o ecosistemas en áreas relativamente pequeñas.
Para la Región del Chicamocha o Región 1, el Variograma sugiere que los puntos (de
registro) separados por distancias cortas están correlacionados y la correlación decrece a
medida que la distancia entre puntos se incrementa. Esto puede verse en el incremento
en el Variograma hasta una distancia residual cercana a 0.3005. En el presente estudio
las variables (incluida en la prueba) están correlacionadas espacialmente, debido a que
el Variograma aparece decreciente a lo largo de las distancias residuales (Lag) posterior
al Rango a0.
Índices de diversidad (Tabla 1-6)
El software DIVA-GIS generó el siguiente resumen para las 59 colectas incluidas en la
muestra de trabajo. El índice de Margalef en este estudio reporta el número de individuos
diferentes al interior de la muestra de trabajo compuesto por 59 accesiones (o entradas).
El índice de Simpson expresa la probabilidad de que dos individuos al azar dentro del
grupo de colectas, sean distintos. El valor de 1 indica baja diversidad.
40
Título de la tesis o trabajo de investigación
Tabla 1-6:
Índices y estimativos de diversidad obtenidos como máximo indicador de
variación ambiental.
Diversidad
Índice
Margalef
Menhinick
Shannon
Simpson
Brillouin
Estimativos
Índice
Chao-1
Chao-2
Jacknife1
Jacknife2
ACE
Valor
14.224
7.681
4.078
1.000
3.128
Valor
1779.500
921.875
117.000
173.983
-1.000
Capítulo 2
41
2. Capítulo 2
2.1 Diversidad molecular de accesiones colombianas de
Lippia alba (Mill.) N.E. Brown
2.1.1 Introducción
Lippia alba: taxa, descripción botánica y diseminación
Taxón: Lippia alba (Mill.) N. E. Br. ex Britton & P. Wilson. Nombres comunes:
[prontoalivio (Colombia), erva-cidreira (Brasil), juanilama (Costa Rica), salvia morada
(Argentina), bushy matgrass, Bushy Lippia Oaxaca lemon verbana, melissa] (En:
http://www.ars-grin.gov/cgi-bin/npgs/html taxon). La mayor parte de la Verbenaceae se
compone de dos grupos ricos en especies: el complejo sensu Verbena/Glandularia con
170 especies, y el complejo Lantana/Lippia/Aloysia con 200–400 especies (Yuan et al.,
2009). Es un arbusto aromático de 1-1,15 metros de altura, con ramas largas, delgadas y
dispuestas en ángulo; hojas opuestas o en grupos de tres, de 1-3 centímetros de largo,
oblongo-elípticas de obtusas a redondeadas, bordes aserrados, superficies cubiertas de
vellosidades, venas prominentes, inflorescencias en capitulo o espigas de 2 centímetros
de largo, flores nectaríferas tubulares de 4.5 mm*1.5 mm, blancas, rosadas o púrpura
suave (Seaforth y Tikasingh, 2008; Castro et al., 2002; Muñoz et al., 2007; En:
http://aplicaciones2.colombiaaprende.edu.co/concursos/ expediciones botanicas/ fotos/
5721351.jpg; Caetano et al., 2011). La reproducción sexual presenta características de
especie alógama, típico en verbenáceas silvestres (Suárez et al., 2008; Caetano et al.,
2011). En L. alba, anormalidades postmeióticas afectan la formación de granos de polen
viables y es determinante de viabilidad y germinación baja de las semillas, presentando
problemas importantes en su reproducción sexual (Muñoz et al., 2006; Caetano et al.,
2011; Herrera et al., 2013). Su forma asexual de reproducción es fácil, rápida y exitosa
(estenopropagación). Como sinónimos para L. alba (Mill.) N.E.Br. están: Lantana alba
Mill., Lippia geminata Kunth., Lantana germinata (Kunth.) Spreng. (Linares, 2007;
Seaforth y Tikasingh, 2008).
Es usada como ingrediente natural en cosmetología (aromaterapia) e industria
farmacéutica (Wezel, 2003; Montanari et al., 2004; Trujillo, 2004; UNCTAD, 2005;
Seaforth and Tikasingh, 2008). Es una de las plantas medicinales más importantes
42
Título de la tesis o trabajo de investigación
utilizadas, entre otros, por pobladores brasileros (Castro et al., 2002; UNCTAD, 2005;
Oliveira et al., 2006); especialmente aprovechada por sus propiedades somáticas,
sedativas, antidepresivas y analgésicas (Schroeder et al., 2004; UNCTAD, 2005;
Hennebelle et al., 2007). El aceite esencial de L. alba tiene además usos como
estomático, antiespasmódico, digestivo, anti-hemorroides y anti-asmático (Vit et al., 2002;
Schroeder et al., 2004; UNCTAD, 2005; Hennebelle et al., 2008).
Lippia alba y L. origanoides han sido las Verbenáceas más ampliamente reportadas
(Stashenko et al; 2003-2004; Jarvis et al., 2006; Hennebelle et al., 2006; Vera et al.,
2010; Jaramillo et al., 2010) como promisorias por los componentes de sus aceites
esenciales (Borneol, camfor, 1,8- cineole, citronellol, geranial, linalool, myrcene, neral,
piperitona, sabineno, 2-undecanona, - muuroleno, - cariofileno, - cubebeno, - elemeno, cadineno). In vitro, las dos especies han demostrado efecto antimicrobiano en humanos
(Vera, 2007; Cardona, 2008-2009; Camacho et al., 2010) y en plantas (Álvarez, 2008;
Erazo 2010; Guerrero, 2010). No obstante, la cantidad de componentes principales de
sus aceites esenciales varían con el sitio de colecta, con diferenciaciones importantes
especialmente en las cantidades de p-cymene, -terpinene, thymol, carvacrol, butyl
hydroxy anisole (Jarvis et al., 2006; Duran, 2007; Vega et al., 2013).
Rango de distribución natural. El clade Verbeneae se distribuye primariamente en
Latinoamérica donde ocurre en un rango amplio de ecosistemas, con un segundo centro
de origen en África (Marx et al., 2010). Inferencias filogenéticas sugieren a Suramérica
como lugar de origen de las Verbenaceae con aproximadamente seis eventos de
colonización, los cuales dieron origen a las especies presentes en el viejo mundo (Marx
et al., 2010). El análisis filogenético realizado por Marx et al. (2010) mostró ocho clades
reconocidos como tribus (Casselieae, Citharexyleae, Duranteae, Lantaneae,
Nerosportoneae, Petreae, Priveae, Verbeneae). Lantaneae y Verbeneae forman un clade
con aproximadamente 2/3 de las especies en el clade Verbeneae.
Su área más probable de origen va de México a Argentina y en las Antillas (Vit et al.,
2002; Arango, 2004; UNCTAD, 2005). La especie se distribuye desde Norteamérica (Ver:
conservation status in U.S. & Canada in NatureServe Explorer database) Centro-Sur de
E.U.A.: Estados Unidos de América- Texas; Norte de Mexico: Mexico - Baja California,
San Luis Potosi, Sinaloa, Tamaulipas, Zacatecas; Sur de Mexico: Mexico- Campeche,
Chiapas, Colima, Guanajuato, Guerrero, Jalisco, Mexico, Michoacán, Nayarit, Oaxaca,
Tabasco, Veracruz, Yucatán. En Suramérica por el Caribe: Islas Caimán; Cuba;
Republica Dominicana; Haití; Jamaica; Puerto Rico; Trinidad & Tobago; Mesoamérica:
Belice; Costa Rica; El Salvador; Guatemala; Honduras; Nicaragua; Panamá. Norte de Sur
America: Guayana Francesa; Guyana; Surinam; Venezuela. Brasil: Brasil - Amazonas,
Bahía, Ceará, Goiás, Maranhao, Mato Grosso, Minas Gerais, Para, Paraná, Pernambuco,
Rio Grande do Norte, Rio Grande do Sul, Rio de Janeiro, Rondonia, Santa Catarina, Sao
Paulo. Sur América occidental: Colombia; Ecuador - Bolívar, Guayas, Los Ríos, MoronaSantiago, Napo; Perú - Amazonas, Huánuco, La Libertad, Lambayeque, Lima, Loreto,
Pasco, Piura, San Martin, Ucayali. Sur de Sur América: Argentina - Buenos Aires, Chaco,
Corrientes, Entre Ríos, Formosa, Jujuy, Misiones, Santa Fe; Paraguay (En:
http://www.ars-grin.gov/cgi-bin/npgs/html/taxon.pl?22367). También se encuentra en
Suráfrica (Cicció y Ocampo, 2006), India (Gurdip et al., 1999) y Australia (En:
http://biocahe.
ala.org.au/occurrences/search?q=lsid:urn:lsid:biodiversity.org.au:apni.
taxon: 313500#.).
Capítulo 2
43
Registros biológicos de L. alba en Colombia. No existe un registro fitogeográfico de L.
alba para Colombia. Los herbarios colombianos reportan 37 colectas en 18
departamentos de Colombia. El Herbario de la Universidad de (1) Antioquia-HUAagenda un ejemplar de L. alba con código 61260 [2008-07-22]. El Herbario Nacional
Colombiano-COL, registra 21 colectas de L. alba en: (2) Santander [Puente Nacional
(Morales A.L. sn), Suaita (Fernández J.L. 21249), Bucaramanga (Jaramillo C., 1).
Bucaramanga, Suaita y Bolívar (Durán et al., 2007), Curití, San Gil, Jordán, Valle de San
José, Aratoca y Pescadero] (En: Informe anual Técnico CENIVAM, 2005), (3)
Cundinamarca [Arbeláez (Salama, A.M. 15, La Vega; Salama A.M. 141), Anolaima], (4)
Tolima [Flandes y Venadillo], (5) Boyacá [Cubará], (6) Antioquia [Puerto Berrio], (7)
Arauca [Saravena, Bolívar, Turbaco y Colorado], (8) Valle del Cauca [Cali y Palmira], (9)
Quindío [Armenia], (10) Cesar [Sn. Martin] (Durán et al., 2007), (11) Bolívar [entre
Sincelejo y Colosó (R. Romero-Castañeda 9295)], (12) Amazonas [Trapecio Amazónico,
entre Puerto Nariño y Loretoyacú, a orillas del río Amazonas (J. M. Duque-Jaramillo
2418)], (13) Guajira [Nazareth (R. Romero-Castañeda 4450), Urbilla, cauce del arroyo en
la salida hacia Ipapure (Carlos Saravia & Donald Jonson 314)], (14) Magdalena
[Municipio de Santa Marta, alrededores de Pueblito, altitud 400 m. (R. RomeroCastañeda 8040)] (García-Barriga, 1975; Linares, 2007). El Herbario Amazónico
Colombiano-COAH- muestra seis registros para L. alba en (15) Putumayo [Mocoa y
Puerto Leguízamo], (16) Caquetá [Florencia], (17) Meta [Mesetas] y (18) Vaupés
[Morichal] (En: http://www.sinchi. org.co/herbariov/ genera_ indice.php). Más 59
accesiones de Lippia alba colectadas en diferentes localidades de dos zonas
agroecológicas colombianas: Región I (Sumapaz) y Región II (Chicamocha). Estas
accesiones forman parte de una colección transitoria (año 2013) establecida en el Centro
Experimental de la Universidad Nacional de Colombia-Sede Palmira-CEUNP.
Descripción citogenética de Lippia alba. La caracterización citogenética hecha por
Dias et al. (2007), Pierre et al. (2010), y Sousa et al., (2011) revela para L. alba un
número variable cromosomas originado por diploidía y poliploidía (Sousa et al., 2011) con
2n=30 cromosomas para el quimiotipo citral, mientras que individuos la carvona mostró
2n=60. Los individuos del quimiotipo linalool revelaron el mismo rango de numero de
cromosomas, de 2n=12 a 2n=60, en diferentes células del mismo individuo, con 2n=44
como el cariotipo más frecuente hallado (Pierre et al., 2011). Dias et al. (2007) detectaron
mediante FISH (Fluorescence In Situ Hybridization) seis sitios 45S rDNA y dos sitios 5S
rDNA, y un análisis meiótico reveló condición cromosómica normal. El número de
cromosomas y la presencia de dos sitios 5S rDNA sugirió un origen poliploide, reportado
por Pierre et al. en 2010 y Sousa et al. (2011) en Brasil, al hallar diferentes citotipos
(diferentes niveles de ploidía e individuos mixoploídes). La mayor parte de la variación
genética del quimiotipo linalool lo describe Pierre et al. (2010) como una consecuencia de
la mixoploidía presente en la especie. La reproducción sexual entre los quimiotipos es
poco probable, sugiriendo aislamiento reproductivo (Pierre et al., 2011). Las
características citogenéticas de L. alba pueden ser marcadores útiles en diferenciación
de especies (Dias et al., 2007), estudios de conservación (Pierre et al., 2011; Sousa et
al., 2011; Santos et al., 2012) y planes-programas de mejoramiento (Santos et al., 2012).
Marcadores moleculares para estudios de diversidad y estructura genética en
plantas. Marcadores moleculares AFLP desarrollados por Vos et al., en 1995 (Beebe et
al., 2001; Larson et al., 2004; Wolf et al., 2004; Grassi et al., 2005; Tero et al., 2005;
Ramalho et al., 2006; Assogbadjo et al., 2006), loci de alloenzimas (Huh et al., 1999;
Culley and Wolfe, 2001; Mariot et al., 2002; Zhang et al., 2002; Wang et al., 2004;
44
Título de la tesis o trabajo de investigación
González et al., 2004; Kang et al., 2005), ISSR (Wang et al., 2003; Xiao et al., 2004;
Sheng et al., 2005; Xie et al., 2005; Ge et al., 2005), ITS del ADN ribosomal (Jaramillo
and Manos, 2001; Ortiz et al., 2005; Suarez et al., 2007), marcadores del ADN del
cloroplasto (cpDNA) (Suárez et al., 2007), RAPD (Wadt et al., 2004; Wang et al., 2003;
del Rio and Bamberg, 2003; Verma et al., 2004; Zhang et al., 2002; Ortiz et al., 2005;
Zucchi et al., 2005), SSR (Zhang et al., 2002; El-Mouei et al., 2011) y Random Amplified
Microsatellite-RAM (Muñóz et al., 2008); han sido usados para investigar los niveles de
variaciones morfométricas, variación genética y estructura genética dentro y entre
poblaciones de especies vegetales promisorias (Assogbadjo et al., 2006). Los resultados
obtenidos con los marcadores moleculares han permitido: (i) proponer estrategias para
conservación y manejo sustentable de algunas especies (ii) determinar evolución
bioquímica, deriva genética, cruzamientos y flujo génico entre poblaciones, (iii) identificar
reservorios potenciales de diversidad de plantas dentro de ecosistemas, (iv) Identificar
factores causantes de cambios en cantidad de variación entre poblaciones y localidades
de centros de diversidad genética (Wang et al., 2003), (v) identificar factores que juegan
papel importante en la distribución geográfica de la diversidad genética (Wang et al.,
2004), (vi) Identificar implicaciones de la estructura genética de una población en
prácticas de restauración (Sheng et al., 2005), (vii) identificar relaciones entre distancias
genéticas y espaciales y (viii) proponer hipótesis acerca del efecto de aislamiento por
distancia como función de restricción de flujo vía dispersión de semilla (Wolf et al., 2004).
La técnica RAM ha sido evaluada en Physalis peruviana (Bonilla et al., 2008), Rubus spp
(Morillo et al., 2005), Psidium guajava (Sanabria et al., 2006), Heliconia spp. (Arcos et al.
2004), Beauveria bassiana (Marmolejo et al., 2008) y Bos taurus raza Hartón del Valle
(Piedrahita et al., 2007). En uchuva, Bonilla et al., (2008) hallaron diversidad genética alta
y accesiones de fruto rojo genéticamente diferenciados y una región geográfica con
variabilidad alta. Muñoz et al., (2008) diferenciaron las especies R. glaucus, R. robustus y
R. urticifolius, detectando duplicados y variabilidad genética alta en R. glaucus. Los
cebadores RAM fueron altamente polimórficos en guayaba y encontraron variabilidad alta
en el valle geográfico del Rio Cauca-Colombia (Sanabria et al., 2006). En el orden
Zingiberales, Arcos et al., (2004) diferenciaron familias, algunos subgéneros y variantes
intra/entre especie.
Diversidad genética y estructura de poblaciones naturales de Lippia spp. La
estructura genética de poblaciones naturales del género Lippia ha sido escasamente
estudiada (Montanari et al., 2004; Viccini et al., 2004-2006; Dias et al., 2005-2006;
Suárez et al., 2007-2008; Yamamoto et al., 2008; Martínez et al., 2008). Análisis previos
utilizando marcadores RAPD fueron efectivos para comprender la diversidad genética de
especies de Lippia spp. y contribuyeron a entender su adaptación al ambiente,
conservación e implicaciones taxonómicas (Viccini et al., 2004; Pereira et al., 2006).
Viccini et al., (2004), evaluaron el grado de diversidad genética en nueve especies de
Lippia (L. corymbosa, L. diamantinensis, L. filifolia, L. florida, L. hermannioides, L.
lupulina, L. rotundifolia, L. rosella y L. sidoides) del sudoeste de Brasil. La distancia
genética interespecífica promedio fue similar para todas las especies y más alta que las
distancias intraespecíficas. Las especies con presencia limitada, mostraron diversidad
interespecífica más baja. El dendograma por el método UPGMA mostró grupos mayores
con una clara diferenciación entre especies (Viccini et al., 2004). En 2012, Santos et al.,
aislaron y caracterizaron ocho loci de microsatélites (SSR) polimórficos en dos
poblaciones brasileras de Lippia alba, los cuales mostraron potencial informativo de
moderado alto para estudios genéticos. Siete de ocho secuencias SSR revelaron
múltiples bandas típicas de especies poliploides sugiriendo genomas tetraploides en L.
Capítulo 2
45
alba. El índice de diversidad de Shannon (H) fueron 2.013 y 1.989, respectivamente. La
heterocigocidad esperada (He) tuvo rango entre 0.407 y 0.718, con media de 0.564 para
la población A; y de 0.234 a 0.704 con media 0.503 en la población B.
Suárez et al. (2007) llevaron a cabo un estudio piloto en Lippia origanoides, L. alba, y L.
citriodora para observar polimorfismos intra e inter-específicos en cuatro regiones del
cpDNA, de ITS del ADN ribosomal y 50 loci ISSR. Tres de cuatro regiones del cpDNA
(petA-psbE, tnrL-trnF and trnL intron) y la región ITS mostraron mayormente variación
interespecífica. Diez y siete regiones ISSR de 50 loci analizados mostraron
polimorfismos, inter e intraespecíficos. La divergencia de secuencias entre pares de
especies en la región del plastidio, mostró rangos de 0.4% a 1%, mientras que la
divergencia en secuencias de regiones ITS fue cerca del 5%. Los resultados obtenidos
sugieren que las regiones ITS, tal como han sido reportadas para otros géneros de
plantas, puede convertirse en una región de elección para estudios de genética de
poblaciones en este tipo de especies. Los valores de diversidad genética descrita por
Suárez et al. (2008) en Lippia origanoides (plantas alógama Verbenaceae relacionada)
fueron: P=86.21%, para porcentaje de loci polimórficos; I=0.453, para índice de
diversidad de Shannon; y HB=0.484 para Heterocigocidad panmitica promedio.
2.1.2 Metodología
Material vegetal. En este trabajo fueron analizados 59 accesiones de Lippia alba
colectadas en diferentes localidades de dos zonas agroecológicas: Región I (Sumapaz
4.3124166N, -74.493694W) y Región II (Chicamocha 6.5683333N, -73.1400277W). Cada
accesión representa una planta individual propagada por estaca. Estas accesiones
forman parte de la colección in vivo-ex situ del Banco de germoplasma de la Universidad
Nacional de Colombia-Sede Palmira. La lista de accesiones con sus respectivos datos de
colecta se muestra en el Anexo C.
Protocolo para extracción de ADN. Se utilizó el protocolo A-2X desarrollado por Vega
& Chacón (2011). El protocolo de extracción de ADN para especies aromáticas llamado
A-2X consta del siguiente Buffer y reactivos:
i.
ii.
iii.
iv.
v.
vi.
vii.
Buffer A-2X: 2% CTAB w/v, 1.5 M NaCl, 20mM EDTA, 100 mM Tris-HCl pH:8 y
1% β-mercapto (adicionado justo antes de usar)
Cloroformo: Isomil alcohol (24:1) v/v
Isopropanol a -20ºC (Idem: 2-propanol)
Gdm+ -solución salina: a) Guanidina Hidrocloride (Gdm HCl) Solución 2M o 4M
Guanidina diluida en agua destilada o b) Guanidina Tiocianato (Gdm SCN)
solución: 2M o 4M
Etanol al 90% y 70%
Buffer TE bajo en sales (10 mM Tris + 1 mM EDTA) pH≥8.0, como solubilizanteantidegradante del ADN, y
RNasa en TE buffer a [1µg/µL].
46
Título de la tesis o trabajo de investigación
Pasos:
1. Se maceraron 100-200 mg de tejido en nitrógeno líquido en un mortero
esterilizado; y se creó un banco de tejido a -20ºC y -40ºC, depositado en el
Laboratorio de Genética de la Universidad Nacional-Palmira.
En cámara de flujo
2. Inmediatamente se transfirió--el tejido macerado en polvo—a un (tubo) Eppendorf
(con capacidad de 2 mL) conteniendo 800 µL de Buffer A-2X,
3. Se homogenizó la muestra suavemente por inversión y se incubó por 30’ a 65ºC.
Agitándose cada cinco minutos (5’),
4. Se adicionó 800µL de Cloroformo: Alcohol Isoamílico (24:1). Mezclando la
muestra vigorosamente; usando Vortex hasta obtener una emulsión. Se centrifugó
a 14400 xg (gravedades) por 10 minutos (10’),
5. Se transfirió el sobrenadante a un tubo Eppendorf de 2mL. Se adicionó 200 µL
de Isopropanol a -20ºC y se mezcló suavemente por inversión.
Fuera de la cámara de flujo
6. Se adicionó 1 µL de Gdm+. Mezclando rápidamente por inversión durante cinco
minutos (5’)
7. Se transfirió todo el contenido (mínimo 700 µL) de la muestra a una columna de
Silica (EconoSpinTM All-in-1 Min Spin Columns Epoch BioLabs) y se centrifugó a
10.000 xg (gravedades) durante cinco minutos (5’). Descartando el centrifugado
contenido en la base-recipiente de la columna. Repitiendo este paso hasta que
drenó completamente,
8. Se lavó la membrana con 500 µL de Etanol al 90% y se centrifugó a 10.000 xg
(gravedades) durante cinco minutos (5’). Descartando el (liquido) centrifugado del
tubo colector. Repitiendo la acción usando Etanol del 70%.
9. Se centrifugó la columna a 10.0000 xg durante 10 minutos o hasta que la
membrana quedó totalmente seca,
10. Se adicionó 100 µL de TE Buffer pH≥8.0 precalentado a 65ºC por cinco minutos,
directamente en el centro de la membrana. Se incubó a 65ºC por cinco minutos y
se centrifugó a 10.000 xg por 1 minuto para colectar el fluido en el tubo colector,
con el ADN,
11. Se repitió el paso 10, pero el tiempo de incubación se redujo a 3 minutos,
12. (Opcional) se adicionan 2 µL de RNasa e incube a 37ºC por 15 minutos.
LA CALIDAD DEL ADN SE REVISÓ EN UN GEL DE AGAROSA AL 0.8%
Amplificación y evaluación de marcadores RAM. El ADN total fue extraído a partir
de100-200 mg. de tejido foliar fresco de cada accesión por el método A-2X (Vega y
Chacón, 2011). La calidad y la concentración del ADN fue evaluada en un gel de agarosa
0.8%. La integridad del ADN fue confirmado mediante electroforesis en gel de
poliacrilamida de 0.8% de agarosa en buffer TBE.
Técnica RAM. Se realizó la PCR para los cebadores ACA-AG-CA-CCA-TG-CT-CGA-GT.
Los productos PCR fueron visualizados en geles de poliacrilamida (37:1) al 7% a 160
voltios por una hora y se hizo tinción con Bromuro de Etidio. La mezcla para la PCR fue:
ADN 10.0 ng/µL + Buffer TAQ 10X 2.50 µL +MgCl2 2.50 µL 25 mM+ DNTP [4.00 µL] +
Capítulo 2
47
Cebador [1.00 µL] + Agua 11.80 µL + TAQ Polimerasa 0.15
termociclador se describen a continuación:
Tabla 2-1:
µL. Los pasos del
Pasos del termociclador
Paso
Tº
t (tiempo)
Etapas
1
95
5 minutos
Desnaturalización
2
95
30 segundos
Desnaturalización
3
TH*
45 segundos
Hibridación
4
72
2 minutos
Extensión
5
37
6
72
7 minutos
7
16
30 minutos
Primer
veces desde el paso 2
Extensión
TH*
ACA-AG-CA
50
CCA-TG-CT
55
GT-CCA
58
TH*= Tiempo de Hibridación
Análisis de datos. Se caracterizaron las bandas por presencia/ausencia en cada
accesión (introducción) y los datos fueron consignados en una matriz binaria de 1/0. Las
frecuencias alélicas y genotípicas (observadas y esperadas); y los valores de Diversidad
Genética por locus (hj=1-p2-q2) y promedio (Hi=Σhj/n) de Nei [D] (1987), fueron
calculadas usando el Programa TFPGA v1.3 (bajado de la red el 15-04-2013). La
discriminación potencial de cada cebador (o grado de variabilidad genética en la
población) fue expresado por el Coeficiente de Simpson (H=Σ(1- Σpi2)/n), donde pi es la
frecuencia del iesimo alelo y n corresponde al número de loci detectados por cada cebador
(Manica-Cattani et al., 2009). Para todos los pares de genotipos, los valores de
similaridad genética fueron calculados usando el coeficiente de Dice-Nei (Kosman y
Leonard, 2005), el cual excluye 0-0 como indicador de similaridad (Manica-Cattani et al.,
2009) y de Ward (1963). Usando matrices de similaridad genética, un dendograma fue
construido acorde a peso de los pares de grupos usando UPGMA (Manica-Cattani et al.,
2009) y el Programa Ward-MLM (Franco et al., 2005; Silva et al., 2010). La relación entre
datos químicos y genéticos fue determinado mediante un AMOVA y análisis canónico
(Manica-Cattani et al., 2009). Los datos fueron analizados usando los programas
SAS/STATv.9.2 y GenAlEx (En: http://biology. anu.edu.au/GenAlEx/ Download.html). El
programa propuesto por Franco et al., (1998), utiliza la estrategia en dos pasos WARDMLM para agrupamiento de observaciones utilizando variables continuas y categóricas
simultáneamente. El programa está dividido en tres bloques de acuerdo a los tres pasos
sugeridos para la clasificación: WARD-MLM1.SAS (calcula la matriz de similaridades y
distancias de Gower); WARD-MLM2.SAS (calcula y grafica el logaritmo de máxima
verosimilitud para número probable de grupos) y WARD-MLM3.SAS (define el número de
grupos).
48
Título de la tesis o trabajo de investigación
2.1.3 Resultados
Diversidad y variabilidad genética entre accesiones de Lippia alba
La Heterocigocidad promedio esperada-He (Heterocigocidad media o Diversidad genética
de Nei) para la muestra de trabajo tuvo un valor medio (0.0 ≤ He=0.2467 ≤ 0.5). Los
valores de Diversidad Molecular-DM-estuvieron en el rango de 0.1219 a 0.3425 para
siete marcadores RAM (Tabla 2-2). Con base en la frecuencia de variantes (Numero
efectivo de alelos [Ae] y Heterocigocidad esperada [He], la diversidad genética por locus
(hj=1-p2-q2) tuvo valores máximos (cercanos a 0.5) en los cebadores ACA-AG-CGA-CCA.
Las frecuencias alélicas y genotípicas (observada y esperada) por cebador y por locus
(dado cebador), se reportan en el Anexo D y el número efectivo de alelos (o número de
alelos esperados en cada locus) se muestra en la Tabla 2-2.
Con base en el número de variantes (polimorfismo [Pj], proporción de loci polimórficos
[P], abundancia de variantes alélicas [A] y promedio de alelos por locus [n]), los siete
cebadores generaron 13.43±4.43 loci, con una proporción de loci polimórficos P=91.48%
dada su frecuencia alélica Pj=q≤0.95/0.99 (Tabla 2-2). Amplificaron siete (de ocho)
cebadores generando bandas consistentes y reproducibles en 94 loci, con 45±9.52
bandas por individuo (Anexo D). Se detectaron tres alelos dominantes únicos y cinco
(alelos dominantes) raros con frecuencia p≤0.05 (Tabla 2-2).
Tabla 2-2:
Parámetros (número efectivo de alelos-Ae y Heterocigocidad EsperadaHe) y variables (Polimorfismos-Pj y alelos por locus) para medir la diversidad genética de
Lippia alba con base en frecuencia y número de variantes.
Número
Cebador
Ae
He
1
2
3
4
5
6
7
ACA
AG
CA
CGA
CCA
TG
GT
0,1960
0,1427
0,0638
1,3930
6,6881
4,7730
1,9180
2,1678
0,3339
0,3425
0,1219
0,1718
0,3317
0,2483
0,1768
0,2467
No.
Loci
19
14
19
9
8
11
14
94
Polimórficos
19
14
13
8
8
10
14
86
Loci
Monomórficos
0
0
6
1
0
1
0
8
n
26,420
37,070
49,310
11,890
23,620
22,450
11,000
25,9657
El grado de variabilidad genética fue alta (H=0.7356). La discriminación potencial de cada
cebador expresado por el Coeficiente de Simpson [H=Σ(1- Σpi2)/n] mostró valores entre
0.2991 en el cebador AG, hasta 0.9632 en el cebador TG (Tabla 2-3).
Capítulo 2
49
Tabla 2-3:
genética.
Valores de Coeficiente de Simpson determinantes de la variabilidad
Cebador
1
2
3
4
5
6
7
Secuencia
ACA
AG
CA
CGA
CCA
TG
GT
No. Loci
19
14
19
9
8
11
14
94
Loci
Polimórficos Monomórficos
19
0
14
0
13
6
8
1
8
0
10
1
14
0
86
8
H
Índice de Simpson
0,7656
0,5634
0,2991
0,9270
0,8001
0,9632
0,8136
0,7331
No obstante, para especies como L. alba y L. origanoides, estos valores (H=0.484
usando ITS) son comunes en Colombia (Suárez et al., 2007-2008). De otro lado, ManicaCattani et al. (2009) hallaron variabilidad genética alta entre accesiones de L. alba
colectadas al sur de Brasil, con valores promedio de 0.565 para ISSR y 0.625 para
RAPD. En la muestra de trabajo las frecuencias alélicas mostraron todos los valores
posibles dentro del rango, un factor que puede considerarse característico de los RAM;
diferente a las frecuencias obtenidas por Manica-Cattani et al. (2009), con valores
extremos altos (>0.90) y bajos (<0.10) en accesiones de Brasil con el mismo tipo
(dominante) de marcador (RAPD e ISSR). De hecho, el poder discriminante de los RAM
ha sido valorada en recursos fitogenéticos locales incluyendo heliconias (Arcos et al.,
2004), árboles nativos de Psidium guajava (Muñoz et al., 2008; Sanabria et al., 2006),
Rubus spp (Muñoz et al., 2008), uchuva (Bonilla et al., 2008), y más recientemente en la
caracterización del Banco Colombiano de germoplasma de maíz (En: R.D. Rojas-Pantoja
‘Evaluación de la diversidad genética en las razas criollas e indígenas de maíz en
Colombia mediante marcadores moleculares tipo RAM’ Trabajo de grado UN-Palmira).
Variabilidad y diversidad genética dentro de (intraespecífica) accesión
La Diversidad genética de Nei [D=0.2722], dentro de accesión fue también de valor
medio sin ser significativamente mayor que el valor de DG entre accesiones [D=0.2467].
Los valores respectivos de Diversidad Molecular-DM-estuvieron en el rango de 0.000 a
0.4776 para siete marcadores RAM (Tabla 2-4). Siete cebadores amplificaron generando
bandas robustas y visibles (Tabla 2-4). Con excepción del cebador GT (100%
monomórfico; DG=0.0000) los cebadores mostraron valores de DG (dentro de accesión)
más altos que los mostrados por DG entre accesiones, es decir, los indicadores de
diversidad y variabilidad genética fueron mayores dentro de accesiones, que entre
accesiones.
50
Tabla 2-4:
Título de la tesis o trabajo de investigación
Valores de diversidad genética intraespecífica.
Primer
1
2
3
4
5
6
7
Secuencia
ACA
AG
CA
CGA
CCA
TG
GT
No. loci
19
14
19
9
8
11
14
94
Loci
Polimórficos Monomórficos
19
0
14
0
13
6
8
1
8
0
10
1
14
0
86
8
Hi
0.3603
0.4298
0.4776
0.1765
0.3252
0.1363
0
0.2722
El valor de Diversidad Molecular-DM-para la muestra fue medio, en la escala de 0 a 0.5
de Diversidad Genética-DG- de Nei (1978) para marcadores RAM.
Discusión principal. El valor de Diversidad Molecular-DM-para la muestra fue
intermedio (0,24), en la escala de 0 a 0.5 de DG de Nei (1978) para marcadores RAM.
Valores DM≤0.332 se consideran medio, como el resultado obtenido. No obstante, para
especies naturalizadas como L. alba y L. origanoides, valores bajos (H=0.484) son
comunes en Colombia (Suárez et al., 2007-2008). En Lippia origanoides, una especies
relacionada del mismo género, Vega y Chacón en 2008 identificaron cuatro poblaciones
de (en la Región Andina de Colombia) estructuradas genéticamente (Ht = 0.32; I = 0.48).
En 2013, Vega et al. obtuvieron valores relativamente altos de DG (0,35≤Hs≤0,37) para
L. origanoides en dos regiones colombianas (Magdalena medio y Chicamocha) con una
diferenciación genética baja, la cual varió de 0.07 a 0.17 entre poblaciones. En
poblaciones brasileras de L. alba, Manica-Cattani et al. (2009) hallaron variabilidad
genética alta entre accesiones de L. alba colectadas al sur de Brasil, con valores
promedio de 0.565 para ISSR y 0.625 para RAPD. Santos et al., (2012) obtuvieron
valores de heterocigocidad esperada (He) entre 0.407 y 0.718, con media de 0.564 para
una población A; y de 0.234 a 0.704 con media 0.503 en una población B usando SSR
(Santos et al., 2012).
Los grados de variabilidad genética en la población (H) se presentan en la Tabla 2-3. En
la muestra de trabajo las frecuencias alélicas mostraron todos los valores posibles dentro
del rango, un factor que puede considerarse característico de los RAM, similar a los
obtenidos por Vega y Chacón (2008) y Vega et al. (2013) en la región del ChicamochaColombia, usando AFLP; diferente a las frecuencias obtenidas por Manica-Cattani et al.
(2009), con valores extremos altos (>0.90) y bajos (<0.10) en accesiones de Brasil con el
mismo tipo (dominante) de marcador (RAPD e ISSR). De hecho, el poder discriminante
de los RAM ha sido valorada en recursos fitogenéticos locales incluyendo heliconias
(Arcos et al., 2004), árboles nativos de Psidium guajava (Muñoz et al., 2008; Sanabria et
al., 2006), Rubus spp (Muñoz et. al., (2008), uchuva (Bonilla et al., 2008), y más
recientemente en la caracterización del Banco Colombiano de Maíz (J. E Muñoz, cita
textual).
Los dos grupos de individuos estudiados revelan un proceso de estructuración genética
de sus poblaciones. El AMOVA (Tabla 2-8) y el estadístico FST=0,054 (p<0,001)
Capítulo 2
51
detectaron que las poblaciones estudiadas aquí se están estructurando genéticamente.
Análisis de Coordenadas Principales-ACP, AMOVA y análisis Bayesiano conducidos por
Suárez et al. (2008) revelaron un bajo nivel de diferenciación genética entre dos
localidades dentro del Cañón del Chicamocha-Colombia, sugiriendo una sola población
de Lippia origanoides. Los niveles de DG en esta población, expresado con base en
porcentaje de loci polimórficos (P=86.21%), el índice de diversidad de Shannon (I=0.453)
y la Heterocigocidad panmitica promedia (HB=0.484), es comparable con los niveles de
DG de otras Verbenáceas alógamas relacionadas (Suárez et al., 2008).
La muestra inicial de colectas en esta prueba fue 83 individuos; esta se redujo al hallare
únicamente 59 individuos con características diferenciables. Cuatro factores ‘pueden’
estar afectando la DG de la muestra de L. alba en Colombia: a) la especie muestra
anormalidades postmeióticas-afectando la formación de granos de polen viables y es
determinante de viabilidad y germinación baja de las semillas (Muñoz et al., 2006),
presentando
problemas
importantes
en
su
reproducción
sexual
(En:
http://www.bdigital.unal.edu.co/ 4959/1/memoria10a%C3%B1os UNmaestriarfn palmira.
2011.pdf.), b) su forma particular de diseminación, en el que se presenta un aspecto
socio-cultural de dispersión por adultos mayores de grupos culturales y étnicos
(Antropocoria) originado por sus propiedades medicinales (etnobotánica), c) su
estenopropagación (o propagación vegetativa fácil y rápida) por esquejes y estacas de
todo tipo de madera y d) régimen extractivo alto sin reemplazo. Más, dispersión
restringida de semilla y/o distancias cortas de movimiento de polen, propuestos por
Suárez et al. (2008) para L. origanoides, una Verbenaceae con características similares a
L. alba.
Cebadores (primers) RAM. Ocho cebadores RAM fueron evaluados en el ADN total de
cada accesión. De ocho, siete produjeron patrones de bandas robustas y reproducibles
de intensidad mediana y alta (Figuras 2-1/2). Los cebadores seleccionados--y usados
para amplificar el ADN de todas las accesiones--rindieron un total de 2655 fragmentos. El
número y tipo de cebador usado en la prueba fue criterio del laboratorio de Biotecnología
de la Universidad Nacional de Colombia-Sede Palmira, con base en su efectividad en la
caracterización de: a) recursos fitogenéticos locales [Physalis peruviana (Muñoz et al.,
2008; Bonilla et al., 2008), Rubus spp, Psidium guajava (Muñoz et al., 2008; Sanabria et
al., 2006) y Heliconia spp. (Arcos et al. 2004; Muñoz et al., 2008)]; b) fitopatógenos
[Beauveria bassiana (Marmolejo et al., 2008; Muñoz et al., 2008)] y c) recursos
zoogenéticos autóctonos [Bos taurus raza Hartón del Valle (Piedrahita et al., 2007;
Muñoz et al., 2008)].
Figura 2-1: Patrón de bandas generadas por el cebador ACA_
Figura 2-2:
Patrón de bandas generadas por el cebador CA_
52
Título de la tesis o trabajo de investigación
Los microsatélies son (marcadores) versátiles en aplicaciones ecológicas (Selkoe &
Toonen, 2006). Pueden ser usados para estimar parámetros de interés ecológico como
tasa de migración, tamaño de población, cuello de botella, parentesco. Son de particular
interés en ecología debido a que permite discernimientos a escala muy fina, sobre
interrogantes ecológicos, siendo además neutros y se ajustan a los principios de herencia
mendeliana. Ventajas: a) fácil preparación de la muestra, generan mucha información
(altas tasas de mutación resultando en altos contenidos de diversidad alélica).
A pesar de sus bondades tienen varios retos y supuestos que complican su análisis
(Selkoe & Toonen, 2006). Los mecanismos de mutación no son claros, presentan alelos
idénticos en secuencia con diferente linaje (descendencia) (Homeoplásia), y problemas
con la amplificación (altas tasas de mutación con múltiples versiones (alelos) y error en
lectura de alelos. Ocurren muchos pasos entre la extracción de ADN y la conformación
de la base de datos. Un error de genotipado de 1% puede llevar a un sustancial número
de genotipos multilocus incorrecto en una serie grande de datos. Las fuentes de error
incluye: amplificación pobre, error interpretativo (banda fantasma), interpretación
incorrecta del patrón de bandas, contaminación o error en la entrada de datos (Selkoe &
Toonen, 2006).
Capítulo 2
53
Bandas únicas y alelos raros. Los cebadores (RAM) detectaron tres alelos dominantes
únicos y cinco-alelos dominantes-raros con frecuencia p≤0.05 (Tabla 2-5). Los alelos
únicos se ubicaron en los loci CA_17/18/19 de la accesión 341 con frecuencia p=0.0085.
Los alelos raros (p≤0.05) se mostraron en el locus ACA_14, accesiones 310-312-320322; locus ACA_17, accesiones 349-353-357; locus ACA-17, accesiones 174-187-189201-357; locus CGA_8, accesiones 174-187-322; locus CGA_9, accesiones 174-187;
locus CCA_3, accesiones 344/5-‘Daza’. El cálculo de frecuencias genotípicas/alélicas
reportó diez alelos únicos con frecuencia q≤0.1302 y cinco alelos raros recesivos. Los
alelos únicos se localizaron todos en el cebador CA, locus 1 a 10 de la accesión 341.
Los-alelos-raros fueron estimados en el cebador AG, locus 6 y 10 de las accesiones 340354-366-370-‘Daza’; locus 7 de las accesiones 340-354-366-‘Daza’; Locus 9 de las
accesiones 340-366-370-‘Daza’; locus 12 de las accesiones 366-367-377-378-‘Daza’; con
frecuencia q≤0.30.
La presencia de alelos raros en un genotipo sugiere el comportamiento de efecto dosis: a
mayor presencia de alelos raros, menor % de energía germinativa en su progenie Murillo,
2000). La heterocigocidad es uno de los mecanismos más eficientes de una población
para lograr mantener alelos raros, evitando su extinción (Murillo, 2000). Individuos con
He alta tienden a producir progenies menos viables (fitness). Valores extremos de He
presentan un menor nivel de adaptabilidad (Murillo, 2000). Alta VG y presencia de alelos
raros, indican la necesidad de grandes colecciones de germoplasma para representar la
DG total de una especie (Igic et al., 2006). En programas de conservación y
mejoramiento se debe utilizar el grado de heterocigocidad como un criterio para
ubicación espacial de individuos (Murillo, 2000). Plantas con alelos raros tienen más
probabilidad de aparearse que aquellas con alelos comunes (Igic, 2006). No obstante, los
alelos raros poseen una influencia mayor en las estimaciones de riqueza alélica, que en
la de heterocigocidad.
Tabla 2-5:
Alelos únicos y alelos raros hallados en la muestra de 59 accesiones de L.
alba colectadas en dos zonas (Chicamocha y Sumapaz-Colombia).
Alelos únicos hallados con frecuencias p≤0,0085
Locus
CA_17
CA_18
CA_19
Frecuencia
alélica
Frecuencias genotípicas
Genotipos
AA
Freq gen (esp)
p2
Aa
Aa
2pq
q2
1
1
58
59
Freq gen (obs)
0,0169
0,9831
Genotipos
AA
Aa
Aa
TOTAL
Freq gen (esp)
p2
2pq
q2
1
59
1
58
Freq gen (obs)
0,0169
0,9831
Genotipos
AA
Aa
Aa
Freq gen (esp)
p2
2pq
q2
1
59
Individuos
1
58
Freq gen (obs)
0,0169
0,9831
Accesión
TOTAL
Individuos
Individuos
hj= (1-p2q2)
P
q
0,0085
0,9915
P
q
0,0085
0,9915
P
q
0,0085
0,9915
0,0169
0,0169
TOTAL
0,0169
341
54
Título de la tesis o trabajo de investigación
Alelos raros con frecuencia p ≤ 0.05
ACA_14
ACA_17
Genotipos
AA
Aa
Aa
TOTAL
Freq gen (esp)
p2
2pq
q2
1
59
Individuos
4
55
Freq gen (obs)
0,0678
0,9322
Genotipos
AA
Aa
Aa
Freq gen (esp)
p2
2pq
q2
1
56
59
Individuos
Freq gen (obs)
ACA_19
CGA_9
CCA_3
0,0508
Genotipos
AA
Freq gen (esp)
p2
Individuos
Freq gen (obs)
CGA_8
3
AA
Freq gen (esp)
p2
0,9492
Aa
2pq
q2
1
54
59
0,0847
Genotipos
Aa
2pq
q2
1
3
56
59
Freq gen (obs)
0,0508
0,9492
Genotipos
AA
Aa
Aa
TOTAL
Freq gen (esp)
p2
2pq
q2
1
59
2
57
Freq gen (obs)
0,0339
0,9661
0,9655
P
q
0,0258
0,9742
P
q
0,0433
0,9567
P
q
0,0258
0,9742
P
q
0,0171
0,9829
P
q
0,0258
0,9742
310-312- 320-322
0,0502
349-353-357
0,0829
174-187-189-201-357
0,0502
174-187-322
0,0336
174-187
0,0502
344-345-Daza
TOTAL
Individuos
Individuos
0,0345
0,0666
TOTAL
0,9153
Aa
q
TOTAL
Aa
5
P
Genotipos
AA
Aa
Aa
TOTAL
Freq gen (esp)
p2
2pq
q2
1
Individuos
3
56
59
Freq gen (obs)
0,0508
0,9492
1
Diversidad genética-Dg. El valor de Dg fue 0.2467, con un significado de valor medio en
la escala de 0 a 5. Para su estimación, las dos colectas (Chicamocha y Sumapaz) fueron
combinadas para su análisis. Se calculó Diversidad genética-Dg-por locus (hj) y por
accesión (H) más los estadígrafos específicos: tamaño promedio de muestra (n), Numero
efectivo de alelos (Ae) Proporción de loci polimórficos (P), Heterocigocidad esperada
(He) y Heterocigocidad promedia por locus (H). La Tabla 2-6 muestra los estadígrafos
por locus para siete cebadores.
Ochenta y seis de 94 loci fueron polimórficos. El número de loci por cebador fue
ACA=CA>AG=GT>TG>CGA>CCA y la proporción de loci polimórficos fue alta (0.907). El
Anexo 2 y las Figuras 8 a 15 detalla el número de loci y valores de DG, expresado como
hj (cuando esta se estima) para cada cebador. Los promedios para los estadígrafos
específicos fueron: nx=25,97; Aex=2,4062; Px=0,9260; Hox=0,4008; Hex=0,3595; y la
Dgx=0,2467. Adicionalmente, se estimaron cuatro estadígrafos específicos con base en
número de alelos por locus dentro de accesión; los valores se reportan en las ‘últimas
cuatro columnas de la Tabla 2-6.
Capítulo 2
55
Tabla 2-6:
Estadígrafos para siete cebadores RAM para 59 colectas de L. alba.
No. loci
Media
Error típico
Mediana
Moda
Desviación estándar
Varianza de la muestra
Curtosis
Coeficiente de asimetría
Rango
Mínimo
Máximo
Suma
Cuenta
Mayor (1)
Menor(1)
Nivel de confianza (95,0%)
13,43
1,67
14,00
19,00
4,43
19,62
-1,50
0,23
11,00
8,00
19,00
94,00
7,00
19,00
8,00
4,10
Tabla 2-7:
Número de loci, polimorfismos y valores de Diversidad Genética-DG.
Estadígrafos específicos (n=número promedio de alelos/locus, Ae=número efectivo de
alelos, P=proporción de loci polimórficos, Ho=Heterocigocidad observada,
He=Heterocigocidad esperada). Y, estadígrafos por accesión (P=Proporción de
accesiones multilocus, H=Heterocigocidad promedio por accesión), más número de
accesiones multiloci y mono locus.
DG (Nei, 1987)
LOCI
Primer
Sec.
1
2
ENTRE
DENTRO
ACCESIONES
Poli
Mono
DG
N
Ae
P
Ho
He
Hi
P
H
multi
Mono
ACA
No.
loci
19
19
0
0,3339
26,42
3,7245
1,0000
0,4478
0,7315
0,3603
0,9831
0,7656
58
1
AG
14
14
0
0,3425
37,07
0,1427
1,0000
0,6283
0,4994
0,4298
0,9661
0,5634
57
2
3
CA
19
13
6
0,1219
49,32
0.0638
0,6842
0,8359
0,1755
0,4776
1,0000
0,2991
59
0
4
CGA
9
8
1
0,1718
11,89
1,3930
0,8889
0,2015
0,9202
0,1765
0,7966
0,9270
47
12
5
CCA
8
8
0
0,3317
23,63
6,6881
1,0000
0,1250
0,9813
0,3252
0,9322
0,8001
55
4
6
TG
11
10
1
0,2483
22,45
0,4339
0,9091
0,3806
0,1363
1,0000
0,9632
59
0
7
GT
14
14
0
0,1768
11,00
2,0549
1,0000
0,1864
1,3048
0,5134
0,0000
0,0000
0,8136
0
59
56
Título de la tesis o trabajo de investigación
Figura 2-3: Diversidad genética (hj=1-p2-q2) para 19 loci del cebador (RAM) ACA_.
Este primer reveló 19 loci y 502 bandas en 59 individuos evaluados. Las frecuencias
alélicas estuvieron en el rango de 0.0502 a 0.5, la DG media (expresada como hj) tuvo
valor bajo (0,3339) ubicándose en el límite superior del intervalo bajo (<=0.33), con
mínima=0.0502 en el locus ACA_17 y máxima=0.5 en el locus ACA_8.
0,6000
0,5000
hj=1-p2-q2
0,4000
0,3000
Series1
0,2000
0,1000
0,0000
1 5 9 13172125293337414549535761656973
Ceabdor ACA_
Figura 2-4: Diversidad genética (hj=1-p2-q2) individual para 14 loci del cebador (RAM)
AG_. Este primer reveló 14 loci y 519 bandas en 59 individuos evaluados. Las
frecuencias alélicas estuvieron en el rango de 0.1462 a 0.5, la DG media tuvo valor bajo
(0,3425) ubicándose en el límite superior del rango bajo (<=0.33), con mínima=0.14662
en el locus AG_14 y máxima=0.50 en el locus AG_5.
Cebador GA_
0,6000
hj=(1-p2-q2)
0,5000
0,4000
0,3000
0,2000
0,1000
0,0000
1 4 7 10 13 16 19 22 25 28 31 34 37 40 43 46 49 52 55
Cebador
Capítulo 2
57
Figura 2-5: Diversidad genética (hj=1-p2-q2) individual para 19 loci del cebador (RAM)
CA_. Este primer reveló 19 loci y 937 bandas en 59 individuos evaluados. Las
frecuencias alélicas estuvieron en el rango de 0.000 a 0.2265, la DG media -expresada
como hj- tuvo el valor más bajo (0,1219) de la clase baja (<=0.33), con mínima=0.0000
en el locus CA_11-16 y máxima=0.2265 en el locus CA_1-10.
Cebador CA_
0,2500
hj=(1-p2-q2)
0,2000
0,1500
0,1000
0,0500
0,0000
1 5 9 13 17 21 25 29 33 37 41 45 49 53 57 61 65 69 73
Cebador CA_
Figura 2-6: Diversidad genética (hj=1-p2-q2) individual para 9 loci del cebador (RAM)
CGA_. Este primer reveló 9 loci y 107 bandas en 59 individuos evaluados. Las
frecuencias alélicas estuvieron en el rango de 0.000 a 0.4945, la DG media-expresada
como hj-tuvo valor bajo (0,1718) en la clase baja (<=0.33), con mínima=0.0000 en el
locus CGA_6 y máxima=0.4945 en el locus CA_7.
Cebador CGA
0,6000
HJ=(1-p2-q2)
0,5000
0,4000
0,3000
0,2000
0,1000
0,0000
1 3 5 7 9 11 13 15 17 19 21 23 25 27 29 31 33 35
Cebador CGA_
58
Título de la tesis o trabajo de investigación
Figura 2-7: Diversidad genética (hj=1-p2-q2) individual para 8 loci del cebador (RAM)
CCA_. Este primer reveló 8 loci y 189 bandas en 59 individuos evaluados. Las
frecuencias alélicas estuvieron en el rango de 0.0502 a 0.4973, la diversidad genética
media -expresada como hj- tuvo el valor bajo (0,3317) ubicándose en el límite superior
del intervalo bajo (<=0.33), con mínima=0.0502 en el locus CCA_3 y máxima=0.4973 en
el locus CcA_5.
Cebador CCA_
0,6000
hj=(1-p2-q2)
0,5000
0,4000
0,3000
0,2000
0,1000
0,0000
1
3
5
7
9 11 13 15 17 19 21 23 25 27 29 31
Cebador CCA_
Figura 2-8: Diversidad genética (hj=1-p2-q2) individual para 11 loci del cebador (RAM)
TG_. Este primer reveló 11 loci y 247 bandas en 59 individuos evaluados. Las
frecuencias alélicas estuvieron en el rango de 0.0 a 0.4755, la diversidad genética media
-expresada como hj- tuvo valor bajo (0,2483) en el intervalo bajo (<=0.33), con
mínima=0.0 en el locus TG_8 y máxima=0.4755 en el locus TG_7.
Cebador TG
0,5000
hj=(1-p2-q2)
0,4000
0,3000
0,2000
0,1000
0,0000
1 3 5 7 9 11 13 15 17 19 21 23 25 27 29 31 33 35 37 39 41 43
Cebadores TG_1-11
Capítulo 2
59
Figura 2-9: Diversidad genética (hj=1-p2-q2) individual para 14 loci del cebador (RAM)
GT_. El cebador reveló 14 loci y 154 bandas en 59 individuos evaluados. La frecuencia
alélica única fue 0,1768, la diversidad genética media-hj-tuvo valor bajo (0,1768)
ubicándose en el rango bajo (<=0.33), con valor único para todos los loci de 0,1768.
hj=(1-p2-q2)
Cebador GT
0,2000
0,1800
0,1600
0,1400
0,1200
0,1000
0,0800
0,0600
0,0400
0,0200
0,0000
1 3 5 7 9 1113151719212325272931333537394143454749515355
Cebadores 1 a 14
Figura 2-10: Diversidad genética promedio general (Hi=0,2467) y por cebador RAM en
59 accesiones de Lippia alba colectadas en dos zonas agroecológicas colombianas.
0,4000
0,3500
hj=(1-p2-q2)
0,3000
0,2500
0,2000
0,1500
0,1000
0,0500
0,0000
ACA
Hi 0,3339
AG
CA
CGA
CCA
TG
GT
Hi
0,3425
0,1219
0,1718
0,3317
0,2483
0,1768
0,2467
60
Título de la tesis o trabajo de investigación
Discusión
Todos los cebadores detectaron diversidad y estructura genética baja en la muestra de
trabajo. Los siete cebadores mostraron valores de hj cercanos a cero, excepto el cebador
AG (hj=0,1466). Cinco de los siete (71,4%) primers tuvieron valores de hj cercanos a 0,5
como valor máximo. El cebador AG obtuvo el rango más alto de hj con min.=0,1466 y
máx.=0,5; y el cebador CA el intervalo más bajo con min.=0,0 y máx.=0,2265. El primer
GT fue neutro con un hj=0,1768. De hecho, los marcadores RAM estuvieron por debajo
de los valores hj obtenidos por Montanari et al., (2004); Viccini et al., (2004-2006); Dias et
al., (2005-2006); Pereira et al., (2006); Yamamoto et al., (2008); y Manica-Cattani et al.
(2009), sugiriendo la presencia de mayor DG (0,62 con ISSR y 0,589 con RAPD) y menor
variabilidad genética-VG en Brasil, usando marcadores (también) dominantes en
poblaciones brasileras de L. alba.
Estructura genética (poblacional)
La Dg dentro de poblaciones (WP) fue significativamente mayor que entre poblaciones
(AP), detectando un proceso común el cual está estructurando (generando una
estructura genética en) las poblaciones estudiadas. La diferenciación poblacional (o
estructural) fue determinada usando un AMOVA y el estadístico FST. La prueba de
diferenciación entre poblaciones usando el AMOVA mostró la menor variación haplotípica
entre poblaciones (Est.Var.AP=0,141) con remanente (95%) distribuida dentro de la
población (Est.Var.WP=2,46). El valor FST fue 0,054 (p<0,001), indicando diferenciación
genética pequeña entre regiones. El mayor aporte a la respuesta obtenida por la prueba
lo generó la fuente de variación entre poblaciones-AP (MS=6,381); no obstante, su
estimativo de varianza fue apenas el 5% de la varianza total estimada (Tabla 2-8).
Tabla 2-8:
Análisis molecular de varianza-AMOVA- y valor de FPT para 58 accesiones
colombianas de L. alba.
Source
Among Pops
Within Pops
Total
Stat
PhiPT
PhiPT max
Phi'PT
df
1
56
57
SS
6,381
137,744
144,125
MS
6,381
2,460
Est. Var.
0,141
2,460
2,601
Proportion
5%
95%
100%
Value
P(rand >= data)
0,054
0,001
0,948
0,057
Los resultados del análisis molecular de varianza con base en la matriz binaria de
distancias para cálculo de PhiPT mostraron los valores porcentuales siguientes:
Capítulo 2
Tabla 2-9:
61
Valores porcentuales por cebador dentro (WI) y entre (AI)
Cebador
ACA
AG
CA
CGA
CCA
TG
GT
Full
Entre(AI)
46%
3%
0%
36%
57%
22%
21%
23%
Dentro (WI)
54%
97%
100%
64%
43%
78%
79%
77%
Estructura jerárquica
Se examinó la estructura jerárquica usando un análisis de conglomerados (UPGMA) y
PCoA de las dos regiones y diferenciación genética entre poblaciones a partir de un
AMOVA (Tabla 7). La matriz de similaridad entre los dos grupos basado en el coeficiente
de Nei&Li se muestra en el Anexos E y F. El clúster (Figura 16) y el PCoA (Figuras 17 y
18) mostraron la existencia de diferentes conglomerados geográficos en cada
subpoblación. El nivel de diferenciación entre las dos poblaciones fue relativamente bajo
pero significativo; igual resultado obtuvo Martínez (2008) en L. origanoides, una especie
relacionada, en la región del Chicamocha-Colombia.
Figura 2-11: Estructura jerárquica de dos poblaciones colombianas de L. alba con base
en polimorfismos generados por siete cebadores RAM.
2. 5
A
v
e
r
a
g
e 2. 0
D
i
s
t
a
n 1. 5
c
e
B
e
t
w 1. 0
e
e
n
C
l
0. 5
u
s
t
e
r
s
0. 0
3 3 3 3 2 2 2 3 3 3 3 3 3 3 3 4 3 3 3 1 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 2 3 3 5 3 3 3 3 3 3 3 3 3 1 1 1 2 3
0 0 0 0 0 1 0 0 4 4 4 5 4 4 5
5 5 5
5 6 6 7 7 6 6 6 6 7 6 7 7 7 5 5 1 1 1 2
2 4
6 5 0 0 1 1 4 4 7 7 8 8 0 4
0 2 1 3 2 7 9 4 2 7 8 0 9 6 6
1 2 8
9 3 9 4 5 2 4 5 0 9 7 8 0 7 3 7 2 3 4 2
0 0
6 4 5 8 0 1 4 5 3 4 7 9 1 1
Na me
of
Ob s e r v a t i o n
or
Cl u s t e r
62
Título de la tesis o trabajo de investigación
Figura 2-12: Región Chicamocha. Análisis de coordenadas principales-PCoA-basado
en polimorfismos RAM y distancia genética de Nei&Li. Tres coordenadas explicaron el
80.49% de la variación hallada por la prueba.
Principal Coordinates (PCoA)
Coord. 2
5 67
21
179 23
22
19
15
1116
20
10
13
14 8 12
4
31 2
Series1
18
Coord. 1
Figura 2-13: Región Sumapaz. Análisis de coordenadas principales-PCoA-basado en
polimorfismos RAM y distancia genética de Nei&Li. Tres coordenadas explicaron el
72,8% de la respuesta obtenida por la prueba.
Principal Coordinates (PCoA)
Coord. 2
1411
16
10 12 5 13
25
9
6
8
7
34
234
15 24 1 32
172133 2931
22
23 26
20 353027
28
Series1
18 19
Coord. 1
Estructura espacial. La estructura espacial a través de poblaciones (combinada), con
valor Omega (Ω=89.096) y de probabilidad [P(rand>=data)] para el correlograma
combinado fue significativo. La relación entre distancias geográficas y las distancias de
Nei usando Mantel fue significativa (p≤0.001), indicando aislamiento por distancia entre
Capítulo 2
63
las subpoblaciones (Figura 2-14/5). Examinando la relación entre distancias geográficas
y distancias genéticas, fue significativa para Sumapaz (p≤0.04) y no significativa para
Chicamocha (p≥0.23); indicando aislamiento por distancia en Sumapaz y lo opuesto, (no
aislamiento por distancia) en Chicamocha. Las Figuras 2-14/5 muestran la estructura
espacial entre (Figura 2-14) y de cada población (Figuras 2-16/7). El correlograma
(Omega=97.136) para la sub-población Sumapaz mostró estructura espacial significativa
[0.001 P(Omega-rand≥Omega-data]. El correlograma (Omega=28.488) para la estructura
espacial de la población Chicamocha no fue significativa [0.098 P(Omega-rand≥Omegadata. El análisis de la estructura espacial global muestra que la subpoblación Sumapaz
está estructurada, mientas que Chicamocha no.
Figura 2-14: Estructura espacial combinada para las dos subpoblaciones (Chicamocha
y Sumapaz).
r
Combined Spatial Structure Analysis
0,200
0,000
-0,200
-0,400
r
U
0,05
0,1
0,15
0,2
0,25
0,3
0,35
0,4
0,45
0,5
L
Distance Class (End Point)
Figura 2-15: Estructura espacial para cada población, Chicamocha y Sumapaz.
Spatial Structure Analysis for each Population
r
0,500
0,000
GD Chicamocha
-0,500
0
0
0
0
0
0
0
Distance Class (End Point)
0
0
1
GD (2) Sumapaz
64
Título de la tesis o trabajo de investigación
Figura 2-16: Correlograma (Omega=97.136) para la estructura espacial de la población
Sumapaz, [0.001 P(Omega-rand≥Omega-data], mostrando ser una población
estructurada.
Results of Spatial Structure Analysis
r
0,500
0,000
r
-0,500
U
0,05
0,1
0,15
0,2
0,25
0,3
0,35
0,4
0,45
0,5
L
Distance Class (End Point)
Figura 2-17: Correlograma (Omega=28.488) para la estructura espacial de la población
Chicamocha no significativo [0.001 P(Omega-rand≥Omega-data], mostrando una
población no estructurada.
Results of Spatial Structure Analysis
r
0,500
r
0,000
U
-0,500
0,05
0,1
0,15
0,2
0,25
0,3
0,35
0,4
0,45
0,5
L
Distance Class (End Point)
Discusión
La estructura genética poblacional se determinó usando los estadígrafos específicos F,
AMOVA, PCoA y Mantel. El análisis de Mantel agrupó los individuos por sus
características o proximidad geográfica. Los análisis de autocorrelación espacial
mostraron un patrón consistente de aislamiento por distancia con un moderado pero
significativo nivel de estructura espacial. Los resultados obtenidos sugieren atender lo
propuesto por Suárez et al. (2008) quienes refieren muestrear este tipo de Verbenaceae
a distancias mayores a 1.2 km obteniendo genotipos diferentes, lo que ayudaría a
preservar los niveles de DG. Las causas de este patrón espacial son desconocidas y
puede estar asociado, entre otros a dispersión restringida de semilla y/o distancias cortas
de movimiento de polen (Suárez et al., 2008).
Capítulo 2
3. Capítulo 3
3.1 Variabilidad
fitoquímica
entre
accesiones
colombianas de Lippia alba (Mill.) N.E. Brown
3.1.1 Introducción
Aceites esenciales/metabolitos secundarios en plantas
Los aceites esenciales son líquidos aceitosos aromáticos olorosos [a veces semilíquido o sólido], obtenidos a partir de material vegetal [flores, brotes, semillas, hojas,
ramas, cortezas, hierbas, maderas, frutos y raíces] (UNCTAD, 2005). Dependiendo
del tipo y calidad, los aceites esenciales pueden ser utilizados en diferentes
industrias. Se aplican principalmente en la industria alimentaria como aromatizantes,
la industria del perfume-fragancias, y la farmacéutica, para agregar sabor y/u olor a
medicamentos. Su uso en el servicio de salud se denomina "aromaterapia" (UNCTAD,
2005). En plantas, la producción de metabolitos secundarios-ms-es de origen
genético y cumplen funciones de atracción/ repulsión (Viccini et al., 2004; Pereira et
al., 2006). No obstante, su expresión está determinada por condiciones pre-existentes
en su hábitat (presencia/ausencia de: fitófagos, otras especies vegetales,
componentes biofísicos, otros posibles). Estos ms están siendo activamente usados
como bioprotectantes con acción _cida [sufijo] sustentables, en actuales sistemas de
producción agrícola alrededor del mundo (Das & Das, 2005). La presencia, el
rendimiento y la composición de los metabolitos secundarios en las plantas,
incluyendo los componentes volátiles y los que ocurren en los aceites esenciales,
pueden verse afectados de varias maneras, desde su formación en la planta hasta su
aislamiento final (Figueiredo et al., 2008). El conocimiento de los factores que
determinan la variabilidad química y el rendimiento en cada especie son muy
importantes (Figueiredo et al., 2008), en los que se incluyen las: (a) variaciones
fisiológicas; (b) condiciones del medio ambiente; (c) variaciones geográficas; (d)
factores genéticos y su evolución; (e) política y condiciones socioculturales y también
(f) cantidad de material vegetal / espacio (Riccardi et al., 2000; Jarvis et al., 2006;
Figueiredo et al., 2008; Palacio y López, 2008).
Orden Lamiales
El potencial fitoquímico de las Lamiales ha sido revaluado en: Eusteralis deccanensis,
la cual posee el quimiotipo patchoulene (Thoppil, 2000), Clerodendron infortunatum L.
(Verbenaceae) es usado como pesticida, siendo un componente de jardines en
Barak-Assam NE-India (Das and Das, 2005), Aegiphila filipens (Verbenaceae) and
Lippia dulcis [Verbenaceae] (Arango, 2004) la cual es usada en medicina tradicional
colombiana, actualmente se prueba como ingrediente en pasta dental (Pino-Benítez y
Valois, 2004), Lippia scaberrima en histoquimica (Combrinck et al., 2007); y Lippia
65
66
Título de la tesis o trabajo de investigación
alba por propiedades antioxidantes y neurosedativa de polifenoles e iridiodeos
(Hennebelle et al., 2007); la decocción filtrada de Phyla scaberrima (Juss. ex Pers)
Moldenke y Stachytarpheta jamaicensis (L.) Vahl; han sido usadas en tratamientos
antipatológicos y alteraciones del ciclo menstrual en humanos (Godínez y Volpoto,
2008).
Composición del aceite esencial de Lippia alba
Su aceite esencial tiene un relativo alto contenido de la carvona, entre 30 y 35%
(Castro et al., 2002). Esto lo convierte en un interesante sustituto de otros aceites
esenciales que contienen este mismo componente. Otro componente químico
importante de L. alba es el limoneno, componiendo más del 25% del aceite esencial
(UNCTAD, 2005). En L. alba, los metabolitos secundarios son flavonoides, alcaloides
y principalmente los aceites esenciales producidos por glándulas epidermales en
células del mesófilo. En investigaciones recientes, los principales compuestos del
aceite esencial observados en hojas son: terpinene, p-cymene, caryophylene,
myrcene, geraniol, and neral (Castro et al., 2002.; Jarvis et al., 2006; Blanco et al.,
2007; Celis et al., 2007; Mesa et al., 2009; Escobar et al., 2010).
Variabilidad química en plantas
Las plantas que viven en hábitats tropicales han tenido que desarrollar variedad de
defensas para protegerse de las enfermedades virales, agentes patógenos, insectos y
mamíferos predadores (Berier et al., s.f.). La biodiversidad de especies de plantas
tropicales, junto con la diversidad química encontrada en cada planta, nos lleva a la
conclusión de que las plantas tropicales son quizás la fuente más valiosa de nuevas
entidades químicas bioactivas (Berier et al., s.f.). En términos de los componentes
fitoquímicos sólo cinco grupos se han definido para más de 100 variedades con base
en consideraciones de carácter regional y organolépticas (Verma et al., 2004).
Variabilidad química de aceites esenciales en Lippia alba
La composición química del aceite esencial de L. alba depende sensiblemente del
origen geográfico de la planta, las condiciones de su cultivo, la edad y la parte de la
planta empleada para la extracción, y de algunos otros factores geobotánicos
(Stashenko et al., 2003). La mayoría de estudios en L. alba son determinaciones
genotípicas (Montanari et al., 2004; Viccini et al., 2004-2006; Dias et al., 2005-2006;
Suárez et al., 2007-2008; Yamamoto et al., 2008; Martínez et al., 2008),
caracterizaciones bioquímicas [quimiotipificación] (Fischer et al., 2004; Hennebelle et
al., 2006) y de morfología externa (Montanari et al., 2004; Hennebelle, 2007).
Individuos de L. alba de la región nororiental de Colombia mostró ser un quimiotipo
nuevo, no descrito previamente en la literatura, conteniendo la carvona [40-57%]
como componente mayoritario en los aceites, seguido de limoneno [24-37%],
biciclosesquifelandreno [2-22%] piperitenona [1-2%], piperitona [0.8-1.2%] y el βburboneno [0.6-1.5%]. El componente principal (en las fracciones volátiles) fue
limoneno [27-77%], seguido por la carvona [14-30%], biciclosesquifelandreno [1-33%]
y el β-burboneno [0.5-6.5%] (Stashenko et al., 2003). El análisis de aceites esenciales
obtenidos de hojas de muestras de Guayana Francesa, Martinica y Guadalupe,
mostraron siete quimiotipos diferentes (Hennebelle et al, 2006), existiendo una
posible conexión entre quimiotipos y morfotipos (Hennebelle et al., 2006). La
interacción GxE de 10 genotipos de cuatro composiciones químicas (quimiotipos) de
L. alba de tres regiones del estado de Sao Paulo (SP), Brasil, tuvieron un rango
amplio de producción de hoja, mientras que el rendimiento de aceites esencial tuvo
una alta y uniforme determinación genotípica. Variación no cualitativa fue detectada
Capítulo 2
para composición química, y la variación cuantitativa fue de baja magnitud. Genotipos
Linalol y limoneno/la carvona
fueron invariablemente más productivos para
rendimiento de aceite que los genotipos citral y mirceno/canfor (Yamamoto et al.,
2008). La variación de aceite esencial de Lippia junelliana de 16 sitios diferentes de
medio oeste Argentino mostró ocho compuestos como componente importante en los
aceites (Juliani et al., 2002). De acuerdo con estos componentes, cuatro quimiotipos
silvestres se han detectado. El quimiotipo primero contenía altos niveles de
ocimenone (54-76%) con cantidades más bajas de mirceno (10%). El segundo se
componía de grandes cantidades de dihidrocarvona (59-80%). Los principales
componentes se encuentran en la tercero,
quimiotipo limoneno (10-40%) y
piperitenona (10-40%) y la cuarta contenía quimiotipo limoneno (41%) y óxido de
piperitenona (26%) (Riccardi et al., 2000; Juliani et al., 2002).
Las especies de L. alba (Miller) N.E. Brown se caracterizan por una gran variabilidad
en morfología y composición química del aceite esencial (Oliveira et al., 2006).
Tavares, et al. (2005), presentan datos sobre la variación cuantitativa de los
principales componentes volátiles de la producción de linalol en un quimiotipo de L.
alba. El contenido de [alpha]-pineno, (Z)-3-hexenilo y [alpha]-gurjunene fue mayor en
las plantas madre cultivadas en el suelo que en las plántulas crecieron en medio MS,
mientras que el contenido de sabineno, mirceno, 1,8 -cineol y p-menta-l, 5,8-trieno fue
menor. La adición de 0.2.3 μM de IAA en el medio fue significativamente mayor para
sabineno y el contenido mirceno. La adición de 0,92 μM de cinetina aumentó
significativamente el 3 (S) - (+)-nivel linalol (Tavares et al., 2005). En Colombia,
Camargo y López (2008) y Palacio y López (2008) evaluaron la respuesta de L. alba a
la disponibilidad de agua; y nitrógeno (Hernández et al., s.f.; Antolinez y López, 2008).
Las variaciones cuantitativas de los principales componentes volátiles de la
producción de linalol en quimiotipo L. alba están asociadas con factores nutricionales
y ambientales (Hernández et al., s.f.; Tavares et al., 2005; Camargo y López, 2008;
Palacio y López, 2008; Antolinez y López, 2008).
La composición química y la actividad antimicrobiana de los aceites esenciales en L.
alba las investigó Oliveira et al., (2006) con en el fin de relacionarlas a sus usos
tradicionales. El análisis de los aceites esenciales que permitió la identificación de dos
especies de Lippia alba, una como quimiotipo mirceno-citral (15% y 37,1%,
respectivamente) y Lippia alba f. intermedia como un quimiotipo citral (22,1%). Los
aceites esenciales de ambas especies fueron activos contra todos los
microorganismos probados (bacterias y hongos) mediante el ensayo de halos de
inhibición con rango desde 1,1 hasta 5,0 cm; probablemente debido a su alto
contenido de monoterpenos oxigenados (51,0% y 40,1%, respectivamente),
representada principalmente por aldehídos y alcoholes. Los datos químicos y
farmacológicos de L. alba obtenido por Oliveira et al., (2006) estuvieron de acuerdo
con la encuesta etnobotánica. En Colombia, EL Centro de Investigaciones en
Biomoléculas-CIBIMOL- de la Universidad Industrial de Santander ha realizado, entre
otros, estudios valorativos en enfermedades tropicales, de bioactividad y principios
activos de Lippia alba como antimicrobial (Bueno y Stashenko, 2009); antiviral
[Ocazionez et al. (2010) en virus del dengue; Meneses et al., (2009) en virus de la
fiebre amarilla], antifúngica (Mesa et al., 2009) en Candida y Aspergillus (Montiel et
al.,2007), en bacterias [Bueno et al., (2009) en M. tuberculosis]; anti-protozoario
(Escobar et al. 2010). Una lista completa de reportes la ubica en
http://tux.uis.edu.co/quimica/ investigacion/centros/cibimol.
La apuesta en la presente investigación fue determinar la variabilidad química de 59
accesiones de Lippia alba de tal forma que permitiera una aproximación a la
estructuración poblacional de la especie en Colombia. Se logró, mediante la
67
68
Título de la tesis o trabajo de investigación
construcción de una matriz de datos químicos que permitió: a) determinar variabilidad
química y b) conformar una base de datos compatible con datos moleculares y
ecológicos que determinó la estructura genética y espacial de la población
colombiana de L. alba.
3.1.2 Metodología
Material vegetal. Muestras de 200 gramos de tejido (excepto raíces) de 59
accesiones de L. alba colectadas en dos zonas agroecológicas de la Región Andina
colombiana (Chicamocha y Sumapaz), conformantes del Banco de Lippia alba del
CEUNP. Las 59 accesiones forman parte del Banco in vivo ex situ de Lippia alba del
Centro Experimental Universidad Nacional-Sede Palmira (CEUNP). Para el estudio,
se consideraron los ecosistemas Bs-T (Bosque seco Tropical) y Bh-T (Bosque
húmedo Tropical), dado que los remanentes de Bs-T contienen familias de plantas
similares a los hallados en Bh-T (IAVH, 1998). Se prospectaron dos agroecosistemas
contrastantes, Cañón del rio Chicamocha y Cañón del rio Sumapaz; regiones con
máximo número de estudios y colectas de L. alba, conteniendo el mayor número de
Zonobiomas y agroecosistemas dentro de zonobioma; incluyendo y los ecosistemas
de Bs-T y Bh-T.
Tamizaje fitoquímico (García, 2003; García et al., 2003; García, 2006; Baldizán et
al., 2006) y estimativos de varianza (Peakall & Smouse, 2006). Pruebas
colorimétricas [por duplicado] con etanol y cloroformo como solventes, valoraron la
presencia (1)-ausencia (0) de siete grupos de metabolitos secundarios contenidos en
la muestra de trabajo, usando el protocolo, equipos y apoyo técnico del Recurso
Humano (Anexo G) adscrito al Laboratorio de Fitoquímica de la Universidad Nacional
de Colombia-Sede Palmira [diciembre de 2012/enero de 2013]. Los estimativos de
varianza entre y dentro de poblaciones e individuos se estimaron usando el algoritmo
desarrollado por Peakall & Smouse (2006). Para dar respuesta a la pregunta de
investigación, se aplicó el Programa GenAlEx (Peakall & Smouse, 2006) a la matriz
de datos de laboratorio (de presencia/ausencia de metabolitos) a partir de pruebas
duales con etanol y cloroformo como solventes, para valorar la presencia (1)-ausencia
(0) de siete metabolitos secundarios [1. derivados de cumarinas, 2. esteroides y
terpenoides, 3. flavonoides, 4. glicósidos cardiotónicos, 5. saponinas, 6. heterósidos y
7. taninos] en 59 accesiones colombianas de Lippia alba. Las pruebas se
desarrollaron entre diciembre de 2012 y enero de 2013, bajo la supervisión del equipo
de trabajo, usando el protocolo (Anexo G) desarrollado por el Laboratorio de
Fitoquímica de la Universidad Nacional de Colombia-Sede Palmira.
Metodologías analíticas. Cuatro metodologías analíticas y dos tipos de datos fueron
usadas para establecer grupos y origen (genético, ambiental o genético-ambiental) de
la variabilidad química en la muestra de trabajo. Se utilizaron dos grupos de
procedimientos, Grupo I (DISTANCE, CLUSTER-TREE) y Grupo II (FACTOR,
PRINQUAL-BIPLOT) de SAS/STATv9.0; la estrategia WARDMLM.SAS y el Programa
GenAlEx (Peakall & Smouse, (2006). Paso I preliminar: Los datos crudos fueron
procesados usando el procedimiento DISTANCE+CLUSTER+TREE de SAS/STAT
v9.0 para hallar los posibles grupos químicos. Paso II. La matriz de datos de
laboratorio (Anexo 1) fue procesada con los procedimientos PRINQUAL y FACTOR
de SAS/STAT v9.0 para obtener un número óptimo de Factores Comunes (variables
sintéticas continuas), conformante de la matriz ultima para el análisis final conjunto
(ecológicos, químicos y moleculares). Una matriz final homologada mediante Factores
Comunes y únicos, fue procesada para determinar el análisis concluyente y número
de grupos. Adicionalmente, se usó la estrategia WARDMLM.SAS para identificar
Capítulo 2
69
número posible de grupos químicos, más un Biplot con PRINQUAL-FACTOR de
SAS/STAT.
La base de la investigación fue el Análisis de Factores con Probabilidad MáximasAFPM. El AFPM calcula estimativos de comunalidad preferentes con valores de
probabilidad máxima (o máxima probabilidad). La forma computacional más simple y
eficiente del Análisis Factor(ial) es el AFPM, el cual es obtenido por la misma vía
como los ACP, con el uso de la opción PRIORS=MAX de SAS/STATV9.0, en
presencia de matrices singulares. La forma usual del análisis es:
proc factor data= SAS-data-set method=principal scree
mineigen=0 priors=max outstat=<libname>;
run;
El Cuadrado de la Correlación Múltiple (CCM) de cada variable con todas las demás
variables fue usado como Estimativo de Comunalidad Preferente [o más importante]
(En: SAS/STATv9.0); y es la base para la conformación final de grupos. El grupo de
metodologías analíticas usadas, son procedimientos accequibles en diferentes
paquetes estadísticos, la dificultad radica en el Programa estadístico a elegir y la
construcción de una macro que los una. En el presente estudio se usó el lenguaje IML
de SAS/STATv9.0.
Interpretación del análisis. La matriz de datos generado por la prueba fueron
analizados usando el algoritmo desarrollado por el Programa GenAlex v.6.5 (Peakall
& Smouse, 2006) para datos binarios (1/0). La matriz de datos estuvo conformada por
58 accesiones (35 de la Región I=Sumapaz y 23 de la Región II= Chicamocha). Los
resultados del análisis de varianza se obtienen a partir de una matriz de distancia
para el cálculo de estadísticos F con base en las formulas:
Fst = AP / (WI + AI + AP) = AP / TOT
Fis = AI / (WI + AI)
Fit = (AI + AP) / (WI + AI + AP) = (AI + AP) /
TOT. Coeficiente de endogamia
Nm = [(1 / Fst) - 1] / 4 Numero efectivo de
migrantes por generación
AP es el Estimativo de varianza entre Poblaciones, WI es la varianza entre Individuos
y WP es el Estimativo de varianza dentro de Poblaciones; por su sigla en inglés. Los
estadígrafos a considerar en el análisis son los estadígrafos de F de la Tabla RD1.
Los valores de Fmax/F’st corresponden al rango de valores mostrados a través de 14
tipos de metabolitos secundarios [ms] valorados con dos tipos de solventes,
cloroformo y etanol.
3.1.3 Resultados
Posibles grupos fitoquímicos presentes en la muestra. El análisis para estimar
varianzas a partir de los datos fitoquímicos se muestra en la Tabla 3-1. La variabilidad
química dentro de individuo [WI=2.060] tuvo indicador alto y entre poblaciones
[AP=0.033], bajo; entre individuos mostró valor cero [AI=0.000]. AP fue la fuente de
variación con mayor peso en la respuesta dada por el ANAVA [MS=2.567]. La
variabilidad entre poblaciones [AP=0.033] fue baja con un FST=0.023
P(rand≥data=0.070) y proporción 2%. No obstante, el estimativo de varianza con
70
Título de la tesis o trabajo de investigación
mayor proporción ocurrió dentro de individuos [WI=98%]; es decir, la mayor
variabilidad se dio dentro de individuo. Los valores máximos y mínimos para FST
fueron 0.597 y 0.038 respectivamente.
Análisis genético con base en variables químicas. El índice de fijación promedio
(Fst) para todas las poblaciones fue de 0.023 con un rango que fluctuó entre 0.038 y
0.597 (p≥0.07). La diferenciación genética entre poblaciones (Fst) generado por la
prueba indica que químicamente las frecuencias alélicas son muy parecidas
(Fst=0.023) en las dos subpoblaciones; es decir, no ha habido diferenciación (Hs=Ht);
mostrando mucho flujo génico y poca deriva génica, corroborando que el flujo génico
ha sido el proceso evolutivo determinante (Nm=10.82). El coeficiente de endogamia
(o de coancestro) debida a apareamientos no aleatorios dentro de poblaciones,
mostró un exceso de heterocigosis (Fis= -0.46) en las poblaciones, como una posible
selección a favor de las heterocigotos.
Tabla 3-1:
Estimativos de Varianza [Est.Var.] para el tamizaje fitoquímico de 58
accesiones colombianas de L. alba, usando el Programa GenAlex v6.5.
Source
Among Pops
Among Indiv
Within Indiv
Total
Df
1
56
58
115
SS
2.567
42.493
119.500
164.560
MS
2.567
0.759
2.060
F-Statistics
Fst
Fis
Fit
Value
0.023
-0.462
-0.429
P(rand >= data)
0.070
1.000
1.000
Fst max
F'st
0.597
0.038
Nm
10.819
Est. Var.
0.033
0.000
2.060
2.093
Proporción
2%
0%
98%
100%
Discusión principal
El grado de diferenciación fitoquímico (por ms) entre poblaciones fue 0.033. Es decir,
las dos poblaciones son muy similares debido probablemente a su forma particular de
diseminación en Colombia (Antropocoria). No obstante, ese 2% indica una porción
mínima de individuos con grado máximo de diferenciación genética a causa de su
carácter alógama y viabilidad baja de semilla; de hecho, los resultados evidencian
reproducción sexual y diferenciación entre poblaciones debido muy probablemente a
alogamia más factores ambientales locales.
Los estimativos de varianza: a) entre regiones, indican poblaciones similares. Los
individuos presentes en cada región son sub-poblaciones; b) entre individuos,
muestran quimiotipos no diferenciables. Entre regiones la diferencia es apenas del
2%, indicando la presencia de una población fragmentada (o sub-poblaciones) en las
regiones evaluadas.
El estimativo de varianza dentro de individuo, indica variabilidad alta (98%) para
contenidos de ms causado por respuesta fisiológica/ambiental. El grado de
diferenciación para contenido de ms (de origen genético) entre y dentro de individuos,
tuvieron valores negativos (Tabla 1). Lo que significa que, químicamente los
Capítulo 2
71
individuos fueron muy parecidos debidos probablemente a su baja capacidad de
reproducción
sexual
y
su
forma
de
propagación-diseminación
(Antropocoria=Diseminación activa de material reproductivo, debido a la acción
humana). Los posibles grupos químicos se visualizan en el Biplot de la Figura 1. El
rango de valores de F nuestra la posibilidad: a) de identificar al menos un morfotipo
especial y b) discriminante de los ms en la conformación final de grupos.
Existen elementos multiplicadores interrelacionados que pueden intervenir en esta
variabilidad, como: determinantes genéticos, ambientales, de herbivoría y patógenos.
L. alba presenta una amplia individualidad genética, mostrando Expresividad variable
causado por caracteres poligénicos. Determinantes genéticos que intervienen en la
respuesta química de L. alba a las condiciones ambientales. Las variantes
interindividuales determinan el rango amplio de adaptabilidad de la especie a
condiciones ambientales en todo el rango altitudinal colombiano. Esta variación en el
genoma es causa de la respuesta a la presencia de ms. L. alba muestra variabilidad
interindividual para contenido de ms dependiente de múltiples factores derivados de
la expresión de la variabilidad biológica interindividual.
Respuestas específicas (obtenidas de la prueba) para:
Número grupos químicos presentes en la muestra de trabajo. La estrategia
WARDMLM.SAS determinó la presencia de nueve grupos fitoquímicos. La Tabla 3-2
y el Anexo 1 muestran el número final de grupos fitoquímicos generados por la
estrategia WARDMLM.SAS, con base en la matriz de datos de laboratorio. La matriz
de datos crudos (de presencia-ausencia) muestran nueve grupos fitoquímicos así:
Grupo I (Accesiones 189-359-360-362/3/4/5); Grupo II (174-201/2-209373/4/5/6/7/8/9); Grupo III (300/1/2/3/4/5); Grupo IV (346-348/9-350/1/2/3/4/356/7);
Grupo V (CEUN-308-310/1/2/3/4-320-322); Grupo VI (3-4-5); Grupo VII (369-370);
Grupo VIII (187-366/7); Grupo IX (340/1/2/3/4/5-347). No obstante, el Biplot de la
Figura 1 sugiere un grupo químico predominante mayoritario [en Colombia] y la
presencia de al menos tres accesiones con características químicas individuales; esto
lo demuestra las presencias/ausencias de ms en las accesiones 201, 213 y 341; las
cuales desplazaron, el centroide hacia la derecha y parte inferior del Biplot. La
localización de estas accesiones dentro del Biplot indica características químicas
propias y distintas al resto de accesiones incluidas en la prueba. Este resultado
sugiere la ‘posible’ colecta de individuos propagados (sexualmente) por semilla; su
verificación se realizó mediante marcadores moleculares RAM.
Tabla 3-2:
Número final de grupos y de individuos por grupo [Peso-Proporción]
obtenidos con la estrategia WARDMLM.SAS.
Grupo
Frecuencia
Peso
Proporción
1
7
7.0000
0.118644
2
10
10.0000
0.169492
3
6
6.0000
0.101695
4
11
11.0000
0.186441
5
9
9.0000
0.152442
6
4
4.0000
0.067797
7
3
3.0000
0.050847
8
3
3.0000
0.050847
9
6
6.0000
0.101695
72
Título de la tesis o trabajo de investigación
Las frecuencia destacadas fueron, en su orden: Taninos>Esteroides&Terpenoides>
Flavonoides; la menor frecuencia estuvo en Heterósidos y Glicósidos cardiotónicos.
Los siete tipos de ms (Tabla 3) valorados colorimétricamente en dos solventes
mostraron frecuencias altas para Taninos (Cloroformo-[C]=0.8136 y Etanol
[E]=0.6949), Esteroides&Terpenoides (C=0.8305 y E=0.6271) y Flavonoides
(C=0.6271 y E=0.7458); y bajas para Heterósidos (C=0.0169 y E=0.000) y Glicósidos
cardiotónicos [C=0.0508 y E=0.0508]. Los derivados de cumarinas y saponinas
mostraron valores intermedios; Los glicósidos y heterósidos fueron excepcionalmente
bajos, 0.05 y 0.01 respectivamente.
En general, las frecuencias [por tipo de solvente] fueron relativamente mayores para
cloroformo, con excepción hecha para flavonoides. En Saponinas, Esteroides y
Taninos, las frecuencias conjuntas (cloroformo y etanol) mostraron valores con
diferencias significativas entre solventes. Los valores de frecuencia fueron: C=0.3729
y E=0.1356 para Saponina; C=0.8305 y E=0.6271 para Esteroides y C=0.8136 y
E=0.6949 para Taninos. En cumarinas, glicósidos y heterósidos las frecuencias
fueron iguales (Tabla 3). Los restantes ms tuvieron frecuencias sin diferencias
significativas entre solventes, verificadas todas estadísticamente. En una escala
arbitraria, frecuencias menores de 0.3, se consideran bajas; entre 0.31 y 0.6,
aceptable; entre 0.6 y 1.0 buenas. Lo valores [de frecuencia] generados por el análisis
dice: Esteroides, flavonoides y taninos, bien; Saponinas, apenas aceptable en el
solvente cloroformo. Cumarinas, Glicósidos y heterósidos, bajas.
Tabla 3-3:
Proporción de individuos con valor [1] de presencia de siete tipos de
metabolito secundario en dos tipos de solvente, ordenados por tipo de solvente:
Cloroformo* y Etanol**.
Metabolitos
Cloroformo*
Etanol
Esteroides &
Terpenoides
Taninos
0.8305
0.6271
0.8136
0.6949
Flavonoides
0.6271
0.7458
Saponina espumídica
0.3729
0.1356
Derivados de Cumarinas
0.1186
0.1186
Glicósidos cardiotónicos
0.0508
0.0508
Heterósidos
Cianogénicos
0.0169
0.0000
Cloroformo
Etanol**
Flavonoides
0.6271
0.7458
Taninos
0.8136
0.6949
Esteroides &
Terpenoides
Saponina espumídica
0.8305
0.6271
0.3729
0.1356
Derivados de Cumarinas
0.1186
0.1186
Glicósidos cardiotónicos
0.0508
0.0508
Heterósidos
Cianogénicos
0.0169
0.0000
Ordenado por etanol
Metabolitos
Capítulo 2
Discusión
El tamizaje fitoquímico demostró la ausencia de alcaloides en Lippia alba (Hernández
et al., s.f.; Morataya et al., 2006; Medina-López et al., 2011). Con valores cero para
alcaloides, la estrategia WARDMLM.SAS definió un número final de nueve grupos
fitoquímicos. La sustentación técnica para el número de grupos la definió la estrategia
en el paso III, generando una lista con los log-Likelihood correspondientes a cada uno
de los posibles números de grupos y el gráfico logL versus número de grupos.
Los valores altos de frecuencia fitoquímica de taninos, esteroides & terpenoides y
flavonoides define los quimiotipos presentes en Colombia. Algunas de las
propiedades medicinales de Lippia alba se deben en efecto al alto contenido de
terpenos livianos como 1,8-cineol, limoneno, b-mirceno (Riccardi et al., 2000). La
actividad antioxidante y/o actividad antimicrobiana son atribuibles a los flavonoides y
fenoles/taninos (Martínez et al., 2002; Núñez et al., 2008; Escamillas et al., 2009;
López M.T. En: http://doymaforma.com el 04/05/2013). La variabilidad fitoquímica
hallada en la muestra de trabajo llevó a clasificarla en los nueve grupos fitoquímicos
detallados en el Anexo 1.
Los valores bajos de frecuencia fitoquímica revelan la existencia de otros posibles
quimiotipos para Colombia. Valores contrastantes (altos y bajos) de frecuencia
fitoquímicos, indican la presencia de un grupo mayoritario o quimiotipo predominante
en Colombia. La estrategia WARDMLM.SAS muestra tres grupos mayoritarios y es
más discriminante que el Biplot. No invalida la información generada por el Biplot,
sino que realiza un análisis más discriminante (detallado). Los resultados obtenidos
por las diferentes pruebas apuntan a que la variabilidad fitoquímica de la muestra de
L. alba, es del tipo fisiológica-ambiental y no existen factores epigenéticos
involucrados. Los análisis genéticos con marcadores RAM lo demuestran.
El resultado sugiere que cloroformo fue mejor solvente, sin poder ser demostrado
técnicamente en este documento. En el tamizaje fitoquímico de las 59 muestras, se
utilizaron estándares conocidos para comparar los posibles metabolitos presentes en
las muestras, muchos de los cuales corresponden a los reportados para Colombia en
la literatura, presentando frecuencias altas esperadas. No obstante, por su carácter
cualitativo, se requieren ajustes para validación de lecturas de cada método de
caracterización de extractos, como tablas colorimétricas, espectrofotómetros y otros
posibles, que permita construir escalas colorimétricas.
Análisis con base en datos de laboratorio procesados mediante Análisis de
Factores con Probabilidad Máxima-AFPM.
El clúster con el método Ward y la distancia de Gower, se aproxima mucho más al
Biplot de la Figura 3-1 y al clúster generado por los marcadores moleculares RAM.
Dos grandes grupos, uno conformado por las accesiones 187-356-340-454-370-367369-341 y el segundo gran grupo conformado por tres subgrupos (Figura 3-2). La
accesión 341 formó completamente independiente. El clúster muestra que son
químicamente iguales entre sí: las accesiones (1- 351); (356-359-360-378);
(362/3/4/5-374/5); (4-320); (174-189-373); (202-209-217-300-302/3/4/5-377); (301346-344), sin estar asociado a región. El Biplot generado por FC se muestra en la
Figura 3-2. Las características del Biplot-FC, son ampliamente distintas a la mostrada
por el Biplot de la Figura 3-3, y mucho más discriminante. El más confiable es que se
asemeje a los reportados por los marcadores moleculares.
73
74
Figura 3-1:
Título de la tesis o trabajo de investigación
Biplot para presencia/ausencia de ms en 59 accesiones de Lippia alba
El procedimiento AFPM (Análisis de Factores con Probabilidad Máxima) usó la opción
PRIORS=MAX en presencia de matrices con correlación singular; los valores propios
de la matriz reducida de correlaciones (Total=10.2698937, Promedio=0.51349468)
definió 11 factores con el criterio de retenibles explicando el 1.1464 de la variación
hallada en la prueba. La matriz de 11 factores incluyendo región, altitud y
Capítulo 2
coordenadas (Norte y Occidente) decimales; se muestra en el Anexo 3 y forma parte
de la matriz final de marcadores conjuntos que definieron la estructura genética y
fitogeográfica de muestras de Lippia alba usada en la prueba. Esta salida (output)
generada por el AFPM se considera la matriz de marcadores fitoquímicos, a partir de
la cual se generó el respectivo Biplot y el clúster Fitoquímico. El Biplot y el clúster
generado por los AFPM se muestran en la Figura 2 y Tabla 4.
Figura 3-2: Grupos fitoquímicos con base en AFPM de SAS/STATv9.0 para datos
netos de laboratorio
75
76
Título de la tesis o trabajo de investigación
Figura 3-3: Biplot para factores con valores de probabilidad máxima [FPM] de
presencia de ms en 59 accesiones colombianas de Lippia alba, incluyendo datos de
localidad [región, altitud, coordenadas].
Discusión
Los factores con probabilidad máxima conformaron un agrupamiento diferente usando
el mismo procedimiento analítico (o metodología analítica). Este agrupamiento se
acerca mucho más a lo explicado por el Biplot de la Figura 1 con base en datos
crudos (de laboratorio). El Biplot y el clúster muestra cuatro tendencias mayores; y
separa los quimiotipos en dos grandes grupos: región Sumapaz y Región
Chicamocha. Se quiso aquí mostrar las homologías dentro de región y posible
división en dos sub-poblaciones, dada por condiciones ambientales. De hecho los
Capítulo 2
grupos químicos finales, los determino la estrategia wardmlm.sas al inicio, teniendo
en cuenta región, altitud, y coordenadas, determino para la Región 1 (Chicamocha),
cuatro grupos; para la Región 0 (Sumapaz): cinco grupos. Lo que el clúster muestra
es la variabilidad WI dentro y entre sub-poblaciones. Las dos sub-poblaciones fueron
inducidas con base en datos de localización y los valores de Est.Var. obtenidos por el
ANAVA. Existe un factor de peso en la prueba, es el factor ambiental que predomina
en Lippia alba y ha sido sustentado por varios autores incluyendo Stashenko et al.
(2003). La variabilidad química neta, se definió plenamente en la Tabla 3-1.
Metodología analítica sustentable para estructurar poblaciones tipo Lippia alba
Matriz de Factores compatible con datos moleculares y ecológicos. La matriz
final de factores se presenta en el Anexo A. Los datos para la matriz final fueron
ordenados alfabéticamente por Factores: ecológicos + fitoquímicos + moleculares; es
decir, la matriz final es la sumatoria del número de factores de probabilidad máxima
obtenidos por cada tipo de marcador. En el presente estudio, el AFPM para datos
fitoquímicos de laboratorio genero 11 Factores. Los cuales se suman a la matriz de
factores ecológicos. Cualquiera de las matrices debe llevar datos de localización; este
estudio incluyó: región, altitud y coordenadas decimal norte y occidente.
Los requisitos fundamentales para la macro, es la inclusión de al menos una variable
continua. Las matrices van una a continuación de otra y lleva su propia nomenclatura
iniciando con el acrónimo ACC, luego va cada una de las variables. Para datos
ecológicos antecede la letra E a cada Factor finalizado ordinalmente, es decir,
EFactor1 [Ecological Factor1] y así sucesivamente. Los individuos [ACC] van
ordenados en la columna A; a partir de la columna B, se colocan las variables de
localización [región, altitud, coordenadas, otros posibles no usados en esta prueba].
El orden de las matrices y factores dentro de cada matiz es arbitrario. El termino
FACTORn debe ir completo, debido a que así es como lo presenta la salida SAS
(output). No hay restricción de uso de mayúsculas o minúsculas. La sustentación de
la metodología analítica para estructurar poblaciones tipo L. alba se hace en la Tesis
doctoral ‘Estructura genética y fitogeográfica de dos sub-poblaciones colombianas de
Lippia alba’.
Se desarrolló una macro en IML permite estimar DG, Est. Var. y patrones espaciales
tomados a partir de datos químicos, ambientales y moleculares como base. El
Análisis de Factores de Probabilidad Máxima) además de eliminar variables que no
dan respuestas, permite obtener estimativos de comunalidad final y conservar al
menos tres variables continuas de localización individual (altitud, coordenadas norte y
oeste); e incluir marcadores moleculares como patrón base en estudios
poblacionales. Una macro en lenguaje matricial IML permitió conformar patrones
espaciales (incluyendo datos de localización) con base en la combinación de al
menos dos grupos de datos (químicos, fisiológicos, ecológicos, otros posibles) más
datos moleculares como patrón base; admitió: a) estimar componentes de varianza,
conformar grupos y definir patrones de distribución espacial. La macro en lenguaje
IML se agrega en formato digital, para uso por otros investigadores.
Una matriz de factores moleculares (sine qua non); mas una matriz de factores
ambientales; mas una matriz de factores fitoquímicos; incluyendo una matriz de
factores fisiológicos, son ideales para definir patrones espaciales de distribución de
especies promisoria nativas y naturalizadas. Una CANCORR preliminar indicaría las
variables fisiológicas: Tasa de asimilación neta-TAN, Coeficiente de Área Foliar-CAF,
Área foliar Específica-AFE, Tasa de Crecimiento Relativo-TCR; como variables
predictoras
potentes
y
de
fácil
estimación.
77
78
Título de la tesis o trabajo de investigación
Figura 3-4: Clúster para presencia de siete metabolitos secundarios en una muestra de 59 accesiones colombianas de Lippia alba, con base en Análisis de
Factores con Probabilidad Máxima [AFPM], incluyendo datos de localidad.
4. Conclusiones y recomendaciones
4.1 Conclusiones
4.1.1 Marcadores ecológicos: conclusión principal
El análisis sugiere reportar la matriz n=40 variables * p=11 CP (Factores o marcadores)
como la matriz de factores (de peso) o ‘Marcadores Ecológicos’. Los Estimativos de
Comunalidad Final-ECF- por variable y por Factor generados en la prueba, son
determinantes de la presencia (permanencia y/o supervivencia) de individuos en los sitios
de muestreo.
Conclusiones específicas
El análisis de Patrones Espaciales permitió definir los atributos estructurales y
funcionales de los ecosistemas. Cuando analizamos datos espaciales, es de gran
importancia describir los patrones espaciales de una variable medida. Para el Factor 1 en
cada una de las dos regiones de las Figuras 2-16 y 2-17 sugiere dos regiones
contrastantes (Chicamocha y Sumapaz); en que las accesiones separadas por distancias
cortas no están correlacionadas. Los biomas Andino, Sub-andino y Basal, actualmente
el http://hermes.humboldt.org.co//ecosistemas/andes/ los describe como ecosistemas
transformados y áreas con cultivos
mixtos de autoconsumo, causado por las
necesidades de supervivencia de sus pobladores.
Se concluye que la autocorrelación espacial marca una pauta importante en ensayos
genéticos de especies promisorias nativas y naturalizadas y facilita el diagnóstico del
requisito de independencia.
La exploración de la estructura espacial (de los residuos) utilizando los semivariogramas
residuales debería ser una práctica rutinaria a añadir a los diagnósticos comunes de los
otros requisitos del análisis. El conocimiento de los niveles y distribución de la diversidad
genética en poblaciones naturales de plantas garantiza la representación de la
variabilidad genética de las poblaciones naturales amenazadas. Parece claro que la
estimación de los niveles de variación genética debe ser un cometido inherente a su
actividad. Muchas veces el material acumulado en bancos de semillas carece de esta
información básica, lo cual puede resultar en colecciones genéticamente poco
representativas que, aunque sean numéricamente muy abundantes, tienen una utilidad
muy limitada de cara a eventuales reforzamientos o reintroducciones poblacionales
(Vilches et al., 2004).
80
Título de la tesis o trabajo de investigación
4.1.2 Marcadores moleculares: conclusión principal
El resultado obtenido aquí y por Suárez et al., (2007-2008), Vega y Chacón (2008) y
Vega et al. (2013) permite afirmar que el territorio colombiano forma parte del centro de
diversidad genética o de distribución natural primario de la especie. Sustentando la tesis
de distribución de la especies, dado que sus centros de origen están asociados con
valores altos de DG y VG y albergan poblaciones de parientes silvestres (En:
http://www.biodiversidad.gob.mx/genes/
centrosOrigen/
Gossypium
/2doInforme
/Segundo% 20informe % 20 Gossypium . pdf). La muestra de trabajo usada en la prueba,
reporta valores intermedios de DG= 0.2467/0.2722 y distribución-adaptación amplia en
Colombia (0-2800 msnm) con valores H=0.7234/0.7331 altos, sugiriendo para este
estudio, la presencia de dos subpoblaciones en proceso de estructuración genética. Las
investigaciones realizadas por Montanari et al., (2004); Viccini et al., (2004-2006); Dias et
al., (2005-2006); Pereira et al., (2006); Yamamoto et al., (2008) y Manica-Cattani et al.
(2009), indican la presencia de mayor variabilidad genética-VG en Brasil comparado con
los resultados obtenidos en esta investigación. Este resultado propone un amplio centro
de origen y distribución primario de la especie que incluye el norte de Suramérica.
Conclusiones específicas
El uso de un marcador molecular tipo, es igual a sustentar el prototipo de distancia e
índices, y es comparable al arquetipo de un programa y marca de un equipo
determinado. En todos los casos, lo importante es lograr un resultado final limpio, es
decir, una salida (output) sustentable.
Los RAM son marcadores dominantes igual que RAPD e ISSR y han sido usados por
otros autores, siendo efectivos valorando RG nativos y promisorios en Colombia y Brasil,
mostrando poder discriminante alto, generando resultados válidos y sustentables. En el
presente estudio, la potencia de los RAM fue valorada mediante marcadores químicos y
ecológicos, incluidos en la prueba. Y, fueron tan efectivos en L. alba como lo fueron en
otras especies vegetales nativas y naturalizadas.
Lippia alba como recurso fitogenético (promisorio naturalizado en Colombia) fue
susceptible de ser analizada mediante RAM. El poder discriminante de RAM fue efectivo
en L. alba sustentado por los resultados generados en otros estudios en la especie (L.
alba) y en RG promisorios. Generaron patrones de bandeo claras y su análisis
comparativo con otros marcadores (químicos y ecológicos) muestran homología en los
resultados. Adicionalmente, el cálculo de DG de Nei fue limpio y claro, evidenciando
resultados obtenidos por otros autores con otro tipo de marcadores dominantes.
Una medida de la eficiencia relativa de un Marcador Molecular no ha sido desarrollada
plenamente. Los análisis comparativos con otro tipo de marcadores (químicos y
ecológicos) usados en la prueba, han determinado su poder discriminante alto y se
ofrece aquí como alternativa analítica de la DG en L. alba.
Los marcadores moleculares-MM-son efectivos pero la lectura de bandas y tipo de
distancia a usar en los agrupamientos inducen subjetividad al procedimiento. El uso
combinado de marcadores tipo, son idóneos para estructurar poblaciones de especies
vegetales (promisorias) del tipo Verbenaceae y minimiza la subjetividad generada por la
técnica de lectura de bandas y tipo de distancia a usar. La combinación de datos
Anexo B. Nombrar el anexo B de acuerdo con su contenido
81
Ecológicos-Moleculares-Químicos-E*M*Q (usando de coordenadas) y la variabilidad
química presente en cada individuo, optimizó (redefinió y configuró mejor) los
agrupamientos; que analizando cada marcador en forma individual. Un análisis factorial
simple determina la potencia de los efectos simples (E, M, Q) y/o de las interacciones
generadas por cada marcador y se convierte en una herramienta válida en el
procedimiento.
La hipótesis de estructura genética moderada para la muestra (de referencia) tiene como
sustento la variación geográfica. Como se hipotetizó, se detectaron niveles moderados
de DG entre individuos. De hecho, los estimativos de DG aquí reportados son
indirectamente comparables a estimaciones previas hechas por otros autores [Suárez et
al. (2007); Montanari et al., (2004); Viccini et al., (2004-2006); Dias et al., (2005-2006);
Pereira et al., (2006); Yamamoto et al., (2008); y Manica-Cattani et al. (2009)], debido al
tipo de marcador usado. Valores altos de DG fueron detectados con niveles altos de
alelos por locus y niveles altos de polimorfismos en alelos a través de individuos.
Niveles intermedios de DG indican aislamientos recientes, impidiendo el desarrollo de
una estructura genética fuerte de cada subpoblación; o flujo génico bajo o inexistente
entre individuos dentro de cada región. El AMOVA indicó una DG mayor dentro que entre
individuos, revelando ausencia o frecuencias bajas de alelos únicos y raros. Las posibles
barreras son de tipo biológico con impacto negativo en la composición génica de L. alba.
Los resultados indican además que los reservorios genéticos no son diferentes. La
pérdida de la DG ha tenido un impacto negativo enorme en el potencial evolutivo de la
especie.
En términos generales, los niveles intermedios de DG es explicada por: i) su forma
particular de diseminación (Antropocoria-etnobotánica-estenopropagación), ii) capacidad
baja de propagación sexual por fertilidad baja de la semilla. Este tipo de diseminación
determina el número bajo de alelos únicos y (alelos) raros; y iii) el régimen extractivo sin
reemplazo el cual está afectando mayormente la DG. A pesar de los factores limitantes,
el análisis mostró un valor remanentes de DG el cual revela que la especie mantiene
valores de incipientes de DG debido a polinización cruzada y producción de semilla. Los
resultados sugieren en todos los casos: a) intercambio de semilla genética, b) bancos de
germoplasma de mínimos individuos con máxima DG, c) Inducir DG.
4.1.3 Marcadores químicos: conclusión principal
No se observaron valores altos de Estimativos de Varianza [Est.Var.] que conduzcan a
establecer Diversidad Genética-DG- entre poblaciones e individuos, a partir de datos
fitoquímicos. Los resultados generados por la prueba apuntan a la presencia de dos subpoblaciones, delimitadas (cada una) por condiciones ambientales locales. Los valores
obtenidos de Fst muestra un estructura moderada entre las subpoblaciones (Fst=0.023
p≤0.07), sustentando el flujo génico mantenido por la especie en las dos regiones
estudiadas y muy probablemente para Colombia.
El valor alto de WI indica la posible presencia de individuos con variantes químicas
importantes. Las Verbenaceae tienden a variaciones fitoquímicas, particularmente Lippia
alba, en la que no se observa diferencias morfológicas entre distintas procedencias
(Riccardi et al., 2000). Estas variaciones fitoquímicas dentro de una misma especie
82
Título de la tesis o trabajo de investigación
botánica, producen cambios en la composición química, aun en plantas localizadas a
distancias cortas (Riccardi et al., 2000). Las accesiones 201, 213 y 341 se muestran
como variantes fitoquímicas importantes, conclusión soportada por los análisis
moleculares con marcadores RAM usados en la prueba (En: Diversidad molecular de 59
accesiones de Lippia alba detectado mediante marcadores RAM).
La cuantificación de ms por espectrometría y cromatogramas son ideales. No obstante, la
construcción de una matriz de presencias/ausencias permitió su unión a matrices de
datos moleculares con marcadores RAM, para la clasificación y conformación final de
grupos y la potencialización del análisis de diversidad molecular de la muestra de trabajo.
Conclusiones específicas
La variabilidad biológica puede ser de origen epigenéticos, genético, ambiental, genéticoambiental, de respuesta a la herbivoría o daño mecánico y latrogénica (por agentes
contaminantes especialmente químicos). Asumiendo, que la Variabilidad Individual [WI]
debe ser menor que la variabilidad entre individuos [AI]; estimar la variabilidad biológica
individual [WI] y la variabilidad biológica máxima total [WT], permitió obtener valores de
referencia poblacionales y establecer valores de variabilidad para la muestra de trabajo.
Las variables de localización [incluyendo las ambientales] explican en gran proporción, la
variabilidad fitoquímica de L. alba. La muestra de trabajo reportó rango muy amplio de
adaptabilidad, desde 0 en Buenaventura-Valle, hasta 2800 metros altitudinales en el
departamento del Cauca; revelado por el valor de variabilidad fitoquímica intra alta (del
98%) hallada en la prueba (WI=.2.060).
Los índices (Fst-Fit-Fis) muestran una estructura moderada de las subpoblaciones,
sustentado el flujo génico mantenido por la especie y un exceso de heterocigosis, como
posible selección a favor de los heterocigotos.
Las características fitoquímicas de las sub-poblaciones estudiadas corresponden a las
reportadas por un grupo significativo de autores colombianos; con variantes fitoquímicas
importantes y valor también importante de Estimativo de Varianza [Est. Var.]. El examen
de los aceites esenciales de la muestra de trabajo mostró que los componentes
terpénicos livianos son mayoritarios, que junto a taninos y flavonoides se constituyeron
en marcadores fitoquímicos. En consecuencia, a) la muestra de trabajo corresponde a los
quimiotipos ya documentados por otros autores para Colombia y muy probablemente
otros no reportados aun; lo cual valida los datos de laboratorio obtenidos; y b) la similitud
observada en la muestra determina el parentesco fitoquímico de las accesiones lo que
permite la conformación de un gran grupo químico dominado por Taninos-terpenoidesflavonoides.
Por lo extenso del análisis y la posible inclusión de muchos datos crudos, una macro en
IML u otro programa, permite conformar patrones espaciales (incluyendo datos de
localización). La combinación de al menos dos grupos de datos (químicos, fisiológicos,
ecológicos, otros posibles) más datos moleculares como patrón base; permite estimar
componentes de varianza, conformar grupos y definir patrones de distribución espacial.
Una matriz de factores moleculares es sine qua non.
.
Anexo B. Nombrar el anexo B de acuerdo con su contenido
83
4.2 Recomendaciones
Los biomarcadores ideales a usar serian: Bioquímicos. Tipo, cantidad y calidad de
aceites esenciales y sus respectivos metabolitos secundarios obtenidos por
espectrometría; Morfofisiológicos. Respuestas fisiológicas (tasas e índices fisiológicos:
TAN, RAF, AFE, IAF, TCR), morfológicas (acumulación y distribución de biomasa) y
citohistológicas (caracterizaciones citogenéticas) a partir del banco in vivo ex situ. Más un
grupo básico de descriptores para Verbenaceae ajustado a L. alba.
4.2.1 Para Marcadores Ecológicos
La Bioprospección de Lippia alba en Colombia, en búsqueda de nuevas entidades
químicas bioactivas, debe hacerse en todo el país a distancias aproximadas de 1-1,2
kilómetros entre sitios de muestreo, usando el software DIVA-GIS.
Se recomienda el uso de marcadores ecológicos, con base en patrones espaciales,
debido a la gran variabilidad intraespecífica (a nivel individual) hallada en Lippia alba,
causada por su sensibilidad alta de la especie a factores bióticos y abióticos.
4.2.2 Para Marcadores Moleculares
Establecer un protocolo-guía o software para lectura de bandas RAM, que maximice el
potencial valorativo de los marcadores RAM. Ello permitirá eliminar la subjetividad
generada por su valoración visual por parte del investigador.
4.2.3 Para Marcadores Químicos
Usar espectrometría acoplada a gases-masas y cromatogramas, genera variables
cuantitativas exactas.
A. Anexo A: matriz de factores de criterio
para 40 variables ecoambientales tomadas
en 59 accesiones colectadas en dos zonas
ecológicas contrastantes BhT y BsT
Factor Pattern
Var
reg
alt
CDN
CDO
cod
bio
clim
coveg
Tmax
Tmin
HRmax
HRmin
VVmax
VVmin
hPamax
hPamin
UVmax
Uvmin
DUmax
DUmin
pH
CO
N
Ca
K
Mg
Na
AI
CICE
CIC
P
Cu
Fe
MN
Zn
B
Ar
Li
A
tex
Factor1
Factor2
Factor3
Factor4
Factor5
Factor6
-0.63693
0.11124
-0.83564
-0.75653
0.52480
-0.50530
-0.31270
0.08237
-0.32545
-0.55359
0.76037
0.74706
0.38858
-0.05062
0.43366
0.71088
-0.01910
-0.13113
0.70567
0.17610
-0.25017
0.29290
0.37223
0.25028
0.13547
0.33035
0.05570
0.07687
0.22492
0.43930
-0.06903
0.33770
0.22498
0.35280
0.21948
-0.07206
0.08317
0.43146
-0.30284
-0.05693
0.26969
0.31066
0.14638
0.18538
0.13609
0.72538
0.70769
0.18211
-0.43351
-0.11443
0.01744
0.10746
-0.43666
-0.33437
0.44430
0.21316
0.60626
0.72756
-0.49422
-0.66272
-0.05462
0.13573
0.14989
0.37821
0.35810
0.30937
-0.00195
0.29395
0.46669
0.29293
-0.05282
-0.04230
0.05507
0.22199
0.12743
0.44519
0.47896
0.33308
-0.47236
0.51097
0.29482
0.03107
0.24144
0.18821
-0.07706
0.00150
-0.12573
0.08977
-0.21541
-0.42458
0.07326
0.02030
0.55103
0.43766
-0.52018
-0.44227
-0.53159
-0.46183
-0.08785
0.10660
0.44263
-0.01649
0.10679
0.63555
0.42481
0.41240
0.00491
-0.39033
0.62761
-0.07992
0.21153
0.26865
-0.22486
-0.05238
-0.03842
0.55208
0.09216
0.22937
-0.19820
0.19993
-0.17759
-0.51297
-0.24843
-0.37049
-0.33220
-0.16163
0.02134
-0.26121
0.41575
0.43523
-0.10778
-0.08087
-0.42039
-0.49954
0.16683
0.00094
0.12041
0.18781
-0.02675
0.06867
0.26311
0.26488
0.32084
0.40803
0.31899
0.27427
0.19168
-0.24267
0.39352
0.23197
0.33185
0.08720
-0.55107
-0.23328
0.24235
0.24247
-0.42364
-0.14603
0.33729
-0.16053
0.26503
0.20078
0.02384
-0.05141
0.12606
0.17702
0.11986
0.27773
-0.48590
-0.30413
0.31055
0.35612
-0.09532
0.17049
0.28800
0.19406
0.25897
0.33828
0.13426
0.35738
0.51992
-0.37599
0.17745
0.01300
0.34823
-0.30774
0.20466
-0.19007
-0.19658
-0.44284
0.29469
0.04379
-0.18642
-0.14001
-0.26222
-0.07863
-0.36096
-0.47338
0.45149
0.03013
0.39903
0.40438
0.21444
0.17041
-0.06986
-0.07946
-0.22941
-0.01795
-0.20641
-0.24430
-0.09810
-0.10745
0.08406
0.12532
-0.24524
-0.10491
0.21776
0.06325
0.04246
-0.18323
-0.38488
0.62949
0.66543
0.05146
0.15891
-0.00873
-0.18957
0.30817
-0.04594
0.54011
-0.32943
0.21604
0.04346
-0.23449
-0.12918
-0.07559
-0.44938
-0.22612
0.39327
-0.34437
Anexo B. Nombrar el anexo B de acuerdo con su contenido
A.
reg
alt
CDN
CDO
cod
bio
clim
coveg
Tmax
Tmin
HRmax
HRmin
VVmax
VVmin
hPamax
hPamin
UVmax
Uvmin
DUmax
DUmin
pH
CO
N
Ca
Anexo A:
85
(Continuación).
Factor7
Factor8
Factor9
Factor10
Factor11
Factor12
-0.14323
0.12713
0.18387
0.30362
0.59365
-0.08116
-0.30875
0.75004
0.04060
0.24123
-0.22230
-0.19249
-0.19527
0.04865
0.14431
-0.00104
0.23068
0.02946
0.18460
0.23769
0.11517
0.20430
-0.25729
-0.05585
0.15417
0.08757
0.04343
0.01440
-0.18643
-0.00589
0.00399
-0.17323
0.01851
-0.02104
-0.09601
-0.16493
0.01336
0.04219
0.04681
0.05059
0.12982
0.05886
-0.00091
-0.18154
-0.07386
-0.17634
0.07742
-0.08889
-0.06818
0.44189
0.04707
0.12041
-0.15515
-0.17607
-0.21928
-0.14793
0.03524
-0.05156
-0.11660
-0.11854
0.01188
-0.21837
-0.13073
0.15332
0.22325
-0.01355
0.31889
-0.14114
-0.04402
-0.12265
-0.22248
-0.23220
0.28557
-0.11557
0.13673
0.01755
0.17536
-0.10041
-0.22920
0.32317
0.13956
0.12768
0.07777
0.12306
-0.19178
-0.22212
-0.12915
-0.20895
0.11213
-0.01024
0.09109
0.16842
-0.13938
-0.17124
0.18839
0.03101
0.01939
-0.19125
-0.14146
-0.21707
0.07650
-0.01073
0.07312
0.04538
0.19993
0.11913
-0.03102
-0.12911
-0.00923
0.11365
-0.07385
-0.02104
-0.02789
0.01303
-0.02262
-0.02053
-0.10313
-0.25433
0.07558
-0.15606
0.02644
0.06443
0.09833
0.09446
-0.21674
-0.00563
-0.04972
-0.05566
0.01090
-0.01217
0.36396
0.31811
-0.07450
-0.09921
-0.08973
-0.18310
-0.07218
-0.09557
-0.04850
0.03761
-0.13481
0.04182
-0.11963
-0.17020
Factor Pattern
K
Mg
Na
AI
CICE
CIC
P
Cu
Fe
MN
Zn
B
Ar
Li
A
tex
A.
Factor7
Factor8
Factor9
Factor10
Factor11
Factor12
-0.18549
0.35262
-0.18464
-0.11109
0.20487
0.00098
0.11695
-0.04172
0.02652
0.35061
0.05151
0.35409
-0.17071
-0.03480
0.13751
-0.29067
0.15234
-0.07964
-0.05765
-0.32936
-0.19002
-0.11939
0.46571
0.49333
0.44203
0.34398
0.38660
0.03144
-0.19493
0.27013
-0.06555
-0.31720
0.13400
0.21921
0.62123
0.07225
-0.01448
-0.02024
0.11101
0.07131
-0.15936
-0.16859
-0.30646
0.19108
0.04293
0.11168
-0.08800
0.04922
0.13290
-0.13082
0.05061
0.19036
-0.12146
-0.14405
-0.09829
0.42940
-0.29069
-0.16328
0.28893
-0.21181
0.25849
0.13218
-0.24746
0.07443
0.46388
0.31261
-0.13661
0.23139
-0.07674
-0.13316
-0.23987
0.18509
0.19882
0.22188
-0.35767
0.16610
-0.05815
-0.13136
0.10110
0.13180
-0.03170
0.19282
-0.02376
0.38317
-0.02338
-0.01170
0.30666
-0.10155
0.01714
0.23365
0.27929
0.17760
-0.04533
-0.23525
0.16942
0.11200
Anexo A:
(Continuación).
86
Título de la tesis o trabajo de investigación
Acc
Factor1
Factor2
Factor3
Factor4
Factor5
Factor6
377
201
374
369
379
375
378
202
189
187
364
174
209
367
365
217
349
363
366
362
370
373
350
346
357
351
360
359
356
358
340
348
342
352
341
344
301
4
347
345
5
3
311
320
-1.67980
-1.62821
-1.49460
-1.46919
-1.45315
-1.37931
-1.36084
-1.27381
-1.12543
-1.12480
-1.04030
-0.99939
-0.98844
-0.98506
-0.94711
-0.90904
-0.86355
-0.65292
-0.59868
-0.49846
-0.47206
-0.44006
-0.42672
-0.39542
-0.29271
-0.23047
-0.22148
-0.20525
-0.17787
-0.15115
-0.09755
-0.05537
-0.01732
0.17097
0.27705
0.46348
0.47495
0.54198
0.72169
0.74278
0.76570
0.81037
0.82555
0.86234
0.86652
-1.35553
-1.33242
-0.88057
0.66354
-0.76288
-1.68720
-0.69123
-0.76218
0.76419
0.87522
0.75922
-0.76250
0.12166
1.00906
1.38011
-0.20570
-0.55623
1.19100
1.59497
1.59909
2.43611
-0.02008
-0.03577
-0.16631
-0.00811
-0.14938
1.68023
1.62129
-1.34636
0.00996
-0.35487
0.11705
-0.57909
-1.06479
-0.69798
-0.25296
-2.39598
0.37820
0.56001
1.02856
1.08908
-0.22484
0.05762
-0.17121
0.41304
-0.06415
-0.07855
0.34551
0.30452
0.38434
-0.64990
0.60924
0.24555
-0.35486
-0.74503
-0.66261
0.55936
0.10906
0.12520
0.62558
0.00124
-0.54649
0.42607
1.14794
1.11626
3.27122
1.49836
0.10143
-1.60341
-0.23733
-0.71269
0.52072
-2.03360
-0.93442
0.11505
0.02882
-0.40836
-0.52330
-0.84816
-0.70261
0.08013
0.20683
-1.28608
-0.67106
-0.10198
-0.61997
-1.43103
-1.57817
-0.73747
-1.27785
0.22741
0.21672
0.20091
0.03121
0.30435
-0.54035
0.33690
-0.56197
-1.12883
-1.51338
-1.43527
-0.48771
-1.50070
-0.56886
-0.60272
-1.07655
1.30663
-0.49735
-0.42534
0.02067
1.36537
1.95171
1.67509
-0.04002
0.84538
1.16400
-1.16143
-1.30460
0.54372
1.99691
1.10652
0.80834
0.79076
-0.43908
1.64618
0.68401
-1.22516
0.58113
1.19786
0.96193
0.08054
1.50137
-0.58061
-2.07011
0.80167
0.55299
0.62778
0.32897
0.71820
-0.00680
-0.13079
0.52172
-0.59816
0.59656
-1.11500
-0.94206
0.89699
0.69486
-0.38617
-0.25158
1.01280
0.46482
0.19866
0.03332
-0.10017
0.44903
-0.94339
-0.28700
-1.69543
-0.63977
-1.13214
0.29236
-0.94121
1.44184
-1.02783
-0.18105
-1.76579
0.39627
1.45772
-0.60677
0.69871
0.26676
1.40539
-2.35487
-1.54075
1.87687
-0.10286
0.98641
0.78187
0.81286
0.48287
0.45920
-0.04969
0.30560
0.24594
0.74902
-0.02266
-0.38679
-0.43425
0.81152
0.57315
0.20130
-0.61307
0.30848
-0.15907
-0.23684
-0.54457
0.33533
-0.15762
0.34818
-0.04249
2.49263
-0.60195
-0.63404
-1.41033
0.82508
-0.07975
2.47736
-1.06034
-0.93880
-1.57769
-0.57525
-1.67129
-0.63486
1.04252
-0.91670
0.03738
-1.41163
-1.61240
-0.25535
-0.31952
1.54766
-0.27044
-0.50472
1.76078
Factor5
0.22257
-0.17792
-1.16226
-1.42566
1.33992
Factor6
-1.56237
-0.21452
-0.24947
0.06982
-0.90510
1
A.
Anexo A:
Acc
354
322
308
300
353
Factor1
0.89192
0.89519
1.11707
1.12044
1.24115
(Continuación).
Factor2
0.51997
0.25755
-0.58714
-1.28598
0.10136
Factor3
0.73313
-0.84130
1.55236
1.21911
0.74659
Factor4
-0.38211
0.56618
-1.27922
-0.80069
0.35773
Anexo B. Nombrar el anexo B de acuerdo con su contenido
314
302
304
305
310
303
313
312
2
377
201
374
369
379
375
378
202
189
187
364
174
209
367
365
217
349
363
366
362
370
373
350
346
357
351
360
359
356
358
340
348
342
352
1.29008
1.31495
1.32934
1.33323
1.33948
1.41566
1.57585
1.61804
1.64973
1.01999
1.00042
0.63799
0.48906
0.96241
0.30397
0.52727
0.66189
-0.50901
-0.68377
-0.68204
0.24063
-0.54863
-0.53752
-0.78962
-0.09885
-0.31831
0.24166
-0.63351
0.21168
1.23948
1.84869
-0.13335
-1.02825
-1.30247
-0.45638
-0.80655
-1.43019
-0.59749
-0.56192
-0.57144
-0.20378
-1.20195
-0.38669
A. Anexo A:
Acc
341
344
301
4
347
345
5
3
Factor1
-0.05486
-0.24739
-0.20645
-1.08566
-0.49582
-0.42232
-0.18920
0.83205
-0.64731
-1.23645
-1.46233
-1.14608
1.30870
-0.97882
0.69720
1.03229
0.54201
0.45236
0.39484
-0.39054
-0.09798
-0.17290
-0.45495
-1.05247
1.56117
-0.43750
0.66061
0.46261
0.38816
0.08911
0.33786
-0.45033
0.53066
-0.47524
1.25140
0.49128
0.23754
0.64848
-0.83578
-0.47312
0.19502
-1.91571
0.43387
-0.16941
-1.96913
-0.37178
-0.43226
-0.40179
1.07095
-0.47205
1.17219
-0.98013
1.48543
1.01397
1.34043
-1.73584
1.81162
-0.33120
-0.22415
1.19662
0.13569
0.08388
0.15678
-0.13283
0.10075
0.24022
1.32574
0.34168
-0.38231
0.17140
-0.44778
0.04708
-0.08482
-0.24716
0.07307
-0.41239
-0.26242
-0.49288
-0.05850
-0.33917
2.24230
-1.10927
0.22054
-0.22156
-0.52289
-0.24120
-0.89244
0.20826
-1.29168
-0.60189
0.18405
-0.07382
2.21662
-1.91839
0.36023
-0.94595
-0.64385
-0.98478
-1.15155
-0.90179
-0.30804
-0.40916
1.33570
0.85640
0.90432
1.09576
-0.96599
0.82361
1.09901
1.00107
2.31233
-0.08238
-0.35688
-0.29872
1.66197
-0.50074
-0.50866
-0.26634
-0.51298
-1.47330
-1.41614
-0.56910
-0.47785
-0.83949
0.08277
-1.26360
0.29630
0.31048
-0.71624
-0.12173
0.75345
-0.73158
-0.06283
0.13991
-0.28123
0.16537
-2.03676
87
-0.16930
-0.55121
-0.35973
-0.99615
-0.35944
-0.89986
0.28945
0.15214
3.33329
0.13790
0.06869
-0.20075
-0.33050
0.20584
0.31229
-0.06728
0.48152
0.14583
0.27637
0.22123
-0.26592
-0.35706
-0.98591
-0.39644
-0.20640
1.30138
-0.07240
-1.32045
-1.48699
2.65115
-1.15542
0.49088
1.00062
0.32066
0.84475
-1.75975
0.57450
-0.11404
-0.46007
0.47744
1.85172
-0.21157
0.55498
2.26389
-0.05406
1.47004
0.42378
0.19883
-0.17613
-0.25835
-1.23867
2.35042
0.34502
0.53103
0.31838
0.19292
0.93185
-0.07203
0.23608
-0.64636
0.05591
-1.00444
-0.88834
0.88359
-0.05034
-0.27676
-0.68369
-0.19926
-0.04062
-0.14432
-0.39867
-0.63620
0.57714
0.87166
-0.18075
-1.41390
-0.87829
-1.17534
0.29957
2.57904
0.71787
-0.23064
-1.16371
-0.16480
-0.46944
0.83813
Factor5
-0.09742
0.12394
0.66677
-1.29875
-2.04263
1.28027
-1.17457
-1.06015
Factor6
-0.57270
0.89414
0.98901
0.42839
1.67288
2.50961
1.14961
-0.65383
(Continuación).
Factor2
-0.46978
-0.75462
1.24866
-0.32395
2.82596
-0.12727
-0.33014
-0.09908
Factor3
-0.89102
-0.14423
-0.07836
3.36086
-1.83799
-0.49050
1.09603
0.89292
Factor4
-1.20749
-0.36304
-1.76246
0.26511
2.05780
-0.37856
-0.98611
-0.67268
88
Título de la tesis o trabajo de investigación
311
320
1
354
322
308
300
353
314
302
304
305
310
303
313
312
2
1.27688
1.37470
1.96763
0.87802
0.40441
0.58200
-0.27984
0.86649
0.10499
-0.77284
-0.82839
-0.54219
0.97869
-0.67853
1.37040
2.80577
-3.54193
0.45368
0.24592
-0.53414
-2.62205
1.18466
-1.35119
0.52548
-1.71047
0.44138
0.46279
-0.33320
-1.29977
-1.81894
-0.19846
1.46108
1.67796
1.64031
-0.29199
-0.35284
0.03955
-1.72458
2.06129
-0.54545
0.85661
-2.40938
0.28840
0.94884
0.48896
0.52896
0.80132
0.72055
0.40883
-0.42623
-1.31522
0.81623
-1.02073
-0.44525
0.95088
0.44843
0.80616
-0.30093
0.65864
-0.80795
-0.14845
-0.66430
-0.62013
1.64609
0.07066
1.75652
-0.68651
2.56785
-0.60351
1.35150
-0.93252
0.55558
-1.43309
0.04681
0.85473
-1.16978
-0.57859
-1.35922
-0.50287
0.68633
1.90831
-1.40257
0.51099
1.07959
2.06406
0.90651
-1.68462
-1.83256
-1.24095
-1.17527
-2.48010
0.97092
0.54678
-1.09311
0.27409
0.84968
0.69830
1.16596
-0.00303
-1.02666
1.04967
-1.00302
Anexo B. Nombrar el anexo B de acuerdo con su contenido
89
B. Anexo B. Patrón de Factores de criterio
por accesión incluyendo coordenadas
decimal (Norte y Oeste).
Accesión CDN CDO
Factor1
312 4.27242 -74.4425 -1.04920
314 4.19217 -74.4850 -1.04113
313 4.25847 -74.4745 -1.03023
304 4.31750 -74.5160 -1.01925
303 4.32053 -74.5150 -1.01466
305 4.31331 -74.5181 -1.01196
302 4.31203 -74.5000 -1.00193
300 4.31242 -74.4937 -0.96849
353 4.29685 -74.6423 -0.96453
310 4.28214 -74.4099 -0.92386
3 4.30997 -74.5261 -0.91719
347 4.30059 -74.8054 -0.88714
322 4.31475 -74.3893 -0.88217
308 4.27697 -74.5261 -0.87427
311 4.27256 -74.4122 -0.85084
301 4.31700 -74.4997 -0.84011
1 4.31331 -74.5181 -0.82326
354 4.28744 -74.6581 -0.80594
320 4.24689 -74.4433 -0.80525
4 4.27697 -74.5444 -0.79870
345 4.28527 -74.8771 -0.78660
341 4.39761 -74.8270 -0.77343
344 4.33510 -74.8557 -0.76987
Accesión CDN
CDO
Factor7
Factor2
Factor5
Factor6
1.54774 0.24963 -0.43272 -0.71864
-0.12411 -0.12511 -0.89208 2.40806
1.16690 0.23714 -0.67355 -0.13495
-0.74804 1.88535 -0.78538 1.27175
-0.21632 2.43835 -0.25742 -0.04871
-0.43887 2.09910 -0.57237 0.33364
-0.54091 2.19504 -0.51102 -0.09648
-0.65377 1.92206 -0.29616 0.31350
0.56688 0.81696 0.30590 -0.79661
1.59418 -0.96358 -1.72525 0.14498
0.01630 -1.10360 0.15036 2.08708
0.65328 -0.83011 1.07380 -0.04090
0.46392 -0.60045 -0.01026 0.21306
0.04192 2.12347 -0.39708 -0.02718
0.18527 -0.96384 -1.31903 -0.41556
-2.02132 1.10341 -1.11297 -0.29008
0.67832 -0.91958 -0.41301 1.97310
0.83361 0.71017 0.23113 -1.33146
0.08562 0.09248 -1.95899 -1.02639
0.30190 -1.38708 0.01473 -1.49660
1.19995 -0.34389 0.93383 0.57234
-0.57696 -0.72551 0.87218 1.76646
-0.14088 -0.07901 0.62802 -0.80918
1.26262
0.69615
0.79716
-0.18656
-0.98510
-0.92286
-0.75299
-1.27788
1.62813
0.09870
1.17454
-2.33040
0.24465
-0.88644
1.28395
-0.00206
2.28105
0.28003
0.77571
0.72605
-1.19403
0.16003
0.56277
Factor8
Factor3
Factor9
Factor4
Factor10
Factor11
312 4.27242 -74.4425 2.85861 0.00887 -1.02652 -0.82059 1.02284
314 4.19217 -74.4850 -0.50390 1.02155 -0.59874 0.29064 -0.87399
313 4.25847 -74.4745 2.33980 0.64797 0.54485 0.82780 -0.86171
304 4.31750 -74.5160 -1.44634 0.68537 -0.17287 0.48673 0.51074
303 4.32053 -74.5150 -0.65819 0.27496 0.26613 1.03380 -0.43812
305 4.31331 -74.5181 -1.06073 -0.04320 0.83230 -0.81553 0.72730
302 4.31203 -74.5000 -0.57365 0.99628 -0.10720 1.23748 -0.22035
300 4.31242 -74.4937 0.34576 1.06162 0.30835 -0.48616 0.89926
353 4.29685 -74.6423 -0.48057 -2.96687 -0.42529 1.07444 0.76381
310 4.28214 -74.4099 0.66847 -0.73576 2.90089 -1.09191 1.35687
3 4.30997 -74.5261 -0.25437 0.68192 0.13619 0.41607 -0.87422
347 4.30059 -74.8054 2.74298 -0.40789 -1.46515 3.06394 1.68145
322 4.31475 -74.3893 0.81332 1.76813 0.59999 1.50486 -1.73050
308 4.27697 -74.5261 0.41546 -1.66235 0.94208 -1.06617 -2.20408
311 4.27256 -74.4122 1.15322 -0.29624 0.05939 0.53514 0.50984
90
Título de la tesis o trabajo de investigación
B. Anexo B. (Continuación)
Accesión CDN
CDO
Factor7
Factor8
Factor9
Factor10
Factor11
301 4.31700 -74.4997 -0.09492 1.15951 -1.50937 -0.95925 1.00971
1 4.31331 -74.5181 0.06824 -0.42333 -0.09998 -0.43899 -2.12879
354 4.28744 -74.6581 0.04871 -3.23976 1.03552 -0.74654 -0.73027
320 4.24689 -74.4433 0.99922 -0.12997 -0.46834 -2.33848 -1.21383
4 4.27697 -74.5444 -1.35950 2.13445 1.87560 2.05910 -0.25603
345 4.28527 -74.8771 0.03780 -0.18593 -0.18455 -0.21925 2.46534
341 4.39761 -74.8270 -0.73105 -0.45643 -0.89911 -0.46932 -0.24535
344 4.33510 -74.8557 -0.83337 -0.31715 0.06045 0.10897 0.96999
Accesión CDN CDO
Factor1
5 4.28267 -74.4116 -0.71999
352 4.30427 -74.6327 -0.70685
342 4.40009 -74.8265 -0.67525
356 4.26807 -74.7026 -0.66800
340 4.39766 -74.8274 -0.61439
358 4.25201 -74.7197 -0.59534
348 4.30694 -74.7180 -0.59029
357 4.25591 -74.7157 -0.52992
351 4.37797 -74.6739 -0.51997
346 4.27328 -74.7528 -0.51547
350 4.36664 -74.6999 -0.45759
349 4.33670 -74.7078 -0.34377
359 5.85631 -73.6810 0.98135
373 7.03761 -73.1763 0.99336
360 6.50125 -73.2308 1.06187
217 6.54047 -73.3701 1.12776
375 7.21694 -73.0012 1.15135
174 7.04219 -73.1718 1.15191
362 6.50158 -73.2354 1.15902
363 6.50053 -73.2149 1.16692
209 6.85619 -73.2481 1.19367
366 6.57208 -73.1500 1.19944
202 7.00878 -73.1699 1.20760
Accesión CDN CDO
Factor7
Factor2
Factor5
Factor6
1.12104 -0.70055 -0.39298 -0.98052
-1.07043 -0.52958 -0.76041 -1.02435
-0.69773 -0.80114 0.80655 -1.33627
-1.50378 -0.96801 0.13153 -1.13658
-0.45258 -0.47245 1.44011 -1.03308
-0.09827 -0.62691 2.11326 0.13676
0.01140 -0.71033 1.00586 0.27106
-0.24981 -0.81292 1.17228 -0.91065
-0.30169 -0.98783 0.83938 1.43306
-0.47765 -1.63618 -0.08081 0.00641
-0.27193 -0.67140 1.82847 -0.11891
-0.96841 -1.22486 1.32244 -0.48136
1.44930 -1.46884 -2.26795 2.05016
0.23629 1.19650 1.66508 3.35732
1.55697 0.37906 -0.80847 -0.66898
-0.48925 0.07832 -0.86847 -0.85966
-1.97941 -0.26543 -0.77531 0.54204
-0.96978 0.60255 -0.35674 -0.34935
1.46107 0.60095 0.47245 0.17217
0.99630 0.17470 -0.16984 -0.02309
-0.19961 0.12437 -0.98527 -1.23299
1.39595 0.61866 0.24538 -0.49304
-1.06105 0.27228 -0.09404 -0.23172
1.61343
0.82938
-0.28519
0.85033
-0.51754
-0.88940
-1.63148
-1.31652
-0.80129
-2.15609
-0.10081
0.28450
-1.13427
1.19222
-0.41181
0.45362
-0.01835
0.66130
0.09351
0.19263
-0.18086
-0.46301
-0.62372
Factor8
Factor3
Factor9
Factor4
Factor10
Factor11
5 4.28267 -74.4116 -1.60205 1.00455 -0.14868 0.74980 0.54935
352 4.30427 -74.6327 -0.46305 0.09667 -2.84046 -0.59500 1.44273
342 4.40009 -74.8265 -0.93018 1.26736 1.46897 0.86252 -0.71086
356 4.26807 -74.7026 -0.95716 -0.69260 -0.99738 0.17232 1.27514
340 4.39766 -74.8274 -0.07877 -0.12004 0.52412 -0.34150 -0.83233
358 4.25201 -74.7197 -0.23640 -0.81296 -0.25900 0.39777 -0.31736
348 4.30694 -74.7180 1.24582 0.65757 -0.29699 -1.21995 0.32873
357 4.25591 -74.7157 -1.08022 -1.54353 0.85865 -0.34690 -0.74948
351 4.37797 -74.6739 -0.03827 0.44970 -0.70142 -0.88271 -0.94392
346 4.27328 -74.7528 0.50165 -0.03606 0.28090 -1.09556 -0.65076
350 4.36664 -74.6999 -0.62582 0.11889 -0.30564 -0.83775 -0.36046
349 4.33670 -74.7078 -0.83831 0.14361 -0.49797 -1.76018 0.16526
359 5.85631 -73.6810 -1.82401 -0.50612 1.66319 0.09770 2.70417
Anexo B. Nombrar el anexo B de acuerdo con su contenido
Accesión CDN CDO
Factor7
Factor8
Factor9
Factor10
91
Factor11
373 7.03761 -73.1763 0.11735 -1.62467 -0.33730 0.69428 0.60308
217 6.50125 -73.2308 -0.95756 -0.78641 -1.02893 1.37065 -0.23623
217 6.54047 -73.3701 -0.12501 0.18002 -0.91162 -0.06564 -0.21079
375 7.21694 -73.0012 0.45193 -0.30602 1.28092 -0.24501 0.36946
174 7.04219 -73.1718 0.56236 -0.02626 0.54458 1.28537 0.62133
362 6.50158 -73.2354 -0.30790 -0.18072 -0.99451 0.66337 -1.08077
363 6.50053 -73.2149 0.12387 0.60277 -1.82312 -0.40980 -0.25285
209 6.85619 -73.2481 -0.51693 0.09922 -0.54667 -0.02756 -0.33612
366 6.57208 -73.1500 -0.60766 0.18762 -1.14768 1.12051 -1.12590
202 7.00878 -73.1699 2.48462 0.36461 0.82301 0.68112 -0.47512
Accesión CDN CDO
378
364
367
189
379
374
201
365
187
377
370
369
7.00553
6.56658
6.57622
6.50078
7.00803
7.02361
7.00717
6.56833
6.59894
7.00900
6.64981
6.62675
-73.1693
-73.1484
-73.1517
-73.2328
-73.1696
-73.1673
-73.1781
-73.1400
-73.1566
-73.1792
-73.1693
-73.1646
Accesión CDN CDO
Factor1
1.24149
1.24477
1.25367
1.26119
1.26271
1.26369
1.27873
1.28227
1.29372
1.29629
1.30391
1.40012
Factor7
Factor2
Factor3
Factor4
Factor5
Factor6
-0.99640
0.38232
0.65232
0.29670
-1.09382
-1.24332
-1.73889
1.05913
0.45553
-1.77558
2.53625
0.15422
0.28617
-0.56185
-0.18417
-0.55815
0.12899
0.08310
-0.16571
0.12591
-0.66101
-0.16340
1.96484
-0.29708
0.60335
-1.21050
-0.23455
-0.82036
0.52726
0.43012
0.14216
0.09353
-1.27732
0.17894
2.90501
0.32719
-0.13857
0.25049
0.04344
-1.04311
0.31508
-0.08721
0.23224
-0.48500
0.42673
0.27218
-0.53828
-0.18766
0.70918
-1.43962
-0.69391
-0.22479
0.48112
0.47473
0.97641
-0.81155
-1.58000
0.91599
1.37173
0.74692
Factor8
Factor9
Factor10
Factor11
378 7.00553 -73.1693 -0.12465 -0.13420 1.77532 0.54464 -0.24083
364 6.56658 -73.1484 0.05654 0.11345 -0.54979 -0.77324 -0.45289
367 6.57622 -73.1517 -0.45092 0.09847 -0.84266 0.26961 -0.51527
189 6.50078 -73.2328 -0.50153 -0.48194 -0.08922 -0.28540 0.01734
379 7.00803 -73.1696 0.73824 -0.40199 0.71575 -0.03071 0.75107
374 7.02361 -73.1673 0.50307 -0.59964 0.86301 0.46949 -0.00021
201 7.00717 -73.1781 0.93333 -0.00834 0.41450 0.23931 0.46037
365 6.56833 -73.1400 -0.82989 -0.14130 -0.06655 -0.01402 -0.82634
187 6.59894 -73.1566 0.11742 0.62710 -0.31233 -0.93820 -0.75248
377 7.00900 -73.1792 0.98507 0.06559 0.40128 0.10330 0.27033
370 6.64981 -73.1693 0.23121 2.77867 1.19359 -2.50946 1.58669
369 6.62675 -73.1646 -0.45117 -0.02877 -0.71445 -0.52997 -0.21400
92
Título de la tesis o trabajo de investigación
C. Anexo C: lista de accesiones con
datos de colecta
Acc
Reg.
gn
mn
sn
go
mo
so
cod
bio
cli
cob
tmax
300
0
1223,00
alt
4,00
18,00
44,70
74,00
29,00
37,30
4,312416667
cdn
-74,493694444
cdo
33,00
1,00
1,00
15,00
35,00
301
0
1259,00
4,00
19,00
1,20
74,00
29,00
58,80
4,317000000
-74,499666667
33,00
1,00
1,00
15,00
35,00
302
0
1151,00
4,00
18,00
43,30
74,00
30,00
0,00
4,312027778
-74,500000000
33,00
1,00
1,00
15,00
27,00
303
0
1393,00
4,00
19,00
13,90
74,00
30,00
53,90
4,320527778
-74,514972222
33,00
1,00
1,00
15,00
28,70
304
0
1350,00
4,00
19,00
3,00
74,00
30,00
57,60
4,317500000
-74,516000000
33,00
1,00
1,00
15,00
29,00
305
0
1272,00
4,00
18,00
47,90
74,00
31,00
5,00
4,313305556
-74,518055556
33,00
1,00
1,00
15,00
30,00
308
0
816,00
4,00
16,00
37,10
74,00
31,00
34,00
4,276972222
-74,526111111
110,00
2,00
3,00
42,00
31,00
310
0
1296,00
4,00
16,00
55,70
74,00
24,00
35,70
4,282138889
-74,409916667
110,00
2,00
3,00
42,00
28,80
311
0
1365,00
4,00
16,00
21,20
74,00
24,00
43,80
4,272555556
-74,412166667
110,00
2,00
3,00
42,00
29,50
312
0
1176,00
4,00
16,00
20,70
74,00
26,00
33,10
4,272416667
-74,442527778
110,00
2,00
3,00
42,00
30,00
313
0
1077,00
4,00
15,00
30,50
74,00
28,00
28,30
4,258472222
-74,474527778
110,00
2,00
3,00
42,00
28,90
314
0
944,00
4,00
11,00
31,80
74,00
29,00
6,10
4,192166667
-74,485027778
33,00
1,00
1,00
15,00
27,60
320
0
1449,00
4,00
14,00
48,80
74,00
26,00
35,80
4,246888889
-74,443277778
110,00
2,00
3,00
42,00
29,10
322
0
1361,00
4,00
18,00
53,10
74,00
23,00
21,40
4,314750000
-74,389277778
33,00
1,00
1,00
15,00
30,00
340
0
264,00
4,00
23,00
51,59
74,00
49,00
38,53
4,397663889
-74,827369444
14,00
2,00
4,00
13,00
35,50
341
0
262,00
4,00
23,00
51,41
74,00
49,00
37,08
4,397613889
-74,826966667
14,00
2,00
4,00
13,00
33,80
342
0
260,00
4,00
24,00
0,31
74,00
49,00
35,52
4,400086111
-74,826533333
14,00
2,00
4,00
13,00
33,40
344
0
249,00
4,00
20,00
6,36
74,00
51,00
20,47
4,335100000
-74,855686111
16,00
2,00
4,00
26,00
29,00
345
0
296,00
4,00
17,00
6,97
74,00
52,00
37,41
4,285269444
-74,877058333
14,00
2,00
4,00
13,00
30,60
346
0
284,00
4,00
16,00
23,82
74,00
45,00
9,96
4,273283333
-74,752766667
14,00
2,00
4,00
13,00
35,40
347
0
283,00
4,00
18,00
2,13
74,00
48,00
19,59
4,300591667
-74,805441667
14,00
2,00
4,00
13,00
37,80
348
0
311,00
4,00
18,00
24,99
74,00
43,00
4,95
4,306941667
-74,718041667
14,00
2,00
4,00
13,00
39,20
349
0
316,00
4,00
20,00
12,13
74,00
42,00
28,23
4,336702778
-74,707841667
14,00
2,00
4,00
13,00
34,70
350
0
374,00
4,00
21,00
59,92
74,00
41,00
59,58
4,366644444
-74,699883333
14,00
2,00
4,00
13,00
35,00
351
0
392,00
4,00
22,00
40,69
74,00
40,00
26,02
4,377969444
-74,673894444
14,00
2,00
4,00
13,00
35,60
352
0
327,00
4,00
18,00
15,36
74,00
37,00
57,82
4,304266667
-74,632727778
14,00
2,00
4,00
13,00
27,20
353
0
326,00
4,00
17,00
48,65
74,00
38,00
32,37
4,296847222
-74,642325000
110,00
2,00
3,00
42,00
27,60
354
0
341,00
4,00
17,00
14,79
74,00
39,00
29,22
4,287441667
-74,658116667
110,00
2,00
3,00
42,00
29,80
356
0
397,00
4,00
16,00
5,04
74,00
42,00
9,33
4,268066667
-74,702591667
14,00
2,00
4,00
13,00
34,20
357
0
285,00
4,00
15,00
21,28
74,00
42,00
56,59
4,255911111
-74,715719444
14,00
2,00
4,00
13,00
35,50
358
0
275,00
4,00
15,00
7,22
74,00
43,00
10,97
4,252005556
-74,719713889
14,00
2,00
4,00
13,00
39,00
1
0
1650,00
4,00
18,00
47,90
74,00
31,00
5,00
4,313305556
-74,518055556
110,00
2,00
3,00
42,00
27,90
3
0
1470,00
4,00
18,00
35,90
74,00
31,00
34,00
4,309972222
-74,526111111
33,00
1,00
1,00
15,00
33,80
4
0
1570,00
4,00
16,00
37,10
74,00
32,00
39,70
4,276972222
-74,544361111
14,00
2,00
4,00
13,00
33,00
5
0
1830,00
4,00
16,00
57,60
74,00
24,00
41,60
4,282666667
-74,411555556
22,00
3,00
5,00
23,00
24,00
Localización [Acc= Accesión; Reg.= Región (0=Chicamocha, 1=Sumapaz); alt.=Altitud; gn=Grados Norte; mn=minutos norte;
sn=segundos norte; go=grados oeste; mo=minutos oeste; so=segundos oeste; cdn=coordenada decimal norte; cdo=coordenada
decimal oeste]. Ecosistemas (IAVH, 1998) [cod=código de clima; bio= ecosistema; cli= clima; cob= cobertura]; Clima [tmax=
temp. Máxima.
Anexo B. Nombrar el anexo B de acuerdo con su contenido
93
C: Anexo C (Continuación)
tmin
Hrmax
Hrmin
Vvmin
hPamax
Uvmin
Dumax
Dumin
pH
CO
N
Ca
K
24,00
70,00
52,00
Vvmax
15,00
8,00
1012,00
hPamin
1011,00
Uvmax
15,20
13,90
259,40
242,30
5,05
2,37
0,21
14,91
0,72
24,00
70,00
52,00
15,00
8,00
1012,00
1011,00
15,20
13,90
259,40
242,30
7,73
1,50
0,13
7,42
0,20
20,00
68,00
66,00
15,00
8,00
1012,00
1011,00
15,20
13,90
259,40
242,30
6,67
2,93
0,25
17,25
0,29
21,00
70,00
69,00
15,00
8,00
1012,00
1011,00
15,20
13,90
259,40
242,30
6,73
3,88
0,33
22,44
0,64
21,00
73,00
68,00
15,00
8,00
1012,00
1011,00
15,20
13,90
259,40
242,30
5,61
5,96
0,51
14,81
0,43
22,00
71,00
70,00
15,00
8,00
1012,00
1011,00
15,20
13,90
259,40
242,30
5,56
2,34
0,20
19,38
0,54
22,00
56,00
51,00
15,00
8,00
1012,00
1011,00
15,20
13,90
259,40
242,30
6,73
3,88
0,33
22,44
0,64
28,00
67,00
65,00
7,00
5,00
1014,00
1013,00
17,20
16,00
258,50
240,70
4,64
2,83
0,24
7,21
1,69
27,90
65,00
63,00
8,00
6,00
1014,00
1013,00
17,00
15,50
258,50
240,70
5,13
1,98
0,17
6,83
0,15
30,00
71,00
70,00
8,00
6,00
1014,00
1013,00
17,00
15,50
258,50
240,70
7,33
3,00
0,26
19,64
1,34
28,80
69,00
67,00
8,00
6,00
1014,00
1013,00
17,00
15,50
258,50
240,70
6,00
2,96
0,26
18,34
1,27
27,10
59,00
55,00
10,00
8,00
1014,00
1013,00
16,80
15,30
258,50
240,90
5,45
7,98
0,69
11,58
0,92
29,00
58,00
56,00
12,00
10,00
1014,00
1013,00
16,80
15,30
258,50
240,90
6,78
1,09
0,09
7,10
0,62
28,00
52,00
51,00
7,00
5,00
1014,00
1013,00
17,20
16,00
258,50
240,70
6,67
2,93
0,25
17,25
0,29
35,20
49,00
48,00
9,00
6,00
1013,00
1012,00
16,10
15,10
255,90
243,40
8,13
1,04
0,09
19,19
2,68
33,70
52,00
48,00
9,00
6,00
1013,00
1012,00
16,10
15,10
255,90
243,40
6,09
6,71
0,58
17,45
0,77
32,80
56,00
53,00
9,00
6,00
1013,00
1012,00
16,10
15,10
255,90
243,40
7,13
0,80
0,07
9,09
1,67
28,50
70,00
69,00
9,00
6,00
1013,00
1012,00
16,10
15,10
255,90
243,40
7,00
2,12
0,18
14,73
1,16
30,20
66,00
63,00
6,00
5,00
1013,00
1011,00
16,10
15,20
247,40
238,30
4,80
4,83
0,42
18,59
1,79
35,20
45,00
44,00
6,00
5,00
1013,00
1011,00
16,10
15,20
247,40
238,30
4,86
1,92
0,17
5,37
0,17
36,20
63,00
61,00
8,00
5,00
1014,00
1011,00
16,10
15,20
249,40
238,10
5,60
2,59
0,22
17,24
0,33
36,50
54,00
38,00
8,00
7,00
1013,00
1011,00
16,30
15,40
249,40
238,10
5,05
2,47
0,21
14,91
0,72
34,60
44,00
42,00
8,00
7,00
1013,00
1011,00
16,30
15,40
249,40
238,10
7,67
0,96
0,08
13,06
2,05
34,60
48,00
43,00
8,00
7,00
1013,00
1011,00
16,30
15,40
249,40
238,10
7,40
1,94
0,17
24,01
0,50
35,40
41,00
38,00
8,00
7,00
1013,00
1011,00
16,30
15,40
249,40
238,10
5,61
5,96
0,51
14,81
0,43
26,50
69,00
62,00
9,00
8,00
1014,00
1011,00
15,80
15,50
250,00
247,30
7,73
1,50
0,13
7,51
0,20
27,30
88,00
87,00
9,00
8,00
1014,00
1011,00
15,80
15,50
250,00
247,30
7,88
4,18
0,36
27,02
1,06
29,60
70,00
68,00
9,00
8,00
1014,00
1011,00
15,80
15,50
250,00
247,30
7,68
1,93
0,17
27,83
1,10
33,40
72,00
70,00
9,00
8,00
1014,00
1011,00
15,80
15,50
250,00
247,30
7,71
0,76
0,07
6,62
0,16
33,10
58,00
53,00
8,00
4,00
1013,00
1011,00
15,80
14,90
249,00
239,10
7,84
1,14
0,10
23,96
0,46
34,80
53,00
50,00
8,00
4,00
1013,00
1011,00
15,80
14,90
249,00
239,10
7,88
4,18
0,36
27,02
1,06
27,00
48,00
47,00
7,00
5,00
1014,00
1013,00
17,20
16,00
258,30
240,80
6,50
6,17
0,83
16,50
0,76
31,20
56,00
53,00
7,00
5,00
1014,00
1013,00
17,20
16,00
258,30
240,80
6,20
6,71
0,58
16,80
0,77
31,00
76,00
75,00
7,00
5,00
1014,00
1013,00
17,20
16,00
258,30
240,80
7,30
0,80
0,07
9,08
1,67
22,00
78,00
77,00
7,00
5,00
1014,00
1013,00
17,20
16,00
258,30
240,80
6,80
2,12
0,18
14,30
1,16
Tmin=temperatura mínima; Hrmax=Humedad relativa máxima; Hrmin=Humedad relativa mínima; Vvmax=Velocidad del viento
máxima; Vvmin=velocidad del viento mínima; hPamax=hPamin=; Uvmax=; Uvmin=; Dumax=; Dumin= Suelo [pH= acidez; CO=
Carbono orgánico; N=nitrógeno; Ca=Calcio; K=Potasio…]
94
Título de la tesis o trabajo de investigación
C. Anexo C: (Continuación)
Mg
Na
AI
CICE
CIC
P
Cu
Fe
Mn
Zn
B
Ar
L
A
text
Margalef
Menhinick
4,95
0,05
0,16
20,79
26,33
109,73
3,52
262,25
46,09
18,48
0,66
13,04
31,34
55,62
3,00
0,49
0,391
0,39
0,11
0,00
8,21
5,26
108,18
1,37
618,98
56,42
13,15
0,27
5,04
13,34
81,62
1,00
0,49
0,391
2,29
0,32
0,00
20,16
24,19
116,00
3,09
208,58
17,74
43,46
0,57
15,04
35,34
49,62
2,00
0,49
0,391
3,66
0,07
0,00
26,81
18,74
116,00
2,13
69,63
18,64
14,69
0,44
21,04
39,34
39,62
2,00
0,49
0,391
1,03
0,25
0,00
16,52
40,22
67,67
4,23
194,06
21,33
10,71
0,67
4,71
21,67
73,62
3,00
0,49
0,391
6,45
0,09
0,00
26,46
28,34
11,95
1,98
192,91
34,70
6,06
0,42
15,04
23,34
61,62
3,00
0,49
0,391
3,66
0,07
0,00
26,81
18,74
16,00
2,13
69,63
18,64
14,69
0,44
21,04
39,34
39,62
2,00
0,49
0,391
5,49
0,09
2,86
17,34
29,93
24,46
1,79
277,54
56,74
15,98
0,34
25,04
23,34
51,62
4,00
0,49
0,391
0,98
0,07
0,26
8,29
11,48
116,00
1,44
189,92
39,62
122,36
0,37
9,04
17,34
73,62
3,00
0,49
0,391
4,72
0,11
0,00
25,80
16,46
116,00
1,52
389,12
247,46
39,37
1,07
13,04
35,34
51,62
2,00
0,49
0,391
3,94
0,18
0,00
23,75
25,46
116,00
8,93
363,25
47,71
16,26
0,48
21,04
29,34
49,62
2,00
0,49
0,391
1,76
0,26
0,25
14,76
50,42
39,02
4,23
290,54
5,60
28,37
0,31
3,04
21,34
75,62
1,00
0,49
0,391
1,64
0,12
0,00
9,47
9,59
30,67
0,77
480,66
82,36
2,58
0,39
11,04
27,34
61,62
3,00
0,49
0,391
2,29
0,32
0,00
20,16
24,19
116,00
3,09
208,58
17,74
43,46
0,57
15,04
35,34
49,62
2,00
0,49
0,391
2,70
0,09
0,00
24,66
10,55
116,00
1,34
4,94
4,32
8,67
0,31
17,04
27,34
55,62
3,00
0,49
0,391
2,90
0,06
0,00
21,19
42,36
74,66
1,09
68,78
14,09
18,58
0,33
1,04
19,34
79,62
1,00
0,49
0,391
1,38
1,01
0,00
13,15
12,22
116,00
0,82
20,35
8,42
5,04
0,39
15,04
25,34
59,62
3,00
0,49
0,391
2,83
0,11
0,00
18,83
13,38
116,00
1,01
21,43
8,55
11,47
0,91
9,04
23,34
67,62
3,00
0,49
0,391
6,68
0,10
0,55
27,70
44,49
116,00
2,47
184,73
116,13
38,59
0,78
22,71
19,67
57,62
4,00
0,49
0,391
0,66
0,06
0,30
6,56
14,41
20,94
0,44
306,04
9,62
2,34
0,33
14,71
35,67
49,62
2,00
0,49
0,391
2,72
0,07
0,00
20,37
27,04
116,00
3,13
208,56
62,59
342,52
0,49
21,04
41,34
37,62
2,00
0,49
0,391
4,95
0,05
0,16
20,79
26,33
109,73
3,52
262,25
46,09
18,48
0,66
13,04
31,34
55,62
3,00
0,49
0,391
2,44
0,06
0,00
17,61
13,16
116,00
0,35
18,19
4,21
3,13
0,50
5,04
7,34
87,62
3,00
0,49
0,391
4,41
0,30
0,00
29,23
21,78
116,00
1,04
19,70
6,53
24,42
0,74
7,04
17,34
75,62
3,00
0,49
0,391
1,03
0,25
0,00
16,52
40,22
67,67
4,23
194,06
21,33
10,71
0,67
4,71
21,67
73,62
3,00
0,49
0,391
0,39
0,11
0,00
8,21
5,26
108,18
1,37
618,98
56,42
13,15
0,27
5,04
13,34
81,62
1,00
0,49
0,391
2,25
0,07
0,00
30,40
19,67
116,00
1,05
27,79
5,74
51,80
0,60
11,04
27,34
61,62
3,00
0,49
0,391
2,42
0,13
0,00
31,47
23,52
16,00
1,13
109,81
36,73
5,99
0,89
31,04
23,34
45,62
4,00
0,49
0,391
0,42
0,10
0,00
7,29
4,53
116,00
0,38
8,95
2,95
6,99
0,18
5,04
15,34
79,62
1,00
0,49
0,391
0,96
0,07
0,00
25,45
11,50
16,00
0,51
7,51
6,93
2,07
0,37
21,04
19,34
59,62
4,00
0,49
0,391
2,25
0,07
0,00
30,40
19,67
116,00
1,05
27,79
5,74
51,80
0,60
11,04
27,34
61,62
3,00
0,49
0,391
2,91
0,06
0,00
21,19
42,36
74,66
1,09
68,78
14,09
18,58
0,33
1,04
19,34
79,62
1,00
0,49
0,391
2,90
0,06
0,00
21,19
42,36
77,40
1,09
68,78
14,09
18,58
0,33
3,00
21,00
79,00
1,00
0,49
0,391
1,38
1,01
0,00
13,15
12,22
116,00
0,82
20,35
8,24
5,04
0,39
17,00
24,00
58,00
3,00
0,49
0,391
2,83
0,11
0,00
18,40
13,38
116,00
1,01
21,43
8,54
11,47
0,91
9,04
22,00
67,00
3,00
0,49
0,391
Suelo [Mg= magnesio; Na=sodio; Al=aluminio; CICE=capacidad de intercambio catiónico efectivo; CIC= Capacidad de
intercambio catiónico; P=Fósforo; Cu=Cobre; Fe= Hierro; Mn= Manganeso; Zn=Cinc; B=Boro; Ar=Arcilla; L=Limo; A=Arena;
text= textura; Índices de Biodiversidad [Margalef=Indice de equitatividad de Margalef; Menhinick= Indice de riqueza de
especies; Shannon= Índice de diversidad de especies; Brillouin= Indice de diversidad; Chai-1=estimado de diversidad especies
de una muestra; Chao-2= estimado de diversidad de especies total; Jacknife-1/2=distribución muestral de un estadígrafo.
D. Anexo D: frecuencias alélicas y genotípicas por cebador
y por locus
Cebador ACA_
Frecuencias genotípicas
Locus
ACA_1
ACA_2
ACA_3
ACA_4
Frecuencias alélicas
Genotipos
AA
Aa
aa
Freq gen (esp)
p2
2pq
q2
1
hj= (1-p2-q2)
TOTAL
Individuos
15
44
59
Freq gen (obs)
0,2542
0,7458
1,0000
TOTAL
Genotipos
AA
Aa
aa
Freq gen (esp)
p2
2pq
q2
1
Individuos
14
45
59
Freq gen (obs)
0,2373
0,7627
1,0000
TOTAL
Genotipos
AA
Aa
aa
Freq gen (esp)
p2
2pq
q2
1
Individuos
32
27
59
Freq gen (obs)
0,5424
0,4576
1,0000
TOTAL
Genotipos
AA
Aa
aa
Freq gen (esp)
p2
2pq
q2
1
Individuos
34
25
59
Freq gen (obs)
0,5763
0,4237
1,0000
p
q
0,1364
0,8636
p
q
0,1267
0,8733
p
q
0,3235
0,6765
p
q
0,3491
0,6509
0,2356
0,2212
0,4377
0,4544
96
Título de la tesis o trabajo de investigación
Locus
ACA_5
ACA_6
ACA_7
ACA_8
ACA_9
ACA_10
ACA_11
ACA_12
Frecuencias genotípicas
Genotipos
AA Aa
aa
Freq gen (esp)
p2 2pq
q2
Individuos
36
23
Freq gen (obs)
0.6102
0.3898
Genotipos
AA Aa
aa
Freq gen (esp)
p2 2pq
q2
Individuos
48
11
Freq gen (obs)
0.8136
0.1864
Genotipos
AA Aa
aa
Freq gen (esp)
p2 2pq
q2
Individuos
26
33
Freq gen (obs)
0.4407
0.5593
Genotipos
AA Aa
aa
Freq gen (esp)
p2 2pq
q2
Individuos
44
15
Freq gen (obs)
0.7458
0.2542
Genotipos
AA Aa
aa
Freq gen (esp)
p2 2pq
q2
Individuos
29
30
Freq gen (obs)
0.4915
0.5085
Genotipos
AA Aa
aa
Freq gen (esp)
p2 2pq
q2
Individuos
51
8
Freq gen (obs)
0.8644
0.1356
Genotipos
AA Aa
aa
Freq gen (esp)
p2 2pq
q2
Individuos
14
45
Freq gen (obs)
0.2373
0.7627
Genotipos
AA Aa
aa
Freq gen (esp)
p2 2pq
q2
Individuos
47
12
Freq gen (obs)
0.7966
0.2034
Frecuencias alélicas
TOTAL
1
59
TOTAL
1
59
TOTAL
1
59
TOTAL
1
59
TOTAL
1
59
TOTAL
1
59
TOTAL
1
59
TOTAL
1
59
p
q
0.3756
0.6244
p
q
0.5682
0.4318
p
q
0.2521
0.7479
p
q
0.4958
0.5042
p
q
0.2869
0.7131
p
q
0.6318
0.3682
p
q
0.1267
0.8733
p
q
0.5490
0.4510
hj=
(1-p2-q2)
0.4691
0.4907
0.3771
0.5000
0.4092
0.4653
0.2212
0.4952
Anexo B. Nombrar el anexo B de acuerdo con su contenido
Locus
ACA_13
ACA_14
ACA_15
ACA_16
ACA_17
ACA_18
ACA_19
Frecuencias genotípicas
Genotipos
AA Aa
aa
Freq gen (esp)
p2 2pq
q2
Individuos
32
27
Freq gen (obs)
0.5424
0.4576
Genotipos
AA Aa
aa
Freq gen (esp)
p2 2pq
q2
Individuos
4
55
Freq gen (obs)
0.0678
0.9322
Genotipos
AA Aa
aa
Freq gen (esp)
p2 2pq
q2
Individuos
10
49
Freq gen (obs)
0.1695
0.8305
Genotipos
AA Aa
aa
Freq gen (esp)
p2 2pq
q2
Individuos
40
19
Freq gen (obs)
0.6780
0.3220
Genotipos
AA Aa
aa
Freq gen (esp)
p2 2pq
q2
Individuos
3
56
Freq gen (obs)
0.0508
0.9492
Genotipos
AA Aa
aa
Freq gen (esp)
p2 2pq
q2
Individuos
18
41
Freq gen (obs)
0.3051
0.6949
Genotipos
AA Aa
aa
Freq gen (esp)
p2 2pq
q2
Individuos
5
54
Freq gen (obs)
0.0847
0.9153
97
Frecuencias alélicas
TOTAL
1
59
TOTAL
1
59
TOTAL
1
59
TOTAL
1
59
TOTAL
1
59
TOTAL
1
59
TOTAL
1
59
p
q
0.3235
0.6765
p
q
0.0345
0.9655
p
q
0.0887
0.9113
p
q
0.4325
0.5675
p
q
0.0258
0.9742
p
q
0.1664
0.8336
p
q
0.0433
0.9567
hj=
(1-p2-q2)
0.4377
0.0666
0.1616
0.4909
0.0502
0.2774
0.0829
98
Título de la tesis o trabajo de investigación
Cebador AG_
Locus
AG_1
AG_2
AG_3
AG_4
AG_5
AG_6
AG_7
Genotipos
Freq gen (esp)
Individuos
Freq gen (obs)
Genotipos
Freq gen (esp)
Individuos
Freq gen (obs)
Genotipos
Freq gen (esp)
Individuos
Freq gen (obs)
Genotipos
Freq gen (esp)
Individuos
Freq gen (obs)
Genotipos
Freq gen (esp)
Individuos
Freq gen (obs)
Genotipos
Freq gen (esp)
Individuos
Freq gen (obs)
Genotipos
Freq gen (esp)
Individuos
Freq gen (obs)
Frecuencias genotípicas
AA
Aa
p2
2pq
11
0.1864
AA
Aa
p2
2pq
17
0.2881
AA
Aa
p2
2pq
14
0.2373
AA
Aa
p2
2pq
35
0.5932
AA
Aa
p2
2pq
44
0.7458
AA
Aa
p2
2pq
54
0.9153
AA
Aa
p2
2pq
55
0.9322
Freq alélica
aa
q2
48
0.8136
aa
q2
42
0.7119
aa
q2
45
0.7627
aa
q2
24
0.4068
aa
q2
15
0.2542
aa
q2
5
0.0847
aa
q2
4
0.0678
TOTAL
1
59
TOTAL
1
59
1.0000
TOTAL
1
59
1.0000
TOTAL
1
59
1.0000
TOTAL
1
59
TOTAL
1
59
TOTAL
1
59
hj=
p
q
0.0980
0.9020
p
q
0.1563
0.8437
p
q
0.1267
0.8733
p
q
0.3622
0.6378
p
q
0.4958
0.5042
p
q
0.7089
0.2911
p
q
0.7396
0.2604
(1-p2-q2)
0.1768
0.2637
0.2212
0.4620
0.5000
0.4127
0.3852
Anexo B. Nombrar el anexo B de acuerdo con su contenido
Locus
AG_8
AG_9
AG_10
AG_11
AG_12
AG_13
AG_14
Genotipos
Freq gen (esp)
Individuos
Freq gen (obs)
Genotipos
Freq gen (esp)
Individuos
Freq gen (obs)
Genotipos
Freq gen (esp)
Individuos
Freq gen (obs)
Genotipos
Freq gen (esp)
Individuos
Freq gen (obs)
Genotipos
Freq gen (esp)
Individuos
Freq gen (obs)
Genotipos
Freq gen (esp)
Individuos
Freq gen (obs)
Genotipos
Freq gen (esp)
Individuos
Freq gen (obs)
Frecuencias genotípicas
AA
Aa
p2
2pq
52
0.8814
AA
Aa
p2
2pq
56
0.9492
AA
Aa
p2
2pq
55
0.9322
AA
Aa
p2
2pq
52
0.8814
AA
Aa
p2
2pq
54
0.9153
AA
Aa
p2
2pq
11
0.1864
AA
Aa
p2
2pq
9
0.1525
99
Freq alélica
aa
q2
7
0.1186
aa
q2
3
0.0508
aa
q2
4
0.0678
aa
q2
7
0.1186
aa
q2
5
0.0847
aa
q2
48
0.8136
aa
q2
50
0.8475
TOTAL
1
59
TOTAL
1
59
TOTAL
1
59
TOTAL
1
59
TOTAL
1
59
TOTAL
1
59
TOTAL
1
59
hj=
p
q
0.6556
0.3444
p
q
0.7745
0.2255
p
q
0.7396
0.2604
p
q
0.6556
0.3444
p
q
0.7089
0.2911
p
q
0.0980
0.9020
p
q
0.0794
0.9206
(1-p2-q2)
0.4516
0.3493
0.3852
0.4516
0.4127
0.1768
0.1462
100
Título de la tesis o trabajo de investigación
Cebador CA_
Locus
CA_1
CA_2
CA_3
CA_4
CA_5
CA_6
CA_7
Genotipos
Freq gen (esp)
Individuos
Freq gen (obs)
Genotipos
Freq gen (esp)
Individuos
Freq gen (obs)
Genotipos
Freq gen (esp)
Individuos
Freq gen (obs)
Genotipos
Freq gen (esp)
Individuos
Freq gen (obs)
Genotipos
Freq gen (esp)
Individuos
Freq gen (obs)
Genotipos
Freq gen (esp)
Individuos
Freq gen (obs)
Genotipos
Freq gen (esp)
Individuos
Freq gen (obs)
Frecuencias genotípicas
AA
Aa
aa
p2 2pq
q2
58
1
0.9831
0.0169
AA
Aa
aa
p2 2pq
q2
58
1
0.9831
0.0169
AA
Aa
aa
p2 2pq
q2
58
1
#¡DIV/0!
0.0169
AA
Aa
aa
p2 2pq
q2
58
1
0.9831
0.0169
AA
Aa
aa
p2 2pq
q2
58
1
0.9831
0.0169
AA
Aa
aa
p2 2pq
q2
58
1
0.9831
0.0169
AA
Aa
aa
p2 2pq
q2
58
1
0.9831
0.0169
Freq alélica
TOTAL
1
59
1.0000
TOTAL
1
59
1.0000
TOTAL
1
59
#¡DIV/0!
TOTAL
1
59
1.0000
TOTAL
1
59
p
q
hj=
(1-p2-q2)
0.2265
0.8698 0.1302
p
q
0.2265
0.8698 0.1302
p
q
0.2265
0.8698 0.1302
p
q
0.2265
0.8698 0.1302
p
q
0.2265
0.8698 0.1302
TOTAL
1
59
p
q
0.2265
0.8698 0.1302
TOTAL
1
59
p
q
0.8698 0.1302
0.2265
Anexo B. Nombrar el anexo B de acuerdo con su contenido
101
Cebador CA_ (Continuación)
Locus
CA_8
CA_9
CA_10
CA_11
CA_12
CA_13
CA_14
Genotipos
Freq gen (esp)
Individuos
Freq gen (obs)
Genotipos
Freq gen (esp)
Individuos
Freq gen (obs)
Genotipos
Freq gen (esp)
Individuos
Freq gen (obs)
Genotipos
Freq gen (esp)
Individuos
Freq gen (obs)
Genotipos
Freq gen (esp)
Individuos
Freq gen (obs)
Genotipos
Freq gen (esp)
Individuos
Freq gen (obs)
Genotipos
Freq gen (esp)
Individuos
Freq gen (obs)
Frecuencias genotípicas
AA
Aa
aa
p2
2pq
q2
58
1
0.9831
0.0169
AA
Aa
aa
p2
2pq
q2
58
1
0.9831
0.0169
AA
Aa
aa
p2
2pq
q2
58
1
0.9831
0.0169
AA
Aa
aa
p2
2pq
q2
59
0
1.0000
0.0000
AA
Aa
aa
p2
2pq
q2
59
0
1.0000
0.0000
AA
Aa
aa
p2
2pq
q2
59
0
1.0000
0.0000
AA
Aa
aa
p2
2pq
q2
59
0
1.0000
0.0000
Freq alélica
TOTAL
1
59
p
q
hj=
(1-p2-q2)
0.2265
0.8698 0.1302
TOTAL
1
59
p
q
0.2265
0.8698 0.1302
TOTAL
1
59
p
q
0.2265
0.8698 0.1302
TOTAL
1
59
p
q
0.0000
1.0000 0.0000
TOTAL
1
59
p
q
0.0000
1.0000 0.0000
TOTAL
1
59
p
q
0.0000
1.0000 0.0000
TOTAL
1
59
p
q
1.0000 0.0000
0.0000
102
Título de la tesis o trabajo de investigación
Cebador CA_ (Continuación)
Locus
CA_15
CA_16
CA_17
CA_18
CA_19
Genotipos
Freq gen (esp)
Individuos
Freq gen (obs)
Genotipos
Freq gen (esp)
Individuos
Freq gen (obs)
Genotipos
Freq gen (esp)
Individuos
Freq gen (obs)
Genotipos
Freq gen (esp)
Individuos
Freq gen (obs)
Genotipos
Freq gen (esp)
Individuos
Freq gen (obs)
Frecuencias genotípicas
AA
Aa
aa
p2
2pq
q2
59
0
1.0000
0.0000
AA
Aa
aa
p2
2pq
q2
59
0
1.0000
0.0000
AA
Aa
aa
p2
2pq
q2
1
58
0.0169
0.9831
AA
Aa
aa
p2
2pq
q2
1
58
0.0169
0.9831
AA
Aa
aa
p2
2pq
q2
1
58
0.0169
0.9831
Freq alélica
TOTAL
1
59
p
q
hj=
(1-p2-q2)
0.0000
1.0000 0.0000
TOTAL
1
59
p
q
0.0000
1.0000 0.0000
TOTAL
1
59
p
q
0.0169
0.0085 0.9915
TOTAL
1
59
p
q
0.0169
0.0085 0.9915
TOTAL
1
59
p
q
0.0085 0.9915
0.0169
Anexo B. Nombrar el anexo B de acuerdo con su contenido
103
Cebador CGA_
Locus
CGA_1
CGA_2
CGA_3
CGA_4
CGA_5
CGA_6
CGA_7
Frecuencias genotípicas
Genotipos
AA Aa
aa
Freq gen (esp)
p2 2pq
q2
Individuos
11
48
Freq gen (obs)
0.1864
0.8136
Genotipos
AA Aa
aa
Freq gen (esp)
p2 2pq
q2
Individuos
15
44
Freq gen (obs)
0.2542
0.7458
Genotipos
AA Aa
aa
Freq gen (esp)
p2 2pq
q2
Individuos
7
52
Freq gen (obs)
0.1186
0.8814
Genotipos
AA Aa
aa
Freq gen (esp)
p2 2pq
q2
Individuos
17
42
Freq gen (obs)
0.2881
0.7119
Genotipos
AA Aa
aa
Freq gen (esp)
p2 2pq
q2
Individuos
11
48
Freq gen (obs)
0.1864
0.8136
Genotipos
AA Aa
aa
Freq gen (esp)
p2 2pq
q2
Individuos
0
59
Freq gen (obs)
0.0000
1.0000
Genotipos
AA Aa
aa
Freq gen (esp)
p2 2pq
q2
Individuos
41
18
Freq gen (obs)
0.6949
0.3051
Freq alélica
TOTAL
1
59
1.0000
TOTAL
1
59
1.0000
TOTAL
1
59
1.0000
TOTAL
1
59
1.0000
TOTAL
1
59
p
q
hj=
(1-p2-q2)
0.1768
0.0980 0.9020
p
q
0.2356
0.1364 0.8636
p
q
0.1149
0.0612 0.9388
p
q
0.2637
0.1563 0.8437
p
q
0.1768
0.0980 0.9020
TOTAL
1
59
p
q
0.0000
0.0000 1.0000
TOTAL
1
59
p
q
0.4477 0.5523
0.4945
104
Título de la tesis o trabajo de investigación
Cebador CGA_ (Continuación)
Locus
CGA_8
CGA_9
Frecuencias genotípicas
Genotipos
AA Aa
aa
Freq gen (esp)
p2 2pq
q2
Individuos
3
56
Freq gen (obs)
0.0508
0.9492
Genotipos
AA Aa
aa
Freq gen (esp)
p2 2pq
q2
Individuos
2
57
Freq gen (obs)
0.0339
0.9661
Freq alélica
TOTAL
1
59
p
hj=
q
(1-p2-q2)
0.0502
0.0258 0.9742
TOTAL
1
59
p
q
0.0336
0.0171 0.9829
Cebador CCA_
Locus
CCA_1
CCA_2
CCA_3
CCA_4
Frecuencias genotípicas
Genotipos
Freq gen (esp)
Individuos
Freq gen (obs)
Genotipos
Freq gen (esp)
Individuos
Freq gen (obs)
Genotipos
Freq gen (esp)
Individuos
Freq gen (obs)
Genotipos
Freq gen (esp)
Individuos
Freq gen (obs)
AA Aa
p2 2pq
22
0.3729
AA Aa
p2 2pq
17
0.2881
AA Aa
p2 2pq
3
0.0508
AA Aa
p2 2pq
24
0.4068
aa
q2
37
0.6271
aa
q2
42
0.7119
aa
q2
56
0.9492
aa
q2
35
0.5932
Freq alélica
TOTAL
1
59
1.0000
TOTAL
1
59
1.0000
TOTAL
1
59
1.0000
TOTAL
1
59
1.0000
p
q
0.2081
0.7919
p
q
0.1563
0.8437
p
q
0.0258
0.9742
p
q
0.2298
0.7702
hj=
(1-p2q2)
0.3296
0.2637
0.0502
0.3540
Anexo B. Nombrar el anexo B de acuerdo con su contenido
105
Cebador CCA_ (Continuación)
Locus
CCA_5
CCA_6
CCA_7
CCA_8
Frecuencias genotípicas
Genotipos
Freq gen (esp)
Individuos
Freq gen (obs)
Genotipos
Freq gen (esp)
Individuos
Freq gen (obs)
Genotipos
Freq gen (esp)
Individuos
Freq gen (obs)
Genotipos
Freq gen (esp)
Individuos
Freq gen (obs)
AA Aa
p2 2pq
42
0.7119
AA Aa
p2 2pq
23
0.3898
AA Aa
p2 2pq
27
0.4576
AA Aa
p2 2pq
31
0.5254
aa
q2
17
0.2881
aa
q2
36
0.6102
aa
q2
32
0.5424
aa
q2
28
0.4746
Freq alélica
TOTAL
1
59
TOTAL
1
59
TOTAL
1
59
TOTAL
1
59
p
q
0.4632
0.5368
p
q
0.2189
0.7811
p
q
0.2635
0.7365
p
q
0.3111
0.6889
hj=
(1-p2q2)
0.4973
0.3419
0.3882
0.4286
106
Título de la tesis o trabajo de investigación
Cebador TG_
Locus
TG_1
TG_2
TG_3
TG_4
TG_5
Frecuencias genotípica
Genotipos
AA Aa
aa
Freq gen (esp) p2 2pq
q2
Individuos
13
46
Freq gen (obs)
0.2203 0.7797
Genotipos
AA Aa
aa
Freq gen (esp) p2 2pq
q2
Individuos
12
47
Freq gen (obs)
0.2034 0.7966
Genotipos
AA Aa
aa
Freq gen (esp) p2 2pq
q2
Individuos
12
47
Freq gen (obs)
0.2034 0.7966
Genotipos
AA Aa
aa
Freq gen (esp) p2 2pq
q2
Individuos
12
47
Freq gen (obs)
0.2034 0.7966
Genotipos
AA Aa
aa
Freq gen (esp) p2 2pq
q2
Individuos
13
46
Freq gen (obs)
0.2203 0.7797
Freq alélica
TOTAL
1
59
1.0000
TOTAL
1
59
1.0000
TOTAL
1
59
1.0000
TOTAL
1
59
1.0000
TOTAL
1
59
p
q
0.1170
0.8830
p
q
0.1075
0.8925
p
q
0.1075
0.8925
p
q
0.1075
0.8925
p
q
0.1170
0.8830
hj=
(1-p2-q2)
0.2066
0.1918
0.1918
0.1918
0.2066
Anexo B. Nombrar el anexo B de acuerdo con su contenido
107
Cebador TG_ (Continuación)
Locus
TG_6
TG_7
TG_8
TG_9
TG_10
TG_11
Frecuencias genotípica
Genotipos
AA Aa
aa
Freq gen (esp) p2 2pq
q2
Individuos
13
46
Freq gen (obs)
0.2203 0.7797
Genotipos
AA Aa
aa
Freq gen (esp) p2 2pq
q2
Individuos
37
22
Freq gen (obs)
0.6271 0.3729
Genotipos
AA Aa
aa
Freq gen (esp) p2 2pq
q2
Individuos
59
0
Freq gen (obs)
1.0000 0.0000
Genotipos
AA Aa
aa
Freq gen (esp) p2 2pq
q2
Individuos
32
27
Freq gen (obs)
0.5424 0.4576
Genotipos
AA Aa
aa
Freq gen (esp) p2 2pq
q2
Individuos
33
26
Freq gen (obs)
0.5593 0.4407
Genotipos
AA Aa
aa
Freq gen (esp) p2 2pq
q2
Individuos
11
48
Freq gen (obs)
0.1864 0.8136
Freq alélica
TOTAL
1
59
TOTAL
1
59
TOTAL
1
59
TOTAL
1
59
TOTAL
1
59
TOTAL
1
59
p
q
0.1170
0.8830
p
q
0.3894
0.6106
p
q
1.0000
0.0000
p
q
0.3235
0.6765
p
q
0.3362
0.6638
p
q
0.0980
0.9020
hj=
(1-p2-q2)
0.2066
0.4755
0.0000
0.4377
0.4463
0.1768
108
Título de la tesis o trabajo de investigación
Cebador GT_
Locus
GT_1
GT_2
GT_3
GT_4
GT_5
Frecuencias genotípicas
Genotipos
AA Aa
aa
Freq gen (esp) p2 2pq
q2
Individuos
11
48
Freq gen (obs)
0.1864 0.8136
Genotipos
AA Aa
aa
Freq gen (esp) p2 2pq
q2
Individuos
11
48
Freq gen (obs)
0.1864 0.8136
Genotipos
AA Aa
aa
Freq gen (esp) p2 2pq
q2
Individuos
11
48
Freq gen (obs)
0.1864 0.8136
Genotipos
AA Aa
aa
Freq gen (esp) p2 2pq
q2
Individuos
11
48
Freq gen (obs)
0.1864 0.8136
Genotipos
AA Aa
aa
Freq gen (esp) p2 2pq
q2
Individuos
11
48
Freq gen (obs)
0.1864 0.8136
Freq alélica
TOTAL
1
59
1.0000
TOTAL
1
59
1.0000
TOTAL
1
59
1.0000
TOTAL
1
59
1.0000
TOTAL
1
59
p
q
0.0980
0.9020
p
q
0.0980
0.9020
p
q
0.0980
0.9020
p
q
0.0980
0.9020
p
q
0.0980
0.9020
hj=
(1-p2-q2)
0.1768
0.1768
0.1768
0.1768
0.1768
Anexo B. Nombrar el anexo B de acuerdo con su contenido
109
Cebador GT_ (Continuación)
Locus
GT_8
GT_9
GT_10
GT_11
GT_12
GT_13
GT_14
Frecuencias genotípicas
Genotipos
AA Aa
aa
Freq gen (esp) p2 2pq
q2
Individuos
11
48
Freq gen (obs)
0.1864 0.8136
Genotipos
AA Aa
aa
Freq gen (esp) p2 2pq
q2
Individuos
11
48
Freq gen (obs)
0.1864 0.8136
Genotipos
AA Aa
aa
Freq gen (esp) p2 2pq
q2
Individuos
11
48
Freq gen (obs)
0.1864 0.8136
Genotipos
AA Aa
aa
Freq gen (esp) p2 2pq
q2
Individuos
11
48
Freq gen (obs)
0.1864 0.8136
Genotipos
AA Aa
aa
Freq gen (esp) p2 2pq
q2
Individuos
11
48
Freq gen (obs)
0.1864 0.8136
Genotipos
AA Aa
aa
Freq gen (esp) p2 2pq
q2
Individuos
11
48
Freq gen (obs)
0.1864 0.8136
Genotipos
AA Aa
aa
Freq gen (esp) p2 2pq
q2
Individuos
11
48
Freq gen (obs)
0.1864 0.8136
Freq alelica
TOTAL
1
59
TOTAL
1
59
TOTAL
1
59
TOTAL
1
59
TOTAL
1
59
TOTAL
1
59
TOTAL
1
59
p
q
0.0980
0.9020
p
q
0.0980
0.9020
p
q
0.0980
0.9020
p
q
0.0980
0.9020
p
q
0.0980
0.9020
p
q
0.0980
0.9020
p
q
0.0980
0.9020
hj=
(1-p2-q2)
0.1768
0.1768
0.1768
0.1768
0.1768
0.1768
0.1768
110
Título de la tesis o trabajo de investigación
E. Anexo E: bandas por individuo para siete cebadores
RAM
Accesión
ACA_1 ACA_2
ACA_3 ACA_4
ACA_5 ACA_6
ACA_7 ACA_8 ACA_9 ACA_10 ACA_11 ACA_12 ACA_13 ACA_14 ACA_15
174
1
1
1
0
1
1
0
1
0
1
0
1
1
0
0
187
1
1
1
0
1
1
0
1
0
1
0
1
1
0
0
189
1
1
1
0
1
1
0
1
0
1
1
1
1
0
0
201
1
1
1
0
1
1
0
1
0
1
1
1
1
0
0
202
1
1
0
0
1
1
0
1
0
1
0
1
0
0
0
209
1
1
0
0
1
1
0
1
0
1
1
1
0
0
0
217
1
1
0
0
1
1
0
1
0
1
0
1
0
0
0
300
1
1
0
0
1
1
0
1
0
1
1
1
0
0
0
301
1
1
0
0
1
1
0
1
0
1
0
1
1
0
0
302
1
1
0
0
1
1
0
1
0
1
1
1
1
0
0
303
1
1
0
0
1
1
0
1
0
1
0
1
1
0
0
304
1
1
1
0
1
1
0
1
1
1
0
1
1
0
0
305
1
0
0
0
1
0
0
1
0
0
1
1
1
0
0
308
1
0
0
1
1
0
0
1
0
1
1
1
1
0
0
310
0
0
1
0
0
0
1
1
1
1
1
1
1
1
0
311
1
0
1
0
0
0
1
1
1
0
1
0
1
0
0
312
0
0
1
0
0
0
1
1
0
0
1
1
1
1
0
313
0
0
1
0
0
0
0
0
0
0
0
0
1
0
0
314
0
0
1
0
0
1
0
1
1
0
0
0
0
0
0
320
0
0
1
0
0
0
0
1
1
0
0
0
1
0
0
322
0
0
1
0
0
0
0
0
0
0
0
0
1
1
0
112
Título de la tesis o trabajo de investigación
Accesión
ACA_1 ACA_2
ACA_3 ACA_4
ACA_5 ACA_6
ACA_7 ACA_8 ACA_9 ACA_10 ACA_11 ACA_12 ACA_13 ACA_14 ACA_15
341
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
342
0
0
0
1
0
1
0
0
0
1
0
0
0
0
1
344
0
0
0
1
0
1
0
0
1
1
0
1
0
0
1
345
0
0
0
1
0
1
0
0
0
1
0
1
0
0
1
346
0
0
0
1
0
1
0
0
0
1
0
1
0
0
1
347
0
1
0
1
0
1
0
0
1
1
0
1
0
0
1
348
0
0
0
1
0
1
0
1
1
1
0
1
0
0
1
349
0
0
0
1
0
1
0
1
1
1
0
1
0
0
1
350
0
0
0
1
0
1
0
0
0
1
0
0
0
0
0
351
0
0
0
1
1
1
0
0
0
1
0
0
0
0
0
352
0
0
0
1
1
1
1
1
1
1
0
1
0
0
0
353
0
1
0
1
1
1
1
1
1
1
1
1
0
0
1
354
0
0
0
1
1
1
1
1
1
1
0
1
0
0
1
356
0
0
0
1
0
1
0
0
0
1
0
0
0
0
0
357
0
0
0
1
0
1
0
1
1
1
0
1
0
0
1
358
0
0
1
1
1
1
1
0
0
1
0
1
1
0
0
359
0
0
1
1
1
1
1
1
1
1
0
1
1
0
0
360
0
0
1
1
1
1
1
1
1
1
0
1
1
0
0
362
0
0
1
1
0
1
1
1
1
1
0
1
1
0
0
363
0
0
1
1
1
1
1
1
1
1
1
1
1
0
0
364
365
366
367
369
370
373
374
375
377
378
379
1
2
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
1
1
1
1
1
1
1
0
1
0
1
1
1
1
1
1
1
1
0
1
1
1
1
0
1
1
1
1
1
1
1
1
1
0
1
1
1
0
1
1
1
1
1
1
1
1
1
1
1
1
1
0
1
1
1
1
1
1
1
1
1
1
1
1
1
0
1
1
1
1
1
1
1
1
1
1
1
1
1
0
1
1
1
1
1
1
1
1
1
1
1
1
1
0
1
0
1
0
1
1
1
1
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
1
1
1
1
1
1
1
1
1
1
1
1
1
1
1
1
0
0
1
0
0
1
1
0
1
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
Anexo B. Nombrar el anexo B de acuerdo con su contenido
Accesión
4
5
Accesión
174
187
189
201
202
209
217
300
301
302
303
304
305
308
310
311
312
313
314
320
322
340
341
342
344
345
346
347
348
349
350
351
113
ACA_1 ACA_2
ACA_3 ACA_4
ACA_5 ACA_6
ACA_7 ACA_8 ACA_9 ACA_10 ACA_11 ACA_12 ACA_13 ACA_14 ACA_15
0
0
0
0
0
0
0
0
0
1
0
0
1
0
0
0
0
1
1
1
1
1
1
1
1
1
1
1
0
0
ACA_16 ACA_17 ACA_18 ACA_19 AG_1 AG_2
AG_3 AG_4 AG_5
AG_6
AG_7
AG_8
AG_9
AG_10
AG_11
AG_12 AG_13
0
0
0
1
1
1
1
1
1
1
1
1
1
1
1
1
1
0
0
0
1
1
1
1
1
1
1
1
1
1
1
1
1
1
1
0
1
1
1
1
1
1
1
1
1
1
1
1
1
1
1
1
0
1
1
0
1
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
0
0
1
1
1
1
1
1
1
1
1
0
1
0
1
0
0
0
0
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
1
1
1
1
1
1
1
1
1
1
1
1
1
0
1
0
1
0
0
1
1
1
1
1
1
1
1
1
1
9
0
0
0
1
0
0
1
1
1
1
1
1
1
1
1
1
1
0
1
0
1
0
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
1
0
0
1
1
1
1
1
1
1
1
1
1
0
0
1
0
1
0
1
1
1
1
1
1
1
1
1
1
1
1
0
0
0
0
1
1
1
1
1
1
0
1
1
1
1
0
0
0
0
0
0
0
0
0
1
1
1
0
1
1
0
1
0
0
0
0
0
0
0
0
0
1
1
1
0
1
1
0
1
0
0
0
0
0
0
1
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
1
0
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
1
0
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
1
0
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
1
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
1
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
1
0
0
0
0
0
0
1
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
0
0
0
1
1
1
1
1
1
1
1
0
1
0
0
0
0
1
0
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
0
1
1
1
1
1
1
1
1
0
1
0
0
0
0
0
0
0
1
1
1
1
1
1
1
1
0
0
0
0
0
0
0
0
0
1
1
1
1
1
1
1
1
0
1
0
0
0
0
0
0
0
1
1
1
1
1
1
1
1
0
1
1
0
0
0
0
0
0
1
1
1
1
1
1
1
1
0
0
0
0
0
0
0
0
0
1
1
1
1
1
1
1
1
0
0
0
0
0
0
0
0
0
1
1
1
1
1
1
1
1
0
114
Título de la tesis o trabajo de investigación
Accesión
ACA_16 ACA_17 ACA_18 ACA_19 AG_1 AG_2
AG_3 AG_4 AG_5
AG_6
AG_7
AG_8
AG_9
AG_10
AG_11
AG_12 AG_13
353
1
1
0
0
0
0
0
0
1
1
1
1
1
1
1
1
0
354
1
0
0
0
0
0
0
0
0
0
0
0
1
1
0
1
0
356
0
0
0
0
0
0
0
0
1
1
1
1
1
1
1
1
0
357
1
1
0
1
0
0
0
0
0
1
1
1
1
1
1
1
0
358
1
0
1
0
0
0
0
0
0
1
1
1
1
1
1
1
0
359
1
0
0
0
0
0
0
0
0
1
1
1
1
1
1
1
0
360
1
0
0
0
0
0
0
0
0
1
1
1
1
1
1
1
0
362
1
0
1
0
0
0
1
1
0
1
1
1
1
1
1
1
0
363
1
0
1
0
0
0
0
1
0
1
1
1
1
1
1
1
0
364
1
0
1
0
1
1
1
1
0
1
1
1
1
1
1
1
0
365
1
0
0
0
1
1
1
1
1
1
1
1
1
1
1
1
0
366
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
367
1
0
0
0
0
0
0
1
1
1
1
1
1
1
1
0
0
369
1
0
0
0
0
0
0
1
1
1
1
1
1
1
1
1
0
370
1
0
0
0
0
0
0
0
0
0
1
1
1
0
1
1
0
373
1
0
0
0
0
0
0
1
1
1
1
1
1
1
1
1
0
374
1
0
1
0
0
0
0
1
1
1
1
1
1
1
1
1
0
375
1
0
1
0
0
0
0
1
1
1
1
1
1
1
1
1
0
377
0
0
0
0
0
0
0
1
1
1
1
1
1
1
0
0
0
378
1
0
1
0
0
0
0
1
1
1
1
1
1
1
1
0
0
379
0
0
1
0
0
0
0
1
0
1
1
1
1
1
1
1
0
1
0
0
1
0
0
0
0
1
0
1
1
1
1
1
1
1
0
2
0
0
1
0
0
1
0
1
1
1
1
1
1
1
1
1
0
3
1
0
1
0
0
0
0
0
0
1
1
1
1
1
1
1
0
4
0
0
0
0
1
0
1
1
1
1
1
1
1
1
1
1
0
5
1
0
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
Accesión
AG_14 CA_1
CA_2
CA_3
CA_4 CA_5
CA_6 CA_7 CA_8
CA_9
CA_10
CA_17
CA_18
CA_19
CGA_1 CGA_2 CGA_3 CGA_4
174
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
1
0
0
187
0
1
1
1
1
1
1
1
1
1
1
0
0
0
1
1
0
1
189
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
1
0
0
201
0
1
1
1
1
1
1
1
1
1
1
0
0
0
1
1
1
1
202
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
0
209
1
1
1
1
1
1
1
1
1
1
1
0
0
0
1
1
1
1
217
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
0
Anexo B. Nombrar el anexo B de acuerdo con su contenido
115
Accesión
AG_14 CA_1
CA_2
CA_3
CA_4 CA_5
CA_6 CA_7 CA_8
CA_9
CA_10
CA_17
CA_18
CA_19
CGA_1 CGA_2 CGA_3 CGA_4
301
1
1
1
1
1
1
1
1
1
1
1
0
0
0
1
1
0
1
302
1
1
1
1
1
1
1
1
1
1
1
0
0
0
1
0
0
0
303
1
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
0
304
1
1
1
1
1
1
1
1
1
1
1
0
0
0
1
1
0
0
305
0
1
1
1
1
1
1
1
1
1
1
0
0
0
1
0
0
0
308
1
1
1
1
1
1
1
1
1
1
1
0
0
0
1
1
0
0
310
0
1
1
1
1
1
1
1
1
1
1
0
0
0
1
0
0
0
311
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
0
312
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
0
313
0
1
1
1
1
1
1
1
1
1
1
0
0
0
1
0
0
0
314
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
0
320
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
1
0
0
322
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
0
340
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
0
341
0
0
0
0
0
0
0
0
0
0
0
1
1
1
0
1
1
1
342
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
1
0
1
344
1
1
1
1
1
1
1
1
1
1
1
0
0
0
0
1
1
1
345
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
0
346
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
1
1
347
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
0
348
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
0
349
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
1
350
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
1
351
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
1
1
352
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
1
0
0
353
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
1
354
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
1
1
356
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
1
357
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
0
358
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
0
359
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
1
0
0
360
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
0
362
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
0
363
1
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
0
364
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
1
0
0
116
Título de la tesis o trabajo de investigación
Accesión
AG_14 CA_1
CA_2
CA_3
CA_4 CA_5
CA_6 CA_7 CA_8
CA_9
CA_10
CA_17
CA_18
CA_19
CGA_1 CGA_2 CGA_3 CGA_4
366
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
0
367
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
0
369
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
0
370
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
1
373
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
0
374
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
375
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
377
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
378
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
379
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
1
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
2
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
3
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
4
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
5
0
1
1
1
1
1
1
1
1
1
1
0
0
0
0
0
0
Accesión
CGA_5 CGA_7 CGA_9 CCA_1
CCA_2 CCA_3
CCA_4 CCA_5 CCA_6 CCA_7
CCA_8
CCA_9
CCA_10 TG_1
TG_2
TG_3
TG_4
174
0
1
1
0
0
0
0
0
0
1
1
1
1
0
0
0
0
187
0
1
1
0
0
0
0
0
0
1
1
1
1
0
0
0
0
189
0
1
0
0
0
0
0
0
0
1
1
1
1
0
0
0
0
201
1
1
0
0
0
0
0
0
0
1
1
1
1
0
0
0
0
202
0
1
0
0
0
0
0
0
0
1
1
1
1
0
0
0
0
209
1
1
0
0
0
0
0
0
0
1
1
1
1
0
0
0
0
217
0
1
0
0
0
0
0
0
0
1
1
1
1
0
0
0
0
300
0
1
0
0
0
0
0
0
0
1
1
1
1
0
0
0
0
301
0
1
0
0
0
0
0
0
0
1
1
1
1
0
0
0
0
302
0
1
0
0
0
0
0
0
0
1
1
1
1
0
0
0
0
303
0
1
0
0
0
0
0
0
0
1
1
1
1
0
0
0
0
304
0
1
0
1
1
1
1
1
1
1
0
0
0
1
1
1
1
305
0
1
0
1
1
1
1
1
1
0
0
0
0
1
1
1
1
308
1
1
0
1
1
1
1
1
1
0
0
0
0
1
1
1
1
310
0
1
0
1
1
1
1
1
1
0
0
0
0
1
1
1
1
311
0
0
0
1
1
1
1
1
1
0
0
0
0
1
1
1
1
312
0
0
0
1
1
1
1
1
1
0
0
0
0
1
1
1
1
313
0
1
0
1
1
1
1
1
1
0
0
0
0
1
1
1
1
0
1
0
0
1
0
0
0
0
0
Anexo B. Nombrar el anexo B de acuerdo con su contenido
117
Accesión
CGA_5 CGA_7 CGA_9 CCA_1
CCA_2 CCA_3
CCA_4 CCA_5 CCA_6 CCA_7
CCA_8
CCA_9
CCA_10 TG_1
TG_2
TG_3
TG_4
320
1
1
0
1
0
0
0
1
1
0
1
1
0
1
0
0
0
322
0
1
0
1
1
1
1
1
1
0
0
0
0
1
1
1
1
340
0
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
341
1
1
0
1
1
1
1
1
1
0
0
0
0
1
1
1
1
342
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
344
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
345
0
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
346
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
347
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
348
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
349
0
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
350
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
351
352
353
354
356
357
358
359
360
362
363
364
365
366
367
369
370
373
374
375
377
378
1
0
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
1
1
1
1
1
1
0
1
1
1
0
1
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
1
1
1
1
1
1
1
1
1
1
1
1
1
1
0
1
1
1
1
1
1
1
0
0
1
1
1
1
1
0
1
0
1
0
1
0
1
1
1
1
1
1
1
1
0
1
1
1
1
1
1
0
0
1
1
0
1
0
1
1
1
1
1
1
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
118
Título de la tesis o trabajo de investigación
Accesión
1
2
3
4
5
CGA_5 CGA_7 CGA_9 CCA_1
CCA_2 CCA_3
CCA_4 CCA_5 CCA_6 CCA_7
CCA_8
CCA_9
CCA_10 TG_1
TG_2
TG_3
TG_4
0
0
0
0
0
0
0
0
0
1
1
1
0
0
0
0
0
0
0
0
1
1
1
1
0
1
0
0
0
0
1
1
1
1
0
1
0
0
0
0
0
0
0
0
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
1
0
0
0
0
0
0
0
0
1
0
0
0
0
0
0
0
1
1
1
0
0
0
0
0
Accesión
TG_5
TG_6
TG_7
TG_9
TG_10 TG_11
NN_1 NN_2 NN_3 NN_4
NN_5
NN_6
NN_7
NN_8
NN_9
NN_10 NN_11 NN_12 NN_13
174
0
0
1
1
1
1
1
1
1
1
1
1
1
1
1
1
1
1
1
187
0
0
1
1
1
1
1
1
1
1
1
1
1
1
1
1
1
1
1
189
0
0
1
1
1
1
1
1
1
1
1
1
1
1
1
1
1
1
1
201
0
0
1
1
1
1
1
1
1
1
1
1
1
1
1
1
1
1
1
202
0
0
1
1
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
209
0
0
1
1
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
217
0
0
1
1
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
300
0
0
1
1
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
301
0
0
1
1
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
302
0
0
1
1
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
303
0
0
1
1
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
304
1
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
Accesión
TG_5
TG_6
TG_7
TG_9
TG_10 TG_11
GT_1 GT_2 GT_3
GT_4
GT_5
GT_6
GT_7
GT_8
GT_9
GT_10 GT_11 GT_12 GT_13
305
1
1
0
0
0
0
1
1
1
1
1
1
1
1
1
1
1
1
1
308
1
1
0
0
0
0
1
1
1
1
1
1
1
1
1
1
1
1
1
310
1
1
0
0
0
0
1
1
1
1
1
1
1
1
1
1
1
1
1
311
1
1
0
0
0
0
1
1
1
1
1
1
1
1
1
1
1
1
1
312
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
313
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
314
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
320
1
1
0
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
322
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
340
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
341
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
342
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
344
0
0
0
0
0
0
1
1
1
1
1
1
1
1
1
1
1
1
1
345
0
0
0
0
0
0
1
1
1
1
1
1
1
1
1
1
1
1
1
346
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
Anexo B. Nombrar el anexo B de acuerdo con su contenido
119
Accesión
TG_5
TG_6
TG_7
TG_9
TG_10 TG_11
GT_1 GT_2 GT_3
GT_4
GT_5
GT_6
GT_7
GT_8
GT_9
GT_10 GT_11 GT_12 GT_13
348
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
349
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
350
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
351
1
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
352
0
0
1
0
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
353
0
0
1
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
354
0
0
1
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
356
0
0
1
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
357
0
0
1
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
358
0
0
1
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
359
0
0
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
360
0
0
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
362
0
0
1
0
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
363
0
0
1
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
364
0
0
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
365
0
0
1
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
366
0
0
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
367
0
0
0
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
369
0
0
1
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
370
0
0
1
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
373
0
0
1
1
1
0
1
1
1
1
1
1
1
1
1
1
1
1
1
374
0
0
1
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
375
0
0
1
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
377
0
0
1
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
378
0
0
1
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
379
0
0
1
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
Accesión
TG_5
TG_6
TG_7
TG_9
TG_10 TG_11
GT_1 GT_2 GT_3
GT_4
GT_5
GT_6
GT_7
GT_8
GT_9
GT_10 GT_11 GT_12 GT_13
1
0
0
1
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
2
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
3
0
0
0
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
4
0
0
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
5
0
1
1
1
0
0
0
0
0
0
0
0
0
0
0
0
0
0
0
120
Título de la tesis o trabajo de investigación
E. Anexo E. matriz de distancias geográficas para a) región
Chicamocha, b) Región Sumapaz
1
CHICAMOCHA
1
0,000
0,444
0,545
0,036
0,033
0,201
0,539
1,291
0,544
0,544
0,543
0,476
0,475
0,471
0,466
0,416
0,392
0,006
0,019
0,244
0,034
0,037
0,034
23
Geographic
Distancie
D
2
0,000
0,124
0,409
0,410
0,273
0,221
0,909
0,123
0,125
0,114
0,033
0,035
0,028
0,023
0,029
0,052
0,439
0,425
0,637
0,411
0,407
0,409
3
0,000
0,509
0,512
0,356
0,143
0,785
0,002
0,003
0,018
0,107
0,115
0,109
0,111
0,143
0,162
0,540
0,527
0,753
0,511
0,509
0,511
4
0,000
0,008
0,166
0,505
1,256
0,509
0,509
0,508
0,442
0,440
0,436
0,432
0,381
0,357
0,030
0,020
0,274
0,002
0,009
0,009
5
0,000
0,171
0,509
1,261
0,511
0,511
0,510
0,443
0,441
0,437
0,433
0,382
0,359
0,030
0,015
0,268
0,009
0,003
0,001
6
0,000
0,339
1,090
0,355
0,355
0,357
0,306
0,307
0,301
0,296
0,244
0,221
0,195
0,186
0,437
0,168
0,169
0,171
7
0,000
0,751
0,145
0,140
0,160
0,223
0,232
0,222
0,221
0,223
0,229
0,534
0,524
0,771
0,506
0,507
0,509
8
0,000
0,787
0,784
0,795
0,888
0,894
0,891
0,894
0,928
0,944
1,285
1,275
1,521
1,257
1,258
1,260
9
0,000
0,005
0,016
0,105
0,113
0,107
0,109
0,142
0,161
0,539
0,526
0,752
0,510
0,508
0,510
10
0,000
0,020
0,109
0,116
0,111
0,112
0,144
0,162
0,539
0,526
0,753
0,511
0,508
0,511
11
0,000
0,094
0,101
0,097
0,099
0,136
0,156
0,538
0,525
0,748
0,510
0,507
0,510
12
0,000
0,009
0,006
0,010
0,062
0,086
0,472
0,457
0,667
0,443
0,439
0,442
13
0,000
0,011
0,014
0,063
0,087
0,471
0,456
0,663
0,442
0,438
0,441
14
0,000
0,004
0,057
0,080
0,466
0,452
0,662
0,438
0,434
0,436
15
0,000
0,052
0,076
0,462
0,448
0,658
0,434
0,430
0,432
16
0,000
0,024
0,411
0,397
0,612
0,383
0,379
0,381
17
0,000
0,388
0,374
0,592
0,359
0,356
0,358
18
0,000
0,017
0,251
0,029
0,033
0,030
19
0,000
0,255
0,019
0,018
0,016
20
0,000
0,274
0,270
0,268
21
22
23
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
0,000
21
0,011 0,000
22
0,010 0,003 0,000 23
Anexo B. Nombrar el anexo B de acuerdo con su contenido
121
E. Anexo E: (continuación)
1
35
Geographic
SUMAPA
Distancie
Z
D(2)
1
2
3
0,000
0,00
0,008
0
0,00
0,006
5
0,000
0,01
0,023
6
0,017
0,01
0,023
6
0,017
0,01
0,024
9
0,018
0,04
0,048
8
0,044
0,09
0,089
6
0,095
0,09
0,091
8
0,096
0,07
0,065
2
0,070
0,06
0,057
4
0,059
0,12
0,121
6
0,121
0,09
0,083
0
0,086
0,11
0,104
0
0,111
0,33
0,344
7
0,338
0,33
0,344
7
0,338
0,33
0,344
7
0,338
0,35
0,363
6
0,356
0,37
0,384
9
0,378
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
0,00
0
0,00
3
0,00
8
0,04
5
0,11
2
0,11
3
0,08
7
0,07
4
0,13
2
0,10
3
0,12
6
0,32
2
0,32
1
0,32
2
0,34
1
0,36
4
0,00
0
0,00
5
0,04
2
0,11
2
0,11
3
0,08
6
0,07
2
0,12
9
0,10
1
0,12
7
0,32
2
0,32
1
0,32
1
0,34
0
0,36
2
0,00
0
0,03
7
0,11
3
0,11
3
0,08
6
0,07
0
0,12
6
0,10
0
0,12
9
0,32
1
0,32
0
0,32
0
0,33
8
0,36
0
0,00
0
0,11
6
0,11
4
0,08
4
0,05
5
0,09
4
0,08
8
0,14
2
0,32
5
0,32
4
0,32
5
0,33
5
0,35
1
0,00
0
0,01
0
0,03
4
0,06
9
0,11
7
0,04
9
0,03
9
0,43
3
0,43
3
0,43
3
0,44
9
0,46
7
0,00
0
0,03
0
0,06
4
0,10
8
0,04
0
0,04
8
0,43
4
0,43
3
0,43
4
0,44
8
0,46
5
0,00
0
0,03
5
0,09
1
0,02
6
0,06
8
0,40
5
0,40
4
0,40
5
0,41
8
0,43
5
0,00
0
0,06
7
0,03
3
0,10
2
0,37
9
0,37
9
0,37
9
0,38
9
0,40
3
0,00
0
0,06
9
0,15
6
0,39
9
0,39
9
0,40
0
0,39
7
0,40
3
0,00
0
0,08
7
0,41
3
0,41
2
0,41
3
0,42
2
0,43
5
0,00
0
0,44
6
0,44
5
0,44
6
0,46
7
0,48
9
0,00
0
0,00
0
0,00
3
0,06
9
0,12
3
0,00
0
0,00
3
0,06
9
0,12
3
0,00
0
0,07
1
0,12
5
0,00
0
0,05
4
0,00
0
20
21
22
23
122
Título de la tesis o trabajo de investigación
1
35
SUMAPA
Geographic
Z
D(2)
Distancie
1
2
3
0,224
0,216
0,213
0,192
0,139
0,149
0,166
0,214
0,229
0,234
0,024
0,033
0,062
0,087
0,21
9
0,20
9
0,20
6
0,18
5
0,13
4
0,14
4
0,16
1
0,20
9
0,22
5
0,22
9
0,01
9
0,02
7
0,06
0
0,09
5
0,218
0,209
0,207
0,186
0,133
0,143
0,160
0,207
0,223
0,228
0,018
0,026
0,057
0,093
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
0,20
4
0,19
4
0,19
1
0,16
9
0,11
9
0,13
0
0,14
7
0,19
5
0,21
1
0,21
6
0,00
8
0,01
5
0,05
3
0,11
0
0,20
2
0,19
3
0,19
0
0,16
9
0,11
7
0,12
8
0,14
5
0,19
3
0,20
9
0,21
4
0,00
5
0,01
3
0,04
9
0,11
0
0,20
0
0,19
1
0,18
9
0,16
9
0,11
5
0,12
5
0,14
2
0,19
0
0,20
6
0,21
1
0,00
0
0,00
9
0,04
5
0,11
1
0,19
4
0,19
1
0,19
6
0,17
9
0,11
0
0,11
8
0,13
2
0,17
7
0,19
1
0,19
5
0,03
7
0,03
3
0,01
8
0,11
5
0,30
9
0,30
3
0,30
2
0,28
1
0,22
4
0,23
3
0,24
8
0,29
3
0,30
7
0,31
1
0,11
3
0,11
9
0,13
5
0,00
2
0,30
8
0,30
3
0,30
3
0,28
2
0,22
3
0,23
1
0,24
6
0,29
0
0,30
4
0,30
8
0,11
3
0,12
0
0,13
2
0,01
0
0,27
8
0,27
3
0,27
4
0,25
4
0,19
3
0,20
1
0,21
6
0,26
0
0,27
4
0,27
8
0,08
6
0,09
2
0,10
2
0,03
3
0,24
8
0,24
6
0,25
0
0,23
2
0,16
5
0,17
2
0,18
6
0,22
8
0,24
1
0,24
5
0,07
0
0,07
3
0,07
2
0,06
7
0,26
0
0,26
6
0,27
7
0,26
5
0,18
5
0,18
9
0,19
8
0,23
0
0,23
9
0,24
2
0,12
6
0,12
5
0,10
3
0,11
7
0,28
1
0,27
9
0,28
3
0,26
5
0,19
8
0,20
5
0,21
9
0,26
0
0,27
3
0,27
6
0,10
0
0,10
4
0,10
5
0,04
8
0,32
9
0,31
9
0,31
5
0,29
2
0,24
4
0,25
4
0,27
0
0,31
7
0,33
2
0,33
6
0,12
9
0,13
7
0,16
0
0,03
9
0,14
2
0,13
4
0,13
1
0,15
5
0,21
6
0,21
1
0,20
2
0,18
0
0,18
0
0,18
1
0,32
1
0,31
4
0,30
8
0,43
1
0,14
2
0,13
4
0,13
1
0,15
4
0,21
6
0,21
0
0,20
2
0,18
0
0,18
0
0,18
1
0,32
0
0,31
3
0,30
7
0,43
1
0,14
3
0,13
5
0,13
1
0,15
4
0,21
6
0,21
1
0,20
3
0,18
1
0,18
2
0,18
3
0,32
0
0,31
4
0,30
8
0,43
1
0,14
0
0,14
8
0,15
9
0,18
7
0,22
5
0,21
7
0,20
3
0,16
7
0,16
1
0,15
9
0,33
8
0,33
1
0,31
7
0,44
7
0,16
0
0,17
7
0,19
5
0,22
3
0,24
5
0,23
5
0,21
9
0,17
5
0,16
4
0,16
1
0,36
0
0,35
2
0,33
3
0,46
6
0,04
8
0,07
8
0,10
7
0,13
1
0,12
4
0,11
3
0,09
6
0,05
0
0,04
1
0,03
9
0,23
8
0,23
0
0,20
8
0,34
1
0,08
8
0,10
4
0,12
5
0,15
3
0,17
3
0,16
3
0,14
8
0,10
8
0,10
0
0,09
9
0,28
8
0,27
9
0,26
2
0,39
4
0,00
0
0,03
1
0,06
2
0,08
4
0,08
5
0,07
6
0,06
3
0,04
2
0,05
1
0,05
5
0,20
0
0,19
2
0,17
6
0,30
7
0,00
0
0,03
1
0,05
3
0,08
2
0,07
7
0,07
0
0,06
9
0,08
1
0,08
6
0,19
1
0,18
4
0,17
4
0,30
1
Anexo B. Nombrar el anexo B de acuerdo con su contenido
1
SUMAPA
Geographic
Z
D(2)
Distancie
1
2
3
0,000
0,028
0,092
0,090
0,090
0,099
0,112
0,116
0,189
0,183
0,180
0,300
0,000
0,084
0,087
0,092
0,114
0,129
0,134
0,169
0,163
0,164
0,279
0,000
0,012
0,030
0,079
0,096
0,101
0,115
0,107
0,092
0,222
0,000
0,018
0,067
0,084
0,089
0,125
0,117
0,100
0,231
0,000
0,049
0,066
0,071
0,142
0,134
0,114
0,247
4
0,000
0,018
0,023
0,190
0,181
0,158
0,291
5
0,000
0,006
0,206
0,197
0,173
0,305
6
0,000
0,211
0,202
0,177
0,310
9
123
7
8
10
11
0,000
0,009
0,045
0,111
20
21
22
23
24
25
26
27
28
29
30
31
32
0,000
33
0,038 0,000
34
0,118 0,133 0,000 35
12
13
14
15
16
17
18
19
20
21
22
23
124
Título de la tesis o trabajo de investigación
F. Anexo F: matriz de distancias genéticas para a) región
Chicamocha, b) región Sumapaz
1 23
CHICAMOCHA
1
0
3
6
12
25
32
22
37
36
37
38
35
32
48
38
33
39
22
37
37
38
35
33
D
2
0
7
9
26
29
23
40
37
40
41
38
35
51
41
36
40
25
40
38
41
38
34
1
23
3
Pop1
4
Binary
Distancie
0
6
25
26
22
37
34
35
34
33
32
48
38
33
39
22
35
35
40
33
33
0
27
20
26
39
36
37
36
37
36
50
40
35
39
24
37
35
42
35
33
1 23
5
0
9
3
20
15
20
19
24
19
27
17
14
18
29
16
18
15
16
14
6
0
12
25
22
25
20
27
26
34
24
21
23
36
21
21
24
21
19
7
0
23
18
21
22
23
18
30
20
17
21
32
19
21
18
19
17
8
0
7
8
7
6
9
11
9
6
12
21
8
8
17
10
12
9 10 11 12 13 14 15 16 17 18 19 20 21 22 23
0
9
10
11
10
14
10
9
7
24
9
9
18
9
7
0
7
6
7
15
9
8
10
23
6
6
15
8
8
0
9
8
16
8
5
13
20
5
5
16
7
9
0
5
15
11
10
16
25
8
8
19
12
12
0
16
6
5
13
20
5
5
14
9
11
0
12
15
13
26
15
15
18
15
19
0
5
11
18
5
5
10
7
11
0
12
17
4
4
11
6
10
0
25
12
10
17
12
8
0
19 0
19 2 0
26 11 13 0
19 6 6 15
25 8 6 15
0
8
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
0 23
Anexo B. Nombrar el anexo B de acuerdo con su contenido
Binary
Distancie Pop1
2
3
4
125
SUMAPAZ D(2)
1
0
6
3
6
18
38
40
42
45
29
28
26
24
32
27
46
24
37
35
23
19
18
19
20
26
21
22
27
22
22
22
21
17
21
27
0
7
4
16
40
38
42
45
29
26
26
20
30
27
42
20
35
37
23
19
20
19
18
24
19
24
27
20
24
24
21
21
19
31
0
5
17
37
39
41
42
28
27
27
23
29
28
47
25
40
38
26
22
21
22
23
29
22
21
28
23
23
19
18
16
20
26
0
16
40
40
40
41
27
26
24
20
26
25
42
22
37
35
23
17
18
19
18
24
17
22
27
20
22
20
17
17
17
29
5
0
34
34
32
31
23
20
18
22
20
33
34
30
41
43
31
25
26
27
28
22
23
32
37
32
32
28
23
25
25
35
6
0
10
16
13
25
24
28
34
28
43
44
40
31
27
43
41
38
39
40
36
45
46
47
42
42
44
43
39
33
45
7
0
14
21
31
30
34
36
36
41
48
34
27
27
41
39
38
39
38
32
41
44
43
40
42
44
43
39
37
43
8
0
13
25
28
28
34
28
35
48
40
29
25
41
35
34
35
36
34
37
44
45
44
44
42
39
35
39
43
9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35
0
22
25
23
29
23
42
41
41
30
28
44
38
37
40
37
35
38
43
48
41
43
41
36
38
34
48
0
7
9
15
9
24
29
23
44
40
28
22
21
24
21
19
26
29
32
25
27
25
22
22
18
32
0
6
12
6
21
24
18
41
37
25
21
22
23
18
16
29
34
35
22
28
26
25
25
15
35
0
10
8
19
26
18
37
35
23
17
16
17
16
14
23
30
31
22
24
28
23
25
19
33
0
14
23
30
18
35
39
25
21
20
21
20
22
23
28
29
22
24
26
21
23
19
31
0
21
24
22
43
39
27
23
24
25
20
18
29
36
39
26
32
28
27
27
17
39
0
41
15
38
32
20
16
17
18
13
23
24
31
22
19
23
23
24
22
18
20
0
30
47
51
33
37
38
37
32
26
39
44
45
36
44
44
43
41
33
55
0
25
25
9
9
10
9
6
14
17
18
19
10
16
20
21
19
13
29
0
10
22
26
25
24
27
33
32
35
34
33
33
39
40
36
38
44
0
18
22
21
22
23
33
26
31
34
27
29
29
34
30
32
38
0
10
9
8
9
15
14
15
16
13
17
17
22
18
20
28
0
3
6
5
15
12
17
20
13
13
19
18
18
14
28
0
3
6
16
11
16
17
14
10
18
17
15
15
25
0
7
17
14
15
18
15
11
21
20
16
18
26
0
10
13
20
21
8
16
18
19
17
11
29
0
19
24
23
16
24
24
25
25
19
35
0
11
16
13
15
11
10
14
20
22
0
11
14
12
12
13
15
25
21
0
15
13
15
14
18
26
14
0
10
12
13
17
11
21
0
14 0
13 5 0
15 7 8 0
17 19 18 22 0
19 15 14 14 28
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
0 35
126
Título de la tesis o trabajo de investigación
G. Anexo G: protocolo usado para
cualificación de metabolitos secundarios.
Laboratorio
fitoquímica
Universidad
Nacional-Palmira
Protocolo Laboratorio de Fitoquímica de la Universidad Nacional de Colombia-Sede
Palmira. Pruebas duales con etanol y cloroformo como solventes, para valorar la
presencia (1)-ausencia (0) de siete metabolitos secundarios [1. derivados de cumarinas,
2. esteroides y terpenoides, 3. flavonoides, 4. glicósidos cardiotónicos, 5. saponinas, 6.
heterósidos y 7. taninos].
Lavar el material vegetal,
En mortero, macere ˜20 gramos de tejido en 20 ml de etanol,
Filtre en embudo (de vidrio) tapado con lana de vidrio y recoja el filtrado en un
Erlenmeyer. Deje escurrir bien,
Al residuo agregue 20 ml de cloroformo y macere bien,
Filtre y recoja el filtrado en embudo de separación. Agregue 10 ml de agua destilada.
Agite y deje reposar. Si no hay buena separación agregue ˜5 ml de solución saturada de
cloruro de sodio. Agite y deje reposar,
Separe la capa (de cloroformo) inferior en un Erlenmeyer y séquela con sulfato de sodio
anhidro,
Decante la solución,
Guarde la fracción etanólica y la clorofórmica para las pruebas de:
Alcaloides. En un tubo de ensayo vierta 1 ml de solución etanólica y otro, 1 ml de
solución clorofórmica. A cada tubo agregue 1 ml de HCl al 5%. Si se forma precipitado,
filtre. Agregue a cada fracción 2-3 gotas de: a) Borchardat (precipitado), b) Dragendorff
(precipitado), Wagner (precipitado). En cada caso la presencia de precipitado indica
REACCIÓN POSITIVA.
Derivados de Coumarinas. A 0.2 ml de solución etanólica-SE y 0.2 ml de solución
clorofórmica-SC, agregue a cada una 3 gotas de éter etílico y agite. En un papel filtro
aplique 2 gotas separadas de la solución etérea etanólica y dos gotas de la solución
etérea clorofórmica, de manera que forme 4 manchas de 1 cm de diámetro aprox. sin
que se toquen entre sí. A una mancha de cada solución, agregue 1 gota de NaOH 1N.
Deje secar. Exponga las manchas a la luz ultravioleta. La aparición de fluorescencia en la
mancha indica REACCIÓN POSITIVA.
128
Título de la tesis o trabajo de investigación
Esteroide y triterpenoides. En beaker de 50 ml agregue 0.3 ml de extracto etanólico.
Evapore el solvente (en baño maría). Deje enfriar. Agregue 0.5 ml de CHHCl3. Agite.
Vierta el contenido en un vidrio de reloj que contraste con el fondo blanco. Agregue 1-2
gotas de anhídrido acético y 1-2 gotas de H2SO4 concentrado. La aparición de colores
verdes, azules, púrpura, rojo, indican REACCIÓN POSITIVA. Tome 0.2 ml de extracto
clorofórmico inicial, vierta en un vidrio de reloj, y siga el procedimiento anterior.
Flavonoides. Evapore 0.5 ml de extracto etanólico y 0.5 de extracto clorofórmico. el
residuo disuélvalo en 1 ml de metanol. Adicione 2 gotas de HCl concentrado y un trocito
de cinta de Mg. Deje hasta que se consuma todo el Mg. El surgimiento de una coloración
rosada, naranja, roja azulosa, violeta, verde, azul; indican PRUEBA POSITIVA.
Glicósidos cardiotónicos. Evapore por separado en un beaker de 50 ml del extracto
etanólico y 0.5 ml del extracto clorofórmico. A los residuos agregue 1 ml de metanol.
Separe cada solución en dos porciones y realice las siguientes pruebas:
A. Reactivo de Kedde. Agregue 2-3 gotas de este reactivo a dos de las soluciones.
Un color azul violeta, indica REACCIÓN POSITIVA.
B. A las soluciones restantes, adicione 3 gotas de una solución recién preparada de
nitroprusiato de sodio al 5% y 3 gotas de NaOH 2N. La coloración violeta intensa
indica REACCIÓN POSITIVA.
Saponina espumídica. En un tubo de ensayo, disuelva 1 ml de extracto etanólico en 15
ml de agua destilada. Agitar vigorosamente por unos minutos. Si la fase espumosa
permanece estable por las de media hora, el resultado se considera POSITIVO.
Heterósidos cianogénicos. En un Erlenmeyer de 125 ml coloque 10 gramos de material
vegetal (fresco) picado, agregue 10 ml de agua destilada y 1 ml de H2SO4 concentrado.
Tapar con u tapón de corcho en el cual está suspendida una tira de papel reactivo de
picrato de sodio, de modo que no quede en contacto con las paredes del fresco ni con el
material. Calentar a baño maría por 15 minutos. Si el papel toma color marrón rojizo,
indica PRUEBA POSITIVA de HCN.
Taninos. A 0.3 ml de cada una de las soluciones a analizar se agregan dos gotas de
solución de cloruro férrico al 2%. Si hay cambio de coloración o aparición de un
precipitado, la prueba es POSITIVA.
Bibliografía
A
Altieri M A, C I Nicholls. 2007. Conversión agroecológica de sistemas convencionales de
producción: teoría, estratégias y evaluación. Ecosistemas 16 (1). Enero 2007.
http://www. revistaecosistemas. net/articulo. asp?Id=457.
Álvarez V. 2008. Caracterización química y evaluación de la actividad inhibitoria de aceite
esencial de un quimiotipo de Lippia Origanoides H.B.K sobre el desarrollo micelial
y la formación de esclerocios de Sclerotium cepivorum BERK. Trabajo de grado
Ingeniería Agronómica, Universidad Nacional De Colombia - Sede Palmira.
Antolinez-Delgado C. A, N. Rodríguez-López. 2008. PLASTICIDAD FENOTÍPICA EN
Lippia alba Y Lippia origanoides (VERBENACEAE): RESPUESTA A LA
DISPONIBILIDAD DE NITRÓGENO. Acta biol. Colomb., Vol. 13 No. 1, 2008 53 –
64.
Antolinez-Delgado C A, N Rodríguez-López. 2008. Plasticidad fenotípica en Lippia alba y
Lippia origanoides (Verbenaceae): respuesta a la disponibilidad de nitrógeno. Acta
biol. Colomb., Vol. 13 No. 1, 2008 53 – 64.
ArangoA
Altieri-Caro S. 2004. EthnobotanA
Altieriical studies in the Central Andes (Colombia): Knowledge distribution of plants use
according to informant´s characteristics. Lyona. Vol 7(2).
Arcos, A. L.; Mondragón, A. J.; Muñoz, J. E., Botero, S. 2004. Colecta y caracterización
molecular con marcadores tipo RAM (Microsatélites aleatorios) de heliconias y
especies relacionadas, p 346-347. En: Congreso Colombiano de Botánica:
Botánica, Diversidad y Cultura, 2, Popayán, Noviembre. Memorias.
Assogbadjo A. E, T. Kyndt, B. Sinsin, G. Gheysen, Van Damme P. 2006. Patterns of
Genetic and Morphometric Diversity in Baobab (Adansonia digitata) Populations
across Different Climatic Zones of Benin (West Africa) Annals of Botany 2006
97(5):819-830.
B
Baldizán A, C Domínguez, DE García, E. Chacón, L Aguilar. Metabolitos secundarios y
patrón de selección de dietas en el bosque deciduo tropical de los llanos centrales
venezolanos. Rev Zoot Trop, Vol 24, No 3, 2006, pp 213-232.
Beebe S, J Rengifo, E Gaitán, M C Duque, J Tohme. J. 2001. Diversity and Origin of
Andean Landraces of Common Bean Crop Sci. 41:854-862.
Berier D E, J C Thomas, S R King s.f. Haman Pharmaceuticals: Integrating Indigenous
Knowledge, Tropical Medicinal Plants, Medicine, Modern Science and Reciprocity
into a Novel Drug Discovery Approach. Shaman Pharmaceuticals, Inc. 213 East
Grand Ave. South San Francisco, CA 94080. En: http://www.nestsci.org/
Science/Special/ feature11.html.
Blanco KM, A.J. Agudelo, J.R. Martínez, E. E. Stashenko, Estudio comparativo de los
aceites esenciales de Lippia alba Mill N.E. Brown cultivada con tres tipos de
compostaje, Scientia et Technica, 2007, año XIII, 33, 231-233.
130
Título de la tesis o trabajo de investigación
Bonilla M. L.; Espinosa K.; Posso A. M.; Vásquez, H.; Muñoz, J. E. 2008. Caracterización
molecular de 43 accesiones de uchuva de seis departamentos de Colombia. Acta
Agron (Palmira) 57(2): 109-115.
Boschker H T S, J J Middelberg. 2002. Stable isotopes and biomarkers in microbial
ecology. FEMS Microbiology Ecology 40 (2002) 85-95.
Bozo L, M Fernández, M López, R Reyes y P Suárez. 2007. Biomarcadores de
contaminación química en comunidades microbianas. Interciencia Jan 2007, vol.
32 nº 1.
Bueno J.G, E. Stashenko, Antimycobacterial natural products an opportunity for the
Colombia biodiversity, Revista Española de Quimioterapia, 2009, Sometido.
Bueno J.G., J.R. Martínez, E. Stashenko, W. Ribón, Anti-tubercular activity of some
aromatic and medicinal plants, grown in Colombia, Biomédica, 2009, 29 (1), 5160.
C
Camargo A.A., N. Rodríguez López. Expresión fenotípica de la asignación de biomasa en
Lippia origanoides y Lippia alba: Repuestas a la disponibilidad de agua en el
suelo, Acta Biológica Colombiana, 2008, 13 (3), 133-148.
Caetano C M, D Y Cañar, E Muñoz (Eds). 2011. 10 AÑOS DE LA MAESTRÍA EN
CIENCIAS BIOLÓGICAS, LÍNEA DE
INVESTIGACIÓN EN RECURSOS
FITOGENÉTICOS NEOTROPICALES. CONTRIBUCIÓN AL ESTUDIO DE LA
BIOLOGIA FLORAL Y REPRODUCCIÓN SEXUAL DE “PRONTO ALIVIO”, Lippia
alba (Mill). N. E. Browne ex Britton & Wilson. Verbenaceae. ISBN “Coordinadores
editoriales. MEMORIAS. En: (En: http://www.bdigital.unal.edu. co/ 4959/ 1/
memoria10a%C3%B1os UNmaestriarfnpalmira.2011.pdf.)
Camacho A M, D A Andrade, S M Ledesma. 2010. Evaluación del efecto antifúngico del
empleo de aceite esencial y extractos de Lippia alba en sus quimiotipos (Valle,
Costa Rica, Cítrica) y Tomillo (Thymus vulgaris) sobre Stromatinia cepivora (Berk)
Wetzel en condiciones del Valle del Cauca Colombia. Trabajo de grado Ingeniería
Agroindustrial, Universidad Nacional De Colombia - Sede Palmira.
Cardona J. 2008. Evaluación de la actividad antifúngica de extractos orgánicos de Lippia
alba y Lippia dulcis Colombia. Trabajo de grado Ingeniería Agronómica,
Universidad Nacional de Colombia Sede Palmira.
Cardona J. 2009. Evaluación in vitro de la actividad inhibitoria Stromatinia cepivorum
Berk de extractos orgánicos de Lippia alba, Lippia origanoides y Phyla dulcis.
Ingeniería Agronómica, Universidad Nacional de Colombia Sede Palmira.
Castro, D.M.; Ming, L.C.; Marques, M.O.M. (2002). Biomass production and chemical
composition of Lippia alba (Mill.) N. E. Brown Britt & Wilson in leaves on different
plant parts in different seasons. Acta Hort. (ISHS) 569, 111-115.
Celis CN, P. Escobar, J.H. Isaza Martínez, E. Stashenko, J.R. Martínez, Estudio
comparativo de la composición química y la actividad biológica de los aceites
esenciales de Lippia alba, Lippia origanoides y Phyla dulcis, especies de la familia
Verbenaceae, Scientia et Technica, 2007, año XIII, 33,103-105.
Cicció J F, R A Ocampo. 2006. Variación anual de la composición química del aceite
esencial de Lippia alba (Verbenaceae). Lankesteriana 6(3). P149-152 (2006).
Combrinck S, G. W Du Plooy, R. I. McCrindle, B. M. Botha. 2007. Morphology and
Histochemistry of the Glandular Trichomes of Lippia scaberrima (Verbenaceae).
Ann Bot (Lond). 2007 Apr 27; 17468110 (P,S,E,B,D).
Bibliografía
131
Culley T.M., Wolfe A.D. 2001. Population genetic structure of the cleistogamous plant
species Viola pubescens Aiton (Violaceae), as indicated by Allozyme and ISSR
molecular markers. Heredity 86:545-556.
D
Das T, A.K. Das. 2005. Inventorying plant biodiversity in hmegardens: a case study in
Barack Valley, ssam, North East India. Current Science 89, 1, 155-163.
del Rio, AH, and JB Bamberg. 2006. Genetic equivalence of putative duplicate
germplasm collections held at CIP and US potato genebanks. Am J. Potato Res
83:279-285.
Dias-Brandão A, L F Viccini, S M Recco-Pimentel. 2005. Meiotic analysis of two putative
polyploid species of Verbenaceae from Brazil. Caryologia 58, 315-9. Contact:
Recco-Pimentel, Shirlei Maria; Univ Estadual Campinas, UNICAMP, Inst Biol,
Dept Biol Celular, CP 6109, BR-13083863 Campinas, SP, Brazil.
Dias-Brandão A, L F Viccini, F R. Gonçalves-Salimena, A L Laforga-Vanzela and S M
Recco-Pimentel. 2007. Cytogenetic characterization of Lippia alba and Lantana
camara (Verbenaceae) from Brazil. Journal of Plant Research. Volume 120,
Number 2 / marzo de 2007.
Doran J W y Parkin B T 1994. Defining Soil Quality for a Sustainable Environment. Soil
Science Society of America, Inc. Special Publication. Number 35. Madison,
Wisconsin, USA.
Dray S. 2011. A New Perspective about Moran’s Coefficient: Spatial Autocorrelations as a
Linear Regression Problem. Geographical Analysis. Vol. 43, Issue 2, Pages 127141.April 2011.
Duque-Villegas A. 1985. ENCUESTA NACIONAL DE PLANTAS MEDICINALES Y
AROMÁTICAS UNA APROXIMACIÓN AL MERCADO DE LAS PMyA EN
COLOMBIA 1. Instituto de Investigación de Recursos Biológicos Alexander von
Humboldt.
Duran D C, L A Monsalve, J R Martínez, E E Stashenko. 2007. ESTUDIO
COMPARATIVO DE LA COMPOSICIÓN QUÍMICA DE ACEITES ESENCIALES
DE Lippia alba PROVENIENTES DE DIFERENTES REGIONES DE COLOMBIA,
Y EFECTO DEL TIEMPO DE DESTILACIÓN SOBRE LA COMPOSICIÓN DEL
ACEITE. Scientia et Technica Año XIII, No 33, Mayo de 2007. UTP. ISSN 01221701 435.
E
El-Mouei R, W Choumane, F Dway, 2011. Molecular characterization and genetic
diversity in genus Citrus in Syria. Int. J. Agric. Biol., 13: 351–356
Erazo L A. 2010. Evaluación in vivo de la actividad inhibitoria sobre Sclerotium
cepivorum, por medio del aceite esencial de Lippia origanoides, en el cultivo de la
cebolla (Alium cepa L) Trabajo de grado Ingeniería Agronómica, Universidad
Nacional De Colombia - Sede Palmira.
Ernst W H O, P J Peterson. 1994. The role of biomarkers in environmental assessment
.4. terrestrial plants. Ecotoxicology 3, 180-192.
Ernst W H O. 1999. Biomarkers in plants. In: Peakall, D.B., Walker, C.H., Migula, P.
(eds.), Biomarkers: A Pragmatic Basis for Remediation of Severe Pollution in
Eastern Europe. Kluwer Academic Publishers, Dordrecht, pp. 135-151.
Escobar P; S M Leal, LV Herrera; J R Martínez; E Stashenko. Chemical composition and
antiprotozoal activities of Colombian Lippia spp essential oils and their major
components. Mem. Inst. Oswaldo Cruz, 2010, 105, 184-190.
132
Título de la tesis o trabajo de investigación
Espinosa G. 2009. Diseño y aplicación de una metodología de evaluación de riesgo
ecológico para fauna terrestre. Investigación ambiental. Ciencia y política pública.
Toxicología Ambiental, Universidad Autónoma de San Luis Potosí. En:
http://www.revista. ne.gob.mx/thesis/view/1.
F
Figueiredo A C, J G Barroso, L G Pedro, J J C Scheffer. 2008. Factors affecting
secondary metabolite production in plants: volatile components and essential oils.
Flavour and Fragrance Journal, Volume 23 Issue 4, Pages 213–226.
Fischer U, R Lopez, E Poell, S Vetter, J Novak, C M Franz. 2004. Two chemotypes within
Lippia alba populations in Guatemala. Flavour and Fragrance Journal 19, 333-5.
Contact: Novak, Johannes; Inst Appl Bot, Univ Vet Med, Vet Pl 1, A-1210, Vienna,
Austria.
Franco J, J Crossa, S Taba, H A Shands. A sampling strategy for conserving genetic
diversity when forming core subsets. Crop Science, v.45, p.1035-1044, 2005.
G
García DE. Evaluación de los principales factores que influyen en la composición
fitoquímica de Morus alba (Linn.). Tesis de Maestría, Estación experimental de
Pastos y Forrajes “Indio Hatuey”, Cuba. 97 pp.
García DE, MG Medina, C Dominguez, A Baldizán, J Humbría, L. Cova. Evaluación
química de especies de leguminosas con potencial forrajero en el Estado de
Tujillo, Venezuea. Rev Zoot Trop, 24 (4): 401-415. 2006.
Godínez D, G Volpato, 2008. Plantas medicinales que se venden en el mercado El Rio,
Camaguey, Cuba. Rev Mex de Biodiverv 79: 243-259.
González J., Cruz-Angón A., Flores-Palacios A., Vovides A.P. 2004. Diversity and
genetic Structure of the Mexican Endemic Epiphyte Tillandsia achyrostachis E.
Morr. Ex Backer var. achyrostachis (Bromeliaceae). Annals of Botany 94:545-551.
Grassi F., Cazzaniga E., Minuto L., Peccenini S., Barberis G., Basso B. 2005. Evaluation
of biodiversity and conservation strategies in Pancrayum maritimum L. for the
Northern Tyrrhenian Sea. Springer Netherlands. Vol 14 Number 9: 2159-2169.
Guerrero A. 2010. Evaluación de los extractos de aceites esenciales de Lippia
origanoides, en el control de hongos fitopatógenos en el cultivo de Ají
Cayena (Capsicum frutescens)en campo. Trabajo de grado Ingeniería
Agronómica, Universidad Nacional de Colombia – Sede Palmira.
Gupta S K, S P S Khanuja, S Kumar. 2001. In vitro micropropagation of Lippia alba.
Current Science, Vol. 81, No. 2, 25 jul. 2001.
H
Hennebelle T, S Sahpaz, C Dermont, J H Bailleul. 2006. The essential oil of Lippia alba:
analysis of samples from French overseas departments and review of previous
works. Chem Biodivers.2006 Oct; 3(10):1116-25.
Hennebelle T, S Sahpaz, B Gressier, H Joseph, F Bailleul. 2007. Antioxidant and
neurosedative properties of polyphenols and iridoids from Lippia alba. Phytother
Res. 2007 Aug 17.
Hennebelle T, S Sahpaz, H Joseph, F Bailleul. 2008. Ethnopharmacology of Lippia alba.
France. J. Ethnopharmacology, 2008 Mar 5; 116(2): 211-222.
Hernández C. 1990. La selva en Colombia. En Selva y Futuro, Eds. C. Hernández, J.
Carrizosa. Sello Editorial, Bogotá. pp. 13-40.
Bibliografía
133
Hernández C, G Hurtado, Q Ortiz, C Walscbulger, 1992. Unidades biogeográficas de
Colombia. En Diversidad biológica de Iberoamérica, Compilado por. G. Halffter.
Mexico. pp.
Hernández-V H, C Bonilla-C, M. Sánchez-O. Efecto de la fertilización nitrogenada en la
producción de biomasa y calidad de aceite esencial en Lippia alba (Miller), Pronto
alivio. S.f.
Herrera A M, C E Carranza, M I Chacón-Sánchez. 2013. Establishment of propagation
methods for growing promising aromatic plant species of the Lippia (Verbenaceae)
and Tagetes (Asteraceae) genera in Colombia. Agron. colomb. vol.31 no.1 Bogotá
ene./abr. 2013.
Huh M.K., Lee H.Y., Huh H.W. 1999. Genetic diversity and population structure of
Gleditsia japonica var. koraiensis in Korea. Bot. Bull. Acad. Sin. (1999) 40:159164.
Hyne RV, W A Maher. 2003. Invertebrate biomarkers: links to toxicosis that predict
population decline. Ecotoxicology and Environmental Safety, 54, 306-374.
I
IAVH (Instituto Alexander von Humboldt). 1998. Bosque seco Tropical (Bs-T) en
Colombia. Programa de Inventario de la biodiversidad Grupo de Exploraciones y
Monitoreo Ambiental GEMA. IAVH 1998.
IAVH. 2004. Informe anual 2003. Proyecto Conservación y Uso Sostenible de la
Biodiversidad de los Andes Colombianos Instituto de Investigación de Recursos
Biológicos Alexander von Humboldt. 107 p.
Igic B, L Bohs, J R Kohn. 2006. Ancient polymorphism reveals unidirectional breeding
system shifts. PNAS january 31, 2006. Vol. 103 no. 5 1359-1363.
J
Jaramillo M A, P S Manos. 2001. Phylogeny and patterns of floral diversity in the genus
Piper (Piperaceae). American Journal of Botany 88(4): 706–716.
Jaramillo B E, E Stashenko, J R Martínez. 2010.Composición química volátil de Satureja
brownei (Sw.) Briq. Revista Cubana de Plantas Medicinales 15(1) 52-63.
Jarvis A, M J Paternina Quijano, A Arcos, H J Rodríguez, C Nagles, N Melo. 2006.
Evaluación Rápida de la Adaptación al Medioambiente de plantas promisorias
medicinales. En: II Segundo Congreso Internacional de Plantas Medicinales y
Aromáticas Universidad Nacional de Colombia Sede Palmira. Octubre 19, 20 y 21
de 2006.
Juliani, H.R. (Jr.), A.R. Koroch, H.R. Juliani, V.S.Trippi and J.A. Zygadlo. 2002
Intraspecific variation in leaf oils of Lippia junelliana (Mold.) Tronc. Biochemical
Systematics and Ecology 30: 163 –170.
K
Kang M., Jiang M., Huang H. 2005. Genetic Diversity in Fragmented Populations of
Berchemelia wilsonii var. pubipetiolata (Rhamnaceae) Annals of Botany 95:11451151. doi: 10.1093/aob/mci125, available online at www.aob.oupjournals.org.
Kosakivska V. 2008. Biomarkers of plants with different types of ecological strategies.
General and Applied Plant Physiology. Vol XXIV, Special Issue, No. 1-2, 2008.
Kosman E, K. J. Leonard. 2005. Similarity coefficients for molecular markers in studies of
genetic relationships between individuals for haploid, diploid, and polyploid
species. Molecular Ecology (2005) 14, 415– 424
Kurian S, Y Grigoryev, S Head, D Campbell, T Mondala and D R. Salomon, 2007.
Applying genomics to organ transplantation medicine in both discovery and
134
Título de la tesis o trabajo de investigación
validation of biomarkers. Published in final edited form as: Int Immunopharmacol.
2007 December 20; 7(14): 1948–1960.
L
Larson S.R., Jones T.A., Jensen K.B. 2004. Population structure in Pseudoroegneria
spicata (Poaceae: Triticeae) modeled by Bayesian clustering of AFLP genotype.
America Journal of Botany. 2004; 91:1789-1801.
Linares J. 2007. Lippia alba(P. Mill.) N.E. Br. ex Britt. & Wilson.
http://www.siac.net.Co/sib/catalogoespecies/especie.do?idBuscar=89&method=di
splayAAT.
M
Manica-Cattani M F, J Zacaria, G Pauletti, L Atti-Serafini, S Echeverrigaray. 2009.
Genetic variation among South Brazilian accesions of Lippia alba Mill.
(Verbenaceae) detected by ISSR and RAPD markers. Braz. J. Biol., 69(3): 375380. 2009.
Mariot A., Di Stasi L.C., dos Reis M.S. 2002. Genetic Diversity in Natural Populations of
Piper cernuum. The Journal of Heredity: 93(5).
Marmolejo D F, R Mejía, I Hurtado, A M Posso, J E Muñoz. 2008. Caracterización
molecular de 15 aislamientos de Beauveria bassiana asociaos con Cosmopolites
y Metamasius en plátano y banano en tres regiones de Colombia. Acta Agron.
Vol. 57 no.3.199-204.
Martínez F. 2008. “Diversidad y relaciones genéticas de plantas promisorias de jardín del
género Lippia en el Departamento de Santander, Colombia”. GIEFIVET-UIS.
Producción científica. Proyectos de investigación finalizados. En: http://cenivam.
uis.edu.co /cenivam/productos/proinvesfinalizados.html
Martínez-Núñez F O, G A Castillo Villamizar, M.I. Chacón S.2008. Genetic diversity within
and among wild and garden aromatic species 6 of the genera Lippia, Aloysia and
Phyla in several locations in northeastern Colombia, Plant Systematics and
Evolution.
Marx H E, N O’Leary, Y-W Yuan, P W-Irwing, D C Tak, M E Múlgura, R G Olmstead.
2010. A molecular phylogeny and classification of verbenaceae. American Journal
of Botany 97(10): 1647-1663.
Medina-López LA, JJ Araya-Barrantes, G Tamayo-Castillo, RM Romero. Comparación de
metodologías de extracción para limoneno y carvona en Lippia alba usando
cromatografía de gases. Ciencia y Tecnología, 27(1y 2): 1-13,2011. ISSN: 03780524.
Meneses R, R. Ocazionez, J.R. Martínez, E. Stashenko, Inhibitory effect of essential oils
obtained from plants grown in Colombia on yellow fever virus replication in vitro.
Annals of Clinical Microbiology and Antimicrobials, 2009, 8 (8), http://www.annclinmicrob.com/content/8/1/8.
Mesa Arango AC, J. Montiel Ramos, B. Zapata, D.C. Durán García, L. Betancur Galvis,
E. Stashenko, Citral and carvone chemotypes from the essential oils of Colombian
Lippia alba (Mill.) N.E. Brown: composition, cytotoxicity and antifungal activity,
Memorias do Instituto Oswaldo Cruz, 2009, 104 (6), 878-884.
Mohammadi S. A, B. M. Prasanna. 2003. Analysis of Genetic Diversity in Crop Plants—
Salient Statistical Tools and Considerations. Crop Sci. 43:1235-1248.
Montanari R.M, L. A. Sousa, M.N. Leite, A.D.F. Coelho, L.F. Viccini, M.B. Stefanini. 2004.
Phenotypical plasticity of the external morphology in Lippia alba (Mill.) N. E. Br. ex
Britt & Wilson in response to level of luminosity and fertilization. Revista Brasileira
de Plantas Medicinais 6, 96-101.
Bibliografía
135
Montiel J, A.C. Mesa Arango, D.C. Durán García, J.G. Bueno, L.A. Betancur Galvis, E.
Stashenko, Evaluación de la actividad anti-candida y anti-aspergillus de aceites
esenciales de Lippia alba (Miller) N.E Brown y su asociación con sus
componentes mayoritarios Scientia et Technica, 2007, año XIII, 33, 243-246.
Morataya-Morales MA. Caracterización farmacopéica de cuatro plantas nativas de
Guatemala Albahaca (Ocimum micranton), Orégano (Lippia graveolens), Salvia
sija (Lippia alba) y Salviyá (Lippia chiapaensis). Tesis para optar al título de
Química Farmaceútica. Universidad de San Carlos de Guatemala, Facultad de
Ciencias Químicas y Farmacia. 2006.
Muñoz A M, C.M Caetano, F A Vallejo, M S Sánchez. 2006. Comportamento meiótico y
descripción morfológica Del pólen de prontoalivio. Acta Agron (Colombia) Vol.
55(2).
Muñoz A M, F A Vallejo, M Sanchez. 2007. Morfología y anatomia de lãs flores y semillas
de pronto alivio. Acta Agron. (Colombia) Vol. 56(1) 2007, p7-11.
Muñoz J E. 2008. Microsatélites amplificados al azar (RAM) en estudios de diversidad
genética vegetal. Acta Agron. Vol. 57, No. 4 (2008).
Mur-Lacambra J. 1992. Contrastes de autocorrelación espacial. Un estudio de Monte
Carlo ESTADISTICA ESPAÑOLA. Vol. 34, Núm. 130, 1992, págs. 285 a 307.
Murillo O, R Salazar. 2000. Relation between the degree of heterozygosity and seed
characteristics of Alnus acuminata. II Simposio sobre avances en la producción de
semillas forestales en América Latina. Memorias Santo Domingo DO. p. 105-108.
Turrialba Centro Agronómico Tropical de Investigación y Enseñanza (CATIE) CR.
2000.
N
Núñez María B., Sánchez Edit G., Bela Alberto, Aguado María I.. DETERMINACIÓN DE
METABOLITOS SECUNDARIOS EN LIPPIA ALBA (MILL.) Y LIPPIA TURBINATA
(GRISEB.) Universidad Nacional del Nordeste. Secretaria General de Ciencia y
Tecnología. Comunicaciones Científicas y Técnicas. 2008.
O
Ocazionez R E; Rocio Meneses; Flor Ángela Torres; Elena Stashenko. Virucidal activity
of Colombian Lippia essential oils on dengue virus replication in vitro. Mem. Inst.
Oswaldo Cruz 2010, 105, 304-309.
Oliveira D R, G G Leitão, S S Santos, H R Bizzo, D L Celuta, S. Alviano, D S Alviano, S G
Leitão. 2006. Ethnopharmacological study of two Lippia species from Oriximiná,
Brazil. Journal of Ethnopharmacology Volume 108, Issue 1, 3 November 2006,
Pages 103-108.
Ortiz–D J., Martínez-M C., Correal E., Simón B., Cenis J.L. 2005. Genetic Structure of
Atriplex halimus Populations in the Mediterranean Basin. Annals of Botany 95:827834.doi: 10.1093/aob/mci086, available online at www.aob.oupjournals.org.
P
Palacio K, N. Rodríguez López, Plasticidad fenotípica de dos poblaciones de Lippia alba
(Verbenaceae) sometidas a déficit hídrico bajo dos condiciones lumínicas, Acta
Biológica Colombiana, 2008, 13 (1), 187-198.
Pascual A N T, K M S Uzuki, F S Almeida, MK Leda, S Odré, S Silva. 2006. Evaluation of
the RAPD profiles from different body parts of Euglossa pleosticta Dressler male
bees (Hymenoptera: Apidae, Euglossina) Neotrop Entomol (on line). 2006. Vol.
35, no. 6, pp.811-817. ISSN 1519-566X.
136
Título de la tesis o trabajo de investigación
Peakal R, P E Smouse. GenAlEx6.5: genetic analysis in Excel. Population genetic
software for teaching and research-an update. BIOINFORMATICS APPLICATION
NOTE. Vol 28 no. 19 2012, pages 2537-2539. doi:10.1093/bioinformatics/bts460.
Pereira Peixoto P. H, F R Gonçalves-Salimena, M de Oliveira Santos, L Da Silva
Garcia, P. M de Oliveira Pierre, L. F. Viccini, W. Campos Otoni. 2006. IN VITRO
PROPAGATION OF ENDANGERED LIPPIA FILIFOLIA MART. AND SCHAUER
EX SCHAUER. In Vitro Cellular and Developmental Biology - Plant 42(6):558-561.
2006.
Pérez E, P Garrido, M Laca. 2008. Indicadores microbiológicos como marcadores de la
contaminación y recuperación de suelos contaminados con queroseno.
Ecosistemas 17(3):133-138.
Piedrahita A M, A Posso, J E Muñoz, L A Álvarez, 2008. Variabilidad genética de Hartón
del Valle mediante RAM. Act Agron Vol 57 no. 1, pp71-76.
Pierre P M O, S M Sousa, L C Davide, M A Machado, L F Viccini. 2011. Karotype
analysis, DNA content and molecular screening in Lippia alba (Verbenaceae). An
Acad Bras Cienc 83: 993–1005.
Pineda-Tenor D, R Jiménez L, R Ramos-Corral, D Lamuño-Sánchez, J A RecioMontealegre, F J García García, R M Gómez-Serranillos. 2011. Variabilidad
Biológica-VB y de Valores de Referencia de Cambio-VRC para una Población
Geriátrica en Pruebas Frecuentes del Laboratorio Clínico y Hematológico.
Sociedad Española de Dirección y Gestión de los Laboratorios Clínicos. XV
Reunión. Toledo 30-31 de marzo de 2011. Resumen-10.
Pino-Benítez N, H. Valois. 2004. Consevation of Biological and cultural Biodiversity in the
Andes and the Amazon basin-Ethnobotany, resource use and Zoology. Lyona,
Volume 7(2) pages 61-69.
Q
R
Ramalho R. 2006. Genetic diversity based on AFLP molecular markers and indicator for
the establishment of core collection for pumpkin (Cucurbita moscata) for north-esta
Brasil. PhD Thesis Universidad Estadual de Norte Fluminenexe (UENF) Campus
dos Goytecazes, RJ, Brasil.
Riccardi G A L, A I A Riccardi, A L Bandoni. Fitoquímica de Verbenaceae (Lippias y
Aloysias) del Nordeste argentino. Universidad Nacional del Nordeste.
Comunicaciones Científicas y Técnicas. 2000.
S
Sanabria H L, M A García, J E Muñóz, H A Díaz. 2006. Caracterización molecular con
marcadores RAM de árboles nativos de Psidium guajava (guayaba) en el Valle del
Cauca. Tesis de Maestría en Ciencias, Universidad Nacional de Colombia-Sede
Palmira.
Santos FR, Lima PF, Priolli RH, Siqueira WJ, Colombo CA. Isolation and characteristics
of eight novel polymorphic microsatellite loci in Lippia alba (Verbenaceae). Am J
Bot. 2012 Aug; 99(8):e301-3. doi: 10.3732/ajb.1100578. Epub 2012 Jul 26.
Schroeder M A, A E López, G C Martínez. 2004. Resultados preliminares del análisis
foliar de Lippia alba (Mill.) N.E. Brown, Pluchea sagittalis (Lamb.) Cab., Petiveria
alliaceae L. y Ocimum salloi Benth. Universidad Nacional del Nordeste.
Comunicaciones Científicas y Tecnológicas 2004. Resumen: A-035.
Seaforth, C. and T. Tikasingh. 2008. A study for the development of a handbook of
selected Caribbean herbs for industry. Technical Centre for Agriculture and Rural
Bibliografía
137
Cooperation (CTA); Inter- American Institute for Cooperation on Agriculture (II
CA). Wageningen, The Netherlands; St. Augustine, Trinidad.
Selkoe K A, R J Toonen. 2006. Microsatellite for ecologists: a practical guide to using and
evaluating microsatellite markers. Ecology Letters, (2006) 9: 615-629.
Sheng Y, Zheng W, Pei K, MA K. 2005. Genetic Variation Within and Among Populations
of a Dominant Desert Tree Haloxylon ammodendron (Amaranthaceae) in China.
Annals of Botany. 96:245-252.
Shugart L R, J F McCarthy, R S. Halbrook. 2006. Biological Markers of environmental and
ecological Contamination: An Overview. Risk Analysis. Volume 12 Issue 3, Pages
353-360.
Silva P D, T C Bastos, L S Azevedo, A T Amaral, A B Passos, R Rodrigues, F Matta.
2010. Quantification of the diversity among common bean accessions using WardMLM strategy. Pesq. Agropec.bras. Brasilia vol 45 no. 10 p 1124-1132.
Singh G, S K Pardey, P A Leclecq, J Sperkova. 1999. Estudies on Essential Oils. Part 15.
GS/MS Analysis of Chemical Constitutiens of leaf oil of Lippia alba (Mill.) from
North India. Journal of Essential Oil Research. Vol. 11, issue 2, 1999.
Sousa S M P, S Silva, G A Torres, L F Viccini. 2009. Chromosome banding and
essential oils composition of Brazilian accessions of Lippia alba (Verbenaceae).
BIOLOGIA Volume 64, Number 4, 711-715.
Sousa S M, G A Torres, L F Viccini. 2012. Kariological studies in Brazilian species of
Lippia L. (Verbenaceae). An Acad Bras Cienc (2002). Comunicaciones Científicas
y Tecnológicas 2004. Resumen: A-035.
Stashenko E E, B E Jaramillo, J R Martínez. 2003. Comparación de la composición
química y de la actividad antioxidante in vitro de los metabolitos secundarios
volátiles de plantas de la familia Verbenaceae. Rev. Acad. Colomb. Cienc. 27
(105): 579-597, 2003.
Stashenko E E, B E Jaramillo, J. R. Martínez. 2004. Comparison of different extraction
methods for the analysis of volatile secondary metabolites of Lippia alba (Mill.)
N.E. Brown, grown in Colombia, and evaluation of its in vitro antioxidant activity, J.
Chromatogram. A, 1025, 93-103 (2004).
Suárez A R, F O Martínez, G A Castillo, M I Chacón. 2007. Molecular Characterization of
Aromatic Species of the Genus Lippia from the Colombian Neotropics.
Proceedings of the International Symposium on Medicinal Nutraceutical Plants.
Ed. A.K. Yadak. Acta Hort. 756, ISHS 2007.
Suárez González A R, F O Martínez-Núñez, G A Castillo-Villamizar, M I Chacón. 2007.
Molecular characterization of aromatic species of the genus Lippia from the
Colombian Neotropics, Acta Horticulturae (ISHS), 756, 129-138.
Suárez-González A R, G A Castillo-Villamizar, M I Chacón. 2008. Genetic diversity and
spatial genetic structure within a population of an aromatic shrub, Lippia
origanoides (Verbenaceae), in the Chicamocha Canyon, northeastern Colombia,
Genetics Research, 90 (6), 455-465.
Sawada, M. 1999. ROOKCASE: An Excel 97/2000 Visual Basic (VB) Add-in for Exploring
Global and Local Spatial Autocorrelation. Bulletin of the Ecological Society of
America, 80(4):231-234.
T
Tarpley L, A.L. Duran, T.H. Kebron, W. Summer. 2005. Biomarkers metabolites capturing
the metabolite variance present in a rice plant developmental period. BMC Plant
Biol. 2005; 5:8.
138
Título de la tesis o trabajo de investigación
Tavares E S, L S Juliao, D Lopez, H R Bizzo, C L S Lage, S G Leiao. 2005. Análise do
óleo escencial de folhas de três quimiotipos de Lippia alba (Mill.) N.E. Br.
(Verbenaceae) cultivados em condições semelhantes. Revista Brasilera de
Farmacognosia 15(1):1-5,Jan./Mar. 2005.
Tero N., Aspi J., Siikamäki P., Jäkäläniemi A. 2005. Local genetic population structure in
an endangered plant species, Silene tatarica (Caryophyllaceae). Heredity 94, 478487.
Thoppil J E. 2000. Essential oil analysis of Eusterialis deccanensis Panigrahi
(Lamiaceae). Boll Chim Farm Jul-Aug; 139(4): 194-5.
Trujillo L. 2004. Useful plants in indigenous Bribri and Cábecar farms, Talamanca, Costa
Rica. Thesis Mag. Sci., CATIE, Turrialba, Costa Rica. 85 p.
U
UNCTAD. 2005. Market Brief in the European Union for selected natural ingredients
derived from native species, Lippia alba (Prontoalivio, Erva cidreira, juanilama,
Melissa). This publication was developed in the context of the UNCTAD / BioTrade
Facilitation. Programme, funded by the Governments of the Netherlands and
Switzerland.
V
Vasseur P, C Cossu-Leguille.
2003. Biomarkers and community indices as
complementary tools for environmental safety. Environmental International 28,
711-717.
Vega N E, M I Chacón. 2011. Isolation of high-quality DNA in 16 aromatic and medicinal
Colombian species using silica-based extraction columns. Agron. colom. 29(3),
349-357. 2011.
Vega N E. 2011. Estructura Poblacional de Lippia origanoides H.B.K. en el Cañón del Rio
Chicamocha (Boyacá & Santander, COL). Universidad Nacional de Colombia
Facultad de Agronomía Bogotá, Colombia. 2011. Trabajo de grado presentado
como requisito parcial para optar al título de: Magister en Ciencias Agrarias.
Vega N.E. and M.I. Chacón-Sánchez. 2012. Genetic structure along an altitudinal
gradient in Lippia origanoides, a promising aromatic plant species restricted to
semiarid areas in northern South America. Ecol. Evol. 2(11), 2669-2681.
Vega N E; W A Delgado and M I Chacón. 2013. Genetic structure and essential oil
diversity of the aromatic shrub Lippia origanoides Kunth (Verbenaceae) in two
populations from northern Colombia. Agron. colomb. [online]. 2013, vol.31, n.1, pp.
5-17. ISSN 0120-9965.
Vera J R, P F Pastrana, K Fernández, A Viña. 2007. Actividad antimicrobiana in vitro de
volátiles y no volátiles de Lippia alba y extractos organicos acuosos de Justicia
pectoralis cultivados en diferentes pisos t{ermicos del departamento del Tolima.
Sciencia et Technica. Año XIII, no. 33, mayo 2007.
Vera P A, J A Olivera, B E Jaramillo, E Stashenko. 2010. Efecto protector Del aceite
esencial de Lippia Alba (Mil.) N.E. Brown sobre toxicidad Del mercuriocromo em
raices de Allium cepa L. Revista Cubana de Plantas Medicinales 15(1) 27-37.
Verma A, N. Kumar, SA Ranade. 2004. Genetic diversity amongst landraces of a
dioecious vegetatively propagated plant, Betelvine (Piper betle L.). J. Biosci. 2004
Sep; 29(3):319-28.
Vit P, Silva, B, Meléndez, P. 2002. Lippia alba N.E.Br. Ficha botánica de interés apícola
en Venezuela, No. 2 Cidrón. REVISTA DE LA FACULTAD DE FARMACIA Vol.
43, 2002.
Bibliografía
139
Vera P A, J A Olivera, B E Jaramillo, E Stashenko. 2010. Efecto protector Del aceite
esencial de Lippia Alba (Mil.) N.E. Brown sobre toxicidad del mercuriocromo em
raices de Allium cepa L. Revista Cubana de Plantas Medicinales 15(1) 27-37.
Verma A, N. Kumar, SA Ranade. 2004. Genetic diversity amongst landraces of a
dioecious vegetatively propagated plant, Betelvine (Piper betle L.). J. Biosci. 2004
Sep; 29(3):319-28.
Vestergaard O, O Hoegh-Guldberg, U Unluata. 2003. UNDERSTANDING CORAL
BLEACHING ACROSS FOUR OCEANS-addressing CBD’s Specific Workplan on
Coral Bleaching. Convention of Biological Diversity (CBD), SBSTTA 8, 10-14 March
2003, Montreal, Extended Abstract.
Viccini L. F, D. C. Souza da Costa, M. A. Machado and A. L. Campos. 2004. Genetic
diversity among nine species of Lippia (Verbenaceae) based on RAPD Markers.
Plant Systematics and Evolution. Volume 246, Numbers 1-2 / abril de 2004.
Viccini L F, P M O Pierre, M M Praça, D C Souza da Costa, E da Costa Romanel, S M de
Sousa, P H Peixoto, P. Salimena, F R Gonçalves, F R Gonçalves Salimena.
2006. Chromosome numbers in the genus Lippia (Verbenaceae). Revista: Plant
Systematics and Evolution, 2006; 256 (1-4) Página(s):171-178 ISSN:03782697.
Vieira R F, L A Skolupa. 1993. Brazilian medicinal plants genebank. Acta Hort. 330:51-58.
Vieira R F. 1999. Conservation of Medicinal and Aromatic Plants in Brazil. J. Janick (ed.),
ASHS Pess, Alexandria, VA.
Vilalta y Perdomo C.J. 2004. Sobre Cómo Enseñar Autocorrelación Espacial. Tecnológico
de Monterrey, Campus Ciudad de México. Tecnológico de Monterrey, Campus
Ciudad de México, y candidato a investigador nacional en el SNI desde 2004.
Vilches Blas, Alicia Roca, José Naranjo, Bernardo Navarro, David Bramwell y Juli
Caujapé-Castells. Estructura espacial de la variación genética de Erysimum
albescens (cruciferae) en gran Canaria: implicaciones para la conservación ex situ
en bancos de germoplasma. Bot. Macaronésica 25: 15:30 (2004). ISSN 0211715015.
Vit P, B Silva, P Meléndez. 2002. Lippia alba N.E.Br. Ficha botánica de interés apícola en
Venezuela, No. 2 Cidrón. REVISTA DE LA FACULTAD DE FARMACIA Vol. 43,
2002.
W
Wang D, J. Shi, S R. Carlson, P, B. Cregan, R W. Ward and B.W. Diers. 2003. A loewcost, High-Throughput Polyacrylamide Gel Electrophoresis System for Genotyping
with Microsatellite DNA Markers. Crop Scie. 43:1828-1832 (2003).
Wezel A. 2003. Plant species diversity at homegardens in humed and semiarid Cuba and
its importance for self-sufficiency of households. Deutscher Trompetag 2003.
Gottingen, October 8-10, 2003.
Wolf P.G., Doche B., Giely L., Taborlet P. 2004. Genetic Structure of Rhododendron
ferrugineum at a Wide Range of Spatial Scales. Journal of Heredity: 95(4):301308.
X
Xiao, L-Q, Xue-Jun Ge, Xun Gong1, Gang Hao, Si-Xiang Zheng. 2004. ISSR Variation in
the Endemic and Endangered Plant Cycas guizhouensis (Cycadaceae). Annals of
Botany 94: 133±138.
Xie G-W., Wang D-L., Yuan Y-M., Ge X-J. 2005. Populations Genetic Structure of
Monimopetalum chinese (Clesteceraceae) an Endangered Endemic Species of
Eastern China. Annals of Botany 95:773-777. doi: 10.1093/aob/mci087, available
online at www.aob.oupjournals.org
140
Título de la tesis o trabajo de investigación
Y
Yamamoto P Y, C A Colombo, J A Azevedo Filho, A L Lourencao, M O Mayo Marques, G
D da Silva Morais, A F Chiorato, A L Melo Martins, W J Siqueira. 2008.
Perfomance of ginger grass (Lippia alba) for traits related to the production of
essential oil. Sci. Agric. (Piracicaba, Braz.), v.65, n.5, p.481-489, Sept./Oct. 2008.
Yuan, Y.-W., Chang Liu, Hannah E. Marx, Richard G. Olmstead. An empirical
demonstration of using pentatricopeptide repeat (PPR) genes as plant
phylogenetic tools: Phylogeny of Verbenaceae and the Verbena complex. Mol.
Phylogenet. Evol. (2009), doi:10.1016/j.ympev.2009.08.029.
Zas-Arregui R, P Martíns Garbín y R de la Mata Pombo. 2008. AUTOCORRELACIÓN
ESPACIAL: UN PROBLEMA COMÚN...MENTE OLVIDADO. Cuad. Soc. Esp.
Cienc. For. 24: 139-145 (2008).
Zhang X, F C Leung, D K O Chan, Y Wu. 2002. Comparative Analysis of Allozyme,
Random Amplified Polymorphic DNA and Microsatellite Polymorphism on Chinese
Native Chickens. Poultry Science 81: 1093-1098.
Zhang S, X Zhanga, 2009. Efect of two composted plant pesticide reissues, incorporated
with Thrichoderma viride, on root-knot nematode in Balloon flower. Agr Sce in
China. Vol 8 Issue 4, April 2009, pages 447-454.
Zoghbi G D, E H A Andrade, A S Santos, M H L Silva, J G S Maia. 1998. Essential oils of
Lippia alba (Mill.) NE Br growing wild in the Brazilian Amazon. Flavour and
Fragrance Journal 13:47-48.
Zucchi M I, J B Pinheiro, L J Chaves, A S G Coelho, M A Couto, L K Morais, R
Venkosvsky. 2005. Estrutura genética e fluxo génico em populações naturais de
cagaita. Pesq. Agropec. Bras vol 40 no 10.