Preguntas esenciales
Anuncio
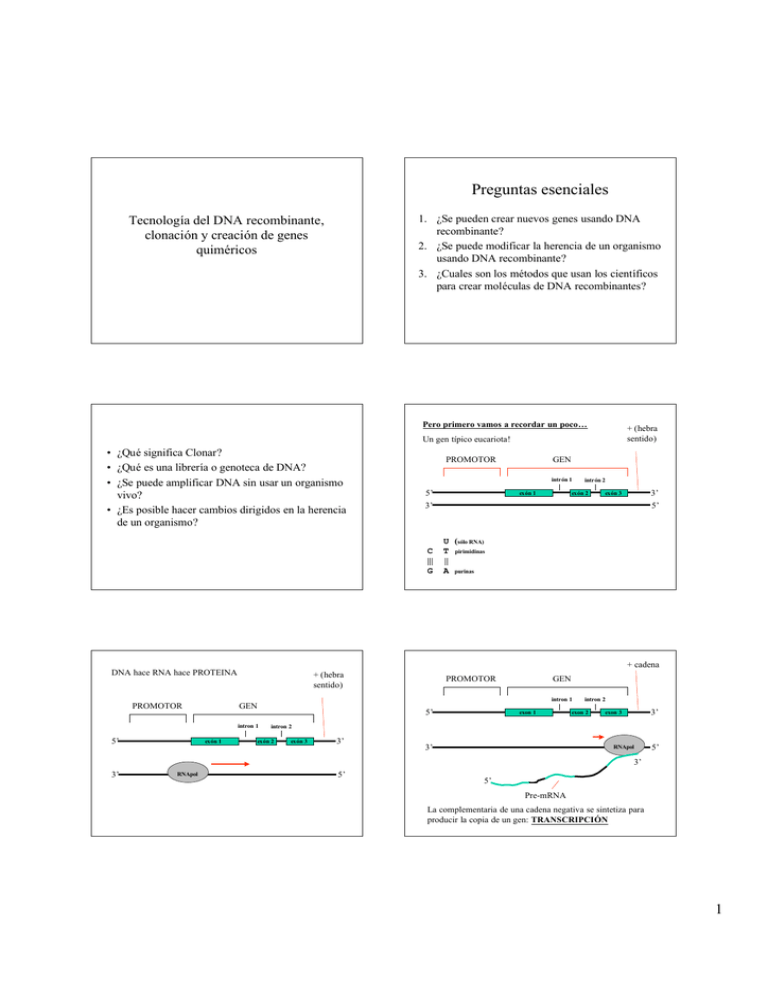
Preguntas esenciales 1. ¿Se pueden crear nuevos genes usando DNA recombinante? 2. ¿Se puede modificar la herencia de un organismo usando DNA recombinante? 3. ¿Cuales son los métodos que usan los científicos para crear moléculas de DNA recombinantes? Tecnología del DNA recombinante, clonación y creación de genes quiméricos Pero primero vamos a recordar un poco… + (hebra sentido) Un gen típico eucariota! • ¿Qué significa Clonar? • ¿Qué es una librería o genoteca de DNA? • ¿Se puede amplificar DNA sin usar un organismo vivo? • ¿Es posible hacer cambios dirigidos en la herencia de un organismo? PROMOTOR intrón 1 5’ + (hebra sentido) 3’ exón 3 5’ U (sólo RNA) T pirimidinas || A purinas PROMOTOR GEN intron 1 GEN 5’ intron 1 exón 1 intrón 2 exón 2 + cadena DNA hace RNA hace PROTEINA 5’ exón 1 3’ C ||| G PROMOTOR GEN exon 1 intron 2 exon 2 3’ exon 3 intron 2 exón 2 exón 3 3’ 3’ RNApol 5’ 3’ 3’ RNApol 5’ 5’ Pre-mRNA La complementaria de una cadena negativa se sintetiza para producir la copia de un gen: TRANSCRIPCIÓN 1 3’ RNApol 5’ 3’ 5’ 3’ mRNA 5’ proteína Pre-mRNA TRADUCCIÓN NH 2 M Splicing o ayuxtamiento S 3’ 5’ 3’ 5’ t RNA G mRNA mRNA RNA Ribosomal ¿Qué significa clonar? OBJETIVOS Clon: una colección de moleculas o células, todos idénticos a una molécula o célula original 1. CLONAR • “Clonar un gen” es hacer copias del mismo, por ejemplo en una población bacteriana • Gen puede ser una copia de un gen original o natural • Gen puede ser una versión alterada del mismo • Tecnología del DNA recombinante lo hace posible en el laboratorio GEN CLON = multiples copias de la misma cosa intron 1 5’ exon 1 intron 2 exon 2 exon 3 3’ Por tanto – si clonamos un gen, significa: restricción •Cortarlo (RESTRICCIÓN) del resto del genoma •Unirlo covalentemente (LIGARLO) dentro de algo que se propague (un VECTOR). •Situar el vector dentro (TRANSFORMARLO) de una célula (HUESPED) que va a propagar el vector por nosotros ligar VECTOR Por ejemplo plasmid Vector recombinante 2 PROPAGACIÓN DEL VECTOR Vector recombinante Cromosoma del huesped •Incremento del número de copias del plásmido TRANSFORMAR •Proliferación celular bacteria Recuerda – DNA procariota no tiene intrones! 3’ 5’ mRNA Si queremos trabajar con un gen y su producto – necesitamos un versión “sin intrones” del gen: Necesitamos hacer una versión en DNA del mRNA 3’ RNApol 5’ 3’ 5’ Pre-mRNA Su nombre es cDNA splicing Esto es: equivalente al RNA 3’ Cuando por ejemplo decimos que hemos clonado y expresado el gen para (por ejemplo hormona de crecimiento humana) queremos decir que hemos clonado el cDNA de la hormona de crecimiento humana 5’ mRNA Figure 9.1.1 Figure 9.1.2 3 Enzimas usadas en tecnología del DNA recombinante Objetivos 1. Clonación 1. DNA polimerasas 2. Enzimas empleados en tecnología del DNA recombinante Reacción general: (dNMP)n + dNTP (dNMP)n+1 + PPi Todas las DNA polimerasas: N 5’ N N 5’ P + P N P N P DNApol N P Necesitan un cebador con un 3’OH libre El DNA sintetizado en la nueva hebra SIEMPRE va en dirección 5’ 3’ P N P P OH 5’ OH P N P 3’ 3’OH 5’ Equivalente a nuestra hebra – para nuestro gen ejemplo OH P.e. DNA polimerasa I de E. coli. P.e. DNA polimerasa I de E. coli. DNA polimerasa dependiente de DNA DNA polimerasa dependiente de DNA 4 P.e. DNA polimerasa I de E. coli. P.e. DNA polimerasa I de E. coli. DNA polimerasa dependiente de DNA DNA polimerasa dependiente de DNA Pero también tiene otras actividades asociadas Pero también tiene otras actividades asociadas p.e. 5’ → 3’ exonucleasa (eliminar los cebadores RNA primers en la replicación del DNA) p.e. 5’ → 3’ exonucleasa (eliminar los cebadores RNA primers en la replicación del DNA) P.e. DNA polimerasa I de E. coli. P.e. DNA polimerasa I de E. coli. DNA polimerasa dependiente de DNA DNA polimerasa dependiente de DNA Pero también tiene otras actividades asociadas Pero también tiene otras actividades asociadas p.e. 5’ → 3’ exonucleasa (eliminar los cebadores RNA primers en la replicación del DNA) p.e. 3’ → 5’ exonucleasa (corrige errores cometidos por la polimerasa) reacción general: 2. RNA polimerasas (NMP)n + NTP (NMP) n+1 + PP i En el lab - del Bacteriofago - RNA polimerasa dependiente de DNA 3’ Alta especificidad para promotores RNApol 5’ 3’ No requiere un cebador 5’ PROMOTOR GEN promotor 5’ 3’ 3’ 5’ (Pre)mRNA RNA se sintetiza en dirección 5’ 3’ 5 3. Transcriptasas reversas 3. Transcriptasas reversas • DNA polimerasa dependiente de RNA – se encuentran en virus (AMW – avian myeloblastosis virus / MMLV – moloney murine leukemia virus. • DNA polimerasa dependiente de RNA – se encuentran en virus (AMW – avian myeloblastosis virus / MMLV – moloney murine leukemia virus. • Algunas transcriptasas reversas también tienen 3’ y/o 5’ actividad exoRNase (degrada RNA después de la transcripción reversa) • Algunas transcriptasas reversas también tienen 3’ y/o 5’ actividad exoRNase (degrada RNA después de la transcripción reversa) • Requieren un cebador • Requieren un cebador 5’ RNA 3’ 5’ RNA 3’ 4. Endonucleasas de restricción 3. Transcriptasas reversas • cebador 5’ DNA polimerasa dependiente de RNA – se encuentran en virus (AMW – avian myeloblastosis virus / MMLV – moloney murine leukemia virus. • Algunas transcriptasas reversas también tienen 3’ y/o 5’ actividad exoRNase (degrada RNA después de la transcripción reversa) • Requieren un cebador Sólo se usan tipo 2 en DNA recombinante Reconoce secuencias palíndromes Cortan dentro – endo - DNA cebador 5’ 5’ RNA 3’ DNA (de nuevo) se sintetiza en dirección 5’ 3’ 5. Ligasa 4. Endonucleasas de restricción Sólo se usan tipo 2 en DNA recombinante Reconoce secuencias palíndromes Cortan dentro – endo - DNA Corrige a la endonucleasa de restricción y requiere ATP Recuerda – se requieren extremos compatibles Funciona mejor en extremos cohesivos que en romos 6 DNA Ligasa G P-AATTC | + | CTTAA-P G GAATTC |||||| CTTAAG ATP /Mg 2+ Ligación de extremos romos usando T4 DNA ligasa, que cataliza la unión dependiente de ATP de moléculas de DNA 6. Quinasas / Fosfatasas Como se hace un cDNA? P _ •Transcriptasa reversa: A P N 5’ P encontrada en virus requiere un cebador P HO OH N 5’ mRNA tiene una cola “poliA” OH P P DNA o RNA ATP AAAAAAAAAA 5’ •Fosfatasa (elimina grupos P) p.e. fosfatasa de tracto intestinal 3’ P mRNA N 5’ HO P DNA or RNA TTTTTTT 5’ AAAAAAAAAA AAAAAAAAAA 5’ 3’ 5’ mRNA Transcriptasa reversa + dNTPs Oligo dT I.e (TTTTTTTTT) TTTTTTT 5’ TTTTTTT 5’ AAAAAAAAAA 5’ 3’ 3’ 5’ AAAAAAAAAA 3’ 3’ 7 1era cadena del cDNA TTTTTTT 5’ 5’ AAAAAAAAAA 3’ 3’ DNA de Doble Cadena, con una cadena equivalente al mRNA TTTTTTT AAAAAA 3’ 3’ 5’ DNA polimerasa + dNTPs 1ra cadena del cDNA TTTTTTT AAAAAA 3’ 5’ 3’ ligar 2nda cadena de cDNA 5’ VECTOR Por ejemplo plasmido cebador = secuencia de reconocimiento de la endonucleasa de restricción Endonucleasas de restricción (EN DETALLE) •Se encuentran en bacterias. •P.e. BamHI Bacillus amyloliquefaciens cepa H Por ejemplo: bacteriófago_ Cromosola propio •En naturaleza – funciona para digerir DNA extraño en la bacteria Me •Reconoce (secuencias palíndromes) secuencias inversas repetidas (tiene la misma secuencia 5’-3’ en cada cadena) Me Me •“restringe” la existencia en la bacteria •Para evitar daño del DNA endógeno, los sitios de restricción están metilados Me Me Me Tipos de endonucleasas Tipo I: Dependiente de ATP Endonucleasas de restricción hidroliza entre el 3’OH y 5’P para dejar un grupo P en el final 5’: 5’ NNNGAATTCNNN N Endonucleasa y metilasa Digiere DNA en sitios al azar Hasta 1 kb del sitio de reconocimiento Tipo II: Independiente de ATP NNNCTTAAGNNN G C P EcoR1 + H2O T A Digiere DNA en el sitio de reconocimiento dependiente de ATP Endonucleasa y metilasa Digiere DNA ~ 25nts de secuencia de reconocimiento 5’NNNG-OH A P Endonucleasa Type III: EcoRI + H 2O T A P P- AATTCNNN NNNCTTAA -P T OH- GNNN 5’ A P T G P C P Cadena - N P P Cadena + 8 Enzimas de restricción Hay varios cientos de endonucleasas de restricción disponibles que producen varios “finales”: 5’ protuberantes P.e. EcoRI 3’ protuberantes P.e. HhaI Romos P.e. SmaI GAATTC |||||| CTTAAG G P-AATTC | | CTTAA-P G GCGC |||| CGCG GCG | C-P CCCGGG |||||| GGGCCC CCC P-GGG ||| ||| GGG-P CCC reconoce una secuencia de DNA de doble cadena específica y corta dentro de esa secuencia romo Se llama el sitio de reconocimiento normalmente 4-8 nucleó nucleótidos Corta fragmentos largos de DNA en piezas Llamada digestió digestión de restricció restricción P-C | GCG Los cortes producidos por las enzimas de restricció restricción tienen simetrí simetría Corta de manera reproducible Se encuentran en bacteria Producen dos tipos de finales cohesivos o romos Enzimas de restricció restricción • hace cortes que se superponen porque reconocen normalmente secuencias simétricas • Llamadas Palí Palíndromes Inverted Palindrome: Palíndrome: la lectura de bases en una cadena en 5’ → 3’ is la misma que la secuencia 5’ → 3’ de la secuencia complementaria Extremos cohesivos: cohesivos: • cada fragmento de DNA (independientemente de la fuente del DNA) generado al cortar un DNA con el mismo E.R. tiene la misma secuencia de bases en los dos finales digeridos (leídos en dirección 5’ a 3’). El nú número de pares de bases en el sitio del enzima de restricció restricción determina la distancia media entre sitios de restricció restricción Los dos fragmentos de DNA distintos se pueden unir por complementareidad de bases para formar una molécula de DNA recombinante Molé Molécula de DNA recombinante: recombinante creación de una nueva combinación de DNA no encontrada en la naturaleza GTAC Es decir la distancia media entre sitios producidos por E.R. = 4 n donde n = número de bases en el sitio kb or kilobase = 1000 pares de bases 9 Se pueden usar algunos “trucos” para crear sitios de restricción De manera práctica – digestión con endonucleasas de restricción del DNA del fago lambda Lambda – un virus que infecta bacteria Genoma linear de DNA de doble cadena de ~ 49Kb Sitio RE lamda genome plasmid Electroforesis en gel de agarosa lamda genome lambda lambda plasmid plasmid +RE +RE plasmid RE RE Tamaño del DNA Con el fin de introducir DNA en un vector necesitamos extremos compatibles EcoRI EcoRI EcoRI EcoRI EcoRI EcoRI EcoRI BamHI EcoRI + BamHI BamHI EcoRI VECTOR P.e. plasmido Usar 2 (no-compatibles) enzimas de restricción: EcoRI BamHI VECTOR EcoRI EcoRI P.e. plasmido EcoRI Problema: Autoligación del vector >> recombinantes ligar EcoRI + BamHI EcoRI BamHI ligar EcoRI BamHI 10







