Centro de Investigaci´on y de Estudios Avanzados del
Anuncio

Centro de Investigación y de Estudios
Avanzados del Instituto Politécnico
Nacional
DEPARTAMENTO DE F ÍSICA
Análisis conformacional del Péptido β
amiloide en membranas de lı́pidos por
medio de la técnica de dinámica
molecular
Tesis que presenta
Fernando Favela Rosales
para obtener el Grado de
Maestro en Ciencias
en la Especialidad de
Fı́sica
Director de tesis: Dr. Francisco Castro Román
México, Distrito Federal
Noviembre, 2008.
Agradecimientos
Primero que todo quiero agradecer a mi asesor, el Dr. Francisco Castro Román,
por haber dirigido esta tesis y haber despertado mi interés en esta área multidiciplinaria llamada biofı́sica, por sus apoyos incluso en los aspectos extra académicos y
por haberme permitido considerarlo como un amigo.
A mis padres Alfredo Favela y Marı́a Guadalupe Rosales† (1944-2008) quienes a
pesar de que no cuento con un trabajo “real” siempre han creı́do en mı́ y en mi inquietud por entender los fenómenos de la naturaleza. A mi madre que donde quiera
que esté seguramente está contenta de ver terminado este trabajo.
A mis hermanos Vero, Fredy, Hugo, Eduardo, Roberto, Julia, Carmela y Catalina por
todo lo que hemos compartido y sino fuera por ese cariño y apoyo que me han dado
nada de esto podrı́a ser posible.
A todos mis sobrinitos que son una lista interminable con quienes he compartido
muchas alegrı́as.
A mis compañeros de generación: Pepe, Abril, Taxi, Migue, Mónica, Gil, Sergio, De
Moure, Aldrin, Carlos, Joel, Estela, Cesaré, Hilario, Karina, Neil y Aizar. Por todos
los momentos “super wow” de estos años, cada uno me dejó enseñanzas positivas.
Siempre estarán en mi memoria sobre todo la compañerita y los “gorditos”.
A Sarayd Limón por hacer mi vida feliz con todos esos bellos momentos que hemos
pasado juntos.
A Alma Tapia porque siempre ha estado conmigo, en las buenas y en las malas y
sé que desde Francia me envı́a su apoyo.
A Rigoberto Cruz, Julián González, Claudio Contreras y Alfredo Gómez por todas
las charlas sobre “cosas de la vida y de la ciencia”.
A Roger Hernández Pinto por proporcionar la Mac. Todo un ejémplo de constancia
y perseverancia.
A la familia Aguayo Candelas por los siempre cálidos recibimientos en Zacatecas y
Nochistlán.
A Javier Berúmen, José Ramı́rez, Ninfa Navarro, Marisa Perea, Yiliana Herrera y
III
Zhenia Hideki por las estrechas relaciones que hemos formado desde hace tanto
tiempo, nunca olvidaré lo que hemos compartido.
A todos los investigadores del Departamento de Fı́sica del CINVESTAV. En especial
a Tonatiuh Matos por todas sus enseñanzas y su forma de transmitir la fı́sica, Miguel
Meléndez por el recibimiento al inicio de mi estadı́a en el DF y Augusto Garcı́a por
mostrar los verdaderos valores de un amante de la filosofı́a de la ciencia.
A José Luis Arauz Lara y sus estudiantes por el agradable trato brindado en mi estancia en el IF-UASLP.
Al grupo de Fı́sica Estadı́stica por el apoyo brindado durante la realización de esta tesis, sobre todo a mis sinodales Pedro Gonzáles Mozuelos y Mauricio Carbajal
Tinoco por sus valiosos comentarios para la versión final y a Martı́n Hernández
Contreras por facilitarme una de sus computadoras.
A las secretarias Patricia Villar, Flor Ibañez y Marı́a de la Luz Rodrı́guez Sandoval
por siempre recibirnos con una sonrisa y hacer sencillos los trámites burocráticos,
gracias por todo.
A Justin A. Lemkul y José Luis Ricardo Chávez por su valiosa ayuda en la parte
técnica de los programas GROMACS y NAMD respectivamente.
Al Centro Nacional de Supercómputo del Instituto Potosino de Investigación Cientı́fica y Tecnológica (IPICyT): Cluster IBM E-1350 (Argentum) y Supercomputadora
Cray XD1 (Unpotosi), porque literalmente esto no hubiera sido posible sin sus recursos.
Al Proyecto Conacyt 60595 “Materia condensada blanda”.
Al CINVESTAV y al CONACYT por permitirme realizar mis estudios de maestrı́a
con sus recursos académicos y económicos respectivamente.
Fernando Favela
México D. F.
17 de diciembre de 2008
Índice general
1. Introducción
1.1. La Vida a Nivel Molecular . . . . . . . . . . . . . . . . . . . . . . . . .
1.2. De la Estructura a la Dinámica . . . . . . . . . . . . . . . . . . . . . .
1.3. Sobre lo que Trata esta Tesis . . . . . . . . . . . . . . . . . . . . . . . .
1
1
3
4
2. Las Membranas
7
2.1. El Modelo del Mosaico Fluı́do de Singer y Nicolson . . . . . . . . . .
7
2.2. Lı́pidos de Membranas . . . . . . . . . . . . . . . . . . . . . . . . . . .
8
2.2.1. Naturaleza de los Lı́pidos . . . . . . . . . . . . . . . . . . . . .
8
2.2.2. Estructura de Lı́pidos en Solución Acuosa . . . . . . . . . . . 12
2.2.3. Fluidez de Membranas y Movimientos de los Lı́pidos en Bicapas 14
2.2.4. Transiciones de Fases . . . . . . . . . . . . . . . . . . . . . . . . 19
3. Las Proteı́nas
3.1. Aminoácidos: Los Bloques Constituyentes de las Proteı́nas . .
3.1.1. Representaciones Estereoquı́micas de los Aminoácidos
3.1.2. Uniones Péptidas . . . . . . . . . . . . . . . . . . . . . .
3.2. La Estructura Tridimensional de las Proteı́nas . . . . . . . . . .
3.2.1. Secuencia o Estructura Primaria . . . . . . . . . . . . .
3.2.2. Estructura Secundaria . . . . . . . . . . . . . . . . . . .
3.3. Otras Conformaciones Hélice . . . . . . . . . . . . . . . . . . .
3.3.1. Estructura Terciaria . . . . . . . . . . . . . . . . . . . . .
3.3.2. Estructura Cuaternaria . . . . . . . . . . . . . . . . . . .
V
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
27
27
27
28
32
32
32
34
38
39
4. El Péptido β Amiloide y La Enfermedad del Alzheimer
4.1. El Péptido Aβ . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . .
4.2. La Agregación en Superficies Lipı́dicas . . . . . . . . . . . . . . . . . .
4.3. Resultados Recientes . . . . . . . . . . . . . . . . . . . . . . . . . . . .
41
42
43
44
5. La Dinámica Molecular
5.1. Haciendo Fı́sica in Silico . . . . . . . . . . .
5.1.1. Los Campos de Fuerza . . . . . . . .
5.1.2. Algoritmos de Integración . . . . . .
5.2. Definición de las Propiedades del Ensamble
5.3. Limitaciones . . . . . . . . . . . . . . . . . .
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
47
47
49
54
56
58
6. Las Simulaciones
6.1. Configuración de los sistemas . . .
6.2. NAMD . . . . . . . . . . . . . . . .
6.2.1. Sistema Aβ40 -DPPC-Agua .
6.2.2. Sistema Aβ40 -DOPC-Agua
6.3. GROMACS . . . . . . . . . . . . . .
6.3.1. Sistema Aβ40 -DPPC-Agua .
6.3.2. Sistema Aβ40 -DOPC-Agua
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
61
61
62
62
65
66
66
67
7. Los Resultados
7.1. Sistemas A1, A2 y A3 . . . . . . . . . . . . . . . . . . . . . . . . . . . .
7.2. Sistemas B1 y B2 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . .
69
69
74
Conclusiones
77
Apéndice A
79
Bibliografı́a
81
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
.
A la memoria de mi madre Marı́a Guadalupe Rosales
Resumen
El péptido β amiloide (Aβ) es el mayor componente encontrado en los depósitos fibrilares en los cerebros de pacientes con la enfermedad de Alzheimer. El Aβ es
un péptido que consiste en una secuencia de aminoácidos que puede ir desde 39 a
43 residuos y que es producido a partir del proceso normal de división de la proteı́na de membrana conocida como proteı́na precursora de amiloide (APP por sus
siglas en inglés). Sin embargo, cuando se ensambla para formar agregados fibrilares,
el Aβ adopta una estructura secundaria de tipo hoja β, caracterı́stica de los depósitos fibrilares amiloides en pacientes con Alzheimer. De esta manera, la formación de
agregados citotóxicos de Aβ requiere que éste experimente cambios conformacionales para adquirir la estructura secundaria tipo hoja β. Debido a la gran insolubilidad
de las fibras de amiloide, ha resultado imposible visualizar experimentalmente los
cambios conformacionales del Aβ con detalle atómico.
En este trabajo se emplea la técnica de simulación por dinámica molecular para
estudiar los cambios conformacionales del péptido Aβ en dos membranas formadas
por fosfolı́pidos diferentes, uno satudado y el otro insaturado. Esto para ver el efecto
del empaquetamiento de las colas hidrofóbicas sobre el péptido.
Se usaron dos paquetes de software de dominio público, NAMD y GROMACS,
que presentan algunas diferencias en cuanto a las técnicas y métodos que emplean
en sus algoritmos. También se empleó un campo de fuerzas distinto en cada uno
de estos paquetes, CHARMM27 en el caso de NAMD y una versión modificada de
GROMOS en GROMACS.
Encontramos que el grado de empaquetamiento, evaluado a partir del área por
lı́pido, juega un papel importante en la evolución de la estructura secundaria del
péptido, presentando éste mayor desarrollo conformacional cuando el modelo de
membrana simulado reproduce mejor el área por lı́pido experimental de la membrana sin péptido.
IX
Abstract
The amyloid β-peptide (Aβ) is a major component found in the amyloid deposits in the brains of Alzheimer’s disease patientes. Aβ is a peptide which can range
in lenght from 39 to 43 residues and is derived from secuential proteolytic cleavage
of the amyloid precursor protein (APP). However, when it assembles to form fibrillar aggregates, the Aβ adopts a secondary structure of type β-sheet, characteristic
of amyloid fibrillar deposits in Alzheimer’s patients. In this way, the formation of
cytotoxic aggregates of Aβ requires a conformational transition to rich β-sheet secondary structure. Because of the amyloid fibril’s extreme insolubility, investigation
of the conformational conversions of Aβ in the atomic detail by means of experimental methods is still intractable.
In the present work we employ molecular dynamics simulations to study the conformational change of Aβ in two different membranes composed from two different
lipids, one saturated and the other insaturated . This is because we want to see the
packing effect of the hydrophobic tail over the peptide.
We used two software packages of public domain, NAMD and GROMACS, that
present some differences concerning the techniques and methods employed in their
algorithms. Also we employed different force fields in these packages, CHARMM27
in the case of NAMD and a modified version of GROMOS in GROMACS.
We found that the degree of packing, evaluated from the area-per-lipid, plays an
important role en the evolution of the secondary structure of the peptide. Aβ presents more conformational development when the model of membrane simulated
reproduces in a better way the experimental area-per-lipid of the membrane without
peptide.
XI
Capı́tulo 1
INTRODUCCIÓN
A biophysicist talks physics to the biologists and biology to
the physicists, but then he meets another biophysicist, they
just discuss women.
Author Unknown
1.1.
La Vida a Nivel Molecular
a biologı́a como sabemos está caracterizada por una increı́ble diversidad de especies, que van desde los enormes mamı́feros a las bacterias microscópicas que
pueden sobrevivir muy por encima del punto de ebullición del agua, sin mencionar
a las plantas. Incluso es más fascinante que esos animales y plantas tienen esencialmente en común sus funciones y estructuras jerárquicas básicas, es decir están
todas constituı́das de células, las unidades independientes más pequeñas clásicamente consideradas para “vivir”, ya sea en la forma de estructuras libres (e.g., bacterias) o como organismos multicelulares. El tamaño de un óvulo humano es del
orden de mm, apreciándose a simple vista. Una bacteria es casi 100 veces más pequeña, mientras que las células nerviosas pueden ser de un metro o más.
Hemos aprendido mucho acerca de la ciencia y biologı́a celular en particular
durante el último siglo; a través del desarrollo de mejores microscopios ópticos y
electrónicos ahora sabemos que dentro de estas estructuras hay elementos incluso
más pequeños como los ribosomas, la mitrocondria y el núcleo con cromosomas
conteniendo nuestro material genético. Esto ha llevado al nacimiento de la biologı́a
molecular que concierne a las moléculas individuales mediadoras de todos los procesos de la vida o escalas nanométricas. En febrero del 2001 el Consorcio de la Secuencia del Genoma Humano y Genómica Celera publicó sus borradores del mapa
de nuestro código genético, colocando a la biologı́a en la era de la información y
marcando el comienzo de una nueva era post-genómica. La ciencia se ha convertido
L
2
1. Introducción
Figure 1.1: La estructura quı́mica de 1,2-dipalmitoil-sn-glicero-3-fosfatidilcolina, también conocida como dipalmitoilfosfatidilcolina, o simplemente “DPPC”. Las colas están dibujadas
como lı́neas en zig-zag y cada vértice representa un grupo CH2 . Es in lı́pido relativamente
tı́pico presente en sistemas biológicos y frecuentemente elegido para simulaciones dado que
los sistemas de DPPC puro están bien documentados experimentalmente.
en un camino muy largo, pero seguimos lejos de la meta del completo entendimiento de las propiedades de los procesos de la vida a partir de la quı́mica y la fı́sica de
las partı́culas participantes.
Una diferencia esencial entre la quı́mica de organismos no vivientes y vivientes
es la mayor complejidad de las moléculas biológicas. Además de los compuestos
orgánicos e inorgánicos más pequeños, las células contienen ensambles macromoleculares complejos que consisten de cientos a millones de átomos. El más famoso es ciertamente el ácido desoxirribonucleico, ADN, en el cual el código genético
está guardado como una secuencia de bases. El diseño único de cada organismo
vivo está descrito en la información de este modelo genético, el cual es usado para sintetizar las proteı́nas en los ribosomas (una parte de la célula). Las proteı́nas
son el caballo de batalla de las células, por ejemplo la fotosı́ntesis en las plantas y
las enzimas digestivas las cuales degradan la comida a compuestos simples. Otros
ejemplos son las proteı́nas de los músculos que producen trabajo mecánico a partir
de reacciones quı́micas y proteı́nas de transporte, las cuales mueven moléculas como el oxı́geno a su sitio de utilización.
Una de las moléculas más frecuentes en esta tesis son los lı́pidos, los cuales son
el principal componente de todas las membranas celulares (ver Figura 1.1).
Estas moléculas relativamente pequeñas tienen una “cabeza” dipolar o algunas
veces cargada que es soluble en agua (hidrofı́lica) y una o dos colas de hidrocarburos
extendidas a las cuales no les “gusta” el agua (hidrofóbica). A mayores concentraciones, esto las hace agregarse y colocar las partes hidrofóbicas una frente a la otra,
mientras que las cabezas solubles están en contacto con el ambiente acuoso. Dependiendo de los tamaños relativos de la cabeza y las colas, se pueden formar micelas
o bicapas.
La membrana exterior que envuelve a la célula de su ambiente es tal vez la es-
1.2. De la Estructura a la Dinámica
3
Figure 1.2: La constitución de la membrana celular. Tı́picamente un lı́pido tiene una cabeza
de átomos parcialmente cargados solubles en agua y una o varias cadenas de hidrocarburos.
La longitud de la molécula es de alrededor de 2 nm. Cuando se agregan pueden formar bicapas estables con un espesor de alrededor de 5 nm orientando las cadenas de frente entre sı́.
Porciones mayores de membranas biológicas usualmente también contienen otras moléculas
como proteı́nas. Estas estructuras son responsables de todas las paredes e interfaces en las
células vivas.
tructura más importante (Figura 1.2), pero todas las membranas definen diferentes
compartimentos en la célula.
Ellas determinan la naturaleza de toda comunicación a través de la interfase
y funcionan como dispositivos de detección, permitiendo y algunas veces inclusive asistiendo la penetración de algunas moléculas pero no otras. Además, proveen
apoyo a las proteı́nas de membrana las cuales trasportan, por ejemplo, iones hacia
adentro y hacia afuera de la célula. Esta función de barrera es crucial para la actividad biológica de la célula; los antibióticos ordinarios usados para combatir bacterias
se adhieren a las proteı́nas que se encuentran en las membranas bacterianas (pero
no en las humanas) y destruyen esta barrera aumentando la permeabilidad de la
membrana, matando ası́ a la bacteria.
1.2.
De la Estructura a la Dinámica
Las primeras estructuras experimentales de proteı́nas y ADN fueron derivadas
de formas cristalizadas de biomoléculas. La cristalografı́a de rayos X sigue siendo
el método disponible de más alta resolución, pero está limitada a proporcionar una
estructura promedio de la molécula en un ambiente muy diferente al de una célula
viva. También ha sido conocido durante mucho tiempo a partir de diversos estudios experimentales que las biomoléculas exhiben fluctuaciones sustanciales que
1. Introducción
4
son esenciales para su actividad biológica y no son capturadas en sus estructuras
promedio. De hecho, esta es una de las principales razones considerablemente menos conocidas acerca de las propiedades detalladas de las membranas biológicas
comparadas por ejemplo al ADN y las proteı́nas; éstas no tienen ninguna estructura de equilibrio bien definida que sea fácil de determinar. Con el avance de nuevos
métodos experimentales como la espectrocopı́a láser y NMR (Nuclear Magnetic Resonance) que también funciona con muestras en solución, mayor atención sin embargo,
ha sido enfocado a la rápida dinámica colectiva e interacciones de macromoléculas
biológicas.
En la mayorı́a de las estructuras biomoleculares ha sido encontrado también que
son fuertemente influenciadas por el medio que las rodea, usualmente el agua el
cual constituye cerca de 70 % de todas las células. Las moléculas de agua cercanas
a superficies como el ADN, proteı́nas o membranas están vinculadas estrechamente a las estructuras, y algunas veces mejor caracterizadas por ser parte de ellas en
lugar de la mayor parte del solvente. La mayorı́a de los procesos biológicos no funcionarı́an sin las moléculas de solvente. Esta importancia viene de las propiedades
muy especiales del agua como un medio altamente polar que apantalla las interacciones moleculares y su red rı́gida de puentes de hidrógeno incluso en la fase
lı́quida.
Nuestro conocimiento de la dinámica biomolecular sigue siendo muy limitado,
aunque es un campo de investigación muy activo. Existen métodos experimentales que pueden resolver átomos individuales, pero usualmente no son capaces de
cuantificar directamente movimientos colectivos complejos. Los métodos indirectos como la espectroscopı́a pueden algunas veces ser aplicados en combinación con
modelos teóricos para los movimientos, pero este enfoque está lejos de ser generalmente aplicable.
1.3.
Sobre lo que Trata esta Tesis
La anterior introducción podrı́a no ilustrar acerca de lo que realmente he estado
haciendo durante estos meses. ¿Dónde entran la fı́sica, las computadoras y las simulaciones?
La mayorı́a de las ciencias experimentales usualmente yacen en un enfoque de
arriba a abajo, es decir, las mediciones son refinadas gradualmente para ser capaces
de observar estructuras más pequeñas y procesos más rápidos hasta que se alcancen los lı́mites técnicos. Si tenemos acceso a computadoras muy potentes se podrı́a
revertir este algoritmo y hacer un modelado de abajo hacia arriba. Esta es la idea central en esta tesis, comenzado de interacciones en pares de átomos, se pueden utilizar
a las computadoras para simular lo que sucede en moléculas biológicas complejas
1.3. Sobre lo que Trata esta Tesis
5
a escalas de tiempo mayores. De esta manera es posible ver movimientos atómicos a un nivel usualmente no accesible para realizar experimentos. El conocimiento
obtenido puede ser utilizado para formular mejores modelos del fenómeno observado. La dinámica molécular está basada en las ideas anteriores y se ha utilizado
para tratar de entender qué sucede a nivel molecular en los inicios de una enfermedad llamada Alzheimer. Esta es la forma más común de demencia, es incurable,
degenerativa y terminal.
Capı́tulo 2
LAS MEMBRANAS
In Darwin’s day, the cell was basically a little blob of Jell-O
enclosed by a membrane. That’s why Darwin didn’t write
about the origin of life; he wrote about the origin of species.
William Dembski
2.1.
El Modelo del Mosaico Fluı́do de Singer y Nicolson
a caracterı́stica esencial del modelo de mosaico fluı́do es que las membranas
biológicas se consideran como estructuras fluı́das cuasiestáticas en las cuales los
lı́pidos y las proteı́nas integrales están acomodadas en forma de mosaico. El modelo
del mosaico fluı́do de Singer y Nicolson (1972), Figura 2.1, ahora es ampliamente
aceptado como el mejor para explicar las propiedades de la membrana celular. Éste
asume que hay una bicapa continua de fosfolı́pidos en la cual están incrustadas proteı́nas globulares.
Las proteı́nas han sido comparadas con icebergs flotando en el mar de la bicapa de
fosfolı́pidos. En el modelo de Danielli Davson se asume un enlace hidrofı́lico entre
los lı́pidos y las proteı́nas, el modelo de Singer y Nicolson considera que la asociación lı́pido-proteı́na es hidrofóbica. La fluidez de la membrana es el resultado de esta
interacción hidrofóbica. Nótese que los fosfolı́pidos y muchas proteı́nas intrı́nsecas
son moléculas anfipáticas, es decir poseen un extremo hidrofı́lico o sea que es soluble en agua y otro hidrófobo o sea que rechaza el agua.
Las proteı́nas globulares de la membrana se consideran de dos tipos, proteı́nas
extrı́nsecas (periféricas) y proteı́nas intrı́nsecas (integrales). Las proteı́nas periféricas son solubles y fácilmente se disocian de la membrana.
L
2. Las Membranas
8
Figure 2.1: El modelo del mosaico fluı́do de Singer y Nicolson (1972).
2.2.
Lı́pidos de Membranas
El marco estructural básico de las membranas biológicas esta conformado por
lı́pidos, siendo los componentes mayoritarios los lı́pidos anfifı́licos conocidos con el
nombre de fosfolı́pidos, moléculas que presentan una cabeza polar y una porción
no polar formada por ácidos grasos de cadena larga. Ellos son los responsables de la
formación de la bicapa la cual a su vez permite la formación de vesı́culas lipı́dicas.
La tendencia de esta doble cadena anfifı́lica de formar una bicapa en soluciones
acuosas es la propiedad fı́sica crucial que determina que se forme una membrana
y permite de esa manera separar los componentes encapsulados de los que quedan
fuera de la vesı́cula.
2.2.1.
Naturaleza de los Lı́pidos
Las membranas biológicas poseen una gran diversidad de lı́pidos de los cuales los componentes principales son los fosfolı́pidos, los glicolı́pidos y el colesterol.
Según el modelo del mosaico fluı́do, los lı́pidos de la membrana suministran un medio fluı́do que permite los movimientos de las proteı́nas de la membrana. Sin embargo, si este fuera el único papel fisiológico de los lı́pidos, bastarı́a con unas pocas
especies moleculares para asegurar un medio fluı́do y no serı́a necesaria la existencia de la gran variedad de especies que se encuentran en las membranas biológicas,
2.2. Lı́pidos de Membranas
9
ya que se han identificado en membranas naturales hasta 1000 moléculas distintas de lı́pidos. Por lo tanto, es posible suponer que ellas cumplen otras funciones
adicionales, aunque no se conocen las funciones especı́ficas desempeñadas por las
distintas clases de lı́pidos, ni se sabe bien porqué y cómo las células mantienen su
contenido y composición lipı́dica relativamente constante. Un ejemplo importante
del avance en el conocimiento de la función de lı́pidos individuales lo constituye
el descubrimiento de que el fosfatidilnositol y sus derivados fosforilados juegan un
papel crucial como precursores del inositol 1,4,5-trisfosfato, un segundo mensajero
que induce liberación de calcio de reservorios intracelulares y que se produce en
respuesta a una gran variedad de estı́mulos internos.
Fosfolı́pidos
Los fosfolı́pidos más comunes son derivados del glicerol, por ello se denominan
glicerofosfolı́pidos o fosfoglicéridos. La estructura general de un glicerofosfolı́pido
(Figura 2.2) se caracteriza por tener esterificados con ácidos grasos los hidroxilos de
los carbones 1 y 2 de la molécula de glicerol, (llamadas posiciones sn-1 y sn-2). Los
ácidos grasos presentes en membranas biológicas pueden tener cadenas de 12 a 26
carbones de largo, con hasta 6 enlaces dobles cis. Sin embargo, los fosfolı́pidos que
se encuentran más frecuentemente en membranas biológicas poseen ácidos grasos
de 14 a 24 carbones y hasta seis enlaces dobles cis (Tabla 2.1). En los lı́pidos naturales
no se encuentran enlaces dobles trans. El hidroxilo en la posición sn-3 de la molécula
de glicerol establece un enlace covalente con un fosfato, lo que le confiere una carga
negativa. El fosfato a su vez forma una unión covalente con una base, la cual puede aportar cargas a la molécula ó tener carga neta cero, de tal modo que la carga
resultante de la molécula de fosfolı́pido será la suma algebraica entre la carga negativa del fosfato y la carga neta de la base. Los glicerofosfolı́pidos mayoritarios de las
membranas biológicas son la fosfatidilcolina, la fosfatidiletanolamina; en menores
cantidades se encuentran la cardiolipina, fosfatidilserina y fosfatidilnositol.
Basándonos en la abundancia de fosfatidilcolina en las membranas biológicas es
el tipo de fosfolı́pido que elegimos para las simulaciones que se presentarán más
adelante. Ya en el primer capı́tulo brindamos una ligera descripción del DPPC por
lo que ahora mostrarémos el otro fosfolı́pido que se utilizó en este trabajo que es el
DOPC y cuya estructura quı́mica se aprecia en la Figura 2.3.
Tomando en cuenta la Tabla 2.1 nos percatamos de que el DPPC tiene ácidos
grasos saturados1 y simétricos, mientras que los ácidos grasos del DOPC son insaturados y simétricos.
1 Un
compuesto saturado no tiene enlaces doble ni triples.
10
2. Las Membranas
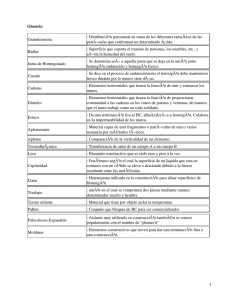
Figure 2.2: Fosfolı́pidos. A: fosfatidilcolina; B: fosfatidiletanolamina; C: fosfatidilserina; D: representación esquemática de un fosfolı́pido con la cabeza hidrófilica (1) y las colas hidrofóbicas (2). Obtenido de: Ácido graso. (2008, 31) de octubre. Wikipedia, La enciclopedia libre.
Glicolı́pidos
Podemos definir otra familia de lı́pidos de membrana, los glicolı́pidos, que se
clasifican como tales por poseer grupos glicosilados, y que en general se encuentran
presentes en menor cantidad en las membranas biológicas que los fosfolı́pidos. Los
glicolı́pidos presentan una distribución altamente asimétrica en membranas biológicas, ya que sólo se encuentran en la superficie externa de las membranas plasmáticas. En tanto los glicolı́pidos de bacterias y plantas son casi todos gliceroglicolı́pidos, vale decir que poseen como base estructural la molécula de glicerol, la mayorı́a
Figure 2.3: La estructura quı́mica de 1,2-dioleoil-sn-glicero-3-fosfatidilcolina, también conocida como dioleoilfosfatidilcolina, o simplemente “DOPC”. Las colas están dibujadas como
lı́neas en zig-zag y cada vértice representa un grupo CH2 .
2.2. Lı́pidos de Membranas
11
Sı́mbolo numérico (*)
Nombre Sistemático
Nombre Común
14:0
16:0
16:1(9)
18:0
18:1 (9); (18:1 (ω-9))
18:2 (9,12); (18:2(ω-6))
18:3 (9,12,15); (18:3(ω-3))
20:0
20:4 (5,8,11,14); (20:4(ω-6))
22:1(13)
22:6(4,7,10,13,16,19); (22:6(ω-3))
24:0
24:1(15); (24:1 (ω-9))
tetradecanoico
hexadecanoico
9-hexadecanoico
octadecanoico
9-octadecenoico
9,12-octadecadienoico
9,12,15-octadecatrienoico
docosanoico
5,8,11,14-eicosatetraenoico
13-docosoenoico
4,7,10,13,16,19-docosahexaenoico
tetracosanoico
15-tetracosenoico
mirı́stico
palmı́tico
palmitoleico
esteárico
oleico
linoléico
α-linolénico
behénico
araquidónico
erúcico
cervónico
lignocérico
nervónico
Table 2.1: Ácidos grasos comunes presentes en membranas vegetales y animales.
∗
El sı́mbolo numérico está representado de modo tal que el primer número corresponde al
número de átomos de carbono, lo que sigue es el número de enlaces dobles, entre paréntesis
se indican las posiciones de éstos. En el caso de ácidos grasos insaturados, se indica además
la nomenclatura alternativa que señala la posición del primer enlace doble a partir del grupo
metilo terminal de la cadena.
de los glicolı́pidos presentes en las membranas de células animales son esfingoglicolı́pidos, ya que en ellos la base estructural es la molécula de esfingosina. Entre
los esfingoglicolı́pidos más comunes se encuentran los cerebrósidos, que contienen
una hexosa neutra como base (por ejemplo, los esfingoglicolı́pidos de la mielina poseen galactosa) y los gangliósidos, con carga neta negativa, en los cuales la base es
un oligosacárido complejo que contiene una o más moléculas de ácido siálico. Los
gangliósidos son especialmente abundantes en la superficie celular de las neuronas;
su función celular no se conoce exactamente, aunque numerosos estudios indican
que están involucrados en diferenciación celular y morfogénesis. Más aún, se han
identificado como sitios de unión para virus, bacterias y toxinas, y parecen estar implicados en la adhesión celular tipo especı́fica.
Colesterol
El tercer componente más abundante en membranas biológicas, además de fosfolı́pidos y glicolı́pidos, es el colesterol (Figura 2.4). Este compuesto, aunque no se
encuentra en bacterias y es poco frecuente en membranas intracelulares tales como
2. Las Membranas
12
las de mitocondrias, núcleo y retı́culo endoplasmático es un componente importante
de las membranas plasmáticas de las células animales.
Figure 2.4: Estructura de la molécula de colesterol. El carácter polar de un extremo de la
molécula lo confiere el grupo OH; el resto de la molécula es no polar y tiene una configuración
plana dada por los cuatro anillos hidrocarbonados.
2.2.2.
Estructura de Lı́pidos en Solución Acuosa
Todos los lı́pidos de las membranas biológicas se caracterizan por poseer una
estructura anfifı́lica; parte de la molécula contiene grupos que le confieren carácter
polar y el resto de la molécula es no polar. Esta caracterı́stica de los lı́pidos los hace
muy poco solubles en agua, de tal modo que al superar una concentración que se
conoce como concentración micelar crı́tica, o c.m.c., los lı́pidos se asocian en estructuras que exponen los grupos polares al medio acuoso y permiten las interacciones de
los grupos no polares entre si, evitando su interacción con el agua. Se conoce este
fenómeno con el nombre del efecto hidrófobo.
La Bicapa Como Estructura de Membranas Biológicas
Las diversas estructuras que adoptan los conjuntos de las moléculas de lı́pidos
en solución acuosa se ilustran en la Figura 2.5. Se aprecia en todas ellas que el con-
2.2. Lı́pidos de Membranas
13
Figure 2.5: Estructuras que adoptan los diversos tipos de lı́pidos en soluciones acuosas a
concentraciones mayores que la concentración micelar crı́tica, c.m.c..
junto expone hacia el medio acuoso las cabezas polares y esconde las regiones no
polares de los lı́pidos en una fase diferente. El tipo de estructura que adoptan las
moléculas de fosfolı́pidos en solución acuosa está determinado por la conformación
geométrica de la molécula y la temperatura. Las membranas biológicas tienen sus
lı́pidos organizados en la estructura de bicapa y se supone que diferentes estructuras moleculares se complementan para conferir mayor estabilidad al sistema.
Estudios de cristales hidratados de fosfolı́pidos saturados, utilizando la técnica
de la difracción de rayos X, indican que la cadena sn-1 está extendida en la configuración todo trans y la región inicial de la cadena sn-2 es paralela a la superficie
de la bicapa, luego la cadena se dobla en el segundo segmento y se hace paralela
a la cadena sn-1 con un desplazamiento de aproximadamente 3 grupos metileno.
Esta estructura de los fosfolı́pidos, que también fué predicha por estudios de resonancia magnética nuclear de deuterio y por difracción de neutrones, existe también
en membranas biológicas, lo que trae como consecuencia que las cadenas de la posición sn-2 tengan un recorrido más largo que las de posición sn-2, lo que tiende a
disminuir la diferencia en largo efectivo de las cadenas. Con base en experimentos
hechos usando lı́pidos que contienen sondas paramagnéticas, se ha propuesto que
para compensar las distintas longitudes de cadenas los fosfolı́pidos se interligan en
el seno de bicapa.
Las cabezas polares de fosfatidilcolina y fosfatiletanolamina, a su vez, tiene una
14
2. Las Membranas
orientación que en promedio es paralela a la superficie de la bicapa tanto en el estado gel como en el lı́quido cristalino. En cambio, los galactocerebrósidos tienen su
cabeza polar perpendicular a la superficie de la bicapa y se proyectan hacia la fase
acuosa. Como lo han indicado estudios de resonacia magnética nuclear de deuterio,
usando los fosfolı́pidos marcados con 2 H en la cabeza polar, la rotación interna de
las cabezas polares esta restringida y es relativamente independiente de la composición de las colas hidrofóbicas. Las cabezas polares cargadas o con carga neta cero
tienen la potencialidad de establecer enlaces de H entre ellas, ya que poseen grupos
donadores tales como los grupos amino y grupos aceptores como los grupos fosfato.
2.2.3.
Fluidez de Membranas y Movimientos de los Lı́pidos en Bicapas
Los lı́pidos presentes en las membranas biológicas tienen la capacidad de experimentar movimientos traslacionales y rotacionales en el plano de la bicapa (Figura
2.6), lo que hace que la bicapa constituya un fluı́do bidimensional. La disipación
de la energı́a cinética de una molécula hacia el medio que la rodea es función de
la fluidez del medio. Podemos en este contexto definir la fluidez de una membrana
como la propiedad que indica con qué facilidad es posible el movimiento (traslacional, rotacional y vibracional) dentro de ella. Es importante destacar que el concepto
de fluidez se refiere especı́ficamente a las propiedades de la región hidrófoba de la
membrana. Por lo tanto, una descripción cuantitativa de la fluidez debe comprender
los movimientos de los lı́pidos en cuanto a velocidad de movimiento de las cadenas y la orientación de ellas con respecto al plano de la bicapa, sin considerar el
movimiento de las cabezas polares. Una membrana muy fluı́da permite mayor movimiento dentro de ella. La fluidez de una membrana es una propiedad macroscópica resultante de la suma de las propiedades individuales de sus componentes y se
podrı́a considerar que tiene una relación inversa a su viscosidad, con la salvedad de
que se trata de un lı́quido anisotrópico.
CONCEPTOS GENERALES. Durante la última década se han estudiado los
movimientos de los lı́pidos tanto en modelos de membrana como en membranas
naturales. Estos estudios se han realizado mediante el empleo de técnicas tales como
la resonancia paramagnética del electrón, la resonancia magnética nuclear, la espectroscopı́a Raman y la fluorescecia. Aunque aún hay alguna controversia acerca de la
interpretación de los resultados obtenidos con estas técnicas, no hay ninguna duda
que ellas han contribuı́do de manera fundamental al entendimiento de la estructura
y dinámica de las bicapas de lı́pidos.
Estos estudios distinguen dos tipos de movimientos de los lı́pidos. El primero
2.2. Lı́pidos de Membranas
15
Figure 2.6: Modelos posibles de movimientos de los lı́pidos en una bicapa. Una molécula de
lı́pido puede, como un todo, moverse lateralmente en el plano de cada monocapa con un coeficiente de difusión traslacional DT del orden de 10−7 cm2 /s y con una frecuencia de salto νj
entre posiciones adyacentes de 108 s−1 . También puede rotar entorno a su eje mayor con un
coeficiente de difusión rotacional DRk y puede efectuar rotaciones limitadas entorno a un eje
perpendicular al plano de la bicapa, con un coeficiente de rotación perpendicular DR⊥ . Las
moléculas de lı́pidos tambien pueden experimentar movimientos de flip-flop de una monocapa a la otra con una frecuencia de eventos, τf−1 , extremadamente baja que alcanza valores de
10−5 s−1 . Los movimientos intramoleculares corresponden a isomerizaciones trans/gauche
entorno a enlaces individuales C-C con una frecuencia media de τj−1 de 1010 s−1 .
concierne a los movimientos intramoleculares de una parte de la molécula con respecto a otra, como por ejemplo rotaciones entorno a enlaces simples y el movimiento
pendular de las cadenas de hidrocarburos. El otro tipo de movimiento es aquel que
involucra a la molécula de lı́pido como un todo y que pueden ser movimientos de
traslación o de rotación. Las moléculas de lı́pidos se pueden trasladar de un punto
a otro en una misma monocapa, fenómeno que puede considerarse como una difusión en un plano. El lı́pido también puede moverse de una monocapa a otra. En
este caso el movimiento involucra traslación y rotación en torno a un eje ya que no
podemos dejar la cabeza polar de lı́pido en el interior de la bicapa. Este movimiento
ha recibido el nombre de flip-flop.
MOVIMIENTOS INTRAMOLECULARES. Estos movimientos incluyen rotaciones entorno a un enlace carbono-carbono simple en un n-alcano. Tomemos el
caso del n-butano como se ilustra en la Figura 2.7. Existen tres conformaciones posibles entre si en 120◦ y que poseen aproximadamente la misma energı́a potencial; sin
embargo las rotaciones no se hacen libremente, necesitándose aproximadamente 3.5
kcal/mol para la interconversión entre una conformación y otra. Estas tres configu-
16
2. Las Membranas
Figure 2.7: Rotaciones del n-butano. A: anti (an); B: gauche-más (g+ ); C: gauche-menos (g− ).
raciones alternadas se denominan: anti (an) o trans (t); gauche - más (g+ o izquierda
- más), y gauche -menos (g− o izquierda - menos). Debido a que la energı́a de interconversión entre estas tres conformaciones no es muy alta, la energı́a térmica es
suficiente para causar rápidas rotaciones en la molécula de manera tal que en cualquier momento podremos encontrar cualquiera de ellas en forma alternada.
En la Figura 2.8 se observa, como los cambios conformacionales descritos anteriormente se traducen en movimientos de las cadenas de hidrocarburos de los
lı́pidos. Para un ácido graso saturado en la conformación todo trans (Figura 2.8A),
una transformación del carbono 9 de anti (an) a gauche - más (g + ) produce un movimiento pendular de la cadena de hidrocarburo (Figura 2.8B). Sin embargo si se
produce en forma consecutiva primero una transformación de an a g + seguida por
una rotación g − , la molécula de lı́pido toma la estructura que se muestra en la Figura
2.8C. Este tipo de cambio conformacional trae como consecuencia un acortamiento
de la cadena en un CH2, lo que equivale a una distancia de 0.127 nm. En promedio,
en una bicapa el número de “distorsiones” de este tipo por cadena varı́a entre dos y
siete, dependiendo de la fluidez de la bicapa.
Es interesante hacer notar aquı́ que rotaciones como las descritas anteriormente
no ocurren en torno a un enlace doble. Sin embargo la presencia de un enlace doble en la cadena disminuye la energı́a necesaria para la rotación del enlace simple
contiguo a solo 2 kcal/mol. La inserción de un enlace doble produce una distorsión
en la conformación de la molécula, como se indica en la Figura 2.8D para un ácido
graso con un enlace doble cis entre los carbones 9 y 10. Si el carbono en la posición siguiente, en este caso el carbono 11, rota a su vez se produce un quiebre denominado
∆tg (Figura 2.8E), esto trae como consecuencia que la molécula ocupe un volumen
2.2. Lı́pidos de Membranas
17
Figure 2.8: A) Una molécula de ácido graso en la conformación todo trans, B) Una transformación del carbono 9 de anti (an) a gauche más (g+ ) produce un movimiento pendular
de la cadena de hidrocarburo, C) sin embargo si se produce en forma consecutiva primero
una transformación de an a g+ seguida por una rotación a g− , la molécula de lı́pido toma
la estructura indicada, D) La inserción de un enlace doble cis produce una distorsión en la
conformación de la molécula; la figura ilustra el caso para un ácido graso con un enlace doble
cis entre los carbones 9 y 10, E) Si el cambono siguiente, en este caso el carbono 11, rota a
su vez se produce un quiebre ∆tg, lo que trae como consecuencia que la molécula ocupe un
volumen menor.
menor, lo que incide directamente en un mayor empaquetamiento de la bicapa.
Se incluye también entre los movimientos intramoleculares aquellos que comprometen varios átomos de carbono a la vez. Los estudios efectuados mediante el
empleo de la táctica de resonancia magnética nuclear indican que los segmentos de
la cadena cercanos al grupo polar están altamente restringidos en sus movimientos, pero a medida que avanzamos hacia el interior de la membrana la posibilidad
de movimientos perpendiculares que comprometen varios átomos de carbono a la
vez se hacen cada vez más frecuentes. En otras palabras, a medida que nos vamos
acercando al centro de la bicapa esta se va haciendo más y mas fluı́da. Se desprende de estas observaciones que, la bicapa se puede homologar a un lı́quido en dos
dimensiones, que además es anisotrópico y presenta una fluidez creciente desde la
superficie hacia el centro de la bicapa.
MOVIMIENTOS TRASLACIONALES. Los movimientos traslacionales de las
2. Las Membranas
18
moléculas de lı́pidos pueden ser de dos clases:
a) Movimientos en el plano de la membrana.
b) Paso de lı́pidos de una monocapa a otra (flip-flop).
Es claro que en el segundo caso la cabeza polar del lı́pido debe atravesar la parte
hidrófoba de la membrana, lo que determina que el proceso de flip-flop conlleve un
gasto de energı́a mucho mayor que el movimiento de los lı́pidos en el plano de la
bicapa.
La difusión lateral de los lı́pidos se ha estudiado mediante el empleo de las técnicas de resonancia paramagnética del electrón, resonancia magnética nuclear y fluorescencia. Se ha determinado experimentalmente que el valor del coeficiente de difusión lateral (DT ) de los lı́pidos está en el intervalo de 10−8 cm2 /s a 10−7 cm2 /s,
dependiendo de la composición de la membrana, como por ejemplo de la presencia
de colesterol, que tiende a disminuir su valor.
Para una esfera de un radio r la relación Stokes-Einstein nos dice que el coeficiente de difusión traslacional DT en función de la viscosidad del medio η está dado por
la ecuación
DT = kB T /6πηr
(2.1)
donde T es la temperatura absoluta y kB es la constante de boltzmann.
Veamos ahora que significa en términos de tiempos y distancias difusionales
un valor de DT = 10−8 cm2 /s. Los tiempos y las distancias se pueden calcular
usando la ecuación de Einstein-Smoluchowki, que nos dice que la distancia media
al cuadrado (hxi2 ) que recorre una molécula en un tiempo t esta dada por
hxi2 = 2DT t
(2.2)
Por lo tanto, la molécula de lı́pido viaja a una distancia igual a 1 µm (10−4 cm) en:
t = 0,5 s
Si el diámetro de la cabeza polar de un lı́pido es aproximadamente 1 nm en medio
segundo la molécula de lı́pido se habrá movido una distancia 1000 veces superior a
su diámetro. En otras palabras, la molécula puede recorrer toda la longitud de una
bacteria tı́pica en medio segundo.
MIGRACIÓN DE LÍPIDOS A TRAVÉS DE LA BICAPA (FLIP/FLOP). ASIMETRÍA DE MEMBRANAS BIOLÓGICAS. La difusión de las moléculas de lı́pidos de una mitad de la bicapa a la otra es extremadamente lenta o simplemente
no ocurre en forma espontánea. En las membranas biológicas estos movimientos
de los lı́pidos se realizan utilizando sistemas enzimáticos especı́ficos. El proceso de
flip-flop de lı́pidos ha recibido considerable atención debido a las asimetrı́as en la
composición lipı́dica que muestran varios tipos diferentes de membranas naturales.
2.2. Lı́pidos de Membranas
19
En la monocapa externa de las membranas plasmáticas de eucariotas se encuentra
un mayor porcentaje de fosfatidilcolina y esfingomielina, mientras que la fosfatidiletanolamina y fosfatidilserina se encuentran preferencialmente en la monocapa
interna. Esta asimetrı́a ha llamado la atención porque se podrı́a esperar que desapareciera con el tiempo, especialmente en aquellas células que son incompetentes
desde un punto de vista de la biosı́ntesis como los eritrocitos. Una explicación posible para el mantenimiento de la distribución asimétrica de lı́pidos en la membrana
celular podrı́a ser que este proceso fuese lento. Esta hipótesis parece cumplirse en
modelos de membrana ya que en ellas estos movimientos son extremadamente infrecuentes. Sin embargo en membranas naturales el proceso de flip-flop puede tener
una vida media del orden de minutos. En aquellos sistemas en donde la migración
de los lı́pidos a través de la bicapa es un proceso rápido, se ha postulado que el
mantenimiento de la asimetrı́a se lleva a cabo en forma enzimática. Las enzimas
responsables de este proceso sólo se han descrito en algunos sistemas celulares, particularmente en eritrocitos. La asimterı́a de los lı́pidos le confiere a las membranas
biológicas un carácter vectorial que puede tener importancia funcional para establecer contactos entre membranas, como los que ocurren en procesos de fusión.
MOVIMIENTOS ROTACIONALES. Las moléculas de lı́pidos pueden efectuar
rotaciones en torno a su eje mayor con un coeficiente de difusión rotacional paralelo,
y pueden efectuar rotaciones limitadas en torno a un eje perpendicular al plano de
la superficie de la bicapa, con un coeficiente de difusión de rotación perpendicular,
como se ilustra en la Figura 2.6. Ambos coeficientes de difusión rotacional alcanzan
valores de 10−9 s−1 en bicapas fluidas. Para una esfera a un volumen V el coeficiente
de difusión rotacional DR se relaciona con la viscosidad del medio η a través de la
ecuación
DR = kT /6ηV = 1/tR
(2.3)
El tiempo de correlación rotacional tR esta dado por el inverso del coeficiente DR
y se expresa en segundos. De los valores experimentales obtenidos para los coeficientes en rotación difusional de 109 s−1 se pueden calcular valores de tiempos de
correlación rotacional para los lı́pidos de 10−9 s.
2.2.4.
Transiciones de Fases
Las moléculas de un fosfolı́pido puro presentan distintas estructuras que dependen del contenido de agua (mesomorfismo liotrópico) y de la temperatura (mesomorfismo termotrópico). En presencia de un exceso de agua, como ocurre en las
células, los lı́pidos puros pueden adoptar distintas configuraciones en función de la
temperatura, ya que la composición del sistema en términos de contenido de agua
está fija.
20
2. Las Membranas
Transiciones de Estados Gel a Lı́quido-Cristalino
La configuración de bicapa, que nos interesa por ser la que adoptan los lı́pidos en
las membranas biológicas, se caracteriza por poseer transiciones de fase, entendiendo como fase un agregado hidratado de fosfolı́pidos cuya formación esta dirigida
por el efecto hidrófobo. La mayor parte de los estudios que han permitido caracterizar las transiciones de fase se han realizado con la técnica de calorimetrı́a diferencial de barrido, aunque se han usado tambien otras técnicas, tales como resonancia
magnética nuclear y del electrón, rayos X, microscopı́a electrónica, espectroscopı́a
infrarrojo con transformadas de Fourier y fluorescencia. La calorimetrı́a diferencial
de barrido es un método termodinámico que permite medir parámetros tales como
temperatura de transición, entalpı́a y entropı́a de transición, en tanto la difracción
de rayos X y la microscopı́a electrónica permiten estudiar la estructura y las dimensiones de las fases. Las técnicas espectroscópicas, y en particular la resonancia
magnética nuclear, permiten estudiar los cambios de la orientación y la dinámica de
los lı́pidos que experimentan una transición de fase. La técnica de espectroscopı́a
infrarrojo con transformada de Fourier es sensible a movimientos más rápidos que
la resonancia magnética nuclear y la resonancia paramagnética del electrón, tales
como las rotaciones trans/gauche, y permite también detectar cambios de fase.
Mediante estos métodos aplicados fundamentalmente al análisis de la bicapa
de fosfolı́pidos, también denominada fase lamelar, se ha determinado que existen
dos fases principales: la fase gel, que ocurre a bajas temperaturas y la fase lı́quidocristalina, que se observa por sobre una temperatura denominada temperatura de
transición, Tt , que es caracterı́stica para cada especie molecular.
La transición de la fase gel a la lı́quido cristalina se debe a la ganancia de entropı́a
configuracional de las cadenas hidrocarbonadas que tiene lugar en el proceso de calentamiento; las cadenas de los lı́pidos poseen la tendencia a pasar de un estado
ordenado, en que están en la configuración todo trans formando un arreglo hexagonal, a uno desordenado como resultado de la ganancia de entropı́a que resulta del
isomerismo rotacional de las cadenas. En cambio la transición de la fase lı́quido cristalina a la fase hexagonal invertida es consecuencia de la tendencia a la curvatura
espontánea de la fase lamelar o de bicapa. Además de la temperatura, cambios en
la fuerza iónica o el pH del medio pueden inducir transiciones de fase debido a la
presencia de grupos ionizables en las cabezas polares que son muy sensibles a estos
cambios del entorno acuoso.
La transición de una bicapa de la fase gel a la fase lı́quido cristalina se acompaña de un aumento de la distancia entre las moléculas, lo que coincide en el empaquetamiento, o número de moléculas por unidad de volumen, disminuye sobre la
temperatura de transición. Esto trae como consecuencia que el grosor de la bicapa
2.2. Lı́pidos de Membranas
21
también disminuya en un 15 % aproximadamente en una transición de fase gel a
lı́quido cristalino. La probabilidad de encontrar configuraciones gauche, que como
ya se indicó se derivan de la configuración trans por rotaciones de 120◦ , varı́a en su
transición de fase. En la fase lı́quido cristalina la probabilidad de encontrar en un
instante dado configuraciones gauche es de 0.2 hasta aproximadamente la mitad de
la cadena, y luego aumenta hasta alcanzar un valor de 0.5 al final de la cadena, que
se encuentra en el centro de la bicapa. Este aumento de configuraciones gauche se
refleja en que el tiempo de correlación rotacional paralelo, en una bicapa en estado
lı́quido cristalino alcanza valores de 1 − 5 × 10−9 s, en tanto el tiempo de correlación perpendicular (Figura 2.6) alcanza valores que corresponden de 2 a 5 veces los
del paralelo. En contraste, en una bicapa en estado gel la probabilidad de encontrar conformaciones gauche es de 0 a 0.1 y los tiempos de correlación rotacional son
100 veces menores que en la fase lı́quido cristalina. Otra manera de visualizar las
diferencias entre ambos estados consiste en caracterizar la frecuencia de las isomerizaciones trans/gauche. Si definimos un tiempo de vida tj como el tiempo promedio
entre saltos conformacionales, tenemos que en una bicapa fluida tj tiene valores de
10−10 s en la parte superior de la cadena y de 10−11 cerca del residuo metilo terminal, en tanto que para una bicapa en estado gel los valores de tj son de 5 a 10 veces
mayores.
Los siguientes parámetros estructurales inciden directamente en la temperatura
de transición de una bicapa formada por una sola especie molecular de fosfolı́pido:
1. El grado de saturación. Para una misma cabeza polar, los fosfolı́pidos saturados presentan mayor temperatura de transición que los insaturados. Un enlace
doble en la cadena sn-2 tiene un efecto mayor que un enlace doble en la misma posición en la cadena sn-I, y un enlace doble cis, como los que presentan
los lı́pidos naturales, influye más que uno trans. Como ya se indicó, la presencia de un enlace doble en la cadena disminuye la energı́a necesaria para la
rotación del enlace simple contiguo a sólo 2 kcal/mol. Esto explica porque un
enlace doble trans, que no desvı́a la cadena, tiene una temperatura de transición menor que su homólogo saturado, aunque produce un menor efecto en la
fluidez de la bicapa que el correspondiente ácido graso insaturado cis, que produce un mayor defecto en el empaquetamiento de la bicapa. Los defectos de
empaquetamiento explicarı́an en parte porque las membranas que contienen
una gran cantidad de lı́pidos no saturados son más fluidas que aquellas que
tienen un alto porcentaje de lipidos saturados. Dentro de los insaturados, influye el número de enlaces dobles y la posición de estos. Tal es ası́ que cuando
se comparan cadenas con el mismo número de átomos de carbono un enlace doble en el centro de la cadena produce la transición a menor temperatura
que si uno se encuentra cerca de cualquiera de los extremos. Al aumentar el
22
2. Las Membranas
número de enlaces dobles la posición en que estos se encuentran es tanto o
más importante que el número de enlaces dobles. Por ejemplo, se obtiene una
Tt semejante para dos enlaces dobles situados cerca de la mitad de la cadena que para tres enlaces dobles con similares ubicaciones. Incluso la presencia
de fosfolı́pidos con ácidos grasos insaturados con muchos enlaces dobles no
aumenta más la fluidez de la bicapa y es posible que incluso la disminuyan
debido al particular empaquetamiento de éstos. Los fosfolı́pidos que tienen
una cadena no saturada y la otra saturada tienen una Tt intermedia entre las
Tt para el mismo fosfolı́pido con ambas cadenas saturadas o no saturadas.
En general, podemos concluir que la presencia de un enlace doble destruye
la cooperatividad de las interacciones del conjunto en el seno de la bicapa, lo
que perturba el empaquetamiento de las cadenas y produce un descenso significativo en la Tt con respecto a la misma molécula conteniendo solo cadenas
saturadas. De esto se desprende que la presencia de un enlace doble perturba
el empaquetamiento de las cadenas, con lo que aumentan las posibilidades de
movimiento y disminuye el orden del sistema, lo que incide en una menor Tt .
2. El largo de la cadena hidrocarbonada. Para fosfolı́pidos saturados cuanto mayor sea el largo de sus cadenas hidrofóbicas, mayor será la temperatura de
transición y la entalpı́a y entropı́a de la transición. Esto se debe a que las fuerzas atractivas de van der Waals entre las cadenas aumentan con el número de
grupos metilenos. En el caso de fosfolı́pidos con ácidos grasos no saturados
el efecto de la longitud de la cadena alifática es menor, ya que se empaquetan
con menor proximidad que los saturados.
3. La naturaleza de la cabeza polar. Si la composición de ácidos grasos saturados
es la misma, las Tt siguen el orden fosfatidiletanolaminas >> fosfatidilserinas
≥ fosfatidilcolinas. Para los mismos grupos polares formando parte de los fosfolı́pidos con dos cadenas de ácido oleico (18:1, cis 9-10) las diferencias en Tt
son menores, pero el orden se mantiene. Se ha atribuı́do este efecto a que la
etanolamina es más pequeña que la colina, lo que permitirı́a un mejor empacamiento de las moléculas. Sin embargo, estudios más recientes indican que el
factor determinante en la mayor temperatura de transición parece ser la capacidad de la fosfatidiletanolamina de formar puentes de H. La estabilización de
la superficie de la bicapa mediante la formación de puentes de H hace que las
moléculas de fosfatidiletanolamina presenten una menor afinidad por el agua
que las de fosfatidilcolina y restringe sus movimientos. La presencia de carga
neta de la cabeza polar disminuye la Tt ya que estas se repelen electrostáticamente, lo que en el caso de fosfatidilserina puede modularse por la presencia
de cationes divalentes como calcio que ejercen un efecto pantalla sobre las car-
2.2. Lı́pidos de Membranas
23
gas electrostáticas y aumentan la Tt . En contraste con el gran cambio en Tt , se
ha encontrado que los valores en las entalpı́as y entropı́as de transición para
una misma longitud de cadena son casi independientes de la composición de
los grupos polares.
Entre otros factores que inciden en la Tt , es interesante destacar que la presencia
del enlace éter en los plasmógenos de fosfatidilcolina y de fosfatidiletanolamina no
afecta significativamente la fluidez ni el grosor de la bicapa, aunque se ha encontrado que la Tt es 2 a 5 ◦ C mayor para un dieter de fosfatidiletanolamina que para el
correspondiente dieter, lo que sugiere un mejor empaquetamiento del dieter. Esto
podrı́a tener relevancia en el caso de las bacterias halofı́licas extremas, que presentan solo lı́pidos con enlaces dieter, y que mantienen a elevadas temperaturas un
grado de fluidez comparable al que presentan otras membranas a temperaturas menores. También podrı́a ser relevante en este contexto que peces que crecen a bajas
temperaturas tienen un menor contenido de plasmógenos y mayor contenido de
ácidos grasos poli-insaturados, tales como ácido cervónico, que los que representan
los mismos peces cuando crecen a mayores temperaturas.
La presencia de colesterol también afecta la Tt de una bicapa, ya que regula su
fluidez y aumenta la estabilidad mecánica de una membrana fluı́da. El grupo OH de
la molécula de colesterol se orienta en la interfase con el medio acuoso de la bicapa
y posee la capacidad de formar enlaces H con el oxı́geno que participa en el enlace
ester sn − 2 del glicerol. A su vez, la cadena alifática de la molécula se inserta en el
seno de la bicapa y puede contribuir a aumentar su fluidez, ya que perturba las interacciones entre las cadenas alifáticas de los fosfolı́pidos. La adición de cantidades
crecientes de colesterol a un fosfolı́pido puro ensancha la transición de fase, reduce
la entalpı́a de transición y eventualmente la elimina a concentraciones más altas. El
colesterol por ser una molécula plana con un esqueleto molecular rı́gido formado
por sus cuatro anillos, restringe las isomerizaciones trans/gauche y los movimientos
angulares de las cadenas de los fosfolı́pidos en una bicapa fluı́da y perturba el empacamiento de las cadenas en la configuración todo trans de una bicapa en estado gel.
Esto trae como consecuencia que el colesterol aumente la probabilidad de encontrar
conformaciones gauche bajo Tt y la disminuye sobre Tt . En contraste con este efecto
perturbador de la región hidrófoba de la bicapa, el colesterol afecta poco las cabezas
polares, ya que primordialmente actúa como un espaciador. Se ha encontrado que
los distintos fosfolı́pidos tienen distintas capacidades de acomodar al colesterol; su
forma se adapta mejor a la de moléculas tales como fosfatidilcolina, esfigomielina
y fosfatidilserina que a la fosfatidiletanolamina. Esto que podrı́a explicar porque en
membranas biológicas se encuentra el colesterol asociado con fosfatidilcolina y esfingomielina en la cara externa de las membranas plasmáticas y es poco frecuente su
24
2. Las Membranas
presencia en membranas internas ricas en fosfatidiletanolamina. De hecho, el colesterol baja la Tt de bicapas de fosfatidiletanolamina presumiblemente por su ruptura
de enlaces de H.
El contenido de colesterol es muy alto en membranas que tienen bajos contenidos de fosfatidiletanolamina, tales como las membranas plasmáticas, y es bajo en
membranas que tienen alto contenido de fosfatidiletanolamina, tales como la membrana mitocondrial y la de retı́culo sarcoplásmico. Mas aún, el colesterol se encuentra siempre en la monocapa opuesta a aquella en que se encuentra la fosfatidiletanolamina, lo que podrı́a deberse a la preferencia del colesterol por asociarse con la
fosfatidilcolina y la esfingomielina sobre la fosfatidiletanolamina. Sin embargo, en
algunas membranas, tales como las de gránulos de zimógeno, gránulos cromafines
y sinaptosomas, el contenido de colesterol y fosfatidiletanolamina es alto, lo que
podrı́a contribuir a hacerlas más inestables, lo que podrı́a a su vez ser requisito necesario para su función.
Relación entre Orden, Movimiento de los Lı́pidos Constituyentes y Fluidez de
una Membrana Biológica
Las transiciones de fases están bien caracterizadas y definidas para especies moleculares únicas, pero no ocurren en membranas biológicas. De hecho, se ha demostrado que las mezclas de los fosfolı́pidos no poseen un comportamiento ideal ya que
experimentan separaciones de fases si sus Tt son muy diferentes. Por lo tanto, si bien
el estudio de sistemas simples ha permitido comprender las propiedades simples
de las bicapas, las membranas más complejas no presentan esta clase de transiciones de fases. Es importante destacar, sin embargo, que todos los estudios realizados
en membranas biológicas indican que a las temperaturas en que operan fisiológicamente todas presentan una bicapa fluı́da. Más aún, en el caso de la Ca2+ −ATPasa
de retı́culo sarcoplásmico se ha demostrado que una membrana fluı́da es requisito
para la función de la enzima. Sin embargo, no todas las membranas biológicas presentan el mismo grado de fluidez, ya que existe un rango incluso en membranas de
una misma célula.
En una bicapa fluida las cadenas alifáticas pueden moverse más rápido que en
una bicapa en estado gel, y además es posible encontrar un conjunto de orientaciones de las cadenas alifáticas. Distintas técnicas, entre ellas la resonancia magnética
nuclear y la resonancia paramagnética del electrón, permiten medir tanto en membranas artificiales como naturales ambos tipos de variables: parámetros de orientación y parámetros dinámicos, entre ellos la velocidad de movimiento de los segmentos y los tiempos de correlación rotacional. Entre las técnicas que permiten la
rapidez de movimientos se encuentra, como ya se indicó, la resonancia magnética
2.2. Lı́pidos de Membranas
25
nuclear, que a través de la medición de tiempos de relajación da información sobre
tiempos de correlación para movimientos moleculares. Con respecto a la caracterización de la orientación, se usa el parámetro de orden, definido a continuación.
PARÁMETRO DE ORDEN. Podemos definirlo como un parámetro estructural
promediado en el tiempo. Es posible caracterizar la posición de una molécula en una
bicapa por el ángulo que forma con la normal al plano de la superficie (Figura 2.6).
El grado de orden de una bicapa esta dado por el promedio en el tiempo del valor
de θ para todas las moléculas que la componen. Sin embargo, el ángulo θ por sı́ mismo no es una forma particularmente conveniente de describir el orden de la bicapa.
Una descripción más conveniente serı́a usar el valor de cos θ pero como existe un
número similar de moléculas en ambos lados de la bicapa, el promedio serı́a igual a
cero ya que se anuları́a los valores positivos y negativos de cos θ a ambos lados de
la bicapa. Para evitar ese problema, se usa el valor de cos2 θ y el promedio se define como hcos2 θi. De esta definición sigue que si todas las moléculas de una bicapa
están alineadas perfectamente paralelas entre ellas, entonces θ = 0 y hcos2 θi = 1.
Si por otra parte todas las moléculas están orientadas al azar, son posibles todos los
valores de θ en las tres dimensiones, y en ese caso hcos2 θi = 1/3. Para simplificar la
notación, se ha definido un parámetro de orden S según la expresión:
S=
1
(3hcos2 θi − 1)
2
(2.4)
Sigue sin más de esta definición que para un sistema perfectamente ordenado en
que θ = 0 el valor de S = 1, y para un sistema totalmente desordenado, en que θ
pueda adoptar todos los valores y hcos2 θi = 1/3, el valor de S = 0.
Usando una variedad de técnicas espectroscópicas, tales como la RMN, la resonancia paramagnética del electrón y la fluorescencia, es posible determinar el valor
de S para bicapas de lı́pidos. Tanto en membranas modelo como en sistemas biológicos se ha encontrado que en bicapas fluı́das el valor de S se mantiene prácticamente
constante desde C2 hasta C9, y aumenta desde C9 hasta el fin de la cadena alifática.
El parámetro de orden alcanza un valor de hasta 0.5 en el seno de una bicapa lı́quido
cristalina y puede alcanzar un valor de 0.9 a 1 en estado gel.
En conclusión, las membranas biológicas se caracterizan por poseer una composición compleja de lı́pidos organizados en la estructura de bicapa fluida, que permite
un intervalo significativo de orientaciones y movimientos de las cadenas alifáticas.
Capı́tulo 3
LAS PROTEÍNAS
The human mind treats a new idea the way the body treats a
strange protein; it rejects it.
Peter B. Medawar
3.1.
Aminoácidos: Los Bloques Constituyentes de las
Proteı́nas
as proteı́nas están compuestas de un conjunto de 19 aminoácidos α y el iminoácido prolina. Para 19 de los 20 aminoácidos comúnmente encontrados en las proteı́nas una estructura general para el estado zwitteriónico1 tiene grupos amino (NH3+ )
y carboxilo (COO− ) ligados a un carbono central llamado carbono α. Los átomos
restantes conectados al carbono α son un átomo de carbono y el grupo R o cadena lateral (Figura 3.1). El término “aminoácido” se utiliza frecuentemente para los
aminoácidos α y también para incluir a la prolina.
Lo que marca la diferencia de un aminoácido con otro es la naturaleza de su cadena lateral. En la Figura 3.2 aparece la lista de los 20 aminoácidos que se encuentran
en las proteı́nas.
L
3.1.1.
Representaciones Estereoquı́micas de los Aminoácidos
Si bien un aminoácido está representado por el diagrama esqueleto de la Figura
3.1, es más revelador, y ciertamente más informativo, imponer una vista estereoquı́mica del arreglo de átomos. En estas vistas se hace un intento de representar las
posiciones de cada átomo en el espacio. Los grupos amino, carboxilo, hidrógeno y
1 Un zwitterión es una molécula que contiene tanto carga positiva como negativa. Los zwitteriones de
los aminoácidos son sales internas; esto es, el protón ácido de la función del ácido carboxı́lico se transfiere
al grupo amino básico.
3. Las Proteı́nas
28
Figure 3.1: a) Modelo de esqueleto de un aminoácido generalizado que muestra los grupos
amino (azul), carboxilo (rojo) y la cadena lateral R enlazados al carbono central α, b) La estructura de la prolina.
R son acomodados tetraédricamente alrededor del carbono α central (Figura 3.3). El
átomo de nitrógeno (azul) es parte del grupo amino (-NH3+ ), los átomos de oxı́geno
(rojo) son parte del grupo carboxilo (-COO− ). Los grupos restantes unidos al carbono α son el átomo de hidrógeno y el grupo R (cadena lateral).
El grupo R es responsable de diferentes propiedades de aminoácidos individuales. Como los aminoácidos constituyen a las proteı́nas, las propiedades del grupo
R contribuyen considerablemente a las propiedades fı́sicas de las proteı́nas. Las estructuras de las 20 diferentes cadenas laterales revela diferencias mayores en, por
ejemplo, tamaño, carga e hidrofobicidad aunque el grupo R siempre está atado al
carbono α.
3.1.2.
Uniones Péptidas
Los aminoácidos se juntan mediante la formación de una unión péptida donde el
grupo amino de una molécula reacciona con el grupo carboxilo de otra. La reacción
esta descrita como una condensación que resulta en la eliminación de agua y la
formación de un dipéptido (Figura 3.4).
Tres aminoácidos se juntan mediante la formación de dos uniones péptidas para formar un tripéptido y la secuencia continúa con la formación de tetrapéptidos,
pentapéptidos y ası́ sucesivamente. Cuando se juntan en series de uniones péptidas, los aminoácidos son llamados residuos para distinguir entre la forma libre y la
forma encontrada en las proteı́nas. Una secuencia corta de residuos es un péptido,
con el término polipéptido aplicado a cadenas más largas de residuos usualmente
3.1. Aminoácidos: Los Bloques Constituyentes de las Proteı́nas
29
Figure 3.2: Propiedades de los 20 aminoácidos proteinogénicos.
de secuencia y longitud conocidas. Dentro de la célula ocurre la sı́ntesis de proteı́nas
en el ribosoma aunque hoy en dı́a la sı́ntesis péptida es posible in vitro vı́a compleja
quı́mica orgánica. Sin embargo, mientras que el quı́mico orgánico lucha para sintetizar un péptido que contiene más de 50 residuos, el ribosoma rutinariamente hace
proteı́nas con 1000 residuos.
3. Las Proteı́nas
30
Figure 3.3: El arreglo espacial de los átomos en la alanina.
Las secuencias de aminoácidos de las proteı́nas son leı́das de izquierda a derecha, es decir, de la terminal N o amino a la terminal C o carboxilo. Los aminoácidos
individuales tienen códigos de tres letras, para guardar espacio en la presentación
de secuencias de proteı́nas largas un código de una sola letra es usado para cada
residuo aminoácido .
Juntar residuos establece una secuencia de proteı́na que esta convencionalmente
dividida en los componentes cadena principal y cadena lateral. La cadena principal,
o columna polipéptida, tiene la misma composición en todas las proteı́nas aunque
podrı́a diferir en extensión - esto es el número de residuos encontrados en la cadena polipéptida. La columna representa la repetición efectiva de uniones péptidas
hechas de los átomos N, Cα y C, con proteı́nas como la insulina que tiene aproximadamente 50 residuos mientras que otras proteı́nas contienen arriba de 1000 residuos
y más de una cadena polipéptida. Mientras que todas las proteı́nas enlazan átomos
de la columna polipéptida similarmente las cadenas laterales presentan un componente variable en cada proteı́na.
3.1. Aminoácidos: Los Bloques Constituyentes de las Proteı́nas
31
Figure 3.4: Cuando dos aminoácidos se juntan se forma una unión péptida. Aquı́ se muestra
como el dipéptido glicil-alanina se forma removiendo una molécula de agua cuando la glicina
se enlaza a la alanina.
Propiedades de la Unión Péptida
La cadena principal o columna de la cadena polipéptida es establecida mediante
la formación de uniones péptidas entre aminoácidos. La columna consiste del enlace
de la amida N, el carbono α y el carbonilo C.
La representación lineal de la cadena polipéptida no transmite la complicación
asociada con las longitudes de unión y los ángulos de los átomos que componen
la unión péptida. La unión péptida formada entre los grupos carbonilo y amino de
dos aminoácidos es una unión única que posee un poco de movilidad intrı́nseca,
esto ocurre debido al carácter de unión doble parcial - una caracterı́stica asociada
con la unión péptida y resonancia entre dos estados cercanamente relacionados.
Una de las consecuencias más importantes de la resonancia es que la longitud de
la unión péptida es más corta de lo esperado para una unión simple C-N. En promedio la longitud de unión péptida es 1.32 Å comparada con 1.45 Å de una unión
ordinaria C-N. En comparación la longitud de unión promedio asociada con una
unión doble C=N es 1.25 Å, enfatizando el carácter intermedio de la unión péptida.
Es importante mencionar que la unión doble parcial entre los átomos de carbono y
nitrógeno restringen la rotación sobre esta unión. Para cualquier columna polipépti-
3. Las Proteı́nas
32
da representada por la secuencia -N-Cα -C-N-Cα -C- solo las uniones Cα -C y N-Cα
exhiben movilidad rotacional. Como un resultado del movimiento restringido sobre
la unión péptida, dos conformaciones relacionadas por un ángulo de 180◦ son posibles. La primera ocurre cuando los átomos Cα son trans a la unión péptida mientras
que la segunda orientación menos favorable ocurre cuando los átomos Cα están en
cis.
3.2.
La Estructura Tridimensional de las Proteı́nas
Los aminoácidos enlazados en una representación plana bidimensional de una
cadena polipéptida no transmiten la belleza del arreglo tridimensional de las proteı́nas. Es la formación de estructura secundaria regular en patrones complicados
de plegado de proteı́na lo que ultimadamente origina las propiedades funcionales
caracterı́sticas de las proteı́nas.
3.2.1.
Secuencia o Estructura Primaria
La estructura primaria es el orden lineal de residuos de aminoácidos a lo largo de
la cadena polipéptida. Surge del enlace covalente de aminoácidos individuales vı́a
uniones péptidas. Ası́, hacer la pregunta ¿Cuál es la estructura primaria de una proteı́na? es simplemente otra forma de preguntar ¿Que es la secuencia de aminoácidos
de las terminales N a la C? Para leer la estructura primaria simplemente trasladamos los códigos de tres o una letra de la izquierda a la derecha, de la terminal amino
a la carboxilo.
Cada proteı́na esta definida por una secuencia única de residuos y todos los niveles subsecuentes de organización (secundaria, terciaria y cuaternaria) se basan en
esta estructura o nivel primario.
3.2.2.
Estructura Secundaria
La estructura primaria lleva a la estructura secundaria; la conformación local de
la cadena polipéptida o la relación espacial de residuos aminoácidos que están muy
cerca en la secuencia primaria. En proteı́nas globulares las tres unidades básicas
de la estructura secundaria son la hélice α, la hoja β y las vueltas. Todas las otras
estructuras representan variaciones sobre una de las antes mencionadas. Dentro de
los 20 diferentes residuos aminoácidos encontrados en proteı́nas hay 780 posibles
permutaciones para un dipéptido. En un polipéptido de tamaño promedio de ∼ 100
residuos el número de secuencias potencial es astronómico.
3.2. La Estructura Tridimensional de las Proteı́nas
33
Figure 3.5: Una hélice α regular. Solo los átomos pesados (C, N y O, pero no el hidrógeno) se
muestran y se omiten las cadenas laterales por claridad.
La Hélice α
La hélice α de mano derecha es probablemente la unidad más conocida y más
identificable de la estructura secundaria. La estructura de la hélice α fué derivada
de estudios de modelado tridimensional hasta la publicación de la estructura cristalográfica de la mioglobina. Esto demostró que las hélices α ocurren en las proteı́nas y
son largas como predijeron los estudios teóricos hechos por Linus Pauling. La hélice
α es el elemento estructural más común encontrado en proteı́nas; en proteı́nas globulares arriba del 30 % de todos los residuos son encontrados en las hélices.
La hélice α regular (Figura 3.5) tiene 3.6 residuos por vuelta con cada desviación
de residuo del residuo anterior de 0.15 nm. Este parámetro es llamado la traslación
por distancia de residuo. Con una distancia de traslación de 0.15 nm y 3.6 residuos
por vuelta, la afinación de la hélice α es simplemente 0.54 nm (esto es 3.6 × 0.15
nm). La afinación es la distancia de traslación entre dos átomos cualquiera correspondientes sobre la hélice. Uno de los mayores resultados de los estudios de modelado tridimensional fue el comprender que la hélice α surge de valores regulares
adoptados por φ y ψ, los ángulos de torsión o diedros (Figura 3.6).
Los valores de φ y ψ formados en la hélice α permiten a los átomos de la columna empaquetarse muy juntos con algunos contactos desfavorables. Lo que es más
3. Las Proteı́nas
34
Figure 3.6: Definición de los ángulos diedros.
importante, este arreglo le permite a algunos de los átomos de la columna formar
puentes de hidrógeno. Los puentes de hidrógeno ocurren entre el oxı́geno carbonilo de la columna (aceptante) de un residuo y el hidrógeno amida (donante) de un
residuo cuatro posiciones más adelante en la cadena polipéptida. Los puentes de
hidrógeno son 0.286 nm de largo entre átomos de oxı́geno y nitrógeno. Es importante notar que en proteı́nas “reales” el arreglo de puentes de hidrógeno muestra
variación en la longitud y el ángulo con respecto a los ejes hélice.
3.3.
Otras Conformaciones Hélice
La hélice 310 es una variación estructural de la hélice α que se encuentra en las
proteı́nas. Las hélices 310 se encuentran frecuentemente en proteı́nas cuando una
hélice α se distorsiona por la presencia de residuos desfavorables, cerca de una región de vuelta o cuando secuencias cortas se pliegan en forma de hélice. En la hélice
310 los puentes de hidrógeno dominates están formados entre los residuos i, i + 3
en contraste a los enlaces (i, i + 4) vistos en la hélice α regular. La designación de
310 se refiere al número de átomos de la columna localizados entre los átomos do-
3.3. Otras Conformaciones Hélice
35
nante y aceptante y el hecho de que hay tres residuos por vuelta. Con tres residuos
por vuelta la hélice 310 es una estructura más angosta y más ajustada en la cual el
potencial para contactos desfavorables entre los átomos de la columna o la cadena
lateral se incrementa.
Mientras la hélice 310 es una estructura más estrecha que la hélice α una tercera posibilidad es una hélice espiral más floja con puentes de hidrógeno formados entre los
grupos CO y NH separados por cinco residuos (i, i + 5). Esta estructura es la hélice
π y en un momento se pensó que no ocurrı́a naturalmente. Sin embargo, ejemplos
de esta estructura de hélice han sido descritos en proteı́nas.
La rareza de esta forma de estructura secundaria surge de ciertas razones. La mayor
limitación es que los ángulos φ/ψ de una hélice π yacen en el borde de lo permitido
en cuanto a energı́a mı́nima y región del gráfico de Ramachandran. El gran radio
de la hélice π significa que los átomos de la columna no hacen contacto tipo va der
Waals a través del eje de la hélice llevando a la formación de un agujero abajo de la
mitad de la hélice que es muy pequeño para ser ocupado por solvente.
La Hebra β
La hebra β , ası́ llamada porque fué la segunda unidad de la estructura secundaria predicha de los estudios de modelado de Pauling y Corey, es una conformación
extendida cuando es comparada con la hélice α. A pesar de su nombre la hebra β
es un arreglo helicoidal, aunque una forma extremadamente elongada con dos residuos por vuelta y una distancia de traslación de 0.34 nm entre átomos similares en
residuos colindantes. Si bien menos fácil de reconocer, esto lleva a una afinación o
distancia de repetición de casi 0.7 nm en una hebra β regular. Una sola hebra β no es
estable principalmente debido al número limitado de interacciones de estabilización
locales. Sin embargo, cuando dos o más hebras forman interacciones de puente de
hidrógeno adicionales un arreglo tipo hoja estable es creado (Figura 3.7). Estas hojas
β resultan en un incremento significante en la estabilidad total y son estabilizadas
por la formación de una columna de puentes de hidrógeno entre hebras adyacentes
que podrı́an envolver residuos ampliamente separados en la secuencia primaria.
Hebras adyacentes pueden alinearse en arreglos paralelos o antiparalelos con la
orientación establecida mediante la determinación de la dirección de la cadena polipéptida de la terminal N a la C. Representaciones caricatura de hebras β hacen fácil
establecer direcciones en estructuras moleculares dado que las hebras β son mostradas frecuentemente como flechas; las puntas de flecha indican la dirección hacia la
terminal C. Estas caricaturas toman diferentes formas pero todas “trazan” el arreglo
de la columna polipéptida y resume los elementos estructurales secundarios encontrados en proteı́nas sin necesidad de mostrar grandes números de átomos.
Las cadenas de poliaminoácidos no forman hojas β cuando se dispersan en solu-
36
3. Las Proteı́nas
Figure 3.7: Dos hebras β adyacentes son unidas mediante puentes de hidrógeno para formar
un pequeño elemento de hoja β. Los puentes de hidrógeno son inter-hebra entre los grupos
vecinos CO y NH.
ción y esto ha entorpecido el estudio de la formación de tales estructuras. Sin embargo, a pesar de esta observación muchas proteı́nas están basada predominantemente
en hebras β. Diferentes representaciones ideales de hebras encontradas en proteı́nas
son frecuentemente distorsionadas retorciendo lo que se origina de una variación
sistemática de ángulos diedros (φ y ψ) hacia mayores valores positivos. El resultado
es un ligero, pero discernible, giro en la cadena polipéptida. Además cuando hebras
de puente de hidrógeno se juntan para formar hojas, distorsiones adicionales ocurren especialmente con mezclas de hebras β paralelas y antiparalelas. En promedio
las hojas β que contienen hebras antiparalelas son más comunes que hojas compuestas enteramente de hebras paralelas. Las hojas antiparalelas frecuentemente se
forman de sólo dos hebras β corriendo en direcciones opuestas mientras que se observa que al menos cuatro hebras β son requeridas para formar hojas paralelas. Las
hebras β se asocian espectacularmente en hojas curvadas extensas conocidas como
barriles.
Los Giros Como Elementos de Estructura Secundaria
Conforme más estructuras de alta resolución son depositadas en los bancos de
datos de proteı́nas se ha permitido a los giros de diferentes proteı́nas ser definidos y
comparados en términos de composición de residuos, ángulos y distancias de unión.
En algunas proteı́nas la proporción de residuos encontrada en los giros puede exceder el 30 % y en vista de este alto valor es improbable que los giros representen
estructuras aleatorias. Los giros tienen el rol universal de habilitar el polipéptido
3.3. Otras Conformaciones Hélice
37
para cambiar de dirección y en algunos casos de regresar ası́ mismo. Los giros de
reversa o curvas surgen de las propiedades geométricas asociadas con estos elementos de la estructura de la proteı́na.
El análisis de la composición de los aminoácidos de los giros revela que las cadenas laterales voluminosas o ramales ocurren con baja frecuencia. En cambio, los residuos con pequeñas cadenas laterales son encontrados preferentemente. Un análisis
de diferentes tipos de giros ha establecido que tal vez existen a lo más 10 conformaciones diferentes sobre la base del número de residuos componentes del giro y los
ángulos φ y ψ asociados con los residuos centrales. Los giros pueden generalmente
ser clasificados de acuerdo al número de residuos que contienen, con el número más
común a ser de tres o cuatro residuos.
Un giro γ contiene tres residuos y frecuentemente enlaza hebras adyacentes de
la hoja β antiparalela. El giro γ está caracterizado por el residuo en la mitad del giro (i + 1) no participante en la unión de hidrógeno mientras que el primer y tercer
residuo pueden formar los puentes de hidrógeno final e inicial de las hebras β antiparalelas. El cambio en la dirección de la cadena polipéptida causado por un giro
γ es reflejado en los valores de φ y ψ para el residuo central. Como un resultado de
su tamaño y flexibilidad conformacional la glicina es un residuo favorecido en esta
posición aunque otros también han sido encontrados.
Los más comunmente encontrados en estructuras de proteı́na son giros de cuatro residuos (giros β). Aquı́ los dos residuos de la mitad (i + 1, i + 2) nunca son
involucrados en los puentes de hidrógeno mientras que los residuos i y i + 3 participarán en los puentes de hidrógeno si un arreglo favorable se forma entre el donor y
el aceptor. Los análisis de estructuras depositadas en las bases de datos de proteı́nas
revelan fuertes preferencias de residuos. En el tipo relativamente común de giro 1β
cualquier residuo puede ocurrir en la posición i, i + 3 con la excepción de que nunca
se encuentra la prolina. La glicina es frecuentemente más encontrada en la posición
i + 3 mientras que la prolina predomina en i + 1. Asn, Asp, Cys y Ser también son
encontrados frecuentemente en giros β como el primer residuo.
Estructura Secundaria Adicional
Más de 50 años de estudio cientı́fico indican que las conformaciones de hélice
α y hoja β son dos grandes tipos de plegado periódico de la columna polipéptida
que son compatibles con las constricciones estéricas planteadas por la naturaleza
plana del enlace péptido de la cadena polipéptida y la naturaleza asimétrica del carbono tetraedro. Si la columna polipéptida de una proteı́na no exhibe periodicidad,
la estructura es conocida como espiral aleatoria o cadena aleatoria. Para cada par
de cadenas que interactúan a través de hojas β se estima el número de puentes de
hidrógeno como el número de puentes β que contribuyen a las interfaces intercade-
3. Las Proteı́nas
38
na de hoja β.
Existen algunas otras formas de estructura secundaria las cuales se adhieren a la
siguiente clasificación :
Hélices: hélice α, hélice 310 , hélice π, hélice β, hélice poliprolina y hélice colágeno.
Extendida: hebra β, giro, horquilla β, bulto β y hebra α
A continuación se muestra la propensidad de los diferentes aminoácidos a presentar alguna de las estructuras antes mencionadas.
A favor de la hélice: Metionina, Alanina, Leucina, Glutamato, Glutamina y
Lisina.
A favor de estructura extendida: Treonina, Isoleucina, Valina, Fenilalanina,
Tirosina y Triptófano
A favor de una estructura desordenada: Glicina, Serina, Prolina, Asparagina
y Aspartato.
Sin preferencia: Cisteı́na, Histidina, Arginina.
3.3.1.
Estructura Terciaria
Al comienzo del 2004 aproximadamente 22000 conjuntos de coordenadas atómicas fueron depositados en bases de datos como el Protein Data Bank (PDB) y este
valor aumenta diariamente con el depósito de nuevas estructuras de organismos
diversos. Mientras algunas de estas proteı́nas están duplicadas o relacionadas, la
mayorı́a representa la determinación de estructuras nuevas usando principalmente
espectroscopı́a de resonancia magnética nuclear. Más de 1000 plegados de proteı́na
han sido descubiertos a la fecha con indudablemente más por venir.
Los archivos PDB contienen información de las posiciones en el espacio de la
amplia mayorı́a de átomos constituyentes de la proteı́na. Identificando las coordenadas x, y y z de cada átomo definimos la molécula entera. Sin embargo además
de las coordenadas de los átomos los archivos PDB también contienen información
que describe la secuencia primaria, el método utilizado para determinar la estructura, el organismo del cual fue derivada la proteı́na, los elementos de la estructura
secundaria, cualquier modificación post-traslacional ası́ como los autores.
3.3. Otras Conformaciones Hélice
39
Estructura Terciaria Detallada
La estructura terciaria representa la cadena polipéptida plegada. Está definida
como el arreglo espacial de residuos aminoácidos que están separados ampliamente en la secuencia primaria o más brevemente como la topologı́a total formada por
el polipéptido. Para proteı́nas globulares pequeñas de 150 residuos o menos la estructura plegada incluye una molécula compacta esférica compuesta de motivos estructurales secundarios con poco de estructura irregular. La estructura desordenada
o irregular en las proteı́nas esta normalmente confinada a las terminales N y C o
más raramente a regiones cerradas dentro de una proteı́na o regiones de enlace que
conectan a uno o más dominios. El preguntar ¿que es la estructura terciaria de una
proteı́na? es sinónimo de preguntar ¿cuál es el plegado de proteı́na?. El plegado
se origina del enlace de estructuras secundarias que forman una molécula globular compacta. Los elementos de la estructura secundaria interactúan vı́a puentes de
hidrógeno, como en las hojas β, pero también dependen de puentes de disulfuro,
interacciones electrostáticas, interacciones de van der Waals, contactos hidrofóbicos
y puentes de hidrógeno entre grupos diferentes de la columna.
3.3.2.
Estructura Cuaternaria
La estructura cuaternaria es el arreglo de múltiples proteı́nas plegadas en un
complejo multi-subunidad. Muchas proteı́nas de hecho están ensambladas de más
de una cadena polipéptida, la cual en el contexto de un ensamble mayor son conocidas como subunidades proteı́nicas. Las enzimas compuestas de subunidades con
diversas funciones son llamadas algunas veces holoenzimas, en las cuales algunas
partes podrı́an reconocerse como subunidades regulatorias y el corazón funcional
es conocido como subunidad catalı́tica. Los cambios en la estructura cuaternaria
pueden ocurrir a través de cambios conformacionales dentro de las subunidades
individuales o a través de la reorientación de las subunidades relativas entre si.
Capı́tulo 4
EL PÉPTIDO β AMILOIDE Y LA ENFERMEDAD DEL
ALZHEIMER
DNA was the first three-dimensional Xerox machine.
Kenneth Boulding
a enfermedad de Alzheimer (EA) es la forma más común de demencia. Esta enfermedad incurable, degenerativa y terminal descrita por primera vez por el psiquiatra alemán Alois Alzheimer en 1901. Generalmente se diagnostica en personas
por arriba de los 65 años de edad, aunque la menos frecuente aparición temprana
de la EA puede ocurrir mucho antes. Una estimación de 26.6 millones de personas
alrededor del mundo fueron afectados con EA en 2006; este número se podrı́a cuatriplicar para 2050.
A pesar de que cada vı́ctima de la EA lo experimenta en una manera única, hay muchos sı́ntomas en común. Los primeros sı́ntomas observables son frecuentemente
relacionados de forma errónea con situaciones relacionadas con la edad o manifestaciones de estrés. En las primeras etapas, el sı́ntoma más comúnmente reconocido
es la pérdida de memoria ası́ como la dificultad para recordar hechos aprendidos
de forma reciente. Cuando un médico ha sido notificado y se sospecha de EA, el
diagnóstico es usualmente confirmado con la evolución del comportamiento y pruebas congnitivas, frecuentemente seguidas de una exploración del cerebro si es posible. Conforme avanza la enfermedad los sı́ntomas incluyen confusión, irritablilidad
y agresión, cambios de humor, pérdida del habla y el apartamiento general de la
vı́ctima conforme pierde los sentidos. Gradualmente se pierden las funciones corporales, finalmente llevando a la muerte. El pronóstico individual es difı́cil de evaluar
ası́ como la duración de la enfermedad. La vida media después del diagnóstico es
aproximadamente siete años. Menos del 3 % de las personas que lo padecen viven
más de catorce años después del diagnóstico.
La causa y progreso de la enfermedad del Alzheimer no esta bien entendida. En
1991, la hipótesis amiloide postula que los depósitos de Aβ son la causa fundamen-
L
4. El Péptido β Amiloide y La Enfermedad del Alzheimer
42
Figure 4.1: Las enzimas actúan sobre la APP (Proteı́na Precursora Amiloide) y la cortan en
fragmentos. El fragmento β-Amilode es crucial en la formación de las placas seniles que aparecen en el Alzheimer.
tal de la enfermedad. Esta teorı́a se origina debido a que el gen para la proteı́na
precursora amiloide (APP) esta localizado en el cromosoma 21, y personas con trisomı́a 21 (Sı́ndrome de Down) exhiben casi universalmente EA cerca de los 40 años
de edad. Los amiloides son agregados fibrosos e insolubles de proteı́nas que comparten rasgos estructurales especı́ficos. El nombre amiloide viene de la identificación
errónea de la sustancia como almidón (amylum en Latı́n). Por un tiempo, la comunidad cientı́fica debatió si los depósitos amiloides eran depósitos de grasas o carbohidratos hasta que finalmente se resolvió que no era ninguno de los dos, sino un
depósito de masas proteı́nicas.
4.1.
El Péptido Aβ
El Aβ es un fragmento de una proteı́na llamada proteı́na precursora amiloide
(APP), una proteı́na transmembrana que penetra a través de la membrana de la
neurona. La APP es crı́tica para el crecimiento, supervivencia y reparación de la
neurona. En el Alzheimer, un proceso desconocido causa que la APP se divida en
fragmentos más pequeños debido a enzimas a través de la proteólisis, ver Figura
4.1.
El plegamiento incorrecto, la formación de agregados y la acumulación del Aβ
en depósitos solubles ha sido implicado como la causa primaria de la muerte neuronal en EA. Además de la EA, más de 20 desórdenes degenerativos que afectan ya
sea el sistema nervioso central (por ejemplo el mal de Parkinson, el mal de Huntington y enfermedades priones transmisibles) o cuestiones periféricas (por ejemplo
la diabetes, cirrosis hepática y enfermedades degenerativas del ojo) comparten la
misma patologı́a general, cada una surge de la agregación de una proteı́na asociada.
Aunque las proteı́nas implicadas en estas enfermedades no comparten estructura o
secuencia homológica, una caracterı́stica definitoria es que todas se agregan en fibri-
4.2. La Agregación en Superficies Lipı́dicas
43
llas insolubles altamente ordenadas que contienen estructuras de hoja β extensivas.
El gran grupo de investigación formado durante los pasados 10 años ha aumentado
significativamente nuestro entendimiento de las vı́as involucradas en la agregación
Aβ y el mecanismo de toxicidad celular. Sin embargo, la base molecular de los eventos primarios durante el proceso de agregación y la naturaleza de las fluctuaciones
estructurales que desencadenan el mal plegamiento y la asociación de Aβ permanecen pobremente entendidos.
El Aβ es un péptido de 39 a 43 residuos aminoácidos derivado del proceso
proteolı́tico de la proteı́na precursora amiloide (APP) transmembranal durante el
metabolismo celular regular. El péptido es anfifı́lico, con una región terminal N
(nitrógeno) hidrofı́lica y una región terminal C (carbono) hidrofóbica que pertenece
al dominio transmembrana de la APP. El montaje de Aβ en depósitos esta acompañado por cambios sustanciales para la conformación péptida. Bajo condiciones
que imitan membrana celular, donde la terminal C del Aβ se encuentra antes de ser
separada de la APP, el Aβ exhibe una cantidad significante de estructura hélice α,
cuando se forma en fribrillas el Aβ las estructuras de hoja β caracterı́sticas encontradas en las fibrillas amiloides. En los buffers fisiológicos a bajas concentraciones de
Aβ, el péptido es principalmente un azar, cadena flexible, con algunos motivos estructurales locales. La aparición de agregados Aβ citotóxicos requiere la formación
de hojas β inter e intramoleculares.
Hay una evidencia creciente de que la membrana celular desempeña un rol central en la mediación de la fibrilogénesis Aβ. Se ha mostrado que los lı́pidos de la
membrana extraı́dos de células cerebrales humanas pueden acelerar la formación
fibril Aβ y de aquı́ que la composición de las membranas lı́pidas controle las interacciones Aβ-lı́pido y la fibrilogénesis [4]. Una caracterı́stica que ha sido bien documentada es el efecto de la carga del grupo cabeza del lı́pido sobre la formación de
fibrilas Aβ. Además de mediar la formación fibril de Aβ, la interacción del péptido
con las membranas celulares también podrı́a servir como una vı́a por la cual se desarrolle toxicidad. Un mecanismo propuesto para la neurotoxicidad del Aβ es la interrupción y despolarización mediada ya sea por formación de canales iónicos o por
un incremento en la conductancia membranal general, que resulta en la alteración
de la homeostasis iónica y desregulación de la trasducción de señales neuronales,
llevando a la muerte de la célula.
4.2.
La Agregación en Superficies Lipı́dicas
Muchas proteı́nas, y en particular muchas proteı́nas amiloidogénicas, son activas
en superficies y las interacciones de estas proteı́nas con las superficies de membra-
4. El Péptido β Amiloide y La Enfermedad del Alzheimer
44
nas lipı́dicas pueden tener profundas consecuencias estructurales. Las interacciones
con lı́pidos pueden inducir cambios funcionales importantes en la estructura de proteı́nas. Por ejemplo las apolipoproteı́nas que están ligadas a lı́pidos son conformacional y funcionalmente distintas de las que no están ligadas. Similarmente, el papel
del lı́pido en el plegado en ensamble de proteı́nas de membrana ha sido relacionado al de un chaperón. En contraste, las proteı́nas globulares activas en la superficie
pueden ser desestabilizadas conformacionalmente mediante interacciones con superficies lipı́dicas y esas interacciones pueden promover agregación de proteı́nas y
formación amiloide.
Es claro entonces que las consecuencias conformacionales de las interacciones
proteı́na-lı́pido son complejas, mostrando dependencia no solo en la naturaleza de
la proteı́na sino también en las propiedades fı́sicas y quı́micas de la superficie lipı́dica en sı́. No es de sorprender que el efecto de las superficies lipı́dicas en la formación amiloide es compleja y multifactorial, reflejando las diferentes capacidades de
las superficies lipı́dicas para estabilizar o desestabilizar un estado monomérico plegado y promover o inhibir interacciones proteı́na-proteı́na.
4.3.
Resultados Recientes
Recientemente han surgido pruebas de que no son los agregados fibrilares sino
la estructura de hoja β dominante de los oligómeros1 y fibrillas intermedias (protofibrillas) las que son neurotóxicas y podrı́an ser el factor patógeno en la EA. Por
ejemplo, Walsh et al. demostró que la microinyección de medio celular abundante en
monómeros y oligómeros pero sin fibrillas amiloides inhibió notablemente la potenciación a largo plazo del hipocampo2 en ratas in vivo. El conflicto entre la hipótesis
amiloide y estas nuevas observaciones emergentes ha estimulado más a los investigadores al estudio de las fases tempranas de ensamblaje de Aβ y a explorar las
propiedades intrı́nsecas de los Aβ y los comportamientos dinámicos conformacionales del monómero Aβ.
Además, ha sido establecido experimentalmente que la mayor estructura secundaria adoptada por el Aβ depende del entorno. El monómero Aβ favorece una estructura de hélice α en un entorno membranal o imitación de membrana como los
detergentes iónicos. La estructura dominante del Aβ en medios membranales o medios tipo membrana, solución acuosa y fibrillas lleva a la postulación de que una
1 Se
dice que una molécula constituye un oligómero cuando los radicales asociados son distintos entre
sı́. La naturaleza orgánica esta llena de estos casos multifuncionales.
2 Parte del cerebro situada en el lóbulo temporal (los seres humanos y otros mamı́feros tienen dos
hipocampos, justo en medio de cada hemisferio cerebral)
4.3. Resultados Recientes
45
conversión de conformación de una hélice α o espira aleatoria a un hoja β sucede
durante (o antes) de la agregación de Aβ. Cambios de conformación similares también han sido encontrados en proteı́nas de otras enfermedades. Ası́, un mecanismo
de estudio riguroso de tal transición conformacional es crı́tico para el entendimiento y desarrollo de enfoques terapéuticos para los desórdenes conformacionales en
general, y la EA en particular.
Desafortunadamente, debido a la extrema insolubilidad de las fibrillas y a que los
oligómeros son altamente propensos a formar agregados, la investigación de los
cambios conformacionales de Aβ a detalle atómico por medio de métodos experimentales sigue siendo intratable. Las simulaciones computacionales con su extremadamente alta resolución temporal y representación a nivel atómico han estado
empezando a usarse de manera muy frecuente para entender las complejas caracterı́sticas de los polipéptidos y en la predicción de las preferencias estructurales.
Capı́tulo 5
LA DINÁMICA MOLECULAR
We‘ve all heard that a million monkeys banging on a million
typewriters will eventually reproduce the entire works of
Shakespeare. Now, thanks to the Internet, we know this is
not true.
Robert Wilensky
5.1.
Haciendo Fı́sica in Silico
La fı́sica provee mucha información y conocimiento acerca de la estructura detallada de la materia a nivel atómico. Esencialmente todas las interacciones y reacciones entre átomos se pueden entender a partir del movimiento de sus electrones
y en principio éstas podrı́an ser calculadas utilizando mecánica cuántica. Las ecuaciones que describen esto son de hecho simples y pueden ser resueltas exactamente,
pero solo para configuraciones extremadamente simples. Cualquier sistema realista
requiere el uso de métodos numéricos aproximados y el uso de computadoras.
Recientemente, los fı́sicos de mentalidad fundamentalista deberı́an ser capaces
de predecir la estructura completa y la dinámica de cualquier sistema, preferiblemente incluyendo el universo, a partir de la mecánica cuántica mediante la solución
numérica de la ecuación de Schrödinger dependiente del tiempo para los electrones
constituyentes y el núcleo atómico. Para macromoléculas como las proteı́nas con miles o millones de átomos esto no es, desafortunadamente, una alternativa. A pesar
del desarrollo extremadamente rápido de las computadoras en las últimas décadas
el poder necesario quizá no vea la luz este siglo.
En la práctica esto no importa mucho. Relativamente pocos procesos, en particular a nivel biomolecular no requieren de ningún enfoque mecanocuántico, a este
nivel el mundo es clásico. Por supuesto no podemos describir los procesos detallados de rompimiento o formación de enlaces sin ella, y algunas de la vibraciones más
5. La Dinámica Molecular
48
rápidas en los enlaces y ángulos podrı́an rondar el borde con la región de mecánica
cuántica, pero aparte de esto la mayorı́a de los movimientos son más que adecuadamente descritos al apegarse a la mecánica clásica. De hecho, la dinámica molecular
probará ser frecuentemente una alternativa mucho mejor a pesar del costo computacional, dado que los modelos están parametrizados para reproducir observaciones
experimentales reales en lugar de simplificar la teorı́as.
Con esta motivación, el enfoque ab initio puede ser remplazado con una parametrización semi-empı́rica de las fuerzas clásicas en el sistema. Dado que esto es muchos órdenes de magnitud más rápido que el resolver la ecuación de Schrödinger
seremos capaces de simular sistemas mucho más grandes por mucho más tiempo.
También es más sencillo llevar a cabo simulaciones bajo condiciones bioquı́micas
realistas, dado que cálculos de mecánica cuántica corresponden a simulaciones en
el vacı́o a temperatura de 0 ◦ K, lo cual es ligeramente diferente del mundo real.
Estas simplificaciones hacen posible estudiar quı́mica usando computadoras en
lugar de experimentos. Dado que las máquinas están basadas en microchips de silicio la descripción in silico ha aparecido en los últimos años, en analogı́a con las
designaciones in vivo / in vitro usadas para resultados bioquı́micos en organismos
vivos y tubos de ensayo, respectivamente. También es usado para otras técnicas que
mezclan biologı́a y computadoras, por ejemplo la bioinformática.
El concepto de dinámica molecular fué originalmente desarrollado por Alder y
Wainwright a principios de los 50’s como una técnica para simular un sistema de
colisión de partı́culas de núcleo duro. Fué extendida más tarde a potenciales continuos y medidas de tiempo uniforme por Rahman. La idea básica es de hecho muy
simple, pero requiere mucho poder de cómputo. La meta es reproducir la evolución
temporal de un sistema de N partı́culas interactuantes (por ejemplo átomos) con
masas mi por solución directa de las ecuaciones de movimiento de Newton,
mi
d2 ri
= Fi
dt2
i = 1, ..., N
(5.1)
donde ri (t) es la posición de la partı́cula i. La fuerza momentánea Fi sobre cada
átomo deberá ser calculada de las interacciones en el sistema. Fi está definida como
la derivada de una función de energı́a potencial V que esta determinada por las
posiciones de todos los átomos,
Fi = −∇ri V (r1 , ..., rN )
(5.2)
El cálculo de este potencial es una parte central del algoritmo. Las ecuaciones implican que la fuerza tratará de mover las partı́culas para alcanzar un estado de baja
energı́a potencial, en la misma manera que la fuerza de gravedad actúa sobre una
manzana para moverla a un estado de menor altura. La aceleración resultante nos
5.1. Haciendo Fı́sica in Silico
49
dice como varı́a la velocidad, y de la variación de la velocidad es posible determinar
posiciones aproximadas de los átomos después de un tiempo muy corto. Este proceso es llamado integración de las ecuaciones de movimiento, repitiendo el cálculo
por un enorme número de pequeños pasos resulta en una trayectoria con la evolución de las posiciones, velocidades y fuerzas sobre todos los átomos durante la
simulación. Si la función de energı́a potencial es una buena aproximación de las
interacciones reales entre las partı́culas, esto puede proporcionar una descripción
extremadamente detallada de las propiedades dinámicas y de equilibrio en el sistema que se estudia. La primera simulación por computadora de una biomolécula
fué realizada en 1977, pero el campo ha avanzado a pasos agigantados. Las escalas
disponibles en tiempo y espacio siguen siendo limitadas en comparación a muchos
procesos biológicos, pero el rendimiento esta prácticamente estallando debido a una
mejor paralelización y software más eficiente. Una parte importante de estos esfuerzos es el desarrollo de software para sumulación con mejor eficiencia, mediante la
introducción de mejores optimizaciones y algoritmos que permitan pasos de tiempo
mayores. Aunque sigue en su infancia, la simulación computacional se está convirtiendo rápidamente en una herramienta poderosa para muchas disciplinas, entre
ellas la quı́mica y la fı́sica.
5.1.1.
Los Campos de Fuerza
Una vez que hemos determinado las ecuaciones de movimiento, la siguiente tarea es definir y calcular la energı́a potencial V ({r}). Existen muchas posibles elecciones para la implementación de esta función, y siempre es un compromiso aproximado entre una descripción tan detallada como sea posible y una que pueda ser
evaluada rápidamente en una computadora. En su apariencia final, junto con los
parámetros de interacción usados en la simulación, es llamado campo de fuerzas.
Muchos campos de fuerzas diferentes han sido desarrollados por varios grupos de
investigación, pero la mayorı́a de ellos están relacionados entre si, como se ilustra
en la Figura 5.1.
Los campos de fuerza All-atom proveen parámetros para cada átomo en el sistema, incluyendo el hidrógeno, mientras que los campos de fuerza united-atom tratan
los átomos de hidrógeno y los de carbono en los grupos metilo (CH3 ) y metileno
(CH2 ) como un solo centro de interacción. Los campos de fuerza Coarse-grained,
que son usualmente utilizados en simulaciones prolongadas de proteı́nas, proveen
inclusive representaciones más abstractas con el fin de incrementar la eficiencia
computacional.
Un potencial que provea extremadamente alta precisión en todas las situaciones
5. La Dinámica Molecular
50
Figure 5.1: Un diagrama simplificado donde se muestra el desarrollo y las relaciones entre los
campos de fuerza generales para la dinámica molecular. Los argumentos para escoger alguno
de ellos son usualmente más cuestion de fe que de ciencia :s
será lento de calcular y no muy útil, ası́ que cuando se aplica a nivel molecular se
hacen muchas aproximaciones. Primero, es subdividido en contribuciones de interacciones ligadas de átomos conectados (por ejemplo enlaces, ángulos y enlaces de
rotación) e interacciones no ligadas entre pares de átomos cercanos entre si. El potencial debido a variaciones en la longitud de enlace entre átomos i y j es usualmente
modelado por un resorte armónico simple,
Venlace (rij ) =
kijb 2
rij − rij0
2
(5.3)
b
0
donde kij
es la constante de fuerza que describe la rigidez del tipo de enlace y rij
es la longitud de equilibrio del enlace. De manera similar, el estrechamiento del
ángulo para los átomos i,j,k (donde i está enlazado a j y j esta enlazado a k) puede
ser descrito por
ϑ 2
kijk
Vángulo (ϑijk ) =
ϑijk − ϑ0ijk
(5.4)
2
ϑ
con ϑ0ijk como el ángulo de equilibrio. La constante de fuerza kijk
determina que tan
difı́cil de distorsionar es el ángulo. En moléculas más grandes también habrá variaciones en el potencial debido a la rotación alrededor del enlace medio en una
5.1. Haciendo Fı́sica in Silico
51
Figure 5.2: Coordenadas internas para las interacciones ligadas: a) r gobierna la deformación
del enlace; b) ϑ representa el término del enlace angular; c) ϕ brinda el ángulo dihedro; d) el
pequeño ángulo α hacia afuera del plano esta gobernado por el ángulo diedro “impropio” ψ.
secuencia de cuatro átomos como se muestra en la Figura 5.2.c.
Esto es un efecto de la configuración geométrica de los orbitales electrónicos.
Aunque la interacción real es bastante complicada, es usualmente modelada con un
potencial diedro armónico simple,
Vdiedro (ϕijkl ) = kjkϕ [1 + cos (nϕ − ϕ0 )]
(5.5)
Solo hay dos alternativas razonables para la posición zero del ángulo. Aquı́ utilizamos la convención bioquı́mica, donde el ángulo ϕ es cero en el estado cis (cuando
los átomos 1 y 4 esta en el mismo lado del enlace).
La multiplicidad n determina el número de mı́nimos durante una rotación completa
y ϕ0 sus posiciones. Para hidrocarburos alifáticos (por ejemplo las colas neutras en
las moléculas lipı́dicas) con cuatro o más carbonos consecutivos algunas veces se
necesita una mejor descripción del potencial la cual tome explı́citamente en cuenta
que los dos estados gauche en ±60◦ son ligeramente menos favorables que el trans
en 180◦ , y también que las transiciones entre gauche y trans son consideredas más
sencillas que cruzar el estado cis debido a la repulsión estérica entre los átomos 1 y
4.
Todas estas interacciones conciernen a los átomos enlazados cercanamente entre si. Esto los hace locales en el espacio y aun más importante es que su número
solo aumentará linealmente con el tamaño del sistema, lo que significa que no son
muy costosos de calcular. En contraste, existen muchas más interacciones no ligadas
5. La Dinámica Molecular
52
entre átomos separados por más de tres enlaces o incluso localizadas en diferentes
moléculas. El cálculo de estas fuerzas es la parte que más tiempo consume en la simulación por dinámica molecular, contando aproximadamente el 90 − 95 % del total
del uso de procesador, incluso si asumimos que todas las fuerzas son entre pares de
partı́culas y despreciamos contribuciones más allá de una distancia de corte. Estas
interacciones no ligadas son usualmente descritas con un término repulsivo importante sobre distancias pequeñas, fuerzas de dispersión atractiva que siempre están
presentes e interacciones electrostáticas entre partı́culas cargadas. Los componentes
dispersivos y repulsivos son frecuentemente combinados en forma de interacciones
de Lennard-Jones,
VLennard-Jones (rij ) =
Cij12
rij12
−
Cij6
rij6
(5.6)
donde Cij6 y Cij12 son parámetros que dependen del tipo de átomos que participan, determinan la atracción y repulsión respectivamente. El potencial resultante
es débilmente atractivo a largas distancias pero fuertemente repulsivo cuando los
átomos empiezan a traslaparse. La repulsión electrostática es mejor descrita por un
término exponencial (interacciones de Buckingham), pero la función exponencial es
muy costosa de calcular en una computadora y usualmente es remplazada por la
ligeramente más simple pero más económica expresión r−12 . Para pares de átomos
cargados la interacción electrostática está dada por
VCoulomb (rij ) =
qi qj
4π0 r rij
(5.7)
donde qi y qj son las cargas. La permitividad del vacı́o es designada por 0 , y r es
la permitividad relativa. A esta última usualmente se le asigna uno, lo cual podrı́a
no ser realista cuando los sistemas simulados como para el caso del agua con un valor experimental 80 veces mayor. Este apantallamiento macroscópico es sin embargo
altamente un efecto de rotación y desplazamiento de todos los dipolos y moléculas.
En dinámica molecular, todos estos movimientos son reproducidos explı́citamente y la restante permitividad microscópica es ası́ considerablemente más pequeña,
acercándose al valor del vacı́o. También es posible argumentar que el valor deberı́a
ser mayor a la unidad, pero en muchos campos de fuerzas esto se corrige implı́citamente parametrizando las cargas para obtener las propiedades de equlibrio correctas. Hay muchos modelos más elaborados para la permitividad relativa, pero
eventualmente uno tiene que aceptar que es una caracterı́stica macroscópica que no
es fácil de representar a nivel atómico.
5.1. Haciendo Fı́sica in Silico
53
Campos de Fuerza de Átomo Unido y Representaciones Reducidas
Hasta ahora hemos asumido que todos los átomos están representados en el modelo. Sin embargo, dado que el número de interacciones no ligadas escala con el
cuadrado del número de sitios de interacción presentes, hay claras ventajas si el
número de sitios de interacción se puede reducir. La forma más simple de hacer esto es hacer una subsuma de algunos o todos lo átomos (usualmente solo los átomos
de hidrógeno) dentro de los átomos a los que están ligados. Un grupo metil serı́a entonces modelado como un solo “pseudo átomo” o un “átomo unido”. Los parámetros de van der Waals y electrostáticos serı́an modificados para tomar en cuenta los
átomos de hidrógeno adyacentes. Es posible un ahorro computacional considerable;
por ejemplo, si el butano es modelado con cuatro pseudo-átomos en lugar de doce
entonces las interacciones de van der Waals entre dos moléculas de butano involucra el cálculo de 16 términos en lugar de 144. Otros hidrocarburos son representados
con frecuencia usando modelos de átomo unido. Muchos de los primeros cálculos
en proteı́nas usaron representaciones de átomo unido. En este caso, no todos los átomos de hidrógeno en la proteı́na son subsumados en sus átomos adyacentes, solo
aquellos que están unidos a átomos de carbono. Los átomos de hidrógeno ligados
a átomos polares como el nitrógeno y oxı́geno son capaces de participar en interacciones de puentes de hidrógeno, los cuales son modelados mucho mejor si estos
hidrógenos se representan explı́citamente.
Un inconveniente con el campo de fuerza es que los centros quirales podrı́an ser capaces de invertirse durante un cálculo. Se encontró que esto es un problema con los
campos de fuerza de átomo unido con proteı́nas. El carbono alfa en la unión péptida
está ligado a un átomo de hidrógeno y a la cadena lateral. Un modelo de campo de
fuerza de átomo unido no incluirı́a explı́citamente el hidrógeno alfa. Desafortunadamente, la estereoquı́mica en el carbono alfa puede entonces invertirse durante un
cálculo. Esto deberı́a evitarse como ocurre naturalmente dado que los aminoácidos
tienen una estereoquı́mica definida. Esta inversión podrı́a prevenirse a través del
uso de un término de torsión impropio (por ejemplo N-C-Cα -R) para mantener la
cadena lateral en la posición relativa correcta.
En un campo de fuerzas de átomo unido el centro de van der Waals es usualmente
asociado con la posición de un átomo pesado, es decir no el hidrógeno. Ası́, para
un grupo CH3 o CH2 el centro de van der Waals estarı́a localizado en el átomo de
carbono. Serı́a más preciso asociar el centro de van de Waals con una posición ligeramente fuera de la posición del carbono, para reflejar la presencia de los átomos de
hidrógeno.
5. La Dinámica Molecular
54
5.1.2.
Algoritmos de Integración
Una vez que las fuerzas en la configuración de los átomos al tiempo t han sido
calculadas el siguiente paso es generar una nueva configuración al tiempo t + ∆t
de acuerdo a la ecuación 5.1. Para un modelo simple de movimiento donde se desea
una solución tan exacta como sea posible, es necesario un algoritmo de alta precisión
para integrar la ecuación 5.1 con pasos pequeños. Sin importar que tan frecuente se
tengan que calcular las fuerzas. La situación para cada sistema usualmente estudiado con dinámica molecular es sin embargo muy diferente. En este caso es inútil
intentar determinar una solución muy detallada para átomos individuales dado que
la dinámica es caótica y los pequeños errores numéricos crecerán exponencialmente y afectarán las trayectorias. A primera vista esto podrı́a parecer terrible ya que
va totalmente en contra del concepto de simulación, pero solo refleja que las propiedades de equilibrio del sistema no son sensibles a los detalles de trayectorias
individuales. Es entonces inútil intentar reproducir los movimientos exactamente.
En lugar de ello, uno deberı́a asegurarse que cualquier parte razonablemente grande que sea extraı́da de una trayectoria será una buena descripción de una partı́cula
con las mismas condiciones iniciales, llamada trayectoria sombra.
Existen muchos algoritmos numéricos con buen funcionamiento por largo tiempo que solo requieren una sola evaluación de la fuerza por cada paso. Una de las
más frecuentemente usadas (y la mejor para la dinámica molecular) fué desarrollada por Verlet y desde entonces se ha vuelto una clase entera de integradores. Esta
basada en la adición y resta de expansiones de Taylor de la dependencia temporal
de las coordenadas ri a los tiempos t − ∆t y t + ∆t,
ri (t − ∆t)
=
ri (t + ∆t)
=
d
(∆t)2 d2
(∆t)3 d3
ri (t) +
ri (t) −
ri (t) + O ∆t4
2
3
dt
2! dt
3! dt
(5.8)
d
(∆t)2 d2
(∆t)3 d3
ri (t) + ∆t ri (t) +
ri (t) +
ri (t) + O ∆t4
dt
2! dt2
3! dt3
(5.9)
ri (t) − ∆t
que junto con la ecuación 5.1 nos brinda
ri (t + ∆t) ≈
vi
≈
−ri (t − ∆t) + 2ri (t) +
∆t2
Fi
mi
1
[ri (t + ∆t) − ri (t − ∆t)]
2∆t
(5.10)
(5.11)
El error de truncamiento que se introduce con este esquema es de orden (∆t)4 para las coordenadas y (∆t)3 para las velocidades. Sin embargo, un problema más
práctico con este enfoque es que las velocidades se obtienen de la diferencia de dos
5.1. Haciendo Fı́sica in Silico
55
Figure 5.3: El esquema de integración Leap-Frog. Las coordenadas ri (cuadros negros) y velocidades vi (cuadros grises) de los átomos son calculadas alternando pasos enteros y medios
de tiempo.
términos de la misma magnitud, haciéndola muy sensible a la precisión numérica y
a los errores de redondeo. Un algoritmo ligeramente modificado, pero teóricamente
equivalente, es el esquema Leap-Frog,
ri (t + ∆t) ≈ ri (t) + ∆tvi (t + ∆t/2)
∆t
vi (t + ∆t/2) ≈ vi (t − ∆t/2) +
Fi
mi
(5.12)
(5.13)
Esta es aún una aproximación a segundo orden a las ecuaciones de movimiento,
pero evita la diferencia entre términos grandes cuando se calculan las velocidades.
El nombre viene de la generación de posiciones y velocidades en pasos de tiempo
completos y medios, respectivamente. Van saltando como ranas entre si como se
muestra en la Figura 5.3.
El mayor inconveniente es que las velocidades están desplazadas de las posiciones por medio paso, pero en el software para dinámica molecular que se ha desarrollado esto se elude promediando las velocidades en más y menos medio paso para
obtener el mismo valor como se obtendrı́a con el algoritmo de Verlet sin errores de
redondeo.
El esquema Leap-Frog es muy atractivo, puede incluso integrar un oscilador armónico con solo seis puntos por periodo. Muchos de los algoritmos numéricos más avanzados requieren más de una evaluación de las fuerzas lo cual esencialmente los
hace inútiles para la dinámica molecular, dado que es la parte que consume más
5. La Dinámica Molecular
56
tiempo en cualquier simulación. Pasos de tiempo razonable cuando se simulan biomoléculas son del orden 1 fs (femtosegundo, 10−15 s), durante el cual un átomo
tı́picamente se mueve cerca de 0,004 nm. Una simulación que cubra 1 ns requerirá 1,000,000 pasos de evaluación de la fuerza e intregración, con cada paso envolviendo quizá 1,000,000 interacciones entre pares de partı́culas para un sistema de tamaño razonable. Aun ası́, con un programa bien optimizado esto es completamente
factible y toma menos de un dı́a en una computadora rápida.
5.2.
Definición de las Propiedades del Ensamble
La mayorı́a de las propiedades observadas en los experimentos no pueden ser
atribuı́das a los átomos individualmente, más bien son propiedades colectivas de
un gran número de átomos, por ejemplo la temperatura y la presión. Es importante
controlarlas en la simulación. Si comenzamos con una proteı́na y agua en una caja
se deberı́a proceder a cerca de 300 ◦ K y presión normal, no a 1500 ◦ K o presiones
termonucleares, lo cual causarı́a que se separara la molécula.
La temperatura T de un sistema está relacionada directamente con la energı́a
cinética promedio de los átomos y el número de grados de libertad Nf . Definimos
una temperatura microscópica momentánea T como
T (t) =
N
1 X
mi vi (t) · vi (t)
Nf kB i=1
(5.14)
donde kB es la constante de Boltzmann. La temperatura efectiva del sistema es
un promedio T = hT i sobre un intervalo de tiempo suficientemente largo. La temperatura inicial del sistema puede obtenerse asignando velocidades aleatorias con
una distribución gaussiana con la amplitud correcta de los átomos. Idealmente, esta
temperatura deberı́a mantenerse durante la simulación, pero debido por ejemplo a
los errores de truncamiento en el algoritmo de integración y el uso de truncamiento de la fuerza la temperatura necesita controlarse cuando se realizan simulaciones
prolongadas. Esto se puede lograr rescalando las velocidades de los átomos para
cada paso de tiempo con un factor calculado λ
s
λ=
1+
∆t
τT
T0
−1
T
(5.15)
donde T0 es la temperatura de referencia y τT una constante de temperatura de acoplamiento. Este esquema de acoplamiento débil producirá una relajación exponencial a la temperatura de referencia. Existen técnicas de acoplamiento de temperatura
5.2. Definición de las Propiedades del Ensamble
57
más elaboradas como el enfoque de Nosé-Hoover que es mejor manteniendo el ensamble estadı́stico del sistema.
No es trivial calcular la presión en sistemas condensados; la ley del gas ideal solo
es válida para densidades muy bajas donde los átomos no interactúan, lo cual no es
el caso de, por ejemplo, el agua. La presión debe obtenerse de la definición básica
de fuerza por unidad de área sobre las paredes de la celda de simulación1 . Esto no
es accesible directamente en la simulación, pero puede ser calculado mediante un
truco, usando el teorema del virial
2K = Ξ
(5.16)
donde K es el promedio del tensor de energı́a cinética traslacional
N
1X
K=
mi vi ⊗ vi
2 i=1
(5.17)
y Ξ el virial del sistema
Ξ=−
N
X
ri ⊗ Fi
(5.18)
i=1
Este virial tiene una componte interna Ξint de las fuerzas interiores al sistema y
una contribución externa Ξext causada por la presión p sobre la superficie de la celda
S
Ξint
= −
N
X
ri ⊗ Fint
i
(5.19)
i=1
Z
Ξext
pN r ⊗ dS
=
(5.20)
S
donde pN es la componente de la presión normal al elemento de superficie dS.
Para la presión isotrópica esto se puede simplificar extrayendo el valor escalar p de
la integral
Z
Z
Ξext = p r · dS = p ∇ · rdV = 3pV
(5.21)
S
V
donde cada pV se le atribuye a las dimensiones x, y y z. En el caso anisotrópico, se tiene una relación similar para cada componente del tensor completo. Esto
proporciona una fórmula para calcular la presión
1 De hecho, la definición más apropiada de presión es la derivada de la energı́a libre con respecto al
volumen, pero esto es equivalente
5. La Dinámica Molecular
58
K − Ξint
(5.22)
V
utilizando las velocidades, posiciones y fuerzas momentáneas en el sistema. La
presión escalar p es igual a un tercio de la traza de este tensor, es decir, el promedio
de las dimensiones x, y y z.
La presión resultante puede ser controlada a través del método de acoplamiento
muy similar a la temperatura, escalando las coordenadas de todas la partı́culas y el
tamaño de la caja cada paso de tiempo con un factor µ (comparar con la Eq. 5.15)
p=2
s
µ=
3
1+
∆t
β(P − P0 )
τp
(5.23)
Para un valor razonable de la compresibilidad del sistema β, esto producirá una
relajación exponencial de la presión, similar al acoplamiento de temperatura débil.
5.3.
Limitaciones
Aunque potencialmente es una técnica poderosa, es importante darse cuenta que
la dinámica molecular como cualquier otro método tiene limitaciones las cuales deben ser consideradas. Primero, las interacciones son enteramente clásicas. Mientras
que usualmente es apropiada, existen sus excepciones. De acuerdo a la mecánica
cuántica los átomos simples podrı́an atravesar las barreras de potencial y no podemos esperar describir las reacciones quı́micas en las cuales se forman y se rompen enlaces. Además, los átomos de hidrógeno son muy ligeros y sus movimientos
están en la frontera con la mecánica cuántica. Aparte de hacer cálculos de mecánica
cuántica, lo cual esta fuera de cuestión, uno podrı́a aplicar términos de corrección
a la energı́a y calor especı́fico de las oscilaciones en los enlaces. Alternativamente,
estos grados de libertad podrı́an ser eliminados y los enlaces tratados como rı́gidos,
aproximando el estado base de un oscilador mecano-cuántico. Esto tiene la ventaja
adicional de que el paso de integracin puede incrementarse significativamente (es
decir de 1 fs a 2 fs).
Si uno intenta simular un sistema aislado, esto significarı́a que muchos de los
átomos experimentarı́an una superficie grande y un lı́mite antinatural a un ambiente vacı́o. Para evitar esto, se utilizan condiciones de frontera periódicas. Los átomos
se colocan en una celda de simulación rectangular (o más general, triclı́nica), y cuando los átomos o vectores de interacción cruzan la frontera simplemente reaparecen
en el lado opuesto, como si el sistema fuera un cristal periódico. Los efectos de esta
aproximación disminuirán conforme el tamaño de la caja aumenta, pero es evidente
en muchas simulaciones.
5.3. Limitaciones
59
Finalmente, la precisión de la simulación es enteramente dependiente de la precisión del campo de fuerzas, el cual contiene varias aproximaciones y parámetros
inciertos. Parecerı́a una buena idea el comenzar con mecánica cuántica y derivar
detalladamente los valores de interacción individuales. Desafortunadamente esto
desprecia otra aproximación importante, se asume que todas las fuerzas actúan entre pares de átomos. Por supuesto uno podrı́a incluir interacciones de tres y cuatro
partı́culas, pero eso serı́a ridı́culamente caro. Una mejor alternativa que funciona
sorprendentemente bien es utilizar parámetros semiempı́ricos que han sido corregidos para parcialmente contribuir por las interacciones de orden mayor mientras
que siguen añadidas del par. Se siguen despreciando efectos dinámicos como polarizaciones en los enlaces. Para mejorar la velocidad del cálculo de las fuerzas, las
interacciones no ligadas son usualmente truncadas más allá de una distancia de 1-2
nm. Esto es una buena aproximación para las interacciones de Lennard Jones, pero
no para las electrostáticas si hay cargas libres en el sistema. Se han implementado
algoritmos como el Particle Mesh Ewald que puede realizar una suma completa de
las fuerzas entre las imágenes periódicas infinitas de todos los átomos, pero debido
a las condiciones periódicas de frontera estas sobre enfatizarán la periodicidad artificial del sistema y algunas veces introducirán un ordenamiento antinatural.
Sin embargo, siempre y cuando se mantengan en mente las aproximaciones y se
revisen cuidadosamente los resultados, la dinámica molecular es un método muy
confiable para estudiar los movimientos presentes en las macromoléculas biológicas, y los efectos de la mayorı́a de las aproximaciones disminuyen entre más grande
sea la escala del fenómeno que se estudia.
Capı́tulo 6
LAS SIMULACIONES
The question is not what you look at, but what you see.
Henry David Thoreau
6.1.
Configuración de los sistemas
E
l Aβ es producto de la división de la proteı́na transmembranal APP. La βsecretasa1 divide el dominio extracelular para generar la terminal N del Aβ y
entonces la γ-secretasa realiza una proteólisis inusual a la mitad del dominio transmembranal de la APP para producir la terminal C del Aβ. Los residuos del 29-40
del Aβ40 son del dominio transmembrana de la APP. Por esta razón en cada uno
de los sistemas que aquı́ presentamos siempre se colocó la LYS-28 en la interfaz
agua-bicapa. Con el objeto de estudiar la evolución conformacional del Aβ40 en dos
membranas diferentes, una con lı́pidos saturados y la otra con lı́pidos insaturados.
Se insertó el Aβ40 en membranas de DPPC y DOPC, los cuales son lı́pidos saturados e insaturados respectivamente. Estos sistemas se estudiaron por simulación de
dinámica molecular usando dos modelos ligeramente diferentes, el de All-atom y
el de United-Atom. Por un lado la simulación de All-atom se llevó a cabo usando el
campo de fuerza CHARMM27 por medio del paquete NAMD, mientras que el modelo de átomo unido se llevó a cabo mediante una versión modificada del campo de
fuerza GROMOS con ayuda del paquete GROMACS. Ası́ pues fueron preparados
un total de cuatro sistemas. Las coordenadas iniciales del Aβ40 con una secuencia
de aminoácidos:
DAEFRHDSGYEVHHQKLVFFAEDVGSNK28 GAIIGLMVGGVV
se obtuvieron de las estructuras RMN determinadas en micelas SDS a PH 5.1 (PDB
1BA4 en el banco de datos de proteı́nas). La estructura y topologı́a de DPPC para
1 Enzimas
que dividen una proteı́na que esta incrustada en la membrana celular.
6. Las Simulaciones
62
Figure 6.1: Estructura inicial del sistema DPPC-Agua, los fosfatos se muestran como esferas color naranja, los demás átomos que componen el lı́pido se muestran en color gris y las
moléculas de agua como pequeñas lineas de color rojo.
realizar simulación con el programa de dinámica molecular NAMD fue obtenida del
sitio web del Dr. Jeff Klauda, respecto a DOPC el Dr. Francisco Castro proporcionó la
estructura y topologı́a. Para el caso de las simulaciones con el programa GROMACS
fueron obtenidas del sitio web del grupo del Dr. Tieleman en el caso de DPPC y del
sitio web del grupo del Dr. Rainer Böckmann en el caso de DOPC.
A continuación describiremos por separado la construcción de los sistemas Aβ40 bicapa-agua para cada uno de los programas utilizados.
6.2.
NAMD
6.2.1.
Sistema Aβ40 -DPPC-Agua
La bicapa lipı́dica proviene de un sistema completamente hidratado y equilibrado durante 50 ns a área constante y que contenı́a inicialmente 36 moléculas de DPPC
(dipalmitoilfosfatidilcolina) por monocapa, para un total de 72 lı́pidos y una razón
de 30:1 aguas:lı́pido. El sistema inicial se puede apreciar en la Figura 6.1.
Dado que el número de aguas que contenı́a el sistema no era sufiente para cubrir
el péptido durante la inserción procedimos a agregarle agua hasta tener una razón
99:1 aguas:lı́pido ya que con este número de moléculas de solvente se cubrı́a ampliamente el péptido. Antes de insertar el péptido se realizó una minimización a 0
6.2. NAMD
63
Figure 6.2: Estructura inicial del Aβ40 con su respectiva correspondencia en representaciones.
a) Se muestra el péptido utilizando el código CPK, se pueden apreciar los diferentes átomos
que lo conforman. b) Se muestra la representación de la estructura secundaria del péptido.
◦
K para eliminar los posibles traslapes entre las moléculas de agua agregadas con
las que ya estaban en el sistema, posteriormente se llevó la temperatura a 323 ◦ K con
el fin de tener el DPPC en una fase lı́quida. Finalmente se realizó una equilibración
del sistema en el ensamble isobárico-isotérmico manteniendo constante el número
de partı́culas (N P T ).
Ya equilibrada la bicapa se procedió a la inserción del péptido. La estructura inicial
del Aβ40 que obtuvimos del banco de datos PDB (Protein Data Bank) se muestra en
la Figura 6.2.
El procedimiento se realizó con la ayuda del programa de visualización VMD.
Traslapamos las estructuras de la bicapa y el Aβ40 de tal manera que la LYS-28 estuviera en la interfaz Agua-DPPC. Identificamos los lı́pidos y las moléculas de agua
que debı́an ser removidos para dejar espacio para colocar el Aβ40 , se retiraron 2
lı́pidos y las moléculas de agua que estaban dentro de una distancia de 3 Å con
respecto al péptido. De la tabla 6.1 podemos apreciar que la carga neta del péptido
es -3, por ello en este punto se le agregaron 3 iones de Na para obtener un sistema
eléctricamente neutro.
Las dimensiones del sistema después del procedimiento anterior fueron las siguientes: x ∼ 44 Å, y ∼ 39 Å y z ∼ 177 Å. El sistema cuenta con 33224 átomos y se
puede apreciar en la Figura 6.3.
Nuevamente se realizó una minimización a 0◦ K para quitar los traslapes entre
6. Las Simulaciones
64
Residuo
N-terminus
Asp-1
Glu-3
Arg-5
Asp-7
Glu-11
Lys-16
Glu-22
Asp-23
Lys-28
C-terminus
Código de una letra
D
E
R
D
E
K
E
D
K
Estado de carga a pH 7
+
+
+
+
-
Table 6.1: Aminoácidos cargados del Aβ40 . Se aprecia que la carga neta del péptido es negativa.
Figure 6.3: Estructura inicial del sistema Aβ40 -DPPC-Agua, la LYS-28 se muestra en color
verde y se puede apreciar que fué colocada correctamente en la interfaz Agua-DPPC. El Aβ40
se muestra utilizando la representación de estructura secundaria.
las moléculas, se llevó la temperatura a 323 ◦ K y posteriormente se realizó una simulación de 50 ns bajo las siguientes condiciones:
El conjunto de parámetros CHARMM27 (MacKerell et al., 1998 Feller and
MacKerell, 2000) fué utilizado junto con el modelo TIP3 para el agua (Jorgensen et
6.2. NAMD
65
al., 1983). El campo de fuerzas corresponde al tipo All-atom.
La elección más natural de un ensamble en simulaciones de membranas y
en particular cuando se estudia la inserción de un péptido es el ensamble N P T dado que el área lateral del sistema Aβ40 /DPPC es desconocida.
La simulación se realizó en el ensamble N P T a T = 323◦ K y P = 1 atm.
Se utilizó un paso de tiempo de 1fs, para controlar la presión P se utilizó el
método del pistón de Langevin con un coeficiente de amortiguamiento de 1 ps−1 y
el periódo del pistón fué de 200 fs.
Se utilizó PME para calcular las fuerzas electrostáticas. El corte a las interacciones de van der Waals se realizó de manera suave2 comenzando en 10 Å y terminando en 11 Å.
Se impusieron condiciones de frontera periódicas (PBC).
Estas condiciones fueron aplicadas a todos los sistemas que se simularon con NAMD
exceptuando la temperatura, ya que para el caso que se describe a continuación se
utilizó 296 ◦ K en lugar de 323 ◦ K.
6.2.2.
Sistema Aβ40 -DOPC-Agua
Esta bicapa proviene de un sistema pre-equilibrado, totalmente hidratado el cual
contenı́a 72 moléculas de DOPC (dioleoilfosfatidilcolina) 36 por monocapa con una
razón de 32:1 aguas:lı́pido. En este caso tampoco era suficiente el agua inicial del
sistema para cubrir el péptido durante la inserción por lo que se aumentó el número
de moléculas de solvente hasta tener una tasa de 89:1 aguas:lı́pido. Se realizó el mismo procedimiento de minimización, aumento de temperatura y equlibración hasta
tener la membrana de DOPC en fase lı́quida a 296 ◦ K. Posteriormente se ancló el
péptido siguiendo el procedimiento descrito en la sección anterior tal que nuevamente la LYS-28 estuviera en la interfaz Agua-DOPC. Fueron removidas también
dos moléculas de DOPC y las aguas que se encontraban dentro de una inmediación de 3 Å del Aβ40 . También se agregaron 3 iones de Na para obtener un sistema
eléctricamente neutro.
Las dimensiones del sistema fueron las siguientes: x ∼ 48 Å, y ∼ 44 Å y z ∼ 140
Å y cuenta con 29095 átomos.
Nuevamente se realizó minimización, aumento de temperatura a 296◦ K y finalmente una simulación de 100ns en el ensamble isobárico-isotérmico bajo condiciones
iguales a las del sistema descrito en la sección anterior con excepción de la temperatura.
2 Aquı́ nos referimos a que el corte no se realiza de forma abrupta sino que se comenienza en 10 Å y se
empieza a llevar el potencial de van der Waals a cero terminando en la distancia de corte 11 Å.
6. Las Simulaciones
66
6.3.
GROMACS
En la utilización de este programa seguimos una metodologı́a un tanto distinta
para configurar los sistemas. Utilizamos la técnica inflategro desarrollada por el Dr.
Tieleman para acomodar los lı́pidos alrededor del péptido y posteriormente hidratar
el sistema hasta cubrir por completo el Aβ40 .
6.3.1.
Sistema Aβ40 -DPPC-Agua
La bicapa DPPC proviene de un sistema pre-equilibrado y totalmente hidratado
que contiene 128 moléculas de DPPC con una razón 28:1 aguas:lı́pido. El método del
Dr. Tieleman parte de tener un sistema sólo con los lı́pidos y el péptido traslapados,
considerando ya la posición de la LYS-28 en la interfaz. Posteriormente se va haciendo un rescalamiento del tamaño del sistema de tal forma que se vayan acomodando
los lı́pidos alrededor del Aβ40 y removiendo los lı́pidos que estén dentro de un radio
de 3 Å. Después de cada rescalamiento se hace una minimización. Durante este proceso se mantiene completamente fijo a el Aβ40 . El rescalamiento se repite hasta que
se tenga una razón de área/lı́pido3 deseada. Posteriormente volvimos a hidratar el
sistema de tal forma que se cubriera todo el péptido y se agregaron 3 iones de Na
para obtener un sistema eléctricamente neutro. Finalmente se tuvo un total de 126
moléculas de DPPC y una razón de 105:1 aguas:lı́pido. Las dimensiones del sistema
después del procedimiento antes mencionado son las siguientes: x ∼ 65 Å, y ∼ 65
Å y z ∼ 150 Å con un total de 46456 átomos.
Se realizó una minimización a 0 ◦ K, se llevó la temperatura a 323 ◦ K para tener el
DPPC en una fase lı́quida y posteriormente se realizó la simulación en el ensamble
N P T durante 100 ns bajo las siguientes condiciones:
Los parámetros del campo de fuerzas desarrollados y empleados por el
Dr. Tieleman fueron aplicados a la bicapa DPPC y el campo de fuerzas Gromos96
53a6 se utilizó en el resto del sistema. El modelo para el agua es SPC. El campo de
fuerzas corresponde al tipo united-atom.
La simulación se realizó en el ensamble N P T a T = 323 ◦ K y P = 1 atm con
un paso de intergración de 2 fs.
Se utilizó el método de acoplamiento de temperatura de Berendsen con una
constante de acoplamiento de 0.1 ps. Cada grupo (péptido, lı́pidos y solvente/ions)
fué acoplado a un baño de temperature separado.
Se utilizó el método de acoplamiento de presión de Berendsen de forma
isotrópica con una presión de referencia de 1 atm y una constante de acoplamiento
3 En
el capı́tulo 7 se describirá este parámetro.
6.3. GROMACS
67
de 0.1 ps.
Las interacciones electrostáticas fueron calculadas utilizando PME.
Se utilizaron condiciones de prontera periódicas (PBC).
Estas condiciones fueron aplicadas a los sistemas que se simularon con GROMACS
exceptuando la temperatura, ya que para DOPC se utiliza una temperatura de 296
◦
K.
6.3.2.
Sistema Aβ40 -DOPC-Agua
La bicapa DOPC-Agua proviene también de un sistema pre-equilibrado totalmente hidratado con 128 moléculas de DOPC, es decir una razón de 37:1 aguas:lı́pido. Se realizó el mismo procedimiento de inflategro del Dr. Tieleman después de
haber traslapado el péptido con los lı́pidos y colocado la LYS-28 en la interfaz.
Se realizó el mismo procedimiento de hidratación y de obtención de neutralidad
eléctrica descritos en la sección anterior. Después de este procedimiento se obtuvo
un sistema con 126 lı́pidos y una razón 117:1 aguas:lı́pido. Las dimensiones del sistema después del procedimiento antes mencionado fueron las siguientes: x ∼ 69 Å,
y ∼ 68 Å y z ∼ 150 Å y cuenta con 47050 átomos.
Se realizó una minimización a 0 ◦ K, se llevó la temperatura a 296 ◦ K para tener el
DOPC en una fase lı́quida y posteriormente se realizó la simulación en el ensamble
N P T durante 100 ns bajo las mismas condiciones descritas en la sección anterior.
En la Tabla 6.2 se describe como se identificarán cada uno de los sistemas de
aquı́ en delante.
Las simulaciones por dinámica molecular fueron realizadas en Centro Nacional
Modelo
A1
A2
A3
B1
B2
Lı́pidos
70 DPPC
70 DOPC
70 DPPC
126 DPPC
126 DOPC
Programa
NAMD
NAMD
NAMD
GROMACS
GROMACS
Tensión Superficial γ (dyn/cm)
0
0
50
0
0
Table 6.2: Sumario de la simulaciones.
de Supercómputo del Instituto Potosino de Investigación Cientı́fica y Tecnológica
(IPICyT) Cluster IBM E-1350 (Argentum) y Supercomputadora Cray XD1 (Unpotosi). Los análisis fueron realizados utilizando las facilidades proporcionadas por
el programa GROMACS. Otro software adicional utilizado fué VMD, para visualizar las estructuras moleculares, y gnuplot para realizar las gráficas de las áreas por
68
6. Las Simulaciones
lı́pido. Para el caso de los análisis de la estructura secundaria se usó el método de
definición de estructura secundaria de proteı́nas (DSSP) [35].
Capı́tulo 7
LOS RESULTADOS
Science... never solves a problem without creating ten more.
George Bernard Shaw
nicialmente se configuraron dos sistemas para analizar las transiciones conformacionales del Aβ40 asociadas con la agregación y neurotoxicidad del Aβ. Dichos
sistemas corresponden a A1 y A2, descritos en el capı́tulo anterior. Dado que existe
un parámetro experimental, que es el área por lı́pido 1 (AL ) para las bicapas DPPC
y DOPC lo podemos utilizar para saber si se encuentran en fase lı́quida o gel. Para
el caso de una bicapa de DPPC se ha reportado experimentalmente una área por
2
2
lı́pido de 62.9 Å y para DOPC de 72.5 Å [6]. Nos dimos a la tarea de monitorear
nuestros sistemas para ver la evolución del parámetro antes mencionado, AL . De la
Figura 7.1 podemos ver que los valores obtenidos de nuestra simulación están muy
por debajo de los valores experimentales, lo que se refleja en un alto grado de empaquetamiento de lı́pidos, comprometiendo la fluidez bidimensional de la membrana.
Por otro lado en el apéndice A se muestran las energı́as totales para cada uno de los
sistemas simulados.
I
7.1.
Sistemas A1, A2 y A3
En el caso de A1 vemos que los parámetros del campo de fuerza CHARMM27
no conducen a una AL acorde al valor experimental, en la Figura 7.2a se aprecia el
ordenamiento de los lı́pidos de la bicapa caracterı́sticos de una fase gel. Para A2 incluso cuando no se tiene el AL en acuerdo con el valor experimental se mantiene una
fase lı́quida (Figura 7.2b), esto se debe a que el DOPC es un lı́pido insaturado y por
lo tanto el “doblez” de las colas hidrofóbicas a nivel de los dobles enlaces impide el
empaquetamiento como el que se da para el DPPC que es saturado y no presenta
doble enlace en las colas hidrofóbicas. Dado que las condiciones necesarias para el
1 Resulta
de dividir el área de una monocapa entre el número de lı́pidos que la conforman
70
7. Los Resultados
Figure 7.1: Evolución de AL para los sistemas A1y A2. En el caso de A1 vemos que AL es ∼ 48
2
2
Å (el valor experimental para DPPC es de 62.9 Å ) lo que se ha reportado como correspondiente a una fase tipo gel. Para A2 a pesar de que el área no corresponde a la experimental
2
(72.5 Å ) se sigue mateniendo una fase lı́quida como se observa en la Figura 7.2.
estudio de los cambios conformacionales del Aβ40 requieren de una fase lı́quida de
la bicapa lipı́dica nos dimos a la tarea de encontrar una manera de garantizar dicho
estado. Diferentes trabajos, como por ejemplo [3], han reportado el problema de que
los parámetros de los diversos campos de fuerza no llevan al valor experimental AL .
Para tratar de mantener A1 a una AL que nos proporcione una fase lı́quida decidimos aplicar una tensión superficial γ a la bicapa y simularlo en el ensamble N P γT .
La tensión que nos brindó un AL más “estable” fue la de 50 dyn/cm y se puede
apreciar la evolución del área por lı́pido en la Figura 7.3b.
Como resultado de la simulación durante 100 ns del sistema con tensión superficial de 50 dyn/cm denominado A3 podemos ver en la Figura 7.3a que predomina
la estructura de hélice, π (hélice 5) y α, durante casi todo el periodo de simulación,
el resto del Aβ40 se mantiene en forma de espiral que como vimos en el capı́tulo
7.1. Sistemas A1, A2 y A3
71
Figure 7.2: a) Sistema A1, construı́do con el lı́pido saturado DPPC y modelado con
CHARMM27 usando NAMD. Se puede apreciar un alto grado de ordenamiento de los lı́pidos, caracterı́stico de una fase gel. b) Sistema A2, construı́do con el lı́pido insaturado DOPC
y modelado con CHARMM27 usando NAMD. En este caso la insaturación debida al doble
enlace de las colas hidrofóbicas del DOPC ayuda a preservar la fase lı́quida, a pesar de la
reducción en el AL .
3 se refiere a la estructura que no exhibe periodicidad. La transición de hélice α a
π se la atribuı́mos a las fluctuaciones del AL ya que se reflejan en un ligero empaquetamiento de las colas hidrófóbicas, las cuales presionan el péptido y esto hace
que los aminoácidos que presentan periodicidad tipo hélice se aprieten y haya más
residuos por cada vuelta. De la Figura 7.4c vemos la evolución del posicionamiento
de Aβ40 .Para que se formen las placas amiloides el Aβ debe salir de la bicapa y autoasociarse.
Ahora analizamos los resultados obtenidos para el sistema A2, formado por el
lı́pido insaturado DOPC. La Figura 7.4b muestra la evolución del AL , la cua está ∼
2
13 Å por debajo del valor experimental, lo cual sugiere, al igual que en el caso
de DPPC, un alto grado de empaquetamiento, sin embargo la figura 7.4b muestra
claramente que a pesar de esto la bicapa no se ordena en fase gel como ocurre con
la bicapa correspondiente al sistema A1 (Figura 7.4a). En la Figura 7.4a se aprecia
72
7. Los Resultados
Figure 7.3: Resultados del sistema A3. a) La evolución de la estructura secundaria del péptido calculada utilizando DSSP. Las estructuras fueron analizadas cada 500 ps, los recuadros
de colores indican el código para las diferentes estructuras secundarias presentes durante la
simulación, las imágenes de las estructuras del Aβ40 estan diseñadas de acuerdo al análisis
de DSSP. b) Evolución del AL a través de simulación. c) Posición del péptido a lo largo de
la simulación con respecto a la interfaz agua-dppc, el Aβ40 se muestra en color negro, los
átomos de fósforo (P) se muestran en color naranja para la mejor distinción de la interfaz,
los óxigenos del las moléculas de agua se muestran en color rojo y las cadenas aciles de los
lı́pidos en color verde.
la evolución de la estructura secundaria del péptido, como en el caso de A3 en su
mayor parte se conserva una estructura tipo hélice, el hecho de la transición de α
a π también lo atribuı́mos a que aunque contamos con una fase lı́quida el valor de
AL está muy por debajo del experimental, sugiriendo una interacción más intensa
7.1. Sistemas A1, A2 y A3
73
Figure 7.4: Resultados del sistema A2. a) La evolución de la estructura secundaria del péptido calculada utilizando DSSP. Las estructuras fueron analizadas cada 500 ps, los recuadros
de colores indican el código para las diferentes estructuras secundarias presentes durante la
simulación, las imágenes de las estructuras del Aβ40 estan diseñadas de acuerdo al análisis
de DSSP. b) Evolución del AL a través de simulación. c) Posición del péptido a lo largo de
la simulación con respecto a la interfaz agua-dppc, el Aβ40 se muestra en color negro, los
átomos de fósforo (P) se muestran en color naranja para la mejor distinción de la interfaz,
los óxigenos del las moléculas de agua se muestran en color rojo y las cadenas aciles de los
lı́pidos en color verde.
entre el péptido y las moléculas de DOPC, conduciendo a un ligero incremento en
el número de aminoácidos por vuelta en la hélice. La Figura 7.4c muestra que al
igual que ocure con la membrana de DPPC el Aβ40 tampoco se libera de la bicapa
de DOPC durante el tiempo de corrida de la simulación.
7. Los Resultados
74
7.2.
Sistemas B1 y B2
Posterior a estas simulaciones decidimos preparar sistemas con bicapas equivalentes utilizando el modelo de átomo unido con el campo de fuerza GROMOS96
53a6 usando el paquete de simulacı́ón GROMACS (sistemas B1 y B2 descritos en el
capı́tulo 6). A continuación mostramos los resultados obtenidos.
En el caso de B1 podemos apreciar en la Figura 7.5b que los parámetros del campo
de fuerza de GROMACS reproducen remarcablemente bien el valor experimental
2
del AL , cuyo valor es de 62.9 Å . En nuestro caso el valor promedio durante el tiem2
po de la simulación es de 61.6 Å . En la Figura 7.5a vemos que el Aβ40 experimenta
grandes cambios conformacionales, con una desaparición casi total de la estructura
de hélice α e incluso, a partir de 65 ns empieza a aparecer la presencia de hoja beta. En la Figura 7.5c se observa que el Aβ40 tiende a estar más cerca de la interfaz
agua-DPPC comparado con el caso de la simulación correspondiente a NAMD y
CHARMM27 (sistema A3). Esta simulación sugiere una mayor tendencia del péptido a salir de la membrana, llevando a cabo al mı́smo tiempo un cambio conformacional en su estructura secundaria de hélice a hoja beta, acorde a la evolución del
péptido en estudios in vitro e in vivo.
Finalmente tenemos el sistema B2. Como se observa en la Figura 7.6b. El AL simulado se encuentra considerablemente debajo del valor experimental pero aun ası́,
el sistema no muestra caracterı́sticas de una fase gel por lo que pudimos realizar el
análisis de la estructura secundaria del Aβ40 en este entorno de membrana (DOPC).
De dicho análisis podemos observar en la Figura 7.6a que se mantiene la estructura
de hélice α a través de los 100 ns pero además a partir de los 8 ns empieza a aparecer
de forma intermitente la estructura de hoja β hasta estabilizarse a partir de los 78 ns.
7.2. Sistemas B1 y B2
75
Figure 7.5: Resultados del sistema B1. a) La evolución de la estructura secundaria del péptido calculada utilizando DSSP. Las estructuras fueron analizadas cada 500 ps, los recuadros
de colores indican el código para las diferentes estructuras secundarias presentes durante la
simulación, las imágenes de las estructuras del Aβ40 estan diseñadas de acuerdo al análisis
de DSSP. b) Evolución del AL a través de simulación. c) Posición del péptido a lo largo de
la simulación con respecto a la interfaz agua-dppc, el Aβ40 se muestra en color negro, los
átomos de fósforo (P) se muestran en color naranja para la mejor distinción de la interfaz,
los óxigenos del las moléculas de agua se muestran en color rojo y las cadenas aciles de los
lı́pidos en color verde.
76
7. Los Resultados
Figure 7.6: Resultados del sistema B2. a) La evolución de la estructura secundaria del péptido calculada utilizando DSSP. Las estructuras fueron analizadas cada 500 ps, los recuadros
de colores indican el código para las diferentes estructuras secundarias presentes durante la
simulación, las imágenes de las estructuras del Aβ40 estan diseñadas de acuerdo al análisis
de DSSP. b) Evolución del AL a través de simulación. c) Posición del péptido a lo largo de
la simulación con respecto a la interfaz agua-dppc, el Aβ40 se muestra en color negro, los
átomos de fósforo (P) se muestran en color naranja para la mejor distinción de la interfaz,
los óxigenos del las moléculas de agua se muestran en color rojo y las cadenas aciles de los
lı́pidos en color verde.
Conclusiones
Utilizando la técnica de simulación por dinámica molecular se estudiaron las
transiciones conformacionales del Aβ40 asociadas con la agregación y la neurotoxicidad del Aβ40 en dos entornos membranales distintos compuestos por lı́pidos de
DPPC y DOPC respectivamente. La elección de estos dos tipos de lı́pidos se hizo
con el objetivo de ver las diferencias en los cambios conformacionales del péptido en función de las propiedades de saturación e insaturación de dichas moléculas
anfifı́licas. Inicialmente se configuraron 2 sistemas (sistema A1, con el lı́pido saturado DPPC, y sistema A2, con el lı́pido monoinsaturado DOPC) para ser simulados
con los parámetros del campo de fuerzas CHARMM27 en el programa de dominio
público NAMD. Observamos que dichos parámetros producen sistemas cuyas áreas
por lipido AL están muy por debajo de los valores reportados experimentalmente.
2
En el caso de A1 se obtiene un valor de 48 Å lo que nos indica que la bicapa se
encuentra en una fase gel. Dicho estado no es adecuado para nuestro estudio ya que
en las membranas de las células es conocido que se cuenta con bicapas lipı́dicas en
fase lı́quida. Para lograr tener los lı́pidos de la bicapa de DPPC en una fase lı́quida optamos por aplicar una tensión superficial (A3) tal que nos diera un AL lo más
cercana posible al valor experimental y obtener ası́ una fase fluida donde poder estudiar la evolución del Aβ40 . En el caso de A2 optamos por realizar la simulación
sin aplicación de tensión superficial a pesar de no obtener un AL acorde al valor
experimental, esto debido a que la membrana conserva la fase lı́quida.
Posteriormente recurrimos a GROMACS y el campo de fuerzas GROMOS96
53a6 donde configuramos 2 sistemas equivalentes a los antes mencionados, que denominamos B1 y B2. En estos sistemas, a diferencia de A1 y A2, notamos una gran
estabilidad para el valor de las AL y en el caso de B1 se obtuvo un valor de aproxi2
madamente 61.5 Å durante el periodo de 100 ns que duró la simulación, lo cual es
bastante cercano al valor experimental reportado en la literatura [6]. En el caso de
B2, a pesar de la estabilidad mencionada en la evolución del área por lı́pido el valor
2
2
61.8 Å es considerablemente inferior al valor experimental reportado de 72.5 Å ,
aunque se conserva la fase fluı́da. De los resultados de las simulaciones podemos
78
Conclusiones
concluir lo siguiente:
El mayor cambio conformacional del péptido Aβ40 se presenta en el sistema
B1, donde se observa una transición de estructura hélice alfa a una conformación con escasa estructura secundaria al cabo de 100 ns. Sin embargo, durante
esta evolución se pudo apreciar la formación de estructura en hoja beta en los
residuos 5-10 durante el intervalo de tiempo de 65 ns a 95 ns. Esto representa
un resultado muy alentador pues la estructura en hoja beta es la estructura
secundaria representativa de los agregados fibrilares tı́picos de la enfermedad
de Alzheimer.
Con excepción del sistema A3, donde se aplicó una tensión de superficie para
obligar al sistema a tener un AL cercana al valor experimental, el sistema B1 es
el único cuya AL reproduce satisfactoriamente el experimento.
En el sistema A3, aun cuando se logró que evolucionara hacia un AL acorde al
experimento después de aplicar una tensión superficial, el péptido mantiene
su estructura secundaria en hélice, presentado dos regiones, una con hélice
alfa y otra con hélice-5, esta última más compacta que la hélice alfa. Esto indica
que la tensión superficial aplicada no es suficiente para corregir las deficiencias
del campo de fuerza. Es verdad que corrige el AL , sin embargo, la membrana
sigue presentando un aspecto de tipo fase gel, como puede apreciarse en la
Figura 7.3c, donde las colas hidrofóbicas del DPPC se encuentran estiradas
mostrando un ordenamiento que dista mucho de una fase lı́quida, como es el
caso de las colas hidrofóbicas del sistema B1.
Para que se formen las placas amiloides, los péptidos Aβ deben salir de la bicapa y autoasociarse. No observamos la salida del péptido de la bicapa durante el tiempo de corrida de nuestras simulaciones. Sin embargo, en el sistema
B1, el cual reproduce mejor que todos las caracterı́sticas de fluidez y AL de la
membrana, el péptido muestra una tendencia hacia la interfaz agua-cabezas
polares, un paso previo quizá hacia su salida de la membrana , lo cual solo podrá verificarse con una corrida de tiempo mucho mayor a los 100 ns de
nuestras simulaciones.
Los campos de fuerza, tanto CHARMM como GROMOS, necesitan de una optimización de sus parámetros con el objetivo de que reproduzcan membranas
cuya AL y fluidez estén en acuerdo con lo que se observa experimentalmente.
Apéndice A
Figure 7.7: Evolución de la energı́a total a través de la trayectoria de 100 ns para los sistemas
simulados con el campo de fuerza CHARMM27 mediante el programa NAMD. Se aprecia
que rápidamente se alcanza el equilibrio.
80
Conclusiones
Figure 7.8: Evolución de la energı́a total a través de la trayectoria para los sistemas simulados con el campo de fuerza GROMOS mediante el programa GROMACS. Se aprecia que el
equilibrio se alcanza posteriormente a los 5 ns de simulación.
Bibliografı́a
[1] Yechun Xu, Jianhua Shen, Xiaomin Luo, Weiliang Zhu, Kaixian Chen, Jianpeng Ma, and Hualiang Jiang, Conformational transition of amyloid βpeptide. PNAS 102, 15 5403-5407 (2005).
[2] Justin A. Lemkul, David R. Bevan, A comparative molecular dynamics analysis of the amyloid β-peptide in a lipid bilayer, Elsevier. 470(2008) 54-63.
[3] Morten O. Jensen, Ole G. Mouritsen and Günther H. Peters, Simulations of a
Membrane-Anchored Peptide: Structure, Dynamics, and Influence on Bilayer
Properties, Biophys J. 86:3556-3575.
[4] Eva Y. Chi, Canay Ege, Amy Winans, Jaroslaw Majewski, Guohui Wu, Kristian
Kjaer, Ka Yee C. Lee, Lipid membrane templates the ordering and induces the
fibrillogenesis of Alzheimer’s disease amyloid-β peptide. Proteins: Structure,
Function, and Bioinformatics, 72, 1-24 (2008).
[5] Erik Lindahl, Computacional modeling of biological membrane and interface
dynamics. Stockholm 2001. Doctoral dissertation royal institute of technology
departament of physics.
[6] John F. Nagle, Stephanie Tristram-Nagle, Structures of lipid bilayer, Biochimica et Biophysica Acta, 1469 (2000) 159-195.
[7] Ramón Latorre, José López-Barneo, Francisco Bezanilla, Rodolfo Llinás,
Biofı́sica y fisiologı́a celular, Universidad de Sevilla secretariado de publicaciones, 1996.
[8] David Whitford, Proteins structure and function, John Wiley & Sons, Ltd, 2005.
[9] Ole G. Mouritsen, Life-as a matter of fat the emerging science of lipidomics, Springer, 2005.
[10] Christopher K. Mathews, K. E. Van Holde, Kevin G. Ahern, Biochemistry, An
imprint of Addison Wesley Longman, Inc, 2000.
82
BIBLIOGRAFÍA
[11] Wei Zhao, Tomasz Rg, Andrey A. Gurtovenko, Ilpo Vattulainen and Mikko
Karttunen, Atomic-Scale Structure and Electrostatics of Anionic Palmitoyloleoylphosphatidylglycerol Lipid Bilayers with N a+ Counterions, Biophys J.
92:1114?1124.
[12] Shirley W. I. Siu, Robert Vácha, Pavel Jungwirth and Rainer A. Böckmann,
Biomolecular simulations of membranes: Physical properties from different
force fields, J. Phys. Chem. 128. 125103 (2008).
[13] Jacob Sonne, Morter O. Jensen, Flemming Y. Hansen, Lars Hemmingsen and
Günther H. Peters, Reparameterization of AII-Atom Dipalmitoylphosphatidylcholine lipid parameters enables simulation of fluid bilayers at zero tension. Biophys J. 92:4157-4167 (2007).
[14] James C. Phillips, Rosemary Braun, Wei Wang, James Gumbart, Emad
Tajkhorshid, Elizabeth Villa, Christophe Chipot, Robert D. Skeel, Laxmikant
Kalé, Klaus Schulten, Scalable molecular dynamics with NAMD. J. Comput.
Chem. 26:1781-1802 (2005).
[15] Brigita Urbanc, Luis Cruz, David B. Teplow and H. Eugene Stanley, Computer
simulations of Alzheimer’s amyloid β-protein folding and assembly. Current
Alzheimer Research 3:493-504 (2006).
[16] Holmes, Clive, Boche, Delphine, Wilkinson, David, Yadegarfar, Ghasem, Hopkins, Vivienne, Bayer, Anthony, Jones, Roy W., Bullock, Roger, Love, Seth,
Neal, James W., Zotova, Elina and Nicoll, James A. R. (2008), Long-term effects of Aβ(42) immunisation in Alzheimer’s disease: follow-up of a randomised, placebo-controlled phase I trial, Lancet, 372, (9634), 216-223.
[17] Benoit Roux, Commentary: Surface tension of biomembranes, Biophys J.
71:1346-1347 (1996).
[18] Fritz Jähnig, What is the surface tension of a lipid bilayer menbrane?
[19] I. Chandrasekhar, D. Bakowies, A. Glättli, P. Hünenberger, C. Pereira and
W. F. Van Gunsteren, Molecular dynamics simulation of lipid bilayers whit
GROMOS96: Application of surface tension. Molecular Simulation 31:543-548
(2005).
[20] Scott E. Feller and Richard W. Pastor, On simulating lipid bilayer with an applied surface tension: Periodic bondary conditions and undulations. Biophys.
J. 71:1350-1355 (1996).
BIBLIOGRAFÍA
83
[21] Teresa K. Attwood, The Babel of Bioinformatics. Science, 290, 5491, 471-473
(2000).
[22] Siu SW, Vácha R, Jungwirth P, Böckmann RA., Biomolecular simulations of
membranes: physical properties from different force fields. J Chem Phys.
128(12):125103 (2008).
[23] Balali-Mooda Kia ; Ashley Richard H. ; Hauss Thomas ; Bradsaw Jeremy
P, Neutron diffraction reveals sequence-specific membrane insertion of prefibrillar islet amyloid polypeptide and inhibition by rifampicin. FEBS letters
579, 5, 1143-1148 (2005).
[24] Peter J. Steinbach , Introduction to
http://cmm.cit.nih.gov/intro simulation/
Macromolecular
Simulation.
[25] Alexander D. Mackerell Jr., Empirical force fields for biological macromolecules: Overview and issues. J. Comput. Chem. 25, 13, 1584-1604 (2004).
[26] Sameer Varma, See-Wing Chiu and Eric Jakobsson, The influence of amino
acid protonation states on molecular dynamics simulations of the bacterial
porin ompf. Biophys J. 90:112-123 (2006).
[27] Canay Ege and Ka Yee C. Lee, Insertion of alzheimer’s Aβ40 peptide into lipid
monolayers. Biophys J. 87:1732-1740 (2004).
[28] Sanders CR, Nagy JK. , Misfolding of membrane proteins in health and disease: the lady or the tiger?. Current opinion in structural biology 10:438-442
(2000).
[29] Murray Coles, Wendy Bicknell, Andrew A. Watson, David P. Fairlie and David J. Craik, Solution structure of amyloid β-peptide(1-40) in a water-micelle
environment. Is the menbrane-spanning domain where we think it is?. Biochem. 37, 11064-11077 (1998).
[30] Georg Büldt and Roland Wohlgemuth, The headgroup conformation of phosoholipids in membranes. J. Membrane Biol. 58, 81-100 (1981).
[31] Salonen IS, Eklund KK, Virtanen JA, Kinnunen PK., Comparison of the effects
of NaCl on the thermotropic behaviour of sn-1ánd sn-3’stereoisomers of 1,2dimyristoyl-sn-glycero-3-phosphatidylglycerol. Biochim. Biophys. Acta. 982,
205-215 (1989).
[32] I. Pascher, S. Sundell, K. Harlos and H. Eibl, Conformation and packing properties of membrane lipis: The crystal structure of sodium dimyristoylphosphatidylglycerol. Biochim. Biophys. Acta 896, 77-88 (1987).
84
BIBLIOGRAFÍA
[33] V. A. Tverdislov, L. V. Yakovenko and A. A. Zhavoronkov, Chirality as a problem of biochemical physics. Russian Journal of General Chemistry 77, 19942005 (2007).
[34] Scott E. Feller, Moleculardynamics simulation of lipid bilayers, ELSEVIER
217-223 (2000).
[35] Kabsch W, Sander C., Dictionary of protein secondary structure: pattern recognition of hydrogen-bonded and geometrical features. Boipolymers. 22, 25772637 (1983).