Tecnología de DNA recombinante
Anuncio

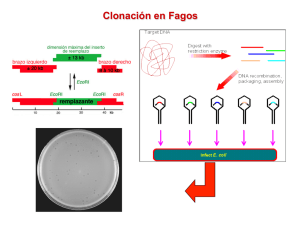
Tecnología de DNA recombinante Esther Zoraya Vega, Ph.D. Certificado en Biotecnología DNA recombinante Es la tecnología que permite combinar DNA de diferentes organismos en una sola molécula de DNA. DNA recombinante Stanley Cohen Herbert Boyer DNA recombinante Enzimas de restricción Son endonucleasas, que pueden romper enlaces fosfodíester en el DNA en secuencias específicas. Enzimas de restricción Enzimas de restricción Las enzimas de restricción reconocen secuencias palindromes de 4 a 8 bases. Enzimas de restricción Las enzimas de restricción no afectan el DNA del organismo que lo produce Mapas de restricción Mapas específicos que se obtienen por enzimas de restricción que se puede generar utilizando cualquier molécula de DNA ó genomas de cualquier organismo. Replicación del DNA- in vitro PCR Descubierta en 1985 por Kary Mullis. Recibío el Premio Novel en 1993. PCR Técnica que permite la amplificación de una secuencia específica de DNA millones de veces. PCR se fundamenta en el fenómeno de la extensión de un “primer” por la polimerasa de DNA descubierto en los 60’s. Ha revolucionado muchas áreas de investigación como: diagnóstico de enfermedades, medicina forense y evolución molecular. PCR PCR utiliza la amplificación enzimática para aumentar el número de copias de cualquier fragmento de DNA hasta 6000 pares de bases de largo. Replicación del DNA In vitro PCR PCR..\Certificado en Biotecnologia\pcrwin.zip PCR Reacción se somete a múltiples ciclos de síntesis que consisten de: Desnaturalización Calentamiento hasta cerca a 94oC, rompiendo los puentes de hidrógeno entre las bases, y la doble hebra de DNA se separa. PCR Hibridización (annealing) La disminución de la temperatura a ~ 65o permite que las sondas (primers) hibridizen con su secuencia complementaria en la hebra sencilla de DNA. La temperatura óptima depende de la proporción de AT a GC en la secuencia de la sonda. PCR Cada ciclo dura ~ 2 minutos. 25 ciclos de síntesis produce, teóricamente, 1,000,000 de copias de la secuencia blanco. 25 ciclos toma aproximadamente 1 hr. PCR Detección Clonaje Generación de múltiples copias de fragmentos de DNA por amplificación ó multiplicación de moléculas de DNA recombinante en un hospedero como una bacteria. Replicación del DNAClonaje del DNA Requisitos de los vectores de clonaje Origen de replicación Marcador dominante – gen que confiera resistencia a antibíotico Lugares únicos de restricción Vectores de clonajepBR322 Plasmidios – se comportan como mini-cromosomas + y pBR322 – tet amp+; fragmentos pequeños ~ 5 kb pUC18 Se producen en # alto de copias. MCS- (polylinker) lugar de inserción DNA Resistencia a amp y lacZ gene En presencia de IPTG y X-gal, las colonias son azules (por la presencia de la enzima activa). Vectores de clonaje Fagos – derivados de fago λ (10-15 kb) Cíclico lítico es posible; lisogénico no Contiene “cos ends” del fago (para hacer λ circular) Presencia de placas en gramas de E. coli indica la presencia de moléculas recombinantes Vectores de clonaje Cosmidos- vectores híbridos artificiales que contienen componentes de y de fagos, aumentando el tamaño del fragmento a clonar a 45 Kb (in vivo como plasmidios; in vitro como fagos) Luego de empacarse, fagos recombinantes son utilizados para infectar bacterias donde se reproducirán como plasmidios Vectores de clonaje YACs - hasta 1000 Kb; llevan centrómeros, origen de replicación y telómeros Vectores de clonaje BACs - basado en 7 Kb factor F de E. coli que actúa como factor F’ (100-300 Kb) Vectores de expresión Vectores de expresión – permite la expresión de secuencias clonadas fusionándolas con “transcription and translation start and stop sequences”. Permite la expresión del DNA clonado en su respectivo hospedero.