Secuenciación del ARN
Anuncio
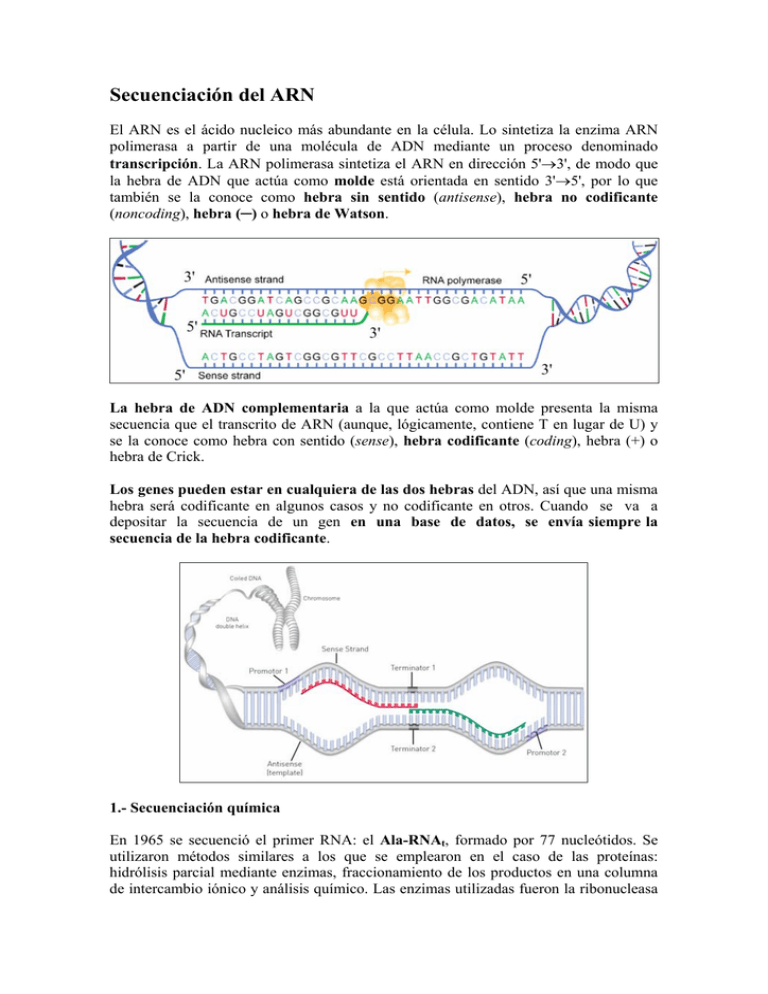
Secuenciación del ARN El ARN es el ácido nucleico más abundante en la célula. Lo sintetiza la enzima ARN polimerasa a partir de una molécula de ADN mediante un proceso denominado transcripción. La ARN polimerasa sintetiza el ARN en dirección 5'→3', de modo que la hebra de ADN que actúa como molde está orientada en sentido 3'→5', por lo que también se la conoce como hebra sin sentido (antisense), hebra no codificante (noncoding), hebra (─) o hebra de Watson. La hebra de ADN complementaria a la que actúa como molde presenta la misma secuencia que el transcrito de ARN (aunque, lógicamente, contiene T en lugar de U) y se la conoce como hebra con sentido (sense), hebra codificante (coding), hebra (+) o hebra de Crick. Los genes pueden estar en cualquiera de las dos hebras del ADN, así que una misma hebra será codificante en algunos casos y no codificante en otros. Cuando se va a depositar la secuencia de un gen en una base de datos, se envía siempre la secuencia de la hebra codificante. 1.- Secuenciación química En 1965 se secuenció el primer RNA: el Ala-RNAt, formado por 77 nucleótidos. Se utilizaron métodos similares a los que se emplearon en el caso de las proteínas: hidrólisis parcial mediante enzimas, fraccionamiento de los productos en una columna de intercambio iónico y análisis químico. Las enzimas utilizadas fueron la ribonucleasa pancreática, que rompe el RNA tras una base pirimidínica (C y U) y la ribonucleasa T1, que escinde la molécula de RNA después de una G o una I (inosina). Poco después, Fred Sanger desarrolló un método basado en la hidrólisis enzimática y la posterior separación y análisis de los oligonucleótidos generados (marcados con 32P) mediante cromatografía bidimensional en papel. De este modo, en 1968, consiguió secuenciar el RNAr de 5S, que tiene una longitud de 120 nucleótidos. Este tipo de secuenciación del ARN puede complicarse debido a la presencia de nucleótidos atípicos o modificados en el ARNt y en el ARNr. Hoy en día, la presencia de nucleótidos modificados se puede detectar con relativa facilidad gracias a la espectrometría de masas. 2.- Secuenciación del ARN a partir del ADNc (EST, RNA-Seq) Si el ARN carece de nucleótidos atípicos o modificados, su secuencia se puede determinar directamente a partir de la secuencia de ADN genómico (ADNg) que lo codifica. En eucariotas, sin embargo, es muy habitual que la secuencia de las moléculas de ARNm que van a ser traducidas no coincida exactamente con la secuencia del ADN genómico, ya que los transcritos primarios de ARN pueden experimentar diversas modificaciones mediante los procesos de maduración del ARN (RNA splicing) o de edición del ARN (RNA editing). Durante la maduración del ARN se eliminan los intrones del transcrito primario y se empalman los exones para generar un ARNm maduro. La edición del ARN es un proceso en el que se pueden añadir, eliminar o sustituir nucleótidos en la secuencia del ARN. En estos casos, la secuencia del ARNm se puede obtener directamente a partir del ADN complementario (ADNc), que es una molécula de ADN sintetizada por la enzima transcriptasa inversa a partir de una molécula de ARNm que actúa como molde. Generación de ADNc a partir de moléculas de ARNm En primer lugar, se extrae todo el ARN celular y se hace pasar a través de una columna de celulosa unida a cadenas de oligo(dT). Las colas de poli(A) de las moléculas de ARNm quedan retenidas en la columna, mientras que el resto del ARN la atraviesa. A continuación se utiliza un tampón de elución que rompe los puentes de hidrógeno entre la cola de poli(A) y las cadenas de oligo(dT) y permite extraer el ARNm de la columna. Este ARNm servirá de molde para que la transcriptasa inversa sintetice una hebra de ADNc utilizando como cebadores oligo(dT), que hibridan con la cola de poli(A). La transcriptasa inversa genera una molécula híbrida ADN/ARN. Esta molécula se trata brevemente con una ribonucleasa que digiere parcialmente la hebra de ARN (algunas transcriptasas inversas tienen actividad ribonucleasa H que, durante el proceso de síntesis de ADN hidrolizan de forma específica el molde de ARN). A continuación, se utiliza una ADN polimerasa para que sintetice la segunda hebra del ADN utilizando los fragmentos cortos de ARN no digeridos como cebadores. Por último, la ADN ligasa se encarga de unir los distintos fragmentos en una única hebra. El resultado de este proceso es la creación de una molécula de ADN de doble hebra con la misma secuencia que el ARNm molde y que se puede secuenciar utilizando cualquiera de las técnicas disponibles. Si se va a secuenciar el ADN mediante el método de Sanger, hay que clonar el ADNc en un vector apropiado para crear genotecas de ADNc que contienen el conjunto de genes transcritos en la muestra original. La secuenciación de estos clones de ADNc genera unas secuencias parciales de cada inserto cuya longitud oscila entre 100 y 800 nucleótidos. Estas secuencias se denominan EST (expressed sequence tags) porque corresponden a regiones del genoma que han sido expresadas. Se pueden obtener EST a partir del extremo 5' o a partir del extremo 3', ya que se pueden diseñar cebadores que hibriden con ambos extremos del vector de clonación. También se puede secuenciar el ADN mediante las técnicas de nueva generación (las plataformas Roche-454, Illumina/Solexa o ABI SOLID). Este método se conoce con el nombre de RNA-seq. En este caso, no es necesario crear una genoteca de ADNc. En vez de ello, se fragmenta el ADNc, se seleccionan los fragmentos con el tamaño deseado y se les añade unos adaptadores especiales que permiten su amplificación por PCR y su posterior secuenciación masiva en paralelo. Los detalles experimentales varían ligeramente, en función de la plataforma de secuenciación que se vaya a utilizar. A partir de cada fragmento de ADNc se obtiene una lectura (read) corta, de entre 30 y 400 nucleótidos. Estas lecturas se pueden comparar directamente con el genoma de referencia o, alternativamente, se pueden ensamblar de novo para crear un mapa transcripcional que nos indica qué genes se han expresado y los niveles de expresión de cada uno. 3.- Secuenciación directa del ARN (Plataforma Helicos) Este método utiliza directamente el ARN como molde, sin necesidad de convertirlo previamente en ADNc. Así se evitan los artefactos relacionados con la actividad de la transcriptasa inversa. Además, se secuencian las moléculas de forma individual (single-molecule technique), con lo que también se evita la etapa de amplificación, bien por clonación in vivo (como en los EST), bien por PCR (como en el caso de la RNASeq). Además, también permite obtener el patrón de expresión génica (el transcriptoma) de manera cuantitativa. Se trata de una secuenciación masiva en paralelo en la que se utiliza una polimerasa especialmente modificada para poder utilizar como sustratos 4 derivados de los nucleótidos naturales denominados "terminadores virtuales" (TV). Los TV están unidos a un fluoróforo y bloqueados en posición 3' por un grupo químico, de modo que en cada ciclo de reacción sólo se puede incorporar un nucleótido. Tanto la unión del fluoróforo como el bloqueo en 3' son reversibles. Terminador virtual fluorescente y bloqueado en su extremo 3' Terminador virtual no fluorescente y desbloqueado en su extremo 3' Las moléculas de ARN que se van a secuenciar tienen que estar poliadeniladas y su extremo 3' debe estar bloqueado. Las moléculas de ARNm ya tienen una cola de poli(A) en su extremo 3'. Al resto de las moléculas de ARN se les puede añadir in vitro la cola de poli(A), utilizando la enzima poli(A)-polimerasa. Tanto las moléculas de ARN poliadeniladas de forma natural (el ARNm) como de forma artificial (los demás ARN) se bloquean en 3' mediante la adición de un residuo de desoxiadenosina (dA) catalizada por la misma poli(A)-polimerasa. Las moléculas de ARN poliadeniladas se introducen en una celda de flujo cuya superficie está recubierta de fragmentos de oligo(dT). La hibridación entre las colas de poli(A) y los oligo(dT) hace que las moléculas de ARN queden fijadas al soporte, con una densidad de 100 millones de moléculas por cm2. La secuenciación se produce durante el proceso de síntesis de la hebra complementaria. Una vez unidas, se lleva a cabo una etapa de "rellenado" (fill) y "sellado" (lock) en la que se añade timidina natural y polimerasa para elongar los oligo(dT) hasta que todos los residuos de adenosina de la cola de poli(A) estén emparejados con una T. Así nos aseguramos de que la secuenciación empezará exactamente en la molécula de ARN y no en su cola de poli(A). Después del "rellenado" se procede al "sellado", que consiste en la adición de 3 TV fluorescentes (A, C y G). No hace falta añadir T porque tras la etapa de rellenado no queda ninguna A sin emparejar. Cada molécula de ARN incorporará el TV adecuado en cada caso. Se excitan los TV fluorescentes y se toma una imagen que permite determinar la posición exacta de cada molécula de oligo(dT) antes de que empiece la síntesis de la hebra complementaria. Después, mediante métodos químicos, se escinde el fluoróforo de cada TV y se desbloquean los extremos 3' para que se puedan incorporar nuevos nucleótidos en el orden que dicta cada molécula de ARN molde. La etapa de síntesis se lleva a cabo mediante ciclos de reacciones. En cada ciclo se añade un TV distinto junto con la polimerasa. Supongamos que se ha añadido C-TV. Este nucleótido se unirá únicamente a aquellas moléculas que presenten una G en el ARN molde. Se lavan los TV no unidos y se ilumina la celda de flujo para excitar los TV. Se toma una imagen que presentará una señal fluorescente en aquellas posiciones donde se haya incorporado el C-TV. Después se escinden químicamente los fluoróforos y se desbloquean los extremos 3' para iniciar un nuevo ciclo de síntesis. Normalmente se completan 120 ciclos de síntesis en los que los nucleótidos se van añadiendo siempre en el mismo orden (por ejemplo C-T-A-G). Examinando la colección de imágenes que se han tomado, en cada posición habrá una sucesión de puntos que indican qué nucleótidos se han incorporado y cuáles no, lo que permite determinar la secuencia de cada molécula de ARN que ha actuado como molde. La longitud media de las lecturas obtenidas es de 33 nucleótidos.