Descargar
Anuncio
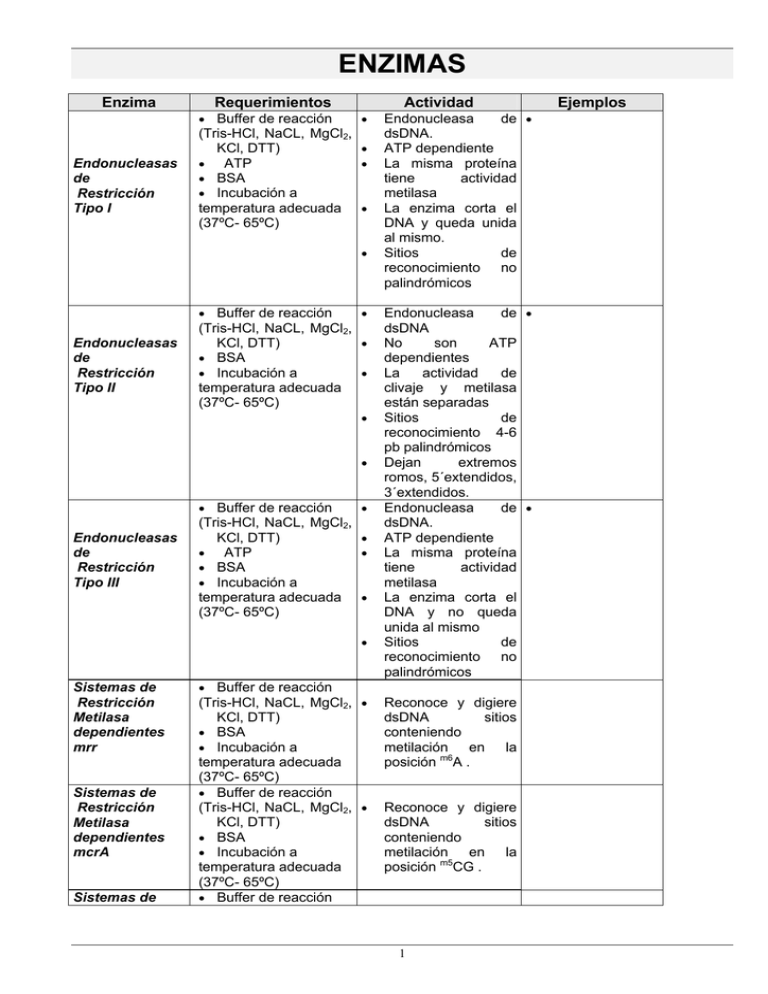
ENZIMAS Enzima Requerimientos Actividad • Buffer de reacción • (Tris-HCl, NaCL, MgCl2, KCl, DTT) • • ATP • • BSA • Incubación a temperatura adecuada • (37ºC- 65ºC) B Endonucleasas de Restricción Tipo I B • • Buffer de reacción • (Tris-HCl, NaCL, MgCl2, KCl, DTT) • • BSA • Incubación a • temperatura adecuada (37ºC- 65ºC) B Endonucleasas de Restricción Tipo II B • • • Buffer de reacción • (Tris-HCl, NaCL, MgCl2, KCl, DTT) • • ATP • • BSA • Incubación a temperatura adecuada • (37ºC- 65ºC) B Endonucleasas de Restricción Tipo III B • Sistemas de Restricción Metilasa dependientes mrr Sistemas de Restricción Metilasa dependientes mcrA Sistemas de Endonucleasa de • dsDNA. ATP dependiente La misma proteína tiene actividad metilasa La enzima corta el DNA y queda unida al mismo. Sitios de reconocimiento no palindrómicos Endonucleasa de • dsDNA No son ATP dependientes La actividad de clivaje y metilasa están separadas Sitios de reconocimiento 4-6 pb palindrómicos Dejan extremos romos, 5´extendidos, 3´extendidos. Endonucleasa de • dsDNA. ATP dependiente La misma proteína tiene actividad metilasa La enzima corta el DNA y no queda unida al mismo Sitios de reconocimiento no palindrómicos • Buffer de reacción (Tris-HCl, NaCL, MgCl2, • KCl, DTT) • BSA • Incubación a temperatura adecuada (37ºC- 65ºC) • Buffer de reacción (Tris-HCl, NaCL, MgCl2, • KCl, DTT) • BSA • Incubación a temperatura adecuada (37ºC- 65ºC) • Buffer de reacción B B B B Reconoce y digiere dsDNA sitios conteniendo metilación en la posición m6A . P P Reconoce y digiere dsDNA sitios conteniendo metilación en la posición m5CG . P 1 P Ejemplos Restricción Metilasa dependientes mcrB (Tris-HCl, NaCL, MgCl2, KCl, DTT) • BSA • Incubación a temperatura adecuada (37ºC- 65ºC) • Buffer de reacción (Tris-HCl, NaCL, MgCl2, KCl, DTT) • BSA • Incubación a temperatura adecuada (37ºC- 65ºC) • • Buffer de reacción • B B B Homing Endonucleasas dam Metilasa • • (Tris-HCl, NaCL, MgCl2, KCl, DTT, EDTA) • BSA • S-adenosilmetionina • Incubación a temperatura adecuada (37ºC- 65ºC) B • Buffer de reacción (Tris-HCl, NaCL, MgCl2, KCl, DTT, EDTA) • BSA • S-adenosilmetionina • Incubación a temperatura adecuada (37ºC- 65ºC) • Buffer de reacción B • (Glicina-KOH, MgCl2, β-mercaptoetanol) • Incubación a 37ºC B B • Exonucleasa III • Buffer de reacción (Tris-HCl, MgCl2,) B B P Endonucleasa de • dsDNA. Sitios de reconocimiento largos (12-40 pb), asimétricos. Secuencias codificantes dentro de intrones (llevan prefijo I-) o inteínas (llevan el prefijo PI-). Digestión y mapeo de genomas grandes. Introduce grupos • metilo en dsDNA en la posición N6 de la Adenina si encuentra la secuencia GATC. Inhibe el corte de endonucleasas que reconocen ese sitio. Bloquea y protege la acción de algunas enzimas. de restricción que reconocen GATC, Ej: MboI (GATC) es sensible, sin embargo Sau3AI (GATC) no es sensible a dam metilación. Bloquea y protege la acción de algunas enzimas. de restricción que reconocen CCAGG o CCTGG. P P Introduce grupos • metilo en dsDNA en la posición C5 de la Citocina interna si encuentra la secuencia CCAGG o CCTGG. P • B Exonucleasa I • B B Metilasa mEcoRI • (Tris-HCl, NaCL, MgCl2, KCl, DTT, EDTA) • BSA • S-adenosilmetionina • Incubación a temperatura adecuada • (37ºC- 65ºC) • Buffer de reacción dcm Metilasa P B B Reconoce y digiere dsDNA sitios conteniendo metilación en la posición Pum5C • P Cataliza la • transferencia de un grupo Metilo de una s-adenosilmetionina (sam) a una de las adeninas de la secuencia de reconocimiento de EcoRI (GAA´TTC) Cataliza la remoción • de nucleótidos de ssDNA en la dirección 3´a 5´. Cataliza la remoción • de nucleótidos de un hidroxilo 3´ terminal de un dsDNA. Dirección 3´a 5´. 2 Bloquea el sitio de reconocimiento de EcoRI Estudio de regiones de DNA por deleciones controladas. • Incubación a 37ºC • • • Exonucleasa Lambda • Buffer de reacción • • (Glicina-KOH, MgCl2,) • BSA • Incubación a 37ºC B B • • Exonucleasa T7 • • • Buffer de reacción (KAc,Tris-Ac, MgAc, DTT) • Incubación a 37ºC • Exonucleasa VII • Buffer de reacción • (Kfosfato, EDTA, βmercaptoetanol) • • Incubación a 37ºC • Buffer de reacción Nucleasa Mung (NaAc, NaCl, ZnSO4) • Incubación a 30ºC Bean B • B • • • Buffer de reacción (CaCl2, NaCl, MgCl2, • Tris-HCl, EDTA) • Incubación a 30ºC B Nucleasa 31 BAL- B B B Los sustratos adecuados son dsDNA con extremos romos o 5´extendidos. La enzima no es activa en dsDNA con extremos 3´extendidos. Cataliza la remoción • de mononucleótidos 5´de un dsDNA Dirección 5´a 3´. Los sustratos adecuados son dsDNA 5´ fosforilados, aunque degrada a menor tasa ssDNA y sustratos no fodforilados. No inicia digestión en nicks o gaps. Cataliza la remoción • de mononucleótidos 5´ de un dsDNA. Dirección 5´a 3´. Degrada tanto extremos 5´ fosforilados como no fosforilados. También degrada RNA y DNA de un híbrido RNA/DNA, pero no actúa sobre dsRNA y ssRNA. Inicia digestión en nicks o gaps. Degrada sdDNA tanto de un extremo 3´ como 5´. Es activa en presencia de EDTA. Degrada extremos simple hebra de moléculas de dsDNA, dsRNA o DNA/RNA, dejándolos romos Dirección 3´a 5´. Degrada extremos 3´y 5´ de un dsDNA Es activa en nicks, gaps y regiones simple hebra de un duplex DNA o RNA. 3 • • Reparación de extremos de DNA extendidos • Mapeo de estructuras secundarias de DNA Mapeo de sitio de restricción en DNA • • Nucleasa SI • Buffer de reacción (NaCl, ZnCl2, Tris-HCl) • Incubación a 37ºC B B • • Buffer de reacción B Dnasa I • (NaAc, MgCl2, Tris-HCl, KCl) • Incubación a 25ºC B • • Buffer de reacción Dnasa RQ1 • (HEPES, KCl, CaCl2, MgCl2) • Incubación a 37ºC • B B B B • Ribonucleasa H • Buffer de reacción (HEPES-KOH, KCl, DTT) • Incubación a 37ºC • Ribonucleasa T1 Ribonucleasa T2 • Buffer de reacción • (Tris-HCl, EDTA) • Incubación a 37ºC • • Buffer de reacción • (NaAc, EDTA) • Incubación a 37ºC • • Buffer de reacción Ribonucleasa A (sin mayores requerimientos) • Incubación a 37ºC • Degrada ssDNA y ssRNA en forma de endonucleasa generando productos con extremos 5´ fosforilados. dsDNA, dsRNA y DNA/RNA son resistentes a la enzima, salvo a concentraciones extremadamente altas de ésta. Degrada dsDNA y ssDNA a oligodeoxiribonucleó tidos. A bajas concentraciones de enzima y tiempos de reacción cortos, introduce nicks en dsDNA. Degrada sdDNA y dsDNA generando oligos con OH-3´. Se suministra libre de Rnasas Endorribonucleasa que hidroliza los enlaces fosfodiester de un RNA hibridado a un DNA, generando productos con OH 3´ y P-5´. No degrada SSRNA, dsDNA o dsRNA Endorribonucleasa que hidroliza ssRNA en la posición 3´ de residuos G. Endorribonucleasa no específica que hidroliza ssRNA, en preferencia en la posición 3´ de residuos A. De origen pancreática, cataliza la despolimerización de ssRNA, hidrolizando específicamente en la posición 3´de una pirimidina (U o C). No hidroliza DNA 4 • • • Reparación de extremos DNA Analizar estructura de hibridos DNA:RNA Remoción del loop generado durante la síntesis de segunda acdena de cDNA • • Nick translation DNAsa Footprinting • Generación de cDNA, utilizada luego de la extracción del RNA Generación de cDNA • • • • • • Generación cDNA Rnasa Protección Generación cDNA Rnasa Protección de de Degradación de RNA en extracción y purificación de ADN • Rnasin • Buffer de reacción (HEPES-KOH, KCl, • DTT) • Incubación a 37ºC • Fosfatasa Alcalina Bacteriana • Buffer de reacción Fosfatasa Alcalina Intestinal • Buffer de reacción (Tris-HCl) γ-32• ATP • Incubación a 65ºC P T4 Polinucleótido Quinasa • Bajar el background previniendo la religación (mayor actividad y mas estable) Remueve fosfatos 3´ y 5´ de DNA y RNA. • Remueve fosfatos 3´ • y 5´ de DNA y RNA Bajar el background previniendo la religación • Cataliza la • transferencia del γfosfato del extremo 5´de un DNA o RNA a un ADP, incorporando un • nuevo fosfato sacado de un ATP. Utilizada para marcar fragmentos de ácidos nucleicos con γ-32-ATP DNA polimerasa 5´- z 3´ z Exo 5´-3´ (dsDNA) Exo 3´- 5´(ssDNA, dsDNA) RNAsa H Marcado de extremos DNA para sonda o para secuenciación. Fosforilar linkers sintéticos que carecen del fosfato 5´ DNA polimerasa 5´3´ Exo 3´- 5´(ssDNA, dsDNA) Primers extension Generación de extremos romos Marcado de extremos recesivos Generación de sondas Generación de sondas por nicktranslation (dietanolamina, 4nitrofenil fosfato, MgCl2) • Incubación a 65ºC B • Buffer de reacción (Tris-HCl, KCl, MgCl2, β-mercaptoetanol) γ-32• ATP • Incubación a 37ºC B P Utilizado enla generación de cDNA • P B Inhibe RNAsas • eucariotas (A, B, C) y RNAsa placentaria No inhibe RNAsa H, Nucleasa SI, RNA polimerasas (T7, Sp6, T3), transcriptasas reversas (AMV, MMLV) Se suministra libre de DNAsas y RNAsas. B P • P z DNA pol I holoenzima (E.coli) z z z z z Klenow DNA Pol z z z z T4 DNA Pol z Mg+2 o Mn+2 pH óptimo 7.4 Agente reductor Como sustrato necesita primers de ssDNA o ssRNA (OH-3´libre) sobre hebra de DNA Mg+2 o Mn+2 pH óptimo 7.4 (buffer fosfato) Agente reductor Como sustrato necesita primers de ssDNA o ssRNA (OH-3´libre) sobre hebra de DNA Mg+2 pH óptimo 8 - 9 Agente reductor P P P P P P P P P P z z z z z z P z z z z z z DNA polimerasa 5´3´ Exo 3´- 5´(ssDNA, 5 z Nick Translation Marcado de moleculas de DNA con extremos 3´ extendidos z dsDNA) z Generación de cDNA con primers al azar z DNA polimerasa 5´3´ z Secuenciación z DNA polimerasa 5´3´ z Amplificación de un fragmento de DNA Para generar plasmidos adecuados para productos de PCR Como sustrato necesita primers de ssDNA (OH-3´libre) sobre hebra de DNA z z T7 DNA pol (sequenace) z z z z z Taq DNA pol z z Mg+2 o Mn+2 pH óptimo 7.6 - 7.8 Agente reductor Como sustrato necesita primers de ssDNA (OH-3´libre) sobre hebra de DNA Mg+2 o Mn+2 pH óptimo 8.3 a temp ambiente. Actividad óptima a 70-74°C Como sustrato necesita primers de ssDNA (OH-3´libre) sobre hebra de DNA Mg +2 pH óptimo 7.6 (MoMLV) pH óptimo 8.3 (AMV) Agente reductor Como sustrato necesita primers de ssDNA o ssRNA (OH-3´libre) sobre hebra de RNA Mg+2 rNTP Secuncia promtora Sutrato: dsDNA P P P P P P P z z P z RNA dependiente DNA pol ( transcriptasa reversa) MoMLV AMV DNA dependiente RNA pol (SP6, T3, T7) z z z z z z z z z P P DNA polimerasa 5´3´ RNAsa H z Generar cDNA RNA polimerasa 5´3´ z Transcripción in vitro Run off Sondas de Rna Utilizadas en sistemas de expresión z z z z Mg+2 o Mn+2 dNTP Sustrato: ssDNA con extremo OH-3´ libre. dsDNA con extremos blunt o 3´recesivos sirven como sustrato en presencia de Co+2 como cofactor z Polimerización de ssDNA a partir de un extremo OH-3´libre Mg+2 ATP (cofactor) Substarto: dsDNA con extremos compatibles o Blunt. Uno de los extremos debe ser OH- 3´ y el otro 5´-P z Cataliza la formación de un enlace fosfodiester entre 3´-OH y un 5´P en los extremos o nicks de DNA P P z Transferasa Terminal z P P z z DNA Ligasa T4 z P P P 6 Determinación de extremos de Rna. z Para generar plasmidos adecuados para productos de PCR z Generar extremos compatibles z Unir fragmentos de DNA doble cadena. z Ejemplo: Plasmido /gen z Reparar nicks z z z DNA Ligasa E. coli z z z RNA Ligasa T4 Mg+2 ATP (cofactor) Substarto: dsDNA con extremos compatibles. Uno de los extremos debe ser OH- 3´ y el otro 5´-P ATP ( cofactor) Sustrato: ssDNA o ssRNA P P z z Cataliza la formación de un enlace fosfodiester entre 3´-OH y un 5´P en los extremos o nicks de DNA Unir fragmentos de DNA doble cadena (menor eficiencia en extremos blunt) z Ejemplo: Plasmido /gen Reparar nicks Cataliza la unión z Circularizar covalente de un 5´-P Molecula RNa. de ssDNA o ssRNA z Ligación de con un 3´-OH de Adapters ssDNA o ssRNA z Determinar extremos de RNa. 7 z





