ARN mensajero
Anuncio

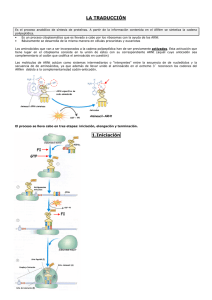
Facultad de Medicina TEMA Genética – 1er Curso 0-3 EXPRESIÓN GÉNICA: TRANSCRIPCIÓN Y TRADUCCIÓN 9 9 9 9 ARN mensajero. La transcripción en eucariotas. Ribosomas. La traducción. ARN mensajero Apartados Síntesis y procesamiento Corte y empalme “Capping” Poliadenilación Estabilidad ARN mensajero Síntesis y procesamiento En eucariotas, el ARNm producido por la ARN polimerasa II actúa como un molde para la síntesis de proteína durante la traducción El ARNm se sintetiza como un precursor, pre-ARNm, en el que se han transcrito tanto los exones como los intrones del gen. Antes de la síntesis de proteína, el pre-ARNm sufre un procesamiento para producir un ARN maduro: - “Splicing” Æ eliminación de intrones - “capping” Æ adición de un nt modificado en 5’ (caperuza) - poliadenilación Æ adición de una cola de hasta 250 adeninas En el núcleo existe una población de moléculas de diferente longitud y diferentes estados de procesamiento: ARN nuclear heterogéneo (hnRNA) ARN mensajero Corte y empalme ARN mensajero Corte y empalme • genes eucariotas (y ARNm) están caracterizados por intrones y exones • exón – parte codificante • intrón – parte no codificante • ARNm son procesados por el espliceosoma, un híbrido de proteína/ARN ensamblados • muchos exones corresponden a dominios de proteínas, una porción auto-plegable de una proteína con su propia actividad ARN mensajero “Capping” y Poliadenilación • El ARNm es procesado por distintos métodos antes de ser traducido a proteína: - caperuza 5’ Gppp, ayuda a la unión al ribosoma, protege de la degradación - cola 3’ poli(A) de 30-200 nucleótidos adenina ARN mensajero “Capping” y Poliadenilación ARN mensajero Estabilidad El ARNm tiene una vida media muy corta en las células. Esto es debido a que las células regulan los niveles de proteínas en el citoplasma principalmente cambiando el ritmo de la transcripción génica. Así pues, estos cambios se reflejan en la cantidad de ARNm asequible para la síntesis de proteínas En células bacterianas la vida media de un ARNm son unos pocos minutos En células eucariotas, suele ser como mucho 6 horas ARN mensajero (I) Objetivos Síntesis y procesamiento El ARN mensajero (ARNm) actúa como un molde para la síntesis de proteínas Se sintetiza en el núcleo por transcripción de los genes que codifican proteínas mediante la ARN polimerasa II. Primero se transcribe como precursor (preARNm), el cual contiene las secuencias intrónicas no codificantes que son posteriormente eliminadas por “splicing”. Al extremo 5’ se le añade una caperuza y al 3’ una cola poli-A. Corte y empalme Este proceso (“splicing”) consiste en la eliminación de los intrones en el ARNm precursor. Las secuencias GT y AG están en los finales de los intrones y son parte de secuencias señal de “splicing” más grandes. En el intrón existe otra señal llamada “branch site”. El proceso conlleva el corte del extremo 5’ del intrón y su unión al “branch site” formando un lazo con cola. El intrón se libera al cortar su extremo 5’ y los exones quedan juntos y se unen. El proceso está catalizado por snRNPs: U1 se une al sitio 5’ de splicing; U2 a la secuencia “branch”; U5 y U4/6 forman un complejo con U1 y U2 llamado espliceosoma que mantiene el ARNm en la posición correcta para el splicing y aporta la actividad enzimática requerida para cortar el intrón y unir los exones. ARN mensajero (y II) Objetivos “Capping” Los ARNm euxariotas son modificados en el extremo 5’ mediante la adición de un nucleótido modificado, 7-metilguanosina, en una unión inusual 5’ Æ 5’ trifosfato al primer nucleótido del ARNm. Esta modificación protege al ARNm de la degradación por 5’ exonucleasas. Poliadenilación Estabilidad La mayoría de los ARNm de eucariotas son modificados en el extremo 3’ por adición de una cola poli A (poliadenilación). El ARNm precursor es cortado alrededor de 20 bases aguas abajo de la secuencia señal de poliadenilación, 5’-AAUAAA-3’, y la polimerasa poli A añade un grupo de adeninas. También protege de las exonucleasas. El ARNm es relativamente inestable comparado con el ARNr y ARNt. Esto permite a las células regular los niveles de proteínas alterando el ritmo de la transcripción. Los ARNm procariotas tienen una vida media mucho más corta que los eucariotas. Términos Cadena codificante (no molde, sentido +) 3’ 5’ ADN G C C A T T T C G A A T C G G T A A A G C T T A 3’ 5’ Cadena molde Transcripción ARN G C C A U U U C G A A U Transcrito, misma secuencia que la cadena codificante Procariotas vs eucariotas Cadena ADN codificante Cadena ADN codificante (x=cualquier base) Transcripción en eucariotas Apartados Introducción ARN polimerasa II ARN polimerasa I ARN polimerasa III Transcripción en eucariotas Introducción • En eucariotas, los genes están normalmente “off” hasta que algo los cambia a “on” • Los promotores de eucariotas tienen una secuencia especial llamada caja TATA: 5’TATA(A/T)A(A/T)3’, a –25 pb, que dirige la unión de un grupo de proteínas denominadas factores de transcripción. • Una vez unidos los factores de transcripción, se une la ARN polimerasa y comienza la transcripción • La terminación ocurre al encontrar una señal AAUAAA Transcripción en eucariotas ARN polimerasa II Transcripción en eucariotas ARN polimerasa II Transcripción en eucariotas ARN polimerasa II Transcripción en eucariotas ARN polimerasa II Transcripción en eucariotas ARN polimerasa II Transcripción en eucariotas ARN polimerasa I Transcribe ARNr 18S, 28S y 5,8S. El promotor contiene dos elementos de secuencia esenciales para una eficiente transcripción: - el core, que solapa el sitio de inicio de la transcripción - una secuencia control aguas arriba en torno a –100 pb Estas secuencias unen ARN polimerasa I y sus factores de transcripción asociados. El core es esencial para que ocurra la transcripción y la secuencia control aguas arriba influye en el nivel de transcripción Una señal de terminación de 18 pb a 600 pb después del final del gen Transcripción en eucariotas ARN polimerasa III Transcribe un grupo de genes cortos que codifican ARNt y el ARNr 5S. Las secuencias promotoras están después del sitio de inicio de la transcripción dentro de la secuencia codificante del gen (regiones de control internas, ICR). Están dentro de las 100 bases desde el sitio de inicio de la transcripción. El gen ARNr 5S contiene una secuencia conocida como caja C, siendo el sitio de unión para los factores de transcripción y la ARN polimerasa III. Existe otra secuencia importante aguas arriba conocida como caja A. Los genes ARNt tienen dos elementos de secuencias conservadas importantes, la caja A y B, que constituyen el ICR, actuando conjuntamente como sitio de unión para los factores de transcripción y la ARN polimerasa III. Una secuencia poli A poco después del final del gen induce la terminación de la transcripción. Transcripción en eucariotas Objetivos Introducción ARN polimerasa II ARN polimerasa I La iniciación es compleja y la terminación no implica estructuras tallo-lazo. Tres ARN polimerasas (I, II, y III) transcriben diferentes grupos de genes. Esta enzima transcribe genes que codifican proteínas. Las secuencias del promotor suelen contener una caja TATA 25 pb aguas arriba del sitio de inicio de la transcripción, donde se une la enzima. Los factores de transcripción (TFII A, B, etc.) se unen al ADN alrededor de la caja TATA en un orden específico y forman una plataforma a la que la ARN polimerasa II se une. Genes sin caja TATA suelen tener otro elemento iniciador. Otros elementos promotores como la caja CAT actúan como sitios de unión para otros factores de transcripción que influyen en el nivel de transcripción. También influyen elementos distantes (potenciadores e inhibidores). No se conoce bien el mecanismo de terminación. Transcribe ARNr 18S, 28S y 5,8S. El promotor contiene dos elementos esenciales para la transcripción: el core, que solapa el sitio de inicio de la transcripción, y una secuencia control aguas arriba en torno a –100 pb. Una señal de terminación de 18 pb se sitúa a 600 pb después del final del gen. ARN polimerasa III Transcribe genes cortos que codifican ARNt y ARNr 5S. Las secuencias promotoras están dentro de la secuencia codificante (regiones de control internas, ICR). El gen ARNt tiene dos elementos de secuencias importantes, la caja A y B; la transcripción del gen ARNr 5S requiere la caja C. Una segunda secuencia llamada caja A es importante. Una secuencia poli A induce la terminación de la transcripción. Ribosomas Apartados Definición Función Composición en procariotas y eucariotas Anatomía de un ribosoma Ribosomas Definición: Complejos grandes de ribonucleoproteínas responsables de la síntesis proteica en todas las células. Ribosomas Función Subunidad grande: cataliza la reacción de la peptidil-transferasa en la síntesis proteica dirigida por el ARNm: formación del enlace peptídico entre los aminoácidos. Subunidad pequeña: descodifica el mensaje del ARNm, controla la fidelidad de la interacción codón-anticodón, se encarga de la translocación del ARNm y ARNt. Ribosomas Composición Procariotas Subunidad 50S ARNr 23S ARNr 5S 35 proteínas RIBOSOMA 70S subunidad 30S RIBOSOMA 80S subunidad 40S ARNr 16S 21 proteínas Eucariotas Subunidad 60S ARNr 28S ARNr 5S ARNr 5.8S 49 proteínas ARNr 18S 33 proteínas Ribosomas Anatomía Anatomía de un Ribosoma • Hay tres lugares en el ribosoma que unen ARNt amino-acil: A = acoge un nuevo ARNt P = aquí se forma el enlace peptídico entre aminoácidos E = sitio de salida para ARNt “vacíos” Ribosomas Objetivos Definición Función Complejos grandes de ribonucleoproteínas responsables de la síntesis proteica en todas las células. Es decir, se trata de estructuras macromoleculares compuestas de ARN ribosomal (ARNr) y proteínas. Están en grandes cantidades en el citoplasma donde traducen el ARNm a proteína Subunidad grande: cataliza la reacción de la peptidil-transferasa en la síntesis proteica dirigida por el ARNm: formación del enlace peptídico entre los aminoácidos. Subunidad pequeña: descodifica el mensaje del ARNm, controla la fidelidad de la interacción codón-anticodón, se encarga de la translocación del ARNm y ARNt. Composición en procariotas y eucariotas Anatomía de un ribosoma Las subunidades grandes y pequeñas son de tamaños característicos, descritos en valores de sedimentación (S). Procariotas: 70S, subunidades 50S y 30S; contienen ARNr 23S, 16S y 5S. Eucariotas: 80S, subunidades 60S y 40S; contienen ARNr 28S, 18S, 5.8S y 5S. Hay tres lugares en el ribosoma que unen ARNt amino-acil:A, acoge un nuevo ARNt; P, donde se forma el enlace peptídico entre aminoácidos; E, sitio de salida para ARNt “vacíos” (no llevan un aminoácido unido). ARN transferente Apartados Papel en la traducción Estructura Unión ARNt - aminoácido ARN transferente Papel en la traducción ARNt son moléculas pequeñas que actúan como adaptadores durante la síntesis proteica. Relacionan la secuencia de nucleótidos del ARNm a la secuencia de aminoácidos del polipéptido. En las células existen ARNt cada uno de los cuales une un determinado aminoácido. Cada ARNt reconoce un codón en el ARNm permitiendo situar su aminoácido en el lugar correcto de la cadena polipeptídica en crecimiento, según especifica la secuencia del ARNm. ARN transferente Estructura La Aminoacil ARNt sintetasa une un aminoácido determinado a un ARNt específico brazo TϕC brazo aceptor brazo opcional brazo DHU brazo anticodón ARN transferente Unión ARNt - aminoácido CH3 S metionina CH2 CH2 H2N ARNtMet CH COOH ? anticodón U A C A U G Codón de inicio ARNm ARN transferente Unión ARNt - aminoácido CH3 metionina CH3 S S CH2 CH2 CH2 H2N CH ATP C AMP + PPi CH2 H2N O CH OH C O O metionina aminoacil ARNt sintetasa ARNtMet ARNt cargado anticodón Objetivos U A C U A C ARN transferente Papel en la traducción ARNt son pequeñas moléculas que transportan los aminoácidos, en un orden especificado por la secuencia de un ARNm, para la síntesis de proteínas. Las células contienen un número de diferentes ARNt cada uno de los cuales une un aminoácido específico. Cada ARNt también se une a un codón específico en el ARNm permitiendo situar su aminoácido en la posición correcta. Estructura El emparejamiento de bases en el ARNt da lugar a una estructura en hoja de trébol compuesta de brazos llamados tallo-lazos. Éstos incluyen: brazo aceptor, punto de unión del aminoácido; brazo anticodón, reconoce los codones en la secuencia del ARNm; brazo DHU, contiene dihirouracilo; brazo opcional; y brazo TϕC, contiene pseudouracilo. La representación de la estructura terciaria sitúa los brazos aceptor y anticodón en los extremos opuestos de la molécula. Unión ARNt - aminoácido La interpretación de la información contenida en el ARNm se lleva a cabo mediante la unión a cada codón de un determinado ARNt que transporta su aminoácido específico. La unión específica ARNt – aminoácido la lleva a cabo la enzima aminoacil ARNt sintetasa. Traducción Apartados Reconocimiento de codones Traducción Iniciación Elongación Terminación Modificaciones post-traduccionales Traducción Reconocimiento de codones Hay una correspondencia entre la unión de un aminoácido a un ARNt y su unión a un determinado codón. Esto asegura que la secuencia de aminoácidos codificada por el ARNm es traducida fielmente. El reconocimiento de codones tiene lugar mediante el anticodón del ARNt por unión codón-anticodón por complementariedad de bases. 4 bases en ADN Æ 64 codones; 3 codones son señales de parada; quedan 61 codones Æ 20 aminoácidos Æ degeneración del código genético. Un ARNt reconoce más de un codón debido a la inestabilidad de la tercera base del codón (“wooble”) (primera del anticodón), que puede unir bases alternativas en esa posición (G≡C, G∼U, inosina≡C, inosina∼A, inosina∼ U). Consecuencia de “wooble” es que sirve para minimizar el efecto de las mutaciones. Traducción Traducción Tres estados: INICIACIÓN, ELONGACIÓN, TERMINACIÓN - Iniciación Æ unión ribosoma a ARNm - Elongación Æ adición repetitiva de aminoácidos - Terminación Æ liberación de la nueva cadena polipeptídica Proteínas accesorias asisten al ribosoma en cada uno de los tres estados La energía requerida proviene de la hidrólisis de GTP y ATP. - GTP Æ movimiento del ribosoma y unión de factores accesorios. - ATP Æ cargar los ARNt y eliminar la estructura secundaria del ARNm. Hasta el 90% del ATP producido en una bacteria se usa para la traducción. Traducción Iniciación Etapa 1: La subunidad ribosomal pequeña une IF1, IF3 e IF2-GTP Etapa 2: La subunidad pequeña se une al ARNm y se mueve al codón de inicio AUG. Traducción Iniciación Etapa 3: Un ARNt iniciador cargado con metionina se une al codón de inicio AUG. Se libera IF3 Etapa 4: Se une la subunidad ribosomal grande. Se libera IF1 e IF2 y se hidroliza GTP. •Hay tres posiciones en el ribosoma que unen ARNt aminoacil A = acepta un nuevo ARNt P = el enlace peptídico se forma aquí E = sitio de salida para ARNt “vacíos” Traducción Elongación complejo aa-tRNA/GTP/EF-Tu Formación del enlace peptídico complejo EF-Tu/GDP met ala Traducción gly Elongación CH3 S CH2 N-terminus H2N CH Peptidiltransferasa CH3 CH2 C N CH C O H2N CH O O Formación del enlace peptídico H C O O P site met A site ala Traducción gly Elongación CH3 S Formación del enlace peptídico CH2 N-terminus H2N CH H CH3 CH2 C N O P site CH C O N CH C O O A site Traducción Terminación E. coli: factores de liberación, RF1 (UAA, UAG) y RF2 (UAA, UGA) Eucariotas: único factor de liberación, eRF Traducción Modificaciones post-traduccionales Después de la traducción, los polipéptidos nuevos sintetizados pueden sufrir una serie de modificaciones antes de ser proteínas funcionales. Se trata fundamentalmente de la unión covalente de grupos químicos y cortes en la cadena polipeptídica. ADICION... pequeños grupos. - metilación - Fosforilación Æ regula la actividad enzimática - acetilación - hidroxilación grandes grupos - glicosilación (lípidos y oligosacáridos) CORTE... (es muy común) - recorte en el final de las proteínas - eliminación de secuencias señal internas o N-terminales - rotura de poliproteínas en péptidos más pequeños ESQUEMA-RESUMEN de la transcripción y traducción en eucariotas Traducción y translocación están acopladas en eucariotas Traducción (I) Objetivos Reconocimiento de codones Es similar en procariotas y eucariotas y se da en tres pasos: INICIACIÓN, ELONGACIÓN Y TERMINACIÓN. Cada estado involucra un grupo de proteínas accesorias. La energía proviene por hidrólisis de ATP y GTP. Traducción Iniciación La complementariedad de bases entre un codón en el ARNm y el anticodón del ARNt asegura que los aminoácidos se sitúan en el orden correcto durante la síntesis proteica. El código genético es degenerado; la mayoría de aminoácidos son codificados por más de un codón. Cada ARNt puede reconocer más de un codón especificando su aminoácido porque la base 5’ en el anticodón puede unir bases alternativas 3’ del codón. Esta posición se conoce como “inestable”. La traducción comienza con la unión de subunidades ribosómicas pequeñas al ARNm a la secuencia Shine-Dalgarno en procariotas y a la caperuza 5’ en eucariotas. La subunidad migra aguas abajo hacia el codón de inicio AUG y el ARNt iniciador metionina (ARNtimet) se une para formar el complejo de iniciación. IF1, 2 y 3 son factores de iniciación bacterianos. IF1 e IF3 evitan la unión de la subunidad grande ribosomal antes de que se complete la iniciación. IF3 lleva el ARNtimet al complejo de iniciación. En eucariotas, participan al menos 9 factores de iniciación. eIF1 y eIF2 tienen papeles similares a IF1 e IF2. Varios factores deshacen la estructura secundaria del ARNm. Traducción (y II) Objetivos Elongación Terminación Después de la iniciación la subunidad grande ribosomal se une al complejo de iniciación formando los sitios A y P. El sitio P es ocupado por ARNtmet. Un segundo ARNt cargado entra al sitio A y la peptidiltransferasa forma un enlace peptídico entre los dos aminoácidos. La ARNt deaciclasa rompe la unión entre metionina y su ARNt dejando un dipéptido unido al segundo ARNt. En procariotas, un factor de elongación, EF-Tu, está asociado con la entrada del ARNt al sitio A. GTP se hidroliza y se libera EF-Tu asociado a GDP. EF-Ts regenera EF-Tu. En eucariotas, eEF1 tiene un papel similar a EF-Tu. Después de la formación del enlace peptídico el ribosoma se mueve al siguiente codón, el dipéptido unido al segundo ARNt se mueve al sitio P expulsando el ARNt iniciador. Un tercer ARNt cargado entra en el sitio A y se repite el ciclo de elongación. La traducción termina cuando un codón de terminación entra al sitio A. Factores de liberación entran al sitio A y causan la liberación del polipéptido. En E. coli, RF 1, 2 y 3 producen la terminación. En eucariotas, una única proteína esta involucrada, eRF. Después de la terminación, el ribosoma se disocia y el ARNm se libera. Modificaciones post-traduccionales Después de la traducción, los polipéptidos pueden ser modificados por la adición de grupos químicos a las cadenas de aminoácidos y a los extremos N y C terminales o por proteólisis. Estas modificaciones pueden ser necesarias para la completa funcionalidad de las proteínas.