Elaboración de mezcla para PCR
Anuncio
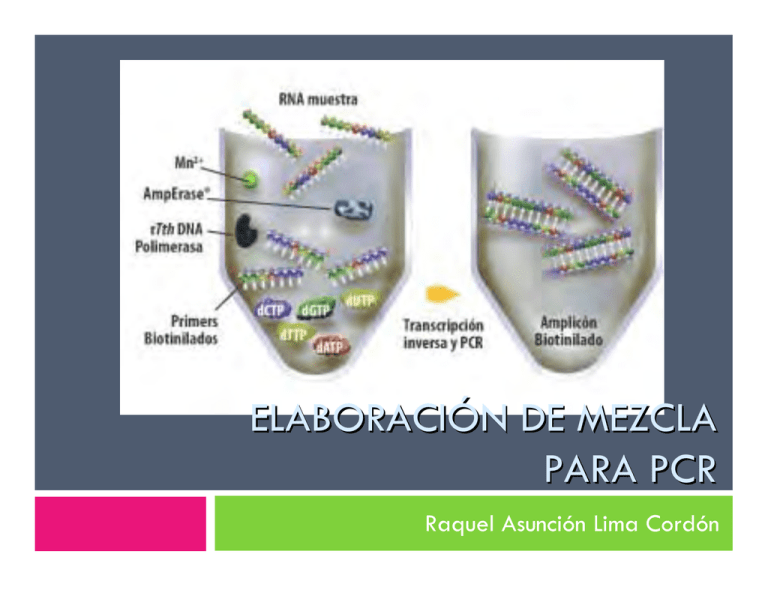
ELABORACIÓN DE MEZCLA PARA PCR Raquel Asunción Lima Cordón PCR Polymerase Chain reaction (reacción en cadena de la polimerasa) Sintetizar muchas veces un fragmento de ADN PCR: simulación de lo que sucede en la célula cuando se sintetiza el ADN PCR Ingredientes necesarios: Polimerasa ADN Oligonucleotidos dNTPs (dinucleotidos) PCR: ingredientes Oligonucleótidos: Uno debe tener la misma secuencia del ADN Segundo debe ser complementario a esa secuencia. Forward y reverse De no ser así, no podría amplificarse el fragmento deseado. PCR: aplicaciones Genética de poblaciones Evolución molecular Genómica Medicina forense PCR: ¿Cómo funciona? Después de haber preparado la mezcla maestra Se coloca en un termociclador Calienta o enfría los tubos a tres temperaturas distintas (ciclos de reacción) 95° (desnaturalización) 40° - 60° (alineamiento) 72° (extensión) PCR: desnaturalización Dobles cadenas de ADN se abren. PCR: alineamiento Se forman y se rompen constantemente los puentes de H entre los oligonucleótidos y el ADN Uniones más estables durarán mas tiempo Polimerasa se une al fragmento de ADN de doble cadena : 5´→ 3´ PCR: extensión Polimerasa alcanza su máxima actividad Continúa la sintesis de los fragmentos de ADN TIPOS DE TECNICAS RAPDs, AFLPs, RFLPs Tener claro el tipo de información que necesitamos Las técnicas se pueden dividir en dos categorías PCRs para la amplificación de un solo sitio conocido del genoma PCRs en los que no es necesario conocer la región que se está amplificando. • PCRs para la amplificación de un solo sitio conocido del genoma Requieren conocer la secuencia que se trabaja Con este tipo es posible hacer filogenias Los datos pueden obtenerse de: Geles que detectan cambios hasta de una sola base SSCP Utilizando enzimas de restricción • PCRs en los que no es necesario conocer la región que se está amplificando. Se desconoce el tamaño del fragmento (s) Se utilizan para determinar polimorfismo genómico Utiliza un solo oligonucleotido con 2 características Pequeño a mediano (6 -18 pb) Secuencia esté presente muchas veces en el ADN ¿Qué se necesita para hacer un PCR? Polimerasa comercial (acompañada de un buffer o amortiguador) MgCl2 (cloruro de magnesio) Oligonucleótidos Agua (debe tener pocas sales, bidestilada) Tubos (0.2 – 0.5 ml) y puntas (libres de ARNasas y ADNasas) Termociclador HACIENDO PCR ANTES DE EMPEZAR Preparar todos los reactivos en alícuotas congeladas (20° C) CONDICIONES DEL PCR Empezar con las mismas condiciones que se hayan reportado para el oligonucleótido que se está utilizando. Realizar pruebas con pocas muestras antes de utilizar todas las muestras de estudio. Concentración inicial Concentración final en la reacción Cantidad para un tubo Cantidad para 10 tubos dNTPs 10 mM 200μM varía de 1 μl acuerdo al magnesio 10 μl Magnesio 25mM 1.5mM puede 3 μl 30 μl 5 μl 50 μl 5 μl 50 μl probarse de 1 a 4 mM 10 μM 1μM puede probarse de 0.1 a 1 μM Oligo forward Oligo reverse 10 μM 1μM puede probarse Enzima 5U/μl 1U 0.2 μl 2 μl Buffer 10X 1X 5 μl 50 μl Agua - - 29.8 μl 298 μl ADN 0.1mg/ml 0.1 mg genómico 11 μl - de 0.1 a 1 μM TEMPERATURAS Y CICLOS Buscar artículos que hayan trabajado con el organismo de interés. Si no hay o bien no funciona se puede probar lo siguiente Desnaturalización inicial: 95°C por 5 – 10 mins 30 ciclos con desnaturalización a 95 °C (30 s), alineamiento a 50 °C (30 s) y extensión a 72 °C por tiempo variable. Extension final 72 °C 10 mins 4 °C LA TEMPERATURA DE ALINEAMIENTO Puede modificarse si el PCR aun no funciona. Se puede calcular la Tm (melting temperature) de cada oligo Varia de la cantidad de dobles o triples enlaces de H http://insilico.ehu.es/tm.php ¿Qué hacer cuando todo empieza a fallar? ¿Cuál es la calidad del ADN que tenemos? ADN contaminado con proteínas que inhiben la reacción de PCR ADN esté degradado: correr una pequeña muestra de ADN en un gel. En la extracción ocurre lo siguiente Rompe tejido: detergente (SDS o CTAB), si quedan restos de SDS la reacción se inhibe. Puede neutralizarse utilizando 0.5% de Tween 20 o 40 Después es necesario deshacerse de todos los componenetes celulares: se usan sales que precipitan proteínas pero no el ADN. Hacer curvas de cloruro de magnesio Revisar la concentración y el manejo de los dNTPs. Revisar el buffer Revisar los oligonucleótidos Algunos ya incluyen el MgCl2 e inhiben la reacción Podrían estar degradados, defectuosos, mal diseñados. Contaminación Trabajar con control negativo GELES DE AGAROSA Métodos para visualizar el PCR Principios básicos de la electroforesis Geles de agarosa o acrilamida (permiten separar fragmentos de acuerdo al tamaño de cada uno) Especie de red con agujeros de tamaños diferentes El ADN es “corrido” por corriente eléctrica hacia el polo positivo (ADN negativo) Fragmentos de un mismo tamaño se agruparan todos juntos formando bandas en el gel ¿Agarosa o acrilamida? Acrilamida Redes con tamaños de poros uniformes Pequeños Útil si los tamaños de los fragmentos son pequeños Separación fina Una sola base de diferencia Son muy laboriosas La tinción con plata Agarosa No forma redes tan uniformes Permite separar moléculas de ADN en un intervalo muy grande Sencillo METODOS PARA GELES DE AGAROSA EQUIPO Cámara de electroforesis Fuente de poder Transiluminador de luz UV Equipo de fotografía Para empezar: Preparar buffer de corrida Con pH requerido TBE (Tris Boratos EDTA): estable y reusable. TAE (Tris Acetatos EDTA): menos estable pero permite obtener mejor separación de bandas La concentración de agarosa dependerá de los tamaños de fragmentos Si se desconoce el tamaño, se puede iniciar con agarosa al 1% Rango efectivo de separacion (Kb) Agarosa (%) 30 - 1 0.5 12 - 0.8 0.7 10 – 0.5 1.0 7 – 0.4 1.2 3 – 0.2 1.5 La agarosa se disuelve en el mismo buffer que se utiliza para la corrida Se calienta hasta ebullición (evitar derramar) Dejar enfriar aprox. 60°C Añadir bromuro de etidio Se intercala en las bases del ADN Brilla con luz UV Es mutágeno y altamente tóxico Verter en el contenedor de geles Colocar peines para formar pozos Dejar enfríar y solidificar Agregar el buffer necesario hasta cubrir el gel CARGANDO EL GEL Colorante de corrida: llevan sustancia espesa (glicerol o sacarosa) que permite que la muestra caiga hacia el fondo del pozo Colorantes: nos dan una idea de cómo van migrando los fragmentos Cuando el gel esté listo, se conectan los cables Cable rojo: polo positivo. Cable negro: polo negativo. El ADN migrará hacia el polo + Se recomienda utilizar 5 volts por cada cm que exista entre los dos electrodos (fragmentos grandes) Fragmentos pequeños se recomienda 90-100 V. Una cámara de 30 cm se correrá a 150 V GRACIAS POR SU ATENCIÓN






