Organización general de las células: Citosol y sistema de
Anuncio

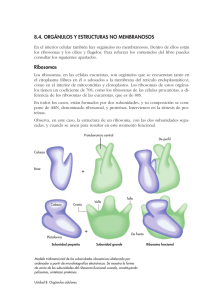
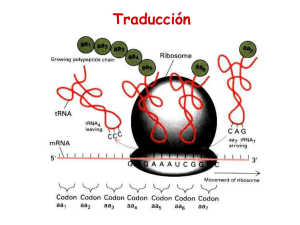
1 Organización general de las células: Citosol y sistema de endomembranas Resumen Tema 11. Ribosomas. Estructura de los ribosomas libres y asociados a membranas. Ribosomas en células eucariotas y procariotas. Polirribosomas. Introducción a la síntesis de proteínas desde la perspectiva del ribosoma. Destino de las proteínas sintetizadas en los ribosomas 1. Ribosomas. Estructura de los ribosomas libres y asociados a membranas • • Sustancia cromidial, material basófilo (Garnier, 1900). Basofilia: difusa, grumos y localizada. Los ribosomas son partículas compactas, adheridas o no a la cara externa de la membranas del RE, que aseguran la síntesis de proteínas uniéndose a los aminoácidos en un orden predeterminado. (Claude y Palade, 1953). • • • • Son muy numerosos y existen en todas las células, excepto en espermatozoides maduros y en los eritrocitos son muy escasos. Aparecen: - Adheridos a la cara externa de la membrana del RE mediante la subunidad mayor y mediado por dos glucoproteínas, riboforina I y II. - Adheridos a la envoltura nuclear externa. - Libres en el citosol, pudiendo encontrarse aislados o formando polisomas o polirribosomas. El hecho de que estén libres o fijos, hace que las proteínas sintetizadas por ellos tengan distintos destinos finales. Ciertas proteínas ribosomales son necesarias para la unión de la subunidad pequeña a la mayor (proteínas estructurales) y otras son necesarias para la síntesis proteica (proteínas funcionales). En las células eucariotas, las subunidades ribosomales son sintetizadas en el núcleo. 1.1. Recambio de los ribosomas • La destrucción de los ribosomas parece ocurrir al azar, sin depender de la antigüedad del ribosoma. 2. Ribosomas en células procariotas y eucariotas 2.1. Procariotas 2.2. Eucariotas • • Ribosomas de plastos: 70S. Ribosomas mitocondriales: 55S - 60S. La subunidad mayor presentan un RNA 4S, equivalente al 5S. 3. Polirribosomas • • • Los ribosomas se asocian en grupos mediante un filamento de mRNA de unos 2 nm de espesor para realizar cualquier tipo de síntesis proteica. El número de ribosomas de un polisoma y la longitud del mRNA que los une depende del peso molecular de la proteína a sintetizar. Las bacterias poseen una velocidad de síntesis proteica mayor. 4. Introducción a la síntesis de proteínas desde la perspectiva del ribosoma • • • • La traducción del mRNA en proteínas depende de moléculas adaptadoras que pueden reconocer y unirse tanto al codón como al aminoácido, los tRNA. El código genético es redundante (diferentes codones determinan un mismo aminoácido): - Muchos de los aminoácidos tienen más de un tRNA - Algunas moléculas de tRNA pueden aparearse con más de un codón (balanceo). El reconocimiento y la unión del aminoácido correcto dependen de las aminoacil-tRNA sintetasas. En el proceso de traducción las sintetasas son tan importantes como los tRNA, es la acción combinada de sintetasas y tRNA la que permite asociar cada codón de la molécula de mRNA con su aminoácido correspondiente. Configuración de los Seres Vivos Resumen Tema 11: Los ribosomas 2 Organización general de las células: Citosol y sistema de endomembranas 4.1. Decodificación del mensaje del ARN • La traducción cuidadosa y rápida de un mRNA a proteína requiere una gran maquinaria molecular, el ribosoma: • Subunidad pequeña enlaza los tRNA a los codones del mRNA. • Subunidad grande cataliza la formación de los enlaces peptídicos que unen los aminoácidos entre si. ¿Pero cómo puede el ribosoma orquestar los movimientos necesarios para la traducción? • Un ribosoma presenta cuatro lugares de unión para moléculas de RNA: uno es para el mRNA y tres (llamados sitios A, P y E) son para moléculas de tRNA. - Inicio de la síntesis proteínas • La traducción del mRNA se inicia con AUG y necesita un metionina-tRNA (bacterias usan formilmetionina). • Los mRNA procariotas son policistrónicos • Los mRNA eucariota son monocistrónicos. - Proceso de elongación de la síntesis de proteínas - Terminación de la síntesis de proteínas • Chaperonas: identifican las proteínas al ser sintetizadas y las van plegando correctamente sintetizando. 5. Destino de las proteínas sintetizadas en los ribosomas - Ribosomas libres: • Proteínas solubles del citoplasma fundamental. • Proteínas periféricas de la membrana plasmática (enzimas, actina, espectrina, etc.). • Proteínas mitocondriales. • Proteínas para los plastos. • Proteínas del interior de los peroxisomas (catalasa, oxidas de aminoácidos, etc.). • Proteínas nucleares (histonas, láminas, etc.) - Ribosomas del retículo endoplasmático rugoso: diferentes destinos (sistema de endomembranas). • Las proteínas recién sintetizadas pueden poseer uno o varios péptidos señal (secuenciales), que sirven para clasificarlas. Configuración de los Seres Vivos Resumen Tema 11: Los ribosomas 1 Organización general de las células: Citosol y sistema de endomembranas Tema 11. Ribosomas. Estructura de los ribosomas libres y asociados a membranas. Ribosomas en células eucariotas y procariotas. Polirribosomas. Introducción a la síntesis de proteínas desde la perspectiva del ribosoma. Destino de las proteínas sintetizadas en los ribosomas 1. Ribosomas. Estructura de los ribosomas libres y asociados a membranas Con microscopía óptica se observa en el citoplasma de muchas células un material basófilo, denominado por Garnier (1900) sustancia cromidial. Garnier notó que la sustancia cromidial variaba de unas células a otras y, dentro de una misma célula, se incrementaba al aumentar el trabajo que debía realizar esa célula; p. ej., en las células acinares pancreáticas la basofilia era más aparente al aumentar la actividad secretora. Para designar este material se suele utilizar el término basofilia, que significa apetencia por colorantes básicos, como hematoxilina. La basofilia viene causada por los ribosomas que, al estar en número considerable, hacen que el colorante básico absorbido sea perceptible al microscopio óptico; sin especificar si los ribosomas están libres o adheridos a membranas, formando retículo endoplasmático rugoso (RER). Así, puede decirse que las células del páncreas son muy basófilas (contienen mucho RER) y que también lo es un eritroblasto (contiene muchos ribosomas libres). En general, la basofilia se manifiesta como: a) difusa, extendiéndose por todo el citoplasma, como en células embrionarias, células plasmáticas o eritroblastos; b) en grumos, formando grumos distribuidos por el citoplasma, como ocurre en hepatocitos y neuronas, y c) localizada, ocupando ciertas áreas del citoplasma, como p. ej., la base de las células acinares del páncreas. Los ribosomas son partículas compactas, adheridas o no a la cara externa de las membranas del RE, que aseguran la síntesis de proteínas uniéndose a los aminoácidos en un orden predeterminado. Su hallazgo fue posible gracias al microscopio electrónico (Claude y Palade, 1953). Se aislaron por centrifugación diferencial (a 100.000 g en gradiente de sacarosa 0,25M y Cl2Mg), determinándose su morfología y composición en procariotas y eucariotas. Constan de dos subunidades (mayor y menor): • La subunidad menor consta de: • Cabeza: abarca un tercio de la subunidad. • Base: Abarca dos tercios de la subunidad. • Plataforma: separada de la cabeza por una cavidad o hendidura. La cabeza y la base se encuentran separadas por una pequeña constricción. • La subunidad mayor consta de: • Protuberancia central: situada entre el tallo y la cresta. • Tallo: proyección alargada. • Valle: situado entre protuberancia central y cresta. • Cresta: proyección corta. Configuración de los Seres Vivos Resumen Tema 11: Los ribosomas 2 Organización general de las células: Citosol y sistema de endomembranas Ensambladas las dos subunidades la protuberancia central queda alineada con la cabeza. Existen diferencias entre los ribosomas de células procariotas y eucariotas. Una célula típica contiene en su citoplasma millones de ribosomas. Existen en todas las células, excepto en espermatozoides maduros y en los eritrocitos son muy escasos. Pueden situarse: • Adheridos a la cara externa de la membrana del RE mediante la subunidad mayor y mediado por dos glucoproteínas, riboforina I y II. • Adheridos a la envoltura nuclear externa. • Libres en el citosol, pudiendo encontrarse aislados o formando polisomas o polirribosomas. El hecho de que estén libres o fijos, hace que las proteínas sintetizadas por ellos tengan distintos destinos finales. Las proteínas sintetizadas por ribosomas libres quedan en el citosol; mientras que las sintetizadas por ribosomas adheridos a RE se transfieren a su lumen para ser destinadas posteriormente a otros orgánulos intracelulares o ser expulsadas fuera de la célula, pero nunca quedan libres en el citosol. En la síntesis proteica se requiere la unión de una subunidad mayor y otra menor, pero no es necesario que sean siempre las mismas, como se demuestra mediante isótopos radiactivos, que marcan una sola subunidad. Al terminar de fabricar una cadena proteica, ambas subunidades se separan y cuando se vuelven a unir las subunidades mayores con las menores para iniciar una nueva cadena, estas uniones ocurren al azar entre el "pool" (o reservorio) de subunidades; por ello es altamente improbable que vuelvan a coincidir las parejas que formaron parte del ribosoma anterior. Ciertas proteínas ribosomales son necesarias para la unión de la subunidad pequeña a la mayor (proteínas estructurales) y otras son necesarias para la síntesis proteica (proteínas funcionales). Se ha conseguido sintetizar proteínas uniendo los dos tipos de subunidades ribosómicas, cada tipo procedente de un individuo diferente. También se ha logrado esta síntesis uniendo dos tipos de subunidades procedentes de especies afines, como la subunidad de 50S de Bacillus subtilis con la de 30S de Escherichia coli, aunque en este caso el rendimiento era mucho menor. Otro experimento efectuado ha sido unir los ribosomas de una especie con el citosol de otras especies, observándose que sólo se realizaba la síntesis proteica cuando ambas especies eran muy afines. En las células eucariotas, las subunidades ribosomales son sintetizadas en el núcleo, por asociación del rRNA recién transcrito con las proteínas ribosomales, las cuales han sido transportadas al núcleo tras ser sintetizadas en el citoplasma. Después, las subunidades ribosomales individuales son reexportadas al citoplasma para que participen en la síntesis de proteínas. 1.1. Recambio de los ribosomas Configuración de los Seres Vivos Resumen Tema 11: Los ribosomas 3 Organización general de las células: Citosol y sistema de endomembranas Los ribosomas deben tener una duración limitada; ya que las células con gran cantidad de ribosomas presentan nucléolo durante toda su vida (como las células acinares del páncreas). Si los ribosomas no se gastaran, no habría necesidad de seguirlos fabricando. Además, estas células van perdiendo la basofilia cuando llevan mucho tiempo sintetizando proteínas. Estos hechos parecen indicar la limitación temporal de la existencia de los ribosomas. De la que, no obstante, no se conoce su duración. La destrucción de los ribosomas parece ocurrir al azar, y no depende, por tanto, de la antigüedad del ribosoma. 2. Ribosomas en células procariotas y eucariotas 2.1. Procariotas Los ribosomas de procariotas, concretamente de Escherichia coli, han sido bien estudiados. Los ribosomas completos son de 70S y tienen un peso molecular de unos 2.500 kDa. En la composición de los ribosomas entran a formar parte rRNA (65%) y proteínas ácidas y básicas (35%). El rRNA forma un doble helicoide en un 60% de su longitud, y quedaría por fuera rodeando la proteína, de ahí la basofilia. La subunidad mayor (50S) presenta tres salientes a modo de picos. Contiene una molécula de RNA de 23S y otra de 5S. La subunidad menor (30S) muestra dos lóbulos y contiene RNA de 16S. Hay 21 proteínas en la subunidad menor y 34 en la mayor, y sólo una de ellas es común a ambas subunidades. Casi todas las proteínas son ricas en aminoácidos básicos y su peso molecular está entre 10 y 30 kDa. Hay agentes inhibidores de la síntesis proteica ribosomal en procariotas que actúan igualmente en eucariotas; es el caso de la puromicina y la actinomicina D. Otros agentes, como el cloranfenicol, la eritromicina, la estreptomicina y la rifamicina, son inhibidores específicos de los ribosomas de procariotas. 2.2. Eucariotas Los ribosomas de eucariotas tienen aproximadamente 80S y un peso molecular de unos 4.200 kDa, aunque existen pequeñas diferencias entre especies. En eucariotas el rRNA, que es el 70% de todo el RNA de la célula, constituye el 40% del peso del ribosoma. La composición en bases de este rRNA es diferente a la del rRNA de procariotas. La subunidad mayor (60S) tiene un RNA de 28S, otro de 5,8S y otro de 5S. La subunidad menor tiene un RNA de 18S, aunque hay pequeñas variaciones dependiendo del organismo en particular. Las proteínas ribosomales difieren de las de procariotas. Hay 33 proteínas en la subunidad menor y 49 en la subunidad mayor, y la mayoría de las proteínas de cada subunidad (o posiblemente todas) son diferentes de las presentes en la otra subunidad. La síntesis proteica de estos ribosomas es inhibida por agentes que también inhiben la síntesis proteica en procariotas (como puromicina y actinomicina D) y por inhibidores específicos, como la alfa-amanitina, la anisomicina y la cicloheximida. En eucariotas también hay ribosomas en las mitocondrias y en los plastos que se asemejan a los de procariotas y, como ellos, son inhibidos por cloranfenicol. Los ribosomas de los plastos son muy semejantes a los ribosomas procarióticos y, como éstos, tienen 70S. Los ribosomas Configuración de los Seres Vivos Resumen Tema 11: Los ribosomas 4 Organización general de las células: Citosol y sistema de endomembranas mitocondriales difieren de ellos en ciertos aspectos. En células animales, los ribosomas mitocondriales son menores que los ribosomas de bacterias y varían algo de unas especies a otras (entre 55S y 60S). Por el contrario, en levaduras y ciliados, los ribosomas mitocondriales suelen ser mayores que los de procariotas. En la subunidad mayor presentan un RNA de 4S, equivalente al de 5S presente en los ribosomas de procariotas y en los ribosomas citoplasmáticos de eucariotas. 3. Polirribosomas Para la síntesis de proteínas, los ribosomas se asocian en grupos mediante un filamento de mRNA de unos 2 nm de espesor, formando polirribosomas o polisomas, que suelen adoptar una configuración en espiral, con la subunidad menor dispuesta hacia el interior de la espiral. El filamento de mRNA pasa por el surco entre las dos subunidades (aunque más bien queda en la subunidad menor). Se han propuesto dos modelos de la posición del mRNA respecto a las subunidades de los ribosomas: entre los dos lóbulos de la subunidad menor, o atravesando ambos lóbulos de la subunidad menor; esto es, una disposición perpendicular a la de la hipótesis anterior. Los ribosomas forman polisomas para realizar cualquier tipo de síntesis proteica: tanto la efectuada por los ribosomas libres, como la realizada por los asociados a membranas (RER). En el RER la subunidad mayor es la que se adosa a la membrana y el surco entre ambas subunidades es paralelo a la superficie de la membrana. El número de ribosomas que forman un polisoma y la longitud del mRNA que los une varían según el peso molecular de la proteína que se va a sintetizar. Así, para sintetizar un polipéptido de 150 aminoácidos se necesita un mRNA con 450 nucleótidos (3 nucleótidos por aminoácido). Como cada triplete de nucleótidos mide 1 nm de longitud, para sintetizar los 150 aminoácidos se necesitarán 150 nm de mRNA. Sobre este mRNA se disponen los ribosomas posibles dejando una distancia entre cada dos ribosomas (entre sus centros) de unos 34 nm. De este modo, un polisoma que sintetice un polipéptido de 150 aminoácidos consta de unos cinco ribosomas. Esta proporción se mantiene para cualquier proteína; así, para sintetizar una proteína de 300 aminoácidos, haría falta un mRNA de 300 nm sobre el que se acomodarían unos 10 ribosomas. La síntesis de la mayoría de las proteínas tarda entre 20 segundos y algunos minutos. Pero incluso durante este periodo de tiempo tan corto, sobre cada una de las moléculas de mRNA que se está traduciendo tienen lugar múltiples inicios de traducción. En cuanto el ribosoma ha traducido una secuencia de nucleótidos que supone un espacio suficientemente grande, un nuevo ribosoma salta sobre el extremo 5' de la molécula de mRNA. Por ello, las moléculas de mRNA que están siendo traducidas generalmente se encuentran en forma de polirribosomas. Este inicio múltiple permite que se puedan sintetizar muchas más moléculas de proteína por unidad de tiempo de las que sería posible si para poder comenzar la síntesis de una proteína se tuviera que terminar la anterior. Para la síntesis de proteínas los ribosomas recorren el mRNA desde un extremo a otro (5’ a 3’). Por cada tres nucleótidos recorridos incorporan un aminoácido a la cadena de proteína que están Configuración de los Seres Vivos Resumen Tema 11: Los ribosomas 5 Organización general de las células: Citosol y sistema de endomembranas sintetizando; aminoácidos que son proporcionados por los tRNAs. Cuando han completado el recorrido, los ribosomas se liberan del mRNA y sueltan la proteína ya terminada. Mientras se esté sintetizando proteína, por cada ribosoma que abandona el polisoma en el extremo final, otro se incorpora en el inicial, de modo que el polisoma mantiene una apariencia estable aunque sus ribosomas cambien. Tanto las bacterias como las células eucariotas utilizan polisomas, pero las bacterias pueden incrementar aún más la velocidad de síntesis proteica. Debido a que el mRNA bacteriano no necesita ser procesado y es accesible físicamente a los ribosomas a medida que se va sintetizando, los ribosomas ya se unen al extremo libre de la molécula de mRNA e inician la traducción antes de que se termine la transcripción del RNA, siguiendo de cerca a la polimerasa en su desplazamiento sobre el ADN. 4. Introducción a la síntesis de proteínas desde la perspectiva del ribosoma La traducción del mRNA en proteínas depende de moléculas adaptadoras que pueden reconocer y unirse tanto al codón como, en otro lugar de su superficie, al aminoácido. Estos adaptadores son un conjunto de moléculas pequeñas de RNA, conocidas como RNA de transferencia (tRNA), que tienen unos 80 nucleótidos de longitud. Normalmente, las moléculas de tRNA se pliegan formando una estructura tridimensional de doble hélice mediante la formación de pares de bases entre diferentes regiones de la molécula. Las moléculas de tRNA presentan cuatro segmentos de doble hélice, produciendo moléculas que tienen un aspecto de hoja de trébol, esquemáticamente. Esa hoja de trébol presenta otro plegamiento añadido, adquiriendo una estructura compacta en forma de “L” que está estabilizada por enlaces de hidrógeno adicionales entre diferentes regiones de la molécula. Hay dos regiones de nucleótidos desapareados, situadas a ambos extremos de la molécula en forma de “L”, que son cruciales para la función del tRNA en la síntesis de proteínas. Una de estas regiones forma el anticodón, un conjunto de tres nucleótidos consecutivos que se aparea con el codón complementario de la molécula de mRNA. El otro es una región corta de cadena sencilla en el extremo 3' de la molécula; es el lugar donde el tRNA se une al aminoácido que corresponde al codón. El código genético es redundante; es decir, que diferentes codones determinan un mismo aminoácido. Esta redundancia implica o bien que muchos de los aminoácidos tienen más de un tRNA, o bien que algunas moléculas de tRNA pueden aparearse con más de un codón. De hecho, realmente ocurren ambas cosas. Algunos aminoácidos tienen más de un tRNA y algunos tRNA están formados de manera que sólo requieren el apareamiento preciso de las dos primeras posiciones del codón, tolerando un desapareamiento (denominado balanceo) en la tercera posición. Este balanceo en el apareamiento de bases explica por qué muchos de los codones alternativos para un aminoácido solamente se diferencian en el tercer nucleótido. El balanceo en el apareamiento de bases hace Configuración de los Seres Vivos Resumen Tema 11: Los ribosomas 6 Organización general de las células: Citosol y sistema de endomembranas posible ajustar los 20 aminoácidos a los 61 codones, con únicamente 31 tipos de moléculas de tRNA. Sin embargo, el número exacto de diferentes tipos de tRNA varía entre unas especies y otras. Cada molécula de tRNA se une específicamente a su aminoácido correspondiente de los 20 posibles. El reconocimiento y la unión del aminoácido correcto dependen de las aminoacil-tRNA sintetasas, que acoplan covalentemente cada aminoácido a su conjunto apropiado de moléculas de tRNA. Hay una sintetasa diferente para cada aminoácido (20 sintetasas en total). Unos nucleótidos determinados, tanto del anticodón como de la zona de unión al aminoácido, permiten a cada enzima sintetasa reconocer su tRNA. En el proceso de traducción las sintetasas son tan importantes como los tRNA, por que es la acción combinada de sintetasas y tRNA la que permite asociar cada codón de la molécula de mRNA con su aminoácido correspondiente. La reacción catalizada por la sintetasa que une el aminoácido al extremo 3' del tRNA es una de las muchas reacciones celulares acopladas a liberación de energía por hidrólisis de ATP, y produce un enlace de alta energía entre el tRNA y el aminoácido. La energía de este enlace se utiliza en una etapa posterior para unir covalentemente el aminoácido a la cadena polipeptídica en crecimiento. 4.1. Decodificación del mensaje del ARN El reconocimiento de un codón por el anticodón de una molécula de tRNA depende del mismo tipo de apareamiento complementario de bases que se produce en la replicación y en la transcripción del ADN. Sin embargo, la traducción cuidadosa y rápida de un mRNA a proteína requiere una gran maquinaria molecular, el ribosoma; que se desplaza sobre el mRNA capturando moléculas de tRNA complementarias, manteniéndolas en posición y uniendo los aminoácidos que transportan, para formar la cadena proteica. La subunidad pequeña enlaza los tRNA a los codones del mRNA, mientras la subunidad grande cataliza la formación de los enlaces peptídicos que unen los aminoácidos entre si en la cadena polipeptídica. Para iniciar la síntesis de una proteína, las dos subunidades se unen sobre una molécula de mRNA, generalmente cerca de su inicio (extremo 5'). Entonces, el ribosoma se desplaza a lo largo del mRNA, traduciendo la secuencia de nucleótidos a secuencia de aminoácidos, leyendo codones de uno en uno y utilizando los tRNA como adaptadores para añadir cada aminoácido en la secuencia correcta sobre el extremo de la cadena polipeptídica en crecimiento. Finalmente, cuando la síntesis de la proteína termina, las dos subunidades del ribosoma se separan. Los ribosomas trabajan con una eficiencia sorprendente: un ribosoma de una célula eucariota añade 2 aminoácidos por segundo a una cadena polipeptídica y los ribosomas de las células bacterianas trabajan aún más deprisa, a una velocidad de 20 aminoácidos por segundo aproximadamente. ¿Pero cómo puede el ribosoma orquestar los movimientos necesarios para la traducción? Configuración de los Seres Vivos Resumen Tema 11: Los ribosomas 7 Organización general de las células: Citosol y sistema de endomembranas Un ribosoma presenta cuatro lugares de unión para moléculas de RNA: uno es para el mRNA y tres (llamados sitios A, P y E) son para moléculas de tRNA. Una molécula de tRNA se mantiene unida fuertemente en los sitios A y P solamente si su anticodón forma apareamientos de bases (permitiendo el balanceo) con un codón complementario de la molécula de mRNA que está unida al ribosoma. Los sitios A y P están suficientemente próximos como para que las dos moléculas de tRNA estén forzadas a formar apareamientos con codones adyacentes en la molécula de mRNA. - Inicio de la síntesis proteínas: El lugar del mRNA donde se inicia la síntesis de proteínas es crucial, ya que determina la pauta de lectura para todo el mensaje. Un error en esta etapa de un solo nucleótido en cualquiera de los dos sentidos causará errores de lectura de todos los codones siguientes, de forma que se producirá una proteína no funcional, cuya secuencia de aminoácidos será completamente errónea. La etapa de iniciación también es de gran importancia en otro aspecto: es el último punto en el que la célula puede decidir si el mRNA ha de traducirse y la proteína ha de ser sintetizada; la velocidad de iniciación determina la velocidad a la que se sintetizará la proteína. La traducción de un mRNA se inicia con el codón AUG, y se necesita un tRNA especial para iniciarla. Este tRNA de iniciación transporta el aminoácido metionina (las bacterias usan la formilmetionina) de modo que todas las proteínas recién sintetizadas tienen metionina como primer aminoácido en su extremo amino. Frecuentemente esta metionina es eliminada posteriormente por una proteasa específica. El tRNA de iniciación es distinto al tRNA que normalmente une metionina. En los eucariotas, el tRNA de iniciación (que está acoplado a la metionina) se carga en primer lugar en la subunidad pequeña del ribosoma junto con unas proteínas adicionales denominadas factores de iniciación. De todos los tRNA cargados que hay en la célula, sólo el tRNA de iniciación es capaz de unirse fuertemente a la subunidad ribosomal pequeña. A continuación, la subunidad ribosomal cargada se une al extremo 5' de una molécula de mRNA, que es reconocida en parte gracias a la caperuza que se encuentra en los mRNA eucariotas. A continuación, la subunidad ribosomal pequeña se desplaza hacia adelante (5' a 3') a lo largo del mRNA buscando el primer AUG. Cuando lo encuentra, algunos factores de iniciación se liberan de la subunidad ribosomal pequeña dejando espacio para ensamblar la subunidad ribosomal grande y completar el ribosoma. Dado que el tRNA de iniciación está unido al sitio P, la síntesis de la proteína puede empezar con la unión del siguiente tRNA acoplado a su aminoácido. En bacterias el mecanismo para seleccionar un codón de inicio es diferente. Los mRNA bacterianos no presentan caperuzas 5' para indicar al ribosoma a partir de dónde comenzar a buscar el inicio de traducción. Por el contrario, presentan secuencias específicas de unión al ribosoma, de hasta 6 nucleótidos de longitud, situadas unos cuantos nucleótidos por delante de los AUG donde comienza la traducción. A diferencia de los ribosomas eucariotas, los ribosomas procariotas pueden unirse directamente a un codón de inicio que se encuentre en el interior de un mRNA, siempre que el Configuración de los Seres Vivos Resumen Tema 11: Los ribosomas 8 Organización general de las células: Citosol y sistema de endomembranas lugar de unión al ribosoma esté precedido por una secuencia de unos cuantos nucleótidos. Como consecuencia de ello, frecuentemente los mRNA procariotas son policistrónicos; es decir, codifican varias proteínas diferentes, todas ellas traducidas a partir de la misma molécula de mRNA. Por el contrario, generalmente un mRNA eucariota sólo contiene información para una sola proteína, es monocistrónico. - Proceso de elongación de la síntesis de proteínas: Una vez se ha iniciado la síntesis de la proteína, cada nuevo aminoácido es añadido a la cadena en crecimiento mediante un ciclo de reacciones: • Etapa 1: Un tRNA que transporta el siguiente aminoácido de la cadena se une al sitio A vacante en el ribosoma mediante el apareamiento de bases con el codón del mRNA expuesto en el sitio A. • Etapa 2: El extremo carboxilo de la cadena polipeptídica se separa del tRNA situado en el sitio P (por rotura del enlace de alta energía entre el tRNA y su aminoácido) y se une, mediante un enlace peptídico, al grupo amino libre del aminoácido que está unido al tRNA en el sitio A. Esta reacción central de la síntesis de proteínas está catalizada por una enzima con actividad peptidiltransferasa, que forma parte del ribosoma. En este caso parece que la parte catalítica del ribosoma no es una de las proteínas sino uno de los rRNA de la subunidad mayor. Se cree que la reacción peptidiltransferasa va acompañada por un desplazamiento de la subunidad grande respecto a la subunidad pequeña, la cual permanece unida al mRNA. Este desplazamiento mueve los dos tRNA a los sitios E y P de la subunidad grande. • Etapa 3: La subunidad pequeña se desplaza exactamente 3 nucleótidos a lo largo de la molécula de mRNA, recuperando su posición inicial respecto a la subunidad mayor, y liberando el tRNA que ocupaba el sitio E. Este ciclo completo, de tres etapas, se repite cada vez que un aminoácido es añadido a una cadena polipeptídica, desde su extremo amino a su extremo carboxilo, hasta que se encuentra un codón de paro. - Terminación de la síntesis de proteínas: El extremo del mensaje codificador de una proteína está indicado por la presencia de algún codón de entre varios de ellos (UAA, UAG o UGA) denominados codones de paro. Estos codones no son reconocidos por ningún tRNA y no especifican ningún aminoácido, sino que le indican al ribosoma que termine la traducción. Unas proteínas denominadas factores de liberación se unen a cualquier codón de paro que alcance el sitio A del ribosoma, y esta unión altera la actividad catalítica peptidiltransferasa del ribosoma, haciendo que ahora catalice la incorporación, al peptidil-tRNA, de una molécula de agua en lugar de un aminoácido. Esta reacción libera el extremo carboxilo del polipéptido en crecimiento de su unión a la molécula de tRNA y, dado que es el único punto de unión del polipéptido en crecimiento al ribosoma, Configuración de los Seres Vivos Resumen Tema 11: Los ribosomas 9 Organización general de las células: Citosol y sistema de endomembranas la proteína completa queda liberada inmediatamente en el citoplasma. El ribosoma libera el mRNA y se disocia en dos subunidades separadas, que pueden ensamblarse de nuevo sobre otra molécula de mRNA e iniciar una nueva ronda de síntesis de proteínas. Muchas proteínas pueden plegarse espontáneamente adquiriendo su forma tridimensional y la mayoría de ellas lo hacen mientras están siendo sintetizadas por el ribosoma. Sin embargo, para plegarse correctamente algunas proteínas necesitan chaperonas. Las chaperonas identifican estas proteínas cuando comienzan a ser sintetizadas por el ribosoma y las van plegando correctamente a medida que se van sintetizando. 5. Destino de las proteínas sintetizadas en los ribosomas Las proteínas sintetizadas por los ribosomas libres pueden ser: proteínas solubles del citoplasma fundamental, proteínas periféricas de la membrana plasmática (enzimas, actina, espectrina, etc.), proteínas con destino a las mitocondrias, proteínas para los plastos, proteínas del interior de los peroxisomas (catalasa, oxidas de aminoácidos, etc.), proteínas nucleares (histonas, láminas, etc.). Las proteínas sintetizadas en los ribosomas del retículo endoplasmático rugoso también tienen diferentes destinos. Las proteínas recién sintetizadas pueden poseer uno o varios péptidos señal, que sirven para clasificarlas de acuerdo con su destino y determinan dónde deben ser exportadas: al RER, a las mitocondrias, cloroplastos, peroxisomas, lisosomas o al núcleo, ya que en la membrana de esos orgánulos existen receptores específicos para cada péptido señal. La ausencia de péptidos señal determina que la proteína quede en el hialoplasma. Cuando hay un solo péptido señal éste suele ser aminoterminal y se ancla en la membrana del orgánulo correspondiente, pero, a menudo, la proteína puede pasar del RER al complejo de Golgi, o del citoplasma fundamental a la membrana mitocondrial externa y de ésta a la matriz mitocondrial. Entonces, este péptido viene seguido por otro o mas péptidos señal de modo que, una vez eliminado el primer péptido señal, el segundo quede en posición amino terminal marcando el próximo destino, y así sucesivamente hasta que la proteína llegue a su destino final. Configuración de los Seres Vivos Resumen Tema 11: Los ribosomas