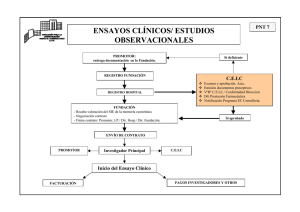
genes de leafy cotiledon2 y sus usos.
Anuncio

19 OFICINA ESPAÑOLA DE PATENTES Y MARCAS C07K 14/415 (2006.01) C12N 15/29 (2006.01) C12N 15/82 (2006.01) C12N 5/04 (2006.01) A01H 5/00 (2006.01) A01H 5/10 (2006.01) ESPAÑA 12 11 Número de publicación: 2 280 352 51 Int. Cl.: TRADUCCIÓN DE PATENTE EUROPEA T3 86 Número de solicitud europea: 01918680 .8 86 Fecha de presentación : 14.03.2001 87 Número de publicación de la solicitud: 1263782 87 Fecha de publicación de la solicitud: 11.12.2002 54 Título: Genes de leafy cotiledon2 y sus usos. 30 Prioridad: 16.03.2000 US 527058 73 Titular/es: The Regents of the University of California 5th Floor, 1111 Franklin Street Oakland, California 94607-5200, US 45 Fecha de publicación de la mención BOPI: 16.09.2007 72 Inventor/es: Harada, John; Kwong, Linda y Matsudaira Yee, Kelly 45 Fecha de la publicación del folleto de la patente: 74 Agente: Zea Checa, Bernabé ES 2 280 352 T3 16.09.2007 Aviso: En el plazo de nueve meses a contar desde la fecha de publicación en el Boletín europeo de patentes, de la mención de concesión de la patente europea, cualquier persona podrá oponerse ante la Oficina Europea de Patentes a la patente concedida. La oposición deberá formularse por escrito y estar motivada; sólo se considerará como formulada una vez que se haya realizado el pago de la tasa de oposición (art. 99.1 del Convenio sobre concesión de Patentes Europeas). Venta de fascículos: Oficina Española de Patentes y Marcas. Pº de la Castellana, 75 – 28071 Madrid ES 2 280 352 T3 DESCRIPCIÓN Genes de leafy cotiledon2 y sus usos. 5 Campo de la invención La presente invención está dirigida a la manipulación genética de plantas. En particular, se refiere a los nuevos genes específicos de embrión útiles para mejorar agronómicamente plantas importantes. 10 15 20 25 30 35 40 Antecedentes de la invención La embriogénesis en plantas superiores es una etapa crítica en el ciclo de vida de la planta en la que se establecen los órganos primarios. El desarrollo del embrión se puede separar en dos fases principales: la fase temprana en la que se establece la organización del cuerpo primario del embrión y la fase tardía que incluye maduración, deshidratación y latencia. En la fase temprana, la simetría del embrión cambia de radial a bilateral, dando lugar a un hipocótilo con un meristemo del vástago rodeado por los dos primordios de los cotiledones en el polo apical y un meristemo de la raíz en el polo basal. En la fase tardía, durante la maduración el embrión alcanza su tamaño máximo y la semilla acumula en almacenaje proteínas y lípidos. La maduración finaliza con la etapa de deshidratación en la que el contenido en agua de la semilla disminuye rápidamente y el embrión pasa al estado metabólico quiescente. La latencia finaliza con la germinación de la semilla, y el desarrollo continúa a partir de las regiones del meristemo del vástago y del meristemo de la raíz. Los mecanismos reguladores precisos que controlan la diferenciación de células y órganos durante la fase inicial de la embriogénesis son bastante desconocidos. Se cree que la hormona de la planta, ácido abscísico (ABA), juega un papel durante la embriogénesis tardía, principalmente en la etapa de maduración inhibiendo la germinación durante la embriogénesis (Black, M. (1991). En ABSCISIC ACID: PHYSIOLOGY AND BIOCHEMISTRY, W. J. Davies y H. G. Jones, eds. (Oxford: Bios Scientific Publishers Ldt.), p. 99-124) y Koornneef, M., y Karssen, C. M. (1994). En ARABIDOPSIS, E. M. Meyerowitz y C. R. Sommerville, eds. (Cold Spring Harbor: Cold Spring Harbour Laboratory Press), p. 313- 334). Se han identificado mutaciones que afectan al desarrollo de la semilla y son insensibles al ABA en Arabidopsis y en maíz. El mutante insensible a ABA (abi3) de Arabidopsis y el mutante vivíparo 1 (vp1) de maíz se detectan principalmente durante la embriogénesis tardía (Mc Carty, et al. (1989) Plant Cell 1:523-532 y Parcy et al. (1994) Plant Cell 6:1567-1552). Tanto el gen de CP1 como los genes ABI3, han sido aislados y se encontró que compartían regiones conservadoras (Giraudat, J. (1995) Current Opinion in Cell Biology 7: 232-238 y McCarty, D. R. (1995). Annu. Rev. Plant Physiol. Plant Mol. Biol. 46:71-93). Se ha mostrado que el producto del gen VP1 que funciona como activador de la transcripción (McCarty, et al. (1991) Cell 66:895-906). Se ha sugerido que ABI3 tiene una función similar. A pesar del reciente progreso en definir el control genético del desarrollo del embrión, se requiere un nuevo progreso en la identificación y análisis de los genes expresados específicamente en el embrión y semilla. La caracterización de dichos genes permitiría la manipulación genética de plantas con una variedad de rasgos deseables. Por ejemplo, se puede usar la modulación de la expresión de los genes que controlen el desarrollo del embrión para alterar rasgos tales como la acumulación en el almacenaje de proteínas en hojas y cotiledones. Alternativamente, se pueden usar promotores genes específicos del embrión o de la semilla para dirigir la expresión de genes heterólogos al embrión o semilla. La presente invención aborda estas y otras necesidades. 45 Breve descripción de los dibujos La Figura 1 es una representación esquemática que muestra los detalles de la estrategia del mapeado de LEC2. 50 55 60 65 Descripción resumida de la invención La invención proporciona un casete de expresión que comprende un promotor unido operativamente a una secuencia polinucleótida heteróloga, o complementaria de la misma, que codifica un polipéptido de LEC2 con por lo menos un 65% de identidad de secuencia con SEC ID NO.2, el polinucleótido induce una morfogénesis embrionaria ectópica o resulta en plantas con semillas no viables, cuando se expresa en una planta. En algunas realizaciones, el polipéptido de LEC2 codificado es SEC ID NO:2. En algunas realizaciones la secuencia del polinucleótido es SEC ID NO:1. El promotor del casete de expresión puede ser constitutivo y/o puede provenir del gen LEC2. Por ejemplo, en una forma de realización, el promotor comprende un polinucleótido por lo menos un 70% idéntico a SEC ID NO:3. El promotor puede ser SEC ID NO:3. En algunas realizaciones, el promotor puede comprender además un polinucleótido por lo menos un 70% idéntico a SEC ID NO:4. El promotor puede comprender además SEC ID NO:4. En algunas realizaciones, el polinucleótido se une al promotor en una orientación antisentido. La presente invención también proporciona un casete de expresión para la expresión de un polinucleótido heterólogo en una célula vegetal. Este casete de expresión comprende un promotor por lo menos un 70% idéntico a SEC ID NO:3 unido operativamente a un polinucleótido heterólogo. En algunas realizaciones, el promotor comprende SEC ID NO:3. En algunas realizaciones, el promotor además comprende un polinucleótido por lo menos un 70% idéntico a SEC ID NO:4. El promotor puede además comprender SEC ID NO:4: 2 ES 2 280 352 T3 5 10 15 20 25 30 35 40 45 50 La invención también proporciona una célula hospedadora que comprende un casete de expresión, que codifica un polipéptido de LEC2 que muestra por lo menos un 65% de identidad de secuencia con SEC ID NO:2. En algunas realizaciones, el ácido nucleico no incluye el cromosoma bacteriano artificial F3H9 (número de acceso a Genbank AG021044). En algunas realizaciones, la célula hospedadora no es una célula de Arabidopsis. El ácido nucleico además puede comprender un promotor unido operativamente al polinucleótido. En algunas realizaciones, el promotor es uno constitutivo. El promotor puede ser un promotor de LEC2, i. e. proceder del gen LEC2. Por ejemplo, en algunas realizaciones, el promotor de LEC2 puede ser por lo menos en un 70% idéntico a SEC ID NO:3. De este modo, el promotor puede comprender SEC ID NO:3. El promotor puede además comprender una secuencia por lo menos en un 70% idéntica a SEC ID NO:4. Por ejemplo, el promotor puede comprender además SEC ID NO:4. En algunas realizaciones, la célula hospedadora se selecciona del grupo que comprende una célula vegetal, bacteriana, de levadura, fúngica o animal. La invención también proporciona un polipéptido aislado que comprende una secuencia de aminoácidos por lo menos un 65% idéntica a SEC ID NO:2, en donde el polipéptido induce morfogénesis embrionaria ectópica o inhibe la producción de semillas en una planta. La invención proporciona un procedimiento para introducir un ácido nucleico aislado en una célula hospedadora que comprende proporcionar un casete de expresión que comprende una secuencia polinucleótida, o complementaria de la misma, que codifica un polipéptido de LEC2 que muestra por lo menos un 65% de identidad de secuencia con SEC ID NO:2, y poner en contacto el ácido nucleico con la célula hospedadora en condiciones que permiten la inserción del ácido nucleico dentro del hospedador. La invención también proporciona un procedimiento de modulación de la transcripción del gen LEC2 que comprende introducir en la célula hospedadora un casete de expresión que comprende un promotor unido operativamente a un polinucleótido heterólogo de LEC2 que codifica un polipéptido de LEC2 por lo menos un 65% idéntico a SEC ID NO:2 y detectar una célula hospedadora con transcripción modulada. En algunas realizaciones, el polipéptido codifica SEC ID NO:2. Por ejemplo, el polipéptido puede ser SEC ID NO:1. En algunas realizaciones, la célula hospedadora es una célula vegetal. En algunas realizaciones, la célula hospedadora no es una célula de Arabidopsis. En algunas realizaciones, se introduce el casete de expresión mediante Agrobacterium. En otra realización, el casete de expresión se introduce mediante cruzamiento sexual. En algunas realizaciones, la modulación de la transcripción resulta en la inducción de características embrionarias en una planta. En algunas realizaciones, se regenera una planta a partir de la célula vegetal. La invención también proporciona un procedimiento para detectar un ácido nucleico en una muestra que comprende (1) proporcionar una molécula de ácido nucleico aislada que consiste en una secuencia polinucleótida, que codifica un polipéptido de LEC2 que muestra por lo menos un 65% de identidad de secuencia con SEC ID NO:2, (2) poner en contacto la molécula de ácido nucleico aislada con una muestra en condiciones que permiten la comparación de la secuencia de la molécula de ácido nucleico aislada con la secuencia de ADN en la muestra, y (3) analizar el resultado de la comparación. En algunas realizaciones, la molécula de ácido nucleico aislada y la muestra se ponen en contacto en condiciones que permiten la formación de un dúplex entre las secuencias de ácido nucleico complementarias. La invención también proporciona una planta transgénica o una célula vegetal transgénica que comprende un casete de expresión que codifica un polipéptido de LEC2 que muestra por lo menos un 65% de identidad de secuencia con SEC ID NO:2. En algunas realizaciones, se regenera una planta a partir de una célula de planta. La secuencia puede ser SEC ID NO:1. En algunas realizaciones, el polipéptido es SEC ID NO:2. El casete de expresión comprende un promotor que unido operativamente al polinucleótido. En algunas realizaciones, el promotor es uno constitutivo. El promotor puede ser un promotor de LEC2, i.e. procedente del gen LEC2. Por ejemplo, en algunas realizaciones, el promotor de LEC2 puede ser por lo menos un 70% idéntico a SEC ID NO:3. De este modo, el promotor puede comprender SEC ID NO:3. El promotor puede comprender además una secuencia por lo menos un 70% idéntica a SEC ID NO:4. Por ejemplo, el promotor puede comprender además SEC ID NO:4. En algunas realizaciones, la secuencia polinucleótida se une al promotor en una orientación antisentido. 55 Definiciones 60 La frase “ácido nucleico” se refiere a un polímero de cadena sencilla o doble de bases desoxiribonucleótidas o ribonucleótidas leídas del extremo 5’ al 3’. Los ácidos nucleicos pueden incluir también nucleótidos modificados que permiten la lectura correcta a través de ellos mediante una polimerasa y no alteran la expresión de un polipéptido codificado por ese ácido nucleico. La frase “secuencia polinucleótica” o “secuencia de ácido nucleico” ambas incluyen las cadenas sentido y antisentido de un ácido nucleico ya sea como cadenas únicas individuales o en dobles cadenas. Incluye, pero no se limita a, plásmidos autoreplicantes, secuencias cromosómicas, y polímeros infecciosos de ADN o ARN. 65 La frase “secuencia de ácido nucleico codificante” se refiere a un ácido nucleico que dirige la expresión de una proteína o péptido específicos. Las secuencias de ácido nucleico incluyen tanto la secuencia de la cadena de ADN que se transcribe a ARN y la secuencia ARN que se traduce a proteína. Las secuencias de ácido nucleico incluyen 3 ES 2 280 352 T3 tanto secuencias de ácido nucleico de longitud completa como secuencias de longitud incompleta derivadas de las secuencias de longitud completa. Además se debería entender que la secuencia incluye codones degenerados de la secuencia o secuencias nativas que se pueden introducir para proporcionar preferencia de codón específica en una célula hospedadora. 5 10 15 El término “promotor” se refiere a determinantes de región o de secuencia localizados upstream o downstream desde el inicio de la transcripción y que están implicados en el reconocimiento y la unión de la ARN polimerasa y de otras proteínas para iniciar la transcripción. Un “promotor vegetal” es un promotor capaz de iniciar la transcripción en células vegetales. Dichos promotores no necesitan ser de origen vegetal, por ejemplo, promotores derivados de virus de las plantas, tales como el promotor CaMV35S, se pueden utilizar en la presente invención. El término “planta” incluye la planta entera, los órganos/estructuras vegetales del vástago, (p.e. hojas, tallos y tubérculos), raíces, flores y órganos/estructuras florales (p.e. brácteas, sépalos, pétalos, estambres, carpelos, anteras y óvulos), semillas (incluyendo embrión, endosperma, cubierta seminal), y frutos (ovario maduro), tejidos vegetales (p.e. tejido vascular, tejido fundamental, y similares) y células (p.e. célula oclusiva, célula huevo, tricomas y similares) y la progenie del mismo. La clase de plantas que se puede usar en el procedimiento de la invención es generalmente tan extensa como la clase de plantas superiores e inferiores sensibles a técnicas de transformación, incluyendo angiospermas (plantas monocotiledóneas y dicotiledóneas), gimnospermas, helechos, y algas multicelulares. Incluye plantas de una variedad de niveles de ploidía, incluyendo aneuploides, poliploides, diploides, haploides y hemizigotos. 20 La frase “célula hospedadora” se refiere a una célula de cualquier organismo. Las células hospedadoras preferidas provienen de las plantas, bacterias, levaduras, hongos, insectos u otros animales. Los procedimientos para introducir secuencias polinucleótidas en los distintos tipos de células hospedadoras son bien conocidas en la materia. 25 La “actividad biológica de un polipéptido” se refiere a cualquier actividad molecular o fenotipo que causa el polipéptido. Por ejemplo, la capacidad para transferir un fosfato a un sustrato o la capacidad de unir una secuencia de ADN específica es una actividad biológica. Una actividad biológica de LEC2 es la capacidad de inducir características embrionarias en órganos vegetales. 30 Una secuencia polinucleótida es “heteróloga a” una segunda secuencia polinucleótida si se origina a partir de una especie foránea, o, si proviene de la misma especie, se modifica de su forma original por la acción del hombre. Por ejemplo, un promotor unido operativamente a una secuencia codificante heteróloga se refiere a una secuencia codificante de una especie diferente de la que deriva el promotor, o, si proviene de la misma especie, una secuencia codificante que es diferente de cualquiera de las variantes alélicas que se encuentran de manera natural. 35 Un polinucleótido “exógeno a” una planta individual es un polinucleótido que se introduce en la planta, o en una generación predecesora de la planta, por cualquier medio diferente del cruzamiento sexual. Ejemplos de medios con los que se puede conseguir esto se describen más abajo, e incluyen la transformación mediada por Agrobacterium, procedimientos biolisíticos, electroporación, técnicas en planta, y similares. 40 45 50 55 60 65 Como se utiliza en el presente documento un “gen específico de embrión” o “gen específico de la semilla” es un gen que preferentemente se expresa durante el desarrollo embrionario de la planta. Para los objetivos de esta memoria, el desarrollo embrionario empieza con las primeras divisiones celulares en el cigoto y continúa a través de la fase tardía del desarrollo embrionario (caracterizado por la maduración, desecación, latencia), y termina con la producción de una semilla madura y desecada. Los genes específicos de embrión se pueden clasificar además como “específicos de la fase temprana” y “específicos de la fase tardía”. Los genes específicos de la fase temprana son aquellos que se expresan en embriones hasta el final de la morfogénesis embrionaria. Los genes específicos de la fase tardía son aquellos que se expresan desde la maduración hasta la producción de una semilla madura y desecada. Un “polinucleótido de LEC2” es una secuencia de ácido nucleico que comprende (o consiste en) una región codificante de aproximadamente 50 a aproximadamente 6800 nucleótidos, algunas veces de aproximadamente 100 a aproximadamente 3000 nucleótidos y a veces de aproximadamente 300 a aproximadamente 1300 nucleótidos, que hibrida con SEC ID NO:1 en condiciones rigurosas (como se definen abajo), o que codifican un polipéptido de LEC2 o un fragmento de al menos 15 aminoácidos del mismo. Los polinucleótidos de LEC2 se pueden identificar también por su capacidad para hibridar en condiciones de baja rigurosidad (p.e. Tm∼40ºC) a sondas de ácido nucleico que tienen la secuencia de SEC ID NO:1, SEC ID NO:1, SEC ID NO:5 (el ADNc de LEC2) y SEC ID NO:6 son ejemplos de polinucleótidos de LEC2. Un “promotor de un gen de LEC2” o “promotor de LEC2” típicamente será de aproximadamente 500 a aproximadamente 4000 nucleótidos de longitud, normalmente de aproximadamente 720 a 3200. Secuencias de promotores ejemplares se muestran como SEC ID NO:3 y SEC ID NO:4. SEC ID NO:3 representa la región 5’ no traducida de LEC2 y SEC ID NO:4 representa la región 3’ no traducida de LEC2. Se puede identificar también un promotor de LEC2 por su capacidad para dirigir la expresión en semillas en la etapa temprana así como también durante la embriogénesis en la etapa temprana y media. El promotor no proporciona una expresión significativa en el tejido de la hoja. Un “polipéptido de LEC2” es una secuencia de aproximadamente 50 a aproximadamente 400, algunas veces de 100 a 150, y preferentemente de 363 residuos de aminoácidos codificados por un polinucleótido de LEC2. Los polipéptidos 4 ES 2 280 352 T3 5 10 15 20 25 30 de LEC2 se caracterizan por la presencia de un dominio B3. Por ejemplo los residuos de aminoácidos del 165 al 277 representan el dominio B3 del polipéptido mostrado en SEC ID NO:2. Se conoce en la materia el dominio B3 y es compartido por otros factores de transcripción incluyendo VIVIPAROUS1 (VP1) ((McCarty, et al. (1989) Plant Cell 1:523-532), AUXIN RESPONSE FACTOR 1 (ARF1) (Ulmasov, T. et al. (1997) Science 276:1865-1868), FUSCA3 (Luerben, H., et al. (1998) Plant J. 15:755-764) y ABI3 (Giraudat, J. et al. (1992) Plant Cell 4, 1251-1261). Los dominios B3 de VP1 (Suzuki, M. et al. (1997) Plant Cell 9:799-807) y ARF1 (Ulmasov, T., et al., supra) han mostrado que son dominios de unión de ADN. El dominio B3 de LEC2 es por lo tanto también un dominio de unión de ADN. Como se usa en el presente documento, un homólogo de un gen particular específico del embrión (p.e., SEC ID NO:1) es un segundo gen en el mismo tipo de planta o en un tipo diferente de planta, que tiene una secuencia polinucleótida de por lo menos 50 nucleótidos continuos que son sustancialmente iguales (determinados como se describe más abajo) a una secuencia en el primer gen. Se cree que, en general, los homólogos comparten un pasado común en la evolución. “Actividad aumentada o mejorada de LEC2 o expresión del gen de LEC2”se refiere a un cambio aumentado en la actividad de LEC2. Ejemplos de dicha actividad o expresión incrementadas incluyen lo siguiente. La actividad de LEC2 o expresión del gen LEC2 se incrementa por encima del nivel de esta en el tipo natural. Plantas control notransgénicas (p.e. se aumenta la cantidad de actividad de LEC2 o la expresión del gen LEC2). La actividad de LEC2 o la expresión del gen de LEC2 se da en un órgano, tejido o célula donde no se detecta normalmente en el tipo natural, plantas control no-transgénicas (i.e. se aumenta la distribución espacial de la actividad de LEC2 o expresión del gen de LEC2). Se aumenta la actividad de LEC2 o expresión cuando la actividad de LEC2 o expresión del gen de LEC2 está presente en un órgano, tejido o célula por un periodo más largo que en el tipo de referencia, controles no-transgénicos (i.e. se aumenta la duración de la actividad de LEC2 o expresión del gen LEC2). Una “secuencia polinucleótida a partir de” un gen particular es una subsecuencia o una secuencia polinucleótida de longitud completa de un gen específico de embrión que, cuando está presente en una planta transgénica, tiene el efecto deseado. Por ejemplo, un efecto es la inhibición de la expresión de un gen endógeno que dirige la expresión de un polinucleótido heterólogo. Una secuencia de longitud completa de un gen particular descrita aquí puede contener aproximadamente un 95%, normalmente por lo menos aproximadamente un 98%, de una secuencia entera mostrada en el Listado de Secuencias de más abajo. El término “tejidos reproductivos” como se utiliza en el presente documento incluye fruto, óvulos, semillas, polen, pistilos, flores o cualquier tejido embrionario. 35 40 45 Un “casete de expresión” se refiere a una estructura de ácido nucleico, que una vez introducida en la célula hospedadora, resulta en la trascripción y/o la traducción de un ARN o polipéptido respectivamente. Las construcciones sentido y antisentido que no están o que no pueden ser traducidas están incluidas específicamente en esta definición. En el caso de tanto la expresión de transgenes como de la inhibición de genes endógenos (p.e. por supresión sentido o antisentido) un experto reconocerá que la secuencia polinucleótida insertada no necesita ser idéntica y puede ser “sustancialmente idéntica” a la secuencia del gen del que derivó. Como se explica más abajo, estas variantes están específicamente cubiertas por este término. En el caso donde la secuencia polinucleótida insertada se transcribe y se traduce para producir un polipéptido funcional, un experto reconocerá que, a causa de la degeneración del codón, un número de secuencias polinucleótidas codificarán el mismo polipéptido. El término “secuencia polinucleótida a partir de” un gen particular específico del embrión, como es LEC2, cubre específicamente estas variantes. Además, el término incluye específicamente secuencias (p.e. secuencias de longitud completa) sustancialmente idénticas (como se describe más abajo) a la secuencia del gen LEC2 y que codifica proteínas que conservan la función del polipéptido de LEC2. 50 En el caso de los polinucleótidos utilizados para inhibir la expresión de un gen endógeno, la secuencia introducida no necesita ser perfectamente idéntica a una secuencia del gen endógeno diana. La secuencia polinucleótida introducida normalmente será por lo menos sustancialmente idéntica (como se describe más abajo) a la secuencia endógena diana. 55 Se dice que dos secuencias de ácido nucleico o polipéptidos son “idénticas” si la secuencia de nucleótidos o residuos de aminoácidos, respectivamente, en las dos secuencias es lo mismo cuando se alinean con la máxima correspondencia como se describe más abajo. En el presente documento el término “complementario a” se usa para dar a entender que la secuencia es complementaria a todo o parte de la secuencia polinucleótida de referencia. 60 65 Se puede dirigir la alineación óptima de secuencias por comparación mediante el algoritmo de homología local de Smith y Waterman Add. APL. Math.2:482 (1981), mediante el algoritmo de homología de alineación de Needleman y Wunsch J. Mol. Biol. 48:443 (1970), mediante la búsqueda por el procedimiento de semejanza de Pearson y Lipman Proc. Natl. Acad. Sci. (U.S.A.) 85:2444 (1988), con implementaciones computerizadas de estos algoritmos (GAP, BESTFIT, FASTA y TFASTA en el Wisconsin Genetics Software, Genetics Computer Group (GCG), 575 Science Dr., Madison, WI), o con inspección. 5 ES 2 280 352 T3 5 10 15 20 25 30 35 40 El “porcentaje de identidad de secuencia” se determina mediante comparación de dos secuencias óptimamente alineadas sobre una ventana de comparación, donde la porción de la secuencia polinucleótida en la ventana de comparación puede comprender adiciones o deleciones (p.e. huecos) comparada con la secuencia de referencia (que no comprende adiciones ni deleciones) para la óptima alineación de las dos secuencias. Se calcula el porcentaje determinando el número de posiciones en la que la base de ácido nucleico o residuo aminoácido idéntico se presenta en ambas secuencias para conseguir el número de posiciones ponderadas, dividiendo el número de posiciones ponderadas por el número total de posiciones en la ventana de comparación y multiplicando el resultado por 100 para conseguir el porcentaje de identidad de secuencia. El término “identidad sustancial” de las secuencias polinucleótidas significa que un polinucleótido comprende una secuencia que tiene por lo menos un 25% de identidad de secuencia. Alternativamente el porcentaje de identidad puede ser cualquier entero entre el 25% y 100%. Más relaciones preferidas incluyen por lo menos un 65%, 70%, 75%, 80%, 85%, 90%, 95%, o un 99% comparadas con una secuencia de referencia utilizando los programas descritos en el presente documento; preferentemente BLAST utilizando parámetros estándares, como se describen más abajo. De acuerdo con esto, las secuencias de LEC2 de la invención incluyen secuencias de ácido nucleico que tienen identidad sustancial con SEC ID NO:1, SEC ID NO:3 y SEC ID NO:4. Las secuencias de LEC2 de la invención también incluyen secuencias polipeptídicas que tienen identidad sustancial con SEC ID NO:2. Un experto reconocerá que estos valores se pueden ajustar de manera apropiada para determinar la identidad correspondiente de proteínas codificadas mediante dos secuencias de nucleótidos teniendo en cuenta la degeneración del codón, la similitud de aminoácido, el posicionamiento del marco de lectura y similares. La identidad sustancial de secuencias de aminoácidos con estos propósitos normalmente significa una identidad de secuencia de al menos un 65%. El porcentaje preferido de identidad de polipéptidos puede ser cualquier entero entre 65% y 100%. Muchas de las realizaciones preferidas incluyen por lo menos un 65%, 70%, 75%, 80%, 85%, 90%, 95%, o un 99%. Las realizaciones más preferidas incluyen un 65%, 66%, 67%, 68%, 69%, 70%, 71%, 72%, 73%, 74% y un 75%. Los polipéptidos que son “sustancialmente similares” comparten secuencias como se indica más abajo excepto que las posiciones de residuos que no son idénticas pueden diferir mediante cambios conservativos de aminoácidos. Las sustituciones conservativas de aminoácidos se refieren a la capacidad de intercambio de residuos que tienen cadenas laterales similares. Por ejemplo, un grupo de aminoácidos que tienen cadenas alifáticas laterales es la glicina, la alanina, la valina, la leucina y la isoleucina; un grupo de aminoácidos que tienen cadenas alifáticas laterales con un grupo hidroxilo es la serina, y la treonina; un grupo de aminoácidos que tienen cadenas laterales que contienen un grupo amido es la asparagina y la glutamina; un grupo de aminoácidos que tienen cadenas laterales aromáticas es la fenilalanina, la tirosina y el triptófano; un grupo de aminoácidos que tienen cadenas laterales básicas es la lisina, la arginina y la histidina; un grupo de aminoácidos que tienen cadenas laterales que contienen un grupo sulfuro es la cisteína y la metionina. Los grupos de aminoácidos de sustitución conservativa son: valina-leucina-isoleucina, fenilalanina- tirosina, lisina-arginina, alanina-valina, ácido aspártico-ácido glutámico, y asparagina-glutamina. Otra indicación de que las secuencias de nucleótidos son sustancialmente idénticas es cuando dos moléculas hibridan entre ellas, o con un tercer ácido nucleico, en condiciones rigurosas. Las condiciones rigurosas dependen de las secuencias y serán diferentes en circunstancias diferentes. Generalmente, las condiciones rigurosas se sitúan a aproximadamente 5ºC por debajo del punto de fusión térmico (Tm) para la secuencia específica a una fuerza iónica y pH definidos. La Tm es la temperatura (bajo fuerza iónica y pH definidos) en la cual el 50% de la secuencia diana hibrida a una sonda perfectamente emparejada. Normalmente, las condiciones rigurosas serán aquellas en las cuales la concentración de sal sea aproximadamente de 0,02 molar a pH 7 y la temperatura sea por lo menos aproximadamente de 60ºC. 45 50 En la presente invención, el ARNm codificado por los genes específicos de embrión de la invención se puede identificar mediante transferencias Nothern en condiciones rigurosas utilizando los ADNc complementarios de la invención o fragmentos de al menos aproximadamente 100 nucleótidos. Para los propósitos de lo aquí expuesto, las condiciones rigurosas para las hibridaciones ARN-ADN son aquellas que incluyen por lo menos un lavado en 0,2X SSC a 63ºC durante 20 minutos, o condiciones equivalentes. El ADN genómico o ADNc que comprende genes de la invención se pueden identificar utilizando los mismos ADNc (o fragmentos de al menos 100 nucleótidos) en condiciones rigurosas, que para el propósito de lo aquí expuesto, incluyen al menos un lavado (normalmente 2) en 0,2X SSC a una temperatura de al menos aproximadamente 50ºC, normalmente aproximadamente a 55ºC, durante 20 minutos, o condiciones equivalentes. 55 Descripción detallada 60 La presente invención proporciona nuevos casetes de expresión útiles para las plantas obtenidas mediante manipulación genética. En particular, la invención proporciona un casete de expresión que comprende un gen de Arabidopsis mencionado aquí como LEC2. Se puede utilizar la modulación de la expresión de este gen para manipular un número de rasgos útiles, tales como la inducción del desarrollo de la semilla. Las secuencias polinucleótidas de los genes de la invención también se pueden utilizar, por ejemplo, para dirigir la expresión de los genes heterólogos deseados en embriones (en el caso de secuencias de promotores) o para modular el desarrollo de embriones o características embrionarias en otros órganos (p.e. aumentando o disminuyendo la expresión del gen en una planta transgénica). 65 En todavía otro uso, se pueden utilizar los casetes de expresión de la invención en el desarrollo de líneas de plantas apomícticas (p.e. plantas en las cuales los procesos de reproducción asexual ocurren en el óvulo, ver, Koltunov, A. Plant Cell 5:1425-1437 (1993) para una discusión sobre apomixis). La apomixis proporciona un nuevo medio para 6 ES 2 280 352 T3 seleccionar y fijar genotipos heterocigotos que no se pueden mantener fácilmente por reproducción tradicional. De este modo, por ejemplo, se pueden obtener y mantener fácilmente líneas nuevas de híbridos con rasgos deseados (p.e. vigor híbrido). Los casetes de expresión de LEC2 de la invención también se pueden utilizar en combinación con otros genes en la producción de líneas de plantas apomícticas. 5 10 Generalmente, la nomenclatura y los procedimientos de laboratorio en la tecnología del ADN recombinante descritos más abajo son aquellos bien conocidos y comúnmente utilizados en la materia. Se utilizan técnicas estándar para la clonación, aislamiento de ADN y ARN, amplificación y purificación. Generalmente, se llevan a cabo reacciones enzimáticas que implican la ADN ligasa, la ADN polimerasa, endonucleasas de restricción y similares según las especificaciones del fabricante. Se llevan a cabo generalmente estas técnicas y varias otras técnicas de acuerdo a Sambrook et al., Molecular Cloning - A Laboratory Manual, 2nd. ed., Cold Spring Harbor Laboratory, Cold Spring Harbor, New York, (1989). Aislamiento de ácidos nucléicos 15 20 25 30 Se puede conseguir el aislamiento de secuencias a partir de genes mediante varias técnicas. Por ejemplo, se pueden utilizar sondas oligonucleótidas basadas en las secuencias descritas en el presente documento para identificar el gen deseado en una librería de ADNc o ADN genómico a partir de unas especies de plantas deseadas. Para construir librerías genómicas, se producen segmentos largos de ADN genómico mediante fragmentación aleatoria, i.e. utilizando endonucleasas de restricción, que se ligan con el ADN del vector para formar concatenarios que se pueden empaquetar en el vector adecuado. Para preparar una librería de ADNc específicos de embrión, se aísla ARNm a partir de embriones y se prepara una librería de ADNc que contiene las transcripciones de genes a partir del ARNm. Se puede cribar también el ADNc o librería genómica utilizando una sonda basada en la secuencia de un gen específico de embrión clonado tal como los polinucleótidos descritos aquí. Se pueden utilizar sondas para hibridar con secuencias de ADN genómico o ADNc para aislar genes homólogos en la misma o diferentes especies de plantas. Alternativamente, se pueden amplificar los ácidos nucléicos de interés a partir de muestras de ácido nucleico mediante técnicas de amplificación. Por ejemplo, la tecnología de la reacción de la polimerasa en cadena (PCR) para amplificar las secuencias de los genes directamente a partir de ARNm, de ADNc, de librerías genómicas o de librerías de ADNc. La PCR y otros procedimientos de amplificación in vitro pueden ser también útiles, por ejemplo, para clonar secuencias de ácido nucleico que codifican para que las proteínas se expresen, para hacer ácidos nucleicos útiles como sondas que detectan la presencia del ARNm deseado en muestras, para secuenciar ácido nucleico, o para otros propósitos. 35 40 45 Se generan cebadores y sondas adecuados para identificar los genes específicos de embrión de tejidos de planta a partir de comparaciones de las secuencias proporcionadas por el presente documento. Para una descripción general de la PCR ver Protocolos de PCR: A Guide to Methods and Applications. (Innis, M, Gelfand, D., Sninsky, J. and White, T., eds.), Academic Press, San Diego (1990). Los cebadores adecuados para la amplificación de la región genómica de LEC2 incluyen los siguientes tres pares de cebadores: D2F - 5’TTTCAGAATACGCAAAAACGAC3’ (SEC ID NO:7) y D2R - 5’AACTATCCTCCCGAGTGACC3’ (SEC ID NO:8); Ef - 5’AGATGGCAAGGATCAACAGG3’ (SEC ID NO:9) y BlastR - 5’CTTGCTTTCGTCCTCGTATATTG3’ (SEC ID NO:10); y F2F - 5’TTTGTGAAGCAAAAT > GGAGC3’ (SEC ID NO:11) y Stop - 5’CGGATGAACCCACGTACG3’ (SEC ID NO:12). Los cebadores adecuados para la amplificación del ADNc de LEC2 incluyen el siguiente par: 5’AAATGGATAACTTCTTACCCTTTCC3’ (SEC ID NO:13) y 5’CGGATGAACCCACGTACG3’ (SEC ID NO:14). Las condiciones de amplificación son normalmente las siguientes. Componentes de reacción: 10 mM de Tris-HCl, pH 8,3, 50 mM de cloruro de potasio, 1,5 mM de cloruro de magnesio, 0,001% de gelatina, 200 µM de dATP, 200 µM de dCTP, 200 µM de dGTP, 200 µM de dTTP, 0,4 µM de cebadores, 100 unidades por ml de polimerasa Taq. Programa: 96 C durante 3 min., 30 ciclos de 96 C durante 45 seg., 50 C durante 60 seg. 72 durante 60 seg. seguido de 72 C durante 5 min. 50 55 60 Se pueden sintetizar también polinucleótidos mediante técnicas bien conocidas como las descritas en la literatura técnica. Ver, i.e., Carruthers et al., Cold Spring Harbor Symp. Quant. Biol. 47:411-418 (1982), y Adams et al., J. Am. Chem. Soc. 105:661 (1983). Entonces se pueden obtener fragmentos de ADN de doble cadena tanto mediante la síntesis de la cadena complementaria y emparejamiento de las cadenas en condiciones adecuadas, como mediante la adición de la cadena complementaria utilizando la ADN polimerasa con una secuencia de cebador adecuada. El género de las secuencias de ácidos nucleicos de LEC2 incluye genes y productos génicos identificados y caracterizados mediante análisis secuencias, secuencias de ácidos nucleicos, incluyendo SEC ID NO:1 y secuencias de proteínas, incluyendo SEC ID NO:2. Las secuencias de LEC2 de la invención incluyen secuencias de ácidos nucleicos que tienen identidad sustancial con SEC ID NO:1. Las secuencias de LEC2 incluyen las secuencias polipéptidicas que tienen identidad sustancial con SEC ID NO:2. Utilización de ácidos nucléicos 65 El análisis genético y molecular de genes implicados en el desarrollo del embrión revela un número de cualidades de la planta que se pueden modular utilizando ácidos nucléicos de LEC2. Los genes LEAFY COTYLEDON (LEC) de Arabidopsis, LEC1, LEC2, y FUSCA3 (FUS3), controlan varios aspectos del desarrollo de la semilla y del embrión (Baumlein, H., et al. (1994) Plant J. 6:379-387; Keith, K., et al., (1994) Plant Cell 6:589-600; Lotan, T., et al., (1998) 7 ES 2 280 352 T3 5 10 15 20 25 30 35 40 Cell 93:1195-1205; Luerben, H., et al. (1998) Plant J. 15:755-764; Meinke, D. W. (1992) Science 258:1647-1650; Meinke, D. W., et al. (1994) Plant Cell 6:1049-1064; Parcy, F., et al. (1997) Plant Cell 9:1265-1277; West, M.A.L., et al. (1994) Plant Cell 6:1731-1745). Los análisis de plantas con mutaciones en estos genes sugieren que son requeridos para el mantenimiento del objetivo de la célula del suspensor en la embriogénesis temprana, la especificación de identidad del cotiledón, la inducción y/o mantenimiento de la fase tardía de maduración, y la supresión del desarrollo postgerminativo durante el desarrollo de la semilla (Harada, J.J. (1997) en ADVANCES IN CELLULAR AND MOLECULAR BIOLOGY OF PLANTS, Volumen 4, Cellular and Molecular Biology of Seed Development, B.A. Larkins y I.K. Vasi, eds. (Dordrecht: Kluver Academia Publishers), pp. 545-592). Por ejemplo, divisiones celulares anormales ocurren en los suspensores de mutantes de lec2 indicando defectos en la embriogénesis temprana (Lotan et al., supra). A diferencia de los embriones de tipo natural, los mutantes de lec2 tienen tricomas en sus cotiledones (Meinke et al.(1994), supra). A causa de que los tricomas son rasgos de la hoja en Arabidopsis, los mutantes tienen cotiledones incompletamente específicos que han revertido parcialmente en las hojas. Aunque el embrión mutante de lec2 puede sobrevivir al secado con sílice (Meinke et al. (1994), supra), partes de los cotiledones son intolerantes a la deshidratación. Estas mismas regiones de los cotiledones son también anormales de manera que no acumulan los ARN expresados normalmente durante la embriógenesis tardía. Además, los embriones del mutante de lec2 tienen activados los meristemos apicales de los vástagos que normalmente son característicos de las plántulas, e indican que el desarrollo postgerminativo ha ocurrido prematuramente. Estas últimas observaciones sugieren que la mutación de lec2 causa defectos en la fase tardía de maduración de la semilla del desarrollo del embrión. A causa de que los genes LEC juegan un papel regulador en muchos aspectos del desarrollo de la semilla, son los reguladores centrales de la embriogénesis. La necesidad de genes LEC en diferentes aspectos de la embriogénesis sugiere que operan para controlar y coordinar el desarrollo de la semilla (Lotan et al., supra). Estudios con el gen LEC1 sugieren que estos genes pueden jugar un papel fundamental en la iniciación del desarrollo de la semilla estableciendo un entorno que permite que ocurra la embriogénesis. Se detecta el ARN de LEC1 en el zigoto pero no en los óvulos de prefertilización, sugiriendo que las funciones del gen se dan en las fases más tempranas del desarrollo embrionario. Normalmente el gen LEC1 se expresa solamente durante el desarrollo de la semilla. Experimentos en los que se manipuló el gen LEC1 para que se expresara en la fase postembrionaria tuvieron como resultado la inducción de la formación del embrión ectópico a partir de células vegetativas (Lotan et al., supra). Juntos, estos resultados sugieren que LEC1 es suficiente para establecer un entorno celular que permite que ocurra la formación del embrión. Diferentes líneas de razonamiento sugieren que LEC2 y FUS3 pueden funcionar de una manera similar a LEC1. Primero, LEC1 es suficiente pero no necesario en la inducción de la formación del embrión. Eso es, los mutantes lec1 aún así producen embriones que sugieren que otros genes pueden tener funciones que se solapan con LEC1 (Lotan et al., supra). Segundo, estudios genéticos muestran que LEC2 interacciona sinérgicamente con LEC1 sugiriendo que estos genes interaccionan físicamente o tienen parcialmente funciones redundantes (Lotan et al., supra; Meinke et al. (1994), supra). LEC2 y FUS3 actúan también sinérgicamente. La interpretación más sencilla de estos resultados es que LEC1, LEC2 y FUS3 juegan papeles fundamentales en la iniciación de la formación del embrión. Las identidades de los genes LEC son consecuentes con sus papeles como genes reguladores. LEC1 codifica un homólogo de la subunidad HAP3 del factor de transcripción de unión a la caja CCAAT (Lotan et al., supra). La proteína FUS3 contiene el dominio B3 del factor de transcripción del viviparous 1/ABA INSENSTIVE3 (Giraudat, J., et al. (1992) Plant Cell 4, 1251-1261; Luessen et al., supra; McCarty, D.R., et al. (1991) Cell Plant 66:895-906). Este dominio B3 contiene un dominio unido a ADN (Suzuki, M., et al. (1997) Plant Cell 9:799-807). 45 Utilización de ácidos nucléicos para inhibir la expresión del gen 50 55 60 65 Las secuencias aisladas preparadas como se describen en este documento, se pueden utilizar para preparar casetes de expresión útiles en varias técnicas. Por ejemplo, se pueden utilizar los casetes de expresión de la invención para suprimir la expresión del gen LEC2 endógeno. La inhibición de la expresión puede ser útil, por ejemplo, para el control de las malas hierbas (mediante la transferencia de una secuencia inhibidora a especies de malas hierbas y permitiéndole transferirse mediante cruzamiento sexual), o para producir frutos con semilla pequeña o no viable. Se pueden utilizar varios procedimientos para inhibir la expresión del gen en plantas. Por ejemplo, se puede utilizar convenientemente tecnología antisentido. Para conseguir esto, se clona un segmento de ácido nucleico a partir de un gen deseado y se une operativamente a un promotor de manera que se transcriba la cadena antisentido del ARN. De esta manera el casete de expresión se transforma en las plantas y se produce la cadena antisentido de ARN. En células vegetales, se ha sugerido que el ARN antisentido inhibe la expresión del gen previniendo la acumulación de ARNm que codifica al enzima de nuestro interés, ver, i.e., Sheehy et al., Proc. Nat. Acad. Sci. USA, 85:8805-8809 (1988), y Hiatt et al., patente US Nº 4.801.340. La secuencia antisentido de ácido nucleico transformada en plantas será sustancialmente idéntica a por lo menos una porción del gen o genes endógenos específicos del embrión a reprimir. La secuencia, sin embargo, no tiene que ser perfectamente idéntica a la expresión inhibidora. Se pueden diseñar los vectores de manera que el efecto inhibidor se aplique a otras proteínas dentro de una familia de genes que muestre homología o una homología sustancial al gen diana. 8 ES 2 280 352 T3 5 10 15 20 25 Para la supresión en antisentido, la secuencia introducida no necesita ser también de longitud completa respecto ya sea el producto primario de transcripción o ARNm completamente procesado. Generalmente, se puede utilizar una mayor homología para compensar el uso de secuencias más cortas. Además, la secuencia introducida no necesita tener el mismo patrón intrón o exón, y la homología de segmentos no codificantes puede ser igualmente efectiva. Normalmente, se debería utilizar una secuencia de entre aproximadamente 30 ó 40 nucleótidos y aproximadamente nucleótidos de longitud completa, aunque se prefiera una secuencia de al menos aproximadamente 100 nucleótidos, es más preferida una secuencia de al menos aproximadamente 200 nucleótidos, y es especialmente preferida una secuencia de al menos aproximadamente 500 nucleótidos. Se pueden utilizar también moléculas de ARN catalítico o ribozimas para inhibir la expresión de genes específicos del embrión. Es posible diseñar ribozimas que se emparejen específicamente con virtualmente cualquier ARN diana y que rompan el esqueleto de fosfodiéster en un lugar específico, inactivando funcionalmente de ese modo el ARN diana. Para llevar a cabo esta ruptura, el ribozima no se altera por si mismo, y de este modo es capaz de reciclar y cortar otras moléculas, convirtiéndose en un enzima verdadero. La introducción de secuencias de ribozimas en los ARN antisentido les confiere una actividad rompedora de ARN, de esta manera incrementan la actividad de las construcciones. Se han identificado varias clases de ribozimas. Una clase de ribozimas derivan de varios ARN circulares que son capaces de romperse y replicarse por si mismos en plantas. Los ARN se replican solos (ARN virales) o con la ayuda de un virus auxiliar (ARN satélites). Ejemplos incluyen ARN del virus de la mancha solar de aguacate y ARN satélites del virus de la mancha anillada del tabaco, el virus del rayado transitorio de la alfalfa, el virus del moteado aterciopelado de tabaco, el virus del moteado de solanum nodiflorum, y el virus del moteado del trébol subterráneo. El diseño y utilización de los ribozimas específicos de ARN diana son descritos en Haseloff et al. Nature, 334:585-591 (1988). Otro procedimiento de supresión es la supresión en sentido. Se ha demostrado que la introducción de casetes de expresión en los cuales el ácido nucleico se configura en la orientación en sentido con respecto al promotor es un medio efectivo para bloquear la transcripción de genes diana. Para un ejemplo de la utilización de este procedimiento para modular la expresión de genes endógenos ver, Napoli et al., The Plant Cell 2:279-289 (1990), y las Patentes U.S. Nos. 5.034.323, 5.231.020 y 5.283.184. 30 35 Generalmente, alguna transcripcion de las secuencias introducidas ocurre cuando se desea la inhibición de expresión. El efecto puede ocurrir cuando la secuencia introducida contiene secuencia no codificante per se, sino solamente las secuencias intrón o no traducidas homólogas a las secuencias presentes en el transcrito primario. La secuencia introducida generalmente será sustancialmente idéntica a la secuencia endógena que se intenta reprimir. Esta mínima identidad será normalmente mayor a aproximadamente un 65%, pero una identidad mayor puede ejercer una represión de la expresión de las secuencias endógenas más efectiva. Se prefiere una identidad sustancialmente mayor de más de aproximadamente un 80%, aunque lo mejor sería de aproximadamente 95% hasta la identidad absoluta. Igual que con la regulación en antisentido, se debería aplicar el efecto a cualquier otra proteína dentro de una familia similar de genes que muestran homología u homología sustancial. 40 45 50 55 Para la supresión en sentido, la secuencia introducida en el casete de expresión, que necesita menos que la identidad absoluta, necesita también no ser de longitud completa, relacionada tanto con el producto primario de transcripción o el ARNm completamente procesado. Se puede preferir esto para evitar la producción concurrente de algunas plantas que sobreexpresadoras. Una mayor identidad en una secuencia de longitud más corta que la completa compensa a una secuencia menos idéntica pero más larga. Además, la secuencia introducida necesita no tener el mismo patrón intrón o exón, y la identidad de los segmentos no codificantes será igualmente efectiva. Normalmente se utiliza una secuencia de los intervalos de tamaño indicados más abajo para la regulación antisentido. Un experto en la materia reconocerá que utilizando tecnología basada en secuencias nucleótidas específicas (i.e. tecnología de supresión en sentido o en antisentido), se pueden suprimir familias de genes homólogos con un solo transcrito sentido o antisentido. Por ejemplo, si se diseña un transcrito sentido o antisentido para conseguir una secuencia que se conserve entre una familia de genes, entonces se pueden suprimir múltiples miembros de una familia de genes. A la inversa, si el objetivo es solamente suprimir un miembro de una familia de genes homólogos, entonces el transcrito sentido o antisentido se debería dirigir hacia secuencias con la mayor varianza entre los miembros de la familia. Otro medio para inhibir la función de LEC2 en una planta es mediante la creación de mutantes dominantes negativos. En esta aproximación, se introducen en una planta polipéptidos de LEC2 mutante, no funcionales, que conservan la capacidad de interactuar con subunidades de tipo natural. 60 Utilización de ácidos nucléicos para aumentar la expresión del gen 65 Se pueden utilizar también secuencias aisladas preparadas como se describe en el presente documento para preparar los casetes de expresión que aumentan o incrementan la expresión del gen LEC2 endógeno. Si se desea la sobreexpresión de un gen, se puede utilizar el gen deseado procedente de especies diferentes para reducir los efectos potenciales de supresión en sentido. La expresión aumentada de los polinucleótidos de LEC2 es útil, por ejemplo, para aumentar el contenido de proteína almacenada en los tejidos de la planta. Dichas técnicas pueden ser particularmente útiles para mejorar el valor nutricional de los tejidos de la planta. 9 ES 2 280 352 T3 5 10 Se puede utilizar cualquiera de los muchos medios bien conocidos en la materia para incrementar la actividad LEC2 en plantas. Es útil aumentar la expresión, por ejemplo, para inducir características embrionarias en plantas o en órganos de plantas. Cualquier órgano puede ser diana, tal como los órganos/estructuras vegetativos del vástago (i.e. hojas, tallos y tubérculos), raíces, flores y órganos/estructuras florales (i.e. brácteas, sépalos, pétalos, estambres, carpelos, anteras y óvulos), semilla (incluyendo embrión, endosperma y el recubrimiento de la semilla) y fruto. Alternativamente se pueden expresar uno o varios genes LEC2 de manera constitutiva (i.e. utilizando el promotor 35S de CaMV) Un experto en la materia reconocerá que los polipéptidos codificados por los genes de la invención, como otras proteínas, tienen diferentes dominios que hacen diferentes funciones. De este modo, las secuencias del gen no necesitan ser de longitud completa, con tal que se exprese el dominio funcional deseado de la proteína. Como se explica más abajo, los polipéptidos de LEC2 están relacionados con el producto del gen FUSCA3, que es un factor de transcripción. De este modo, sin estar vinculado a ningún mecanismo o teoría particular, LEC2 es probable que actúe como un modulador transcripcional. 15 Se cree que la actividad de unión del ADN, y, por lo tanto, la función de activación de la transcripción, de los polipéptidos de LEC2 está modulada por un motivo aminoácido conocido como el dominio B3, según se describe más abajo. Por ejemplo, las posiciones de aminoácidos del 165 al 277 representan el dominio B3 de SEC ID NO:2. De este modo, los polipéptidos de la invención conservarán a menudo estas secuencias. 20 Modificación de los genes endógenos LEC2 25 30 Se conocen bien los procedimientos para introducir mutaciones genéticas en los genes de la planta y seleccionar plantas con los rasgos deseados. Por ejemplo, se pueden tratar semillas u otro material de la planta con una sustancia química mutagénica, de acuerdo con las técnicas estándares. Dichas sustancias químicas incluyen, pero no se limitan a, lo siguiente: dietil sulfato, etilenimina, etilmetanosulfonato, N-nitroso-N-etilurea. Alternativamente, se pueden utilizar la radiación ionizante procedente de fuentes tales como, rayos X o rayos gamma. Se pueden diseñar también fácilmente cadenas de proteínas modificadas mediante varias técnicas de ADN recombinante bien conocidas por aquellos expertos en la materia y descritas, por ejemplo, en Sambrook et al., supra. Se puede utilizar también hidroxilamina para introducir mutaciones de una sola base en la región codificante del gen (Sikorski, et al., (1991). Meth. Enzymol. 194:302-318). Por ejemplo, las cadenas pueden cambiar de la secuencia que se encuentra de manera natural a nivel de estructura primaria por sustituciones, adiciones, deleciones de aminoácidos y similares. Se pueden utilizar estas modificaciones en un número de combinaciones para producir la cadena proteica final modificada. 35 40 45 50 55 Alternativamente, se puede utilizar la recombinación homóloga para inducir modificaciones del gen diana mediante dirigiendo específicamente el gen LEC1 in vivo (ver, generalmente, Grewal and Klar, Genetics 146:1221-1232 (1997) y Xu et al., Genes Dev.10:2411-2422 (1996)). Se ha demostrado la recombinación homóloga en plantas (Puchta et al., Experientia 50:277-284 (1994), Swoboda et al., EMBO J. 13:484-489 (1994); Offringa et al., Proc.Natl. Acad. Sci. USA 90:7346-7350 (1993); y Kempin et al., Nature 389:802-803 (1997)). Para aplicar la tecnología de recombinación homóloga a los genes de la invención, se preparan in vitro mutaciones en las porciones seleccionadas de unas secuencias del gen LEC2 (incluyendo las regiones 5’ upstream, 3’ downstream, e intragénicas) como las descritas en el presente documento y a continuación se introducen en la planta deseada utilizando técnicas estándar. Desde que se sabe que la eficiencia de la recombinación homóloga es dependiente de los vectores usados, el uso de vectores de direccionamiento de genes bicistrónicos como los descritos por Mountford et al., Proc. Natl. Acad. Sci. USA 91:4303-4307 (1994); y Vaulont et al., Transgenic Res. 4:247-255 (1995) son utilizados convenientemente para incrementar la eficiencia de selección de la expresión del gen LEC2 alterado en plantas transgénicas. El gen mutado interaccionará con el gen diana de tipo natural de manera que tendrá lugar la recombinación homóloga y la sustitución dirigida del gen de tipo natural ocurrirá en las células de la planta transgénica, teniendo como resultado la supresión de la actividad de LEC2. Alternativamente, se pueden utilizar oligonucleótidos compuestos de un fragmento continuo de residuos de ARN y ADN en una conformación de duplex con las caperuzas (“caps”) de horquillas dobles en los extremos. Se diseña la secuencia de ARN/ADN para que se alinee con la secuencia del gen LEC2 diana y para que contenga el cambio del nucleótido deseado. La introducción de un oligonucleótido quimérico en un plásmido de T-ADN extracromosómico resulta en la conversión eficiente y específica del gen LEC2 dirigida por moléculas quiméricas en un número pequeño de células vegetales transformadas. Se describe este procedimiento en Cole-Strauss et al., Science 273:1386-1389 (1996) y Yoon et al., Proc. Natl. Acad. Sci. USA 93:2071-2076 (1996). 60 65 En otras realizaciones, se pueden utilizar los promotores derivados de los genes LEC2 para conducir la expresión de genes heterólogos de una manera específica del embrión o específica de la semilla, de tal modo que los productos del gen deseado están en el embrión, semilla, o fruto. Se podrían utilizar genes estructurales adecuados para este propósito que incluyen genes codificantes de proteínas útiles para incrementar el valor nutricional de la semilla o del fruto. Ejemplos incluyen genes codificantes de enzimas que participan en la biosíntesis de antioxidantes tales como la vitamina A, vitamina C, vitamina E y melatonina. Otros genes codificantes de proteínas adecuadas son aquellos que participan en la modificación de ácidos grasos, o en la biosíntesis de lípidos, proteínas y carbohidratos. Todavía pueden ser otros genes aquellos que codifican proteínas que participan en la biosíntesis de auxina y del análogo de la auxina 10 ES 2 280 352 T3 para incrementar el tamaño del fruto, genes codificantes de compuestos farmacéuticamente útiles, y genes codificantes de productos de resistencia en plantas para combatir infecciones fúngicas u otras infecciones de la semilla. 5 10 15 Normalmente, se identifican a los promotores analizando las secuencias en 5’ de un clon genómico correspondiente a los genes específicos del embrión descritos en el presente documento. Se pueden utilizar características de las secuencias del promotor para identificar al promotor. Se han estudiado ampliamente las secuencias que controlan la expresión del gen eucariota. Por ejemplo, elementos de la secuencia del promotor incluyen la secuencia consenso de la caja TATA (TATAAT), que normalmente es de 20 a 30 pares de bases upstream del lugar de inicio de la transcripción. En la mayoría de los ejemplos se requiere la caja TATA para la iniciación precisa de la transcripción. En plantas, más lejos del upstream de la caja TATA, en posiciones de -80 a -100, normalmente hay un elemento del promotor con una serie de adeninas rodeando al trinucleótido G (o T) N G. J. Messing el al., en GENETIC ENGINEERING IN PLANTS, pag. 221-227 (Kosage, Meredith y Hollaender, eds. (1983)). Los expertos en la materia conocen varios procedimientos para identificar y caracterizar regiones del promotor del ADN genómico vegetal (ver, i.e., Jordano, et al., Plant Cell, 1:855-866 (1989); Bustos, et al., Plant Cell, 1:839-854 (1989); Green, et al., EMBO J. 7:4035-4044 (1988); Meier, et al., Plant Cell 3:309-316 (1991); y Zhang, et al., Plant Physiology 110:1069-1079 (1996). Preparación de vectores recombinantes 20 25 30 35 40 45 Para utilizar secuencias aisladas en las técnicas descritas más arriba, se preparan vectores de ADN recombinante adecuados para la transformación de las células vegetales. Son bien conocidas las técnicas para la transformación de una amplia variedad de especies de plantas superiores y están descritas en la literatura técnica y científica. Ver, por ejemplo, Weising et al., Ann. Rev. Genet. 22:421-477 (1988). Una secuencia de ADN que codifica el polipéptido deseado, por ejemplo una secuencia de ADNc que codifica una proteína de longitud completa, preferentemente se combinará con secuencias reguladoras de inicio de la transcripción y de la traducción que dirigirán la transcripción de la secuencia a partir del gen en el tejido de destino de la planta transformada. Por ejemplo, para la sobreexpresión, se puede usar un fragmento del promotor de la planta que dirigirá la expresión del gen en todos los tejidos de una planta regenerada. Estos promotores se denominan promotores “constitutivos” en el presente documento, y se activan bajo la mayoría de las condiciones ambientales y de los estados de desarrollo o diferenciación celular. Ejemplos de promotores incluyen la región de iniciación de la transcripción del virus (CaMV) 35S del mosaico de la coliflor, el promotor -1’ o -2’ derivado del ADN-T de Agrobacterium tumafaciens, y otras regiones de iniciación de la transcripción a partir de varios genes de plantas conocidas por aquellos que son expertos en la materia. Alternativamente, el promotor de la planta puede dirigir la expresión del polinucleótido en un tejido específico (promotores específicos del tejido) o si no pueden ser promotores inducibles bajo control ambiental más preciso. Ejemplos de promotores específicos de tejido bajo control de desarrollo incluyen el promotor que inicia la transcripción solamente en ciertos tejidos, tales como el fruto, las semillas o las flores. Como se observa arriba, los promotores de los genes LEC2 descritos en el presente documento son particularmente útiles para dirigir la expresión de los genes manera que un producto del gen deseado se localice en embriones y semillas. Otros promotores adecuados incluyen aquellos provenientes de genes que codifican proteínas de reserva o proteína de membrana de cuerpo lipídico, oleosina. Ejemplos de condiciones ambientales que pueden afectar a la transcripción por promotores inducibles incluyen condiciones anaeróbicas, temperatura elevada, o la presencia de luz. Si se desea una expresión correcta de polipéptido, se debería incluir una región de poliadenilación en el extremo 3’ de la región codificante. La región de poliadenilación puede derivar del gen natural, a partir de una variedad de genes de otra planta, o a partir de ADN-T. 50 El vector que comprende las secuencias de los genes (i.e. promotores o regiones codificantes) normalmente comprenderá un gen marcador que confiere un fenotipo seleccionable en células vegetales. Por ejemplo, el marcador puede codificar para resistencia a biocida, particularmente resistencia a antibiótico, tal como resistencia a kanamicina, G418, bleomicina, higromicina, o resistencia a herbicida, tal como resistencia al clorosulforon o Basta. 55 60 65 Las secuencias de ácidos nucleicos de LEC1 se expresan recombinantemente en células vegetales para potenciar e incrementar los niveles de polipéptidos de LEC2 endógenos. Alternativamente, se utilizan construcciones antisentido de LEC2 u otras (descritas arriba) para suprimir niveles de expresión de LEC. Se puede preparar una variedad de diferentes construcciones de expresión, tales como casetes de expresión y vectores adecuados para la transformación de las células vegetales. Las técnicas para transformar una amplia variedad de plantas superiores son bien conocidas y están descritas en la literatura técnica y científica Ver, p. e., Weising et al., Ann. Rev. Genet. 22:421-477 (1988). Una secuencia de ADN que codifica para un polipéptido de LEC2, i.e., una secuencia de ADNc que codifica una proteína de longitud completa, se puede combinar con secuencias reguladoras de la transcripción que actúan en cis (promotor) y que actúan en trans (potenciador) para dirigir el tiempo, tipo de tejido y niveles de transcripción en los tejidos deseados de la planta transformada. Se pueden utilizar también elementos de control de la traducción. La invención proporciona un casete de expresión que comprende un ácido nucleico de LEC2 unido operativamente a un promotor que, en una realización preferida, es capaz de conducir la transcripción de la secuencia codificante 11 ES 2 280 352 T3 5 de LEC2 en plantas. El promotor puede derivar, p.e., de fuentes vegetales o víricas. El promotor puede ser, p.e., constitutivamente activo, inducible o específico de tejido. Se pueden escoger diferentes promotores en la construcción de casetes de expresión recombinantes, de vectores, de transgénicos y se pueden emplear para dirigir diferencialmente la expresión del gen, p.e. en algunos o en todos los tejidos de la planta o animal. Normalmente, como se ha discutido arriba, los promotores deseados se identifican analizando las secuencias 5’ de un clon genómico correspondiente a genes específicos de embrión descritos en el presente documento. Promotores constitutivos 10 15 20 25 Se puede utilizar un fragmento de promotor para que dirija la expresión del ácido nucleico de LEC2 en todas las células o tejidos transformados, p.e. como aquellos de una planta regenerada. Dichos promotores se refieren en el presente documento como promotores “constitutivos” y son activos en la mayoría de las condiciones ambientales y estados del desarrollo o diferenciación celular. Se refiere como promotores “constitutivos” aquellos promotores que conducen la expresión continuamente bajo condiciones fisiológicas y son activos bajo la mayoría de condiciones ambientales y estados del desarrollo o diferenciación celular. Ejemplos de promotores “constitutivos” incluyen aquellos provenientes de virus que infectan plantas, tales como la región de iniciación de la transcripción del virus mosaico de la coliflor (CaMV) 35S (ver, p.e., Dagless (1997) Arch.Virol.142:183-191; el promotor 1’ ó 2’ derivado del ADNT de Agrobacterium tumafaciens (ver, p.e., Mentiste (1997) supra; O’Grady (1995) Plant Mol. Biol.29:99-108); el promotor del virus mosaico del tabaco; promotor del virus mosaico de escrofularia (ver, i.e., Maiti (1997) Transgenic Res. 6:143-156); promotores de actina, tal como el promotor del gen de la actina de Arabidopsis (ver, i.e., Huang (1997) Plant Mol. Biol. 1997 33:125-139); promotores del gene de (Adh) alcohol deshidrogenasa (ver, i.e., Millar (1996) Plant Mol. Biol.31:897-904); ACT11 de Arabidopsis (Huang et al. Plant Mol. Biol. 33:125-139 (1996)), Cat3 de Arabidopsis (GenBank Nº U43147, Zhong et al.,Mol. Gen. Genet. 251:196-203 (1996)), el gen que codifica para la proteína desaturasa portadora de estearoil-acilo de Brassica napus (Genbank Nº X74782, Solocombe et al., Plant Phisiol. 104:1167-1176 (1994)), GPc1 del maiz (GeuBaok Nº X15596, Martínez et al., J. Mol. Biol. 208:551-565 (1989)), Gpc2 del maiz (Genbank Nº U45855, Manjunath et al., Plant Mol. Biol. 33:97-112 (1997), otras regiones de inicio de la transcripción conocidas por los expertos en la materia. Ver también Holtorf (1995) “Comparación de diferentes promotores constitutivos e inducibles para la sobreexpresión de transgenes en Arabidopsis thaliana,” Plan Mol. Biol. 29:637-646. 30 Promotores Inducibles 35 Alternativamente, un promotor vegetal puede dirigir la expresión del ácido nucleico de LEC2 bajo la influencia de condiciones ambientales ó condiciones del desarrollo cambiantes. Ejemplos de condiciones ambientales que pueden afectar a la transcripción mediante promotores inducibles incluyen condiciones anaeróbicas, temperaturas elevadas, sequía, o la presencia de luz. Dichos promotores se describen en el presente documento como promotores “inducibles”. Por ejemplo, la invención incorpora el promotor inducible por sequía proveniente del maíz, (Busk (1997) supra); el promotor inducible por el frío,la sequía, y alta concentración de sal proveniente de la patata (Kirch (1997) Plant Mol. Biol. 33:897-909). 40 45 50 55 Alternativamente, promotores vegetales que son inducibles bajo exposición a hormonas vegetales, tales como auxinas, que se utilizan para expresar los ácidos nucleicos. Por ejemplo, la invención puede usar el fragmento de promotor de elementos E1 sensible a auxina (AuxREs) en la haba de soja (Glycine max L) (Liu (1997) Plant Physiol. 115:397-407); el promotor GST6 de Arabidopsis que responde a auxina (también responde al ácido salicílico y al peróxido de hidrogeno) (Chen (1996) Plant J. 10:955-966); el promotor parC por auxina proveniente del tabaco (Sakai (1996) 37:906-913); un elemento que responde a biotina vegetal (Streit (1997) Mol. Plant Microte Interact. 10:933937); y el promotor sensible al ácido abscísico de la hormona del stress (Sheen (1996) Science 274:1900-1902). Los promotores vegetales que son inducibles por la exposición a reactivos químicos que se pueden aplicar a la planta, tales como herbicidas o antibióticos, se utilizan también para expresar los ácidos nucleicos de la invención. Por ejemplo, se puede utilizar el promotor In2-2 del maíz, activado por un herbicida Safener de bencenosulfonamida (De Veylder (1997) Plant Cell Physiol.38:568-577), la aplicación de diferentes herbicidas safener induce diferentes patrones de expresión del gen, incluyendo la expresión en la raíz, hidátodos, y el meristemo apical del tallo. La secuencia codificante de LEC2 puede estar también bajo el control de, p.e., un promotor inducible por tetraciclina, p.e. como el descrito con plantas de tabaco transgénicas que contienen el gen de la arginina descarboxilasa de la Avena sativa L. (avena) (Masgrau (1997) Plant J. 11:465-473); o, un elemento que responde al ácido salicílico (Stange (1997) Plant J. 11:1315-1324). Promotores específicos de tejido 60 65 Alternativamente, el promotor vegetal puede dirigir la expresión del polinucleotido en un tejido específico (promotores específicos de tejido). Los promotores específicos de tejido son elementos de control transcripcional que son activos solamente en células o tejidos particulares en momentos específicos durante el desarrollo de la planta, como son los tejidos vegetativos o los tejidos reproductivos. Los promotores provenientes de los genes LEC2 son particularmente útiles para la dirección específica de tejido de expresión del gen de manera de que se genere el producto del gen deseado solamente o preferentemente en embriones o semillas, como se describe más abajo. 12 ES 2 280 352 T3 5 Ejemplos de promotores específicos de tejido bajo control del desarrollo incluyen promotores que inician la transcripción solamente (o principalmente solamente) en ciertos tejidos, tales como los tejidos vegetativos, p.e., raíces ú hojas, o tejidos reproductivos, tales como el fruto, óvulos, semillas, polen, pistilos, flores o cualquier tejido embrionario. Los promotores específicos del tejido reproductivo podrían ser, p.e. específicos del óvulo, específicos del embrión, específicos del endoesperma, específicos del integumento, específicos de la semilla y de la envoltura de la semilla, específicos del polen, específicos del pétalo, específicos del sépalo o alguna combinación de los mismos. 10 Los promotores específicos de semilla adecuados derivan de los siguientes genes: MAC1 del maíz, (Sheridan (1996) Genetics 142:1009-1020); Cat3 del maíz (GenBank Nº. L05934, Abler (1993) Plant Mol. Biol. 22:101311038); viviparous-1 de Arabidopsis (Genbank Nº. U93215); atmycl de Arabidopsis (Urao (1996) Plant Mol. Biol. 32:571-57; Conceicao (1994) Plant 5:493-505); napA de Brassica napus (GenBank Nº J02798, Josefsson (1987) JBL 26:12196-1301); la familia del gen napina de Brassica napus (Sjodahl (1995) Planta 197:264-271). 15 Se puede utilizar también el gen BEL1 específico del óvulo descrito en Reiser (1995) Cell 83:735-742, GenBank Nº U39944. Ver también Ray (1994) Proc. Natl. Acad. Sci, USA 91:5761-5765. El promotor FIE1 específico del huevo y de la célula central es útil también como promotor específico del tejido reproductivo. 20 25 30 35 40 45 Se utilizan también promotores específicos de sépalos y pétalos para expresar ácidos nucleicos de LEC2 de una manera específica del tejido reproductivo. Por ejemplo, el gen homeótico APETALA1 (AP1) de la flor de Arabidopsis codifica un presunto factor de la transcripción que se expresa en el primordio joven de la flor, y más tarde se localiza en sépalos y pétalos (ver, p. e., Gustafson-Brown (1994) Cell 76:131-143; Mandel (1992) Nature 360:273-277). Es útil también un promotor, para AP2, relacionado con un gen homeótico floral que es necesario para el desarrollo normal de sépalos y pétalos en los verticilos florales (ver, p. e., Drews (1991) Cell 65:991-1002; Bowman (1991) Plant Cell 3:749-758). Otro promotor útil es el que controla la expresión del gen (ufo) de los órganos florales inusuales de Arabidopsis, cuya expresión se limita a la unión entre los primordios de sépalos y pétalos (Bossinger (1996) Development 122:1093-1102). Se ha identificado un promotor específico del polen de maíz en el maíz (Guerrero (1990) Mol. Gen. Genet. 224:161168). Se describen otros genes específicamente expresados en el polen, i.e. por Wakely (1998) Plant Mol. Biol. 37:187192; Ficker (1998) Mol. Gen. Genet. 257:132-142; Kulikauskas (1997) Plant Mol. Biol. 34:809-814; Treacy (1997) Plant Mol. Biol. 34:603-611. Otros promotores adecuados incluyen aquellos provenientes de genes que codifican proteínas embrionarias de reserva. Por ejemplo, el gen que codifica la proteína de reserva 2S de Brassica napus, Dasgupta (1993) Gene 133:301302; la familia 2s de genes de proteína de reserva de la semilla en Arabidopsis; el gen que codifica la oleosina de 20kD en Brassica napus, GenBank Nº M63985; los genes que codifican la oleosina A, Genbank Nº U09118, y, oleosina B, Genbank Nº U09119, de la haba de la soja; el gen que codifica la oleosina de Arabidopsis, Genbank Nº Z17657; el gen que codifica la oleosina 18kD del maíz, GenBank Nº J05212 Lee (1994) Plant Mol. Biol. 26:1981-1987; y, se puede usar el gen que codifica la proteína de bajo peso molecular rica en azufre de haba de soja, Choi (1995) Mol Gen, Genet. 246:266-268. El promotor E8 específico de tejido proveniente del tomate es particularmente útil para dirigir la expresión del gen de manera que se localice el producto deseado en los frutos. Se puede utilizar un promotor del tomate activo durante la maduración del fruto, senescencia y abscisión de las hojas y, en menor extensión, de las flores (Blume (1997) Plant J. 12:731-746). Otros promotores ejemplificativos incluyen el promotor específico del pistilo en el gen SK2 de la patata (Solanum tuberosum L), que codifica una endoquinasa básica específica del pistilo (Ficker (1997) Plant Mol. Biol. 35:425-431); el gen Blec4 del guisante (Pisum sativum cv. Alaska), activo en el tejido epidérmico de los ápices vegetativos y del vástago floral de la alfalfa transgénica. Esto hace que sea una herramienta útil para dirigir la expresión de genes foráneos en la capa epidérmica de los vástagos en crecimiento activo. 50 55 60 65 Se pueden usar también una variedad de promotores específicamente activos en tejidos vegetativos, como hojas, tallos, raíces y tubérculos, para expresar los ácidos nucleicos de LEC2 de la invención. Por ejemplo, promotores que controlan la patatina, la principal proteína de reserva del tubérculo de patata, ver, p.e. Kim (1994) Plant Mol. Biol.26:603-615; Martin (1997) Plant J. 11:53-62. Se puede utilizar también el promotor ORF13 de Agrobacterium rhizogenes que muestra una actividad alta en raíces (Hansen (1997) Mol. Gen Genet. 254:337-343. Otros promotores específicos del tejido vegetativo útiles incluyen: el promotor de tarina del gen que codifica una globulina proveniente de una familia de proteínas de taro corn superior (Colocasia esculenta L. Schott), tarina (Becerra (1995) Plant Mol. Biol. 28:137-144); el promotor de curculina activo durante el desarrollo de taro corn (de Castro (1992) Plant Cell 4:1549-1559) y el promotor para el gen TobRB7 específico de la raíz del tabaco, cuya expresión se localiza en el meristemo de la raíz y en las regiones inmaduras del cilindro central (Yamamoto (1991) Plant Cell 3:371-382). Se pueden utilizar promotores específicos de la hoja, como los promotores de la ribulosa difosfato carboxilasa (RBCS). Por ejemplo, los genes del tomate RBCS1, RBCS2 y RBCS2A se expresan en hojas y en plántulas que crecen expuestas a la luz, solamente RBCS1 y RBCS2 se expresan en el desarrollo de los frutos del tomate (Meier (1997) FEBS Lett. 415:91-95). Se pueden usar unos promotores de la ribulosa difosfato carboxilada expresada casi exclusivamente a altos niveles en las células de mesófilo en los bordes de la hoja y vainas foliares, descritos por Matsuoka (1994) Plant J. 6:311-319. Otro promotor específico de la hoja es el promotor del gen de la proteína de unión de la clorofila a/b de cosecha bajo condiciones de luz, ver, p.e. Shiina (1997) Plant Physiol. 115:477-483; Casal 13 ES 2 280 352 T3 5 10 15 (1998) Plant Physiol. 116:1533-1538. El promotor del gen relacionado con myd de Arabidopsis thaliana (Atmyb5) es específico de la hoja, descrito por Li (1996) FEBS Lett. 379:117-121. El promotor de Atmyb5 se expresa en tricomas de la hoja en desarrollo, en estipulas, y en células epidérmicas en los márgenes de hojas de roseta jóvenes y caulinares, y en semillas inmaduras. El ARNm de Atmyb5 aparece entre la fertilización y la 16ª etapa celular del desarrollo embrionario y persiste más allá de la etapa del duramen. Se puede utilizar también un promotor de la hoja identificado en el maíz por Busk (1997) Plant J. 11:1285-1295. Otra clase útil de promotores específicos de tejido vegetativo son los promotores meristemáticos (punta de la raíz y ápice del vástago). Por ejemplo; Se pueden utilizar los promotores de “SHOOTMERISTEMLESS” y “SCARECROW”, que son activos en el desarrollo del vástago y en los meristemos apicales de la raíz, descritos por Di Laurenzio (1996) Cell 86:423-433; y, Long (1996) Nature 379:66-69. Otro promotor útil es aquel que controla la expresión del gen HMG2 de 3-hidroxi-3-metilglutaril coenzimaA reductasa, cuya expresión se limita a los tejidos meristemáticos y florales (zona secretora del estigma, granos de polen maduros, tejido vascular del gineceo, y óvulos fertilizados) (ver, p.e., Enjuto (1995) Plant Cell. 7:517-527). Son útiles también los genes relacionados con kn1 del maíz y otras especies que muestran expresión específica del meristemo, ver, p.e., Granger (1996) Plant Mol. Biol. 31:373-378; Kerstetter (1994) Plant Cell 6:1877-1887; Hake (1995) Philos Trans. R. Soc. Lond. B. Biol. Sci.350:4551. Por ejemplo, el promotor KNAT1 de Arabidopsis thaliana. En el ápice del vástago, se localiza el transcrito de KNAT1 ante todo en el meristemo apical de la raíz; la expresión de KNAT1 disminuye durante la transición floral y se limita a al córtex del tallo inflorescente (ver, p.e., Lincoln (1994) Plant Cell 6:1859-1876). 20 Un experto reconocerá que un promotor específico de tejido puede dirigir la expresión de las secuencias unidas operativamente tanto en tejidos diferentes del tejido diana. De este modo, un promotor específico de tejido como los utilizados en el presente documento es aquel que dirige la expresión preferentemente en el tejido diana, pero podría dirigir también alguna expresión en otros tejidos. 25 30 35 40 45 50 55 60 65 En otra realización, un ácido nucleico LEC2 se expresa a través de un elemento transponible. Esto tiene en cuenta la expresión constitutiva, todavía periódica e infrecuente del polipéptido constitutivamente activo. Esta invención proporciona también el uso de promotores específicos de tejido derivados de virus que pueden incluir, p.e., el promotor subgenómico del tobamovirus (Kumagai (1995) Proc. Natl. Acad. Sci. USA 92:1679-1683; el virus baciliforme del tungro del arroz (RTBV), que se replica solamente en las células del floema en plantas de arroz infectadas, con su promotor que dirige la expresión fuerte del gen reportero específico del floema; el promotor del virus mosaico del nervio de mandioca (CVMV), con la actividad más alta en elementos vasculares, en células mesofílicas de la hoja y en las puntas de las raíces (Verdaguer (1996) Plant Moll. Biol. 31:1129-1139). Producción de plantas transgénicas Se pueden introducir construcciones de ADN de la invención en el genoma del hospedador de la planta deseada mediante una variedad de técnicas convencionales. Por ejemplo, se pueden introducir la construcción de ADN directamente en el ADN genómico de la célula de la planta utilizando técnicas tales como la electroporación y la microinyección de protoplastos de la célula vegetal, o se pueden introducir las construcciones de ADN directamente en el tejido vegetal utilizando procedimientos balísticos, como es el bombardeo de partículas de ADN. Alternativamente, las construcciones de ADN se podrían combinar con regiones flanqueantes adecuadas de ADN-T e introducir en un vector convencional del hospedador de Agrobacterium tumefaciens. Las funciones virulentas del hospedador de Agrobacterium tumefaciens dirigirán la inserción de la construcción y del marcador contiguo en el ADN de la célula vegetal, cuando la bacteria infecta a la célula. Las técnicas de microinyección son conocidas en la materia y están bien descritas en la literatura científica y de patentes. Se describe la introducción de construcciones de ADN mediante la precipitación con polietilenglicol en Paszkowski et al. Embo J. 3:2717-2722 (1984). Se describen las técnicas de electroporación en Fromm et al., Proc. Natl. Acad. Sci. USA 82:5824 (1985). Se describen las técnicas de transformación balística en Klein et al., Nature 327:70-73 (1987). Están bien descritas en la literatura científica técnicas de transformación mediadas por Agrobacterium tumefaciens, incluyendo el desarme y utilización de vectores binarios en la literatura científica. Ver, por ejemplo Horsch et al., Science 233:496-498 (1984), y Fraley et al., Proc. Natl. Acad. Sci. USA 80:4803 (1983). Se pueden cultivar las células vegetales trasformadas mediante cualquiera de las técnicas de transformación anteriores para regenerar una planta entera que posea el genotipo transformado y de este modo el fenotipo deseado, como es la falta de semillas. Dichas técnicas de regeneración dependen de la manipulación de ciertas fitohormonas en un medio de cultivo de crecimiento de tejido, normalmente dependiendo de un marcador biocida y/o herbicida que ha sido introducido junto con las secuencias nucleótidas deseadas. Se describe la regeneración de plantas a partir de protoplastos cultivados en Evans el al., Protoplasts Isolation and Cultura, Handbook of Plant Cell Cultura, pp. 124176, McMillilan Publishing Company, New Cork, 1983; y Binding, Regeneration of Plants, Plant Protoplasts, pp. 2173, CRC Press, Boca Raton, 1985. Se puede obtener también la regeneración a partir del callo de la planta, explantes, órganos, o partes de estos. Generalmente dichas técnicas se describen en Klee et al., Ann. Rev.of Plant Phys. 38:467486 (1987). 14 ES 2 280 352 T3 5 10 15 20 25 30 35 40 45 50 55 Se pueden utilizar los ácidos nucleicos para conferir rasgos deseados esencialmente a cualquier planta. De este modo, la invención ha utilizado un amplio número de especies, incluyendo especies del género Asparagus, Atropa, Avena, Brassica, Citrus, Citrullus, Capsicum, Cucumis, Cucurbita, Daucus, Fragaria, Glycine, Gossypium, Helianthus, Heterocallis, Hordeum, Hyosoyamus, Lactuca, Linum, Lolium, Lycopersicon, Malus, Manihot, Majorana, Medicago, Nicotiana, Oryza, Panieum, Pannesetum, Persea, Pisum, Pyrus, Prunas, Raphanus, Secale, Senecio, Sinapis, Solanum, Sorghum, Trigonella, Triticum, Vitis, Vigna, y, Zea. Los genes LEC2 son particularmente útiles en la producción de plantas transgénicas del género Brassica. Ejemplos incluyen brécol, coliflor, coles de Bruselas, colza, y similares. Uso y Expresión Recombinante de LEC2 en Combinación con otros Genes Los ácidos nucleicos de LEC2 se pueden expresar junto con otros genes estructurales o reguladores para conseguir un efecto deseado. Se puede transformar una célula o planta, tal como una célula transformada o una planta transgénica, manipulada o reproducida para coexpresar tanto el nucleótido de LEC2 y/o el polipéptido de LEC2, como otro gen o producto génico. Alternativamente, se pueden coexpresar dos o más ácidos nucleicos LEC2 juntos en la misma planta o célula. Los ácidos nucleicos de LEC2 cuando se expresan en el tejido reproductivo o vegetativo de la planta, pueden inducir la morfogénesis del embrión ectópico. De esta manera, en una realización, un ácido nucleico de LEC2 se expresa en una conformación sentido en una planta transgénica para inducir la expresión de estructuras similares al embrión ectópico. Por ejemplo, hojas distintas de tipo natural, órganos de plantas con estructuras similares al embrión ectópico no poseen ni crecen tricomas en ellos. Morfológicamente, estas estructuras similares a las hojas se asemejan más a cotiledones embrionarios que a hojas. El LEC2 se puede coexpresar con un gen o ácido nucleico que incremente la masa del tejido reproductivo, p. e., aumenta el tamaño del fruto, la masa de semilla, la proteína de semilla o los aceites de semilla. Por ejemplo, la coexpresión de ácido nucleico antisentido a genes ADC, tales como los genes AP2 y RAP2 de Arabidopsis, aumentaría drásticamente la masa de la semilla, la proteína de la semilla y los aceites de la semilla; ver, p. e. Jofuku, et al., WO 98/07842; Okamuro (1997) Proc. Natl. Acad. Sci. USA 94:7076-7081; Okamuro (1997) Plant Cell 9:37-47; Jofuku (1994) Plant Cell 6:1211-1225. De este modo, la coexpresión de un ácido nucleico de LEC2 para inducir la expresión ectópica de células y tejidos embrionarios, junto con otro ácido nucleico y/o proteína vegetales, tales como el ácido nucleico antisentido AP2 que aumenta la masa de la semilla, genera una célula, tejido, o planta (p. e. una planta transgénica) con la masa de fruto y de semilla incrementadas, mayores rendimientos de las proteínas de reserva embrionarias, y similares. Alternativamente, la coexpresión de ácidos nucleicos antisentido a LEC2 y LEC1 es útil para inhibir la semilla en el fruto. Los ácidos nucleicos de LEC2 se pueden expresar en células vegetales reproductivas o vegetativas y en tejidos que no tienen la capacidad de producir genes ADC funcionales, como son los genes AP2 y RAP2. El ácido nucleico de LEC2 se puede expresar en una planta transgénica ADC “knockout”. Alternativamente, el ácido nucleico se puede expresar en una célula, tejido o planta que se exprese un ácido nucleico ADC mutante o producto del gen. La expresión del ácido nucleico de LEC2 en cualquiera de estos modelos de ADC no funcionales producirá también una célula, tejido o planta con las masas de fruto y de semilla incrementadas, mayores rendimientos de proteínas de reserva embrionarias, y similares. Como se discute arriba, los ácidos nucleicos de LEC2 son útiles también para la producción de plantas apomícticas. Por ejemplo, los ácidos nucleicos de LEC2 se pueden expresar en combinación con los ácidos nucleicos de LEC1 como los descritos por ejemplo por Lotan, et al., Cell 93:1195-1205 (1998) y en USSN 09/026.221. Alternativamente, los ácidos nucleicos FIE (p.e., PCT WO99/09676 y Ohad, N. et al., (1999) Plant Cell 11(3):404-16) y/o FUSCA3 (Luerben, H., et. al., Plant J.15:755-764) se pueden combinar también con los ácidos nucleicos de LEC1 y/o de LEC2. En estas realizaciones, se usan las construcciones que proporcionan la expresión de un ácido nucleico de LEC2 en combinación con las construcciones que llevan a la expresión de LEC1 o FUS3 y/o la inhibición de la expresión de FIE. Se discuten más abajo los medios para dirigir la expresión a los tejidos deseados, como son los gametofitos femeninos o los óvulos. Fusiones del Gen LEC2 Las secuencias de ácidos nucleicos de LEC2 incluyen también fusiones entre dos o más genes LEC2. Se pueden fusionar diferentes dominios de diferentes genes. Las fusiones de los genes LEC2 se pueden unir directamente o se pueden juntar mediante aminoácidos adicionales que unen las dos o más partes de la fusión. 60 65 Se pueden generar fusiones de genes mediante técnicas básicas de ADN recombinante como las descritas más abajo. La selección de fusiones de genes dependerá del fenotipo deseado producido por la fusión de los genes. Por ejemplo, si se desean fenotipos asociados a un dominio de una proteína de LEC2 con los fenotipos asociados a un dominio diferente de una segunda proteína de LEC2, se crearía la fusión del primer dominio de la proteína de LEC2 con el segundo dominio de LEC2. La fusión se puede testar posteriormente in vitro o in vivo para los fenotipos deseados. 15 ES 2 280 352 T3 El experto en la materia reconocerá que el casete de expresión se incorpora de manera estable a las plantas transgénicas y se confirma que es operativo, se puede introducir en otras plantas por cruzamiento sexual. Se puede utilizar cualquiera de las técnicas estándar de reproducción, dependiendo de las especies que se vayan a cruzar. 5 Ejemplos Los siguientes ejemplos sólo sirven para ilustrar, pero no para limitar la invención reivindicada. Ejemplo 1 10 Este ejemplo muestra la identificación y aislamiento de los genes LEC2. 15 20 25 El alelo lec2-1 provenía de una población de plantas de ecotipo Ws-0 sometidas a mutación con ADN-T (Feldmann, K.A., et al.,(1987). Mol.Gen. Genet. 208:1-9). Sin embargo, el gen de LEC2 no está etiquetado en esta línea de plantas. Se obtuvieron otros alelos mutantes no etiquetados de los genes LEC2 en los ecotipos Ws-4, DLM1, CUC3, y CPT9, del Institut National de la Recherche Agronomique. Se hizo el mapa de LEC2 genéticamente en base a su posición en el cromosoma 1. Se identificaron recombinantes con DISTORTED1 (DIS1) y DIS2 en el ecotipo Ler, que se situaron en el mapa aproximadamente a 17 cm al norte y 5 cM al sur de LEC2, respectivamente. La Figura 1 muestra los detalles de la estrategia del mapeo, incluyendo el número de recombinantes entre LEC2 y de cada uno de los marcadores. Se hizo el mapa de LEC2 en un clon F1A10 del cromosoma artificial bacteriano (BAC). El mapeado de las terminaciones de clones de cósmidos preparados a partir de este BAC estrechó la región que contenía LEC2 a una región de 23 kb. Se preparó una librería de cósmidos a partir del ADN F1A10 de BAC. Se hibridó un fragmento BamHI de 9 kb derivado del cósmido 80 de esta librería en esta región de 23 kb en una transferencia en gel que contenía ADNs genómicos de los tipos naturales Ws-0 y Ws-4 y los mutantes lec2, p.e. lec2-1, DLM1, CUC3 y CPT9. La transferencia identificó un fragmento de restricción EcoRI de 3 kb en plantas de tipo natural que era polimórfico en los genomas de lec2-1 y de CPT9, mostrando que LEC2 podía comprender por lo menos una parte de este fragmento. 30 35 40 Para confirmar esta hipótesis, la secuenciación nucleótida del fragmento de EcoRI de 3 kb y la secuenciación parcial del fragmento BamHI de 9 kb revelaron que el fragmento de EcoRI de 3 kb contiene la región 5’ de un gen como predijo el programa de análisis Genscan (http://CCR-081.mit.edu/GENSCAN.html). El análisis de esta secuencia utilizando las investigaciones Blast de bases de datos públicos (http//www.ncbi.nlm.nih.gov/BLAST/) reveló una secuencia parcial de BACF3H9 no ordenada ni anotada (ver Figura 1). Una región contigua de la secuencia nucleótida proveniente de este BAC correspondía a parte del fragmento de EcoRI de 3 kb y proporcionó la terminación 3’ del gen. El análisis Blast de la secuencia del gen LEC2 (SEC ID NO:1) mostró homología con el dominio B3 de FUS3 y de ABI3. De este modo, LEC2 es un regulador transcripcional del desarrollo embrionario. La secuencia de aminoácidos del polipéptido codificado por LEC2 es aproximadamente un 56% idéntica a FUS3 sobre 103 aminoácidos. Las identidades se encuentran de un lado al otro del dominio de B3 de FUS3 (Número de acceso a Genbank AF016265.1). La mayor identidad encontrada en la investigación BLAST fue de un 60% de identidad sobre 95 aminoácidos con el dominio B3 de la proteína de la zanahoria C-ABI-3 (Número de acceso a Genbank AB005558.1). 45 Los residuos de aminoácidos del 165 al 277 de SEC ID NO:2 representan el dominio B3 de LEC2. El dominio B3 de LEC2 está codificada por las posiciones 3682-3690, 3848-3922, 4817-4917, 5101-5147, 5257-5330 y 5679-5681 de SEC ID NO:1. 50 Un análisis molecular de la secuencia genómica (SEC ID NO:1) revela muchas características. Por ejemplo, el codón de inicio ATG está en la posición 3196 y el codón de parada está en la posición 5973. El final del ADNc está en la posición 6166. Hay 6 exones en el gen. Las posiciones de los exones en SEC ID NO:1 son 3196-3690, 3848-3922, 4817-4917, 5101-5147, 5257-5330, y 5679-5975. 55 Ejemplo 2 Este ejemplo muestra la secuenciación de nucleótidos de dos alelos mutantes de LEC2. Ambos mutantes tienen deleciones en la región de la secuencia codificante que codifica el dominio B3. 60 65 La secuenciación nucleótida del alelo LEC2 del CUC3 mutante reveló una deleción de 11 pares de bases comparada con la secuencia de LEC2 de tipo natural. Esta deleción termina en una alteración del marco de lectura y de este modo del fenotipo mutante. La secuenciación nucleótida de los alelos LEC2 del DLM1 mutante reveló una deleción de 33 pares de bases comparada con la secuencia LEC2 de tipo natural. Esta deleción es la causa del fenotipo mutante. 16 ES 2 280 352 T3 REIVINDICACIONES 5 1. Casete de expresión que comprende un promotor unido operativamente a una secuencia polinucleótida heteróloga, o una complementaria a la misma, que codifica un polipéptido LEC2 que muestra por lo menos un 65% de identidad de secuencia con SEC ID NO:2, donde el polinucleótido induce morfogénesis embrionaria ectópica, o resulta en plantas con semillas no viables cuando se expresa en una planta. 2. Casete de expresión de la reivindicación 1, donde el polipéptido LEC2 es SEC ID NO:2. 10 3. Casete de expresión de la reivindicación 4, donde la secuencia polinucleótida es SEC ID NO:1. 4. Casete de expresión de la reivindicación 1, donde el promotor es un promotor constitutivo. 15 5. Casete de expresión de la reivindicación 1, donde el promotor proviene de un gen LEC2. 6. Casete de expresión de la reivindicación 1, donde el promotor comprende un polinucleótido por lo menos en un 70% idéntico a SEC ID NO:3. 20 25 7. Casete de expresión de la reivindicación 1, donde la secuencia polinucleótida se une al promotor en una orientación antisentido. 8. Casete de expresión para la expresión de un polinucleótido heterólogo en una célula vegetal, donde el casete de expresión comprende un promotor por lo menos un 70% idéntico a SEC ID NO:3 y donde el promotor se une operativamente a un polinucleótido heterólogo. 9. Casete de expresión de la reivindicación 4 o de la reivindicación 8, donde el promotor comprende SEC ID NO:3. 30 10. Casete de expresión de la reivindicación 4 o reivindicación 8, en el que el promotor además comprende un polinucleótido por lo menos un 70% idéntico a SEC ID NO:4. 11. Casete de expresión de la reivindicación 10, donde el promotor comprende SEC ID NO:4. 35 12. Célula hospedadora que comprende un casete de expresión de acuerdo con cualquiera de las reivindicaciones 1 a 11. 13. Célula hospedadora de la reivindicación 12, donde la célula es una célula vegetal, una célula bacteriana, una célula de levadura, una célula fúngica o una célula animal. 40 14. Polipéptido aislado que comprende una secuencia aminoácido. (a) por lo menos un 65% idéntica a SEC ID NO:2, donde el polipéptido induce la morfogénesis embrionaria ectópica o inhibe la producción de semillas en una planta. 45 15. Anticuerpo capaz de unirse al polipéptido aislado de la reivindicación 14. 16. Procedimiento para introducir un ácido nucleico aislado en una célula hospedadora que comprende: (a) proporcionar un casete de expresión de acuerdo con la reivindicación 1; y 50 55 (b) poner en contacto el ácido nucleico con la célula hospedadora bajo condiciones que permiten la inserción del ácido nucleico en la célula hospedadora. 17. Procedimiento para modular la transcripción del gen LEC2, comprendiendo el procedimiento, introducir en una célula hospedadora un casete de expresión que comprende un promotor unido operativamente a un polinucleótido heterólogo de LEC2, el polinucleótido heterólogo de LEC2 codificando un polipéptido LEC2 por lo menos un 65% idéntico a SEC ID NO:2; y detectar una célula con transcripción modulada. 18. Procedimiento de la reivindicación 17, donde el polinucleótido codifica SEC ID NO:2. 60 19. Procedimiento de la reivindicación 18, donde el polinucleótido es SEC ID NO:1. 20. Procedimiento de la reivindicación 17, donde el casete de expresión es introducido mediante Agrobacterium. 65 21. Procedimiento de la reivindicación 17, donde el casete de expresión es introducido mediante cruzamiento sexual. 17 ES 2 280 352 T3 22. Procedimiento de la reivindicación 17, donde la modulación de la transcripción resulta en la inducción de características embrionarias en una planta. 5 23. Procedimiento de la reivindicación 17, donde la modulación de la transcripción resulta en la inducción del desarrollo de la semilla. 24. Procedimiento de la reivindicación 17, donde la célula hospedadora es una planta. 25. Procedimiento de la reivindicación 24, donde una planta se regenera a partir de una célula vegetal. 10 26. Procedimiento para detectar un ácido nucleico aislada en una muestra, que comprende (a) proporcionar una molécula de ácido nucleico aislada que consiste en una secuencia nucleótida que codifica un polipéptido que muestra por lo menos un 65% de identidad de secuencia con SEC ID NO:2, 15 (b) poner en contacto la molécula de ácido nucleico con una muestra bajo condiciones que permiten una comparación de la secuencia de la molécula de ácido nucleico aislada con la secuencia de ADN en la muestra; y (c) analizar el resultado de la comparación. 20 27. Procedimiento de la reivindicación 26, donde la molécula aislada de ácido nucleico y la muestra se ponen en contacto bajo condiciones que permiten la formación de un dúplex entre las secuencias de ácidos nucleicos complementarias. 25 28. Célula vegetal transgénica o planta transgénica que comprende un casete de expresión de acuerdo con cualquiera de las reivindicaciones de 1 a 11. 29. Planta que se ha regenerado a partir de una célula vegetal de acuerdo con la reivindicación 28. 30 35 40 45 50 55 60 65 18 ES 2 280 352 T3 19 ES 2 280 352 T3 LISTA DE SECUENCIAS SEC ID NO:1 secuencia genómica de LEC2 5 10 15 20 25 30 35 40 45 50 55 60 65 1 ES 2 280 352 T3 5 10 15 20 25 30 35 40 45 50 55 60 65 2 ES 2 280 352 T3 5 10 15 20 25 SEC ID NO:2 polipéptido de LEC2 30 35 40 45 50 SEC ID NO:3 promotor 5’ de LEC2 55 60 65 3 ES 2 280 352 T3 5 10 15 20 25 30 35 40 45 50 55 60 65 4 ES 2 280 352 T3 5 10 15 20 25 30 35 40 45 50 55 60 65 5 ES 2 280 352 T3 5 10 15 SEC ID NO:4 promotor 3’ de LEC2 20 25 30 35 40 45 50 55 60 SEC ID NO:5 ADNc de LEC2 65 6 ES 2 280 352 T3 5 10 15 20 25 30 35 40 45 50 55 SEC ID NO:6 gen LEC2 del sitio de inicio de la traducción al sitio de la poliadenilación 60 65 7 ES 2 280 352 T3 5 10 15 20 25 30 35 40 45 50 55 60 65 8 ES 2 280 352 T3 5 10 15 20 25 30 35 40 45 50 55 60 LISTADO DE SECUENCIAS 65 <110> Harada, John Kwong, Linda Matasudaira Lee, Kelly The Regents of the University of California 9 ES 2 280 352 T3 5 10 15 20 25 30 35 40 45 50 55 <120> Genes LEAFY COTYLEDON2 y sus usos <130> 023070-111600PC <140> WO PCT/US01/08219 <141> 14-03-2001 <150> US 09/527.058 <151> 16-03-2000 <160> 14 <170> PatentIn Ver. 2.1 <210> 1 <211> 6766 <212> ADN <213> Arabidopsis sp. <220> <223> Secuencia genómica de LEAFY COTYLEDON2 (LEC2) <220> <221> misc_feature <222> (3196)..(3198) <223> codón de inicio ATG <220> <221> exon <222> (3196)..(3690) <220> <221> exon <222> (3848)..(3922) <220> <221> exon <222> (4817)..(4917) <220> <221> exon <222> (5101)..(5147) <220> <221> exon <222> (5257)..(5330) <220> <221> exon <222> (5679)..(5975) <220> <221> misc_feature <222> (6166) <223> final de ADNc 60 65 10 ES 2 280 352 T3 <400> 1 5 10 15 20 25 30 35 40 45 50 55 60 65 11 ES 2 280 352 T3 5 10 15 20 25 30 35 40 45 50 55 60 65 <210> 2 <211> 362 12 ES 2 280 352 T3 5 10 <212> PRT <213> Arabidopsis sp. <220> <223> Polipéptido de LEAFY COTYLEDON2 (LEC2) <220> <221> DOMINIO <222> (165)..(277) <223> dominio B3 de unión de ADN <400> 2 15 20 25 30 35 40 45 50 55 60 65 13 ES 2 280 352 T3 5 10 15 20 25 30 <210> 3 <211> 3195 <212> ADN <213> Arabidopsis sp. <220> <223> promotor 5’ de LEC2, región 5’ no traducida 35 40 45 50 55 60 65 14 ES 2 280 352 T3 <400> 3 5 10 15 20 25 30 35 40 45 50 55 60 65 <210> 4 <211> 1000 <212> ADN 15 ES 2 280 352 T3 <213> Arabidopsis sp. <220> <223> promotor 3’ de LEC2, región 3’ no traducida 5 <400> 4 10 15 20 25 30 35 40 <210> 5 <211> 1296 <212> ADN <213> Arabidopsis sp. <220> <223> ADNc de LEC2 <400> 5 45 50 55 60 65 16 ES 2 280 352 T3 5 10 <210> 6 <211> 2968 <212> ADN <213> Arabidopsis sp. <220> <223> Gen LEC2 desde el sitio de inicio de la traducción al sitio de poliadenilación <400> 6 15 20 25 30 35 40 45 50 <210> 7 <211> 22 <212> ADN <213> Secuencia Artificial <220> <223> Descripción de Secuencia Artificial: cebador D2F de amplificación de región genómica <400> 7 55 60 tttcagaata cgcaaaaacg ac 22 <210> 8 <211> 20 <212> ADN <213> Secuencia Artificial <220> <223> Descripción de Secuencia Artificial: cebador D2R de amplificación de región genómica 65 17 ES 2 280 352 T3 <400> 8 aactatcctc ccgagtgacc 5 10 15 20 <210> 9 <211> 20 <212> ADN <213> Secuencia Artificial <220> <223> Descripción de Secuencia Artificial: cebador Ef de amplificación de región genómica <400> 9 agatggcaag gatcaacagg 20 25 30 20 <210> 10 <211> 23 <212> ADN <213> Secuencia Artificial <220> <223> Descripción de Secuencia Artificial: cebador BlastR de amplificación de región genómica <400> 10 cttgctttcg tcctcgtata ttg 35 40 23 <210> 11 <211> 20 <212> ADN <213> Secuencia Artificial <220> <223> Descripción de Secuencia Artificial: cebador F2F de amplificación de región genómica <400> 11 45 tttgtgaagc aaaatggagc 50 55 20 <210> 12 <211> 18 <212> ADN <213> Secuencia Artificial <220> <223> Descripción de Secuencia Artificial: cebador Stop de amplificación de región genómica <400> 12 60 65 cggatgaacc cacgtacg 18 <210> 13 <211> 25 <212> ADN <213> Secuencia Artificial 18 ES 2 280 352 T3 <220> <223> Descripción de Secuencia Artificial: cebador de amplificación de ADNc 5 <400> 13 aaatggataa cttcttaccc tttcc 10 15 25 <210> 14 <211> 18 <212> ADN <213> Secuencia Artificial <220> <223> Descripción de Secuencia Artificial: cebador de amplificación de ADNc <400> 14 20 cggatgaacc cacgtacg 18 25 30 35 40 45 50 55 60 65 19