Transporte de proteínas
Anuncio

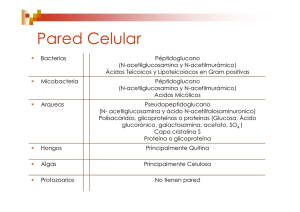
Sistemas de secreción Nature Reviews Microbiology 5, 839-851 (November 2007) Transporte de moléculas al exterior del citoplasma Translocación. Transporte de proteínas intra o a través de la membrana. Exportación. Cuando la proteína es translocada al periplasma. Secreción. Cuando la proteína es transportada al medio extracelular, dentro de otra célula o a la superficie celular. Excreción. Transporte extracelular de moléculas que nos son de origen proteico (por ejemplo productos de la fermentación). Transporte de proteínas Ocurre en las células de los tres dominios de la vida. La tercera parte de las proteínas de las células es secretada a través o insertada en las membranas. Bacterias Gram positivas solo en la membrana citoplasmática. Bacterias Gram negativos también ocurre en la membrana externa. Bacterias fotótrofas además en las membranas fotosínteticas. Mechanisms of regulated unconventional protein secretion Walter Nickel & Catherine Rabouille Nature Reviews Molecular Cell Biology 10, 148-155 (February 2009) Células eucariotas en las membranas de los organelos. Bacterias patógenas en la membrana citoplasmática de las células hospederas y las toxinas en su sitio blanco. Secreción de proteínas Enzimas hidrolíticas. Lipoproteínas periplásmicas. Toxinas. Apéndices de superficie. Proteínas integrales de membrana: ~Transporte. ~Producción de energía. ~División celular. ~Receptores de señales extracelulares. ~Biogénesis. Nature Reviews Microbiology 5, 883-891 (November 2007) Sistemas generales de secreción Sistema Sec (General Secretory Pathway “GSP”). Sistema de translocación y exportación de proteínas no plegadas. Sistema Tat (Twin arginine translocation). Sistema de translocación y exportación de proteínas plegadas. Translocasa YidC. Sistema de translocación de proteínas de Membrana Interna. Nature 406, 575-577 (10 August 2000) Sistema Sec General Secretory Pathway (GSP). Sistema de translocación y exportación de proteínas no plegadas. Componentes: 1. 2. 3. 4. Péptido líder Proteína chaperona (SecB) Complejo de proteínas de unión (SecYEG) ATPasa citoplasmática (SecA) Sistema Sec SecYEG + Sec A= translocasa. Es la responsable del movimiento de la proteína a través de la membrana citoplasmática. El complejo SecYE forma un canal conocido como translocón o canal conductor de proteínas (CCP). SecG, estimula el transporte y SecD, SecF y yajC son regulatorias. Péptido líder (secuencia líder o secuencia señal). La proteína que será translocada es sintetizada con este péptido y es removido durante la translocación. Peptidasa del péptido señal (SPaseI). Libera la proteína translocada del translocón. Nature Reviews Microbiology 5, 839-851 (Nov 2007) Sistema Sec 1. Reconocimiento y guía a la proteína (translocación co-traduccional y post-traduccional) 2. Translocación. 3. Liberación y maduración. Nature Reviews Microbiology 5, 839-851 (Nov 2007) Translocación post-traduccional. Sistema Sec. MENSAJE BIOQUÍMICO, Vol. XXVII (2003) 1) La proteína chaperona SecB se une a una preproteína naciente en el citosol, estabilizando su conformación no plegada. 2a) El complejo SecB-preproteína es dirigido hacia la translocasa SecYEG unida a SecA (3a). 2b) El complejo SecB-preproteína se asocia a la proteína SecA y se dirige a SecYEG en la MI (3b). Translocación post-traduccional. Sistema Sec. MENSAJE BIOQUÍMICO, Vol. XXVII (2003) 4) Se une la secuencia señal a SecA, lo cual estabiliza la interacción SecB-SecA. 5) La preproteína se transfiere a SecA. La unión de ATP por SecA promueve el inicio de la translocación y la liberación de SecB del complejo ternario. La hidrólisis del ATP permite el paso de la proteína a través del translocón. SecA y SecY se muestran como monómeros y el extremo carboxilo de SecA, donde se cree que SecB se une (etapas 1-4 y 12). Translocación de la pre-proteína dependiente de SecA Contornos irregulares indican cambios conformacionales. La línea gruesa de color naranja representa a la presecreción de proteínas y el rectángulo naranja representa el N-terminal del péptido de señal. D: ADP T: ATP. Nature Reviews Microbiology 5, 839-851 (Nov 2007) Translocación co-traduccional. Signal Recognition Particle (SRP) La translocación de proteínas es mediada por una ribonucleoproteína (SRP), su receptor (FtsY) en la membrana y el complejo SecYEG. Requiere la hidrólisis de GTP en la SRP y FtsY para liberar la proteína naciente a través de SecYEG. Nature Reviews Microbiology 4, 537-547 (July 2006) Sec en diferentes organismos <> Nature Reviews Microbiology 4, 537-547 (July 2006) Composición de la translocasa Sec en los dominios de la vida Nature Reviews Microbiology 4, 537-547 (July 2006) Sistema Tat Twin arginine translocation (Tat). Sistema de translocación y exportación de proteínas plegadas. Se ha encontrado en: La membrana citoplasmática en bacterias y arqueas. Membranas tilakoides de los cloroplastos en plantas. Posiblemente en la membrana interna de la mitocondria. Nature Reviews Molecular Cell Biology 2, 350-356, 2001. Componentes twin-arginine translocase (Tat) Peptido señal (SRRxFLK) en el extremo amino terminal. 3 Dominios Amino terminal, cargada positivamente. Hidrofóbico Carboxilo terminal Annu. Rev. Microbiol. 2006. 60:373–95 Corte de la peptidasa Péptido señal Biochemical Society Transactions (2007) Volume 35, part 5 Nomenclatura de aminoácidos Aminoácido Código de tres letras Código de una letra Alanina Ala A Arginina Arg R Asparagina Asn N Ácido aspártico Asp D Cisteína Cys C Glutamina Gln Q Ácido glutámico Glu E Glicina Gly G Histidina His H Isoleucina Ile I Leucina Leu L Lisina Lys K Metionina Met M Fenilalanina Phe F Prolina Pro P Serina Ser S Treonina Thr T Triptófano Trp W Tirosina Tyr Y Valina Val V Proteínas TatABC Tat translocasa: Proteínas membranales (TatA, TatB y TatC). TatA forma el poro por medio de subunidades . TatB tiene función semejante a TatC sin embrago no se encuentra presente en todos los microorganismos. TatC funciona en el reconocimiento de las proteínas. Annu. Rev. Microbiol. 2006. 60:373–95 Presencia de Tat B TatA (verde) TatB (amarillo) TatC (púrpura) A) E. coli K-12 B) Bacillus subtilis C) Halobacterium sp. D) Tilakoides de maíz Tat B pudiera servir como mediador entre Tat A y C. Modelo Tat Annu. Rev. Microbiol. 2006. 60:373–95 Translocasa YidC Translocación e inserción de proteínas de Membrana Interna. Las proteínas de membrana interna (IMP) pueden translocarse e insertarse a través de tres formas propuestas: A. Ruta de YidC. B. Ruta YidC-SRP ? C. Ruta translocasa Sec-YidC o SecSRP-YidC EMBO reports 4, 10, 939–943 (2003) Proteínas en las membranas Hay dos tipos de proteínas integrales de membrana en las membranas celulares: Las que contienen regiones transmembranales (TM) a-hélices ampliamente distribuidas. Las que poseen múltiples plegamientos β, que se encuentran predominantemente en la membrana externa de las bacterias Gram negativas y de la membrana externa mitocondrial y de cloroplastos de las eucariotas. YidC insertasa dependiente e independiente de Sec Nature Reviews Microbiology 6, 234-244 (March 2008) La insertasa YidC tienen la función de promover la inserción de proteínas de membrana en conjunción con la translocasa Sec (o SecSRP) o independiente de ella. YidC como única vía de inserción de proteínas de MI Durante la inserción en la membrana los segmentos transmembranales de la proteína recién sintetizada se unen a YidC, la cual facilita el desplazamiento lateral de los segmentos hidrofóbicos dentro de la bicapa y/o asiste en el plegamiento y ensamblaje. YidC también actúa como proteína insertasa de membrana independiente de Sec de ciertas pequeñas proteínas de membrana. Estas proteínas se unen directamente a YidC o posiblemente utilizan el sistema SPR y FtsY para la unión. Aún se desconoce que determina que las proteínas recién sintetizadas empleen un sistema Sec dependiente o Sec dependiente. PMF: fuerza protón motriz. Inserción por las translocasas Sec y YidC en la MI Microbial Cell Factories 2008 7:10 YidC interactúa con los segmentos TM de dos maneras Nature Reviews Microbiology 6, 234-244 (March 2008) En el modelo secuencial, el primer segmento TM es liberado de YidC para entrar en la fase lipídica antes de unirse a la siguiente hélice TM (a). YidC interactúa con los segmentos TM de dos maneras Nature Reviews Microbiology 6, 234-244 (March 2008) En el modelo de ensamblaje de sitio, YidC tiene un papel importante en el acomodo de las regiones TM en un paquete (b). El conjunto de los segmentos TM se libera de YidC y entran en la bicapa lipídica al mismo tiempo. En ambos casos, los segmentos TM pasan el canal SecYEG uno a la vez mientras que YidC actúa como un acompañante transfiriendo los dominios TM en la bicapa lipídica. Sistemas de secreción en bacterias BMC Microbiology 2009, 9(Suppl 1):S2 Propuesta. Biogénesis de la membrana externa FL(LP) y LPS LPP Omp (Omp85) Nature Reviews Microbiology 4, 57-66, 2006 Pie de figura. Biogenesis of the outer membrane. After synthesis at the inner leaflet of the inner membrane (IM), both lipopolysaccharide (LPS) and phospholipids (PL) are flipped to the outer leaflet of the IM. MsbA is required for the translocation of LPS and possibly phospholipids across the IM, but helical transmembrane domains have also been shown to translocate phospholipids. How both LPS and phospholipids travel from the IM to the outer membrane(OM), and how LPS is flipped to its outer leaflet, remains unknown, although the OM protein (OMP) Imp is required for the assembly of LPS. Both OM lipoproteins and OMPs are synthesized in the cytoplasm and are translocated across the IM by the Sec translocon. After undergoing lipid modification and signal-sequence processing (not shown), OM lipoproteins interact with the ATP-binding cassette (ABC) transporter LolCDE, which hands them over to the periplasmic chaperone LolA. LolA escorts OM lipoproteins across the periplasm and delivers them to the OM-assembly site, the OM lipoprotein LolB. After translocation through the Sec machinery, OMPs are transported to the OM by an unknown mechanism, although the periplasmic chaperones Skp, DegP and SurA have been implicated. At the OM, the YaeT/YfgL/YfiO/NlpB complex assembles OMPs by an unknown mechanism. Nature Reviews Microbiology 4, 57-66, 2006 Principales sistemas de secreción en bacterias Gram negativas Nature Reviews Microbiology 7, 703-714, 2009 Chaperonas Secreta estructuras de virulencia como las fimbrias P y tipo 1 en E. coli uropatógena. 1. Las subunidades son exportadas individualmente a través de la membrana por el sistema Sec y la región amino terminal de la secuencia señal es procesada. 2. En el periplasma cada subunidad se une a proteínas chaperonas periplásmicas. The EMBO Journal (2005) 24, 2075–2086 Chaperonas 3. El complejo chaperona-subunidad migra a la proteína portera (usher) que se encuentra en la membrana externa y forma un canal. 4. La chaperona se une al usher y transfiere la subunidad a este. Más chaperonas transfieren las subunidades para ensamblar el filamento a través del usher. EMBO reports 7, 7, 734–738 (2006) SSTV. Autotransportadores Transporta proteínas con diferentes funciones: proteasas, tóxinas, invasinas, adhesinas. 1. El péptido líder dirige la secreción vía el sistema Sec y se procesa en la cara periplasmática de la membrana interna (MI). 2. El dominio b del intermediario periplásmico adquiere la conformación de barril b y se inserta en la membrana externa (ME) para formar el poro. 3. Se transloca el dominio pasajero a la superficie. SSTII Principal vía para la secreción de proteínas en bacterias Gram negativas: enzimas hidrolíticas y toxinas. Emplea el sistema Sec para transportar las proteínas la periplasma y la secuencia líder es removida por proteasas en la cara externa de la MI. La región amino terminal de la secuencia señal es procesada. SSTII 1. Las proteínas se pliegan en una forma cercana a la nativa. 2. Son excretadas por un sistema llamado secretón que consiste de proteínas localizadas en la MI y en la ME que presentan dominios en el periplasma. 3. Las proteínas a secretar pasan a través del poro formado por la proteína GspD (secretina) y que es estabilizado por las lipoproteínas chaperonas GspS para su correcto plegamiento y actividad. 4. En el modelo de la toxina de V.cholerae Las proteínas Gsp E , L y M regulan la secreción de la extracelular comunicando la fosforilación o hidrólisis del ATP entre la MI y el poro. 5. Las proteínas GspG, H, I, J y K son procesadas por Gsp O y forman una estructura de pilus, principalmente GspG y se postula que actúa empujando la toxina a través del poro con movimientos de contracción. SSTII El sistema de secreción tipo II es empleado para la biogénesis de la fimbria tipo IV y para el flagelo de las arqueas. SSTI La secreción proteína se da en un solo paso desde el citosol hasta el exterior de la célula. Este mecanismo lo realizan la mayoría de las bacterias para la secreción de toxinas y exoenzimas (proteasas y lipasas). SSTI 1. La secuencia exportar es reconocida por la secuencia señal del extremo carboxilo terminal por el transportador ABC en la MI. 2. El transportador ABC interactúa con las proteínas de fusión membranales (MFP). 3. Cuando se forma el complejo Proteína-ABC-MFP, se produce una conexión con la proteínas de los factores de membrana externa (OMF’s) y se forma un poro de salida. 4. En la toxina a-hemolisina (HlyA) de E. coli interacciona con el trasportador ABC (HlyB) y la proteína de fusión trimérica en un proceso dependiente de la fuerza protón motriz (FPM). La translocación de HlyA requiere la interacción entre HlyD y el transportador trimérico TolC, así como de la hidrólisis de ATP. SSTIII Sistema de secreción que ocurre en un solo paso y está asociado a la secreción de factores de virulencia en bacterias patógenas de humanos, animales y plantas (Bordetella, Chlamydia, Erwinia, E. coli, Pseudomonas, Ralstonia, Rhizobia, Salmonella, Shigella, Xanthomonas y Yersinia), así como en la biogénesis flagelar. SSTIII (inyectisoma) Nature Reviews Microbiology 4, 811-825 (November 2006) Tanto el flagelo, como el translocón de secreción de moléculas efectoras, son sistemas complejos que requieren de más de 20 proteínas que se ensamblan en largas estructuras macromoleculares que atraviesan ambas membranas bacterianas, y en los sistemas de virulencia, también la membrana plasmática eucarionte. Esta compuesto por proteínas de MI relacionadas a las del cuerpo basal del flagelo y proteínas que formar anilllos en la MI y en la ME. La translocación de las proteínas se lleva a cabo por el interior de las estructuras y se requiere la energía de la hidrólisis del ATP. Secreción de factores de virulencia SSTIV Sistema versátil que se emplea tanto para la secreción de ácidos nucleicos como proteínas. Se encuentra en bacterias Gram positivas y Gram negativas. Puede ser Sec dependiente (toxina de Bordetella pertusis ) o Sec independiente. Nature Reviews Microbiology 7, 703-714, 2009 SSTIV Nature Reviews Microbiology 7, 703-714, 2009 SSTVI Bacterias Gram negativas. Inyectisoma. Es requerido para los factores de virulencia en patógenos de humanos, animales y plantas. Presente en bacterias simbiontes (fijación de N2) y no simbiontes formadoras de biopelículas) Se asemeja al SSTIII y SSTIV con la presencia probable de chaperonas. THE EMBO JOURNAL (2009) 28, 309 - 310 Infección por Rhizobium (SSTIII y SSTIV) Nature Reviews Microbiology 7, 312-320, 2009 SSTVII (ESX) Presente en Mycobacterium. SecA-1 (GSP) SecA-2 (Vía alternativa) Tat Sistemas especializados (ESAT-6-, SNM-, ESX-, or type VII secretion) Nature Reviews Microbiology 5, 883-891, 2007