Regulación de la Expresión Genética
Anuncio

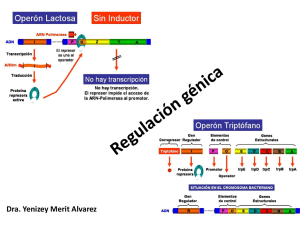
Facultad de Química, UNAM 1630 Genética y Biología Molecular Regulación de la Expresión Genética Unidad 8 En el flujo de la información desde DNA hasta una proteína activa, hay varios pasos regulatorios. Eucariontes Procariontes ... si no hay síntesis del RNAm, el resto de los pasos y la regulación no pueden operar. Proteína Activa La regulación a nivel transcripcional es la más importante cuando se habla de regulación de la expresión genética. 1 GENES CONSTITUTIVOS: Se expresan de manera continua en una célula. Sus productos (RNA / proteínas) tienen funciones en el metabolismo básico celular. Ejemplos: GENES INDUCIBLES. Se expresan solamente en determinadas condiciones del desarrollo o en respuesta al ambiente externo (nutrientes, estrés, etc). Ejemplos: POR LO TANTO, PARA QUE HAYA REGULACIÓN DE LOS GENES TIENE QUE HABER REGULACIÓN POSITIVA Y NEGATIVA GENE CON Regulación positiva (+) El gene “se enciende” (se transcribe o se transcribe mas) GENE CON Regulación negativa (-) El gene “se apaga” (no se transcribe o se transcribe menos) 2 OPERÓN En bacterias, muchos genes se organizan en OPERONES y su regulación ocurre de manera coordinada. En un operón, la expresión de un grupo de genes estructurales está regulada por los mismos elementos de control (promotor y operador) y por genes reguladores. Los genes estructurales están acomodados contiguamente en el DNA. genes sitios de reguladores control genes estructurales P O La lactosa es un disacárido que se debe hidrolizar para ser utilizado: GALACTOSIDASA LACTOSA GALACTOSA GLUCOSA La β-galactosidasa hidroliza a la lactosa para generar glucosa y galactosa. La glucosa es metabolizada. La β-galactosidasa convierte parte de la lactosa a alolactosa 3 La βgalactosidasa se encuentra en niveles muy bajos si no hay lactosa en el medio. Su producción se induce cuando se agrega lactosa al medio y se elimina la glucosa de éste. Se elimina la lactosa Se añade lactosa Tiempo Los operones están formados por genes estructurales y una región de control Genes regulador Regiones de control Genes estructurales Genes estructurales o = región operadora p = promotor Galactosidasa Permeasa Transacetilasa 4 El represor unido al sito operado previene la transcripción de los genes z, y, a El represor unido al sito operado previene la transcripción de los genes z, y, a Galactosidasa El inductor se une al represor y entonces éste ya no se une al DNA Permeasa Transacetilasa La poca alolactosa que se formó actúa como inductor. 5 6 REGULACIÓN FINA DEL OPERÓN LAC Aun en ausencia del represor, el promotor del operón lac no es muy fuerte, por lo que requiere la actividad de otras proteínas. Activador CRP. cAMP receptor protein Si los niveles de glucosa son altos. Si los niveles de glucosa son bajos. AMP cíclico Cuando los niveles de AMPc se incrementan, se une a la proteína CRP El complejo CRP-AMPc se une al promotor del operón de lactosa y causa un giro en el DNA que facilita la unión de la RNA polimerasa al promotor. 7 El promotor del operón Lac contiene un sitio de unión para la proteína CRP Sitio Unión a CRP Sitio Unión a RNA Pol CRP Sitio Unión a CRP El complejo CRP-AMPc se une al promotor del operón de lactosa facilita la unión de la RNA polimerasa al promotor y se incrementa 50 veces la transcripción. Regulación del operón de lactosa. Regulación Negativa. Represor Promotor Operador No se sintetiza RNA RNA Polimerasa Proteína Represor Activo No hay lactosa, hay glucosa, el represor está activo y el operón está apagado 8 Promotor Sitio de Unión a CRP Operador RNA Polimerasa Proteína CRP Inactiva Represor Inactivo Hay lactosa, hay glucosa. Los niveles de AMPc son bajos. La síntesis del RNAm lac es pobre. Regulación positiva. Inducción Promotor Sitio de Unión a CRP RNA Polimerasa Operador Proteín a CRP Activa Represor Inactivo Proteína CRP Inactiva Hay lactosa, glucosa es baja, niveles de AMPc son altos. Se sintetiza mucho RNAm lac 9 Operón de triptofano Este operón incluye cinco genes de enzimas involucradas en la biosíntesis de triptofano. Bajo control del promotor (Ptrp) y del operador (Otrp) El represor se une a triptofano y este complejo se une al operador reduciendo la transcripción 70 veces aproximadamente. 10 En eucariontes, la regulación de la expresión genética se puede dar a distintos niveles. Eucariontes Procariontes 11 Los promotores de los genes eucariontes son más complejos que los procariontes. Muestran menor conservación en los elementos de reconocimiento de las RNA polimerasas. Inicio de la transcripción 18 - 25 nts Caja TATA: TATA(A/T)A(A/T) *URE (Elementos regulatorios “río arriba”). Son sitios de unión de otras proteínas (factores de transcripción) que facilitan la unión de la RNA polimerasa y la transcripción de ese gen. De 100 a 200 pb del inicio. Enhancers (Sec. Intensificadoras). Regiones en el DNA que están alejadas por más de 1000 pb del sitio de inicio y que activan al promotor para que ocurra una transcripción más eficiente. La caja TATA funciona como señal para la unión de la proteína TBP (TATA-binding protein). La unión de TBP al DNA causa una torsión de éste, facilitando la apertura de la doble hélice. El factor de transcripción IID (TFIID) se une a promotores que contienen la caja TATA a través de la proteína TBP. Se forma un complejo multiproteíco en el promotor que permite la asociación de proteínas que están unidas a otras regiones en el promotor. 12 Regulación de la transcripción en eucariontes. Además de los factores de transcripción generales que forman el complejo basal de transcripción, hay otras proteínas que se unen con alta afinidad a motivos específicos en los promotores, en los elementos regulatorios y en las regiones intensificadoras. • Se unen fuertemente al DNA • Activan la transcripción Estas dos actividades distintas residen en dominios discretos y bien caracterizados de los factores de transcripción. Además, pueden tener dominio de dimerización, pues es frecuente que actúen como homodímeros o heterodímeros. Algunos factores de transcripción también tiene un dominio de unión a un ligando, por ejemplo, a una hormona. Dominio de Unión al DNA Dominio de Dimerización Dominio de Transactivación 13 Dominios de Unión al DNA Dominio Hélice-vuelta-Hélice. 4 Hélices alfa. Las hélice 2 y 3 están separadas por un giro β de tal manera que quedan en ángulo recto. Una o dos hélices se unen al surco mayor del DNA Ejemplo: Factores de transcripción homeóticos. Los factores de transcripcion homeóticos regulan la expresión de genes durante el desarrollo embrionario. Mutante Antenepedia en Drosophila melanogaster Estos factores de transcripción se encuentran altamente conservados en eucariontes. La posicion de sus genes en el cromosoma se encuentra en el mismo orden de las regiones del embrión cuyo desarrollo controlan. 14 Dominios de Unión al DNA Dominio dedo de Zinc Se forma un asa de 12 aminoácidos que contiene 2 His y 2 Cys. Estos AA coordinan a un ión Zn2+ Esta estructura se repite tres o más veces a lo largo del dominio. Ejemplo: Factor de transcripción Sp1 Dominios de Dimerización Zippers de Leucina Contiene una región rica en Leu (cada 7 aa, hay Leu). Se forma una superficie hidrofóbica en una hélice α Esta estructura se forma en las dos subunidades que forman el dímero. Hay interacciones hidrofóbicas entre ellas. 15 Regulación de la expresión genética en eucariontes por microRNAs Los microRNAs son sintetizados por la RNA pol II • Se sintetizan como precursores largos (primicroRNA). • Se añade el CAP al extremo 5’. • Se poliadenilan en el 3’. • Son procesados por nucleasas para formar pre-microRNA Regulación de la expresión genética en eucariontes por microRNAs • Exportación de los pre-microRNAs al citoplasma. • Procesamiento por la nucleasa DICER. • Se elimina la región del asa. • RNA duplex asimétrico. • mRISC separa las cadenas de RNA. • Generación del miRNA maduro. 16 Función de los microRNAs en la regulación de la expresión génica El miRNA maduro se aparea por complementaridad de bases al RNAm blanco, generalmente en la región 3’-UTR provocando: 1. Represión de la traducción 2. Degradación del RNAm • Aprox. 2000 miRNAs identificados hasta la fecha en humanos. • Regulan aprox. 33% de los RNAm. • El tamaño de un miRNA maduro varía de 20 – 22 nts. • Un miRNA puede regular muchos RNAm. 17