Síntesis de Proteínas
Anuncio

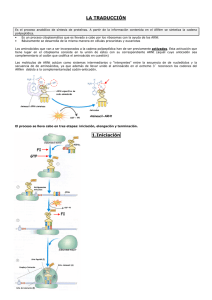
Síntesis de Proteínas Vamos a ver donde confluyen distintos tipos de moléculas, una gran cantidad de moléculas, mucho mayor que las que vimos en procesos como la replicación del ADN. La fidelidad de este sistema va a permitir que la expresión del mensaje que está contenido en el ADN se haga de buena manera. Vamos a ver una etapa importante en la traducción de proteínas que tiene que ver con la activación de los amino ácidos. Esta etapa es la responsable de dar la fidelidad del proceso de síntesis de proteínas. Habíamos visto que en la replicación del ADN teníamos enzimas que tenían la propiedad de corregir errores. Aquí vamos a ver que va a participar una enzima que es la aminoacil ARNt sintetasa y que está involucrada en la activación del a. ácido. Esta enzima va a poder distinguir el ARNt correspondiente al a. ácido. Este es un paso esencial ya que es el ARNt el que va a traer los amino ácidos y los va a ir adicionando de acuerdo al código que está en el ARNm. De manera tal que cargar el ARNt con su correspondiente amino ácido es esencial porque si falla hay una alteración en la proteina. Ahí tienen un cuadro donde podemos comparar las 3 etapas que hemos visto y cuales son los elementos que participan en cada una de ellas. En la replicación tenemos la ADN polimerasa, en la transcripción participa la ARN polimerasa y en la traducción vamos a ver que la verdadera fábrica de la formación de proteínas corresponde a los ribosomas. De todas las moléculas que participan, aquí tenemos más o menos las más importantes: el ARNm que lleva el mensaje, el ARNt es el intermediario y tenemos los ribosomas que van estructurados por ARNr y proteínas ribosomales. Es una fábrica que participa activamente en la traducción. También hay proteínas no ribosomales entre las cuales están los factores de traducción. Para que se den cuanta de la magnitud y complejidad de este proceso, les muestro esta diapo donde ustedes ven que en el caso de la E. coli, si ustedes secan la bacteria, osea le sacan el agua, pueden ver que dentro de todos los componentes que participan en la traducción, el 35% del peso seco de la bacteria corresponde a los elementos que participan en la traducción. Eso significa 20.000 ribosomas, 200.000 ARNt y 100.000 proteínas y cofactores. Aquí está la estructura de esta fábrica que está compuesta por 2 subunidades. Tanto procariontes como eucariontes son muy parecidas, simplemente cambia el tamaño: una subunidad más grande y una más chica y sirve como estructuras en las cuales se reconoce específicamente al ARNm que luego va a reconocer al ARNt en sus distintas etapas: cuando llega con el amino ácido o cuando está en la posición para formar el enlace peptídico o cuando está listo para salir. Asociado a ello hay una zona por donde va a ir saliendo la cadena polipeptídica hacia el exterior donde después está en condiciones de adoptar la forma 3D y realizar su función. Una de las características de este proceso es que la traducción comienza en el extremo 5’ del ARNm y luego de reconocida esta zona avanza hacia el extremo 3’. Construyendo mensajeros sintéticos con distintos tipos de combinaciones, se llegó a la conclusión de que este mensajero era posible de leerlo a través de tripletes de nucleótidos que tenían la información para un determinado amino ácido. Con estudios de marcaje se pudo determinar que las proteínas son sintetizadas desde el amino terminal hacia el carboxilo terminal. Se llegó también a la conclusión de que el código es degenerado porque distintos códigos (tripletes) pueden codificar para un mismo amino ácido. Aquí por ejemplo, para tratar de definir en qué sentido se leía un mensajero, se construye un ARNm sintético que es un poli A con una terminación de citosina en el extremo 3’. De manera tal que si se lee de 5’3’ (ya se sabía que se leía en tripletes) se debería repetir los amino ácidos correspondientes, osea, en este caso tendríamos que teber lisina, lisina, lisina y terminar con asparagina. Si se lee apartir del 3’, el primer triplete en vez de ser AAA es CAA que codifica para glutamina, osea, se hace una proteína distinta. Para saber en qué sentido de sintetizaba una proteína, se van marcando distintos tipos de proteínas y después se aislan los péptidos y ser cortan. Al contar la radioactividad incorporada en los fragmentos, los que tenían el amino ácido terminal, el carboxilo terminal, eran los que tenían una mayor incorporación de radioactividad. Eso indicaba que la síntesis ocurría hacia el carboxilo terminal. Una etapa central de la síntesis de proteínas, es formar el enlace peptídico entre 2 amino ácidos. Si ustedes tratan de hacer esto en condiciones in-vitro, toman amino ácidos y esperan que entre ellos se forme el enlace, eso no ocurre porque está muy desfavorecido termodinámicamente. De manera tal que la ___ necesita activar a la molécula para que esta reacción pueda ocurrir. A lo largo de los distintos capítulos hemos visto que hay que transportar determinados grupos, por ejemplo, los grupos acilo nunca se transportaban solos, sino que lo hacía junto a la CoenzimoA. Entonces teníamos el grupo acetilCoA. Eso es normalmente porque la hidrólisis del grupo transportador provee la energía necesaria para que la reacción ocurra. Otro ejemplo análogo sería en la síntesis de glucógeno donde la glucosa necesitaba ser activada por UDP. Ahí también poníamos un nucleótido. Aquí se hace algo análogo: vamos a activar los amino ácidos con un ARNt. Por qué creen que activamos los amino ácidos con ARNt y no con un nucléotido como UDP? Necesitamos que ese amino ácido activado ahora pueda ser depositado en la fábrica de proteínas. Esta fábrica, para poder sintetizar proteínas debe leer un mensaje y ese mensaje es un polímero de nucleótidos entonces necesitamos una molécula que pueda leer ese mensaje. Lo mejor, es que sea otra cadena de polinucleótidos para así poder interaccionar complementariamente con el ARNm. Por eso tiene sentido escoger una molécula que cumpla esas 2 funciones: por un lado logramos activar el amino ácido y por otro lado nos permite reconocer el sitio donde tiene que ser depositado el amino ácido. Entonces el amino ácido es activado por el ARNt y por otro lado esta misma estructura, en otro sitio, va a tener la posibilidad, por complementariedad, de poder leer cada uno de esos tripletes y así poder codificar para un determinado amino ácido. Distintos tripletes son capaces de codificar para un mismo amino ácido. Los que están en rojo (en la diapo de “código genético” ) con señales de término: UAA, UAG y UGA. Vamos a ver que cuando lleguemos a la etapa de término de la producción no existen ARNt que lean eso; el ribosoma y el mensajero quedan como detenidos esperando que llegue un ARNt. Lo que va a llegar ahí son proteínas que son factores de término. Resumiendo, el código está compuesto por tripletes, a cada triplete se le denomina codón y cada uno de ellos codifica para un amino ácido. El código genético es degenerado porque existen varios codones o tripletes para 1 amino ácido. No hay superposición: un nucleótido solamente pertenece a un único triplete. La lectura “sin comas” se refiere a que tenemos un mensajero que está en condiciones de ser leído, en el caso de eucariontes ha sido procesado, se han modificado sus extremos. La importancia que tiene este sistema es que el código es bastante universal. Parte de lo que permite que existan varios codones para cada amino ácido da cuenta la hipótesis de balanceo en que estos distintos nucleótidos puedan interactuar con más de una base en su correspondiente codón. Vamos a analizar algunas características del ARNt. Es una cadena de ribo nucleótidos que a su vez tiene muchas modificaciones. Tiene bases que son modificadas, bases que son distintas. El apareo de las bases no es el que normalmente conocemos, eso ocurre así porque eso va a permitir mantener el arreglo tridimensional muy particular del ARNt. Vamos a tener un ARNt por cada amino ácido entonces van a ver variaciones estructurales en cada uno de ellos. Pero básicamente todos tienen esta misma estructura. La variación tiene sentido porque el ARNt es cargado o activado por su correspondiente amino ácido manteniendo la fidelidad del proceso. Y esa labor la hace la enzima aminoacil ARNt sintetasa. La enzima por un lado reconoce al ARNt y por otro el amino ácido y por eso también las variaciones que hay en el ARNt porque la enizma va a ser específica para un solo ARNt. Ella va a tener un arreglo tridimensional que le va a permitir en algunos casos con moléculas en distintos sitios o de repente con mezclas de interacción en distintos sitios. En esta zona va a estar ubicado el anitcodón que va a ser la parte que interacciona con su correspondiente codón. Si bien estas son las que leen, son importantes las estructuras que están asociadas a ella para que pueda tener el arreglo tridimensional perfecto para que calce con ese codón y porque también hay algunas aminoacil ARNt sintetasas que reconocen el correspondiente ARNt por el arreglo tridimensional que ellos tienen en la zona del anticodón. La otra característica es que tiene un brazo libre con este triplete CCA y este es el sitio donde uno va a adicionar el amino ácido. Por lo tanto este intermediario va a traer el amino ácido con su extremo 3’ activado y con su ____________ (23’ 45) para poder leer el mensaje del mensajero. Aquí ven ahora la equivalencia de esta “caricatura”, pero en la realidad. Este es el arreglo estructural que tiene el ARNt (en color se ven las zonas homólogas). Va a tener este extremo libre donde se une al amino ácido y de acuerdo a la estructura que adquiera en esta zona le va a permitir poder interactuar con la aminoacil ARNt sintetasa. Ustedes pueden ver distintos tipos de ARNt para distintos tipos de amino ácidos donde cambian levemente pero básicamente todos tienen la misma estructura. Y resumiendo algunas características de estas moléculas, son pequeñas cadenas de ribonucleótidos que van entre 73 y 93. Vamos a encontrar en ellas bases no típicas ( lo típico es lo que se encuentran en el ADN y en el ARN). Muchas de ellas están metiladas o bien dimetiladas lo cual produce cambios en la hidrofobicidad. Normalmente esos cambios en la hidrofobicidad van a permitir 2 cosas: por un lado que este ARNt pueda organizarse en su estructura típica y la otra que pueda interaccionar con una zona que le corresponda de la aminoacil ARNt sintetasa. El extremo 5’ está fosforilado y el 3’ tiene el triplete CCA que va a transportar el amino ácido. Parte de su estructura tiene doble hélice dando una forma de L. El anticodón consiste básicamente en la secuencia a una pirimidina, pirimidina y una purina modificada. Ahora vamos a ver dentro de las proteínas no ribosomales que no forman parte de la estructura del ribosoma a la proteína muy importante que es la amioacil ARNt sintetasa. Vamos a tener una enzima por cada uno de los ARNt y de los amino ácidos. Una de las etapas más importante en la que participa esta enzima es en la activación de los amino ácidos. Para activar el amino ácido lo vamos a metilar es decir, lo vamos a asociar a un grupo AMP y ese AMP proviene de un ATP liberándose un pirofosfato. La liberación del pirofosfato permite que con la hidrólisis se libere energía que hace que esta reacción sea favorable desde el punto de vista energético. Ese AMP va a quedar asociado al amino ácido el cual posteriormente va a ser unido al ARNt. De esa manera logramos activar el amino ácido y luego al ARNt es cargado con su correspondiente aminoácido participando en la traducción. Aquí tenemos el proceso que les acabo de describir: tenemos la molécula de ATP con su nucleosido y trifosfato y por otro lado el amino ácido con su grupo amino y el grupo carboxilo. La enzima entonces lo que hace toma parte de esta estructura que corresponde al AMP y se lo une mediante enlace éster al grupo carboxilo por lo tanto queda un aminoail adenilato. De esta manera nos queda activo este grupo y se libera el pirofosfato. Luego viene el ARNt que tiene en su extremo 3’ la secuencia CCA y aquí tenemos en este nucleótido en el extremo 3’ el azúcar, la ribosa con su extremo 2’ y 3’ con un grupo OH. Viene ahora la aminoacil ARNt sintetasa (todo está ocurriendo en esa enzima) y ella toma el amino ácido (?) y se lo adiciona a uno u otro OH que está presente en la ribosa. En este caso a los del grupo 2’ y por lo tanto nos queda el 2’ Aminoacil ARNt. Y de esta manera tenemos un ARNt que está activado o cargado con su correspondiente amino ácido. Hasta este momento, el anticodón ha servido para que el ARNt sea reconocido por otras sintetasas como su correspondiente ARNt. Ahora, una vez que se está en esta condición, ahora puede ser transportado y llevado al ribosoma y que pueda incorporarse a la síntesis de la proteína cuando el anticodón pueda reconocer su correspondiente codón. La aminoacil ARNt activa tanto al amino ácido como al ARNt. Hay distintos tipos de aminoacil ARNt sintetasas, de clase I y clase II. Pueden ver las de clase I cuales son los amino ácidos que activa y las de clase II. Además las de clase I son monómeros y las de clase II son dímeros. Normalmente la de clase I incorpora el amino ácido en la posición 2’ de la ribosa del ARNt y las de clase II lo hacen en el OH de posición 3’. También hay diferencias en cuanto al tamaño que distingue a un amino ácido de otro. Pueden ver el ARNt y su correspondiente aminoacil ARNt sintetasa que está asociado a él. Normalmente algunos de estos ARNt la cola con la secuencia CCA adquiere una determinada orientación tridimensional que también sirve de reconocimiento de la enzima, ella puede reconocer de su correspondiente ARNt cual es la orientación que tiene el extremo CCA. Pueden ver la estructura de la clase II formando complejos correspondientes de ARN. Normalmente las de tipo I corresponde a amino ácidos grandes y son en su mayoría hidrofóbicos y las de tipo II corresponden a amino ácidos pequeños. Si se dan cuenta, ya aquí hay algunos elementos que van permitiendo la fidelidad del mensaje. Primero, está la enzima que permite reconocer su correspondiente ARNt. Por otro lado, para poder ______ (32’ 50) los amino ácidos ya deja, ésta por ejemplo, descarta a todo el resto que corresponde a las del tipo II porque son más pequeños entonces dentro de los grandes y que sean hidrofóbicos va a tener que entrar ahora a incorporar otro tipo de ese ____ (?) para no confundirlo, es decir, aquellos amino ácidos que tienden a ser parecidos dentro del mismo grupo que selecciona la sintetasa. De manera tal, la alta fidelidad de la síntesis de la proteína va a estar dada por el ARNt que llegue con su adecuado amino ácido y para que llegue el ARNt con su amino ácido adecuado, la aminoacil ARNt sintetasa selecciona por un lado al amino ácido y por el otro al ARNt. Ahora, cómo logra realizar esto? Mediante su disposición estructural. En su estructura, esta enzima tiene 2 sitios estructurales: en un sitio es donde va a ocurrir la activación del amino ácido, osea, la parte donde toma el AMP del ATP y lo incorpora al amino ácido y se forma el aminoacil adenilato y una vez que el amino ácido está activado viene otro sitio de reconocimiento que corresponde a un sitio de hidrólisis, es decir, si el amino ácido al cual se activó recién es un amino ácido equivocado, hay un segundo sitio como de edición en el cual se le saca su molécula de AMP o de ARNt y se deja inactivo. En el sitio donde va a ocurrir la activación, es un sitio que tiene una disposición tridimensional adecuada para que quepa su correspondiente amino ácido. Cuando el resto de los amino ácidos que se asemejan a él son distintos en tamaño la enzima prácticamente no va a tener problema en seleccionarlo. Hay amino ácidos que tienen ______ en tamaño (35’20). Como les decía, para todos aquellos que tengan un tamaño mayor que este sitio quedan inmediatamente excluidos porque no pueden entrar al sitio para activarse. Hay otros que pudieran tener el mismo tamaño, pero teniendo el mismo tamaño muchas veces tienen grupos que son distintos. Supongamos que el amino ácido tiene grupos que son hidrofóbicos y los otros que tienen un tamaño parecido tienen grupos hidrofílicos. Esas diferencias de interacción química permite también eliminarlo. Si de todas maneras se inserta y es activado después la enzima lo mete al sitio de hidrólisis. Si cabe en el sitio de hidrólisis, lo hidroliza. Entonces por ejemplo, la aminoacil ARNt sintetasa para la treonina y para su correspondiente ARNt tiene más problemas que otra aminoacil ARNt sintetasa porque si tiene que activar al ARNt con la treonina, ésta se parece bastante a la valina y a la serina. La treonina tiene en la cadena lateral un grupo metilo y acá un grupo OH. La valina se parece a éste porque tiene 2 grupos metilo, de manera tal que esto lo hace muy parecido en cuanto a tamaño. En el caso de la serina también tiene un grupo OH como la treonina pero carece de un grupo metilo. Como estos son parecidos en tamaño, pueden caber en un sitio de activación. Se ve a la aminoacil ARNt sintetasa capturando al ARNt para la treronina y cuando reconoce ese ARNt, incorpora la treonina, pero pudiera ser que llegase la valina. Pero en ese sitio, la enzima tiene grupos que pueden interaccionar con el grupo OH formando puentes de hidrógeno. Si se mete ahí la valina, el grupo es hidrofóbico, por lo tanto no puede interactuar ese sitio con el que interacciona con el grupo OH, por lo tanto no entra, no cabe. Sin embargo la serina sí, porque también tiene un grupo OH por lo tanto va a poder interaccionar con la estructura y formar puentes de hidrógeno para capturar el amino ácido. La enzima en esta primera etapa sí se puede equivocar y a éste lo puede activar incorporándole el AMP y uniéndolo al ARNt. Pero ahí viene la segunda etapa: entonces la ARNt lo activa acá y lo deja unido al _____ (39’12) lo mueve a este otro sitio ______ o interaccionaba con el sitio de hidrólisis. Y como este es pequeño, cabe en el sitio de hidrólisis y lo hidroliza. Entonces así evita equivocarse. Otro caso el la aminoacil ARNt sintetasa de la isoleucina que también puede confundirse de repente con la valina. Como habíamos visto, la valina tiene 2 grupos metilo y en este caso la isoleucina tiene 3 grupos metilo y los 2 amino ácidos tienen grupos hidrofóbicos. En el otro caso, se confundía con la serina porque tenía un grupo hidrofílico igual que la treonina, entonces las 2 eran capturadas. En este caso la enzima se puede equivocar con la valina porque semeja un poquito el tamaño y tiene también una cadena lateral hidrofóbica, pero como es más pequeña porque carece un grupo metilo, puede caber en el sitio de hidrólisis; la isoleucina no porque no cabe en el sitio de hidrólisis y ha sido activada. El sitio de hidrólisis es como el equivalente a la ADN polimerasa en cuanto a su actividad correctora de errores. Algunos ARNt reconocen la zona que interacciona con el codón. En otros casos hay zonas de la estructura del ARNt que en este caso interactúan de manera muy activa con la aminoacil ARNt sintetasa que reconoce ______ (42’05). Se ha visto en estudios, por ejemplo, que el par guanina - uracilo que están en la posición 3 y 70 es muy importante para reconocer el ARNt para la alanina. Si este par lo cambian y en vez de que quede guanina – uracilo queda citosina – guanina, ahora eso corresponde a una estructura que es reconocida por el ARNt para la cisteína. Hay otros casos que hay zonas del ARNt que ustedes pueden ir descartando otras zonas, sacar el ARNt y llegan a una estructura mínima como ésta, una microhélice. Esta microhélice todavía sigue siendo suficiente para ser reconocida como el ARNt para la alanina. Y si se dan cuenta, aquí _____________(43’ 07) interacción para reconocer la alanina. En este mismo _____________ prácticamente se ha sacado toda la estructura del ARNt pero se ha mantenido el par guanina-uracilo y basta para que se reconozca como su correspondiente ARNt. Bueno, en otros casos hay ARNt que son 2º Módulo Clase de Síntesis de Proteínas Algunas características que conviene destacar son aquellas que revelan que los ribosomas juegan un rol muy activo en el procesos de síntesis de proteínas, lo otro es después analizar con un poquito más de detalle cuales son las etapas propiamente tales de la síntesis que vamos a ver que corresponden a una etapa de inicio luego a una de elongación y a una de término. Aquí vamos a ver que además del mensajero correspondiente de los tRNA que está en los aá y de la estructura de los ribosomas, además de ellos son muy importante las proteínas, que son distintos tipos de factores proteícos que van a participar guiando la etapa de inicio, la de elongación y participando en la etapa de término de la síntesis de la proteína, después al final vamos a ver una lista de algunos antibióticos que van a tener distintos efectos letales sobre los procariontes o algunas células eucariontes , algunos alteran algunos elementos que participan en la síntesis de proteínas, muchos de ellos los sitios de los ribosomas en que al destruir todo el aparataje de la síntesis de proteínas hace que el organismo sea… Bueno, ustedes pueden ver si hacen un gel en 2 dimensiones, ustedes pueden darse cuenta q los RNA estan formados por distintos tipos de proteínas, en este gel esta la subunidad 30S y en el otro gel la subunidad 50S, q forman parte de esta estructura además de los nucleótidos q forman el RNA ribosomal también proteíco. Hay estructuras o distintos tipo de RNA, 5S, 16S esa es una visión tridimensional, aquí hay distintas características de los RNA, respecto a los tipos, cantidad de nucleótidos, cuan estables son, en el caso de los RNA ribosomales, son bastante estables y los tRNA tb. Ahí ustedes pueden ver un arreglo tridimensional, ahora c/u de estos lo pueden descomponer y lo pueden disociar tanto como los RNA q forman parte de ellos como a su vez los de las proteínas. Hay 1 Japonés q trabajó mucho en esto, en disociar los componentes proteícos y las cadenas de ribonucleótidos y posteriormente losgró diseñar y reconocer la secuencia exacta de ir agregando c/u de estos componentes para lograr restablecer todo del ribosoma para poder determinar propiedades funcionales que en los ribosomas son muy importantes, Ustedes ven aquí el ribosoma de los eucariontes q es un poco mayor, pero algunas de las estructuras de sus correspondientes RNA ribosomales son muy análogos a las estructuras que tienen los procariontes, el de eucariontes es bastante más grande, pesa un poco más del doble, éstos RNA son bastante homólogos el de 18S con el de 16S, el 5.8S es único de eucariontes, esa es una diferencia importante respecto a los de procariontes. Claves que se obtuvieron en distintas experiencias experimentales que revelaron la importancia q tiene los ribosomas en la síntesis de proteínas y q no cumplen solo un mero rol estructural, y una es cuando logran disgregar los distintos componentes, si uno interfiere a nivel de 16S y altera algunos enlaces esto provoca la inhibición de la síntesis de proteínas, en los experimentos de reconstrucción de los RNA ribosomales en el caso del 30S, si hay una proteína q no agregamos, eso repercute en que hay una disminución en la síntesis de proteínas. El 16S a q subunidad corresponde? Por ejemplo, si tomamos un ribosoma y lo centrifugamos vamos a ver q en un determinado punto no precipita más y se le pone 80S al coeficiente de sedimentación esto lo podemos separar y tenemos uno de 40S y de 60S por centrifugación, si c/u de éstos además lo disociamos entre sus componentes proteícos y los ribosomas, tú te puedes dar cuenta q aquí los q forman esta estructura q sedimenta en 40S tu lo puedes descomponer en 30 proteínas y un RNA q si lo centrifugas tiene un coeficiente de 18S, por lo que esta subunidad esta formada x un solo RNA el ribosomal más 30 proteínas. La unidad de 60S q es la mayor en el caso de los eucaroiontes, tu te das cuanta q está formada x 40 proteínas y distintos tipos de RNA, uno más grande q es de 28S, uno más pequeño de 5S y uno de 5,8S. En el caso de este q estamos analizando (procarionte) se vio q era un equivalente al de 18S de los eucariontes. Cuando se altera el de 16S en este caso que es el equivalente a 18S en eucariontes, cuando se recontruye el 30S, pero no se agregan todas las proteínas , disminuye la síntesis, después analizando la secuencia del 16S en procariontes se dan cuanta q este está involucrado en la selección del sitio de partida, después se ve q un sitio prima de RNA interacciona en una zona altamente conservada (quiere decir que tiene + ó – la misma secuencia) del RNA de 23S. En algunos casos lograron recomponer los ribosomas sin las proteínas, es decir solo con la estructura de los RNA ribosomales, en esas condiciones todavía se podía catalizar la formación del enlace peptídico, x lo cual algunos dicen q dentro de la estructura de los ribosomas el RNA tiene más importancia q las proteínas o a su vez reafirma el concepto de q los RNA fueron antes que las proteínas xq sobre el RNA se tuvo q hacer la primera proteína y eso significa que xa la formación de esta el enlace peptídico se tuvo que realizar en ausencia de éstas. Después se vio q este RNA es esencial para la actividad transferasa (23S) y por otro lado q la mayororía de los antibióticos ejercen su actividad xq alteran la traducción a nivel de los RNA ribosomales, todo esto indica que los ribosomas tienen un rol muy importante tanto en el proceso de la traducción junto con los tRNA y los RNA mensajeros, además de las proteínas que forman parte de las distintas etapas de la traducción. Esta microfotografía revela una de las diferencias importantes q hay entre ribosomas eucariontes y procariontes y es la cadena naciente del mensajero cuando esta siendo transcrito a partir de la cadena de DNA y se une una cadena de poliribosomas?? Y a esta cadena que se van uniendo a medida que se va saliendo el mensajero y lo comienzan a leer , es decir, aquí tenemos la cadena de DNA y aquí ribosomas q están leyendo un mensajero y si tuviera la posibilidad de teñir podríamos ver cadenas de proteínas que en un lado serían más largas q en el otro. Estructuralmente a los ribosomas se les han podido reconocer 3 sitios distintos y 2 sitios q catalizan 2 actividades muy importantes, una es reconocer la interacción entre codón y anticodón donde va a llegar el tRNA y va a poder interactuar con su correspondiente anticodón y la otra actividad muy importante es q una vez q llega el tRNA con su correspondiente aá es el poder catalizar la formación del enlace peptídico q va a permitir ir construyendo la proteína, Esos 2 sistemas le van a dar los tRNA?’?, hay un sitio de salida se denomina E x EXIT del tRNA q va quedando vacío. De nuevo, aquí tenemos la estructura del tRNA del ribosoma, vamos a tener el sitio donde va allegar el tRNA cargado, un sitio donde va a estar la cadena naciente donde se va a formar después el enlace peptídico, un sitio de salida donde se va a ubicar el tRNA que ha quedado sin su aá y posteriormente se libera, un sitio en donde se va a poder leer en el mensajero, la llegada del tRNA para q interactúe con su correspondiente codón esas son las zonas importantes que vamos a ir encontrando en el ribosoma para que con la interacción de todos ellos pueda ocurrir de manera normal la síntesis de proteínas, ahora hay un sitio q es como un canal que es por donde va saliendo la proteína y de esa manera impide que las cadenas laterales interactúen entre sí y eventualmente la proteína adopte una estructura tridimensional antes de tiempo, eso lo va a ir haciendo a medida q va saliendo. Vamos a ver ahora las distintas etapas de inicio de la síntesis de proteínas, después la de elongación y la de término. Tenemos q recordar algunas cosas importantes, la etapa de inicio en los eucariontes se inicia con el tRNA para metionina, es decir el codón AUG es el codón de inicio y el q reconoce a la metionina, en procariontes se hace de la misma manera, pero es con una formal metionina, ha sido formulada, luego en procariontes existe una secuencia llamada Shine Delgarno q permite interactuar con el mensajero, y al ocurrir esto permite q se reconozca donde está el AUG que se debe leer, ustedes se dan cuenta que esta es la secuencia que está presente en el 16S que reconoce esta secuencia en el mensajero, de esta manera se va a reconocer cual es el AUG a partir del cual va a ser traducida la proteína, si ustedes modificaran esto e indujeran mutaciones no se sabría desde donde partir en procariontes. Cuando nosotros determinamos cual es el AUG significa q tenemos q extraer el tRNA q codifica para la formal metionina??y en el otro sitio se vana ir formando los enlaces peptídicos q van a ir creando una cadena. Siempre es a través del 16S en procariontes. Ahora ustedes se pueden dar cuenta q existen distintos tipos de procariontes y ahí se ve una zona que es más rica en purina y que se aparean con la secuencia complementaria en el RNA ribosomal de 16S, esto permoite identificar al AUG o tb en procariontes se localiza al TUG??como partida para partir la síntesis de las proteínas. Ahora en eucariontes esto es distinto, ahora nos vamos a detener en detalle, es + ó – lo mismo q ocurre en procariontes, aquí tenemos un resumen, aquí tenemos el mensajero y para que éste pueda leerse lo que va a ocurrir ahora es que van a participar varios factores de inicio desde el 1 al 5, vamos a necesitar energía q va a ser aportada por la hidrólisis de GTP y por lo tanto es muy importante la participación de GTP, vamos a necesitar el tRNA de inicio y la subunidad de 40S, luego necesitamos que ese mensajero quede asociado a la subunidad pequeña del ribosoma para que posteriormente se localice adecuadamente el tRNA de inicio con el codón de inicio, una vez q ocurre esto con el conjunto de los distintos factores de inicio se forma toda la estructura del ribosoma y en esas condiciones comienza la etapa de elongación con la participación de factores de elongación y se empieza a alargar la cadena polipeptídica hasta q vamos a llegar a 1 codón de término y la traducción de la proteína va a llegar hasta ahí. Aquí tienen ustedes los distintos factores de inicio q forman parte de la etapa de inicio, van a haber 2 q les voy a pedir q recuerden con detalle q son el 2 y el 4, el factor de inicio 4 normalmente se denomina 4F está compuesto x 3 distintas subunidades la E, A y G, con la ayuda de todos estos factores se va a dar por completada la etapa de inicio. Para q se inicie la síntesis de la proteína tenemos q tener disociados los ribosomas, estos tienen una alta afinidad para asociarse por lo tanto debemos promover una disociación entre ellos, para eso participan 2 factores de inicio el 1 y el 3, éstos ayudan a mantener disociada la subunidad mayor de la menor y de esa manera estos factores capturan a uno de los componentes de la síntesis de proteínas a las subunidad de 40S, otro de los componentes de la síntesis de proteína va a corresponder al tRNA de inicio, para eso va a participar otro factor de inicio que va a corresponder al factor de inicio 2 y éste debe estar asociado a GTP, éste captura al tRNA de inicio q codifica para metionina y se forma este complejo tRNA de metionina con el factor de inicio 2 con el GTP, ahora éstos van a unirse a la subundad pequeña del ribosoma que podía estar disociada de la unidad mayor xq se habían unido a el el factor de inicio 1 y el 3, y de esta manera se forma este complejo de preinicio que esta dado por la subunidad pequeña por el tRNA de metionina y por los factores de inicio 1,3 y 2, éste último está asociado a GTP xq posteriormente la hidrólisis de éste va a aportar la energía necesaria para que se forme y se complete la primera etapa de inicio de la traducción de proteínas. Primero el factor de inicio 1 y 3 son los q se unen al 40S para evitar q se asocie a la subunidad mayor, ahora el factor de inicio 2 trae al tRNA de inicio para pegarlo ahí, a la subunidad pequeña. Entonces, tenemos la subunidad pequeña, el tRNA de inicio, lo q necesitamos ir agregando para tener toda la maquinaria para la síntesis de proteínas es tener el mensajero q necesitamos leer y después vamos a necesitar la otra subunidad grande para q se forme todo el ribosoma y ahí vamos a tener al mensajero, al tRNA de inicio y solo nos va a faltar traer los otros tRNA q corresponden a los codones q siguen al marco de lectura del mensajero correspondiente. Resumiendo, en esta etapa tenemos q el GTP se une al factor de inicio 2, en estas condiciones el factor 2 puede capturar al tRNA de inicio y ahora éste se une a la subunidad 40S q está asociada a los factores 1 y 3. Ahora tenemos la otra etapa q es la de traer a este complejo de inicio al mensajero, para traer a este último va a ser importante la participación de factor de inicio 4, este es una proteína q está compuesta por 3 subunidades, q son 4E, 4A y 4G, normalmente lo que ocurre es que la 4E y la 4A están unidas a la 4G, en algunos textos a todo este complejo se le denomina factor de inicio 4R, es una proteína con 3 monómeros, lo q va a ocurrir es q el mensajero en los eucariontes tiene una modificación en el extremo 5’ y después una modificación del factor ……………. Metil guanosina, para proteger el extremo 5’, en el extremo 3’ tiene un poli A. Ahora además de protegerlo de acciones exonucleasas, este K nos da una señal de reconocimiento del mensajero para adherirlo a la subunidad pequeña de 40S, entonces ahora el factor de inicio 4 se va a pegar acá, el reconocimiento de esta subunidad está dado fundamentalmente por la subunidad E del factor de inicio 4 y la subunidad A va a permitir si aquí esta el CAP normalmente el primer AUG es el que se va a corresponder con el codón de inicio, entonces para poder llegar aquí y ubicarlo en esta zona del ribosoma pequeño lo va a hacer la subunidad A q tiene una actividad helicasa, que le va a permitir ir recorriendo el mensajero, entonces tiene varias subunidades y de éstas la E es la q se une a CAP, la 4A es la q le permite tener actividad helicasa y utilizando ATP va a poder recorrer ese mensajero y ahí va a quedar el mensajero en la subunidad 40S, la 4G es una subunidad de soporte (anclaje) a la cual se une y mantiene el complejo del trímero y tb interactúa con las proteínas y el RNA del ribosoma y poder ubicarlo donde corresponde. Entonces al principio teníamos disociada la subunidad pequeña y para eso contribuían el factor 1 y 3, cuando está disociado se le agrega el tRNA de inicio q llega facilitado x el factor 2 q tiene el GTP, x otro lado vemos q tenemos el mensajero y este es capturado x el factor de inicio 4 y este se une a la zona CAP y esta zona es reconocida fundamentalmente x la subunidad E y la A de el factor 4 es la q va a permitir recorrer el mensajero, para ubicar el tRNA de inicio con el correspondiente AUG, aquí hay un factor de inicio 4d y éste es una proteína q activa a la helicasa, es decir, activa al 4A para q el mensajero se pueda ubicar en el lugar correspondiente, si ustedes se dan cuenta nosotros tenemos q mover o este ribosoma tiene q recorrer esta zona xa poder acoplar el tRNA al AUG, esto se ha logrado con el conjunto de los factores de inicio 1,3,2 y 4, ahora lo q vamos a necesitar es q alguien traiga a la subunidad más grande y el q la trae es otro factor de inicio, en este caso va a actuar el factor de inicio 5 q va a traer a la subunidad mayor del ribosoma para permitir q se forme el complejo 80S q va a ser cuando está armada toda la fábrica y ya sabemos desde q punto tenemos q empezar a traducir la proteína, todo eso se logra con la energía aportada x la hidrólisis del GTP. Como les había dicho el factor 4 había logrado ubicar la subunidad E, acá en la zona q correspondía al RNA y después la subunidad A q tiene actividad helicasa con el grupo de ATP permitió q este se moviera y pudiera ubicarse el tRNA de inicio en el codón de inicio, una vez q se establece esto salen como va a venir el factor de inicio 5 con la subunidad mayor para formar el complejo del ribosoma se necesita sacar el factor de inicio 1 y 3 q mantienen disociadas a las 2 subunidades, salen esos, se hidroliza el GTP y el factor de inicio 5 trae a la subunidad mayor y ahí se forma el complejo de inicio y ahora estamos en condiciones de alongar la cadena peptídico. Hay 2 puntos de regulación q son importantes en la etapa de inicio de la síntesis de proteínas que es los factores de inicio 2 y 4, para q ocurra la primera etapa de inicio, es indispensable q el factor 2 aporte energíaen forma de GTP, una vez q participa en esa etapa y sale, sale como GDP, por lo tanto para poder estar en condiciones de volver a iniciar la otra síntesis de proteínas, vamos a tener q volver a capturar otra subunidad pequeña y capturar nuevamente el tRNA de inicio, para poder hacer eso el factor de inicio 2 tiene q estar asociado a GTP, y por lo tanto como estaba como GDP tenemos q intercambiarlo x GTP y esto lo hace otra proteína q se denomina GEF o como la subunidad 2B de este factor 2, esta proteína simplemente intercambia GDP x GTP, de esta manera cada vez q vea un factor de inicio 2 con GDP lo intercambia x GTP y queda nuevamente activado el factor de inicio 2 y esta en condiciones de ir nuevamente a capturar otro tRNA de inicio, si x algún motivo uno modificara esta actividad o la muta y ésta no puede intercambiar GTP, nos quedaría permanentemente inactivo el factor de inicio 2 y por lo tanto ese individuo no podría hacer síntesis de proteínas. El factor de inicio 4 está formado x este complejo, es un trímero, la subunidad G es la q agarra a la E y a la A, la E es la q permite reconocer la estructura CAP del mensajero para ubicarla en la subunidad pequeña del ribosoma y la subunidad A es la q va a permitir ubicar ese mensajero en el AUG de partida, ahora, para poder hacer q esta estructura se una acá tenemos q hay una proteína q se une al 4E q señala cuando es BP (x binding protein), es una proteína q se une a la E, si esa proteína 4E, esta solita se puede asociar a la proteína G y tb a la subunidad A y formar el trímero activo. Si la 4E está unida a la proteína BP, ésta no puede formar el trímero por lo tanto estaría inactivo, para poder formar este complejo hay q separarla de esa proteína de unión, de su binding protein y ésta se saca fosforilándola, por lo tanto cuando uno activa la traducción de proteínas se tiene q fosforilar esa binding protein para q libere al 4E, cuando ocurre esto ésta se puede unir y formar este complejo, ahora si además esta 4 E es fosforilada con esa modificación covalente tiene la disposición tridimensional q favorece la afinidad x la zona CAP, por lo tanto a nivel del factor 2 necesitamos q éste esté asociado a GTP, si está asociado a GDP tenemos q intercambiarlo x GTP, a nivel del factor 4 la subunidad E debe estar sola formando el trímero para poder reconocer CAP y posteriormente ubicar el AUG. Para q se separen el F4 de la 4BP, esta última debe ser fosforilada, pero tb puede fosforilar a las 2. De manera tal q muchas hormonas o factores de crecimiento q estimulan la proliferación, si se va a estimular la célula se va a dividir y para esto debe multiplicarse su material proteíco, x lo tanto deben estimular la síntesis de proteínas y de esa manera se puede usar el factor de crecimiento derivado de las plaquetas, etc y todo esto induce la activación de kinasas q fosforilan a la proteína de unión de 4E, al fosforilar a la binding protein pueden liberar a la 4E y eso permite formar el complejo activo del factor de inicio 4. Entonces para acoplar la subunidad pequeña y los factores de inicio?? Ocurrieron factores de inicio el 1 y el 3, luego el factor 2, el mensajero q es traído por el factor 4 finalmente es traída la subunidad mayor x el factor 5, ahora lo q se necesita es traer los respectivos tRNA cargados con el aá correspondiente de acuerdo al codón q lee el mensajero. En esta etapa de elongación van a participar pocas proteínas q son factores de regulación y éstas ahora lo q van a hacer es capaturar a los tRNA cargados con los aá y traerlos al ribosoma, van a favorecer el anclaje de ese tRNA al ribosoma y van a favorecer q ocurra la reacción q cataliza el enlace peptídico, eso tb va a ser con la colaboración de GTP, esto factores de elongación tb usan GTP, una vez q se forma el enlace peptídico necesitamos q el ribosoma se mueva para volver a dejar el sitio donde llegan los tRNA cargados, eso tb sucede con la hidrólisis del GTP (esa traslocación). Aquí vemos q juegan un rol importante 2 factores de elongación el 1 y el 2, en el caso del 1 ocurre algo muy análogo a lo q ocurre con el factor de inicio 2, si ustedes se dan cuenta éste permite q se una el aminoacil q esta cargado con 1 aá y éste tiene asociado GTP la catálisis de enlace peptídico va a ser catalizado x la hidrólisis del GTP, para q esto pueda ocurrir nuevamente el factor de inicio 1 después de q ocurra esa etapa va a quedar unido a GDP y necesitamos cambiar este GDP x GTP y ahí tenemos la otra subunidad del factor de elongación 1 q es la q es βγ para q vuelva a restablecer el GTP . Después se necesita mover al ribosoma para q siga avanzando x la cadena del mensajero y esa traslocación tb va a ser dependiente del GTP, todo lo q corresponde a la síntesis de la proteína misma es catalizada x la energía q aporta la hidrólisis del GTP. Por un lado si ustedes se dan cuenta en esta etapa una va vez q ubicamos el codón de inicio (tRNA de inicio) estamos listos para traer los tRNA cargados con aá, aquí en la etapa de elongación el factor de inicio 1 trae al tRNA cargado con su correspondiente aá, éste se ubica en este sitio y aquí se cataliza la formación del enlace peptídico, para poder tener los respectivos tRNA con sus correspondientes aá cargados en una etapa anterior, en otro lado esta la aminoacil tRNA sintetasa qdice tengo este tRNA vacío q es para alanina agarro la alanina y lo pego y los voy tirando para allá, después viene otra aminoacil tRNA sintetasa q es de ác. Glutámico y agarra el tRNA del glutámico y el aá de este y los uno y después lo tira, entonces todos estos tRNA cargados están ahora disponibles para q venga 1 factor de elongación un asociado a GTP para q los tome y los lleve hacia los ribosomas, cuál va a ser el q se una aquí? El q tenga complementariedad de bases con el codón q está en el mensajero. La hidrólisis del GTP q estaba asociado al factor de inicio 1 aporta la energía para catalizar la formación del enlace peptídico y una vez q se produce este enlace entre la cadena antigua y el nuevo aá q entró, ahora tenemos q correr esto para acá y de esa manera vamos a dejar libre al sitio A, este movimiento ocurrió mediante la hidrólisis asociada al factor de elongación 2 q esta asociado a GTP, y ahora su misión es volver a recibir otro tRNA q va a venir asociado al factor de elongación 1. Ahora, siguiendo esta lectura vamos a llegar a un momento en q el codón q sigue es un codón de término y para este no hay ningún tRNA por lo tanto lo q se une acá es una proteína q se denomina factor de término o RF x releaising factor, como se une ahí la enzima q hace la catálisis no forma un enlace peptídico, sino q une este a una molécula de agua, crea un enlace con la molécula de agua y con eso se suelta la molécula de proteína q estaba naciendo, como se suelta la proteína aparecen el factor 1 y el 3 para disociar todo este ribosoma, tener la subunidad pequeña y volver a iniciar todo el proceso. Resumiendo la elongación requiere de proteínas q son factores de elongación, cada tRNA activado con sucorrespondiente aá, o sea un aminoacil tRNA es conducido al ribosoma x un factor de elongación 1, el 1α q está unido a GTP, el GTP va a aportar la energía necesaria para esta etapa, la regeneración del GTP asociado al factor 1α se lo otorga la subunidad βγ del factor 1, el péptido q estaba en el sitio P se va a mover al sitio A catalizado x la peptidil transferasa proceso conocido como transpeptidación, es decir, cuando se forma el enlace peptídico y posteriormente el movimiento de la cadena polipeptídica de la cadena q esta naciendo q está en el sitio A para volver a quedar en el sitio P se conoce como traslocación en esta etapa colabora el factor de elongación 2 q venía con GTP con su correspondiente hidrólisis, de esa manera vuelve a quedar abierto el sitio A, disponible para q venga otro tRNA cargado con su correspondiente aá q pueda ser reconocido x el codón correspondiente del mensajero. Tenemos 3 codones a los cuales se pueden unir estas proteínas de término, nuevamente la unión de estos factores de término va asociada a GTP, o sea si no hay GTP no hay posibilidad de sintetizar proteínas y para poder tener GTP necesitamos tener ribosa y ésta la tenemos del ciclo de las pentosas, o sea sale de la glucosa por lo tanto es importante tener glucosa, xq si no tenemos esto no hay síntesis de proteínas , la peptidil transferasa es la q forma el enlace peptídico, se activa y ahora transfiere el polipéptido al agua y con eso lo libera desde el ribosoma ,el tRNA al q estaba asociada la cadena polipéptidica como salió después de unirse al agua queda solo y por lo tanto en ese estado descargado sale del sitio P GTP se hidroliza usando la energía necesaria, se libera el RNA del ribosoma y aparecen los factores 1 y 3 para disociar el ribosoma en las subunidades 40S y 60S y de esa manera después queda el 40S asociado al factor 1 y al 3, al cual ahora se le van a acoplar el factor 2 q está unido a GTP y q ya agarró al tRNA de inicio y se empieza a formar otra vez el complejo de inicio con la colaboración del factor de inicio 4 del RNA, después va a venir la subunidad mayor con el factor de inicio 5 y así se van sintetizando las proteínas. Aquí ustedes ven una tabla con típicos antibióticos q actúan a nivel de la síntesis de proteínas, algunos de ellos son específicos para procariontes, otros tb actúan en eucariontes, el cloranfenicol liquida a las bacterias xq inhibe la peptidil transferasa, o sea impide q se forme en enlace peptídico, la estreptomicina inhibe la iniciación, la tetraciclina inhibe la unión del aminoacil tRNA a la subunidad pequeña del ribosoma, la puromicina se parece a un aminoacil tRNA con lo cual interfiere la transferencia del péptido, con lo cual se termina antes, si uds ven aquí tienen la estructura de la puromicina y aquí tienen la estructura del tRNA para la tirosina, y se parecen bastante, entonces en vez del tRNA se mete el antibiótico y hasta ahí no más llegó la síntesis de la proteína. En el caso de la toxina histérica q en el tiempo atrás causó tantas muertes si uds se dan cuenta, esta toxina cataliza una modificación covalente del factor de elongación 2 lo ADP-ribosila y al hacer esto lo inactiva e impide q este factor de elongación se una a GTP y pueda ir a colaborar con la elongación y hasta ahí quedó la síntesis de proteínas. La puromicina se parece mucho al tRNA con la tirosina. ¿Quiénes están asociados a GTP? El factor de inicio 2, el factor de elongación 1 y el 2 y tb el … para unirse al RF ¿Dónde solo se ocupa ATP en la síntesis de proteínas? Cuando la actividad helicasa del factor de inicio 4 requiere mover el mensajero para ubicar el sitio de inicio, esa es la única etapa q usa ATP, en todas las otras necesitamos GTP. En la etapa de activación se requiere ATP para activar a los aá.