ESTUDIO ESTRUCTURAL PROTEÍNAS
Anuncio

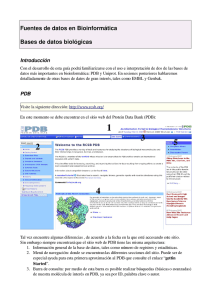
TALLER DE VISUALIZACIÓN MOLECULAR. ESTUDIO ESTRUCTURAL DE MACROMOLÉCULAS Curso 2006/07 Coordinadora del Proyecto: Rosa M León Area de Conocimiento: Bioquímica y Biología Molecular Dpto. Química y Ciecia de Materiales Proyecto de Innovación Docente Area de Bioquímica y Biología Molecular Introducción Se han secuenciado genomas de cientos de organismos incluyendo virus, bacterias, hongos, plantas y animales. El siguiente paso es estudiar la función de tales secuencias. Para ello se están utilizando herramientas informáticas que localizan las secuencias que codifican para proteínas e intentan comprender cómo y cuáles son sus estructuras y funciones más probables, esto ha dada lugar a la aparición de una nueva disciplina “La bioinformática” que abarca el manejo de estas bases de datos y herramientas. Además de la información sobre la estructura primaria hay bases de datos que incluyen la estructura tridimensional de muchas macromoléculas, principalmente proteínas, que han sido determinadas por difracción de Rayos X. La visualización de estas estructuras con programas de visualización molecular, que permiten al usuario rotar las moléculas para verlas desde todas las perspectivas posibles, cambiarlas en tamaño, ver detalles de unos pocos átomos, aporta gran cantidad de datos y facilita la compresión de la estructura y función de estas macromoléculas. El objetivo de esta práctica es aprender las nociones básicas para realizar el análisis estructural de un proteína basándonos en su secuencia. 1. Caracterización fisicoquímica de la proteína conociendo su secuencia Partiendo de la secuencia de una proteína dada y utilizando el servidor de proteómica: http://www.expasy.org, se pretende obtener información fisicoquímica, funcional y estructural de dicha proteína. 2 Proyecto de Innovación Docente Area de Bioquímica y Biología Molecular Para ello utilizaremos el programa ProtParam (/ExPASy Proteomics tools/Primary Structure Analysis): caracteriza físico-químicamente una proteína, dándonos información sobre el número de aa que la componen, su masa molécular, composición de aa, punto isoeléctrico. 2. Comparación de la secuencia dada con las secuencias conocidas utilizando el progrma BLASTtp del NCBI. Al que podemos acceder desde (/ExPASy Proteomics tools/Similaritary Searches) o (http://www.ncbi.nlm.nih.gov/). Dicho programa reconoce posibles dominios conservados que proporcionan información acerca de su posible función. Seleccionar Protein-Protein BLAST (blastp). Pegar la secuencia al recuadro de search y enviar la búsqueda mediante el comando BLAST. Los resultados pueden tardar algunos minutos, se accede a ellos a través del comando FORMAT 3 Proyecto de Innovación Docente Area de Bioquímica y Biología Molecular 3. Localización de las coordenadas PDB de la proteína problema. Las coordenadas XYZ de los átomos de una macromolécula son la información necesaria para representar su estructura. Las coordenadas de las macromoléculas cuya información se ha resuelto por RX, RMN o microscopía electrónica se encuentran depositadas en Protein Data Bank (PDB) (http://www.rcsb.org). Aunque es posible concectarse a este sitio a través de diferentes organismos como el NCBI (National Centre for Biotechnological information-http://www.ncbi.nlm.nih.gov) o el EBI (European Bioinformatics Institute-http://www.ebi.ac.uk). Se puede reclamar el fichero PDB de una proteína usando el código pdb o en nombre de la proteína y la especie a la que pertenece. Si entramos en el PDB podemos. Download files. Web downloads. Podremos descargar pdb file uncompressed. Esto requiere instalar el Java Runtime Environment versión 4.0 o superior. 4. Programas de visualización y manejo de estructuras Hay disponibles un buen número de programas de visualización molecular de libre distribución que permiten la lectura de ficheros de coordenadas en distintos formatos para visualizar estructuras de moléculas. Entre dichos formatos destacan el de la base de datos de proteínas de Brookhaven (Protein Data Bank) adoptado por la IUPAC, y el formato MOL de Tripos Inc. (USA), uno de los principales proveedores de software de modelado molecular. 4 Proyecto de Innovación Docente Area de Bioquímica y Biología Molecular El ViewerLite Weblab. Permite representar la estructura tridimensional de las moléculas utilizando sus coordenadas en formato pdb. Una vez cargada la molécula puede verse de varias manera (varillas, bolas, etc...), puede girarse, pueden verse u ocultarse átomos, puentes de H, etc.. El Swiss-Pdb Viewer. Permite también represetar la estructura tridimensional de las moleculas concociendo sus coordenadas pdb. Además permite superponer varias proteínas, ver intracciones entre ellas o entre proteínas y ligandos, manipularla (hacer manipulaciones puntuales), medir distancias y ángulos entre átomos. Con estos programas podemos hacer un estudio estructural básico de proteínas - Abrir el fichero PDB con Weblab - Visualizar la proteína con sus estructuras secundarias - Describir tipos de estructura secundaria - Número de hélices alfa y/o hebras beta - Estructura supersecundaria y dominios estructurales - Tipo de grupo prostético: aminoácios implicados en su unión - Distribución de aa hidrofóbicos e hidrofílicos - Otras características estructurales destacables Por ejemplo si queremos ver: El citocromo c de caballol, cuyas coordenadas pdb son1hrc.pdb, las introducimos en el buscador del pdb y obtendremos un fichero, que podremos abrir con el programa viewer little: - Primero Seleccionamos el submenú File seguido de Open. Nos aparecerá una nueva ventana donde seleccionaremos el fichero de coordenadas pdb que nos interese abrir. La estructura de la molécula se visualiza en otra ventana. - La estructura de la molécula se visualiza en otra ventana. 5 Proyecto de Innovación Docente Area de Bioquímica y Biología Molecular Cadena de aa Grupos prostéticos u otros grupos H2O - Aparece un menú principal (Toolbar) en el que, a la izquierda, encontramos las herramientas de translación, aproximación, torsión y rotación de la molécula.; tal como se muestra en la imagen siguiente. Se puede acceder a la secuencia aminoacídica o nucleótica de la macromolécula seleccionando en el menú principal el submenú Window seguido de New Hierarchy Window. Seleccionamos como estilo de la proteína cintas solidas Ocultamos agua, esqueleto y cadenas laterales 6 Proyecto de Innovación Docente Area de Bioquímica y Biología Molecular Si cambio el color del fondo y selecciono todo los elementos y puedo pegar la figura en cualquier programa. El programa me permite además ver el grupo prostético, en este caso el grupo hemo, los aminoácidos con los que interacciona directamente dicho grupo prostético etc, - Con backbone y lateral chains activadas puedo visualizar que aa interaccionan con el grupo prostético, substrato u otros ligandos cristalizados junto a la proteína. En este caso His18 y Met 80. Si oculto todos los aminoácidos excepto estos dos y los marco con la herramienta label (hay que seleccionar el aa en la ventana descriptiva). 7 Proyecto de Innovación Docente Area de Bioquímica y Biología Molecular CUESTIONARIO Nombre y apellidos: 1- Caracterísiticas fisico-químicas de la proteína desconocida: 2- Identidad de la proteína. Nombre, especie, número de acceso 3- Presenta algún dominio funcional definido? 4- Cita otras proteínas con las que tenga alta homología según el NCBI 5- Dibuja o adjunta la imagen tridimensional de la proteína problema? 6- Presenta algún grupo prostético? 7- Indica número de hélices alfa u hojas beta que puedes distinguir en su estructura. 8